Method Article
ペプチド生合成酵素を調べた水素重水素交換質量分析(HDX-MS)プラットフォーム
要約
ランチペプチド合成酵素は、ペプチド天然物の生合成中に多段階反応を触媒する。ここでは、ランチペプチド合成酵素の立体ダイナミクス、ならびにペプチド天然物生合成に関与する他の同様の酵素を研究するために使用できる、連続的なボトムアップ、水素重水素交換質量分析(HDX-MS)ワークフローについて述べています。
要約
水素重水素交換質量分析(HDX-MS)は、酵素の立体構造変化と酵素基質相互作用の生物物理特性解析に対する強力な方法です。その多くの利点の中で、HDX-MSは少量の材料しか消費せず、酵素/基質標識を必要とせずに近い天然条件下で行うことができるので、大きな酵素および多タンパク質複合体に対しても酵素立体構造ダイナミクスに関する空間的に解決された情報を提供することができます。この方法は、D2Oで調製した緩衝液への目的の酵素の希釈によって開始される。これは、重水素(N-D)とのペプチド結合アミド(N-H)中のプロチウムの交換を引き起こす。所望の交換時点で、反応アリコートがクエンチされ、酵素がペプチドにタンパク質化され、ペプチドが超高性能液体クロマトグラフィー(UPLC)によって分離され、各ペプチドの質量の変化(重水素に対する水素交換による)がMSによって記録される。各ペプチドによる重水素の取り込みの量は、そのペプチドの局所水素結合環境に強く依存する。酵素交換重水素の非常に動的な領域に存在するペプチドは非常に迅速に存在するのに対し、整然とした領域に由来するペプチドは、はるかにゆっくりと交換を受ける。このようにして、HDXレートは局所的な酵素立体構造ダイナミクスについて報告する。異なるリガンドの存在下で重水素の取り込みレベルへの摂動は、リガンド結合部位をマッピングし、アロステリックネットワークを同定し、酵素機能における立体構造ダイナミクスの役割を理解するために使用することができる。ここでは、ランチペプチドと呼ばれるペプチド天然物の生合成をより深く理解するためにHDX-MSを使用した方法を説明します。ランチペプチドは、従来の構造生物学のアプローチでは研究が困難な大規模で多機能な構造動的酵素によって翻訳後に修飾される遺伝的にコードされたペプチドです。HDX-MSは、これらのタイプの酵素の機械特性を調査するための理想的で適応可能なプラットフォームを提供します。
概要
タンパク質は、フェムト秒スケールの結合振動から、何秒間にわたって起こり得るタンパク質ドメイン全体の再配置に至るまで、時間スケールで異なる立体構造をサンプリングする構造的に動的な分子である1。これらの立体構造変動は、しばしば酵素/タンパク質機能の重要な側面です。例えば、リガンド結合によって誘導される立体構造変化は、触媒に必要な活性部位残基を組織し、連続的な運動機構で基質結合部位を定義し、反応性中間体を環境から遮蔽するか、またはアロステリックネットワークを介して酵素機能を調節することによって、酵素機能を調節するために非常に重要である。最近の研究では、立体構造ダイナミクスは進化を通して保存することができ、保存された分子運動への摂動は基質特異性の変化および新しい酵素機能の出現と相関することができることを示している2,3。
近年、水素-重水素交換質量分析(HDX-MS)は、リガンド結合や変異形成などの摂動にタンパク質の立体構造の景観がどのように応答するかを調査する強力な技術として急速に出現している4、5、6、7。典型的なHDX-MS実験(図1)では、目的のタンパク質をD2Oで調製したバッファーに入れ、溶媒交換可能なプロトンを重水素に置換する。ペプチド結合のアミド部分の交換速度は、pH、局所アミノ酸配列、およびアミド8の局所構造環境に強く依存する。水素結合相互作用に関与しているアミド(αヘリックスやβシートに存在するものなど)は、バルク溶媒にさらされているタンパク質の非構造化領域のアミドよりもゆっくりと交換します。したがって、重水素の取り込みの程度は、酵素の構造の反映である。立体構造が動的な酵素、またはリガンド結合時に構造的な転移を起す酵素は、測定可能なHDX応答をもたらすと予想されるであろう。
構造化アミドの遅い為替レートの機械化基準を図25,8,9に示します。HDXを受けるためには、特定の酸/塩基化学機構を介してHDX交換を触媒する溶媒分子が交換可能なアミドにアクセスできるように、まず一時的に展開された立体構造をサンプリングする必要があります。最終的に、化学為替レート(kchem)と折り畳み速度(kオープンおよびkクローズ)の相対マグニチュードは、実験5,8で測定されたHDXレートを決定する。この単純な運動モデルから、重水素取り込みの程度が基礎となる立体ダイナミクス(kopenとk close)を反映することは明らかです。ほとんどのHDX-MS実験は、交換反応に続いて、目的のタンパク質がペプチドに消化され、各ペプチドによる重水素取り込みが質量7の増加として測定されるボトムアップワークフローで行われる。このように、HDX-MSは、酵素立体構造ダイナミクスへの摂動をペプチドの局所空間スケールにマッピングすることを可能にし、摂動が目的の酵素の異なる領域のダイナミクスをどのように変化させるかを評価することを可能にする。
タンパク質構造力学を解明するためのHDX-MSアプローチの利点は数多くあります。まず、この方法は、第四級構造10の系において少量の天然タンパク質またはタンパク質複合体に対して実施することができる。ボトムアップHDX-MSワークフローが目的のタンパク質配列をカバーする十分な数の確実に同定されたペプチドを提供する限り、アッセイに使用される酵素調製物が高度に精製される11,12である必要さえありません。さらに、HDX-MSは、単一分子蛍光研究13で用いられるような部位特異的タンパク質標識を必要とせずに、近い在来条件下での立体構造ダイナミクスに関する情報を提供することができ、また、調査できるタンパク質またはタンパク質複合体(核磁気共鳴[NMR]分光法などのアプローチを困難にする)にサイズ制限はない。最後に、時間分解HDX-MS法は、X線結晶学15、16、17、18で研究することが困難である本質的に障害を持つタンパク質を研究するために使用することができる。HDX-MSの主な制限は、データが低い構造分解能であるということです。HDX-MSデータは、立体ダイナミクスが変化している場所を指し示し、結合された立体構造の変化を明らかにするのに役立ちますが、観察された変化を駆動する正確な分子メカニズムに関する多くの洞察を提供することはあまりありません。電子捕獲解離法とタンパク質HDX-MSデータの組み合わせにおける最近の進歩は、交換部位を単一アミノ酸残基19にマッピングする約束を示しているが、HDX-MSデータによって転送される構造モデルを明確にするために、生化学的および構造的研究をフォローアップすることが依然としてしばしば必要である。
以下、HDX-MSアッセイの開発のための詳細なプロトコルが20を提示される。以下に示すサンプル調製プロトコルは、一般的に、水性バッファーに良好な溶解性を示す任意のタンパク質に適用できる必要があります。より特殊なサンプル調製方法およびHDX-MSワークフローは、洗浄剤またはリン脂質21、22、23、24の存在下でのアッセイが必要以上にタンパク質に対して利用可能である。HDX-MS データ収集のインストゥルメンタル設定は、液体クロマトグラフィー システムに結合された高分解能の四重極時間質量分析計について説明されています。同様の複雑性と分解能のデータは、市販の液体クロマトグラフィー質量分析(LC-MS)システムの中から収集できます。市販のソフトウェアパッケージを用いたデータ処理の重要な側面も提供される。また、より広範な HDX-MS コミュニティ12の推奨事項と一致するデータ収集と分析のガイドラインも示します。本記載のプロトコルは、HalM2の動的構造特性を研究するために用いられる、抗菌ペプチド天然物20の多段階成熟を触媒するランチペプチド合成酵素である。我々は、HDX-MSを用いて、これまでの特徴付けが排除された基質結合部位およびアロステリック特性を明らかにする方法を示す。タンパク質HDX-MSに関する他のいくつかのプロトコルが近年25,26で公開されている。現在の作業と共に、これらの以前の貢献は、読者に実験計画のいくつかの柔軟性を提供する必要があります。
プロトコル
1. 重質試薬および酵素ストック溶液の調製
- D2 O(99.9%原子画分D)で100-200x濃縮ストック溶液としてHDX反応(バッファー、塩、基質、リガンドなどを含む)に必要な試薬を調製します。バッファーストック溶液の少なくとも 50 mL を準備します。
注意:HalM2の特性評価のために、次のソリューションを用意しました:500 mMMgCl 2、100 mMトリス(2-カルボクセチル)ホスフィン(TCEP)、750 mM ATP(HEPESバッファ)、800 mM HEPES pD 7.1、500 μM HalA2、および500mNPM AMPP. - 在庫液を凍結し、乾燥に乾燥させます。
- D2Oに再溶解し、再乾燥サイクルを少なくとも1回繰り返して、交換可能なプロトンの多くをできるだけ多くのデュテロンに置き換える。
- 重水素化HEPESバッファストックのpDを、集中NaOD/DClで所望の値に調整し、次の関係を念頭に置いて27:
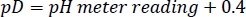
注:アミドHDXレートは、溶液のpL(pL = pHまたはpD)5に強く依存しています。実験間のわずかなpLドリフトを避けるために、バッファーストックソリューションの異なるバッチを同じ方法で準備、保存、使用する必要があります。 - 300 μL HDX アッセイに必要な各試薬の量を計算し、-80 °Cで単一使用アリコートとして保存します。
- 遠心フィルター(表)または同等のデバイスを使用して、プロティア化酵素貯蔵バッファーに濃縮酵素ストック溶液(約100-200 μM)を調製します。
注:正確な緩衝液と遠心フィルターの分子量カットオフは、目的のタンパク質/酵素に依存します。HalM2は、50 mM HEPES、pH 7.5、100 mM KCl、および10%グリセロールに保存されています。10 kDaフィルターを用いて濃縮酵素を調製した。 - 酵素を単一使用部分にアリコートし、-80°Cで保存する。
注: これは停止点になる場合があります。セクション1に記載されているすべてのストックソリューションは、HDX反応の前に準備することができます。-80 °Cで保存すると、ほとんどの酵素/重水素ストック溶液は、何ヶ月もの間安定しています。
2. HDXクエンチボリュームのキャリブレーション
- 第1項で調製した重水素化試薬および濃縮酵素ストックを用いて、D2Oで300μLのHDX反応を調製します。
- 1-5 μMの最終酵素濃度を使用してください。
- 50-100 mM以上の最終的な重水素化HEPESバッファ濃度を使用してください。
- 他の成分の濃度が、所望の酵素活性/機能を維持するのに十分であることを確認してください。
- 1 L の HDX クエンチ溶液 (100 mM リン酸、0.8 M グアニジン-HCl、pH 1.9) を準備します。凍結し、50 mLの部分(長期在庫用)と1 mLの部分(単一の使用アリコートの場合)の両方で保存します。
注:クエンチバッファーの正確な組成は、ボトムアップHDX-MSワークフロー(ステップ3.3.3)のタンパク質分解工程で使用される酵素によって異なります。ここで与えられたクエンチバッファーは、HDX-MSの最も一般的に使用されるプロテアーゼであるペプシンと互換性があります。別のプロテアーゼを使用する場合は、プロテアーゼサプライヤーに確認して、バッファーの相溶性を確認してください。 - クエンチHDX反応混合物の最終的なpLを2.3のpHメーター読み取り値に調整するために必要なクエンチバッファの量を調整します。
注:ペプチドアミドN-H結合の溶媒H/D交換速度は、酸触媒と塩基触媒の両方に影響されるpH依存性プロセスです。最小為替レートは、pH 2.5(pHメーターの読み取り値 = 50:50 H 2O:D2 O混合物の場合は2.3)の値で発生します。したがって、2.5付近の最終的なpL値は、ボトムアップLC-MS分析中に生じる水素バック交換を最小限に抑え、それによってペプチド内の重水素標識を保存する。- ステップ2.1のHDX反応混合物の50 μLと50 μLのクエンチバッファーを混合し、マイクロチップ電極でクエンチ混合液のpLを測定します。
- 必要に応じてクエンチ溶液の体積を増やし、最終的なpHメーターの読み取り値を2.3に調整します。
- 適切なクエンチ量が決定されたら、HDX反応(ステップ2.1)の新鮮な50 μLアリコートを使用して数回クエンチプロセスを繰り返し、固定量のクエンチバッファを追加した際に一貫した最終pLが達成されるようにします。
3. ボトムアップLC-MSワークフローの参考サンプルの作成と最適化
- 0.5 mLチューブ内の三重化に関心のあるタンパク質のための未解製の参考サンプルを準備します。最終反応混合物条件が本物のHDX反応(ステップ2.1)で使用されるものと同一であることを確認し、反応は、H2Oでも調製した試薬ストック溶液を用いてH2Oで調製されることを除いて。
- ステップ 2.3 のように、適切な量のクエンチバッファーを追加して、最終的な pH を 2.5 に調整して、サンプルをクエンチします。フラッシュは液体窒素でサンプルを凍結し、分析の準備ができるまで-80 °Cで保管します。
- ボトムアップ LC-MS ワークフローを使用して、プロティアード酵素参照サンプルを分析します。
注: これらの手順を実行する前に、データ収集に使用する LC-MS システムは適切に調整され、使用できる状態にしておく必要があります。ボトムアップ LC-MS ワークフローのすべてのステップのタイミングと温度は、サンプル間のバック交換の違いを最小限に抑えるために厳密に制御する必要があります。このプロトコルで使用される MS インストルメンテーション (資料表 そして サポート情報)、ほとんどのステップは計測器ソフトウェアを通して制御できます。正確な複製の収集を確実に行うために、ワークフローの手順をできるだけ多く自動化することをお勧めします。- 冷凍庫から個々の酵素参照サンプル(ステップ3.2のように調製)を取り出し、水浴中で1分間37°Cで解凍します。
- 冷凍庫からサンプルを取り出して解凍した後、正確に2分で、クエンチされた参照サンプルの40 μL部分を、ペプシン(酸安定プロテアーゼ)で機能する定常相を含む超高性能液体クロマトグラフィー(UPLC)カラム(2.1 x 30 mm、300 Å、5 μM)に注入します。
- H2O(pH = 2.5)の0.1%のギ酸を溶媒として使用して、15°Cで3分間100μL/minの流量でサンプルを消化します。
- ペプシンカラムから0.4°Cに保持されたC18トラップカラムに溶出して、ペプチドペプチドを回収し、逆入れを最小限に抑えます。
- トラップカラムから脱塩されたペプチドをC18分析カラム(1mm x 100 mm、1.7 μM、130 Å)に渡し、0.4 °Cで操作して消化ペプチドを分離します。
注: ステップ 3.3.3-3.3.5 は、HDX-MS データ収集に使用される特定の LC-MS システムで自動化できます。あるいは、これらのステップは、一貫して低いバック交換を達成するために各ステップのタイミングと温度を慎重に制御する必要があることを念頭に置いて、独立して実行することができます。 - C18カラムをアセトニトリル/水/0.1%のギ酸溶媒系でエルプします。ペプチドの分離を最大化し、ペプチドペプチドの重水素標識を維持するために、目的のタンパク質の LC 勾配を最適化します。
注: グラデーションの溶出の詳細については、 サポート情報を参照してください。 - ペプチド消化をエレクトロスプレーイオン化(ESI)質量分析に供する。
注: サポート情報 に記載されているソース条件は、ほとんどのペプチドに対して十分なイオン化を提供します。- MS機器でイオン化したら、窒素を緩衝ガスとして用いてガス相イオン移動度分離を行い、方法のピーク容量を増強する。
- イオン移動度分離の後、低衝突エネルギー(4 V)と高衝突エネルギー(21-40V)の交互サイクルを含むMSE ワークフローにペプチド前駆体イオンを適用します。
注: 交互に低衝突エネルギーレジームと高衝突エネルギーレジームを使用すると、MSMS データ(高衝突エネルギー)と同時に MS データ(低衝突エネルギー)を収集できます。これにより、前駆体イオンとそれぞれのフラグメントイオンの時間相関が可能になります。この相関は、セクション4に記載されている自信のあるペプチド同定に不可欠です。 - 少なくとも20,000の解決力を持つ質量分析計を使用して、ペプチド前駆体およびフラグメントイオンを検出します。
- 同時にデータ取得と同時に、[Glu-1]-フィブリノペプチドB(GluFib)外部標準のMSデータを取得する。
注: ステップ 3.3.7 で説明されている MS ワークフローは MSE プロトコルと呼ばれます。ボトムアップ HDX-MS に適した MSE プロトコルのインストゥルメンタル設定は 、サポート情報に記載されています。
- LC-MS データの品質を評価します。
注:上記のプロトコルとサポート情報で提供されるインストゥルメンタル設定を使用して、リファレンスサンプルは、約1 x 108の最大信号強度を持つ総イオンクロマトグラムを生成する必要があります。3-9 minの間に多くのペプチドが溶け出しているはずです(図3A-C)。 - 40 μL のブランクサンプル(水中の0.1%のギ酸)を注入して、ペプシンおよび分析用C18カラムを洗浄します。
注: 一般的に、2~ 3 個のブランクで十分です。 - 参照タンパク質サンプルの各三重化について、ステップ 3.3.1~3.3.9 を繰り返します。
4. 参照データの処理とペプチドリストの定義
- プロテオミクスソフトウェア(材料表)を使用して生のMSEデータ(ステップ3.3.7)を分析します。プロテオミクスソフトウェアを使用して、ライブラリ|タンパク質配列データバンクは、目的のタンパク質のアミノ酸配列をインポートしてタンパク質データベースを定義する。
注: このステップの目的は、目的のタンパク質から得られたペプチドの参照 MS データを検索し、MSMS データ (MS データと同時に取得) を使用して、推定ペプチドの識別を検証することです。 - 目的のタンパク質配列に名前を付けます。タンパク質配列をインポートします (FASTA 形式)。このソフトウェアは、データベースタンパク質のインシリコ消化を行い、LC-MSデータの検索に使用されるペプチドのリストを生成します。
- 処理パラメータを定義します([ライブラリ]メニューにあります)。データ取得タイプとしてエレクトロスプレー MSEを選択します。[電荷 2 の質量をロック] フィールドで、[Glu-1] -フィブリノペプチド B (GluFib) の 2+イオンの m/z に785.8426と入力し、[終了] をクリックします。
- ワークフローパラメータを定義します(ライブラリメニューの下にあります)。
- 検索タイプ としてエレクトロスプレー MSE を選択します。 ワークフロー | データベース検索クエリ の見出しで、ステップ 4.2 で作成したデータベースタンパク質を [データバンク ]フィールドで選択します。
- プライマリ消化試薬を非特異的に変更し、Ctrlボタンを押したままカルバミドメチルCをクリックして[固定改変試薬]フィールドをクリアします。
- オプションに移動して出力ディレクトリを指定|オートメーションセットアップ|アイデンティティ E.Apex 3D およびペプチド 3D 出力とイオンアカウンティング出力のチェックボックスをオンにして、目的のディレクトリを指定します。
- 参照サンプル データを処理します。
- プロテオミクスプラットフォームワークスペースの左側のツールバーで、 マイクロティタープレートを右クリックして新しいプレートを作成します。マイクロティタープレートの3つのウェル(セクション3で収集された各参照サンプルに1つずつ)をハイライトします。1つの井戸で左クリックし、3つの井戸に保持し、ドラッグします。
- 右クリックして[ 生データを追加]を選択します。表示されるウィンドウで、セクション 3 の 3 つの参照ファイルを含むディレクトリに移動し、それらを同時に選択します。
- Nextをクリックし、ステップ 4.3 で定義した処理パラメータを選択します。Nextをクリックし、ステップ 4.4 で定義したワークフロー パラメータを選択します。次に[完了]をクリックします。
- 未加工データ、処理パラメータ、およびワークフローパラメータがプレート上の各ウェルに割り当てられると、井戸は青色で表示されます。ウェルを選択し、右クリックして最新の 生データを処理するを選択します。ウィンドウの右下隅をクリックして、データの処理を追跡します。「 実行するジョブなし 」というメッセージが表示されると、処理は完全に完了します。
- データ処理が完了すると、プレート内のウェルが緑色に変わります。ウェルを右クリックし、[ ワークフロー結果の表示] を選択します。参照データ ファイルごとに別のウィンドウが開きます。
- データを検査して、参照サンプルデータ内のMSシグナルの大部分が、対象タンパク質のインシリコ消化から予測されるペプチドに正常にマッピングされたことを確認します。一致したペプチドは、出力スペクトルで青色に着色されます(図4)。[OK] フィルターをダブルクリックし、パーセントカバレッジが 99% を超えているか確認します。
注: 処理時に、データ出力は、ファイル拡張子(raw_data_file_name_IA_final_peptide) で、ステップ 4.5 で指定したディレクトリに自動的に保存されます。 - 追加のしきい値を得る場合は、HDX処理ソフトウェア(材料表)に、プロテオミクスのソフトウェア出力をインポートします。
- HDX 処理ソフトウェアウィンドウの左隅にある [データ ] をクリックします。 PLGSの結果をインポート してクリックし、追加アイコンをクリックします。適切なディレクトリに移動して、ステップ 4.9 から処理済みデータ ファイルを選択します。
- [次へ]をクリックし、最低連続したイオン≥2、質量誤差= 5 ppm、ファイル閾値= 3のパラメータを指定します。[完了] をクリックします。
- しきい値パラメーターに問題がなければ、HDX プロジェクトを保存します。すべてのHDXデータは、分析と表示のためにこのプロジェクトにインポートされます。
注: 次のセクションで説明する重水素交換サンプルは、同一の LC-MS ワークフローで処理する必要があります。したがって、HDXアッセイ(セクション5)を進める前に、サンプル調製(セクション2)、ボトムアップLC-MSワークフロー(セクション3)およびデータ処理ワークフロー(セクション4)が、標的タンパク質の所望の再現性および配列カバレッジを提供していることを確認してください。これらのプロセスのいずれかを変更してカバレッジを改善する必要がある場合は、ステップ2.1に戻り、新しい参照サンプルを三重に準備し、セクション2-4を繰り返し(プロトコルに必要な調整をしながら)各ペプチドを再現的に生成して検出できるようにすることをお勧めします。
5. HDX反応の実施
- HDX反応のためのワークスペースを準備します。
- アリコート前のクエンチバッファーは、適切に標識された0.5 mLチューブに入った。各時点、複製、および分析されるすべての生化学的状態に対して異なるチューブを用意します。HDX反応の50 μL部分の最終的なpH測定量を2.3の値に調整するために必要なステップ2.2からの適切な量のクエンチバッファを使用してください。
- 0.5 mLの槽を簡単に遠心分離し、すべてのクエンチバッファーをチューブの底部に移します。チューブを氷の上に置きます。
- 液体窒素で小さなデュワーを充填し、ワークスペースに隣接して保ちます。
- HDX 反応を準備します。必要な交換ポイント数(所望のタイムポイントごとに1つの50 μLアリコート)を収集するのに十分な反応量があることを確認してください。タイムスケールで3-4桁以上の少なくとも4-5のタイムポイントを収集します(例えば、15 s、60 s、300 s [5分]、1,800 s [30分]、および14,400 s [4時間]のクエンチ時間は、ほとんどの酵素の交換ダイナミクスを十分にカバーします)。
- D2Oのステップ1.1から全ての重水素成分(マイナス酵素)をプレミックスする。
- 検査される生化学的状態(遊離酵素、酵素+リガンド、酵素+阻害剤など)ごとに、少なくとも三重でHDX反応を調製する。
- 酵素を添加する前に、25°Cの温度管理水浴中に反応混合物を10分間インキュベートします。
注:HDXアッセイへのプロチウムの添加を最小限に抑えるために、酵素は濃縮ストック溶液(〜100-200 μM、ステップ1.6)として調製する必要があります。 - 酵素を1~5μMの最終濃度に加えたら、タイマーを開始します。200 μL ピペットを使用して溶液を慎重かつ迅速に混合し、サンプル中に酵素が均等に分散されるようにします。
- 希望の交換時間ポイントで、HDX反応から50 μLのアリコートを取り除き、0.5 mLチューブ内の事前に引用された氷冷クエンチバッファーと素早く均等に混合します。
注:混合ボリュームと混合手順は、2.3の望ましい最終的なクエンチpLがすべてのサンプルで迅速に達成されることを確実にするために、できるだけ正確かつ再現可能である必要があります。クエンチバッファー氷を冷たく保つことは、酵素の変性時の交換を最小限に抑えるのに役立ちます。 - HDXサンプルを焼き付けた直後に、チューブをキャップし、液体窒素でフリーズします。
- すべてのアッセイが完了するまで時間を収集し続け、サンプルを-80°C冷凍庫に移して保管します。
注: これは停止点になる場合があります。すべてのHDXのタイムポイントを収集した後、LC-MS分析の準備ができるまで、サンプルは-80°Cで保存することができます。理想的には、3倍のHDX反応は、同じ日に関心のあるすべての生化学的状態に対して行われるべきである。少なくとも、特定の生化学的状態に対するすべての複製HDX反応は、同じ日に並行して実行する必要があります。
- すべての急がいった HDX タイム ポイントを収集した後、サンプルを、ステップ 3.3 で説明したように開発された最適化されたボトムアップ LC-MS ワークフローに適用します。サンプル間に適切な数のブランクを含むランダム化された順序で HDX サンプルを注入して、ペプチドの持ち越しが最小限になるようにします。
注: HDX データは MSE モードで収集する必要はありません。したがって、高衝突エネルギーセグメント(ステップ3.3.7.2)はMSデューティサイクルから削除する必要があります。これは、ステップ 3.3 で説明したワークフローに対する変更のみです。 - 収集中の HDX データの品質を評価します。
- 非全イオンクロマトグラムの全イオンクロマトグラムに存在するクロマトグラフィーピークが、重水素化サンプルで同じ保持時間に現れるようにします(図3D-F)。
- 質量スペクトルが参照および重水素化されたサンプルから特定の時間間隔で合計された値が、重水素化の証拠を示していることを確認します(すなわち、個々のペプチドの同位体エンベロープの変化は、重水素化サンプルの高いm/z値に[図6])。
6. HDX データの処理
- ステップ 4.11 で作成した HDX プロジェクトに HDX データ をインポートするには、データ |上部のツールバーの MS ファイル 。
- 必要に応じて 「新規状態 」と 「新規暴露 」をクリックして、分析に関連する生化学的状態(例えば、遊離酵素、酵素+リガンドなど)と重水素暴露時間を定義します。
- [新規 Raw]をクリックして、分析する HDX データ ファイルを選択します。インポートされる各生データ ファイルに、適切な交換時間と生化学的状態を割り当てます。
注: データは、一括でインポートおよび処理することも、すべて一度に処理することもできます。プロジェクトにデータを追加しても、そのプロジェクト内で以前に実行された分析は元に戻りません。
- データ ファイルを追加したら、[ 完了 ] をクリックしてデータ処理を開始します。少しの遅延の後、ソフトウェアは、ユーザーが続行する前に、データを保存する必要があるかどうかを尋ねます。[ はい] をクリックします。
注:初期処理は、分析されるサンプルの数、最終的なペプチドリスト(ステップ4.10)に含まれるペプチドの数、クロマトグラフィーウィンドウのサイズ、およびスペクトル取得の頻度に応じて、数時間かかることがあります。 - 必要に応じて、[ 構成] メニューの処理パラメータを変更して、イオン検索パラメータを変更します。特定のプロジェクト内のすべてのデータに対して同じイオン検索パラメータを使用してください。
7. HDXデータの分析と可視化
注:生データの初期処理が完了すると(ステップ6.2)、HDX処理ソフトウェアは、分析された各生データファイルのペプチドリスト(ステップ4.10で生成)からペプチドを配置します。リスト内のペプチドの同位体分布が生データファイルに配置されると、HDX処理ソフトウェアは各同位体を「スティック」で表します( 図6C-Eのように)。次に、特定のペプチドのスティックの相対強度を使用して、参照スペクトルに対する重水素取り込みの計算が行われます。HDX処理ソフトウェアは、ほとんどのペプチドに「スティック」を適切に割り当てるという素晴らしい仕事をしていますが、重水素取り込み値の大幅な手動キュレーションが必要です。
- ペプチド重水素摂取値の分析
- ペプチドリストの最初のペプチドを選択し、ビューメニューから積み上げスペクトルプロットを開きます。積み上げスペクトルプロットウィンドウで上下にスクロールすると、選択したペプチドの質量スペクトルが重水素交換時間の関数として表示されます(図7D,E)。
- マウスクリックを使用してスティックを割り当て、割り当て解除して、適切な同位体分布がデータに配置され、各同位体のピークが割り当てられていることを確認します(割り当てられたスティックは青色に表示されます)。スタックされたスペクトルプロット (図 7D,E)のスペクトルをクリックすると、アクティブなデータビューアウィンドウでスティックを割り当て/割り当て解除できるようになります (図7C)。
- 積み上げスペクトルプロットウィンドウの上部にある充電状態を切り替えて、各充電状態のスティック割り当てを確認します。
- 対象の生化学的状態ごとに、ステップ 7.1.1~7.1.3 を繰り返します。生化学的状態は、積み上げスペクトルプロットウィンドウの上部で切り替えることもできます。最も正確な重水素取り込み差の測定のために、各生化学的状態のための電荷状態の同じセットにスティックが割り当てられていることを確認してください。
- ペプチドリストのペプチドごとにステップ7.1.1-7.1.4を繰り返します。
- カバレッジマップを使用して、ペプチド重水素取り込み値の標準偏差を確認します。
- 対象となるタンパク質のアミノ酸配列に沿ってマッピングされたペプチドリストの各ペプチドを表示する ビュー メニューからカバレッジマップにアクセスします(図8C)。相対標準偏差(Daの単位)に従ってペプチドを着色します。
- 相対標準偏差が高い外れ値ペプチドを視覚的に検索します。カバレッジ マップの外れ値ペプチドをクリックして、積み上げスペクトルプロット (図8B) とデータ ビューア ウィンドウ (図 8A) にターゲット ペプチドを入力します。
- 積み重ねられたスペクトルプロットを使用して、外れ値ペプチドのすべての電荷状態とすべての時間ポイントが適切にスティックを割り当てられていることを注意深く確認してください。
注: ほとんどの場合、標準偏差が大きいペプチド(>0.3 Da)は、ソフトウェアによって適切に割り当てられていない同位体ピークを有します( 図8Bに示されています)。欠落しているスティックを割り当てると、一般にペプチドの相対標準偏差を <0.3 Da に縮小できます。 - 相対標準偏差を0.3Da未満に減らすことができない場合は、リストからペプチドを非表示にします。
- 目的のタンパク質の構造モデルにマッピングするための2つの生化学的状態間のHDX差データをエクスポートします。
- カバレッジ マップの関心の差を表示します。カバレッジ マップを右クリックして、差分データを.csv ファイルにエクスポートします。データ |に移動して状態データを (.csv形式で) エクスポートします 。 メインツールバーで 状態データをエクスポート します。
注: 差分データと状態データ ファイルの適切なフォーマットは 、サポート情報 に記載されています。 - 目的のタンパク質の差分データ、状態データ、および pdb ファイルを Deuteros28にインポートします。[99% 信頼区間] を選択し、[ 合計を有効にする] を選択してデータを処理します。
メモ:マテロスを実行するには、MATLABをPCにインストールする必要があります。Deuterosは、データセット内の反復測定値を使用して、各ペプチドの取り込みデータの標準偏差を計算します。この標準偏差は、プロットに表示される有意な交換の信頼区間を定義するために使用されます。 - PyMOLオプションで、輸出取り込み|のエクスポートを使用して、重要な交換の違いの領域を、PyMOLソフトウェアを使用して目的のタンパク質のpdb構造にマッピングするPymolスクリプトを生成します。
注: このプロトコルで説明されているワークフローを使用すると、特定のペプチドに対する有意な重水素取り込み差の 99% 信頼区間は、通常 0.3-0.5 Da です。すべての交換時間ポイントで合計される差の 99% 信頼区間は、通常 0.7-1.0 Da です。
- カバレッジ マップの関心の差を表示します。カバレッジ マップを右クリックして、差分データを.csv ファイルにエクスポートします。データ |に移動して状態データを (.csv形式で) エクスポートします 。 メインツールバーで 状態データをエクスポート します。
結果
タンパク質分解消化の品質とサンプル注射の各セットのワークフローの再現性を評価する必要があります。したがって、HDX-MSアッセイを行う前に、目的のタンパク質のタンパク質分解、逆相液体クロマトグラフィーと気相イオン移動度を用いたペプチドの分離、およびMSを用いたペプチドの検出に有効な条件を確立することが不可欠です。この目的のために、対象となるタンパク質(重水素の不在時に採取した)の参照サンプルをまず調査する必要がある(セクション3)。 図3A-C のクロマトグラフィーデータは、HalM2ランチペプチド合成酵素の3つの参照サンプルに対するイオンクロマトグラム(TIC)の合計を示しています。TIC は、質量スペクトルスキャンに含まれるすべての m/z 値におけるすべてのイオンカウントの合計における時間依存変化です。 図 3D-Fに、3 ~ 8 分の間の TIC の詳細なビューを示します。 図 3G-I に示す質量スペクトルは、各 TIC のわずかな時間間隔 (5.0 ~ 5.1 分) にわたるすべての質量スペクトルの合計を表します。 図 3の次の機能に特に注意する必要があります。
まず、勾配の終点の9.3分近くの大きなピークは、HalM2の不完全に消化された(したがって、より大きく、より疎水性の)断片を表す(図3A−C)。消化はHalM2濃度を下げることで効率を高めることができたが、これはペプチドシグナル強度とシグナル:ノイズ比を減少させる。消化はまたpepsinカラムとの接触時間(3分、プロトコルステップ3.3.3から)を増やすことによってより効率的にすることができる。ただし、接触時間が増加すると、より多くのバック交換が行われます。最終的には、これらのパラメータは、目的の配列カバレッジ、シグナル強度、および重水素保持を提供するために、目的のタンパク質のバランスを取る必要があります。次に、TIC プロファイルの形状と強度は同じである必要があります( 図 3D-Fのように)。これは、HalM2のタンパク質分解性消化が再現可能であり、3つの参照サンプルすべてにおいて同様の効率であることを示唆している。重水素交換サンプルの同様の消化は、同様の保持時間を持つペプチドの同じセットを生成することが期待されます。第 3 に、特定の時間間隔における質量スペクトルも同様である必要があります (図 3G-I)。クロマトグラフィー分離の5.0〜5.1分間隔での質量スペクトルの素早い視覚的比較は、各サンプルの質量スペクトル信号が実際に非常に類似していることを示しており、各サンプルに類似したペプチドが存在し、C18カラムから同様の時間に溶出しているという確信を与えます。同様の迅速な目視検査は、クロマトグラム全体で他の時間間隔で行う必要があります。
参照サンプルのLC-MSデータの類似性を視覚的に検証した後、プロテオミクスソフトウェアは、目的のタンパク質のアミノ酸配列に由来するペプチドのMSデータを検索するために使用される。タンパク質配列データバンク(対象となるタンパク質のアミノ酸配列)、プロトコルステップ4.2-4.4に記載されている処理およびワークフローパラメータを定義した後、各々の参照サンプルを処理して、目的のタンパク質由来の予測ペプチドと一致するペプチドペプチドを検出する。非重水素化された HalM2 参照サンプルに対するプロテオミクスソフトウェア出力の例を 図 4に示します。特に以下の機能に注意する必要があります。まず、処理およびワークフローパラメータで定義された値に従って、目的のタンパク質内の特定のペプチドに一致するMSシグナルは、底部スペクトルで青色に着色されます。参照サンプルが「正常に」処理されると、大部分のシグナルは青色で表示されます ( 図 4のデータの場合と同様)。さらに、左上のパネルで、「OKフィルター」が緑色のチェックマークに切り替えられることを確認します。これを行うと、右上パネルの上部バー( 図4の青色で強調表示されている)の統計には、高い信頼度スコアを与えるペプチドのみが反映されます。これらのペプチドは、ppm質量精度が低く、複数のフラグメントイオン、およびフラグメントイオン保持時間と親イオン保持時間の間の良好な相関を示し、かつ確実な同定であると考えられる。示されているHalM2の参照サンプルでは、1,421個のペプチドが高いスコアで検出され、HalM2配列の99.6%をカバーしました。プロトコルセクション3で説明されているボトムアップLC-MSワークフローを使用して、ほとんどのタンパク質でほぼ100%の配列カバレッジを達成できるはずです。さらに、全てのペプチドに関する追加の有用な統計は、質量誤差、前駆体保持時間、フラグメント数(生成イオン)、フラグメントイオンの完全なリスト、イオン移動性ドリフト時間など、右上パネルに表されています。この情報は、最も確実に同定されたペプチドに常に焦点を当てるべき結果の解釈に有用である。
最終的なペプチドリストは、HDX処理ソフトウェアの追加の閾値によって決定されるべきである。参照データを分析してペプチドリスト(プロトコルセクション4)を生成した後、出力をHDX処理ソフトウェアにインポートして追加のしきい値を設定する必要があります。出力ファイルは、プロトコルステップ 4.5 で定義されたディレクトリに格納され、ファイル拡張子は 「..._IA_final_peptide.csv。HDX処理ソフトウェアに出力をアップロードすると、ペプチドの完全なリストが「ペプチドプレビュー」パネル(図5)に表示され、ウィンドウの右下隅にペプチド、シーケンスカバレッジ、冗長性の数が表示されます。これらの値は、追加のしきい値が適用されるとリアルタイムで変化します。 [次へ] をクリックすると、指定したフィールドでしきい値を設定できます。最も重要なフィルタは、すべてのペプチドを3つの参照サンプルのそれぞれに配置する必要がある「ファイルしきい値」、値<10 ppmに設定する必要がある「最大MH+誤差(ppm)」、および「 最小連続製品」であり、これはペプチドがMSMS(すなわち、高衝突エネルギー)フェーズ中に少なくとも2つの連続した断片イオンを生成する必要がある。
HDX-MSアッセイの最も重要な技術的制限は、重水素交換サンプルが(プロティアードバッファー付き)消し込まれるとすぐに発生するプロチウムに対する重水素のバック交換です。ワークフローのプロテアーゼ消化およびLC-MS部分の間、バック交換が継続されます。バック交換は避けられませんが、すべてのポストクエンチステップのpH、温度、およびタイミングがプロトコルに記述されているように慎重に制御されている場合、重水素ラベルの60%-70%を維持することができます。このため、アッセイの初期段階では、ペプチドのほとんどが重水素を保持していることを確認することが不可欠です。これは、重水素サンプルの生データと参照サンプルの生データを比較することで、迅速に達成できます。参照サンプルの再現性を確保するために行われたように (図 3)、重水素交換と非重膜参照サンプルの TIC を比較し、プロファイルの形状が互いに類似していることを確認します。さらに、両方のサンプルで同じクロマトグラフィー時間間隔にわたって質量スペクトルを合計します。重水素交換サンプル中のペプチドのほとんどは、同位体分布のm/z値の上昇に向けた明らかな変化を示すべきである( 図6)。このデータは、重水素ラベルのかなりの割合が、酸クエンチ、ペプシン消化、およびLC-MSデータ収集の過程を通じて維持されていることを示しています。
ワークフローが十分な重水素保持を提供していることを確認した後、重水素の取り込み量を定量化する必要があります。したがって、重水素交換サンプルの生データはHDX処理ソフトウェアにインポートされます。HDX処理ソフトウェアは、参照サンプルと最終ペプチドリストを使用して、各生データファイル内のペプチドリストからペプチドを見つけ、それぞれの同位体ピークに「スティック」を割り当てます。図 7に示す HalM2 の代表的なデータでは、正常に割り当てられたスティックは、すべてのスペクトルで青色で表示されます。スティックを割り当てた後、同位体分布の重心 m/z 値はソフトウェアによって決定され、非重奏参照サンプルに対する重水素取り込みの計算に使用されます。各ペプチドの重水素交換値は手動でチェックする必要があります。図7では、現在選択されているペプチド(HIDKLTVGL、HalM2残基110−118にまたがる)がメインデータビューアの左側のパネルに示されている(図7A)。他のパネルは、このペプチドに関連付けられたHDXデータを示す。ペプチドリスト内の任意のペプチドをクリックすると、そのペプチドからHDXデータを他のパネルに取り込みます。図7Bは、ペプチド110−118の重水素取り込みプロットを示す。このデータ・セットには、4 つの交換時間ポイント(0.5、5、30、および 240 分)が含まれています(3 重単位で収集)。交換データは、2つの生化学的状態について収集された:遊離のHalM2酵素(赤)とAMPPNPとその基質ペプチドHalA2(青)に複合化されたHalM2酵素。時間経過全体にわたってペプチド110-118に有意な重水素取り込み差と反復測定の高精度に注意してください(誤差バーは図7Bのプロットに示されています)。一般に、取り込み測定における同様の精度は、ほとんどのペプチドに対して得られ、このプロトコルで提示されるワークフローの再現性を強調する。リガンド結合(図7Bの青曲線)の際、HalM2の110-118領域のペプチド結合アミドは、明らかに重水素交換から有意な保護を受け、110-118領域のアミノ酸がリガンド結合時に安定的に構造化されていることを示唆している。この領域のその後の変異生成は、前駆体ペプチドHalA220に結合する役割を示した。図 7に示されているのは、HalM2 状態 (図 7D)と HalM2:AMPPNP:HalA2 状態 (図 7E)のスタックスペクトルプロットです。これらのプロットでは、交換時間が下から上に増加します。より長いポイントでの重水素交換時のペプチド110~118の質量増加は、目視検査から明らかである。また、ペプチド110-118が完全にリガンドされた状態(図7E)よりも、HalM2状態(図7D)でより重水素を取り込むことも明らかです。必要に応じて、図 7D または図 7E のいずれかのサブプロットをクリックすると、メイン データ ビューアのスティックの割り当てを手動で変更できます (図 7C)。スティックの割り当て/割り当て解除時に、HDX処理ソフトウェアは重水素の取り込み値を再計算し、すべてのプロットがリアルタイムで更新されます。同様に、パネル B の個々のデータポイントをクリックすると、手動でスティックを割り当てるためのパネル C のデータビューアーが表示されます。
各ペプチドにスティックが割り当てられ、すべての生化学的状態の時間差が交換された後、測定された取り込み値の標準偏差をチェックする必要があります。これは、カバレッジ マップと積み上げスペクトル プロットで最も簡単に実行できます (図 8)。カバレッジ マップ (図 8C)で、対象の状態と交換時間を選択し、相対取り込み標準偏差を選択します。被覆マップ内のペプチドは、測定された重水素取り込み値の標準偏差に従って着色されます。このように、同位体が誤って割り当てられているペプチドを視覚的に識別することは非常に容易になります。カバレッジマップで外れ値ペプチド(高標準偏差)をクリックすると、メインデータビューアと積み上げスペクトルプロットに外れ値ペプチドのHDXデータが表示されます。各時間ポイントと充電状態のスティックの割り当ては、測定された取り込み値を修正するために必要に応じて変更することができます。繰り返しますが、HDX処理ソフトウェアは、スティックの割り当てが変更されるとリアルタイムですべてのデータ表示を更新します。
HDXデータセットを完全にキュレーションした後、各ペプチドの重水素取り込み差測定の重要性は、データ解釈の前に考慮する必要があります。Engenと同僚29によって記述されたアプローチを用いて、我々は、上記プロトコル20に記載されたワークフローを用いて、HalM2タンパク質に対して0.1±0.1Daの平均取り込み値を推定した。この値は、同様のボトムアップ、連続交換 HDX ワークフロー29、30に対して他のユーザーが報告したエラーと一致しています。最近、ポリティスと同僚は、HDXデータセット28の著しい取り込みの違いを迅速に決定するための有用なオープンソースツール(MATLABに実装)であるDeuterosを開発しました。代表入力ファイル(「State_Data_for_Deuteros」と「Difference_Data_for_Deuteros」)は、サポート情報に含まれています。このプロトコル(表)で説明されているHDX処理ソフトウェアから直接エクスポートした場合(資料一覧)、差分と状態のデータファイルは、Deuterosによるファイル読み取りに適した形式になります。HDX データが別の LC-MS システムで生成される場合、サポート情報で提供されるデータ ファイルに似たデータ ファイルを手動で作成する必要があります。
Dueteros ワークスペースを図 9に示します。データが Deuteros にインポートされると、ユーザーは目的の信頼限界 (>98% 推奨) を選択し、[ インポートと計算] をクリックします。フラット化されたデータ マップの場合は、データ タイプの カバレッジ とカラー スケールの 絶対 値を選択します。[ プロット] をクリックします。林図の場合、データタイプとして バイナリフィルタ を選択し、目的の信頼フィルタを選択し、合計を有効にします。 プロットをクリックすると、Deuterosは、アミノ酸配列内の位置とその重水素取り込み値の関数として、交換時の各時点ですべてのペプチドをプロットします。有意な交換を示すペプチドは、計算される差に応じて、ペプチドがそれぞれ多かれ少なかれ重水素を占めるかどうかに応じて、赤または青色で着色される。各プロットで報告される有意値( 図9にプロットされたデータの0.39から0.72 Daの範囲)は、データセット内に存在する反復測定値から決定される各ペプチドについて測定された取り込み差の標準偏差から計算されます。信頼区間は、個々のプロットに対して破線のバーとして表示されます。有効にすると、「Sum」は、各ペプチドの各時点で測定された取り込み差を単純に加算します。最後に、目的のタンパク質の3次元構造に関する取り込みデータの可視化のために、PyMOLオプションで 輸出取り込み からエクスポートを選択し、 をエクスポートします。Deuterosは、目的のタンパク質のオープンpdbファイルを含むPyMOLワークスペースにドラッグアンドドロップできるPyMOLスクリプトを生成します。
このプロトコルで提示されたHDX-MSワークフローは、遺伝的にコードされたペプチド(HalA2)20に一連の翻訳後修飾を触媒する酵素(HalM2)の生化学的性質を特徴付けるために使用された。図10において、HALM2:AMP-PNP複合体へのHalA2前駆体ペプチドの結合に関する代表的なHDX-MSの結果が示されている。パネル A は HalM2 カバレッジ マップを示しており、色は [HalM2:AMPPNP] 生化学的状態と [HalM2:AMPPNP:HalA2] 状態との相対的な取り込み差を示します。これらの取り込みの違いをHalM2酵素の相同性モデルにマッピングするために使用されたデウテロス(図10B,C)。赤色に着色されたペプチドは、HalA2結合に対する重水素摂取の減少を示し、これらの領域のHalM2が前駆体ペプチドの結合に直接関与する可能性があることを示唆している。この仮説を調べるには、領域I-IIIを変異させ、変異型酵素の運動特性を調べた。領域IおよびIIIの突然変異は、両方とも、HalA2ペプチド結合親和性において有意な摂動を導き、HalM2のこれらの領域がペプチド基質と直接相互作用しているか、またはHalA2結合を可能にする構造を形成するために必要であることを示唆している。対照的に、領域IIの変異は、HalA2結合親和性に影響を及ぼさなかったが、この変異体は触媒活性をほとんど欠いていた。この発見の1つの説明は、HalA2結合時に観察された領域IIの組織が、酵素を活性化する立体構造変化を引き起こすということです。なお、この研究の前に、HalM2-HalA2結合モードまたはシステムの触媒的に関連する立体構造変化に関する情報は、主にHalM2とHalA2の両方の大きなサイズと柔軟な性質が構造研究を妨げてきたため、利用可能な情報は存在しなかった。このように、HalM2ランチペプチド合成酵素に関するこれらの代表的なデータは、HDX-MSを使用して、高分解能構造データがない場合でも、構造的に動的な酵素系の機能的に関連する領域を迅速に見つけることができる方法を示している。
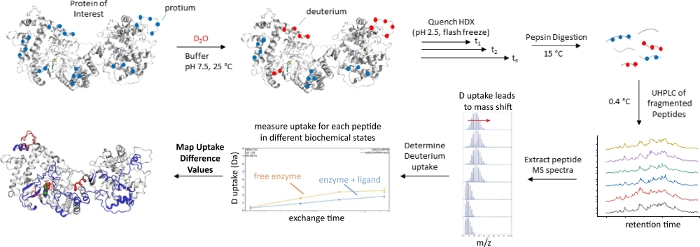
図 1: 継続的な交換、ボトムアップ HDX-MS ワークフロー。詳細については、テキストを参照してください。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図2:HDX速度は、タンパク質の立体構造ダイナミクス(kopenおよびk近接)およびN-D結合に対するタンパク質骨格におけるN-H結合の化学的交換のpH依存速度(kchem)に依存する。この図の大きなバージョンを表示するには、ここをクリックしてください。

図3:HalM2参照サンプルの三重化のための代表的なLC-MSデータ。各パネルの右上の数字は、イオンの合計数を表します。各行には、異なる参照サンプルのデータが表示されます。最初のカラム(A-C)は、イオンクロマトグラム(TIC)の合計を示しています。9.3分の大きなピークは、大きな未消化ペプチドを表します。中央の列(D-F)は、3 分から 8 分の間の TIC の詳細なビューを示しています。プロファイルの形状の良好な一致に注意してください, クロマトグラム全体にわたって各参照サンプル内のペプチド信号の同様の基礎混合物を示します.3番目の列(G-I)は、クロマトグラフィーの走行時間の5.0~5.1分の間に記録された質量スペクトルをすべて合計して生成した各基準サンプルの質量スペクトルを示しています。このデータを目視で調べると、各サンプルで同じペプチドの多くが検出されています。 この図の大きなバージョンを表示するには、ここをクリックしてください。
図4:HalM2参照サンプルの代表的なプロテオミクスソフトウェア出力。左上のパネルは、MSデータで検出されたHalM2由来ペプチドの完全なリストを示しています。「OKフィルタ」は緑のチェックマークを表示しています。これにより、信頼度スコアに従ってデータをフィルタリングし、低信頼のペプチド同定を除去します。右パネルの上部バー(青色でハイライト表示)は、高得点のペプチドセットの累積統計量を示しています。最も重要な統計は、検出されたペプチドの数(この場合は1,421)と配列カバレッジ(この場合は99.6%)です。すべてのペプチドの追加統計も右上のパネルに示されています。このデータ テーブルの各列は並べ替え可能です。下部パネルには、予測された HalM2 由来のペプチドペプチドと一致したすべての MS シグナルが表示されます。MS信号の大部分は割り当てられており、青色になっていることに注意してください。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図 5: HDX 処理ソフトウェアでの追加しきい値3つのHalM2リファレンスサンプルのそれぞれについて、プロテオミクスのソフトウェア出力がHDX処理ソフトウェア(左パネル)にインポートされます。データをインポートした後、追加のしきい値が実行されます (右側のパネル)。「最小連続生成物」分野とは、ペプチド中の隣接するペプチド結合の切断によって生成される断片イオンをいう。「最小MH+誤差」パラメータは、ユーザーが許容質量精度を定義することを可能にし、「ファイルの閾値」は、ユーザーが3つの参照ファイルすべてで検出されたペプチドのみにペプチドを制限することを可能にする。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図6:HalM2参照サンプル(赤)とハルM2サンプルを5分間(緑色)のD2Oバッファでインキュベートした代表的なデータ。どちらのサンプルも、プロトコルセクション3で説明されているボトムアップLC-MSワークフローに従った。左側の列は、6.0~6.1 分の時間枠で合計された質量スペクトルを示しています。3つの右の列は、重水素化されたサンプル(緑色、上)の高いm/z値へのシフトがペプチド信号のほとんどに明らかである同じ質量スペクトルのより近いビューを示しています。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図 7: HDX 処理ソフトウェアのワークスペースのスクリーンショット(A)プロテオミクスソフトウェアによるHalM2参照サンプルの分析から得られたHalM2由来ペプチドのリスト(図3)とHDX処理ソフトウェアにおけるその後の閾値(図4)現在選択されているペプチド(HIDKLTVGL、HalM2残基110−118にまたがる)は青色で強調表示される。(B) 対象となる2つの生化学的状態に対する重水素取り込み曲線(交換時間の関数として):遊離のHalM2酵素と、AMPPNPと前駆体ランチペプチドに結合したHalM2酵素、HalA2。(C)アクティブに選択されたサンプルの質量スペクトル (この場合、未処理の HalM2 参照サンプルの 1 つ)。パネル C の青いスティックは、初期データ処理時に HDX 処理ソフトウェアによって適切に割り当てられていない場合、必要に応じて手動で割り当て/割り当てを解除できます。(D,E)HalM2 状態(D)および HalM2:AMPPNP:HalA2 状態 (E) の積み上げスペクトル プロット。重水素の取り込みの時間依存性の増加は、2つの生化学的状態の取り込み差と同様に、容易に見える。この図の大きなバージョンを表示するには、ここをクリックしてください。

図8:測定した取り込み値の標準偏差を最小限に抑える。HDX処理ソフトウェア(パネルC)のカバレッジマップは、測定された重水素の取り込み値に大きな標準偏差を持つペプチドを迅速に同定する便利な手段を提供します。この仮定の場合、HalM2由来ペプチドの一部の同位体ピーク(青スティック)は、残基110~118に及ぶ110-118に対して、5分交換時間ポイント(パネルBのグレーのスティック)に割り当てられていない。これは5分交換時間に測定された重水素取り込み値の大きい標準偏差に至る。大きな標準偏差は、カバレッジマップ(パネルC)の110-118ペプチドの青色、取り込みプロット(パネルA)のデータポイント散乱と大誤差バーから容易に明らかである。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図 9: 作業スペースこの例では、取り込み差値をHDX処理ソフトウェアで計算し、HalM2:AMPPNP:HalA2状態の重水素取り込み値をHalM2:AMPPNPの状態から差し引いた。比較の目的は、ペプチド(HalA2)結合がHalM2:AMPPNP複合体の構造ダイナミクスをどのように変化させたかを可視化することであった。データ・セットには、4 つの交換時間ポイント (0.5、5、30、および 240 分) が含まれていました。交換時間の各時点における各ペプチドの取り込み差は、ウッズプロットに示されています。ウッズプロットの着色ペプチドは、データセットに存在する反復測定値の標準偏差と、Deuterosで選択されたユーザ定義の信頼限界によって定義されるように、有意な取り込み差を示すペプチドを示しています。 この図の大きなバージョンを表示するには、ここをクリックしてください。
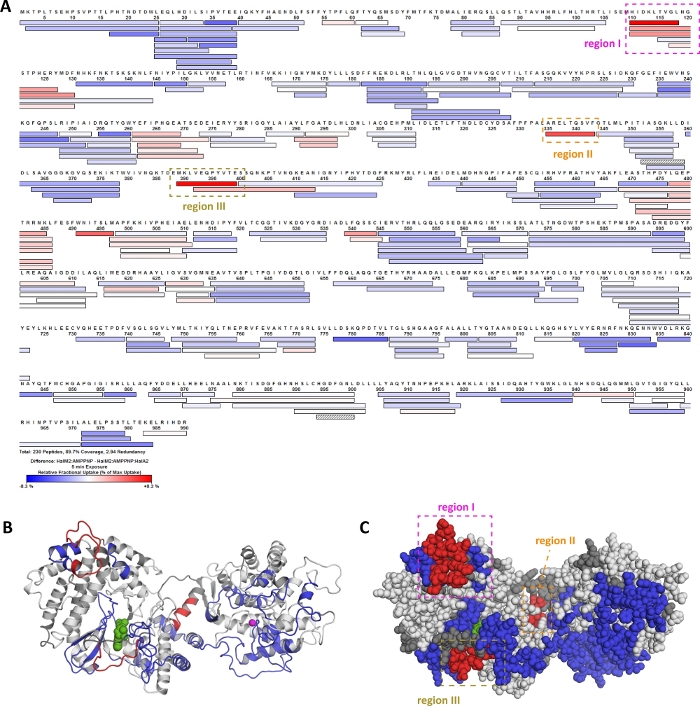
図10:HDX-MSデータガイドランチペプチド合成酵素HalM2におけるペプチド結合およびアロステリック活性化の機能分析。HalA2前駆体ペプチドとHalM2ランチペプチド合成酵素との結合時の重水素取り込み変化を調査した。5分交換反応後の各ペプチドの取り込み差がプロットされる(A)。このプロットは、HDX処理ソフトウェアで生成されました。赤と青に着色されたペプチドは、HalA2ペプチドの存在下でそれぞれ重水素の取り込みが少なくなります。これらのHDXの「ホットスポット」は、ハルM2相同性モデル(B,C)にマッピングされています。有意な交換差を受けているペプチドの同定は、デウテロスで決定された。異なる値を相同性モデルにマッピングするために使用されるPyMOLスクリプトは、Deuterosで生成されました。 この図の大きなバージョンを表示するには、ここをクリックしてください。
ディスカッション
このプロトコルで提示されるHDX-MSワークフローは、タンパク質中の構造的に動的な要素の空間分布をマッピングし、これらのダイナミクスが摂動(リガンド結合、酵素変異生成など)にどのように変化するかを調査するための非常に堅牢なプラットフォームを提供します。HDX-MSは、立体構造ダイナミクスの調査に一般的に使用される他の構造生物学アプローチに対して、いくつかの明確な利点を有する。最も顕著なのは、少量のタンパク質のみが必要です。ここに記載されているワークフローを用いて、1 μMタンパク質の1 mLサンプルは、5つの交換時間ポイントを含むHDX反応を3倍にするのに十分な材料を提供します。さらに、対象となるタンパク質にサイズ制限は事実上なく、タンパク質複合体はHDX-MSアプローチにも同様に適しています。サイズ制限は、ペプチドペプチドをクロマトグラフィー的に分解できる程度、およびm/zおよびイオン移動性寸法でのみ制限されます。このように、多くのタンパク質/タンパク質複合体に対して、HDX-MSはタンパク質とタンパク質相互作用の相互作用の相互作用、リガンド結合部位、立体構造ダイナミクス、およびアロステリックネットワークに関する貴重な情報を提供します。最後に、HDX反応は、タンパク質の部位特異的な標識/エンジニアリングを必要とせずに、穏やかでほぼネイティブの条件下で行われ、内因性活性が確実に保持されるようにするのに役立ちます。このアプローチの主な制限事項(目的のタンパク質に関して提供する機械化情報の観点から)は、ペプチドレベルのHDXデータが本質的に低い空間分解能であるということです。したがって、データは二次構造の変化が起こっていると推測するのに十分であるが、構造摂動の機構的影響は、より高い分解能(例えば、NMR、cryoEM、またはX線結晶)構造、計算研究、および/または生化学的研究を完全に解釈する必要がある。
結果の再現性と信頼性を確保するためには、プロトコルのいくつかの重要な側面を念頭に置く必要があります。まず、HDX反応中の重水素交換の程度と、作業および解析中のバック交換の程度は、pH、時間、および温度に大きく依存します。したがって、バッファー、HDX 反応、および LC-MS メソッドの準備手順は、データ セット間でのこれらの物理パラメーターの変動を最小限に抑えるために、可能な限り体系的にする必要があります。可能な限り、比較される生化学的状態のHDX反応は同じ日に同じ研究者によって行われるべきであり、これらのサンプルのLC-MSデータは同じ溶媒のバッチで連続して収集されるべきである。時間が経つにつれて、ペプシン消化の効率も低下するので、特にペプシンカラムが他の多くのタイプのサンプルを消化するために使用されている場合、比較的狭い時間枠内で消化および分析されるサンプルに最適です。標準タンパク質を用いてペプシンカラムの消化効率を定期的にチェックするのが良い習慣です。これを達成するために、新しく消化されたサンプルを古いサンプルと比較できる専用のHDX処理ソフトウェアプロジェクトを設定して、同じターゲットペプチドのセットが同じ強度で検出されることを確認することができます。
このプロトコルで示されているワークフローは、多くの酵素/タンパク質システムに適切なデータを提供する必要がありますが、最適化の潜在的な点がいくつかあります。まず、HDX反応の時間スケールを変更して、より高速/遅いダイナミクスをキャプチャすることができます。最も顕著なのは、多くの酵素は数秒以内に完全に交換される非常に動的な要素を含むことです。これらの極めて動的な要素が関心を持つ場合、定常状態のメソッド、連続交換HDX-MSが文献31に報告されている。第二に、LC法の条件は、消化効率(最終的にはシーケンスカバレッジを決定する)および重水素保持(最終的には方法の感度を決定する)を変更するために容易に変更することができます。ペプシン消化の持続時間と温度を考慮すると、流量が遅く、温度が高いほど、タンパク質のより徹底的な消化が好まれます。また、HDXアッセイにおけるタンパク質濃度は、シグナル強度が低すぎると増加したり、ペプシン消化が非効率的な場合に減少したりすることができる。分析C18カラム上のペプチドを分離するために使用されるアセトニトリル勾配を考慮すると、より速い勾配は重水素ラベルを保持しますが、消化された反応混合物に存在する消化ペプチドのクロマトグラフィー分解能を犠牲にします。消化器内に存在するペプチドが少ない小さなタンパク質(約200個のアミノ酸)の場合、より速いLC勾配を実装しやすくなる。HalM2(約1,000アミノ酸)のような大きなタンパク質の場合、混合物中のより多くのペプチドによって生成される追加のスペクトルの複雑さを解決するために、より長い勾配が必要です。この後者のシナリオでは、ガス相イオン移動性分離を含めることで、解析のピーク容量を劇的に向上させることができます。イオンモビリティ分離を含むのは、ノイズ比に対する信号の低減を犠牲にします。最後に、HDXサンプルのMSデータをMSE モードで収集する必要はありません。MSE デューティサイクルのMSMS部分(すなわち、高衝突エネルギーセグメント、ステップ3.3.7.2)は、ペプチドリスト(プロトコルセクション4)を定義するために参照サンプルにのみ必要です。したがって、HDXサンプルは、信号:ノイズ比を増加させるためにMS専用モードで分析する必要があります。
このプロトコルで説明されているワークフローが正常に動作している場合、完全に交換されたペプチドは、60%-70%の相対的な重水素取り込み値を示す必要があります。重水素の取り込みがこれよりかなり低い(溶媒暴露された、保護されていないペプチド)場合、最も可能性の高い説明は、サンプルのpHがワークアップ/分析の一部の部分の間に変化していることです。このシナリオでは、マイクロチップ電極を使用して、HDXアッセイのpHとクエンチ反応アリコートを注意深く監視する必要があります。LC-MS溶媒のpHもチェックする必要があります。この問題の発生を最小限に抑えるために、アッセイに必要なすべてのバッファーおよび試薬の濃縮ストック溶液を準備し、保管することを強くお勧めします(プロトコルセクション1に概説)。
近年、HDX-MSはタンパク質の構造力学を調査する強力な分析ツールとして登場しています。HDX実験用に設計および最適化された市販のLC-MSシステムの開発(この研究で使用され、材料に記載されているシステムなど)は、強力なソフトウェアパッケージと相まって、HDX-MSアプローチを多くの学術および産業ラボに拡張し、15年前のニッチ技術をユーザーフレンドリーな分析プラットフォームに変えました。空間分解能の限界にもかかわらず、HDX-MSはタンパク質運動に対して非常に定量的で再現性の高い測定を提供し、他のアプローチでは調査が困難な立体構造動的酵素の研究に理想的です。これらの特性のために、HDX-MSは、生物学と医学の多くの基本的な分野で関連性を持つ障害および動的タンパク質系の構造生物学に不可欠なニッチを埋めます。したがって、HDX-MS分析は、近い将来の構造生物学者の武器庫の重要なツールであり続けると予想されます。
開示事項
開示するものは何もありません。
謝辞
この研究は、カナダの自然科学工学研究評議会、ケベック自然とテクノロジー、カナダイノベーション財団、マギル大学のスタートアップ資金によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| [glu-1]-fibrinopeptide B (Glu-Fib) | BioBasic | NA | |
| 0.5 mL Amicon Ultracel 10k centrifugal filtration device (Millipore) | Milipore Sigma | UFC501096 | |
| acetonitrile | Fisher | A955-1 | |
| AMP-PNP | SIGMA | A2647-25MG | |
| ATP | SIGMA | a2383-5G | |
| D2O | ALDRICH | 435767-100G | |
| formic acid | Thermo Fisher | 28905 | |
| guanidine-HCl | VWR | 97063-764 | |
| HEPES | Fisher | BP310-1 | |
| Magnesium chloride | SiGMA-Aldrich | 63068-250G | |
| Potassium chloride | BioBasic | PB0440 | |
| potassium phosphate | BioBasic | PB0445 | |
| TCEP Hydrochloride | TRC Canada | T012500 | peptide was synthesized upon request |
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| software | |||
| Deuteros | Andy M C Lau, et al | version 1.08 | |
| DynamX | Waters | version 3.0 | |
| MassLynx | Waters | version 4.1 | |
| Protein Lynx Global Server (PLGS) | Waters | version 3.0.3 | |
| PyMOL | Schrödinger | version 2.2.2 | |
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| Instrument and equipment | |||
| ACQUITY UPLC BEH C18 analytical Column | Waters | 186002346 | |
| ACQUITY UPLC BEH C8 VanGuard Pre-column | Waters | 186003978 | |
| ACQUITY UPLC M-Class HDX System | Waters | ||
| HDX Manager | Waters | ||
| microtip pH electrode | Thermo Fisher | 13-620-291 | |
| Waters Enzymate BEH column or Pepsin solumn | Waters | 186007233 | |
| Waters Synapt G2-Si | Waters |
参考文献
- Karplus, M., McCammon, J. A. Molecular dynamics simulations of biomolecules. Nature Structural & Molecular Biology. 9 (9), 646-652 (2002).
- Campbell, E., et al. The role of protein dynamics in the evolution of new enzyme function. Nature Chemical Biology. 12 (11), 944-950 (2016).
- Tokuriki, N., Tawfik, D. S. Protein dynamism and evolvability. Science. 324 (5924), 203-207 (2009).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass Spectrometry Reviews. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chemical Society Reviews. 40 (3), 1224 (2011).
- Katta, V., Chait, B. T. Conformational changes in proteins probed by hydrogen-exchange electrospray-ionization mass spectrometry. Rapid Communications in Mass Spectrometry. 5 (4), 214 (1991).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Science. 2 (4), 522 (1993).
- Smith, D. L., Deng, Y., Zhang, Z. Probing the non-covalent structure of proteins by amide hydrogen exchange and mass spectrometry. Journal of Mass Spectrometry. 32 (2), 135-146 (1997).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Xiao, K., et al. Revealing the architecture of protein complexes by an orthogonal approach combining HDXMS, CXMS, and disulfide trapping. Nature Protocols. 13 (6), 1403-1428 (2018).
- Smith, D. L., Deng, Y., Zhang, Z. Probing the non-covalent structure of proteins by amide hydrogen exchange and mass spectrometry. Journal of Mass Spectrometry. 32 (2), 135-146 (1997).
- Masson, G. R., et al. Recommendations for performing, interpreting and reporting hydrogen deuterium exchange mass spectrometry (HDX-MS) experiments. Nature Methods. 16 (7), 595-602 (2019).
- Liu, J., et al. An Efficient Site-Specific Method for Irreversible Covalent Labeling of Proteins with a Fluorophore. Scientific Reports. 5, 16883 (2015).
- Marion, D., et al. Overcoming the overlap problem in the assignment of proton NMR spectra of larger proteins by use of three-dimensional heteronuclear proton-nitrogen-15 Hartmann-Hahn-multiple quantum coherence and nuclear Overhauser-multiple quantum coherence spectroscopy: application to interleukin 1.beta. Biochemistry. 28 (15), 6150-6156 (1989).
- Balasubramaniam, D., Komives, E. A. Hydrogen-exchange mass spectrometry for the study of intrinsic disorder in proteins. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1834 (6), 1202-1209 (2013).
- Goswami, D., et al. Time window expansion for HDX analysis of an intrinsically disordered protein. Journal of the American Society for Mass Spectrometry. 24 (10), 1584-1592 (2013).
- Keppel, T. R., Howard, B. A., Weis, D. D. Mapping Unstructured Regions and Synergistic Folding in Intrinsically Disordered Proteins with Amide H/D Exchange Mass Spectrometry. Biochemistry. 50 (40), 8722-8732 (2011).
- Mitchell, J. L., et al. Functional Characterization and Conformational Analysis of the Herpesvirus saimiri Tip-C484 Protein. Journal of Molecular Biology. 366 (4), 1282-1293 (2007).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Hydrogen/deuterium exchange mass spectrometry with top-down electron capture dissociation for characterizing structural transitions of a 17 kDa protein. Journal of the American Chemical Society. 131 (35), 12801-12808 (2009).
- Habibi, Y., Uggowitzer, K. A., Issak, H., Thibodeaux, C. J. Insights into the Dynamic Structural Properties of a Lanthipeptide Synthetase using Hydrogen-Deuterium Exchange Mass Spectrometry. Journal of the American Chemical Society. 141 (37), 14661-14672 (2019).
- Hebling, C. M., et al. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Analytical Chemistry. 82 (13), 5415-5419 (2010).
- Duc, N. M., et al. Effective application of bicelles for conformational analysis of G protein-coupled receptors by hydrogen/deuterium exchange mass spectrometry. Journal of the American Society for Mass Spectrometry. 26 (5), 808-817 (2015).
- Canul-Tec, J. C., et al. Structure and allosteric inhibition of excitatory amino acid transporter 1. Nature. 544 (7651), 446-451 (2017).
- Reading, E., et al. Interrogating Membrane Protein Conformational Dynamics within Native Lipid Compositions. Angewandte Chemie International Edition. 56 (49), 15654-15657 (2017).
- Hentze, N., Mayer, M. P. Analyzing protein dynamics using hydrogen exchange mass spectrometry. Journal of Visualized Experiments. (81), e50839 (2013).
- Lento, C., et al. Time-resolved ElectroSpray Ionization Hydrogen-deuterium Exchange Mass Spectrometry for Studying Protein Structure and Dynamics. Journal of Visualized Experiments. (122), e55464 (2017).
- Schowen, K. B., Schowen, R. L. Solvent isotope effects on enzyme systems. Methods in Enzymology. 87, 551-606 (1982).
- Lau, A. M. C., Ahdash, Z., Martens, C., Politis, A. Deuteros: software for rapid analysis and visualization of data from differential hydrogen deuterium exchange-mass spectrometry. Bioinformatics. 35 (7), 3171-3173 (2019).
- Houde, D., Berkowitz, S. A., Engen, J. R. The utility of hydrogen/deuterium exchange mass spectrometry in biopharmaceutical comparability studies. Journal of Pharmaceutical Sciences. 100 (6), 2071 (2011).
- Burkitt, W., O'Connor, G. Assessment of the repeatability and reproducibility of hydrogen/deuterium exchange mass spectrometry measurements. Rapid Communications in Mass Spectrometry. 22 (23), 3893-3901 (2008).
- Rob, T., Gill, P. K., Golemi-Kotra, D., Wilson, D. J. An electrospray ms-coupled microfluidic device for sub-second hydrogen/deuterium exchange pulse-labelling reveals allosteric effects in enzyme inhibition. Lab on a Chip. 13, 2528-2532 (2013).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved