Method Article
氢-铀交换质谱仪(HDX-MS)平台,用于研究肽生物合成酶
摘要
在肽天然产品的生物合成过程中,兰西肽合成器催化多步反应。在这里,我们描述了一个连续的,自下而上的,氢-钚交换质谱仪(HDX-MS)的工作流程,可用于研究兰西肽合成器的构象动力学,以及肽天然产品生物合成中涉及的其他类似酶。
摘要
氢-铀交换质谱仪(HDX-MS)是酶构象变化和酶基质相互作用生物物理特征的有力方法。HDX-MS 的诸多优点中,仅消耗少量材料,无需酶/基质标签即可在近原生条件下进行,并且可以提供酶构象动力学空间解决信息−甚至用于大型酶和多蛋白复合物。该方法由将兴趣酶稀释到 D2O 中准备的缓冲器中启动。这触发了肽键(N-H)与铀(N-D)的蛋白交换。在所需的交换时间点,反应液被淬火,酶被蛋白酶分解成肽,肽通过超性能液相色谱 (UPLC) 分离,并且每个肽的质量变化(由于氢气交换为铀)由 MS 记录。每种肽吸收的钚量在很大程度上取决于该肽的局部氢结合环境。肽存在于酶交换的非常活跃的区域非常快,而从有序区域提取的肽的交换速度要慢得多。通过这种方式,HDX 速率报告了局部酶构象动力学。然后,在不同配体存在的情况下,对脱脂吸收水平进行扰动,可用于绘制配体绑定位点的地图,识别同位素网络,并了解构象动力学在酶功能中的作用。在这里,我们说明了我们如何使用HDX-MS来更好地了解一种叫做兰西肽的肽天然产品的生物合成。Lanthipeptides 是基因编码肽,通过大型、多功能、符合性动态酶进行转化后进行修饰,这些酶很难用传统的结构生物学方法进行研究。HDX-MS 为研究这些类型的酶的机械特性提供了一个理想且适应性的平台。
引言
蛋白质是结构动力学分子,在时间尺度上采样不同的构象,从女性到第二级的键振动到整个蛋白质域的重新排列,这些排列可以在多秒1内发生。这些构象波动通常是酶/蛋白质功能的关键方面。例如,由配体结合引起的构象变化通常对于调节酶功能至关重要,无论是组织催化所需的活动站点残留物、在顺序动能机制中定义基板结合位点、保护反应中间体免受环境影响,还是通过全石器网络调节酶功能。最近的研究还表明,在整个进化过程中可以保存符合性动力学,而对保存分子运动的扰动可以与基质特异性的变化和新酶功能2、3的出现相关。
近年来,氢-铀交换质谱仪(HDX-MS)迅速成为探索蛋白质构象景观如何对配体结合或突变4、5、6、7等扰动做出反应的有力技术。在典型的HDX-MS实验(图1)中,一种感兴趣的蛋白质被放入D2O制备的缓冲器中,这触发了可溶剂交换质子与 deuteria 的替代。肽债券的交换率在很大程度上取决于pH度、局部氨基酸序列和8号辣椒的局部结构环境。从事氢粘结相互作用的中位(如存在于α螺旋和β片中)的交换速度比暴露于散装溶剂的蛋白质非结构化区域中的中位要慢。因此,铀吸收的程度反映了酶的结构。符合动态的酶,或在配体结合后进行结构转换的酶,将有望产生可测量的HDX响应。
结构化中间体缓慢汇率的机械基础见图25、8、9。为了进行HDX,结构区域必须首先对展开的构象进行瞬时采样,以便通过特定的酸/碱基化学机制促进HDX交换的溶剂分子能够进入可交换的中间体。最终,化学汇率(k化学)的相对幅度和折叠和重折叠率(k打开和k关闭)决定了实验5、8中测量的HDX汇率。从这个简单的动能模型中,很明显,铀吸收的程度将反映潜在的构象动力学(如k开和k关闭的定义)。大多数HDX-MS实验都是在自下而上的工作流程中进行的,在交流反应之后,兴趣蛋白被消化成肽,每个肽的吸乳量被测量为质量7的增加。通过这种方式,HDX-MS 允许在肽的局部空间尺度上映射酶构象动力学的扰动,使研究人员能够评估扰动如何改变感兴趣的酶不同区域的动力学。
HDX-MS方法用于阐明蛋白质结构动力学的优点是多种多方面的。首先,该方法可以在具有第四纪结构10的系统中用少量的原生蛋白质或蛋白质复合物进行。只要自下而上的HDX-MS工作流程提供足够数量的自信识别肽,涵盖感兴趣的蛋白质序列,就根本不需要在检测中使用的酶制剂进行高度纯化。此外,HDX-MS可以提供近原生条件下的构象动力学信息,而无需像单分子荧光研究13中那样使用部位特异性蛋白质标签,并且对可以研究的蛋白质或蛋白质复合物没有大小限制(这使得核磁共振[NMR]光谱学等方法具有挑战性)7,14。最后,可以采用时间解决的HDX-MS方法来研究内在紊乱的蛋白质,这些蛋白质很难用X射线晶体学15、16、17、18来研究。HDX-MS 的主要限制是数据结构分辨率低。HDX-MS 数据可用于指出构象动力学的变化位置,并揭示耦合的构象变化,但它们通常不会提供对驱动观测变化的精确分子机制的深入了解。电子捕获分离方法与蛋白质HDX-MS数据相结合的最新进展表明,将交换点绘制为单一氨基酸残留物19有希望,但还需要进行后续生化和结构研究,以便为HDX-MS数据转发的结构模型提供清晰度。
下面,提出了一个详细的协议,为开发HDX-MS检测是20。下面介绍的样品制备协议通常应适用于在水性缓冲中表现出良好溶解性的任何蛋白质。更专业的样品制备方法和HDX-MS工作流程可用于蛋白质比需要检测在洗涤剂或磷脂的存在21,22,23,24。HDX-MS 数据收集的仪器设置被描述为高分辨率四倍飞行时间质谱仪与液相色谱系统相结合。可以在一些市售的液相色谱-质谱 (LC-MS) 系统中的任何一个系统上收集具有类似复杂性和分辨率的数据。还提供了使用市售软件包进行数据处理的关键方面。此外,我们亦提供与更广泛的HDX-MS社区12的建议相一致的数据收集及分析指引。所述协议用于研究HalM2的动态结构特性,HalM2是一种促进抗菌肽天然产品20的多步成熟。我们说明了如何使用 HDX-MS 来揭示基板绑定站点和未采用以前特征的异体特性。近年来,关于蛋白质HDX-MS的其他几项协议已经发表25,26。结合目前的工作,这些早期的贡献应该为读者提供一些实验设计的灵活性。
研究方案
1. 准备脱毒试剂和酶库存解决方案
- 将 HDX 反应所需的试剂(包括任何缓冲器、盐、基板、配体等)准备为 D2O 中的 100 −200x 浓缩库存解决方案(99.9% 原子分数 D)。准备至少 50 mL 的缓冲库存解决方案。
注:对于HalM2的描述, 准备了以下解决方案: 500 mM MgCl2,100 mM 三 (2- 卡盒乙基) 磷酸 (TCEP), 750 mM ATP (在 HEPES 缓冲区), 800 m HEPES pD 7.1, 500μM 哈拉2 和 500 m M AMPPNP. - 冻结和溶解库存解决方案的干燥。
- 在 D2O 中重新溶解,并重复至少一次的同化周期,以尽可能多地用脱硫剂替换尽可能多的可交换质子。
- 将已除名的 HEPES 缓冲库存的 pD 调整为具有集中 NAOD/DCl 的预期值,请记住以下关系27:
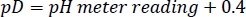
注:中间硬盘速率在很大程度上取决于解决方案(pL = pH或pD)5的pL。需要以相同的方式准备、存储和使用不同批次的缓冲库存解决方案,以避免实验之间出现轻微的 pL 漂移。 - 计算 300 μL HDX 测定所需的每个试剂的数量,并在 -80 °C 下作为一次性引用存储。
- 使用离心滤清器(材料表)或等效设备,在催化酶存储缓冲器中准备浓缩酶库存溶液(约 100 −200 μM)。
注:确切的缓冲器和离心过滤器分子重量的截止取决于感兴趣的蛋白质/酶。HalM2 储存在 50 mM HEPES、pH 7.5、100 mM KCl 和 10% 甘油中。10 kDa 过滤器用于制备浓缩酶。 - 将酶加入一次性使用部分,并储存在-80°C。
注意:这可能是一个停止点。第 1 节中描述的所有库存解决方案均可在 HDX 反应之前准备。如果存储在 -80 °C,大多数酶/脱酸库存解决方案将稳定数月。
2. HDX 淬力音量校准
- 使用第 1 节中准备的脱脂试剂和浓缩酶库存,在 D2O 中准备 300 μL HDX 反应。
- 使用1−5微米的最终酶浓度。
- 使用至少 50− 100 mM 的最终脱水 HEPES 缓冲浓度。
- 确保其他组件的浓度足以保持所需的酶活性/功能。
- 准备 1 升 HDX 淬斗溶液(100 m M 磷酸盐,0.8 M 瓜尼丁-HCl,pH 1.9)。冻结和存储在 50 mL 部分(长期库存)和 1 mL 部分(一次性报价)。
注:淬头缓冲器的确切组成将取决于自下而上的 HDX-MS 工作流(步骤 3.3.3)的蛋白酶。此处给出的淬热缓冲器与 pepsin 兼容,肽是 HDX-MS 最常用的蛋白酶。如果使用其他蛋白酶,请与蛋白酶供应商核实,以确保缓冲区兼容性。 - 校准淬火缓冲器的体积,将淬火 HDX 反应混合物的最终 pL 调整为 2.3 的 pH 表读数值。
注:肽在N-H键中的溶剂H/D汇率是一个pH依赖的过程,同时受酸和碱基催化。最低汇率为 pH 2.5 值(50:50H2 O:D 2 O 混合物的 pH 仪表读数 =2.3)。因此,接近 2.5 的最终 pL 值将最大限度地减少自下而上的 LC-MS 分析过程中发生的氢背交换,从而保留肽中的 deuterium 标签。- 将步骤 2.1 中的 HDX 反应混合物的 50 μL 与 50 μL 的淬火缓冲液混合,用微尖电极测量淬火混合物的 pL。
- 根据需要增加淬当解决方案的体积,将最终 pH 值读数调整为 2.3。
- 确定适当的淬火量后,使用 HDX 反应(步骤 2.1)中的新鲜 50μL aliquot 多次重复淬火过程,以确保在添加固定数量的淬火缓冲后实现一致的最终 pL。
3. 编制参考样本并优化自下而上的 LC-MS 工作流程
- 在 0.5 mL 管中为感兴趣的三位一体蛋白质准备未去位的参考样本。确保最终反应混合物条件与正宗 HDX 反应(步骤 2.1)中使用的条件相同,但响应是在 H2O 中使用也用 H2O 制备的试剂库存解决方案准备的。
- 通过添加适当的淬头缓冲器以将最终 pH 调整为 2.5,将样品按步骤 2.3 进行淬名。闪光将样品冻结在液氮中,储存在-80°C,直到准备好分析。
- 使用自下而上的 LC-MS 工作流分析蛋白酶参考样本。
注意:在执行这些步骤之前,应正确校准用于数据采集的 LC-MS 系统,并准备使用。必须严格控制自下而上 LC-MS 工作流程中所有步骤的时间和温度,以最大限度地减少样品之间背部交换的差异。使用本协议中使用的MS仪器(材料表 和 支持信息),大部分步骤可以通过仪器软件进行控制。为了确保精确复制的收集,建议尽可能多地自动实现工作流程中的步骤自动化。- 从冰柜中取出单个酶参考样品(按步骤 3.2 制备),在 37 °C 下解冻,在水浴中解冻 1 分钟。
- 在从冰柜中取出样品并解冻后整整 2 分钟,将淬火参考样品的 40 μL 部分注入超性能液相色谱 (UPLC) 列 (2.1 x 30 mm、300+ 、 5 μM),其中含有含有肽(酸稳定蛋白酶)的静止相位。
- 在 15 °C 时以 100μL/min 的流速消化样品 3 分钟,使用 H2O 中的 0.1% 的甲酸(pH = 2.5) 作为溶剂。
- 收集化粪肽,因为它们从肽列中流出到 C18 陷阱柱上,保持在 0.4 °C,以最大限度地减少背部交换。
- 将脱盐的化粪肽从陷阱柱传递到 C18 分析柱(1 mm x 100 mm、1.7 μM、130+)保持并操作在 0.4 °C 以分离化粪肽。
注:用于 HDX-MS 数据采集的某些 LC-MS 系统可以自动执行步骤 3.3.3−3.3.5。或者,这些步骤可以独立执行,请记住,需要仔细控制每个步骤的时间和温度,以实现持续的低背交换。 - 用丙酮三甲酸/水/0.1%的丙酸溶剂系统将C18列进行优雅。优化 LC 梯度以获得感兴趣的蛋白质,以最大限度地分离和保存败血肽中的 deuterium 标签。
注:梯度弹性详细信息在 支持信息中提供。 - 将消化液受制于电溅电传 (ESI) 质谱。
注: 支持信息 中提供的源条件将为大多数消化肽提供足够的电流。- 在MS仪器中电离后,使用氮作为缓冲气体进行气相离子移动分离,以提高该方法的峰值容量。
- 离子移动分离后,将消化肽前体离子受制于 MSE 工作流,该工作流涉及低碰撞能量 (4 V) 和高碰撞能量 (21−40 V) 的交替循环。
注:交替的低碰撞和高碰撞能量系统允许与 MSMS 数据(高碰撞能量)同时收集 MS 数据(低碰撞能量)。这反过来又允许前体离子与各自的碎片离子的时间相关性。这种相关性对于第 4 节中描述的自信肽识别至关重要。 - 使用具有至少 20,000 解析能力的质量分析仪检测肽前体和碎片离子。
- 在数据采集的同时,获取 [Glu-1] -纤维肽 B (GluFib) 外部标准的 MS 数据。
注:步骤 3.3.7 中描述的 MS 工作流称为 MSE协议。支持信息中提供了适合自下而上的 HDX-MS 的 MSE协议的完整工具设置。
- 评估 LC-MS 数据的质量。
注:使用上述协议和支持信息中提供的仪器设置,参考样本应产生总离子色度图,最大信号强度约为1×108。在3−9分钟(图3A−C)之间应该有很多怀疑论者肽。 - 注入 40 μL 的空白样品(水中 0.1% 的丙酸),以清洁肽和分析 C18 列。
注:一般来说,2−3个空白应该足够了。 - 每个三方参考蛋白样本重复步骤 3.3.1−3.3.9。
4. 处理参考数据并定义肽列表
- 使用蛋白质组学软件(材料表)分析原始 MSE数据(步骤 3.3.7)。使用蛋白质组学软件,导航到库|蛋白质序列数据库通过导入感兴趣的蛋白质的氨基酸序列来定义蛋白质数据库。
注:此步骤的目标是搜索从兴趣蛋白中提取的怀疑肽的参考 MS 数据,并使用 MSMS 数据(与 MS 数据同时获取)来验证任何假定肽识别。 - 给感兴趣的蛋白质序列起个名字。导入蛋白质序列(以 FASTA 格式)。该软件将对数据库蛋白质进行硅化消化,以生成用于搜索LC-MS数据的肽列表。
- 定义处理参数(位于 库 菜单下)。选择 电子喷雾 MSE 作为数据采集类型。在充电 2 字段 的锁定质量 中,输入 785.8426, 用于 [Glu-1] -纤维肽 B (GluFib) 的 2+ 离子的 m/z,然后单击 完成。
- 定义 工作流 参数(位于 库 菜单下方)。
- 选择 电子喷雾 MSE 进行搜索类型。在 工作流程 下| 数据库搜索查询 标题,选择数据 库 字段中第 4.2 步创建的数据库蛋白质。
- 将主文摘试剂更改为非特定,并通过在单击卡巴米多姆甲基 C时按住Ctrl按钮清除固定修饰剂试剂字段。
- 通过导航到选项来指定输出目录|自动化设置|身份 E.检查Apex 3D 和肽 3D 输出和离子会计输出的框,并指定所需的目录。
- 处理参考样本数据。
- 在蛋白质组学平台工作区的左侧工具栏上,通过右键单击 Microtiter 板创建新板。突出显示微油板中的三口井(第 3 节收集的每个参考样本一口)。左键单击一口井,按住并拖至三口井。
- 右键单击并选择 添加原始数据。在显示的窗口中,导航到包含第 3 节中的三个参考文件的目录,并同时选择它们。
- 单击 下一步 ,然后选择步骤 4.3 中定义的处理参数。单击 下一步 ,然后选择步骤 4.4 中定义的工作流参数。然后点击 完成。
- 一旦将原始数据、处理参数和工作流程参数分配给板上的每口井,油井将呈蓝色。选择油井,右键单击并选择 处理最新的原始数据。单击窗口的右下角以跟踪数据的处理。消息 出现后,无需运行任何工作 ,处理将完全完成。
- 数据处理完成后,板中的油井将变绿。右键单击油井并选择 查看工作流结果。每个参考数据文件将打开一个单独的窗口。
- 检查数据,以确保参考样本数据中的大多数 MS 信号都成功地映射到从兴趣蛋白的硅中消化预测的肽。匹配肽将在输出光谱中为蓝色(图 4)。双击 OK 过滤器 ,检查覆盖率是否大于 99%。
注:处理后,数据输出将通过步骤 4.5 中指定的目录中的文件扩展(raw_data_file_name_IA_final_peptide)自动保存。 - 将蛋白质组学软件输出导入 HDX 处理软件(材料表),以获得额外的阈值。
- 单击 HDX 处理软件窗口左角 的数据 。单击 导入 PLGS 结果 并单击添加图标。通过导航到适当的目录,从步骤 4.9 中选择已处理的数据文件。
- 单击 "下一步" 并指定以下参数:最小连续离子≥ 2、质量错误 = 5 ppm 和文件阈值 = 3。点击 完成。
- 一旦满足阈值参数,保存 HDX 项目。所有 HDX 数据都将导入此项目进行分析和显示。
注:需要用相同的LC-MS工作流程处理下一节中描述的交换样品。因此,在进行 HDX 检测(第 5 节)之前,确保样本制备(第 2 节)、自下而上的 LC-MS 工作流(第 3 节)和数据处理工作流(第 4 节)提供目标蛋白质所需的可重复性和序列覆盖范围。如果需要更改其中任何一个过程以改善覆盖范围,建议返回到步骤 2.1,在三脚架中准备新的参考样本,并重复第 2 −4 节(同时对协议进行必要的调整),以确保每个肽可以重复生成和检测。
5. 进行高清反应
- 为 HDX 反应准备工作空间。
- 将淬灭前缓冲区放入正确标记的 0.5 mL 管中。为要分析的每个时间点、每个复制品和每个生化状态准备不同的管子。使用所需的步骤 2.2 中的适当量淬热缓冲器,将 HDX 反应中 50μL 部分的最终 pH 值调整为 2.3。
- 简要离心机 0.5 mL 浴缸将所有淬热缓冲器转移到管子底部。把管子放在冰上。
- 用液氮填充小德瓦尔,并保持与工作空间相邻。
- 准备HDX反应。确保有足够的反应量来收集所需的交换时间点数(每个所需时间点一个 50μL 的单数)。收集至少 4 −5 个时间点超过 3−4 个数量级的时间尺度(例如,15 秒、60 秒、300 秒 [5 分钟]、1,800 秒 [30 分钟] 和 14,400 秒 [4 h] 的淬泄时间为大多数酶提供了足够的交换动态覆盖)。
- 在 D2O 中预混合步骤 1.1 中的所有去除组件(减去酶)。
- 要检查每个生化状态(自由酶、酶+利根德、酶+抑制剂等),请至少用三分位制备HDX反应。
- 在添加酶之前,在温度控制的水浴中将反应混合物在 25 °C 中孵化 10 分钟。
注意:酶应作为浓缩库存溶液(约 100− 200 μM,步骤 1.6)进行制备,以便最大限度地减少在 HDX 检测中添加钚。 - 将酶添加到 1−5 μM 的最终浓度后,启动定时器。使用 200 μL 移液器小心快速混合溶液,以确保酶均匀地分布在样品中。
- 在所需的交换时间点,从 HDX 反应中取出 50 个 μL 别名,并快速均匀地与 0.5 mL 管中预先引用的冰冷淬热缓冲器混合。
注意:混合体积和混合过程必须尽可能精确和可重复,以确保所有样品中快速实现所需的最终淬斗 pL。保持淬热缓冲冰冷将有助于尽量减少酶变性时的背部交换。 - 在淬火HDX样品后,立即将管子盖上盖子,并在液氮中闪光冻结。
- 继续收集时间点,直到完成所有检测,然后将样品转移到 -80 °C 冰柜进行存储。
注意:这可能是一个停止点。收集所有 HDX 时间点后,样品可存储在 -80 °C,直到准备好进行 LC-MS 分析。理想情况下,应在同一天对所有生化感兴趣的状态执行三倍HDX反应。至少,所有针对特定生化状态的复制 HDX 反应都应在同一天并行运行。
- 在收集所有淬火的 HDX 时间点后,将样品受受于步骤 3.3 中描述的优化自下而上的 LC-MS 工作流。以随机顺序注入 HDX 样品,样品之间有适当数量的空白,以确保任何肽的结运量都极小。
注:HDX 数据无需在 MSE 模式下收集。因此,高碰撞能量段(步骤 3.3.7.2)应从 MS 值周期中删除。这应该是对步骤 3.3 中描述的工作流程的唯一更改。 - 评估正在收集的 HDX 数据的质量。
- 确保未去除的参考样本的总离子色谱图中存在的色谱峰值同时出现在被除名的样品中(如图 3D−F)。
- 确保从参考和被除名的样品中总结出特定时间间隔的质谱显示脱硫的证据(即,在脱硫样品中将单个肽的同位素包络转移到更高的 m/z 值 [图 6])。
6. 处理高清数据
- 通过单击数据|,将 HDX 数据导入第 4.11 步创建 的 HDX 项目顶部工具栏中的 MS文件 。
- 根据需要单击 "新状态 "和 "新暴露", 以定义与分析相关的生化状态(如自由酶、酶 + 配体等)和钚暴露时间。
- 单击 "新原始" 以选择要分析的 HDX 数据文件。将适当的交换时间和生化状态分配给导入的每个原始数据文件。
注:数据可以分批导入和处理,也可以同时全部输入和处理。向项目添加数据不会撤消之前在该项目中执行的任何分析。
- 添加数据文件后,单击 "完成 "以开始数据处理。短暂延迟后,软件会询问用户是否希望在继续之前保存数据。单击 "是"。
注:初始处理可能需要长达几个小时,具体取决于分析样本的数量、最终肽列表中有多少肽(步骤 4.10)、色谱窗口的大小以及光谱采集频率。 - 如果需要,更改 配置 菜单中的处理参数以更改离子搜索参数。确保对给定项目中的所有数据使用相同的离子搜索参数。
7. HDX 数据的分析和可视化
注:完成原始数据的初始处理(第 6.2 步)后,HDX 处理软件将在所分析的每个原始数据文件中从肽列表(以步骤 4.10 生成)中找到肽。一旦列表中肽的同位素分布位于原始数据文件中,HDX 处理软件将用"棒"表示每个同位素(如 图 6C−E)。然后,用于计算特定肽棒的相对强化度,以计算相对于参考光谱的 deuterium 吸收量。虽然 HDX 处理软件在正确分配"棒"到大多数肽方面做得令人钦佩,但仍需要对 deuterium 吸收值进行大量人工策展。
- 导出两个生化状态之间的 HDX 差异数据,用于映射感兴趣的蛋白质的结构模型。
- 显示覆盖图中的兴趣差异。右键单击覆盖映射,将差异数据导出到.csv文件。通过导航到数据|导出状态 数据 (以.csv格式)主要工具栏中的 出口状态数据 。
注: 在支持信息中提供了差额数据和国家数据文件的适当格式。 - 将差异数据、状态数据和感兴趣的蛋白质的 pdb 文件导入 Deuteros28中。选择 99% 的置信区间,选择 启用总和,并处理数据。
注:必须安装在PC上才能运行德特罗斯。Deuteros 将使用数据集中的复制测量来计算每个肽的吸收数据的标准偏差。此标准偏差将用于定义重大交换的置信区间,该区间将显示在绘图上。 - 根据 PyMOL 选项,选择 出口吸收 | 导出 生成Pymol脚本,使用PyMOL软件将具有显著交换差异的区域映射到感兴趣的蛋白质的pdb结构上。
注意:使用本协议中描述的工作流程,给定肽在单个时间点显著的 deuterium 吸收差异的 99% 置信区间通常为 0.3−0.5 Da。所有交换时间点所总结的差额的 99% 置信区间通常为 0.7 −1.0 Da。
- 显示覆盖图中的兴趣差异。右键单击覆盖映射,将差异数据导出到.csv文件。通过导航到数据|导出状态 数据 (以.csv格式)主要工具栏中的 出口状态数据 。
结果
有必要评估蛋白催化消化的质量以及每组样品注射工作流程的可重复性。因此,在进行HDX-MS检测之前,必须为兴趣蛋白的蛋白分析、使用反相液相色谱和气体相离子流动性分离肽以及使用MS检测肽建立有效条件。为此目的,应首先调查感兴趣的蛋白质的参考样本(在没有贫血的情况下收集)(第3节)。图3A−C中的色谱数据显示了HalM2兰西肽合成酶三个参考样本的总离子色谱图(TICs)。TIC 是质量光谱扫描中包含的所有 m/z 值中所有离子计数的总和的时间依赖变化。图3D−F显示了3至8分钟之间的TIC的更近距离视图。图 3G 中显示的质谱−I表示每个 TIC 中小时间间隔(从 5.0 到 5.1 分钟)的所有质谱的总和。应特别注意图3中的以下特征。
首先,梯度末端接近9.3分钟的大峰值表示HalM2(图3A−C)的未完全消化(因此,较大且更疏水)碎片。通过降低HalM2浓度可以提高消化效率,但这会降低肽信号强度和信号:噪声比。通过增加与 pepsin 列的接触时间(从 3 分钟起,协议步骤 3.3.3),消化还可以提高效率。然而,增加接触时间将导致更多的回交换。最终,这些参数需要平衡感兴趣的蛋白质,以提供所需的序列覆盖,信号强度和静留。其次,TIC 轮廓的形状和强度应相似(如 图 3D−F)。这表明HalM2的蛋白解消化是可重复的,在所有三个参考样本中都有类似的效率。期望对交换的样品进行类似的消化,就会产生具有类似保留时间的相同组肽。第三,在给定时间间隔内的质谱也应该相似(图3G−I)。对色谱分离5.0−5.1分钟时间间隔的累计质谱进行快速的视觉比较表明,每个样本中的质量光谱信号确实非常相似,这使人们相信每个样本中都存在类似的肽,并且在C18列的类似时间进行回流。在色谱图的其他时间间隔内,应进行类似的快速目视检查。
在目视验证参考样本的LC-MS数据中的相似性后,蛋白质组学软件用于搜索MS数据,寻找从感兴趣的蛋白质的氨基酸序列中提取的肽。在定义了蛋白质序列数据库(感兴趣的蛋白质的氨基酸序列)、协议步骤 4.2 −4.4 中描述的处理和工作流程参数后,对每个单独的参考样本进行处理,以检测与从感兴趣的蛋白质中提取的预测肽相匹配的多肽。 图4中显示了未去位HalM2参考样本的蛋白质组学软件输出示例。应特别注意以下功能。首先,根据处理和工作流程参数中定义的值与感兴趣的蛋白质中的特定肽匹配的任何 MS 信号都将在底部光谱中为蓝色。如果参考样本"成功"处理,大多数信号将呈蓝色( 如图 4中的数据一样)。此外,在左上面板中,确保将"OK 过滤器"切换到绿色复选标记。完成此任务后,右上面板顶部条中的统计数据( 图 4中以蓝色突出显示)将仅反映那些提供高置信分数的肽。这些肽具有较低的ppm质量精度,多个片段离子,以及片段和母离子保留时间之间的良好相关性,被认为是自信的识别。对于显示的 HalM2 参考样本,检测到 1,421 多肽的高分,占 HalM2 序列的 99.6%。使用协议第 3 节中描述的自下而上的 LC-MS 工作流,大多数蛋白质应实现近 100% 的序列覆盖。此外,在右上面板中还列出了每个肽的其他有用统计数据,包括质量错误、前体保留时间、片段(产品离子)数量、按名称分列的片段离子完整列表以及离子移动漂移时间。这些信息对于解释结果很有用,结果应该始终集中在最自信的多肽上。
最终肽列表应通过 HDX 处理软件中的附加阈值来确定。在分析生成肽列表的参考数据(协议第 4 节)后,应将输出导入 HDX 处理软件以进行附加阈值。输出文件将存储在协议步骤 4.5 中定义的目录中,文件扩展为"..._IA_final_peptide.csv"。将输出上传到 HDX 处理软件后,肽的完整列表将显示在"肽预览"面板(图 5),肽的数量、序列覆盖和冗余将显示在窗口的右下角。应用附加阈值时,这些值将实时更改。单击 下一步后,阈值可以在指示字段中设置。最关键的过滤器是"文件阈值",它要求所有肽都位于三个参考样本中的每一个, 应设置为值 ±lt;10 ppm 的"最大 MH+ 误差(ppm)"和"最小连续产品",这要求肽在 MSE 工作流的 MSMS(即高碰撞能量)阶段(协议步骤 3.3.7.2)期间产生至少两个连续的片段离子。
HDX-MS 检测最重要的技术限制是,一旦脱脂交换样品被淬火(用质子缓冲器),就会对钚进行回交换。在工作流程的蛋白酶消化和 LC-MS 部分,背部交换继续。回换是不可避免的,但如果按照协议中描述的pH度、温度和所有淬门后步骤的时间得到严格控制,则可以保持 60%− 70% 的 deuterium 标签。因此,在检测开发的早期阶段,必须检查大多数肽是否保留了铀。通过将被除名样本的原始数据与参考样本的原始数据进行比较,可以快速实现此目的。为确保参考样本(图3)的可重复性,我们比较了交换和未除损的参考样本的 TIC,确保配置文件的形状彼此相似。此外,在两个样本中,将质谱相加于相同的色谱时间间隔。交换样品中的大多数肽在同位素分布上应表现出明显的变化,以达到更高的m/z值(如 图6)。这些数据表明,在整个酸淬灭、肽消化和LC-MS数据收集过程中,相当一部分的 deuterium 标签一直被保留着。
在验证工作流程是否提供了足够的留存后,需要量化"因此,用于交换铀样品的原始数据导入 HDX 处理软件中。HDX 处理软件将使用参考样本和最终确定的肽列表,在每个原始数据文件中定位肽列表中的肽,并将"棒"分配给每个同位素峰值。在 图 7中显示的 HalM2 的代表性数据中,成功分配的棒在所有光谱中均为蓝色。分配棒后,同位素分布的中心 m/z 值将由软件确定,并用于计算相对于未去除的参考样本的 deuterium 吸收量。应手动检查每个肽的铀交换值。在 图 7中,当前选定的肽 (HIDKLTVGL, 跨越 HalM2 残留物 110−118) 显示在主数据查看器的左面板 (图 7A)中。其他面板显示与此肽相关的 HDX 数据。单击肽列表中的任何肽都会在其他面板中填充该肽中的 HDX 数据。 图7B 显示了肽110−118的排泄物吸收图。此数据集包含四个交换时间点:0.5、5、30 和 240 分钟(以三分法收集)。为两种生化状态收集了交换数据:自由哈尔姆2酶(红色)和与AMPPNP及其基质肽HalA2(蓝色)复合的HalM2酶。请注意,在整个时间过程中,肽 110−118 的显著吸收差异以及复制测量的高精度( 图 7B中的绘图中显示错误条)。一般来说,大多数肽的吸收测量精度相似,这突显出本协议中的工作流程的可重复性。在配体结合( 图7B中的蓝色曲线)下,HalM2 110−118区域的肽键显然受到大量保护,免受铀交换的侵害,这表明110−118区域的氨基酸在配体结合后结构更加稳定。该地区随后的诱变表明,它对前体肽HalA220具有约束力。 图7 中还显示了HalM2状态(图7D)和哈尔姆2:AMPPNP:哈拉2状态(图7E)的堆叠光谱图。在这些图中,交换时间从底部增加到顶部。从目视检查可以明显看出,在较长时间点交换时肽110−118的量增加。同样明显的是,肽110−118在HalM2状态(图7D)中比在全配体状态(图7E)中吸收更多的铀。如有必要,单击 图 7D 或图 7E中的任何子图,将允许用户手动修改主数据查看器(图 7C)中的棒的分配。在坚持分配/分配后,HDX 处理软件将重新计算 deuterium 吸收值,所有绘图将实时更新。同样,单击面板 B 中的单个数据点将填充 C 面板中的数据查看器以进行手动粘合分配。
在为每个肽分配棒并交换所有生化状态的时间点后,应检查测量的吸收值的标准偏差。这是最容易执行与覆盖图和堆叠光谱图 (图 8).在覆盖图(图 8C)中,选择感兴趣的状态和交换时间,然后选择相对吸收标准偏差。覆盖图中的肽将根据测量的 deuterium 吸收值的标准偏差进行着色。通过这种方式,将很容易在视觉上识别具有同位素棒错误分配的肽。单击覆盖映射中的离群值肽(具有高标准偏差)将填充主要数据查看器和堆叠的光谱图,以及来自离群值肽的 HDX 数据。然后,可以根据需要修改每个时间点和充电状态的棒分配,以纠正测量的吸收值。同样,HDX 处理软件将随着棒分配的更改而实时更新所有数据显示。
在完全策划了 HDX 数据集之后,在数据解释之前,需要考虑对每种肽的 deuterium 吸收差异测量的重要性。使用 Engen 和同事29描述的方法,我们估计使用上述协议 20 中描述的工作流程,HalM2 蛋白质的平均吸收值为 0.1 ±0.1Da 。此值与其他人报告的类似自下而上、连续交换 HDX 工作流29、30的错误一致。最近,Politis 和同事开发了 Deuteros,这是一种有用的开源工具(在 MATLAB 中实施),用于快速确定 HDX 数据集28中的显著吸收差异。Deuteros 的代表输入文件("State_Data_for_Deuteros"和"Difference_Data_for_Deuteros")包含在支持信息中。如果直接从本协议(材料表)中描述的 HDX 处理软件导出,则差额和国家数据文件将具有适当的格式供 Deuteros 进行文件读取。如果在不同的 LC-MS 系统上生成 HDX 数据,则必须手动构建类似于支持信息中提供的数据文件。
二重奏工作空间显示在图 9 中。一旦数据导入 Deuteros,用户将选择所需的置信度限制(建议为 98%),然后单击"导入和计算"。对于扁平化数据地图,请选择数据类型和颜色比例的绝对覆盖范围。单击绘图。对于树林绘图,选择二进制滤镜作为数据类型,所需的置信度滤清器并启用总和。点击情节后,Deuteros 将在每个交换时间点绘制所有肽,作为它们在氨基酸序列中的位置和其 deuterium 吸收值的函数。根据计算的差额,具有显著交换作用的肽将分别被涂成红色或蓝色,这取决于肽是否分别占用了或多或少的肽。每个绘图中报告的意义值(图 9中绘制的数据中从 0.39 到 0.72 Da 不等)是根据从数据集中存在的复制测量结果确定的每种肽的吸收差异的标准偏差计算的。在每个绘图中,置信区间显示为破折号条。如果启用,"Sum"只需添加每个肽在每个时间点测量的吸收差异。最后,为了可视化兴趣蛋白的三维结构的吸收数据,选择在PyMOL选项下的出口吸收,然后出口。Deuteros 将生成一个 PyMOL 脚本,该脚本可以拖入 PyMOL 工作区,其中包含感兴趣的蛋白质的开放 pdb 文件。
本协议中提出的HDX-MS工作流程用于描述一种酶(HalM2)的生化特性,该酶将一系列转化后修饰催化到基因编码肽(HalA2)20上。在 图 10中,显示了具有代表性的 HDX-MS 结果,显示 HalA2 前体肽与 HalM2:AMP-PNP 复合物的结合。面板 A 显示 HalM2 覆盖图,其中颜色表示 [哈尔姆2:AMPPNP] 生化状态与 [哈尔姆2:AMPPNP:HalA2] 状态之间的相对吸收差异。Deuteros 用于将这些吸收差异映射到 HalM2 酶的同源模型(图10B,C)上。彩色红色肽表示对 HalA2 结合的二氧化硫吸收量减少,表明 HalM2 的这些区域可能直接参与前体肽的结合。为了研究这一假设,对I-III区域进行了变异,并研究了变异酶的动能特性。第一和第三区域的突变都导致 HalA2 肽结合亲和力出现重大扰动,表明 HalM2 的这些区域要么直接与肽基质相互作用,要么需要形成使 HalA2 绑定的结构。相比之下,第二区域的突变对HalA2结合亲和力没有影响,但这种突变几乎没有催化活性。这一发现的一个解释是,在HalA2结合上观察到的区域II的组织触发了激活酶的构象变化。应当指出,在这项研究之前,没有关于HalM2-HalA2绑定模式或系统中催化相关构象变化的信息,主要是因为HalM2和HalA2的体积大且性质灵活,排除了结构研究。因此,这些关于HalM2兰西肽合成酶的代表性数据说明了如何利用HDX-MS快速定位结构动态酶系统的功能相关区域−即使没有高分辨率结构数据。
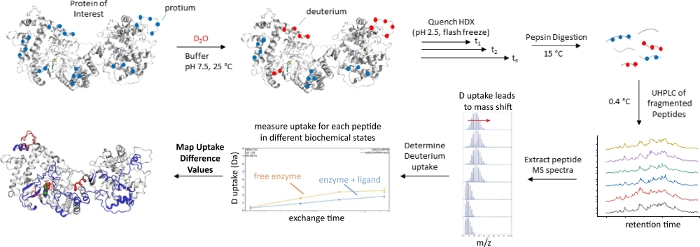
图1:持续交换,自下而上的HDX-MS工作流程。有关详细信息,请参阅文本。 请单击此处查看此图的更大版本。

图2:HDX汇率取决于蛋白质构象动力学(k开和k关闭)和N-H键在N-D键(k化学)的蛋白质骨干中的化学交换的pH依赖率。请单击此处查看此图的更大版本。

图3:三脚架HalM2参考样本的代表性LC-MS数据。每个面板右上角的数字表示总离子计数。每行显示不同参考样本的数据。第一列(A−C)显示总离子色谱 (TICs)。9.3 分钟的大峰值代表大的未消化肽。中间列(D−F)显示 3 到 8 分钟之间对 TIC 的更近距离视图。请注意配置文件形状的良好一致性,这表示整个色谱中每个参考样本中肽信号的类似潜在混合物。第三列(G−I)显示通过汇总色谱运行时间点 5.0 至 5.1 分钟之间记录的所有质谱生成的每个参考样本的质谱。对这些数据的目视检查表明,每个样本中检测到许多相同的肽。 请单击此处查看此图的更大版本。
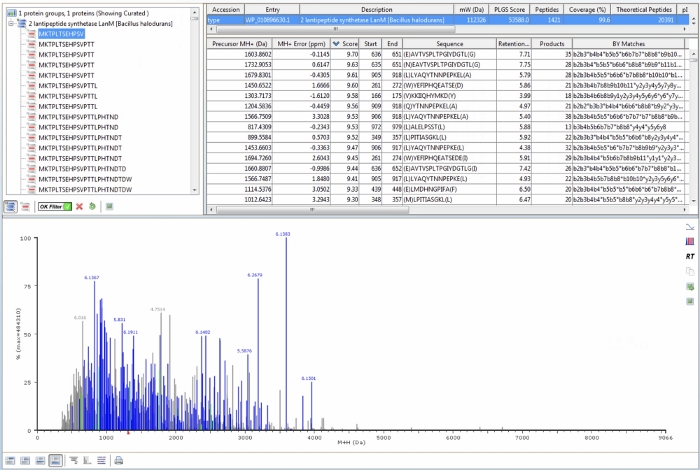
图4:HalM2参考样本的代表性蛋白质组学软件输出。左上面板显示 MS 数据中检测到的 HalM2 衍生肽的完整列表。请注意,"确定筛选器"显示绿色复选标记。这会根据置信评分过滤数据,并去除低置信度肽识别。右面板的顶部条(以蓝色突出显示)显示高分的多肽集的累积统计数据。最重要的统计数据是检测到的肽数量(在此例中为 1,421)和序列覆盖率(在此例中为 99.6%)。每个肽的其他统计数据也显示在右上面板中。此数据表中的每个列都是可排序的。底部面板显示与预测的 HalM2 衍生化粪肽匹配的所有 MS 信号。请注意,大多数 MS 信号被分配,并且是蓝色的。 请单击此处查看此图的更大版本。

图 5:HDX 处理软件中的其他阈值。三个 HalM2 参考样本中每个样本的蛋白质组学软件输出导入 HDX 处理软件(左面板)中。导入数据后,执行额外的阈值(右面板)。"最小连续产品"字段是指肽中相邻肽键裂开产生的碎片离子。"最小 MH+ 错误"参数允许用户定义可接受的质量精度,"文件阈值"允许用户仅将肽限制在所有三个参考文件中检测到的肽。 请单击此处查看此图的更大版本。

图6:HalM2 参考样本(红色)和在 D2O 缓冲器中孵育的 HalM2 样本的代表数据 5 分钟(绿色)。这两个样本都受协议第 3 节中描述的自下而上的 LC-MS 工作流程的影响。左列显示在 6.0 −6.1 分钟时间窗口上总结的质谱。三个右列显示相同质谱的更近视图,在大多数肽信号中,脱硫样品(绿色、顶部)中向更高 m/z 值的转换是显而易见的。 请单击此处查看此图的更大版本。

图 7:HDX 处理软件中工作空间的屏幕截图。(A) 从用蛋白质组学软件(图3)分析HalM2参考样本以及随后在HDX处理软件(图4)中阈值中获得的HalM2衍生肽列表。当前选定的肽(HIDKLTVGL,跨越 HalM2 残留物 110−118)以蓝色突出显示。(B) 两种生化感兴趣状态的铀吸收曲线(作为交换时间的函数):自由哈尔姆2酶和与AMPPNP和前体兰西肽(HalA2)结合的HalM2酶。(C) 主动选取样本的质量谱(在此例中,未减记的 HalM2 参考样本之一)。如果 HDX 处理软件在初始数据处理过程中没有正确分配蓝棒,则可以根据需要手动分配/分配。(D, E)哈尔姆2状态(D)和哈尔姆 2:AMPPNP:哈拉2 状态(E)的堆叠光谱图。久间依赖时间的增加很容易看出,两种生化状态之间的吸收差异也显而易见。 请单击此处查看此图的更大版本。

图8:在测量的吸收值中最大限度地减少标准偏差。HDX 处理软件 (面板 C) 中的覆盖图提供了一种方便的手段,可快速识别在测量的 deuterium 吸收值中具有较大标准偏差的肽。在这个假设的情况下,HalM2衍生肽的一些同位素峰值(蓝色棒)跨越残留物110−118未分配5分钟交换时间点(面板B中的灰色棒)。这导致在 5 分钟交换时间点测量的 deuterium 吸收值出现大标准偏差。从覆盖图 (面板 C) 中的 110 −118 肽的蓝色以及吸收图(面板 A) 中的数据点散射和大误差栏来看,大标准偏差显而易见。 请单击此处查看此图的更大版本。

图9:德特罗斯工作空间。在此示例中,通过从 HalM2:AMPPNP 状态中减去 HalM2:AMPPP:HalA2 状态的精华吸收值,在 HDX 处理软件中计算吸收差值。比较的目的是可视化肽(HalA2)结合如何改变HalM2:AMPPNP复合物的结构动力学。数据集包含 4 个交换时间点(0.5、5、30 和 240 分钟)。伍兹的地块说明了每个交换时间点每个肽的吸收差异。伍兹地块中的彩色肽表示多肽具有显著的吸收差异,这由数据集中存在的复制测量标准偏差和在 Deuteros 中选择的用户定义的置信度限制所定义。 请单击此处查看此图的更大版本。
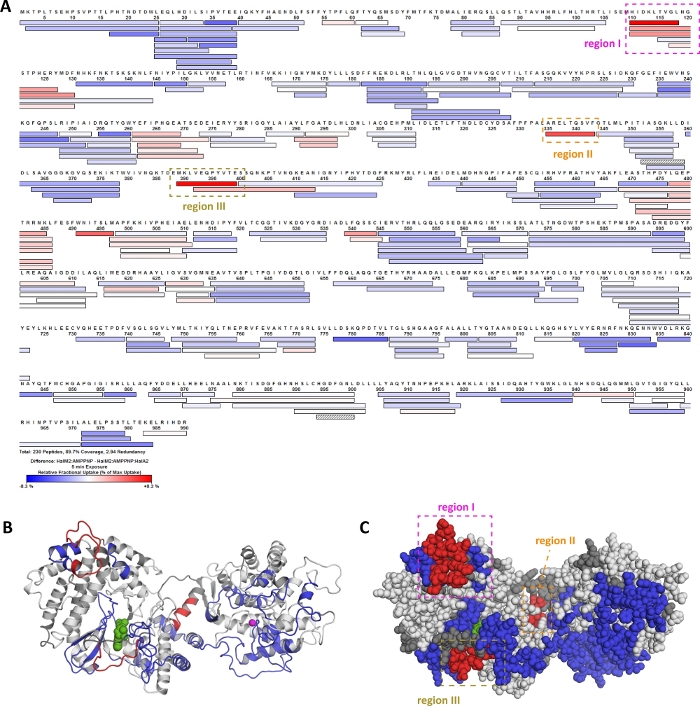
图10:HDX-MS数据指南在兰西肽合成酶HalM2中肽结合和异体激活的功能分析。调查了HalA2前体肽与HalM2兰西肽合成酶结合后,宫内吸收变化。绘制了 5 分钟交换反应后每个肽的吸收差异(A)。此绘图生成于 HDX 处理软件中。在HalA2肽的存在下,红色和蓝色的肽分别受到越来越少和更多的吸收。这些 HDX"热点"映射到 HalM2 同源模型(B,C)上。在德乌特罗斯确定了发生重大交换差异的肽的鉴定。用于将差异值映射到同源模型的 PyMOL 脚本生成于 Deuteros 中。 请单击此处查看此图的更大版本。
讨论
本协议中介绍的 HDX-MS 工作流程为绘制蛋白质结构动态元素的空间分布图和调查这些动力学如何因扰动(脂结合、酶突变等)而变化提供了非常强大的平台。HDX-MS 与其他通常用于研究构象动力学的结构生物学方法相比具有几个明显的优势。最值得注意的是,只需要少量的蛋白质。使用此处描述的工作流程,1 mL 1 μM 蛋白质样本为包含 5 个交换时间点的三方 HDX 反应提供了足够的材料。此外,兴趣蛋白几乎没有大小限制,蛋白质复合物同样符合HDX-MS方法。尺寸限制仅受化粪肽在色谱仪上以及 m/z 和离子移动维度的解析范围的限制。因此,对于许多蛋白质/蛋白质复合物,HDX-MS 将提供关于蛋白质-蛋白质交互接口、配体结合位点、构象动力学和异构网络的宝贵信息。最后,HDX反应是在温和、近原生条件下进行的,无需对蛋白质进行特定部位的标记/工程,这应有助于确保内生活性得到保留。该方法的主要局限性(就它提供的关于感兴趣的蛋白质的机械信息而言)是肽水平HDX数据本质上是低空间分辨率的。因此,这些数据足以推断二次结构正在发生变化,但结构扰动的机械影响需要更高的分辨率(如NMR、低温或X射线晶体)结构、计算研究和/或生化研究来充分解释。
为了确保结果的可重复性和可靠性,应牢记议定书的几个关键方面。首先,HDX 反应期间的 deuterium 交换程度以及工作和分析期间的背部交换程度在很大程度上取决于 pH、时间和温度。因此,准备缓冲器、HDX 反应和 LC-MS 方法的程序必须尽可能系统化,以最大限度地减少数据集中这些物理参数的变化。如果可能,同一研究人员应在同一天对生化状态进行HDX反应比较,并且应用同一批溶剂连续几天收集这些样本的LC-MS数据。随着时间的推移,肽消化的效率也会降低,因此,在相对狭窄的时间窗口内进行消化和分析的样品是最佳选择−特别是如果使用肽柱来消化许多其他类型的样品。使用标准蛋白质定期检查肽柱的消化效率是好的做法。为此,可以建立一个专门的HDX处理软件项目,将新消化的样品与较旧的样品进行比较,以确保检测到具有类似强化度的目标肽。
虽然本协议中的工作流程应为许多酶/蛋白质系统提供足够的数据,但有几个潜在的优化点。首先,可以修改 HDX 反应的时间尺度,以捕获更快/更慢的动态。最值得注意的是,许多酶将包含高度动态的元素,在几秒钟内完全交换。如果这些极具活力的元素是感兴趣的,方法的预先稳定状态,连续交换HDX-MS已报告在文献31。其次,LC 方法条件可以随时更改,以更改消化效率(最终决定序列覆盖范围)和 deuterium 保留(最终确定方法的敏感性)。考虑到肽消化的持续时间和温度,较慢的流速和较高的温度有利于更彻底地消化蛋白质。如果信号强度过低,HDX检测中的蛋白质浓度也会增加,如果肽消化效率过低,蛋白质浓度也会降低。考虑到用于分离分析 C18 列上的消化肽的丙酮三聚氰化物梯度,更快的梯度将保留 deuterium 标签,但以消化反应混合物中存在的消化肽的色谱分辨率为代价。对于消化中含有较少的消化肽的较小蛋白质(+200氨基酸),更快的LC梯度可能更容易实现。对于较大的蛋白质,如HalM2(+1,000氨基酸),需要更长的梯度来解决混合物中较多肽产生的额外光谱复杂性。在后一种情况下,加入气体相离子移动分离有助于显著提高分析的峰值容量。离子移动分离的加入是以信号与噪声比略有降低为代价的。最后,应注意,HDX 样本的 MS 数据不需要在 MSE 模式下收集。MSE 值周期的 MSMS 部分(即高碰撞能量段,步骤 3.3.7.2)仅对参考样本进行定义肽列表(协议第 4 节)。因此,应以 MS 专用模式分析 HDX 样本,以增加信号:噪声比。
如果本协议中描述的工作流程运行正常,完全交换肽应显示相对的 deuterium 吸收值为 60%−70%。如果发现 deuterium 吸收率大大低于此(对于溶剂暴露的、未受保护的肽),最可能的解释是样本的 pH 在工作/分析的某些部分发生变化。在这种情况下,应使用微尖电极仔细监测 HDX 检测的 pH 和淬火反应引用。还应检查LC-MS溶剂的pH。为了尽量减少此问题的发生,强烈建议准备和存储检测所需的所有缓冲器和试剂的集中库存解决方案(如协议第 1 节所述)。
近年来,HDX-MS已成为研究蛋白质结构动力学的强大分析工具。为 HDX 实验设计和优化的商用 LC-MS 系统(如本研究中使用的系统和材料中列出的系统)的开发,以及强大的软件包,将 HDX-MS 方法扩展到许多学术和工业实验室,并将 15 年前曾经的利基技术转变为更人性化的分析平台。尽管空间分辨率有限,HDX-MS 仍对蛋白质运动提供高度定量和可重复的测量,非常适合研究难以用其他方法研究的构象动态酶。由于这些特点,HDX-MS填补了无序和动态蛋白质系统结构生物学中的重要利基,这些系统在生物学和医学的许多基本领域都具有相关性。因此,在可预见的未来,HDX-MS分析有望成为结构生物学家武器库中的一个重要工具。
披露声明
我们没有什么可透露的。
致谢
这项工作得到了加拿大自然科学和工程研究理事会、魁北克自然与技术基金会、加拿大创新基金会和麦吉尔大学创业基金的支持。
材料
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| [glu-1]-fibrinopeptide B (Glu-Fib) | BioBasic | NA | |
| 0.5 mL Amicon Ultracel 10k centrifugal filtration device (Millipore) | Milipore Sigma | UFC501096 | |
| acetonitrile | Fisher | A955-1 | |
| AMP-PNP | SIGMA | A2647-25MG | |
| ATP | SIGMA | a2383-5G | |
| D2O | ALDRICH | 435767-100G | |
| formic acid | Thermo Fisher | 28905 | |
| guanidine-HCl | VWR | 97063-764 | |
| HEPES | Fisher | BP310-1 | |
| Magnesium chloride | SiGMA-Aldrich | 63068-250G | |
| Potassium chloride | BioBasic | PB0440 | |
| potassium phosphate | BioBasic | PB0445 | |
| TCEP Hydrochloride | TRC Canada | T012500 | peptide was synthesized upon request |
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| software | |||
| Deuteros | Andy M C Lau, et al | version 1.08 | |
| DynamX | Waters | version 3.0 | |
| MassLynx | Waters | version 4.1 | |
| Protein Lynx Global Server (PLGS) | Waters | version 3.0.3 | |
| PyMOL | Schrödinger | version 2.2.2 | |
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| Instrument and equipment | |||
| ACQUITY UPLC BEH C18 analytical Column | Waters | 186002346 | |
| ACQUITY UPLC BEH C8 VanGuard Pre-column | Waters | 186003978 | |
| ACQUITY UPLC M-Class HDX System | Waters | ||
| HDX Manager | Waters | ||
| microtip pH electrode | Thermo Fisher | 13-620-291 | |
| Waters Enzymate BEH column or Pepsin solumn | Waters | 186007233 | |
| Waters Synapt G2-Si | Waters |
参考文献
- Karplus, M., McCammon, J. A. Molecular dynamics simulations of biomolecules. Nature Structural & Molecular Biology. 9 (9), 646-652 (2002).
- Campbell, E., et al. The role of protein dynamics in the evolution of new enzyme function. Nature Chemical Biology. 12 (11), 944-950 (2016).
- Tokuriki, N., Tawfik, D. S. Protein dynamism and evolvability. Science. 324 (5924), 203-207 (2009).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass Spectrometry Reviews. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chemical Society Reviews. 40 (3), 1224(2011).
- Katta, V., Chait, B. T. Conformational changes in proteins probed by hydrogen-exchange electrospray-ionization mass spectrometry. Rapid Communications in Mass Spectrometry. 5 (4), 214(1991).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Science. 2 (4), 522(1993).
- Smith, D. L., Deng, Y., Zhang, Z. Probing the non-covalent structure of proteins by amide hydrogen exchange and mass spectrometry. Journal of Mass Spectrometry. 32 (2), 135-146 (1997).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Xiao, K., et al. Revealing the architecture of protein complexes by an orthogonal approach combining HDXMS, CXMS, and disulfide trapping. Nature Protocols. 13 (6), 1403-1428 (2018).
- Smith, D. L., Deng, Y., Zhang, Z. Probing the non-covalent structure of proteins by amide hydrogen exchange and mass spectrometry. Journal of Mass Spectrometry. 32 (2), 135-146 (1997).
- Masson, G. R., et al. Recommendations for performing, interpreting and reporting hydrogen deuterium exchange mass spectrometry (HDX-MS) experiments. Nature Methods. 16 (7), 595-602 (2019).
- Liu, J., et al. An Efficient Site-Specific Method for Irreversible Covalent Labeling of Proteins with a Fluorophore. Scientific Reports. 5, 16883(2015).
- Marion, D., et al. Overcoming the overlap problem in the assignment of proton NMR spectra of larger proteins by use of three-dimensional heteronuclear proton-nitrogen-15 Hartmann-Hahn-multiple quantum coherence and nuclear Overhauser-multiple quantum coherence spectroscopy: application to interleukin 1.beta. Biochemistry. 28 (15), 6150-6156 (1989).
- Balasubramaniam, D., Komives, E. A. Hydrogen-exchange mass spectrometry for the study of intrinsic disorder in proteins. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1834 (6), 1202-1209 (2013).
- Goswami, D., et al. Time window expansion for HDX analysis of an intrinsically disordered protein. Journal of the American Society for Mass Spectrometry. 24 (10), 1584-1592 (2013).
- Keppel, T. R., Howard, B. A., Weis, D. D. Mapping Unstructured Regions and Synergistic Folding in Intrinsically Disordered Proteins with Amide H/D Exchange Mass Spectrometry. Biochemistry. 50 (40), 8722-8732 (2011).
- Mitchell, J. L., et al. Functional Characterization and Conformational Analysis of the Herpesvirus saimiri Tip-C484 Protein. Journal of Molecular Biology. 366 (4), 1282-1293 (2007).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Hydrogen/deuterium exchange mass spectrometry with top-down electron capture dissociation for characterizing structural transitions of a 17 kDa protein. Journal of the American Chemical Society. 131 (35), 12801-12808 (2009).
- Habibi, Y., Uggowitzer, K. A., Issak, H., Thibodeaux, C. J. Insights into the Dynamic Structural Properties of a Lanthipeptide Synthetase using Hydrogen-Deuterium Exchange Mass Spectrometry. Journal of the American Chemical Society. 141 (37), 14661-14672 (2019).
- Hebling, C. M., et al. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Analytical Chemistry. 82 (13), 5415-5419 (2010).
- Duc, N. M., et al. Effective application of bicelles for conformational analysis of G protein-coupled receptors by hydrogen/deuterium exchange mass spectrometry. Journal of the American Society for Mass Spectrometry. 26 (5), 808-817 (2015).
- Canul-Tec, J. C., et al. Structure and allosteric inhibition of excitatory amino acid transporter 1. Nature. 544 (7651), 446-451 (2017).
- Reading, E., et al. Interrogating Membrane Protein Conformational Dynamics within Native Lipid Compositions. Angewandte Chemie International Edition. 56 (49), 15654-15657 (2017).
- Hentze, N., Mayer, M. P. Analyzing protein dynamics using hydrogen exchange mass spectrometry. Journal of Visualized Experiments. (81), e50839(2013).
- Lento, C., et al. Time-resolved ElectroSpray Ionization Hydrogen-deuterium Exchange Mass Spectrometry for Studying Protein Structure and Dynamics. Journal of Visualized Experiments. (122), e55464(2017).
- Schowen, K. B., Schowen, R. L. Solvent isotope effects on enzyme systems. Methods in Enzymology. 87, 551-606 (1982).
- Lau, A. M. C., Ahdash, Z., Martens, C., Politis, A. Deuteros: software for rapid analysis and visualization of data from differential hydrogen deuterium exchange-mass spectrometry. Bioinformatics. 35 (7), 3171-3173 (2019).
- Houde, D., Berkowitz, S. A., Engen, J. R. The utility of hydrogen/deuterium exchange mass spectrometry in biopharmaceutical comparability studies. Journal of Pharmaceutical Sciences. 100 (6), 2071(2011).
- Burkitt, W., O'Connor, G. Assessment of the repeatability and reproducibility of hydrogen/deuterium exchange mass spectrometry measurements. Rapid Communications in Mass Spectrometry. 22 (23), 3893-3901 (2008).
- Rob, T., Gill, P. K., Golemi-Kotra, D., Wilson, D. J. An electrospray ms-coupled microfluidic device for sub-second hydrogen/deuterium exchange pulse-labelling reveals allosteric effects in enzyme inhibition. Lab on a Chip. 13, 2528-2532 (2013).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。