Method Article
MALDI-TOF質量分析およびカスタムデータベースを使用すると、ユニークな洞窟環境(カーチェナー洞窟、AZ、USA)を常在菌を特徴づけるために
要約
This work details procedures for rapid identification of bacteria using MALDI-TOF MS. The identification procedures include spectrum acquisition, database construction, and follow up analyses. Two identification methods, similarity coefficient-based and biomarker-based methods, are presented.
要約
MALDI-TOF mass spectrometry has been shown to be a rapid and reliable tool for identification of bacteria at the genus and species, and in some cases, strain levels. Commercially available and open source software tools have been developed to facilitate identification; however, no universal/standardized data analysis pipeline has been described in the literature. Here, we provide a comprehensive and detailed demonstration of bacterial identification procedures using a MALDI-TOF mass spectrometer. Mass spectra were collected from 15 diverse bacteria isolated from Kartchner Caverns, AZ, USA, and identified by 16S rDNA sequencing. Databases were constructed in BioNumerics 7.1. Follow-up analyses of mass spectra were performed, including cluster analyses, peak matching, and statistical analyses. Identification was performed using blind-coded samples randomly selected from these 15 bacteria. Two identification methods are presented: similarity coefficient-based and biomarker-based methods. Results show that both identification methods can identify the bacteria to the species level.
概要
Matrix-assisted laser desorption/ionization time-of-flight (MALDI-TOF) mass spectrometry (MS) has been shown to be a rapid and reliable tool for identification of bacteria at the genus, species, and in some cases, strain levels1-4. MALDI-TOF MS ionizes biological molecules (typically proteins) that originate from cell surfaces, intracellular membranes, and ribosomes from bacterial whole cells or protein extracts1,5. The resulting peaks form characteristic patterns or “fingerprints” of the bacteria analyzed1. Identification of bacteria is based on these mass-to-charge “fingerprints”.
Two of the most commonly used identification strategies are library-based and bioinformatics-based strategies1. Library-based approaches involve comparing the mass spectra of unknowns to previously collected mass spectra of known bacteria in databases/libraries for identification. Commercially available software, such as BioNumerics, Biotyper, and SARAMIS software packages, as well as open source software tools, such as SpectraBank6, are available to facilitate the comparison and quantification of similarity between mass spectra of unknowns and reference bacteria. Bioinformatics-based approaches usually rely on fully sequenced genomes of bacteria for identification. In contrast to library-based approaches which do not involve identification of the biological nature of particular peaks, bioinformatics-based approaches involve protein identification1.
The majority of recent MALDI fingerprint-based studies have used library-based approaches to identify bacteria1. Library-based approaches require construction of databases and comparison of the similarity between mass spectra. Studies show that many experimental procedures, such as medium3,7, cultivation time8, sample preparation method3, and matrix used9, affect the mass spectra obtained. Furthermore, some closely-related species and strains generate spectra with only subtle differences. Thus, library-based approaches require rigorously standardized procedures to generate highly reproducible mass spectra between replicates. Minor variations in protocols may compromise the efficacy of identification, especially at the subspecies and strain levels1,3,10. However, neither manufacturer-provided reference databases nor reported custom databases include visually documented procedures for database construction and/or application of a data analysis pipeline. For this reason, the objective of this work was to develop, apply, and demonstrate a comprehensive and detailed procedure for library-based bacterial identification using MALDI-TOF MS.
In this demonstration, mass spectra of 15 bacteria isolated from a karstic environment (Kartchner Cavern, AZ, USA) were collected and imported into software to construct a model database. Data processing and the analysis pipeline were detailed using the model database. Finally, mass spectra of blind-coded bacteria which were randomly selected from these 15 bacteria were collected again and compared to the reference spectra in the model database for identification. Results show that bacteria can be correctly identified either based on similarity coefficients or potential biomarkers/peak classes.
プロトコル
注意 :どのような環境からの未確認細菌は病原性とすることができ、適切なバイオセーフティプロトコルを使用して、慎重に処理する必要があります。ライブ文化との仕事は生物学的安全レベル2(BSL-2)の手順を使用して、クラスII安全キャビネットで実行する必要があります。 BSL-2手続きの詳細については、CDC / NIHのタイトルのマニュアル、「微生物学的および生物医学研究所でバイオセーフティ、「ページ33-38で利用可能です。ドキュメントは、オンラインで利用可能ですhttp://www.cdc.gov/biosafety/publications/bmbl5/BMBL.pdf 。白衣/ガウン、安全眼鏡、及びニトリルまたはラテックス手袋など適切な個人用保護具(PPE)は、着用しなければならない。標準微生物学的慣行や注意事項に従わなければならず、バイオハザード廃棄物は適切に廃棄しなければならない。
このデモで使用される細菌は、カーチェナー洞窟から単離した、AZ、USA、ドライ鍾乳石、フロー石、湿った鍾乳石や鍾乳石のドリップ( 表1)を含む4つの環境から。すべての分離株は、16S rDNAの塩基配列決定によって同定され、25%グリセロール、R 2 B培地中で-80℃で保存した。すべての実験は、室温で完了した。
注:私たちは、データベース構築と未知数の質量スペクトルのための質量スペクトルを取得するために、同じサンプル調製方法を使用することをお勧めします。試料調製法は、スペクトルの品質および再現3に影響することが以前に示されている。異なる試料調製方法を使用すると、(株レベルで、例えば 、)より高い分類学的解像度が所望される場合は特に、未知数の誤った識別を引き起こし得る。
MALDIターゲット1.沈着
注意 :タンパク質抽出物を得るためのいくつかのプロトコルは、酸とのguidに従って利用されなければならない有機溶媒の使用を必要とするそれぞれの材料安全データシート(MSDS)に含まれるelines情報。適切なPPEは、アセトニトリルなどの毒性、引火性溶剤、かなりの量で作業するときに使用する必要があります着用しなければならないと種類とボリューム使用される化学物質の( 例えば 、白衣/ガウン、手袋、安全眼鏡、および呼吸器保護により変動するため、ギ酸およびトリフルオロ酢酸などの腐食性の酸、)。
- ステンレス製MALDIターゲットプレート上に、それを乾燥させ(必要に応じて、以前に記載されているプロトコル11-13を用いて得られた)は生細胞を含まないタンパク質抽出μlの預金1。 1μlのマトリックス溶液(αシアノ-4-ヒドロキシ桂皮酸溶液)を用いて乾燥させたタンパク質抽出物をオーバーレイし、それを乾燥させる。
- 各生物学的反復のために、技術的反復(5から20技術的反復)の適切な数を見つける。ここでは、それぞれの生物学的複製のための10の技術的複製物および3生物学的複製ワットを発見ERE準備。
注意:我々は、タンパク質抽出試料調製方法を使用している場合に磨かMALDI鋼ターゲットプレートを使用することをお勧めします。グランド鋼ターゲットプレートを使用すると、個々のサンプルウェルの外側の異なるサンプルの混合を拡散意図しない引き起こすことがある。 - 預金1μlの較正用ターゲットプレート上の標準とそれを乾燥させる。 1μlのマトリックス溶液とオーバーレイし、それを乾燥させる。
- 陰性対照として、ターゲットプレート上に堆積物2μlのマトリックス溶液。
2.マススペクトルの取得
- 窒素レーザー(λ= 337nmで)を備えたMALDI-TOF質量分析計を使用したBruker FlexControlソフトウェアを使用して動作する。
- 100ショット単位で500レーザーショットの蓄積によって正の線形モードにおける各質量スペクトルを収集する。 20 kVのイオン源1の電圧を設定します。イオン源2電圧18.15 kVのに。と9.05 kVのにレンズ電圧。これらのパラメータは楽器-speciであることに注意してくださいFICと最適な結果を得るために、他の楽器に調整が必要になる場合があります。
- 2からの充電あたりの20kDaに自動化されたスペクトル評価のための質量電荷範囲を設定します。重心ピーク検出アルゴリズムを使用してください。 100ダでの最小分解能しきい値を設定します。 100での最小強度しきい値を設定2.しきい値:ノイズ比(N S)に信号を設定します。
3.データベース構築
- データベース設計
- 「新しいデータベースウィザード」を使用してBioNumerics 7.1で新しいデータベースを作成します。
- 「実験タイプ」パネルでコマンドを使用して、 例えば 、のMALDIスペクトル実験タイプを作成します。
- 「データベース設計パネル」を使用してレベルを作成します。 「データベース」メニューの「レベル>新しいレベルを追加... "コマンドを使用して新しいレベルを追加。ここで、「種」レベル、「生物学的複製 "レベル」と「技術複製」レベル、respectivを作成エリー。
- 生の質量スペクトルのインポートと前処理
- 「ファイル」メニューの「エクスポート>マススペクトル」コマンドをクリックしてFlexAnalysisを使用して.txtファイルとして生の質量スペクトルをエクスポートします。
- 技術的反復のレベルのデータベースに生の質量スペクトル(.txtファイル)をインポートします。
- 生の質量スペクトル前処理を行います。
- インポートとリサンプル(二次フィッティングアルゴリズムを使用して)。
- (50点の大きさと転がりディスク付き)ベースライン減算を実行します。
- (20ポイントと10ポイントのベータのウィンドウサイズとカイザー窓)なめらかな、ノイズ[連続ウェーブレット変換(CWT)]を計算し、第二のベースラインの減算(200ポイントの大きさと転がりディスク)を行う。
- [10:ノイズ比(N S)の最小信号CWT]ピークを検出する。
- 前処理した後、そのようなピークを含むピークリストなどの各マススペクトルの特徴的なパターンを保存大きさ、ピーク強度、S:データベース内のN など 。
- 複合質量スペクトルを作成する
- 「分析」メニューの「要約... "コマンドを使用して、前処理済みのスペクトルから複合スペクトルを作成します。目標レベルとして「生物学的複製」を選択してください。
- ここでは、「生物学的複製」レベルでの分離株の3つの複合質量スペクトルで、その結果、そのコロニーのための複合質量スペクトルを得るために、同じコロニーの10技術的反復の質量スペクトルを兼ね備えています。
- ここでは、それは「種」レベルで分離するための一つの複合スペクトルを作成するために3複合スペクトルをまとめる。
注:複合スペクトルは、技術的反復のポイントバイポイント平均である。 95%未満(初期設定)の平均値との類似性(ピアソン相関)を複製する合成から除外される。彼らは、75%で存在する場合、複合スペクトル上のピークのみ(と呼ばれ含まの複製のデフォルト設定)。生物学的複製のために、これらの設定は、それぞれ、90および60%であった。
4.マススペクトルデータ解析
- データベース内のエントリを選択し、「比較」パネルに「新しい比較を作成する」コマンドをクリックして、比較を作成。
- ここで、比較·分析を表示するには「技術の複製」および/または「生物学的複製」のレベルで質量スペクトルを使用しています。
- 類似性に基づくクラスター分析および多次元スケーリング(MDS)
- 色でグループを作成します。 3生物学的複合マススペクトルを選択し、対応する分離株のためのグループを作成するには、「グループ」メニューの「選択範囲から新しいグループを作成」コマンドをクリックします。自動的にこれら3質量スペクトルに使用する色を指定します。
- あるいは、「でコマンドを使用して対応する色で、フィールドの状態を定義するこの定義されたフィールドに基づくグループ化がこのグループのために定義された同じ色を使用するように、データベースエントリ」パネル。
- クラスター分析を行います。 「クラスタリング」メニューの「クラスター分析の計算」コマンドをクリックします。比較設定の1ページ目で、「ピアソン相関」を選択し、デフォルトなどの他のパラメータを残す。 2ページ、「UPGMA」を選択します。その後、「完了」をクリックします。
- 「統計」メニューの「多次元スケーリング... "コマンドを使用して、MDSプロットを取得します。
- ピークマッチング
- 「実験」のパネルで、スペクトルタイプ「MALDI」をクリックします。その後、「レイアウト>の表示画像」を選択します。スペクトルは、ゲルのバンドとして示されている。
- 「スペクトラ」メニューの「ピークマッチングを行い "コマンドを使用してピークマッチングを行います。
- ピーククラスの識別
- 主成分分析(PCA)を実行する。強調表示」実験MALDI」の実験タイプ ""パネルと使用し、「PCAを実行するために統計的「メニュー」でコマンドを「主成分分析...。
- 2-Wayクラスタリングを実行します。 「比較」ウィンドウの「統計>マトリックスマイニング」を...クリックします。ピークのクラスに一致するピークの強度は、異なる色(ヒートマップ)を用いて表される。
カスタムデータベース5.細菌同定
- 類似性係数に基づく方法
- 比較を作成し、ステップ4.3.3で説明したように「技術の反復 "レベルで質量スペクトルに基づく系統樹を生成する。類似性の比較のために系統樹を保存します。
- 「選択したエントリの識別>分析 "未知の質量スペクトルを選択し、クリックします。識別ダイアログボックスが表示されます。
- 「比較に基づく「分類器の種類(またはストアドclassifを選択IER)と「次へ」をクリックします。次のページで、「次の「参照比較として保存樹形図を選択し、[OK]をクリックします。
- 識別方法として「基本類似性」を選択し、「次へ」をクリックします。
- 採点方法として「最大類似性」を選択してください。各パラメータの適切なしきい値と最小差分値を入力し、「次へ」をクリックします。
- 計算が完了すると、識別ウィンドウが表示されます。 「結果」パネルで、最高の未知の一致するデータベースのメンバーが一覧表示されます。
- 識別プロジェクトを保存し、「識別プロジェクト」パネルで、「クロス検証解析」コマンドを使用して識別を検証する。
- 潜在的なバイオマーカーに基づく方法
- ピークのクラスを定義します。 「マトリックスマイニング」ウィンドウで、共通の特性を共有するピークのセットを選択し、speciとしてこれらのピークを定義する使用してFICピーククラス(潜在的なバイオマーカー)」スペクトル>ピーククラスタイプの管理...」「比較ウィンドウ」で。
- ここでは、すべての15の分離株のために隔離するごとに、特定のピークのクラスを定義します。
- 未知数の質量スペクトルを選択し、前述のように定義されたピークのクラスにこれらのスペクトルのピークと一致する。
結果
このデモで構築したデータベースは、「すべてのレベル」、「種」、それぞれ「生物学的複製」と「技術複製」、( 図1A)を含む、最高に最低レベルから、4つのレベルを持っていた。 「技術の複製」レベルは技術的反復のすべての前処理されたスペクトルを含んでいた。 「生物学的複製」と「種」レベルはコンポジット(サマリー)スペクトルを含んでいた。 「すべてのレベル」は、すべての技術的複製スペクトルだけでなく、すべての複合スペクトルを含んでいた。
スペクトラム集約手順は代表的ピークを用いて、図1に示されている。各メンバー質量スペクトルは、薄い灰色の線として表示されます。複合スペクトルを赤色に着色線として表されている。隣接するピークは容易に目視検査( 図1B)を可能にするために別の色でマークされています。
30回の反復の質量スペクトルの再現性(3つの生物学的複製物、10テクニカルレプリケートそれぞれ)を算出し、表1に示されている。最も再現性バチルス種 Bについて98.0±1.4であり、最も低い再現Curvibacter種 ( 表1)89.4±7.8であった。
生物学的レプリケートレベルでのクラスター分析は、複雑な質量スペクトルデータに階層構造の可視化を容易にした。 図2に示されるように、生物学的複製物を一緒にクラスタ化され、細菌の15種は15クラスターを形成した。密接に関連する種、例えば、 バチルス·エスピー。 、B、D、及びEは、一緒にクラスター化する傾向があった。しかし、異常値、例えば、 バチルス·エスピー。 CおよびFは、また観察された。 「技術と生物」レベルでの質量スペクトルに基づくMDSプロットを図3に示す。MDS Pロットはこれらの細菌のスペクトル間の類似性の明確な、3次元可視化をもたらした。技術的複製物および生物学的複製物の両方が同様のグループ分け( 図3のAおよびB)を示した。
ピークマッチングは、質量スペクトルにおけるピークのセットを区別するために使用した。一定の許容値(x軸上の点)、リニア許容値(ppm)を、ピーク検出率を含むピークマッチングパラメータは、ユーザによって指定される必要がある。 m / zを×位置公差=定数公差+線形公差:定数公差及び線形公差が方程式を用いて、ピークの位置公差を計算するために使用される因子である。増加のm / zを使用すると、一定の許容範囲の重要性が減少する。ピーク検出率は、ピークがスペクトルの定義されたレートよりもためのその位置に見出される場合にのみ、最大クラスが行われたことを意味する。 1つ以上のパターンのピークは、ピーククラスを表す。例えば、ピーク検出率equalsは10%、最大クラスがスペクトルの10%を超える位置にピークを持っている場合にのみ行うことができる。これは技術的反復とセットで低有病率のピーク(通常ノイズピーク)を除外します。セットは生物学的複製の複合スペクトルに基づいている場合、低有病率のピークはすでに複合スペクトルの作成中に除外されてきたように、この数は低くする必要があるかもしれない。このデモンストレーションでは、ピークマッチングが「テクニカルレプリケート "レベルで質量スペクトルを使用して実行し、これらのパラメータの値は、それぞれ1.9、550および10%であった。選択されたパラメータに基づいて、ピークは、異なるピーク群を生じる、一致または一致しないと考えられた。ピーク検索結果の例は、単一の単離物の30回の反復(Bacillus種 A)を用いて、図4に示されている。マッチングの結果は、生強度が色として存在するテーブルとして可視化した。ブルーは、低強度を示し、赤は高いIを示し、ntensity。ピークマッチング結果に基づいて、ユーザは、フォローアップ分析を容易ピークのクラスを定義することができる。
両方の主成分分析(PCA)( 補足図1)との双方向のクラスタリングは、複合ピーククラスを分析するために使用され得る。 「テクニカルレプリケート"レベルで質量スペクトルを用いて、代表的な双方向のクラスタリング結果を、 図5に示されている。二つの樹状図が示されている。一つは、m / z値の隣にあり、他の細菌のエントリの上に( 図5)。ピーク強度は、緑色が低強度を示し、赤色は高い強度を示した色で表現した。例えば、 バチルス·エスピー。 AとF は、B。SPと非常に少数のピーククラスを共有。 B及びD( 図5)。精密検査は、B種ことを示した。 BおよびDはまた、種特異的ピーククラス( 図5)のセットを有する。これらの結果は、特定のピークセットが証明書を共有することを示しているアイン特性は種レベルの潜在的なバイオマーカーとして定義することができる。例えば、13のピークセットはB.属に属する。 Dが選択され、B種のピーククラス(潜在的なバイオマーカー)として定義した。 2152.5、2894.9、3420.8、4302.0、4339.9、4629.2、5189.4、5448.4、5878.7、6388.8、6838.8、6931.1、及び7849.1( 表2)を含む、D、。異なる単離物のピークのクラスは、異なる色( 補足図2)で示すことができる。それぞれの具体的なピークのクラスは、表2にまとめた分離します。定義されたピークのクラスは、さらに手動で彼らはさらに100 AUの最小強度を持つすべての技術的反復に登場したことを確認するためにチェックした、ピークのクラスのサブセットはまた、細菌の特徴付けを容易にするために格納されている可能性があります亜種および/または歪みレベルで、例えば、非病原性株から病原性株を区別するため、および/または抗生物質耐性/感受性を検査する。
識別に関してENT ">は、ブラインドコード化された単離物の質量スペクトルを収集し、データベース内の参照質量スペクトルと同様に前処理された。類似性係数の比較に基づいて同定するために、パラメータ値が最大類似度を含む、指定された最小類似が指定されていない95.0%及び87%での平均的な類似性で。( すなわち 、チェックしないまま)。最小差分値が最大類似し、平均類似性の両方の5のように設定した。これらの値は、さらに増加するように最適化する必要があるかもしれない正答率6図は、類似性係数( 図6)の比較に基づいて識別結果を示す。マッチング結果は、このブラインドコード化された細菌は、おそらくB.種であることを示唆した。A.この識別プロジェクトとの比較に基づいて類似係数は、さらにクロス確認( 補足図3によって検証した)。交差検定は、25%の被覆率で試験した。より高い被覆率を用いて、例えば、50%または100%は、識別の信頼性を高めることができるが、特に大規模なデータベースの場合、完了するまでに非常に長い時間を要する。すべてのテストされたクラスは、100%真のポジティブおよび0%偽陰性( 補足図3)を有している。興味深いことに、ピークのクラスの比較に基づいて識別プロジェクトのためのクロスバリデーションは、類似性係数の比較に基づくものよりもはるかに高速である。識別は、ピーククラスマッチング( 補足図4)に基づいて完成させることができる。しかし、ピークのミスマッチ( 補足図4)が観察された。ミスマッチは、それぞれのタンパク質中のアミノ酸の交換に起因する質量シフトに起因し得る。一致していないピークはまた種レベルでの差別はありませんが、この株または分離株に特有のピークがある可能性があります。一緒になって、O類似性係数およびバイオマーカーベース - - 容易にここで説明試料調製、スペクトル取得、およびデータ分析のワークフローを使用して、カルスト環境から種レベルで細菌を同定することができるウル結果は、両方の識別方法があることを示唆している。

図1.データベース構築及び質量スペクトルサマライズこのデモ(A)で構築したデータベースの構造。 Aminobacter種A(B)の10技術的反復からピークを用いて、ピーク要約のイラスト。
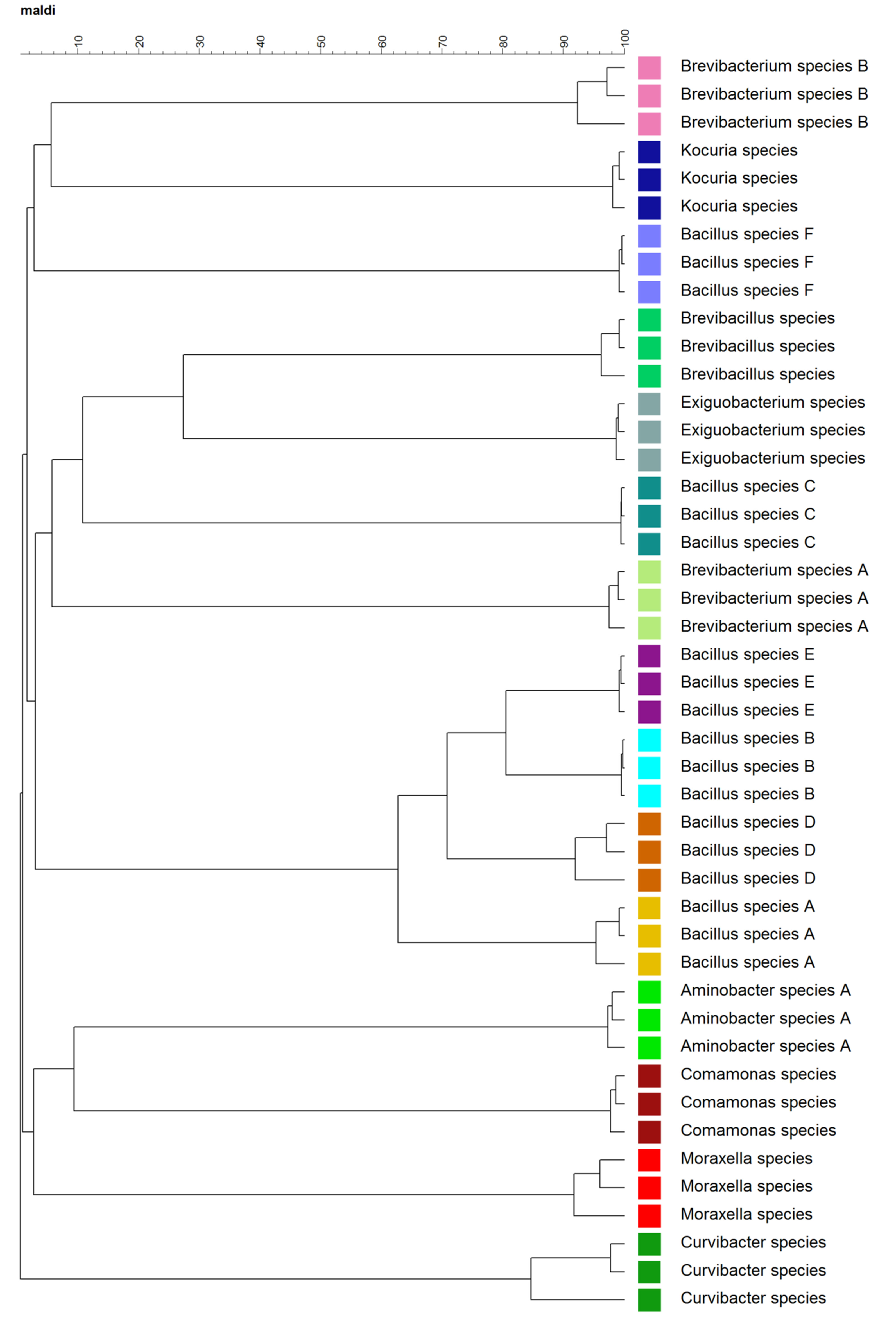
複合の図2.デンドログラム生物学的な複製レベルでの質量スペクトル。データセットは、それぞれの種のための3つの複合スペクトルを持つ15種類の種のスペクトルが含まれています。それぞれの種は色でコード化した。

多次元スケーリング(MDS)30それぞれの種のためのスペクトル(A)と、それぞれの種のための3つの複合スペクトル(B)との生物学的複製レベルの技術的複製レベルでの質量スペクトルの表現は。色は同じ色としてコード化された。図図2で使用される。

図4.ピークマッチングテーブルの説明図。表は技術的レプリケートのleveにおけるバチルス種 Aの質量スペクトルに基づいて生成されたL。ピークマッチングパラメータの値は、それぞれ線形公差定数公差1.9、550、ピーク検出率は10%であった。ブルーは、低ピーク強度を示し、赤は高いピーク強度を示している。 図の拡大版をご覧になるにはこちらをクリックしてください。

図5.双方向クラスタリングの図。図テクニカルレプリケートレベルで質量スペクトルを使用して生成された。分離株の色は、図2で使用したのと同じ色としてコード化された。ピーク強度は、色、グリーンの意味低強度と赤の意味は、高強度で表されます。 図の拡大版をご覧になるにはこちらをクリックしてください。
キープtogether.withinページ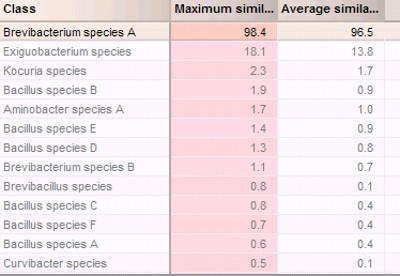
カスタムデータベースを使用して、類似性係数の比較に基づいて、図6細菌同定。
| IDのA | ソース | 最寄りの相対的なB /門/クラス | 受託#(最も近い相対B) | %の類似性 | BioNumericsキー | 再現性(%) |
| D2 | ドライ鍾乳石 | バチルス種。 E-257 /ファーミキューテス | FJ764776.1 | 98.8 | バチルス種のA | 94.9±4.0 |
| D7 | ドライspeleot裾 | バチルス種。 GGC-P3 /ファーミキューテス | FJ348039.1 | 99.0 | バチルス種B | 98.0±1.4 |
| F1 | フロー石 | バチルスナイアシン株 M27 /ファーミキューテス | KC315764.1 | 99.2 | バチルス種C | 96.5±2.4 |
| F4 | フロー石 | バチルス種。 GGC-P5A1 /ファーミキューテス | FJ348046.1 | 99.1 | バチルス種D | 89.8±8.8 |
| F9 | フロー石 | バチルス種。 OSS 19 /ファーミキューテス | EU124558.1 | 99.4 | バチルス種E | 96.5±1.9 |
| R10 | 鍾乳石ドリップ | バチルス種。 K1 /ファーミキューテス | GU968734.1 | 99.8 | バチルス種F | 95.4±3.9 |
| D11 | ドライ鍾乳石 | ブレビバチルス·ブレビス株 IMAU80218 / ファーミキューテス | GU125635.1 | 99.5 | ブレビ種 | 94.3±5.8 |
| F14 | フロー石 | エキシグオバクテリウム·エスピー ZWU0009 /ファーミキューテス | JX292087.1 | 99.3 | エキシグオバクテリウム種 | 96.5±2.5 |
| M7 | モイスト鍾乳石 | ブレビバクテリウム·エスピー N78 /放線菌 | HQ188605 | 97.6 | ブレビバクテリウム種A | 97.5±2.0 |
| M14 | モイスト鍾乳石 | コクリアrhizophila株 Ag09 /放線菌 | EU554435.1 | 100 | コクリア属 | 95.2±4.1 |
| M15 | モイスト鍾乳石 | ブレビバクテリウム·エスピー MN3-3 /放線菌 | JQ396535.1 | 99.5 | ブレビバクテリウム種B | 92.1±4.9 |
| R4 | 鍾乳石ドリップ | SP Aminobacter。 KC-EP-S4 /αプロテオバクテリア | FJ711220.1 | 99.9 | 種A Aminobacter | 95.4±2.7 |
| F5 | フロー石 | コマモナステストステロニ歪みNBRC 12047 /βプロテオバクテリア | AB680219 | 100 | コマモナス種 | 96.4±2.6 |
| R8 | 鍾乳石ドリップ | Curvibacterデリケート/βプロテオバクテリア | AB680705 | 97.0 | Curvibacter </ em>の種 | 89.4±7.8 |
| F8 | フロー石 | モラクセラ属。 19.2 KSS /γプロテオバクテリア | HE575924.1 | 99.9 | モラクセラ種 | 92.6±4.9 |
細菌はカーチェナー洞窟、AZ、USAから単離され、16S rDNAの塩基配列決定を用いて同定した。二つのプライマー、27fに(5 'AGA GTT TGA TCC TGG CTC AG 3')および1492r(5 'TAC GGT TAC CTT GTT ACG ACT T 3')は、ほぼ1400 bpの長16S rRNA遺伝子配列を得るために使用した。
B NCBIデータベースのBLAST検索に基づいています。
報告されたCの値は、1標準偏差±30回の反復(3生物学的複製10技術的反復で各)の平均相関係数である。
デモで使用される表1.細菌分離株。
| BioNumericsキー | ピーククラス/潜在的なバイオマーカー(DA) |
| バチルス種のA | 2152.5、2224.9、2595.8、2894.9、2921.3、3380.5、3496.3、3515.0、3733.5、4302.0、4340.0、4385.8,4763.9、4910.6、5189.4、5227.0、5634.6、5769.6、5892.8、6301.4、6756.2、6789.4、6990.3、7029.5、7466.3 |
| バチルス種B | 2152.5、2941.2、3196.9、3262.7、3352.9、3420.8、3733.5、3925.2、4302.0,4339.9、4629.2、4713.4、4859.3、4900.6、5189.4、5227.0、5541.8、5878.7、6388.8、6524.0、6704.5、6838.8、7142.7、7317.5、7466.3、 7849.1、9263.9、9721.2 |
| バチルス種C | 2588.0、3361.8、4330.4、5173.0、5847.6、6332.0、6524.0、6720.3 |
| バチルス種D | 2152.5、2894.9、3420.8、4302.0、4339。9、4629.2、5189.4、5448.4、5878.7、6388.8、6838.8、6931.1、7849.1 |
| バチルス種E | 2152.5、2224.9、2941.2、3180.1、3380.5、4302.0、4339.9、4705.3、5878.7、6356.6、6735.1、6756.2 |
| バチルス種F | 3308.6、3367.8、3567.5、4279.7、4489.2、4629.2、4727.9、4751.7、5067.7、6614.7、6919.7、7130.9 |
| ブレビ種 | 2133.3、2611.0、4263.3、4302.0、4859.3、4900.6、5080.2、5219.0、5847.6、6775.7、7529.4、9721.2 |
| エキシグオバクテリウム種 | 2588.0、3053.3、3420.8、3695.5、4263.3、5133.1、5173.0、5248.8、6104.8、6605.3、6804.4、6838.8、7390.2 |
| ブレビバクテリウム種A | 3053.3、6104.8、6146.5 |
| コクリア属 | 3080.0、4366.6、5080.2、5163.8、5207.1、5892.8、6160.0、6197.5、6445.0、7433.7 |
| ブレビバクテリウム種B | 3222.8、3330.4、3367.8、4330.4、4350.4、4795.3、4995.7、5731.6、6445.0、6735.1,7487.3 |
| 種A Aminobacter | 2133.3、2562.4、3361.8、3410.4、4289.2、4629.2、4662.0、4869.8、6064.1、6221.0、6720.3、6789.4,6818.8、7216.1、7447.4 |
| コマモナス種 | 2806.0、2921.4、3246.5、4350.4、4727.9、5607.5、5666.3、6221.0、6488.3、7317.5、9362.6 |
| Curvibacter種 | 2868.6、3453.2、4319.8、5133.1、6292.4、6903.4、7433.7 |
| モラクセラ種 | 3011.2、5698.0、6720.3、7064.8、7366.6 |
それぞれの種のために定義され、表2のピーククラス(潜在的なバイオマーカー)(DA)。
補足図1番目におけるマススペクトルの原理成分分析(PCA)電子テクニカルレプリケートレベル(A)とピーククラス(B)の色は、 図2で使用したのと同じ色としてコード化された。
補足図2双方向クラスタリングに基づいて選択されたピークのクラスの実例。同じ標識を有するピークのクラスが同じ色で着色されている。
補足図BioNumericsのカスタムデータベースに基づいて識別プロジェクト3.クロス検証結果。
カスタムデータベースを使用してピークマッチングに基づく補足図4.細菌識別。
ディスカッション
このデモでは、特性評価およびMALDI-TOF MSおよびカスタム·データベースを使用して、細菌の同定の詳細な手順を示した。例えば、伝統的な分子的方法、16S rDNAの塩基配列決定と比較して、MALDI-TOF MSベースのフィンガープリント法は、多様な細菌のより迅速な識別を容易にする。 、そのロバスト性のため、この技術は、広く環境から臨床現場1,14-16細菌、ウイルス、真菌および酵母を特徴付けるために使用される。また、MALDI-TOF MSは、いくつかの場合において、より高い分類学的解像度1を得たことが報告されている。例えば、 バチルス·エスピー。 、B、D、及びEは、一緒に( 図2)をクラスタ化する傾向があるものの、明確に分離し、異なるB.種のスペクトルの間の類似性。 80%未満である( 図2)であった。対照的に、これらの分離株の16S rDNA配列は、これらの分離株を区別するために使用することができなかった高い類似性を有していた種レベルで。 B. SP の 16S rDNA配列。 B.種の配列ながらBおよびDは、多重アラインメント分析に基づいて99%の類似性を有する。 AとEは、B種の配列に、それぞれ、95%および96%の類似性を示す。 BおよびD.外れも観察された。例えば、 バチルス·エスピー。 CとFが離れて他のB. SPからグループ化された。 ( 図2)。外れ値の分離株の出現は質量スペクトルのクラスタリング解析は必ずしも系統関係を確立していないことを示します。分離株からの環境は、質量スペクトルのクラスタに影響を与える可能性が得られた。湿った鍾乳石とB.属から単離された、例えば、 ブレビバクテリウム種B及びコクリア種。鍾乳石のドリップから単離されたFが一緒に( 表1、図2)クラスタ化する傾向があったが、それ以上の研究は、これはISOLの大きなコレクションで観察されているかどうかを調べるために必要とされているのATE。
このライブラリベースの技術は、いくつかの制限があります。キャラは通常、データベースに基づいています。現在の商用データベースは、主に、細菌株、特に病原性のもので構成されている。これらの商用データベースは、臨床微生物ラボの設定で最も有用である。環境単離物ならびにウイルス、真菌および酵母を特徴付けるために、カスタムデータベースが大ひずみのコレクションを使用して構築される必要がある。フォローアップ分析で使用されるパラメータは、特に亜種および菌株レベルで、分類学上の解像度を向上するために最適化される必要があり得る。例えば、S:Nこのデモンストレーションのピーク検出に使用されるこの値は、種レベルの同定に適しているが、歪みレベル識別のため、この値は低くする必要があるかもしれ10であった。これらの処理パラメータと同様に、データ処理のワークフローは時々ユーザー定義の多くのソフトウェアパッケージであり、例えば、ClinProToolsとBionume以来RICSは、適切なワークフローのパラメータ値と選択の最適化は、おそらく、データ分析を最適化するために必要とされる。このデモでは、ピークマッチングパラメータ、識別プロジェクトで使用される閾値は、クロスバリデーション正しい識別率を向上させるために必要なすべての最適化。これらのパラメータを最適化するための方法および/またはプロシージャを見つけるには私たちの研究室に非常に興味深い。例えば、1つのアプローチは、我々は、MALDI-TOFデータ取得17自動最適化するために最近使用された統計的要因計画を伴うかもしれない。追加の将来のアプリケーションとMALDI-TOF MSベース微生物フィンガープリンティングの強化は、広く利用可能の建設、環境細菌および/ または非細菌の微生物の大規模なデータベースだけでなく、混合培養物18と微生物群集の特徴付けを含む。
開示事項
Authors Vranckx and Janssens are employees of Applied Maths NV, the manufacturer of data analysis software used in this video. Applied Maths NV provided select software modules highlighted in this video as well as a portion of the publication costs associated with this video.
謝辞
This work was supported by the New College of Interdisciplinary Arts and Sciences at Arizona State University, Applied Maths NV, and by the National Science Foundation (ROA Supplement to Award No. MCB0604300). Any opinions, findings, and conclusions or recommendations expressed in this material are those of the author(s) and do not necessarily reflect the views of the National Science Foundation.
資料
| Name | Company | Catalog Number | Comments |
| α-cyano-4-hydroxy-cinnamic acid | ACROS Organics | 163440050 | ≥ 97%, CAS 28168-41-8 |
| MALDI calibration kit | Aigma-Aldrich | MSCAL1-1KT | This kit also contains acetonitrile, trifluoroacetic acid , sinapinic acid, etc. |
| MALDI target plate | Bruker Daltonics | 280800 | Polished Steel |
| Bruker Microflex LRF MALDI-TOF mass spectrometer | Bruker Daltonics | ||
| Bruker FlexControl software | Bruker Daltonics | version 3.0 | |
| Bruker FlexAnalysis software | Bruker Daltonics | version 3.0 | |
| Bionumerics software | Applied Maths | version 7.1 |
参考文献
- Sandrin, T. R., Goldstein, J. E., Schumaker, S. MALDI TOF MS profiling of bacteria at the strain level: A review. Mass Spectrom Rev. 32 (3), 188-217 (2013).
- Siegrist, T. J., et al. Discrimination and characterization of environmental strains of Escherichia coli by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry (MALDI-TOF-MS). J Microbiol Meth. 68 (3), 554-562 (2007).
- Goldstein, J. E., Zhang, L., Borror, C. M., Rago, J. V., Sandrin, T. R. Culture conditions and sample preparation methods affect spectrum quality and reproducibility during profiling of Staphylococcus aureus with matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Lett Appl Microbiol. 57 (2), 144-150 (2013).
- Benagli, C., et al. A rapid MALDI-TOF MS identification database at genospecies level for clinical and environmental Aeromonas strains. Plos One. 7 (10), (2012).
- Sauer, S., Kliem, M. Mass spectrometry tools for the classification and identification of bacteria. Nat Rev Microbiol. 8 (1), 74-82 (2010).
- Bohme, K., et al. SpectraBank: An open access tool for rapid microbial identification by MALDI-TOF MS fingerprinting. Electrophoresis. 33 (14), 2138-2142 (2012).
- Walker, J., Fox, A. J., Edwards-Jones, V., Gordon, D. B. Intact cell mass spectrometry (ICMS) used to type methicillin-resistant Staphylococcus aureus: media effects and inter-laboratory reproducibility. J Microbiol Meth. 48 (2-3), 117-126 (2002).
- Ruelle, V., El Moualij, B., Zorzi, W., Ledent, P., De Pauw, E. Rapid identification of environmental bacterial strains by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Commun Mass Sp. 18 (18), 2013-2019 (2004).
- Sedo, O., Sedlacek, I., Zdrahal, Z. Sample Preparation Methods for Maldi-MS Profiling of Bacteria. Mass Spectrom Rev. 30 (3), 417-434 (2011).
- Swatkoski, S., Russell, S., Edwards, N., Fenselau, C. Analysis of a model virus using residue-specific chemical cleavage and MALDI-TOF mass spectrometry. Anal Chem. 79 (2), 654-658 (2007).
- Freiwald, A., Sauer, S. Phylogenetic classification and identification of bacteria by mass spectrometry. Nat Protoc. 4 (5), 732-742 (2009).
- Drevinek, M., Dresler, J., Klimentova, J., Pisa, L., Hubalek, M. Evaluation of sample preparation methods for MALDI-TOF MS identification of highly dangerous bacteria. Lett Appl Microbiol. 55 (1), 40-46 (2012).
- Lasch, P., et al. MALDI-TOF mass spectrometry compatible inactivation method for highly pathogenic microbial cells and spores. Anal Chem. 80 (6), 2026-2034 (2008).
- Usbeck, J. C., Kern, C. C., Vogel, R. F., Behr, J. Optimization of experimental and modelling parameters for the differentiation of beverage spoiling yeasts by Matrix-Assisted-Laser-Desorption/Ionization Time-of-Flight Mass Spectrometry (MALDI-TOF MS) in response to varying growth conditions. Food Microbiol. 36 (2), 379-387 (2013).
- Del Chierico, F., et al. MALDI-TOF MS proteomic phenotyping of filamentous and other fungi from clinical origin. J Proteomics. 75 (11), 3314-3330 (2012).
- Vitale, R., Roine, E., Bamford, D. H., Corcelli, A. Lipid fingerprints of intact viruses by MALDI-TOF/mass spectrometry. Bba-Mol Cell Biol L. 1831 (4), 872-879 (2013).
- Zhang, L., Borror, C. M., Sandrin, T. R. A designed experiments approach to optimization of automated data acquisition during characterization of bacteria with MALDI-TOF mass spectrometry. Plos One. 9 (3), (2014).
- Christner, M., Rohde, H., Wolters, M., Sobottka, I., Wegscheider, K., Aepfelbacher, M. Rapid identification of bacteria from positive blood culture bottles by use of matrix-assisted laser desorption-ionization time of flight mass spectrometry fingerprinting. J ClinMicrobiol. 48 (5), 1584-1591 (2010).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved