Method Article
使用MALDI-TOF质谱和自定义数据库的表征细菌土著一个独特的洞穴环境(卡切内岩洞,AZ,USA)
摘要
This work details procedures for rapid identification of bacteria using MALDI-TOF MS. The identification procedures include spectrum acquisition, database construction, and follow up analyses. Two identification methods, similarity coefficient-based and biomarker-based methods, are presented.
摘要
MALDI-TOF mass spectrometry has been shown to be a rapid and reliable tool for identification of bacteria at the genus and species, and in some cases, strain levels. Commercially available and open source software tools have been developed to facilitate identification; however, no universal/standardized data analysis pipeline has been described in the literature. Here, we provide a comprehensive and detailed demonstration of bacterial identification procedures using a MALDI-TOF mass spectrometer. Mass spectra were collected from 15 diverse bacteria isolated from Kartchner Caverns, AZ, USA, and identified by 16S rDNA sequencing. Databases were constructed in BioNumerics 7.1. Follow-up analyses of mass spectra were performed, including cluster analyses, peak matching, and statistical analyses. Identification was performed using blind-coded samples randomly selected from these 15 bacteria. Two identification methods are presented: similarity coefficient-based and biomarker-based methods. Results show that both identification methods can identify the bacteria to the species level.
引言
Matrix-assisted laser desorption/ionization time-of-flight (MALDI-TOF) mass spectrometry (MS) has been shown to be a rapid and reliable tool for identification of bacteria at the genus, species, and in some cases, strain levels1-4. MALDI-TOF MS ionizes biological molecules (typically proteins) that originate from cell surfaces, intracellular membranes, and ribosomes from bacterial whole cells or protein extracts1,5. The resulting peaks form characteristic patterns or “fingerprints” of the bacteria analyzed1. Identification of bacteria is based on these mass-to-charge “fingerprints”.
Two of the most commonly used identification strategies are library-based and bioinformatics-based strategies1. Library-based approaches involve comparing the mass spectra of unknowns to previously collected mass spectra of known bacteria in databases/libraries for identification. Commercially available software, such as BioNumerics, Biotyper, and SARAMIS software packages, as well as open source software tools, such as SpectraBank6, are available to facilitate the comparison and quantification of similarity between mass spectra of unknowns and reference bacteria. Bioinformatics-based approaches usually rely on fully sequenced genomes of bacteria for identification. In contrast to library-based approaches which do not involve identification of the biological nature of particular peaks, bioinformatics-based approaches involve protein identification1.
The majority of recent MALDI fingerprint-based studies have used library-based approaches to identify bacteria1. Library-based approaches require construction of databases and comparison of the similarity between mass spectra. Studies show that many experimental procedures, such as medium3,7, cultivation time8, sample preparation method3, and matrix used9, affect the mass spectra obtained. Furthermore, some closely-related species and strains generate spectra with only subtle differences. Thus, library-based approaches require rigorously standardized procedures to generate highly reproducible mass spectra between replicates. Minor variations in protocols may compromise the efficacy of identification, especially at the subspecies and strain levels1,3,10. However, neither manufacturer-provided reference databases nor reported custom databases include visually documented procedures for database construction and/or application of a data analysis pipeline. For this reason, the objective of this work was to develop, apply, and demonstrate a comprehensive and detailed procedure for library-based bacterial identification using MALDI-TOF MS.
In this demonstration, mass spectra of 15 bacteria isolated from a karstic environment (Kartchner Cavern, AZ, USA) were collected and imported into software to construct a model database. Data processing and the analysis pipeline were detailed using the model database. Finally, mass spectra of blind-coded bacteria which were randomly selected from these 15 bacteria were collected again and compared to the reference spectra in the model database for identification. Results show that bacteria can be correctly identified either based on similarity coefficients or potential biomarkers/peak classes.
研究方案
注意 :在任何环境不明的细菌可能是致病的,必须使用适当的生物安全协议,谨慎处理。与活的文化工作,必须在II类生物安全柜使用生物安全2级(BSL-2)的程序进行。有关BSL-2程序的更多信息,请在CDC / NIH手册中标题为"生物安全微生物和生物医学实验室,"33-38页。该文件可在网上http://www.cdc.gov/biosafety/publications/bmbl5/BMBL.pdf 。适当的个人防护装备(PPE),包括实验室大衣/袍,护目镜和腈或乳胶手套,必须佩戴。标准的微生物实践和注意事项必须遵循的,和生物危险废物,必须进行适当的丢弃。
在这个演示中使用细菌分离卡切内岩洞,AZ,USA,从4的环境中,包括干钟乳石,流石,湿润钟乳石和钟乳石滴( 表1)。所有分离物的16S rDNA序列鉴定,并保持在-80℃下在25%的甘油,R 2 B培养基。所有实验均在室温下完成。
注:我们推荐使用相同的样品制备方法获得大规模光谱数据库建设和未知的质谱。样品制备方法以前已证明影响光谱质量和再现性3。使用不同的样品制备方法可能会导致不正确的识别未知的,特别是当更高分类分辨率( 例如 ,在应变水平)是期望的。
1.沉积在MALDI靶
注意 :几个协议,以获得蛋白质提取物需要使用的酸和有机溶剂必须按照GUID被利用包含在各自的材料安全数据表(MSDS)elines和信息。合适的PPE必须佩戴并且将异基于化学品的使用的类型和量( 例如 ,实验室外套/服,手套,安全眼镜,和呼吸保护必须用显著量的有毒的,易燃的溶剂,如乙腈中工作时使用的,和腐蚀性酸,如甲酸和三氟乙酸)。
- 存1微升不含存活细胞到不锈钢MALDI靶盘,并允许它以干燥(使用适当的,先前所描述的协议11-13得到的)的蛋白质提取物。覆盖干燥的蛋白质提取物与1μl的基质溶液(α氰基-4-羟基肉桂酸溶液),并允许其干燥。
- 对于每一个生物重复,临场技术重复(5〜20技术复制)的适当数量。在这里,发现10技术复制的每个生物重复和3个生物学重复瓦特ERE准备。
注意:我们建议使用蛋白提取的样品制备方法时,使用抛光不锈钢MALDI靶板。使用地面钢靶板可能引起扩散和不同样品的无意混合单个样品井之外。 - 存1微升校准标准到目标板,并让其干燥。覆盖用1微升基质溶液,并允许它干燥。
- 存2微升基质溶液到目标板作为阴性对照。
2.质谱采集
- 使用MALDI-TOF质谱仪装有氮激光器(λ= 337纳米),并使用布鲁克FlexControl软件操作。
- 由500激光投100拍摄的增量积累收集每个质谱正线性模式。设置在离子源1电压20千伏;离子源2的电压为18.15 kV的;和透镜电压,以9.05千伏。需要注意的是,这些参数是乐器特定FIC,可能需要其他手段的调整,以获得最佳的效果。
- 从每次充电2至20 kDa的设定批量荷范围自动频谱评估。使用质心的峰值检测算法。定在100大的最小分辨率阈值。在2阈值100设定的最小强度阈值:将信号噪声比(N S)。
3.数据库建设
- 数据库设计
- 创建BioNumerics 7.1一个新的数据库使用"新建数据库向导"。
- 创建谱的实验类型, 例如,的Maldi,在"实验类型"面板中使用的命令。
- 创建使用"数据库设计面板"的水平。使用添加了新的水平,在"数据库"菜单中的"等级>添加新的水平..."命令。在这里,打造"种"的水平,"生物重复"水平"和"技术复制"的水平,respectiv伊利。
- 导入和预处理原质谱
- 导出原始质谱为使用FlexAnalysis在"文件"菜单中单击"导出>质谱"命令.txt文件。
- 导入原料质谱(.txt文件)转换为数据库中的技术重复的水平。
- 预处理原料质谱。
- 导入和重采样(使用二次拟合算法)。
- 执行基线减法(与滚动光盘50点的尺寸)。
- 计算噪音[连续小波变换(CWT),流畅(Kaiser窗以20分的窗口大小和10点测试),并执行第二基线减法(滚动光盘200点大小)。
- 检测峰[CWT具有最小的信噪比(S:N)10]。
- 预处理后,保存每个质谱的特征模式,例如含峰峰列表大小,峰强度,S:N 等 ,在数据库中。
- 创建复合质谱
- 创建从"分析"菜单中的"总结..."命令预处理光谱复合光谱。选择"生物重复"的目标水平。
- 这里,结合同殖民地的10技术复制质谱产生复合质谱为殖民地,从而导致该菌株在"生物重复"三级复合质谱。
- 在这里,总结一下三复合光谱创建一个复合谱是孤立的"种"的水平。
注意:合成频谱是逐点平均的技术重复的。具有相似性(Pearson相关),以低于95%(缺省设置)的平均复制被排除在复合材料。上的复合光谱峰仅称为如果它们存在于75%(在包括重复的默认设置)。对于生物学重复,这些设置分别为90和60%,分别为。
4.质谱数据分析
- 选择在数据库中的条目,并创建由"比较"面板中单击"创建新的比较"命令比较。
- 这里,可使用质谱在"技术复制"和/或"生物复制"的水平,显示的比较和分析。
- 基于相似性的聚类分析和多维标度(MDS)的
- 创建颜色组。选择三个生物复合质谱,然后单击"创建新组的选择"命令,在"组"菜单中创建一个组对应的分离。指定自动用于这三种质谱一种颜色。
- 另外,定义域指出使用的命令,相应的颜色"数据库条目"面板,使得在此基础上定义的字段中的任何分组使用该组中定义的相同的颜色。
- 进行聚类分析。在"群集"菜单中单击"计算聚类分析"命令。在比较设置页1,选择"Pearson相关",离开其他参数为默认值。在第2页,选择"UPGMA"。然后单击"完成"。
- 获得"统计"菜单中的MDS阴谋使用"多维尺度..."命令。
- 峰匹配
- 在"实验"面板点击谱型"的Maldi"。然后选择"布局>显示图像"。光谱被示为凝胶带。
- 请在"光谱"菜单中的"做峰匹配"命令峰匹配。
- 峰值类鉴定
- 执行主成分分析(PCA)。突出"实验MALDI"在实验型","面板,并使用"主成分分析..."命令,在"统计"菜单中执行PCA。
- 执行双向聚类。点击"比较"窗口中的"统计>矩阵挖掘"......。的匹配峰值类的峰的强度使用不同的颜色(热图)表示。
5,细菌鉴定与自定义数据库
- 相似系数为基础的方法
- 创建一个比较和按步骤4.3.3描述生成基于质谱在"技术复制"级树状图。保存树状的相似性比较。
- 选择一个未知的质谱,并单击"分析>确定入选作品"。出现在鉴定对话框。
- 选择"比较基础"分类型(或存储classifIER),然后点击"下一步"。在接下来的页面中,选择保存的树状图作为参考比较,然后单击"下一步"。
- 选择"基本相似性"作为一种识别方法,然后单击"下一步"。
- 选择"最大的相似性"的评分方法。键入适当的阈值和最低值的差值为每个参数,然后单击"下一步"。
- 一旦计算完成,将出现识别窗口。在"结果"面板中,数据库的成员最符合未知列。
- 保存该项目鉴定和验证使用"交叉验证分析"命令识别,在"识别项目"面板。
- 潜在的生物标志物为基础的方法
- 定义峰值类。在"黑客帝国矿业"窗口中,选择组峰具有共同的特点,并确定这些峰作为特定使用FIC峰值类(潜在生物标志物)"谱>管理高峰类的类型......"中的"比较窗口"。
- 在这里,定义特定的每个隔离所有15株峰值类。
- 选择未知数的质谱和匹配这些光谱的峰到峰定义的类,如前所述。
结果
在此演示构建的数据库有四个层次,从最高到最低的水平,其中包括"各级","物种","生物重复"和"技术复制",分别为( 图1A)。 "技术复制"级别包含的技术复制所有的预处理光谱。在"生物重复"和"物种"的水平包含复合(摘要)光谱。 "各级"包含了所有的技术重复谱以及所有的复合光谱。
频谱聚合过程使用的是有代表性的峰, 如图1所示。每个成员质谱显示为细灰线。该合成频谱被表示为着色为红色的线。相邻的峰均标有不同的颜色,以使用户更容易目视检查( 图1B)。
30次重复的质谱的再现性(3生物学重复,每次用10技术重复)进行了计算,并示于表1,最高的再现性为98.0±1.4为芽孢杆菌种B和最低再现性为89.4±7.8为Curvibacter种( 表1)。
聚类分析在分层结构中的复杂的质谱数据的生物复制水平便利的可视化。 如图2,生物学重复聚集在一起,和细菌的15种形成15簇。密切相关的物种,例如,B。藻。 A,B,D和E,倾向于聚集在一起。然而,异常值,例如,B。藻。 C和F,也观察到。基于所述质谱在"技术与生物"电平的MDS曲线示于图3中 。MDS P大量的产生,这些细菌谱之间的相似性明确,3-D可视化。两个技术重复和生物学重复表现出类似的分组( 图3的A和B)。
峰匹配被用来区分套质谱峰。峰匹配参数,包括恒定公差(在x轴分),线性公差(PPM)和峰值检测率需要由用户指定。不断的宽容和线性宽容是用来计算使用的公式峰的位置公差的因素:位置公差=常数宽容+线性公差×M / Z。随着M / Z,不断容忍的重要性减弱。峰值检测率意味着只有一个峰被发现在该位置以上的频谱的定义的速率,峰值类制成。在一个或多个图案的峰代表一个峰类。例如,如果峰值检出率EQUALS 10%,峰值类可以只作了若光谱的10%以上具有在该位置的峰值。这不包括低流行高峰(通常噪声峰值)的一组与技术复制。如果该组是基于生物学重复的复合光谱,这个数字可能需要较低的低发病率的峰已经被过滤掉的创建复合光谱的期间。在这个演示中,利用质谱在"技术复制"的水平进行峰匹配以及这些参数的值是1.9,550和10%,分别为。根据所选参数,峰被认为是匹配的或不匹配,从而导致不同的峰值组。的峰值匹配结果的一个例子示于图4使用单一分离物的30次重复(Bacillus 物种的A)。匹配结果进行形象化为一表,其中原始强度是存在作为颜色。蓝色表示低强度红色表示较高的Intensity。根据峰值匹配结果,用户可以定义峰值类便于后续分析。
两个主成分分析(PCA)( 补充图1)和双向聚类可用于分析复杂峰类。使用质谱在"技术复制"电平的代表双向聚类结果示于图5中 ,两个系统树图被示出。一个是毗邻m / z值,而另一个是上面的细菌的条目( 图5)。峰强度由颜色,其中绿色表示低强度红色表示强度高表示。例如,B。藻。 A和F与B SP分享很少峰值类。 B和D( 图5)。仔细检查发现,B.藻。 B和D也有套种特异性峰类( 图5)的。这些结果表明,特定峰值集共享证书 AIN特性可以被定义为物种水平潜在生物标志物。例如,13峰集属于B.藻。 D组选择并定义为B.藻峰类(潜在生物标志物)。 D,包括2152.5,2894.9,3420.8,4302.0,4339.9,4629.2,5189.4,5448.4,5878.7,6388.8,6838.8,6931.1,7849.1和( 表2)。峰类不同菌株可以显示在不同的颜色( 补充图2)。特异于每个峰的类分离物被列于表2。定义峰类被进一步人工检查,以确保它们出现在所有技术重复具有100欧此外最小强度,峰类的子集也可以被存储以便于细菌的表征在亚种和/或应变的水平,例如,从非致病株区分致病株和/或检查的抗生素抗性/敏感性。
ENT">对于识别,盲编码分离株的质谱收集和预处理中相同的方式在数据库中的参考质谱。对于基于相似系数的比较识别,参数值被指定,包括最大相似在95.0%,并在87%的平均相似性。未指定的最小相似度( 即 ,任其发展)的最小差异值被设置为5同时为最大相似度和平均相似性。这些值可能需要进一步优化,以提高的正确的识别率。 图6示出了基于相似系数( 图6)的比较鉴别结果,匹配结果表明,此盲编码细菌是最有可能的B.藻。A.本鉴定项目的基础上的比较相似系数由交叉验证( 补充图3进一步验证)。交叉验证,在25%的覆盖率进行测试。使用更高的覆盖率,例如,50%或100%时,可以提高识别的信心,但需要更长的时间来完成,特别是对大型数据库。所有被测试的类有100%的真阳性和0%的假阴性( 补充图3)。有趣的是,交叉验证用于基于峰类的比较识别的项目比那些基于相似系数的比较快的多。识别也可以基于峰值类匹配( 补充图4)完成。然而峰的失配进行观察( 补充图4)。的失配可能是由于质量转变,从在相应的蛋白质氨基酸交换导致。不被匹配的峰也可以是峰值不在判别在物种水平,但是特定于该菌株或分离。两者合计,邻UR结果表明,这两种识别方法 - 相似系数和生物标志物为基础的 - 可以很容易地识别病菌,在使用该样品制备岩溶环境,频谱收购种的水平,并在这里描述的数据分析流程。

图1.数据库建设和质谱总结在这个演示(A)构造数据库结构。插图使用高峰从Aminobacter种A(B)10技术重复峰值汇总。
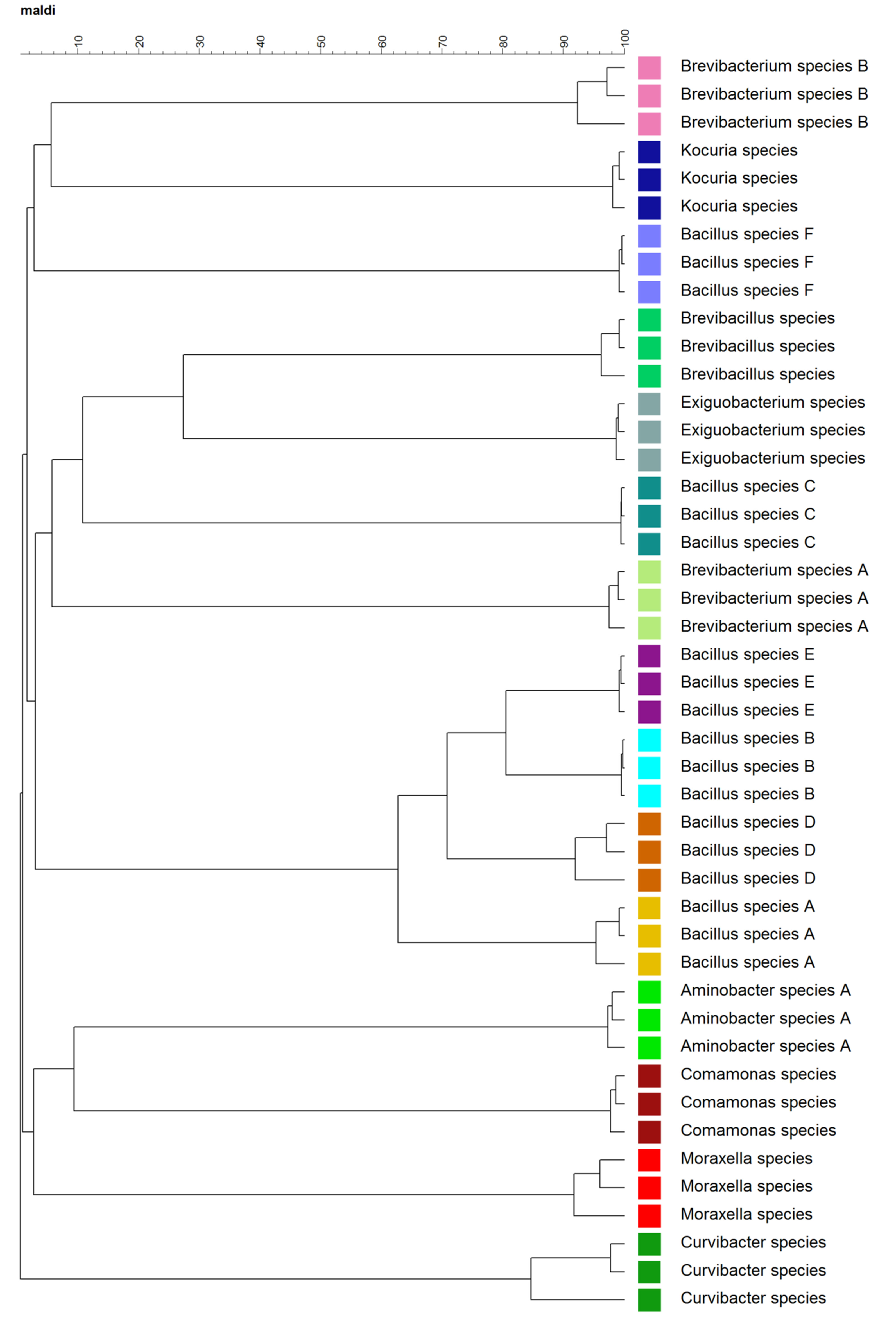
图2.树状复合体质谱在生物复制水平。该数据集包含15个不同的物种与三复合光谱每个物种光谱。每个品种被编码的一种颜色。

图3.多维标度(MDS)的质谱在含有30谱对于每个物种(A)和具有三个复合谱对于每个物种(B)的生物的复制水平的技术的复制水平的表示。颜色被编码为相同的颜色如使用图2中。

图4.峰匹配表的示图。表是基于杆菌种A的在技术复制LEVE质谱生成升。的峰匹配参数的值分别为1.9为恒定公差,550为线性公差和峰值检测率10%,分别为。蓝色表示低峰强度和红色表示较高的峰值强度。 请点击此处查看图的放大版本。

图5的双向聚类的示意图。图是使用质谱在技术复制水平产生。分离的颜色, 如图2中使用的编码为相同的颜色。峰值强度由颜色,绿色的含义低强度红色含义强度高表示。 请点击此处查看图的放大版本。
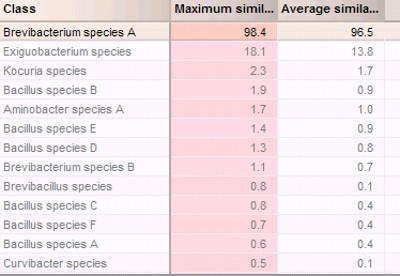
基于使用自定义数据库相似系数的比较图6.细菌鉴定。
| 一个 ID | 来源 | 关系最近的亲属B /语系/类 | 加入#(最近的相对二) | %相似 | BioNumerics关键 | 再现性(%) |
| D2 | 干钟乳石 | 芽孢杆菌 。 E-257 /厚壁菌门 | FJ764776.1 | 98.8 | 芽孢杆菌物种 | 94.9±4.0 |
| D7 | 干speleot下摆 | 芽孢杆菌 。 GGC-P3 /厚壁菌门 | FJ348039.1 | 99.0 | 芽孢杆菌 B | 98.0±1.4 |
| F1 | 流石 | 烟酸芽孢杆菌 M27 /厚壁菌门 | KC315764.1 | 99.2 | 芽孢杆菌 Ç | 96.5±2.4 |
| F4 | 流石 | 芽孢杆菌 。 GGC-P5A1 /厚壁菌门 | FJ348046.1 | 99.1 | 芽孢杆菌 ð | 89.8±8.8 |
| F9 | 流石 | 芽孢杆菌 。 OSS 19 /厚壁菌门 | EU124558.1 | 99.4 | Ë种芽孢杆菌 | 96.5±1.9 |
| R10 | 石钟乳滴 | 芽孢杆菌 。 K1 /厚壁菌门 | GU968734.1 | 99.8 | 芽孢杆菌 ˚F | 95.4±3.9 |
| D11 | 干钟乳石 | 短短芽孢杆菌菌株IMAU80218 / 厚壁菌门 | GU125635.1 | 99.5 | 短短芽孢种 | 94.3±5.8 |
| F14 | 流石 | 微小杆菌属。 ZWU0009 /厚壁菌门 | JX292087.1 | 99.3 | 种微小杆 | 96.5±2.5 |
| M7 | 潮湿的钟乳石 | 短杆菌 。 N78 /放线菌 | HQ188605 | 97.6 | 短物种 | 97.5±2.0 |
| M14 | 潮湿的钟乳石 | Kocuria rhizophila应变Ag09 /放线菌 | EU554435.1 | 100 | Kocuria种 | 95.2±4.1 |
| M15 | 潮湿的钟乳石 | 短杆菌 。 MN3-3 /放线菌 | JQ396535.1 | 99.5 | 短杆菌种B | 92.1±4.9 |
| R4 | 石钟乳滴 | Aminobacter藻。 KC-EP-S4 /α-变形菌 | FJ711220.1 | 99.9 | Aminobacter物种 | 95.4±2.7 |
| F5 | 流石 | 睾丸酮丛毛单应变NBRC 12047 /β-变形菌 | AB680219 | 100 | 丛毛单品种 | 96.4±2.6 |
| R8 | 石钟乳滴 | Curvibacter的美/β-变形菌 | AB680705 | 97.0 | Curvibacter </ em>的种类 | 89.4±7.8 |
| F8 | 流石 | 莫拉菌。 19.2 KSS /γ-变形菌 | HE575924.1 | 99.9 | 莫拉种 | 92.6±4.9 |
细菌从卡切内岩洞,AZ,美国孤立和利用16S rDNA序列分析鉴定。两个引物,27F(5'AGA GTT TGA TCC TGG CTC AG 3')和1492R(5'TAC GGT TAC CTT GTT ACG ACT T第3'),被用来获得近1400碱基对长度的16S rRNA基因序列。
b根据BLAST搜索的NCBI数据库。
Ç报导的值是30次重复(3次生物学重复的每个有10技术重复)±1标准偏差的平均相关系数。
表1.细菌分离株在演示中使用。
| BioNumerics关键 | 山顶类/潜在生物标志物(DA) |
| 芽孢杆菌物种 | 2152.5,2224.9,2595.8,2894.9,2921.3,3380.5,3496.3,3515.0,3733.5,4302.0,4340.0,4385.8,4763.9,4910.6,5189.4,5227.0,5634.6,5769.6,5892.8,6301.4,6756.2,6789.4,6990.3,7029.5,7466.3 |
| 芽孢杆菌 B | 2152.5,2941.2,3196.9,3262.7,3352.9,3420.8,3733.5,3925.2,4302.0,4339.9,4629.2,4713.4,4859.3,4900.6,5189.4,5227.0,5541.8,5878.7,6388.8,6524.0,6704.5,6838.8,7142.7,7317.5,7466.3, 7849.1,9263.9,9721.2 |
| 芽孢杆菌 Ç | 2588.0,3361.8,4330.4,5173.0,5847.6,6332.0,6524.0,6720.3 |
| 芽孢杆菌 ð | 2152.5,2894.9,3420.8,4302.0,4339。9,4629.2,5189.4,5448.4,5878.7,6388.8,6838.8,6931.1,7849.1 |
| Ë种芽孢杆菌 | 2152.5,2224.9,2941.2,3180.1,3380.5,4302.0,4339.9,4705.3,5878.7,6356.6,6735.1,6756.2 |
| 芽孢杆菌 ˚F | 3308.6,3367.8,3567.5,4279.7,4489.2,4629.2,4727.9,4751.7,5067.7,6614.7,6919.7,7130.9 |
| 短短芽孢种 | 2133.3,2611.0,4263.3,4302.0,4859.3,4900.6,5080.2,5219.0,5847.6,6775.7,7529.4,9721.2 |
| 种微小杆 | 2588.0,3053.3,3420.8,3695.5,4263.3,5133.1,5173.0,5248.8,6104.8,6605.3,6804.4,6838.8,7390.2 |
| 短物种 | 3053.3,6104.8,6146.5 |
| Kocuria种 | 3080.0,4366.6,5080.2,5163.8,5207.1,5892.8,6160.0,6197.5,6445.0,7433.7 |
| 短杆菌种B | 3222.8,3330.4,3367.8,4330.4,4350.4,4795.3,4995.7,5731.6,6445.0,6735.1,7487.3 |
| Aminobacter物种 | 2133.3,2562.4,3361.8,3410.4,4289.2,4629.2,4662.0,4869.8,6064.1,6221.0,6720.3,6789.4,6818.8,7216.1,7447.4 |
| 丛毛单品种 | 2806.0,2921.4,3246.5,4350.4,4727.9,5607.5,5666.3,6221.0,6488.3,7317.5,9362.6 |
| Curvibacter种 | 2868.6,3453.2,4319.8,5133.1,6292.4,6903.4,7433.7 |
| 莫拉种 | 3011.2,5698.0,6720.3,7064.8,7366.6 |
表2.峰值类(潜在生物标志物)(道尔顿)为每个物种所定义。
补充图1的质谱中的第主成分分析(PCA)Ë技术复制水平(A)和峰类(B)中。如在图2中使用的颜色被编码为相同的颜色。
补充图2.基于聚类双向选定峰的类的图示。具有相同标记峰类着色用相同的颜色。
补充图的基础上在BioNumerics自定义数据库识别项目3.交叉验证结果。
基于使用自定义数据库峰匹配补充图4.细菌鉴定。
讨论
该演示展示了表征和鉴定用MALDI-TOF MS和一个自定义数据库细菌的详细过程。相较于传统的分子生物学方法,例如,16S rDNA序列分析,MALDI-TOF MS的基于指纹的方法促进更快速鉴定多样的细菌。因为它的耐用性,这种技术被广泛用于从环境和临床环境1,14-16表征细菌,病毒,真菌和酵母。此外,MALDI-TOF MS已经报道,得到,在一些情况下,更高分类分辨率1。例如,B。藻。 A,B,D和E,虽然倾向( 图2)聚集在一起,被清楚地分离和不同B. SP的频谱之间的相似性。小于80%( 图2)。与此相反,这些菌株的16S rDNA序列具有高的相似性,这不能被用来区分这些菌株在种的水平。 B. sp。的16S rDNA序列。 B和D有99%的相似性的基础上的多个对准的分析,而B. sp。的序列。 A和E显示出95%和96%的相似性,分别给B. sp。的序列。 B和D离群还观察到。例如,B。藻。 C和F分组远离其他B.藻。 ( 图2)。离群株的出现表明,质谱的聚类分析并不一定建立亲缘关系。获得从该隔离的环境也可能影响质谱聚类。例如, 短杆菌种B以及从潮湿的钟乳石和 B菌中分离Kocuria种。 F,可以是从钟乳石滴分离倾向于聚集在一起( 表1,图2),但还需要进一步研究,以检查是否这是在分离和提纯的较大集合观察阿泰。
这个库为基础的技术也有一定的局限性。表征通常是基于数据库。目前的商业数据库主要由细菌菌株,特别是那些致病的。这些商业数据库是最有用的临床微生物学实验室中的设置。为了表征环境分离以及病毒,真菌和酵母,自定义数据库,需要使用大应变的集合来构建。在后续的分析中使用的参数也可以根据需要进行优化,以提高生物分类的分辨率,特别是在亚种和应变水平。例如,在S:N用于峰检测在这个示范为10,该值是适当的物种级别标识,但对于应变水平鉴定,这个值可能需要被降低。由于这些处理参数以及数据处理的工作流程,有时用户定义在许多软件包,例如,ClinProTools和BionumeRICS,参数值和选择适当的工作流的优化可能需要优化数据的分析。在这个演示中,峰匹配参数,用于识别项目的阈值,并交叉验证所有需要优化,以提高正确识别率。要找到一个方法和/或过程来优化这些参数是极大的兴趣,我们的实验室。例如,一种方法可能涉及统计因子设计,我们最近用于优化MALDI-TOF自动数据采集17。另外未来的应用和增强的MALDI-TOF MS的基于微生物指纹包括建造广泛可用的,较大的环境的细菌和/或非细菌微生物以及混合培养18和微生物群落的表征的数据库。
披露声明
Authors Vranckx and Janssens are employees of Applied Maths NV, the manufacturer of data analysis software used in this video. Applied Maths NV provided select software modules highlighted in this video as well as a portion of the publication costs associated with this video.
致谢
This work was supported by the New College of Interdisciplinary Arts and Sciences at Arizona State University, Applied Maths NV, and by the National Science Foundation (ROA Supplement to Award No. MCB0604300). Any opinions, findings, and conclusions or recommendations expressed in this material are those of the author(s) and do not necessarily reflect the views of the National Science Foundation.
材料
| Name | Company | Catalog Number | Comments |
| α-cyano-4-hydroxy-cinnamic acid | ACROS Organics | 163440050 | ≥ 97%, CAS 28168-41-8 |
| MALDI calibration kit | Aigma-Aldrich | MSCAL1-1KT | This kit also contains acetonitrile, trifluoroacetic acid , sinapinic acid, etc. |
| MALDI target plate | Bruker Daltonics | 280800 | Polished Steel |
| Bruker Microflex LRF MALDI-TOF mass spectrometer | Bruker Daltonics | ||
| Bruker FlexControl software | Bruker Daltonics | version 3.0 | |
| Bruker FlexAnalysis software | Bruker Daltonics | version 3.0 | |
| Bionumerics software | Applied Maths | version 7.1 |
参考文献
- Sandrin, T. R., Goldstein, J. E., Schumaker, S. MALDI TOF MS profiling of bacteria at the strain level: A review. Mass Spectrom Rev. 32 (3), 188-217 (2013).
- Siegrist, T. J., et al. Discrimination and characterization of environmental strains of Escherichia coli by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry (MALDI-TOF-MS). J Microbiol Meth. 68 (3), 554-562 (2007).
- Goldstein, J. E., Zhang, L., Borror, C. M., Rago, J. V., Sandrin, T. R. Culture conditions and sample preparation methods affect spectrum quality and reproducibility during profiling of Staphylococcus aureus with matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Lett Appl Microbiol. 57 (2), 144-150 (2013).
- Benagli, C., et al. A rapid MALDI-TOF MS identification database at genospecies level for clinical and environmental Aeromonas strains. Plos One. 7 (10), (2012).
- Sauer, S., Kliem, M. Mass spectrometry tools for the classification and identification of bacteria. Nat Rev Microbiol. 8 (1), 74-82 (2010).
- Bohme, K., et al. SpectraBank: An open access tool for rapid microbial identification by MALDI-TOF MS fingerprinting. Electrophoresis. 33 (14), 2138-2142 (2012).
- Walker, J., Fox, A. J., Edwards-Jones, V., Gordon, D. B. Intact cell mass spectrometry (ICMS) used to type methicillin-resistant Staphylococcus aureus: media effects and inter-laboratory reproducibility. J Microbiol Meth. 48 (2-3), 117-126 (2002).
- Ruelle, V., El Moualij, B., Zorzi, W., Ledent, P., De Pauw, E. Rapid identification of environmental bacterial strains by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Commun Mass Sp. 18 (18), 2013-2019 (2004).
- Sedo, O., Sedlacek, I., Zdrahal, Z. Sample Preparation Methods for Maldi-MS Profiling of Bacteria. Mass Spectrom Rev. 30 (3), 417-434 (2011).
- Swatkoski, S., Russell, S., Edwards, N., Fenselau, C. Analysis of a model virus using residue-specific chemical cleavage and MALDI-TOF mass spectrometry. Anal Chem. 79 (2), 654-658 (2007).
- Freiwald, A., Sauer, S. Phylogenetic classification and identification of bacteria by mass spectrometry. Nat Protoc. 4 (5), 732-742 (2009).
- Drevinek, M., Dresler, J., Klimentova, J., Pisa, L., Hubalek, M. Evaluation of sample preparation methods for MALDI-TOF MS identification of highly dangerous bacteria. Lett Appl Microbiol. 55 (1), 40-46 (2012).
- Lasch, P., et al. MALDI-TOF mass spectrometry compatible inactivation method for highly pathogenic microbial cells and spores. Anal Chem. 80 (6), 2026-2034 (2008).
- Usbeck, J. C., Kern, C. C., Vogel, R. F., Behr, J. Optimization of experimental and modelling parameters for the differentiation of beverage spoiling yeasts by Matrix-Assisted-Laser-Desorption/Ionization Time-of-Flight Mass Spectrometry (MALDI-TOF MS) in response to varying growth conditions. Food Microbiol. 36 (2), 379-387 (2013).
- Del Chierico, F., et al. MALDI-TOF MS proteomic phenotyping of filamentous and other fungi from clinical origin. J Proteomics. 75 (11), 3314-3330 (2012).
- Vitale, R., Roine, E., Bamford, D. H., Corcelli, A. Lipid fingerprints of intact viruses by MALDI-TOF/mass spectrometry. Bba-Mol Cell Biol L. 1831 (4), 872-879 (2013).
- Zhang, L., Borror, C. M., Sandrin, T. R. A designed experiments approach to optimization of automated data acquisition during characterization of bacteria with MALDI-TOF mass spectrometry. Plos One. 9 (3), (2014).
- Christner, M., Rohde, H., Wolters, M., Sobottka, I., Wegscheider, K., Aepfelbacher, M. Rapid identification of bacteria from positive blood culture bottles by use of matrix-assisted laser desorption-ionization time of flight mass spectrometry fingerprinting. J ClinMicrobiol. 48 (5), 1584-1591 (2010).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。