Method Article
استخدام MALDI-TOF الطيف الكتلي وقاعدة بيانات مخصصة لتوصيف البكتيريا الأصلية إلى فريد كهف البيئة (Kartchner الكهوف، AZ، الولايات المتحدة الأمريكية)
In This Article
Summary
This work details procedures for rapid identification of bacteria using MALDI-TOF MS. The identification procedures include spectrum acquisition, database construction, and follow up analyses. Two identification methods, similarity coefficient-based and biomarker-based methods, are presented.
Abstract
MALDI-TOF mass spectrometry has been shown to be a rapid and reliable tool for identification of bacteria at the genus and species, and in some cases, strain levels. Commercially available and open source software tools have been developed to facilitate identification; however, no universal/standardized data analysis pipeline has been described in the literature. Here, we provide a comprehensive and detailed demonstration of bacterial identification procedures using a MALDI-TOF mass spectrometer. Mass spectra were collected from 15 diverse bacteria isolated from Kartchner Caverns, AZ, USA, and identified by 16S rDNA sequencing. Databases were constructed in BioNumerics 7.1. Follow-up analyses of mass spectra were performed, including cluster analyses, peak matching, and statistical analyses. Identification was performed using blind-coded samples randomly selected from these 15 bacteria. Two identification methods are presented: similarity coefficient-based and biomarker-based methods. Results show that both identification methods can identify the bacteria to the species level.
Introduction
Matrix-assisted laser desorption/ionization time-of-flight (MALDI-TOF) mass spectrometry (MS) has been shown to be a rapid and reliable tool for identification of bacteria at the genus, species, and in some cases, strain levels1-4. MALDI-TOF MS ionizes biological molecules (typically proteins) that originate from cell surfaces, intracellular membranes, and ribosomes from bacterial whole cells or protein extracts1,5. The resulting peaks form characteristic patterns or “fingerprints” of the bacteria analyzed1. Identification of bacteria is based on these mass-to-charge “fingerprints”.
Two of the most commonly used identification strategies are library-based and bioinformatics-based strategies1. Library-based approaches involve comparing the mass spectra of unknowns to previously collected mass spectra of known bacteria in databases/libraries for identification. Commercially available software, such as BioNumerics, Biotyper, and SARAMIS software packages, as well as open source software tools, such as SpectraBank6, are available to facilitate the comparison and quantification of similarity between mass spectra of unknowns and reference bacteria. Bioinformatics-based approaches usually rely on fully sequenced genomes of bacteria for identification. In contrast to library-based approaches which do not involve identification of the biological nature of particular peaks, bioinformatics-based approaches involve protein identification1.
The majority of recent MALDI fingerprint-based studies have used library-based approaches to identify bacteria1. Library-based approaches require construction of databases and comparison of the similarity between mass spectra. Studies show that many experimental procedures, such as medium3,7, cultivation time8, sample preparation method3, and matrix used9, affect the mass spectra obtained. Furthermore, some closely-related species and strains generate spectra with only subtle differences. Thus, library-based approaches require rigorously standardized procedures to generate highly reproducible mass spectra between replicates. Minor variations in protocols may compromise the efficacy of identification, especially at the subspecies and strain levels1,3,10. However, neither manufacturer-provided reference databases nor reported custom databases include visually documented procedures for database construction and/or application of a data analysis pipeline. For this reason, the objective of this work was to develop, apply, and demonstrate a comprehensive and detailed procedure for library-based bacterial identification using MALDI-TOF MS.
In this demonstration, mass spectra of 15 bacteria isolated from a karstic environment (Kartchner Cavern, AZ, USA) were collected and imported into software to construct a model database. Data processing and the analysis pipeline were detailed using the model database. Finally, mass spectra of blind-coded bacteria which were randomly selected from these 15 bacteria were collected again and compared to the reference spectra in the model database for identification. Results show that bacteria can be correctly identified either based on similarity coefficients or potential biomarkers/peak classes.
Protocol
الحذر: بكتيريا مجهولة الهوية من أي بيئة قد تكون المسببة للأمراض ويجب التعامل معها بحذر باستخدام بروتوكولات المناسبة للسلامة الأحيائية. يجب إجراء عمل مع الثقافات الحية في مجلس الوزراء من الدرجة الثانية للسلامة الأحيائية باستخدام السلامة البيولوجية المستوى 2 (BSL-2) الإجراءات. مزيد من المعلومات حول 2 BSL-إجراءات متاحة في بعنوان دليل CDC / NIH، "السلامة الأحيائية في الميكروبيولوجية الطبية الحيوية والمختبرات،" صفحات 33-38. والوثيقة متاحة على الانترنت في http://www.cdc.gov/biosafety/publications/bmbl5/BMBL.pdf . معدات الحماية الشخصية المناسبة (PPE)، بما في ذلك المختبر المعاطف / العباءات، ونظارات السلامة، والنتريل قفازات اللاتكس أو، يجب أن ترتديه. يجب اتباع الممارسات والاحتياطات القياسية الميكروبيولوجية، ويجب التخلص من النفايات biohazardous بشكل مناسب.
تم عزل البكتيريا المستخدمة في هذه التظاهرة من Kartchner الكهوف،AZ، الولايات المتحدة الأمريكية، من أربع بيئات، بما في ذلك speleothem تجف، حجر التدفق، speleothem رطبة وبالتنقيط الهوابط (الجدول 1). تم تحديد جميع العزلات التي كتبها 16S rDNA التسلسل والاحتفاظ بها في -80 درجة مئوية في 25٪ الجلسرين-R 2 B المتوسطة. تم الانتهاء من جميع التجارب في RT.
ملاحظة: نوصي باستخدام نفس العينة طريقة التحضير للحصول على الأطياف الشامل لبناء قاعدة البيانات والأطياف الشامل من المجاهيل. وقد تبين طريقة تحضير عينات سبق أن تؤثر على نوعية الطيف واستنساخ 3. باستخدام طريقة مختلفة وإعداد العينات قد يسبب تحديد غير صحيح من المجاهيل، القرار التصنيفية وخصوصا عندما العالي (على سبيل المثال، على مستوى السلالة) هو المطلوب.
1. ترسب على MALDI الهدف
الحذر: عدة بروتوكولات للحصول على مستخلصات البروتين تتطلب استخدام الأحماض والمذيبات العضوية التي يجب أن تستخدم وفقا ارشدelines والمعلومات الواردة في ورقة بيانات سلامة المواد الخاصة بكل منها (MSDS). المناسبة PPE يجب أن ترتديه وسوف تختلف بناء على نوع وحجم المواد الكيميائية المستخدمة (على سبيل المثال، والمعاطف مختبر / العباءات، والقفازات، ونظارات السلامة، وحماية الجهاز التنفسي يجب أن تستخدم عند التعامل مع كميات كبيرة من السامة، والمذيبات القابلة للاشتعال، مثل الأسيتونتريل، والأحماض تآكل، مثل الأحماض الفورميك وtrifluoroacetic).
- إيداع 1 ميكرولتر استخراج البروتين لا تحتوي على خلايا قابلة للحياة (تم الحصول عليها باستخدام المناسبة والبروتوكولات التي سبق وصفها 11-13) على الفولاذ المقاوم للصدأ لوحة الهدف MALDI واتركه حتى يجف. تراكب استخراج البروتين المجفف مع 1 ميكرولتر حل مصفوفة (α-cyano-4-هيدروكسي-السيناميك حل حامض)، واتركه حتى يجف.
- لكل تكرار البيولوجي، بقعة عدد مناسب من مكررات الفنية (5 إلى 20 مكررات الفنية). هنا، بقعة 10 مكررات الفنية لكل تكرار البيولوجي و3 مكررات البيولوجية ثيحرث إعدادها.
ملاحظة: نوصي باستخدام مصقول MALDI وحة الهدف الصلب عند استخدام استخراج البروتين طريقة تحضير العينة. باستخدام لوحات الهدف الصلب الأرض قد يسبب انتشار وخلط غير مقصود من عينات مختلفة خارج الآبار عينة الفردية. - إيداع 1 ميكرولتر مستوى calibrant على لوحة الهدف واتركه حتى يجف. تراكب مع 1 ميكرولتر حل المصفوفة واتركه حتى يجف.
- إيداع 2 ميكرولتر حل مصفوفة على لوحة الهدف كعنصر تحكم السلبية.
2. قداس الأطياف اكتساب
- استخدام مطياف الكتلة MALDI-TOF مجهزة ليزر النيتروجين (λ = 337 نانومتر) وتشغيلها باستخدام البرمجيات بروكر FlexControl.
- جمع كل الطيف الشامل في الوضع الخطي الإيجابية التي كتبها تراكم 500 طلقات الليزر في 100 الزيادات النار. تعيين الجهد مصدر أيون 1 إلى 20 كيلو فولت. مصدر أيون 2 الجهد إلى 18.15 كيلو فولت. والجهد العدسة إلى 9.05 كيلو فولت. لاحظ أن هذه الثوابت هي صك SPECIالمرورية وقد تتطلب التعديل على الصكوك الأخرى للحصول على أفضل النتائج.
- تعيين نطاق الكتلة إلى تهمة لتقييم الطيف الآلي من 2 إلى 20 كيلو دالتون لكل تهمة. استخدام خوارزمية الكشف الذروة النقطه الوسطى. تعيين دقة عتبة الحد الأدنى عند 100 دا. تعيين إشارة إلى نسبة الضوضاء (S: N) العتبة 2. تعيين عتبة كثافة الحد الأدنى عند 100.
البناء 3. قاعدة البيانات
- تصميم قاعدة البيانات
- إنشاء قاعدة بيانات جديدة في BioNumerics 7.1 باستخدام "معالج قاعدة بيانات جديد".
- إنشاء نوع التجربة الطيف، على سبيل المثال، MALDI، وذلك باستخدام الأوامر في "أنواع تجربة" لوحة.
- خلق مستويات استخدام "قاعدة بيانات لوحة تصميم". إضافة مستويات جديدة باستخدام "مستوى> إضافة مستوى جديد ..." الأمر في القائمة "قاعدة بيانات". هنا، خلق "الأنواع" مستوى، مستوى "تكرار البيولوجي" "و" تكرار الفني "مستوى، respectivاعل.
- استيراد وتجهيزها الخام أطياف الكتلة
- تصدير الأطياف كتلة الخام كما ملفات .txt باستخدام FlexAnalysis بالضغط على "تصدير> الطيف الشامل" الأوامر في القائمة "ملف".
- استيراد أطياف الكتلة الخام (ملفات .txt) في قاعدة البيانات في مستوى مكررات التقنية.
- Preprocess أطياف الكتلة الخام.
- استيراد وإعادة تشكيله (باستخدام خوارزمية المناسب من الدرجة الثانية).
- إجراء الطرح الأساس (مع قرص المتداول مع حجم 50 نقطة).
- حساب الضوضاء [المستمر المويجات تحول (CWT)]، على نحو سلس (كايزر النافذة مع حجم إطار 20 نقطة وبيتا من 10 نقطة)، وإجراء الطرح خط الأساس الثاني (المتداول القرص مع حجم 200 نقطة).
- كشف قمم [CWT مع الحد الأدنى من إشارة إلى نسبة الضوضاء (S: N) من 10].
- بعد تجهيزها، وتوفير أنماط مميزة من كل الطيف الشامل، مثل قوائم الذروة التي تحتوي الذروةالأحجام، وشدة الذروة، S: N، وما إلى ذلك، في قاعدة البيانات.
- خلق مركب أطياف الكتلة
- خلق أطياف مركب من أطياف preprocessed باستخدام "لخص ..." الأمر في القائمة "تحليل". اختيار "تكرار البيولوجي" كما المستوى المستهدف.
- هنا، والجمع بين أطياف الشامل من 10 مكررات الفنية من نفس المستعمرة أن تسفر عن الطيف الكتلي مركب لتلك المستعمرة، مما أدى إلى ثلاثة أطياف الكتلة مركب لأنه وجد عزلة على مستوى "تكرار البيولوجي".
- هنا، تلخيص أطياف مركب من ثلاثة إلى إنشاء واحدة الطيف مركب لذلك عزل على مستوى "الأنواع".
ملاحظة: الطيف المركب هو متوسط نقطة بنقطة من مكررات التقنية. يعيد مع تشابه (ارتباط بيرسون) لمتوسط أقل من 95٪ (الإعداد الافتراضي) مستثناة من مركب. ويطلق على قمم على أطياف مركب فقط إذا كانت موجودة في 75٪ (الإعداد الافتراضي) من مكررات المدرجة. لمكررات البيولوجية، كانت هذه الإعدادات 90 و 60٪ على التوالي.
4. تحليل قداس الطيف البيانات
- حدد الإدخالات في قاعدة البيانات وإنشاء مقارنة بالضغط على "إنشاء المقارنة الجديدة" القيادة في "مقارنات" لوحة.
- هنا، استخدم أطياف الشامل في "تكرار الفني" و / أو مستويات "تكرار البيولوجي" لإظهار المقارنات والتحليلات.
- التحليل العنقودي القائم على التشابه والقياس متعدد الأبعاد (MDS)
- إنشاء مجموعات مع الألوان. حدد البيولوجي أطياف الكتلة مركب ثلاثة وانقر على "إنشاء مجموعة جديدة من اختيار" الأمر في القائمة "المجموعات" لإنشاء مجموعة لعزل المقابل. تعيين اللون المستخدم تلقائيا لهذه الأطياف الشامل الثلاثة.
- بدلا من ذلك، وتحديد ولايات الميدان مع الألوان المقابلة باستخدام الأوامر في "إدخالات قاعدة بيانات "لوحة مثل أن أي تجمع على أساس هذا المجال المحدد يستخدم نفس اللون محددة لهذه المجموعة.
- أداء التحليل العنقودي. انقر على "حساب التحليل العنقودي" الأمر في القائمة "تجميع". على مقارنة إعدادات صفحة 1، حدد "ارتباط بيرسون"، وترك غيرها من المعالم كما الافتراضي. في الصفحة 2، حدد "UPGMA". ثم انقر فوق "إنهاء".
- الحصول على مؤامرة MDS باستخدام "التحجيم متعدد الأبعاد ..." الأمر في القائمة "الإحصاء".
- مطابقة الذروة
- انقر على نوع الطيف "MALDI" في "تجارب" لوحة. ثم حدد "تخطيط> مشاهدة الصورة". وتظهر الأطياف كما العصابات هلام.
- أداء مطابقة ذروة باستخدام "هل مطابقة ذروة" الأمر في القائمة "الأطياف".
- تحديد الطبقات الذروة
- لتحليل المكون الرئيسي (PCA). تسليط الضوء على "MALDI "نوع التجربة في" التجريبية "لوحة واستخدام" تحليل مكونات الرئيسية ... "القيادة في" القائمة الإحصائية "لتنفيذ PCA.
- أداء اتجاهين المجموعات. انقر على "الاحصائيات> التعدين ماتريكس" ... في "مقارنة" نافذة. شدة القمم تتناسب مع الطبقات الذروة يتم تمثيل ألوان مختلفة (خريطة الحرارة).
5. تحديد البكتيريا مع قاعدة البيانات المخصصة
- تشابه طريقة المستندة إلى معامل
- إنشاء المقارنة وتوليد dendrogram على أساس أطياف الشامل على مستوى "تكرار الفني" كما هو موضح في الخطوة 4.3.3. حفظ dendrogram للمقارنة التشابه.
- تحديد الطيف الكتلي غير معروف، وانقر على "تحليل> تحديد الإدخالات المحددة". يظهر مربع الحوار تحديد الهوية.
- حدد "مقارنة على أساس" نوع المصنف (أو classif المخزنةهيئة الإنصاف والمصالحة) وانقر على "التالي". في الصفحة التالية، اختر dendrogram حفظها وعلى سبيل المقارنة المرجعية ومن ثم انقر فوق "التالي".
- اختيار "التشابه الأساسي" كطريقة تحديد ثم انقر فوق "التالي".
- اختيار "التشابه الأقصى"، كما طريقة التهديف. اكتب في قيم حدية مناسبة والقيم الفرق الحد الأدنى لكل معلمة ومن ثم انقر فوق "التالي".
- وبمجرد الانتهاء من الحسابات، يظهر إطار تحديد الهوية. في "نتائج" لوحة، يتم سرد أعضاء من قاعدة البيانات التي تطابق أفضل المجهول.
- حفظ المشروع تحديد والتحقق من صحة الاستدلال باستخدام "عبر التحقق من صحة التحليل" القيادة في لوحة "مشروع تحديد".
- محتمل طريقة القائم على العلامات البيولوجية
- تحديد فئات الذروة. في الإطار "مصفوفة تعدين"، حدد مجموعات من قمم تقاسم الخصائص المشتركة وتحديد هذه القمم كما SPECIدروس المرورية الذروة (المؤشرات الحيوية المحتملة) باستخدام "أطياف> إدارة أنواع الطبقة الذروة ..." في "نافذة مقارنة".
- هنا، وتحديد الطبقات الذروة محددة لعزل لجميع العزلات 15 لكل منهما.
- حدد أطياف الشامل من المجاهيل وتطابق قمم هذه الأطياف إلى الطبقات الذروة محددة كما هو موضح سابقا.
النتائج
قواعد البيانات التي شيدت في هذه المظاهرة كان أربعة مستويات، من أعلى إلى أدنى مستوى، بما في ذلك "جميع المستويات"، "الأنواع"، "تكرار البيولوجي" و "تكرار الفني"، على التوالي (الشكل 1A). وتضمن مستوى "التقني تكرار" جميع أطياف preprocessed من مكررات التقنية. و"تكرار البيولوجي" والمستويات "الأنواع" احتواء مركب (ملخص) الأطياف. "جميع المستويات" احتوت على جميع الأطياف تكرار الفني وكذلك جميع أطياف مركب.
وترد الإجراءات الطيف التلخيص في الشكل 1 باستخدام قمم التمثيلية. يظهر كل الطيف الكتلي عضو كخط رمادي رقيقة. ويمثل الطيف مركب كخط الملونة باللون الأحمر. يتم وضع علامة القمم المجاورة مع لون مختلف للسماح أسهل الفحص البصري (الشكل 1B).
تم حساب = "jove_content"> واستنساخ أطياف الشامل لل30 مكررات (ثلاثة مكررات البيولوجية، ولكل منها 10 مكررات الفنية) وترد في الجدول 1. وكان أعلى استنساخ 98.0 ± 1.4 بالنسبة للأنواع بكتيريا B، وأدناها كان استنساخ 89.4 ± 7.8 لCurvibacter الأنواع (الجدول 1).
التحليل العنقودي في التصور مستوى تكرار البيولوجي سهلت من الهيكل الهرمي في البيانات أطياف الإعلام المعقدة. كما هو مبين في الشكل 2، مكررات البيولوجية تتجمع معا، و 15 نوعا من البكتيريا تشكيل 15 مجموعات. وثيقي الصلة، على سبيل المثال، B. ليرة سورية. A، B، D، E و، تميل إلى التجمع معا. ومع ذلك، القيم المتطرفة، على سبيل المثال، B. ليرة سورية. C وF، وقد لوحظت أيضا. وترد المؤامرات MDS على أساس أطياف الشامل على المستويات "التقنية والبيولوجية" في الشكل (3). MDS صحققت الكثير واضح، 3-D التصور من أوجه التشابه بين أطياف هذه البكتيريا. كلا مكررات الفنية ومكررات البيولوجية أظهرت تجمع مماثل (الشكل 3 ألف وباء).
وقد استخدم مطابقة ذروة لتمييز مجموعات من القمم في أطياف الشامل. المعلمات مطابقة الذروة، بما في ذلك التسامح ثابت (النقاط على محور س)، والتسامح الخطي (جزء في المليون) ومعدل اكتشاف ذروة تحتاج إلى أن تكون محددة من قبل المستخدم. التسامح المستمر والتسامح الخطية هي العوامل المستخدمة لحساب التسامح موقف القمم باستخدام المعادلة: موقف التسامح = التسامح ثابت + التسامح الخطية × م / ض. مع زيادة م / ض، على أهمية التسامح المستمر يقلل. معدل اكتشاف ذروة يعني أن إلا إذا تم العثور على الذروة في هذا المنصب لأكثر من نسبة محددة من الأطياف، يرصد فئة الذروة. ذروة على أنماط واحد أو أكثر تمثل فئة الذروة. على سبيل المثال، إذا كان EQUA معدل اكتشاف الذروةليرة سورية 10٪، وفئة الذروة يمكن أن يتم إلا إذا كان أكثر من 10٪ من أطياف لها قمم في هذا المنصب. ويستثنى من هذا قمم الانتشار المنخفض (عادة قمم الضجيج) في مجموعة مع مكررات التقنية. إذا كان قائما على مجموعة أطياف مركب من مكررات البيولوجية، وهذا العدد قد تحتاج إلى أن تكون أقل كما تم بالفعل تصفية قمم انتشار منخفضة من خلال إنشاء أطياف مركب. في هذه المظاهرة، تم إجراء مطابقة ذروة باستخدام أطياف الشامل على مستوى "تكرار الفني" وكانت القيم من هذه المعلمات 1.9، 550 و 10٪ على التوالي. استنادا إلى معايير محددة، واعتبرت القمم كما مطابقة أو غير مطابقة، مما أدى إلى مجموعات مختلفة الذروة. ويرد مثال من نتائج التصفح الذروة في الشكل 4 باستخدام 30 مكررات لعزل واحد (الأنواع العصوية A). وتصور نتائج مطابقة كجدول فيها شدة الخام موجودة والألوان. الأزرق يدل على كثافة منخفضة والأحمر يشير ط عاليةntensity. وبناء على نتائج مطابقة الذروة، يمكن للمستخدمين تحديد الطبقات الذروة التي تسهل يحلل المتابعة.
كلا تحليل المكون الرئيسي (PCA) (الشكل التكميلي 1) واتجاهين تجميع يمكن استخدامها لتحليل الطبقات الذروة المعقدة. ويرد تمثيلية في اتجاهين نتيجة التجميع باستخدام أطياف الشامل على مستوى "تكرار الفني" في الشكل 5. وتظهر اثنين من dendrograms. واحد هو القادم لم القيم / ض والآخر هو فوق إدخالات البكتيريا (الشكل 5). ومثل كثافة الذروة التي الألوان التي الأخضر يشير إلى كثافة منخفضة والأحمر يشير إلى كثافة عالية. على سبيل المثال، B. ليرة سورية. ألف وF مشاركة عدد قليل جدا من الطبقات الذروة مع B. ليرة سورية. B و D (الشكل 5). وأظهرت وثيقة الفحص أن B. ليرة سورية. يكون B و D أيضا مجموعات من الطبقات الذروة أنواع محددة (الشكل 5). هذه النتائج تشير إلى أن مجموعات الذروة محددة تبادل سيرت ويمكن تعريف الخصائص عين كما المؤشرات الحيوية المحتملة على مستوى الأنواع. على سبيل المثال، ثلاثة عشر مجموعات الذروة تابعة لB. ليرة سورية. وقد تم اختيار D والذي يعرف بأنه الطبقات الذروة (المؤشرات الحيوية المحتملة) من B. ليرة سورية. D، بما في ذلك 2152.5، 2894.9، 3420.8، 4302.0، 4339.9، 4629.2، 5189.4، 5448.4، 5878.7، 6388.8، 6838.8، 6931.1، 7849.1 و(الجدول 2). يمكن أن تظهر الطبقات الذروة من العزلات المختلفة في ألوان مختلفة (الشكل التكميلي 2). فصول الذروة محددة لكل عزلها تم جدولتها في الجدول 2. تم فحص الطبقات ذروة المعرفة مزيد يدويا للتأكد من أنها ظهرت في جميع مكررات الفنية مع الحد الأدنى من شدة 100 للاتحاد الافريقي وعلاوة على ذلك، يمكن أيضا أن يتم تخزين مجموعات فرعية من الطبقات الذروة لتسهيل توصيف البكتيريا في السلالة و / أو مستويات التوتر، على سبيل المثال، لتمييز السلالات المسببة للأمراض من سلالات غير المسببة للأمراض و / أو لفحص المضادات الحيوية المقاومة / الحساسية.
والأنف والحنجرة "> وفيما يتعلق الهوية، تم جمع أطياف الشامل من العزلات العمياء مرمزة وpreprocessed بنفس الطريقة كما أطياف الكتلة المرجعية في قواعد البيانات. لتحديد على أساس المقارنة بين معاملات التشابه، تم تحديد قيم المعلمة، بما في ذلك الحد الأقصى للتشابه في 95.0٪ ومتوسط التشابه (87٪). لم يتم تحديد الحد الأدنى للتشابه (أي تركت لحالها). وتم تعيين قيم الاختلاف الحد الأدنى إلى 5 لكل من أقصى التشابه ومتوسط التشابه. قد تحتاج هذه القيم ليكون الأمثل على مواصلة زيادة معدل التحديد الصحيح الشكل 6 يوضح نتائج تحديد بناء على مقارنات بين معاملات التشابه (الشكل 6). وتشير نتيجة مطابقة أن هذه البكتيريا أعمى مرمزة كان على الأرجح B. س. A. مشروع تحديد هذا على أساس المقارنة بين تم التحقق من صحة معامل التشابه مزيدا من عبر التحقق من صحة (الشكل التكميلي 3 ). تم اختبار التحقق من صحة عبر في تغطية 25٪. باستخدام تغطية أعلى، على سبيل المثال، 50٪ أو 100٪، ويمكن أن تزيد من الثقة لتحديد الهوية، ولكنه يأخذ وقتا أطول من ذلك بكثير لإكمال، وخاصة بالنسبة للقواعد البيانات الكبيرة. جميع الفئات اختبار لها ايجابيات حقيقية 100٪ والسلبيات الكاذبة 0٪ (الشكل التكميلي 3). ومن المثير للاهتمام، والمصادقة عبر لمشاريع الهوية على أساس المقارنة بين الطبقات الذروة هو أسرع بكثير من تلك القائمة على المقارنة بين معامل التشابه.ويمكن أيضا أن يكتمل تحديد على أساس مطابقة الدرجة الذروة (الشكل التكميلي 4). ومع ذلك لوحظ عدم التطابق من قمم (الشكل التكميلي 4). قد يكون راجعا إلى التحول الشامل الناتجة عن تبادل الأحماض الأمينية في البروتينات منها عدم التطابق. يمكن للقمم عدم مطابقة أيضا أن تكون القمم التي لا التمييزية على مستوى الأنواع ولكن هي محددة لهذه السلالة أو عزل. أخذت معا، سالنتائج اور تشير إلى أن كلا من وسائل تحديد الهوية - معامل التشابه والقائم على العلامات البيولوجية - يمكن التعرف بسهولة البكتيريا على مستوى الأنواع من بيئات الكارستية باستخدام إعداد العينات وحيازة الطيف، وتحليل البيانات سير العمل ووصف هنا.

الشكل 1. بناء قاعدة بيانات والأطياف التلخيص الشامل هيكل قواعد البيانات التي شيدت في هذه التظاهرة (A)؛ التوضيح الذروة تلخيص باستخدام قمم من 10 مكررات الفنية للAminobacter الأنواع A (B).
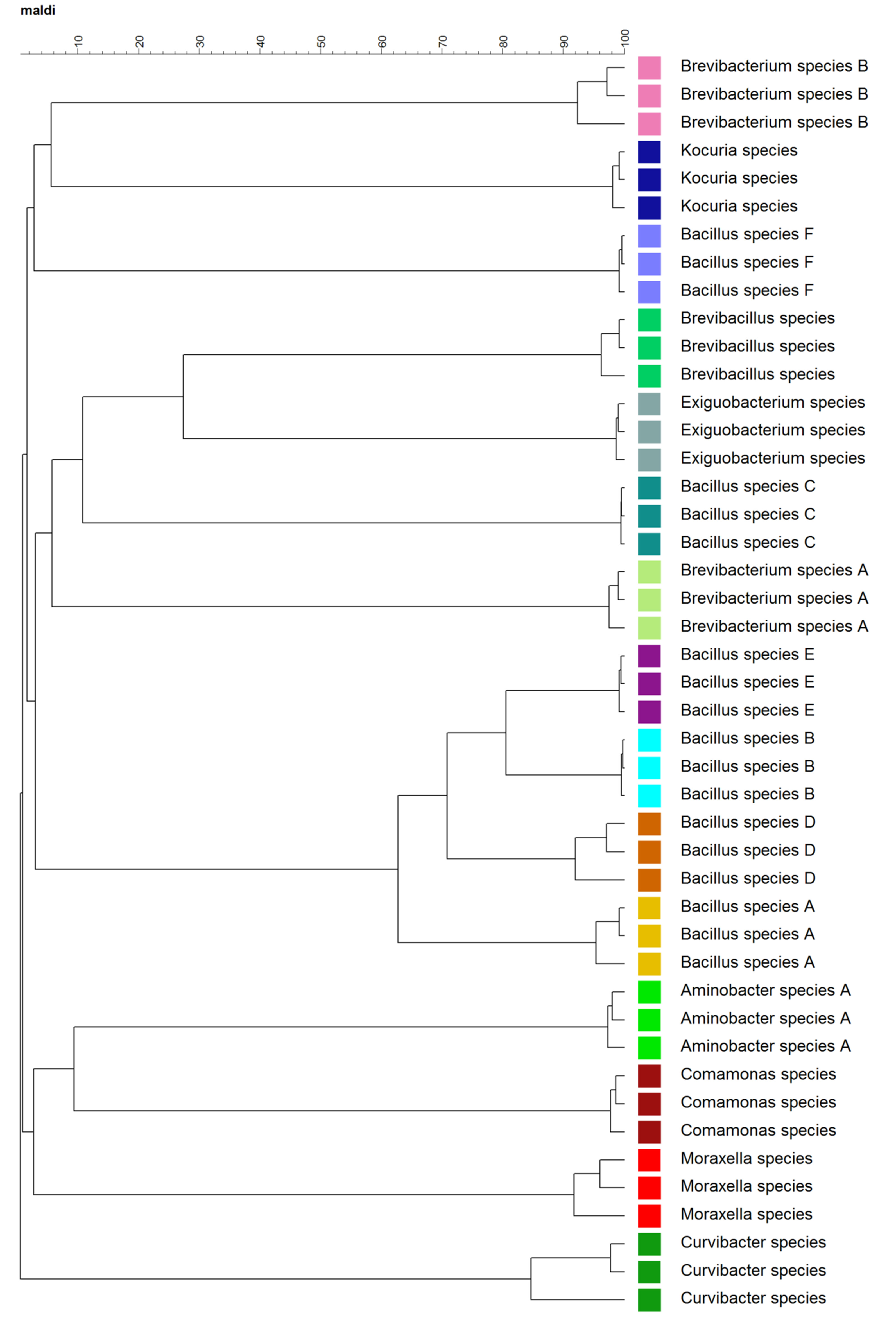
الشكل 2. Dendrogram من مركبأطياف الشامل على مستوى تكرار البيولوجي. ويتضمن مجموعة من البيانات أطياف 15 نوعا مختلفة مع ثلاثة أطياف مركب لكل الأنواع. تم ترميز كل الأنواع مع اللون.

الشكل 3. متعدد الأبعاد التحجيم (MDS) تمثيل الأطياف الشامل على مستوى تكرار التقني مع 30 الأطياف لكل نوع (A) ومستوى تكرار البيولوجي مع ثلاثة أطياف مركب لكل نوع (B). تم ترميز الألوان كما نفس الألوان كما تستخدم في الشكل 2.

الشكل 4. مثال على طاولة الذروة مطابقة. تم إنشاء الجدول على أساس أطياف الشامل الأنواع العصوية A في LEVE تكرار الفني ل. وكانت القيم من المعلمات مطابقة الذروة 1.9 للتسامح المستمر، 550 للتسامح الخطي و 10٪ للمعدل اكتشاف الذروة، على التوالي. الأزرق يشير إلى انخفاض كثافة الذروة والأحمر يشير إلى كثافة عالية الذروة. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم.

الشكل 5. مثال على اتجاهين المجموعات. تم إنشاؤها باستخدام الشكل أطياف الشامل على مستوى تكرار التقني. تم ترميز الألوان من العزلات كما نفس الألوان المستخدمة في الشكل (2). ويمثل شدة الذروة من الألوان، الأخضر معنى منخفضة الكثافة ومعنى الأحمر كثافة عالية. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم.
FO: المحافظة على together.within صفحة = "دائما"> 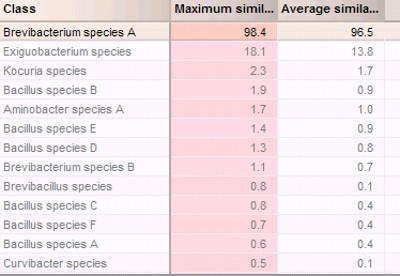
الرقم 6. تحديد بكتيريا على أساس المقارنة بين معامل التشابه باستخدام قاعدة بيانات مخصصة.
| ID ل | مصدر | اقرب النسبية ب / الأسرة في اللغات / الفئة | انضمام # (ب اقرب النسبية) | ٪ تشابه | مفتاح BioNumerics | استنساخ (٪) |
| D2 | speleothem الجافة | عصية س. E-257 / افيرميكوتس | FJ764776.1 | 98.8 | عصية الأنواع A | 94.9 ± 4.0 |
| D7 | speleot الجافةتنحنح | عصية س. GGC-P3 / افيرميكوتس | FJ348039.1 | 99.0 | الأنواع عصية B | 98.0 ± 1.4 |
| F1 | الحجر تدفق | عصية النياسين توتر M27 / افيرميكوتس | KC315764.1 | 99.2 | الأنواع عصية C | 96.5 ± 2.4 |
| F4 | الحجر تدفق | عصية س. GGC-P5A1 / افيرميكوتس | FJ348046.1 | 99.1 | الأنواع عصية D | 89.8 ± 8.8 |
| F9 | الحجر تدفق | عصية س. OSS 19 / افيرميكوتس | EU124558.1 | 99.4 | الأنواع عصية E | 96.5 ± 1.9 |
| R10 | بالتنقيط الهوابط. | عصية س. K1 / افيرميكوتس | GU968734.1 | 99.8 | الأنواع عصية F | 95.4 ± 3.9 |
| D11 | speleothem الجافة | Brevibacillus سلالة القصيرة IMAU80218 / افيرميكوتس | GU125635.1 | 99.5 | الأنواع Brevibacillus | 94.3 ± 5.8 |
| F14 | الحجر تدفق | س Exiguobacterium. ZWU0009 / افيرميكوتس | JX292087.1 | 99.3 | الأنواع Exiguobacterium | 96.5 ± 2.5 |
| M7 | speleothem رطبة | المتقاصرة ليرة سورية. N78 / شعاويات | HQ188605 | 97.6 | المتقاصرة الأنواع A | 97.5 ± 2.0 |
| M14 | speleothem رطبة | سلالة Kocuria rhizophila Ag09 / شعاويات | EU554435.1 | 100 | الأنواع Kocuria | 95.2 ± 4.1 |
| M15 | speleothem رطبة | المتقاصرة ليرة سورية. MN3-3 / شعاويات | JQ396535.1 | 99.5 | المتقاصرة الأنواع B | 92.1 ± 4.9 |
| R4 | بالتنقيط الهوابط. | Aminobacter ليرة سورية. KC-EP-S4 / α-متقلبات | FJ711220.1 | 99.9 | Aminobacter الأنواع A | 95.4 ± 2.7 |
| F5 | الحجر تدفق | Comamonas سلالة التستوستيرونية NBRC 12047 / β-متقلبات | AB680219 | 100 | الأنواع Comamonas | 96.4 ± 2.6 |
| R8 | بالتنقيط الهوابط. | الناعم Curvibacter / β-متقلبات | AB680705 | 97.0 | Curvibacter < / م> الأنواع | 89.4 ± 7.8 |
| F8 | الحجر تدفق | الموراكسيلة ليرة سورية. 19.2 KSS / γ-متقلبات | HE575924.1 | 99.9 | الأنواع الموراكسيلة | 92.6 ± 4.9 |
تم عزل البكتيريا من Kartchner الكهوف، AZ، الولايات المتحدة الأمريكية، وحددت باستخدام 16S rDNA التسلسل. اثنين الاشعال، 27F (5 'AGA GTT TGA TCC TGG CTC AG 3') و1492r (5 'TAC GGT TAC CTT GTT ACG ACT T 3') استخدمت، للحصول على ما يقرب من 1،400 BP-16S طول تسلسل الرنا الريباسي الجينات.
(ب) بناء على بحث BLAST من قاعدة البيانات NCBI.
ج ذكرت القيم هي متوسط معاملات الارتباط من 30 مكررات (ثلاثة مكررات البيولوجية ولكل منها 10 مكررات الفنية) ± انحراف معياري واحد.
الجدول 1. البكتيريا المعزولة المستخدمة في المظاهرة.
FO: الصفحة كسر قبل = "دائما">
| مفتاح BioNumerics | فصول الذروة / المؤشرات الحيوية المحتملة (دا) |
| عصية الأنواع A | 2152.5، 2224.9، 2595.8، 2894.9، 2921.3، 3380.5، 3496.3، 3515.0، 3733.5، 4302.0، 4340.0، 4385.8،4763.9، 4910.6، 5189.4، 5227.0، 5634.6، 5769.6، 5892.8، 6301.4، 6756.2، 6789.4، 6990.3، 7029.5، 7466.3 |
| الأنواع عصية B | 2152.5، 2941.2، 3196.9، 3262.7، 3352.9، 3420.8، 3733.5، 3925.2، 4302.0،4339.9، 4629.2، 4713.4، 4859.3، 4900.6، 5189.4، 5227.0، 5541.8، 5878.7، 6388.8، 6524.0، 6704.5، 6838.8، 7142.7، 7317.5، 7466.3، 7849.1، 9263.9، 9721.2 |
| الأنواع عصية C | 2588.0، 3361.8، 4330.4، 5173.0، 5847.6، 6332.0، 6524.0، 6720.3 |
| الأنواع عصية D | 2152.5، 2894.9، 3420.8، 4302.0، 4339.9، 4629.2، 5189.4، 5448.4، 5878.7، 6388.8، 6838.8، 6931.1، 7849.1 |
| الأنواع عصية E | 2152.5، 2224.9، 2941.2، 3180.1، 3380.5، 4302.0، 4339.9، 4705.3، 5878.7، 6356.6، 6735.1، 6756.2 |
| الأنواع عصية F | 3308.6، 3367.8، 3567.5، 4279.7، 4489.2، 4629.2، 4727.9، 4751.7، 5067.7، 6614.7، 6919.7، 7130.9 |
| الأنواع Brevibacillus | 2133.3، 2611.0، 4263.3، 4302.0، 4859.3، 4900.6، 5080.2، 5219.0، 5847.6، 6775.7، 7529.4، 9721.2 |
| الأنواع Exiguobacterium | 2588.0، 3053.3، 3420.8، 3695.5، 4263.3، 5133.1، 5173.0، 5248.8، 6104.8، 6605.3، 6804.4، 6838.8، 7390.2 |
| المتقاصرة الأنواع A | 3053.3، 6104.8، 6146.5 |
| الأنواع Kocuria | 3080.0، 4366.6، 5080.2، 5163.8، 5207.1، 5892.8، 6160.0، 6197.5، 6445.0، 7433.7 |
| المتقاصرة الأنواع B | 3222.8، 3330.4، 3367.8، 4330.4، 4350.4، 4795.3، 4995.7، 5731.6، 6445.0، 6735.1،7487.3 |
| Aminobacter الأنواع A | 2133.3، 2562.4، 3361.8، 3410.4، 4289.2، 4629.2، 4662.0، 4869.8، 6064.1، 6221.0، 6720.3، 6789.4،6818.8، 7216.1، 7447.4 |
| الأنواع Comamonas | 2806.0، 2921.4، 3246.5، 4350.4، 4727.9، 5607.5، 5666.3، 6221.0، 6488.3، 7317.5، 9362.6 |
| الأنواع Curvibacter | 2868.6، 3453.2، 4319.8، 5133.1، 6292.4، 6903.4، 7433.7 |
| الأنواع الموراكسيلة | 3011.2، 5698.0، 6720.3، 7064.8، 7366.6 |
الجدول 2. الطبقات الذروة (المؤشرات الحيوية المحتملة) (دا) المعرفة لكل الأنواع.
الشكل التكميلي 1. تحليل المبدأ المكون (PCA) من أطياف الشامل في اله المستوى الفني تكرار (A) والطبقات الذروة (B). تم ترميز الألوان كما نفس الألوان المستخدمة في الشكل 2.
يتم تلوين الشكل التكميلي 2. مثال على الطبقات الذروة اختيارهم بناء على اتجاهين المجموعات. فصول الذروة لها نفس التسمية مع نفس اللون.
الشكل التكميلي 3. النتائج عبر المصادقة على مشروع تحديد استنادا إلى قواعد البيانات المخصصة في BioNumerics.
الشكل التكميلي تحديد 4. بكتيريا على أساس مطابقة ذروة استخدام قواعد البيانات المخصصة.
Discussion
وأظهرت هذه التظاهرة الإجراءات التفصيلية لتوصيف وتحديد البكتيريا باستخدام MALDI-TOF MS وقاعدة بيانات مخصصة. وبالمقارنة مع الأساليب الجزيئية التقليدية، على سبيل المثال، طرق بصمة 16S rDNA التسلسل، يستند إلى MS-MALDI-TOF تسهيل التعرف أكثر سرعة من البكتيريا متنوعة. بسبب قوة لها، ويستخدم على نطاق واسع هذه التقنية لتحديد خصائص البكتيريا والفيروسات والفطريات والخمائر من البيئة والمرافق الصحية 1،14-16. وعلاوة على ذلك، تم الإبلاغ عن MALDI-TOF MS على تحمل، في بعض الحالات، دقة أعلى التصنيفية 1. على سبيل المثال، B. ليرة سورية. A، B، D، E و، على الرغم تميل إلى التجمع معا (الشكل 2)، تم فصل بشكل واضح والتشابه بين أطياف مختلفة B. ليرة سورية. كان أقل من 80٪ (الشكل 2). في المقابل، كان تسلسل 16S rDNA من هذه العزلات التشابه عالية، والتي لا يمكن استخدامها للتمييز بين هذه العزلاتعلى مستوى الأنواع. تسلسل 16S rDNA من B. ليرة سورية. B و D لديهم 99٪ تشابه على أساس تحليل محاذاة متعددة، في حين أن تسلسل B. ليرة سورية. ألف وE تظهر 95٪ و 96٪ تشابه، على التوالي، إلى متواليات من B. ليرة سورية. وقد لوحظت أيضا B و D. ايسكنون. على سبيل المثال، B. ليرة سورية. C وF تجميعها بعيدا عن الآخرين B. ليرة سورية. (الشكل 2). ظهور العزلات غير النموذجية يشير إلى أن تحليل تجميع أطياف الشامل لا تضع بالضرورة علاقات النشوء والتطور. وقد تم الحصول على البيئة من الذي يعزل قد تؤثر أيضا أطياف التجميع الشامل. على سبيل المثال، المتقاصرة الأنواع B والأنواع Kocuria التي تم عزلها من speleothem رطبة وB. ليرة سورية. F الذي تم عزله من الهوابط بالتنقيط تميل إلى التجمع معا (الجدول 1، الشكل 2)، ولكن هناك حاجة إلى مزيد من الأبحاث لدراسة ما إذا كان هذا لوحظ في مجموعة أكبر من ISOLآتش.
يحتوي هذا الأسلوب القائم على المكتبة أيضا بعض القيود. عادة ما يستند التوصيف على قواعد البيانات. وتتكون قواعد البيانات التجارية الحالية أساسا من السلالات البكتيرية، وخاصة تلك المسببة للأمراض. هذه قواعد البيانات التجارية هي الأكثر فائدة في ضبط مختبر الميكروبيولوجيا السريرية. لتوصيف العزلات البيئية، فضلا عن الفيروسات والفطريات والخمائر، تحتاج قواعد البيانات المخصصة التي يتم بناؤها باستخدام مجموعات سلالة كبيرة. قد تحتاج أيضا إلى المعايير المستخدمة في التحاليل المتابعة ليكون الأمثل لزيادة دقة التصنيف، وخاصة على السلالة ومستويات التوتر. على سبيل المثال، S: N المستخدمة للكشف عن الذروة في هذه المظاهرة كان 10. هذه القيمة هي مناسبة لتحديد مستوى الأنواع، ولكن لتحديد مستوى الضغط، وقد تحتاج هذه القيمة الى الانخفاض. وبما أن هذه المعلمات التجهيز فضلا عن معالجة البيانات سير العمل في بعض الأحيان التي يحددها المستخدم في العديد من حزم البرامج، على سبيل المثال، ClinProTools وBionumeRICS، من المرجح أن تكون هناك حاجة إلى التحسين من القيم المعلمة واختيار مسارات العمل المناسبة لتحسين تحليل البيانات. في هذه المظاهرة، المعلمات مطابقة الذروة، والقيم عتبة المستخدمة في مشروع تحديد الهوية، وعبر التحقق من صحة يطلب من جميع الأمثل لتحسين معدلات تحديد الصحيحة. لإيجاد طريقة و / أو إجراء لتحسين هذه المعايير هي ذات أهمية كبيرة لمختبرنا. على سبيل المثال، نهج واحد قد تنطوي على تصميم مضروب الإحصائي، والتي كنا مؤخرا لتحسين MALDI-TOF الآلي الحصول على البيانات 17. وتشمل التطبيقات المستقبل إضافية وتعزيز يستند إلى MS-MALDI-TOF الميكروبية البصمات بناء على نطاق واسع، وقواعد البيانات أكبر من البكتيريا البيئية و / أو الكائنات الدقيقة غير البكتيرية وكذلك توصيف ثقافات مختلطة 18 و المجتمعات الميكروبية.
Disclosures
Authors Vranckx and Janssens are employees of Applied Maths NV, the manufacturer of data analysis software used in this video. Applied Maths NV provided select software modules highlighted in this video as well as a portion of the publication costs associated with this video.
Acknowledgements
This work was supported by the New College of Interdisciplinary Arts and Sciences at Arizona State University, Applied Maths NV, and by the National Science Foundation (ROA Supplement to Award No. MCB0604300). Any opinions, findings, and conclusions or recommendations expressed in this material are those of the author(s) and do not necessarily reflect the views of the National Science Foundation.
Materials
| Name | Company | Catalog Number | Comments |
| α-cyano-4-hydroxy-cinnamic acid | ACROS Organics | 163440050 | ≥ 97%, CAS 28168-41-8 |
| MALDI calibration kit | Aigma-Aldrich | MSCAL1-1KT | This kit also contains acetonitrile, trifluoroacetic acid , sinapinic acid, etc. |
| MALDI target plate | Bruker Daltonics | 280800 | Polished Steel |
| Bruker Microflex LRF MALDI-TOF mass spectrometer | Bruker Daltonics | ||
| Bruker FlexControl software | Bruker Daltonics | version 3.0 | |
| Bruker FlexAnalysis software | Bruker Daltonics | version 3.0 | |
| Bionumerics software | Applied Maths | version 7.1 |
References
- Sandrin, T. R., Goldstein, J. E., Schumaker, S. MALDI TOF MS profiling of bacteria at the strain level: A review. Mass Spectrom Rev. 32 (3), 188-217 (2013).
- Siegrist, T. J., et al. Discrimination and characterization of environmental strains of Escherichia coli by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry (MALDI-TOF-MS). J Microbiol Meth. 68 (3), 554-562 (2007).
- Goldstein, J. E., Zhang, L., Borror, C. M., Rago, J. V., Sandrin, T. R. Culture conditions and sample preparation methods affect spectrum quality and reproducibility during profiling of Staphylococcus aureus with matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Lett Appl Microbiol. 57 (2), 144-150 (2013).
- Benagli, C., et al. A rapid MALDI-TOF MS identification database at genospecies level for clinical and environmental Aeromonas strains. Plos One. 7 (10), (2012).
- Sauer, S., Kliem, M. Mass spectrometry tools for the classification and identification of bacteria. Nat Rev Microbiol. 8 (1), 74-82 (2010).
- Bohme, K., et al. SpectraBank: An open access tool for rapid microbial identification by MALDI-TOF MS fingerprinting. Electrophoresis. 33 (14), 2138-2142 (2012).
- Walker, J., Fox, A. J., Edwards-Jones, V., Gordon, D. B. Intact cell mass spectrometry (ICMS) used to type methicillin-resistant Staphylococcus aureus: media effects and inter-laboratory reproducibility. J Microbiol Meth. 48 (2-3), 117-126 (2002).
- Ruelle, V., El Moualij, B., Zorzi, W., Ledent, P., De Pauw, E. Rapid identification of environmental bacterial strains by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Commun Mass Sp. 18 (18), 2013-2019 (2004).
- Sedo, O., Sedlacek, I., Zdrahal, Z. Sample Preparation Methods for Maldi-MS Profiling of Bacteria. Mass Spectrom Rev. 30 (3), 417-434 (2011).
- Swatkoski, S., Russell, S., Edwards, N., Fenselau, C. Analysis of a model virus using residue-specific chemical cleavage and MALDI-TOF mass spectrometry. Anal Chem. 79 (2), 654-658 (2007).
- Freiwald, A., Sauer, S. Phylogenetic classification and identification of bacteria by mass spectrometry. Nat Protoc. 4 (5), 732-742 (2009).
- Drevinek, M., Dresler, J., Klimentova, J., Pisa, L., Hubalek, M. Evaluation of sample preparation methods for MALDI-TOF MS identification of highly dangerous bacteria. Lett Appl Microbiol. 55 (1), 40-46 (2012).
- Lasch, P., et al. MALDI-TOF mass spectrometry compatible inactivation method for highly pathogenic microbial cells and spores. Anal Chem. 80 (6), 2026-2034 (2008).
- Usbeck, J. C., Kern, C. C., Vogel, R. F., Behr, J. Optimization of experimental and modelling parameters for the differentiation of beverage spoiling yeasts by Matrix-Assisted-Laser-Desorption/Ionization Time-of-Flight Mass Spectrometry (MALDI-TOF MS) in response to varying growth conditions. Food Microbiol. 36 (2), 379-387 (2013).
- Del Chierico, F., et al. MALDI-TOF MS proteomic phenotyping of filamentous and other fungi from clinical origin. J Proteomics. 75 (11), 3314-3330 (2012).
- Vitale, R., Roine, E., Bamford, D. H., Corcelli, A. Lipid fingerprints of intact viruses by MALDI-TOF/mass spectrometry. Bba-Mol Cell Biol L. 1831 (4), 872-879 (2013).
- Zhang, L., Borror, C. M., Sandrin, T. R. A designed experiments approach to optimization of automated data acquisition during characterization of bacteria with MALDI-TOF mass spectrometry. Plos One. 9 (3), (2014).
- Christner, M., Rohde, H., Wolters, M., Sobottka, I., Wegscheider, K., Aepfelbacher, M. Rapid identification of bacteria from positive blood culture bottles by use of matrix-assisted laser desorption-ionization time of flight mass spectrometry fingerprinting. J ClinMicrobiol. 48 (5), 1584-1591 (2010).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved