Method Article
L'uso di MALDI-TOF spettrometria di massa e un database personalizzato per caratterizzare batteri indigeni ad un unico ambiente di Cave (Kartchner Caverns, AZ, USA)
In questo articolo
Riepilogo
This work details procedures for rapid identification of bacteria using MALDI-TOF MS. The identification procedures include spectrum acquisition, database construction, and follow up analyses. Two identification methods, similarity coefficient-based and biomarker-based methods, are presented.
Abstract
MALDI-TOF mass spectrometry has been shown to be a rapid and reliable tool for identification of bacteria at the genus and species, and in some cases, strain levels. Commercially available and open source software tools have been developed to facilitate identification; however, no universal/standardized data analysis pipeline has been described in the literature. Here, we provide a comprehensive and detailed demonstration of bacterial identification procedures using a MALDI-TOF mass spectrometer. Mass spectra were collected from 15 diverse bacteria isolated from Kartchner Caverns, AZ, USA, and identified by 16S rDNA sequencing. Databases were constructed in BioNumerics 7.1. Follow-up analyses of mass spectra were performed, including cluster analyses, peak matching, and statistical analyses. Identification was performed using blind-coded samples randomly selected from these 15 bacteria. Two identification methods are presented: similarity coefficient-based and biomarker-based methods. Results show that both identification methods can identify the bacteria to the species level.
Introduzione
Matrix-assisted laser desorption/ionization time-of-flight (MALDI-TOF) mass spectrometry (MS) has been shown to be a rapid and reliable tool for identification of bacteria at the genus, species, and in some cases, strain levels1-4. MALDI-TOF MS ionizes biological molecules (typically proteins) that originate from cell surfaces, intracellular membranes, and ribosomes from bacterial whole cells or protein extracts1,5. The resulting peaks form characteristic patterns or “fingerprints” of the bacteria analyzed1. Identification of bacteria is based on these mass-to-charge “fingerprints”.
Two of the most commonly used identification strategies are library-based and bioinformatics-based strategies1. Library-based approaches involve comparing the mass spectra of unknowns to previously collected mass spectra of known bacteria in databases/libraries for identification. Commercially available software, such as BioNumerics, Biotyper, and SARAMIS software packages, as well as open source software tools, such as SpectraBank6, are available to facilitate the comparison and quantification of similarity between mass spectra of unknowns and reference bacteria. Bioinformatics-based approaches usually rely on fully sequenced genomes of bacteria for identification. In contrast to library-based approaches which do not involve identification of the biological nature of particular peaks, bioinformatics-based approaches involve protein identification1.
The majority of recent MALDI fingerprint-based studies have used library-based approaches to identify bacteria1. Library-based approaches require construction of databases and comparison of the similarity between mass spectra. Studies show that many experimental procedures, such as medium3,7, cultivation time8, sample preparation method3, and matrix used9, affect the mass spectra obtained. Furthermore, some closely-related species and strains generate spectra with only subtle differences. Thus, library-based approaches require rigorously standardized procedures to generate highly reproducible mass spectra between replicates. Minor variations in protocols may compromise the efficacy of identification, especially at the subspecies and strain levels1,3,10. However, neither manufacturer-provided reference databases nor reported custom databases include visually documented procedures for database construction and/or application of a data analysis pipeline. For this reason, the objective of this work was to develop, apply, and demonstrate a comprehensive and detailed procedure for library-based bacterial identification using MALDI-TOF MS.
In this demonstration, mass spectra of 15 bacteria isolated from a karstic environment (Kartchner Cavern, AZ, USA) were collected and imported into software to construct a model database. Data processing and the analysis pipeline were detailed using the model database. Finally, mass spectra of blind-coded bacteria which were randomly selected from these 15 bacteria were collected again and compared to the reference spectra in the model database for identification. Results show that bacteria can be correctly identified either based on similarity coefficients or potential biomarkers/peak classes.
Protocollo
Attenzione: i batteri non identificati da qualsiasi ambiente possono essere patogeni e devono essere trattati con cautela utilizzando protocolli di biosicurezza adeguate. Lavora con fermenti vivi deve essere eseguita in un armadio biosicurezza Classe II utilizzando biologica Livello di sicurezza 2 (BSL-2) procedure. Maggiori informazioni su BSL-2 procedure sono disponibili nel manuale CDC / NIH intitolato "biosicurezza in microbiologiche e Biomediche Laboratories," pagine 33-38. Il documento è disponibile online all'indirizzo http://www.cdc.gov/biosafety/publications/bmbl5/BMBL.pdf . Adeguati dispositivi di protezione individuale (DPI), tra cui camici / camici, occhiali di sicurezza e guanti di lattice o di nitrile, deve essere indossato. Pratiche microbiologiche standard e precauzioni devono essere seguite, e rifiuti biologici devono essere eliminati in modo appropriato.
I batteri utilizzati in questa manifestazione sono stati isolati da Kartchner Caverns,AZ, USA, da quattro ambienti, tra cui speleothem secca, pietra flusso, speleothem umido e stalattiti gocciolatoio (Tabella 1). Tutti gli isolati sono stati identificati mediante sequenziamento del 16S rDNA e conservati a -80 ° C in 25% glicerolo-R media 2 B. Tutti gli esperimenti sono stati completati a RT.
Nota: Si consiglia di utilizzare lo stesso metodo di preparazione del campione per acquisire spettri di massa per la costruzione di database e gli spettri di massa di incognite. Metodo di preparazione del campione è stato dimostrato in precedenza di influenzare la qualità dello spettro e riproducibilità 3. Utilizzando un metodo di preparazione del campione diverso può causare non corretta identificazione di incognite, soprattutto quando a risoluzione più elevata tassonomica (es, a livello del ceppo) è desiderato.
1. Deposizione sul MALDI destinazione
Attenzione: Diversi protocolli per ottenere estratti di proteine richiedono l'uso di acidi e solventi organici che devono essere utilizzati in conformità con guidElines e le informazioni contenute nei rispettivi Schede di sicurezza dei materiali (MSDS). Adeguato DPI devono essere indossati e varia in base al tipo e il volume di prodotti chimici utilizzati (ad esempio, camici / camici, guanti, occhiali di sicurezza, e la protezione delle vie respiratorie devono essere utilizzati quando si lavora con grandi quantità di solventi infiammabili, tossiche, come acetonitrile, e acidi corrosivi, come acidi formico e trifluoroacetico).
- Deposito 1 ml estratto proteico che non contiene cellule vitali (ottenute utilizzando opportuni protocolli, descritte in precedenza 11-13) su una piastra segnale MALDI acciaio inox e lasciarlo asciugare. Sovrapporre l'estratto proteico secco con una soluzione di matrice 1 ml (soluzione di acido-4-idrossi cinnamico α-ciano-), e lasciarlo asciugare.
- Per ogni replica biologica, individuare un numero adeguato di repliche tecnici (da 5 a 20 replicati tecnici). Qui, individuare 10 replicati tecnici per ogni replica biologica e 3 repliche biologiche were preparati.
Nota: Si consiglia di utilizzare una piastra segnale MALDI acciaio lucido quando si utilizza il metodo di preparazione del campione di estrazione di proteine. Utilizzando piastre di acciaio di destinazione terra può causare la diffusione e la miscelazione involontaria di diversi campioni di fuori dei singoli pozzetti del campione. - Deposito 1 ml calibrante di serie sulla piastra segnale e lasciare asciugare. Sovrapposizione con la soluzione di matrice 1 ml e lasciare asciugare.
- Deposito soluzione di matrice 2 microlitri sulla piastra porta come controllo negativo.
2. Mass Spectra Acquisition
- Utilizzare uno spettrometro di massa MALDI-TOF dotato di un laser di azoto (λ = 337 nm) e gestito utilizzando il software Bruker FlexControl.
- Raccogliere ogni spettro di massa in modalità lineare positiva da accumulo di colpi 500 laser con incrementi colpo 100. Impostare la tensione della sorgente di ioni 1 a 20 kV; sorgente di ioni 2 tensione 18.15 kV; e la tensione lenti a 9,05 kV. Si noti che questi parametri sono strumento di specific e potrebbe richiedere una regolazione su altri strumenti per ottenere risultati ottimali.
- Impostare l'intervallo di massa-a-charge per la valutazione dello spettro automatica da 2 a 20 kDa per carica. Utilizzare l'algoritmo di rilevamento di picco baricentro. Impostare la soglia minima risoluzione a 100 Da. Impostare il rapporto segnale rumore (S: N) soglia 2. Impostare la soglia minima di intensità a 100.
3. Database di costruzione
- Progettazione di database
- Creare un nuovo database in BioNumerics 7.1 utilizzando la "procedura guidata Nuovo database".
- Creare un tipo di esperimento spettro, ad esempio, Maldi, utilizzando i comandi nel pannello "Tipi di esperimento".
- Creare i livelli utilizzando il "pannello di progettazione del database". Aggiungere nuovi livelli utilizzando il "Livello> Aggiungi nuovo livello ..." comando nel menu "Database". Qui, creare "specie" di livello, "replica biologica" "e" replica tecnici "livello, respectively.
- Importazione e pre-elaborazione spettri di massa grezza
- Esportare gli spettri di massa crudo come file .txt utilizzando FlexAnalysis cliccando sul pulsante "Esporta> Spettro di massa" comando nel menu "File".
- Importare gli spettri di massa grezza (.txt) nel database del livello dei replicati tecnici.
- Pre-elaborazione spettri di massa grezza.
- Importazione e resample (utilizzando un algoritmo quadratico raccordo).
- Eseguire una sottrazione della linea di base (con un disco di rotolamento con una dimensione di 50 punti).
- Calcola rumore [continua Wavelet Transformation (CWT)], liscia (Finestra Kaiser con una dimensione della finestra di 20 punti e beta di 10 punti), ed effettuare una seconda sottrazione di base (disco a rotazione con dimensioni di 200 punti).
- Rileva picchi [CWT con un segnale minimo di rumore (S: N) di 10].
- Dopo la pre-elaborazione, salvare modelli caratteristici di ogni spettro di massa, come le liste contenenti picco piccodimensioni, intensità dei picchi, S: N, ecc, nel database.
- Creazione di spettri di massa composita
- Creare spettri composito da spettri già preparati usando il comando "Riassumere ..." nel menu "Analisi". Scegliere la "replica biologica" come livello target.
- Qui, unire spettri di massa di 10 replicati tecnici della stessa colonia per produrre uno spettro di massa composito per quella colonia, con conseguente tre composito spettri di massa per isolare che a livello "replica biologica".
- Qui, riassumere i tre spettri composito per creare uno spettro composito per isolare che a livello "Specie".
Nota: Lo spettro composito è la media punto per punto dei replicati tecnici. Replica con una similitudine (Pearson di correlazione) alla media di inferiore al 95% (impostazione di default) sono esclusi dal composito. Peaks sul spettri compositi sono chiamati solo se sono presenti nel 75% (impostazione di default) dei replicati inclusi. Per le repliche biologiche, queste impostazioni sono state 90 e 60%, rispettivamente.
Analisi 4. Messa Spectrum dati
- Selezionare le voci nel database e creare un confronto facendo clic sul comando "Crea nuovo confronto" nel pannello "Confronti".
- Qui, utilizzare gli spettri di massa al "replicare tecnici" e / o livelli "replicati biologica" per visualizzare i confronti e analisi.
- Analisi cluster basato somiglianza e scalabilità multi-dimensionale (MDS)
- Creare gruppi di colori. Selezionare i tre biologica spettri di massa composita e fare clic su "Crea nuovo gruppo dalla selezione" comando nel menu "gruppi" di creare un gruppo per la corrispondente isolato. Designare un colore usato automaticamente per questi tre spettri di massa.
- In alternativa, definire gli stati di campo con i colori corrispondenti utilizzando i comandi del "Database voci pannello "tale che qualsiasi raggruppamento basato su questo campo definito utilizza lo stesso colore definito per questo gruppo.
- Eseguire l'analisi cluster. Fare clic sul comando "Calcola cluster analysis" nel menu "Clustering". In Impostazioni di confronto Page 1, selezionare il "Pearson di correlazione" e lasciare gli altri parametri di default. A pagina 2, selezionare "UPGMA". Quindi fare clic su "Fine".
- Ottenere una trama MDS utilizzando il "ridimensionamento Multi-dimensionale ..." comando nel menu "Statistica".
- Corrispondenza Peak
- Cliccate sul tipo di spettro "Maldi" nel pannello "Esperimenti". Quindi selezionare "Layout> Immagine". Spectra sono presenti come bande gel.
- Eseguire corrispondenza picco con il comando "Do corrispondente picco" nel menu "Spectra".
- Individuazione delle classi di punta
- Effettuare analisi delle componenti principali (PCA). Evidenziare il "Maldi "tipo di esperimento nel" sperimentale "del pannello e utilizzare la funzione" Analisi delle Componenti Principali ... "comando nel menu" statistico "per eseguire PCA.
- Eseguire due vie clustering. Fare clic su "Statistiche> mining Matrix" ... nella finestra "Confronto". L'intensità dei picchi corrispondenti alle classi di punta è rappresentata utilizzando colori diversi (mappa di calore).
5. Batteri identificazione con un database personalizzato
- Metodo coefficiente a base di somiglianza
- Creare un confronto e generare un dendrogramma basato su spettri di massa a livello di "replicare tecnici" come descritto al punto 4.3.3. Salvare il dendrogramma per il confronto di similarità.
- Selezionare uno spettro di massa sconosciuta, e fare clic su "Analisi> Identificare le voci selezionate". Verrà visualizzata la finestra di dialogo di identificazione.
- Selezionare la "base di confronto" tipo classificatore (o un classif memorizzataier) e cliccare su "Avanti". Nella pagina successiva, scegli il dendrogramma salvato come un confronto di riferimento e quindi fare clic su "Avanti".
- Scegliere la "somiglianza di base" come un metodo di identificazione e quindi fare clic su "Avanti".
- Scegliere la "somiglianza Massimo" come metodo di punteggio. Digitare appropriati valori soglia e valori minimi di differenza per ogni parametro e quindi fare clic su "Avanti".
- Una volta che i calcoli sono stati completati, viene visualizzata la finestra di identificazione. Nel "Risultati" pannello, vengono elencati i membri del database che meglio corrispondono l'ignoto.
- Salvare il progetto di identificazione e validare l'identificazione con il "analisi cross-validazione" comando nel pannello "progetto di identificazione".
- Potenziale metodo basato biomarcatore-
- Definire le classi di picco. Nella finestra "Matrix Mining", selezionare serie di cime che condividono caratteristiche comuni e definire questi picchi come speciclassi fic picco (potenziali biomarkers) usando "Spectra> Gestisci tipi di classe picco ..." nella "finestra di confronto".
- Qui, definire classi di picco specifici per ogni isolano per tutti i 15 isolati.
- Selezionare spettri di massa di incognite e abbinare i picchi di questi spettri alle classi picchi definiti come precedentemente descritto.
Risultati
Le basi di dati costruiti in questa manifestazione ha avuto quattro livelli, dal più alto al più basso livello, tra cui "tutti i livelli", "Species", "replica biologica" e "replicare tecnici", rispettivamente (Figura 1A). La "replica tecnici" livello conteneva tutti gli spettri di pre-elaborato replicati tecnici. La "replica Biologica" e livelli di "specie" contenevano il (sommario) spettri composito. "Tutti i livelli" contenevano tutta la replica spettri tecnica così come tutti gli spettri composito.
Procedure Spectrum riepilogo sono mostrati in figura 1 con picchi rappresentativi. Ogni spettro di massa membro appare come una sottile linea grigia. Lo spettro composito è rappresentato come una linea colorata in rosso. Picchi adiacenti sono contrassegnati con un colore diverso per consentire facile ispezione visiva (Figura 1B).
La riproducibilità degli spettri di massa dei 30 replicati (tre repliche biologiche, ciascuna con 10 replicati tecnici) sono stati calcolati e sono mostrati in Tabella 1. La riproducibilità massima era 98,0 ± 1,4 per Bacillus specie B, e il più basso riproducibilità era 89.4 ± 7.8 per Curvibacter specie (Tabella 1).
Analisi cluster a livello di visualizzazione replica biologica facilitato la struttura gerarchica dei dati complessi spettri di massa. Come mostrato in Figura 2, repliche biologiche raggruppati insieme, e 15 specie di batteri formate 15 cluster. Specie strettamente collegate, per esempio, B. sp. A, B, D, ed E, tendevano a raggrupparsi. Tuttavia, valori anomali, ad esempio, B. sp. C e F, sono stati osservati anche. Piazzole MDS basati sugli spettri di massa a livello "tecniche" e biologiche sono mostrati in Figura 3. MDS plotti hanno prodotto una chiara visualizzazione 3-D delle somiglianze tra spettri di questi batteri. Sia replicati tecnici e repliche biologiche mostrato un gruppo simile (Figura 3 A e B).
Corrispondente picco è stato utilizzato per distinguere insiemi di picchi negli spettri di massa. Parametri corrispondenti Peak, tra cui la tolleranza costante (punti l'asse x), la tolleranza lineare (ppm) e tasso di rilevamento di picco devono essere specificati dall'utente. Tolleranza costante e tolleranza lineare sono i fattori utilizzati per calcolare la tolleranza di posizione dei picchi utilizzando l'equazione: tolleranza di posizione = tolleranza costante + tolleranza lineare × m / z. Con l'aumento m / z, l'importanza della tolleranza costante diminuisce. Tasso di rilevamento di picco significa che solo se un picco è trovato in quella posizione per più del tasso definito degli spettri, viene fatta una classe di picco. Un picco su uno o più modelli rappresenta una classe di picco. Ad esempio, se il tasso di rilevamento di picco equals 10%, una classe di picco può essere fatto solo se più del 10% degli spettri hanno picchi a posizione. Ciò esclude picchi di bassa prevalenza (di solito picchi di rumore) in un set con repliche tecnici. Se l'apparecchio è basato sulla spettri composita di repliche biologiche, questo numero può essere necessario inferiore come picchi a bassa prevalenza sono già stati filtrati durante la creazione degli spettri composito. In questa dimostrazione, corrispondente picco è stato eseguito utilizzando spettri di massa a livello "replica tecnici" ed i valori di questi parametri sono stati 1,9, 550 e 10%, rispettivamente. Sulla base di parametri selezionati, picchi sono stati considerati come corrispondenti o non corrispondenti, con conseguente diversi gruppi di picco. Un esempio di coincidenze picco è mostrato in Figura 4 utilizzando 30 replicati di un singolo isolato (specie Bacillus A). I risultati corrispondenti sono stati visualizzati come una tabella in cui sono presenti come colori le intensità prime. Blu indica bassa intensità e rosso indica alta intensity. Sulla base dei risultati di picco corrispondenti, gli utenti possono definire classi di punta che facilitano follow-up analisi.
Entrambi analisi delle componenti principali (PCA) (Figura supplementare 1) e due vie raggruppamento può essere utilizzato per analizzare le classi di picco complesse. Un risultato di clustering bidirezionale rappresentante utilizzando spettri di massa a livello "replica tecnici" è mostrato in Figura 5. Due dendrogrammi sono mostrati. Uno è accanto ai valori m / z e l'altro è sopra le voci batteri (Figura 5). Intensità di picco è stata rappresentata da colori in cui verde indica bassa intensità e rosso indica ad alta intensità. Ad esempio, B. sp. A ed F condividono poche classi di punta con B. sp. B e D (figura 5). Un attento esame ha dimostrato che B. sp. B e D hanno anche insiemi di classi di picco specie-specifici (Figura 5). Questi risultati indicano che le serie di punta specifici condivisione cert caratteristiche ain possono essere definiti come potenziali biomarcatori specie a livello. Ad esempio, tredici insiemi picco facente B. sp. D sono stati selezionati e definiti come classi di picco (potenziali biomarkers) di B. sp. D, tra cui 2.152,5, 2894,9, 3420,8, 4302,0, 4339,9, 4629,2, 5189,4, 5448,4, 5878,7, 6388,8, 6838,8, 6.931,1 e 7.849,1 (Tabella 2). Classi di picco di diversi isolati possono essere visualizzati in diversi colori (Figura integrativa 2). Classi di picco specifici per ogni isolato sono stati tabulati in Tabella 2. Classi di picco definita sono stati controllati ulteriormente manualmente per assicurare che sono apparsi in tutte le repliche tecniche con una intensità minima di 100 au Inoltre, sottoinsiemi di classi di punta potrebbe anche essere memorizzati per facilitare la caratterizzazione dei batteri le sottospecie e / o livelli di deformazione, per esempio, per distinguere i ceppi patogeni da ceppi non patogeni e / o per esaminare antibiotico resistenza / sensibilità.
ent "> Per quanto riguarda l'identificazione, spettri di massa di isolati ciechi codificati sono stati raccolti e preelaborazione nello stesso modo come spettri di massa di riferimento nei database. Per identificazione basata sul confronto dei coefficienti di similarità, i valori dei parametri sono stati specificati, compresa la massima somiglianza al 95,0% e la somiglianza media al 87%. somiglianza minima non è stato specificato (cioè, lasciato incontrollato). I valori minimi di differenza sono stati fissati come 5 sia per la massima affinità e similarità media. Questi valori possono avere bisogno di essere ulteriormente ottimizzato per aumentare il tasso di corretta identificazione. Figura 6 mostra i risultati di identificazione basate sul confronto dei coefficienti di similarità (Figura 6). Il risultato corrispondente suggerito che questo batterio cieca-coded era probabilmente B. sp. A. Questo progetto identificazione basata sul confronto di Coefficiente di somiglianza è stato ulteriormente convalidato dal convalida incrociata (figura complementare 3 ). La convalida incrociata è stata testata con una copertura del 25%. Utilizzando una copertura superiore, ad esempio, 50% o 100%, possono aumentare la fiducia di identificazione, ma richiede un tempo molto più lungo per completare, soprattutto per grandi database. Tutte le classi hanno testato 100% veri positivi e 0% di falsi negativi (Figura 3) supplementare. È interessante notare, convalida incrociata per progetti identificazione basate sul confronto di classi di picco è molto più veloce di quelle basate sul confronto di coefficiente di similitudine.L'identificazione può anche essere compilato considerando corrispondente classe di picco (figura complementare 4). Tuttavia disallineamenti dei picchi sono stati osservati (figura complementare 4). Le discordanze possono essere dovute ad uno spostamento di massa derivante da scambi aminoacidi nelle rispettive proteine. I picchi non si allinea potrebbero anche essere i picchi che non sono discriminante a livello di specie, ma sono specifici per questo ceppo o isolano. Nel loro insieme, our risultati suggeriscono che entrambi i metodi di identificazione - coefficiente di somiglianza e basati biomarcatori-- possono facilmente identificare i batteri a livello di specie da ambienti carsici con la preparazione del campione, l'acquisizione dello spettro, e il flusso di lavoro di analisi dei dati descritto qui.

Figura 1. costruzione di database e di massa spettri riepilogo Struttura di database costruiti in questa dimostrazione (A).; Illustrazione del picco riepilogo con picchi di 10 repliche tecniche di Aminobacter specie A (B).
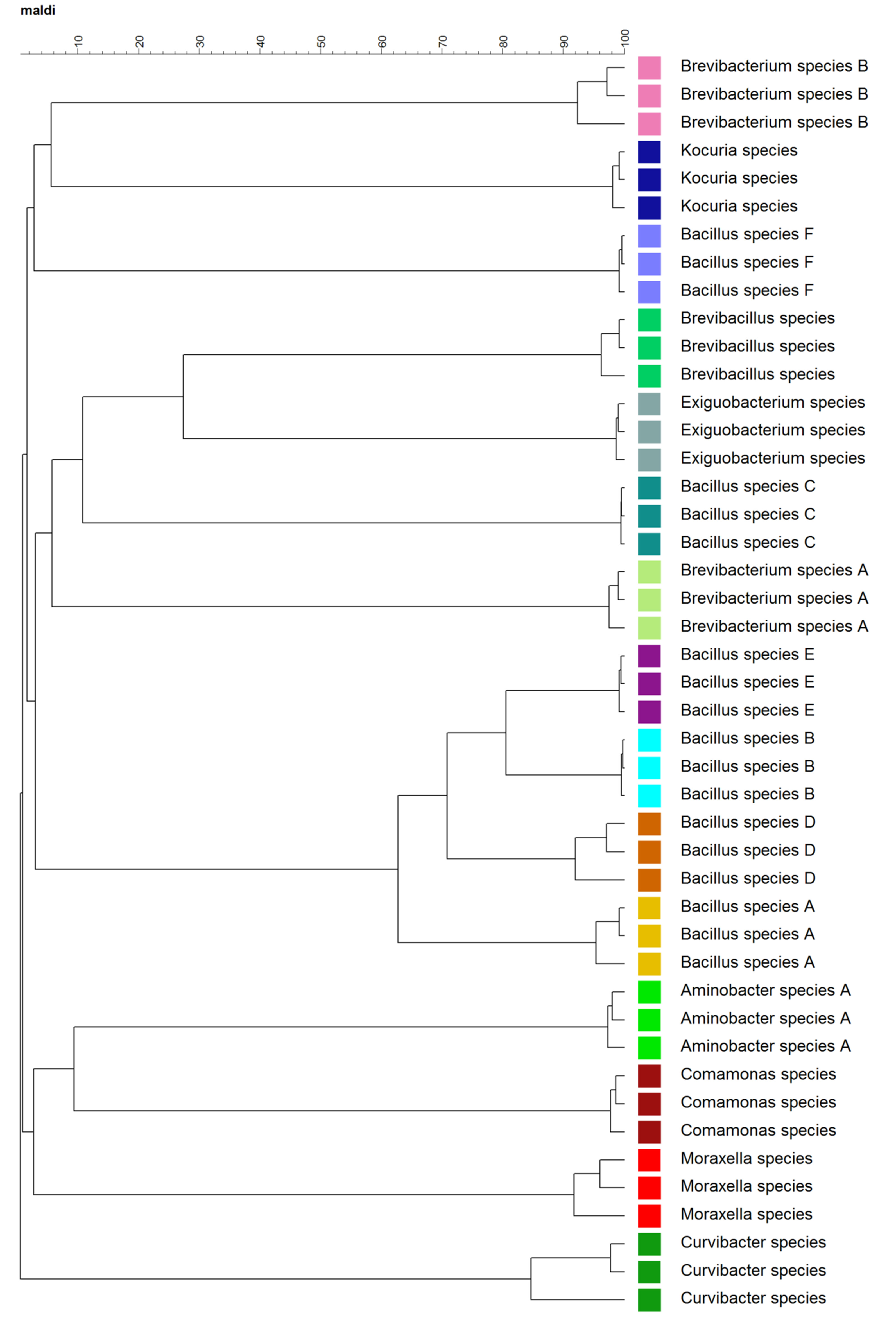
Figura 2. Dendrogramma del compositospettri di massa a livello di replica biologica. L'insieme di dati contiene spettri di 15 specie diverse con tre spettri composito per ciascuna specie. Ogni specie era codificati con un colore.

Figura 3. ridimensionamento Multi-dimensionale (MDS) rappresentazioni di spettri di massa a livello replicare tecnica con 30 spettri per ogni specie (A) e il livello di replicare biologica con tre spettri composito per ogni specie (B). I colori sono stati codificati come gli stessi colori usati nella Figura 2.

Figura 4. Un esempio di tabella di corrispondenza dei picchi. Tabella è stata generata sulla base di spettri di massa di specie Bacillus A alla replica leve tecnicol. I valori dei parametri corrispondenti di punta erano 1.9 per la tolleranza costante, 550 per la tolleranza lineare e del 10% per il tasso di rilevamento di picco rispettivamente. Blu indica bassa intensità di picco e rosso indica ad alta intensità di picco. Clicca qui per vedere una versione più grande della figura.

Figura 5. Un esempio di due vie di clustering. Figura stata generata utilizzando spettri di massa a livello tecnico replica. Colori di isolati sono stati codificati come gli stessi colori usati nella Figura 2. Intensità del picco è rappresentato da colori, significato verde bassa intensità e significato rossa ad alta intensità. Clicca qui per vedere una versione più grande della figura.
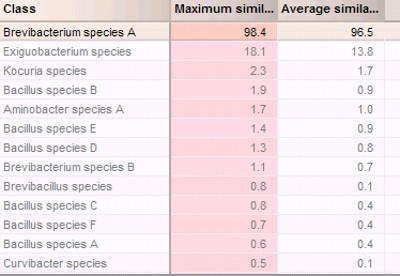
Figura di identificazione 6. Batterio basata sul confronto di coefficiente somiglianza con database personalizzato.
| ID un | Fonte | Vicina relativa b / Phylum / Class | Adesione # (più vicino parente b) | % Somiglianza | Chiave BioNumerics | Riproducibilità (%) |
| D2 | Speleothem Dry | Bacillus sp. E-257 / Firmicutes | FJ764776.1 | 98,8 | Bacillus specie A | 94.9 ± 4.0 |
| D7 | Speleot Dryorlo | Bacillus sp. GGC-P3 / Firmicutes | FJ348039.1 | 99.0 | Specie Bacillus B | 98.0 ± 1.4 |
| F1 | Pietra Flusso | Bacillus niacina ceppo M27 / Firmicutes | KC315764.1 | 99,2 | Specie Bacillus C | 96.5 ± 2.4 |
| F4 | Pietra Flusso | Bacillus sp. GGC-P5A1 / Firmicutes | FJ348046.1 | 99.1 | Specie Bacillus D | 89.8 ± 8.8 |
| F9 | Pietra Flusso | Bacillus sp. OSS 19 / Firmicutes | EU124558.1 | 99,4 | Specie Bacillus E | 96.5 ± 1.9 |
| R10 | Gocciolamento Stalactite | Bacillus sp. K1 / Firmicutes | GU968734.1 | 99,8 | Specie Bacillus F | 95.4 ± 3.9 |
| D11 | Speleothem Dry | Brevibacillus ceppo brevis IMAU80218 / Firmicutes | GU125635.1 | 99,5 | Specie Brevibacillus | 94.3 ± 5.8 |
| F14 | Pietra Flusso | Sp Exiguobacterium. ZWU0009 / Firmicutes | JX292087.1 | 99,3 | Specie Exiguobacterium | 96,5 ± 2,5 |
| M7 | Speleothem Moist | Brevibacterium sp. N78 / Actinobacteria | HQ188605 | 97.6 | Brevibacterium specie A | 97.5 ± 2.0 |
| M14 | Speleothem Moist | Ceppo Kocuria rhizophila AG09 / Actinobacteria | EU554435.1 | 100 | Specie Kocuria | 95.2 ± 4.1 |
| M15 | Speleothem Moist | Brevibacterium sp. MN3-3 / Actinobacteria | JQ396535.1 | 99,5 | Brevibacterium specie B | 92.1 ± 4.9 |
| R4 | Gocciolamento Stalactite | Aminobacter sp. KC-EP-S4 / α-Proteobacteria | FJ711220.1 | 99,9 | Aminobacter specie A | 95.4 ± 2.7 |
| F5 | Pietra Flusso | Comamonas ceppo testosteroni NBRC 12047 / β-Proteobacteria | AB680219 | 100 | Specie Comamonas | 96.4 ± 2.6 |
| R8 | Gocciolamento Stalactite | Delicati Curvibacter / β-Proteobacteria | AB680705 | 97.0 | Curvibacter </ em> specie | 89.4 ± 7.8 |
| F8 | Pietra Flusso | Moraxella sp. 19.2 KSS / γ-Proteobacteria | HE575924.1 | 99,9 | Specie Moraxella | 92.6 ± 4.9 |
a batteri sono stati isolati da Kartchner Caverns, AZ, USA e identificati mediante sequenziamento del 16S rDNA. Due primer, 27F (5 'AGA GTT TGA TCC TGG CTC AG 3') e 1492r (5 'TAC GGT TAC CTT GTT ACG ACT T 3'), sono stati utilizzati per ottenere quasi 1.400 bp lunghezza 16S sequenze di geni rRNA.
b Sulla base di una ricerca BLAST del database NCBI.
c I valori riportati sono i coefficienti medi di correlazione di 30 repliche (tre repliche biologiche ciascuna con 10 replicati tecnici) ± una deviazione standard.
Tabella 1. I batteri isolati di utilizzare in dimostrazione.
| Chiave BioNumerics | Classi Peak / potenziali biomarcatori (DA) |
| Bacillus specie A | 2.152,5, 2224,9, 2595,8, 2894,9, 2921,3, 3380,5, 3496,3, 3515,0, 3733,5, 4302,0, 4340,0, 4385.8,4763.9, 4910,6, 5189,4, 5227,0, 5634,6, 5769,6, 5892,8, 6301,4, 6756,2, 6789,4, 6990,3, 7029,5, 7.466,3 |
| Specie Bacillus B | 2.152,5, 2941,2, 3196,9, 3262,7, 3352,9, 3420,8, 3733,5, 3925,2, 4302.0,4339.9, 4629,2, 4713,4, 4859,3, 4900,6, 5189,4, 5227,0, 5541,8, 5878,7, 6388,8, 6524,0, 6704,5, 6838,8, 7142,7, 7317,5, 7466,3, 7.849,1, 9263,9, 9.721,2 |
| Specie Bacillus C | 2.588,0, 3361,8, 4330,4, 5173,0, 5847,6, 6332,0, 6524,0, 6.720,3 |
| Specie Bacillus D | 2.152,5, 2894,9, 3420,8, 4302,0, 4339.9, 4629,2, 5189,4, 5448,4, 5878,7, 6388,8, 6838,8, 6931,1, 7.849,1 |
| Specie Bacillus E | 2.152,5, 2224,9, 2941,2, 3180,1, 3380,5, 4302,0, 4339,9, 4705,3, 5878,7, 6356,6, 6735,1, 6.756,2 |
| Specie Bacillus F | 3.308,6, 3367,8, 3567,5, 4279,7, 4489,2, 4629,2, 4727,9, 4751,7, 5067,7, 6614,7, 6919,7, 7.130,9 |
| Specie Brevibacillus | 2.133,3, 2611,0, 4263,3, 4302,0, 4859,3, 4900,6, 5080,2, 5219,0, 5847,6, 6775,7, 7529,4, 9.721,2 |
| Specie Exiguobacterium | 2.588,0, 3053,3, 3420,8, 3695,5, 4263,3, 5133,1, 5173,0, 5248,8, 6104,8, 6605,3, 6804,4, 6838,8, 7.390,2 |
| Brevibacterium specie A | 3.053,3, 6104,8, 6.146,5 |
| Specie Kocuria | 3.080,0, 4366,6, 5080,2, 5163,8, 5207,1, 5892,8, 6160,0, 6197,5, 6445,0, 7.433,7 |
| Brevibacterium specie B | 3.222,8, 3330,4, 3367,8, 4330,4, 4350,4, 4795,3, 4995,7, 5731,6, 6445,0, 6735.1,7487.3 |
| Aminobacter specie A | 2.133,3, 2562,4, 3361,8, 3410,4, 4289,2, 4629,2, 4662,0, 4869,8, 6064,1, 6221,0, 6720,3, 6789.4,6818.8, 7216,1, 7.447,4 |
| Specie Comamonas | 2.806,0, 2921,4, 3246,5, 4350,4, 4727,9, 5607,5, 5666,3, 6221,0, 6488,3, 7317,5, 9.362,6 |
| Specie Curvibacter | 2.868,6, 3453,2, 4319,8, 5133,1, 6292,4, 6903,4, 7.433,7 |
| Specie Moraxella | 3.011,2, 5698,0, 6720,3, 7064,8, 7.366,6 |
Tabella 2. classi di picco (i potenziali biomarcatori) (Da) definiti per ciascuna specie.
Figura supplementare 1. Principio analisi delle componenti (PCA) degli spettri di massa in the livello tecnico replicare (A) e le classi di picco (B). I colori sono stati codificati come gli stessi colori usati nella Figura 2.
Figura complementare 2. Un esempio di classi picchi selezionati sulla base del bidirezionale clustering. Classi picco avente la stessa etichetta vengono colorate con lo stesso colore.
Figura supplementare 3. Risultati Cross-validazione del progetto di identificazione sulla base dei database personalizzati in BioNumerics.
Figura supplementare identificazione 4. Batterio basato sulla corrispondenza di picco utilizzando database personalizzati.
Discussione
Questa dimostrazione ha mostrato le procedure dettagliate di caratterizzazione e identificazione di batteri utilizzando MALDI-TOF MS e un database personalizzato. Rispetto ai metodi tradizionali molecolari, per esempio, metodi di impronte 16S rDNA sequenziamento, MS MALDI-TOF facilitano più rapida identificazione di diversi batteri. A causa della sua robustezza, questa tecnica è ampiamente utilizzata per caratterizzare i batteri, virus, funghi e lieviti dall'ambiente e in ambito clinico 1,14-16. Inoltre, MALDI-TOF MS è stato segnalato per permettere, in alcuni casi, a risoluzione più elevata tassonomica 1. Ad esempio, B. sp. A, B, D, ed E, pur tendendo a raggrupparsi (Figura 2), erano chiaramente separati e la somiglianza tra gli spettri di diversa B. sp. era inferiore al 80% (Figura 2). Al contrario, le sequenze 16S rDNA di questi isolati avevano alta somiglianza, che non potrebbero essere utilizzati per differenziare questi isolatia livello di specie. Le sequenze 16S rDNA di B. sp. B e D hanno 99% di somiglianza basata sull'analisi allineamento multiplo, mentre le sequenze di B. sp. A ed E mostrano 95% e il 96% di somiglianza, rispettivamente, alle sequenze di B. sp. B e D. I valori anomali sono stati anche osservati. Ad esempio, B. sp. C e F raggruppati lontano da altri B. sp. (Figura 2). La comparsa di isolati outlier indica che l'analisi di clustering di spettri di massa non significa necessariamente stabilire relazioni filogenetiche. L'ambiente da cui sono stati ottenuti isola può anche influenzare la massa spettri clustering. Ad esempio, Brevibacterium specie specie Kocuria che sono stati isolati da speleothem umido e B. sp B e. F che è stato isolato da stalattite gocciolamento tendeva a raggrupparsi insieme (Tabella 1, Figura 2), ma sono necessarie ulteriori ricerche per verificare se questo è osservata in un gruppo più ampio di isolAtes.
Questa tecnica basata biblioteca ha anche alcune limitazioni. Caratterizzazione è di solito basato su database. Database commerciali correnti sono costituite principalmente da ceppi batterici, in particolare quelli patogeni. Questi database commerciali sono più utili in ambienti di laboratorio di microbiologia clinica. Per caratterizzare isolati ambientali così come i virus, funghi e lieviti, database personalizzati devono essere costruiti utilizzando grandi collezioni di deformazione. I parametri utilizzati nelle analisi di follow-up può anche avere bisogno di essere ottimizzato per aumentare la risoluzione tassonomica, soprattutto ai sottospecie e livelli di deformazione. Ad esempio, l'S: N utilizzato per il rilevamento di picco in questa dimostrazione era 10. Questo valore è appropriato per l'identificazione livello di specie, ma per l'identificazione livello di deformazione, questo valore può essere necessario abbassare. Dal momento che questi parametri di lavorazione e trattamento dei dati dei flussi di lavoro sono a volte definito dall'utente in molti pacchetti software, ad esempio, ClinProTools e BionumeRICS, una ottimizzazione dei valori dei parametri e la selezione dei flussi di lavoro adeguati sarà probabilmente necessario per ottimizzare l'analisi dei dati. In questa dimostrazione, i parametri corrispondenti di picco, i valori di soglia utilizzati nel progetto di identificazione e la convalida incrociata tutte ottimizzazione necessari per migliorare i tassi di identificazione corrette. Per trovare un metodo e / o la procedura di ottimizzare questi parametri è di grande interesse per il nostro laboratorio. Ad esempio, un approccio potrebbe coinvolgere disegno fattoriale statistica, che abbiamo usato recentemente per ottimizzare MALDI-TOF automatico di acquisizione dati 17. Ulteriori applicazioni e valorizzazione del fingerprinting microbico basato su MS MALDI-TOF futuri includono la costruzione di ampiamente disponibili, database più grandi di batteri ambientali e / o di microrganismi non-batterici e caratterizzazione di colture miste 18 e comunità microbiche.
Divulgazioni
Authors Vranckx and Janssens are employees of Applied Maths NV, the manufacturer of data analysis software used in this video. Applied Maths NV provided select software modules highlighted in this video as well as a portion of the publication costs associated with this video.
Riconoscimenti
This work was supported by the New College of Interdisciplinary Arts and Sciences at Arizona State University, Applied Maths NV, and by the National Science Foundation (ROA Supplement to Award No. MCB0604300). Any opinions, findings, and conclusions or recommendations expressed in this material are those of the author(s) and do not necessarily reflect the views of the National Science Foundation.
Materiali
| Name | Company | Catalog Number | Comments |
| α-cyano-4-hydroxy-cinnamic acid | ACROS Organics | 163440050 | ≥ 97%, CAS 28168-41-8 |
| MALDI calibration kit | Aigma-Aldrich | MSCAL1-1KT | This kit also contains acetonitrile, trifluoroacetic acid , sinapinic acid, etc. |
| MALDI target plate | Bruker Daltonics | 280800 | Polished Steel |
| Bruker Microflex LRF MALDI-TOF mass spectrometer | Bruker Daltonics | ||
| Bruker FlexControl software | Bruker Daltonics | version 3.0 | |
| Bruker FlexAnalysis software | Bruker Daltonics | version 3.0 | |
| Bionumerics software | Applied Maths | version 7.1 |
Riferimenti
- Sandrin, T. R., Goldstein, J. E., Schumaker, S. MALDI TOF MS profiling of bacteria at the strain level: A review. Mass Spectrom Rev. 32 (3), 188-217 (2013).
- Siegrist, T. J., et al. Discrimination and characterization of environmental strains of Escherichia coli by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry (MALDI-TOF-MS). J Microbiol Meth. 68 (3), 554-562 (2007).
- Goldstein, J. E., Zhang, L., Borror, C. M., Rago, J. V., Sandrin, T. R. Culture conditions and sample preparation methods affect spectrum quality and reproducibility during profiling of Staphylococcus aureus with matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Lett Appl Microbiol. 57 (2), 144-150 (2013).
- Benagli, C., et al. A rapid MALDI-TOF MS identification database at genospecies level for clinical and environmental Aeromonas strains. Plos One. 7 (10), (2012).
- Sauer, S., Kliem, M. Mass spectrometry tools for the classification and identification of bacteria. Nat Rev Microbiol. 8 (1), 74-82 (2010).
- Bohme, K., et al. SpectraBank: An open access tool for rapid microbial identification by MALDI-TOF MS fingerprinting. Electrophoresis. 33 (14), 2138-2142 (2012).
- Walker, J., Fox, A. J., Edwards-Jones, V., Gordon, D. B. Intact cell mass spectrometry (ICMS) used to type methicillin-resistant Staphylococcus aureus: media effects and inter-laboratory reproducibility. J Microbiol Meth. 48 (2-3), 117-126 (2002).
- Ruelle, V., El Moualij, B., Zorzi, W., Ledent, P., De Pauw, E. Rapid identification of environmental bacterial strains by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Commun Mass Sp. 18 (18), 2013-2019 (2004).
- Sedo, O., Sedlacek, I., Zdrahal, Z. Sample Preparation Methods for Maldi-MS Profiling of Bacteria. Mass Spectrom Rev. 30 (3), 417-434 (2011).
- Swatkoski, S., Russell, S., Edwards, N., Fenselau, C. Analysis of a model virus using residue-specific chemical cleavage and MALDI-TOF mass spectrometry. Anal Chem. 79 (2), 654-658 (2007).
- Freiwald, A., Sauer, S. Phylogenetic classification and identification of bacteria by mass spectrometry. Nat Protoc. 4 (5), 732-742 (2009).
- Drevinek, M., Dresler, J., Klimentova, J., Pisa, L., Hubalek, M. Evaluation of sample preparation methods for MALDI-TOF MS identification of highly dangerous bacteria. Lett Appl Microbiol. 55 (1), 40-46 (2012).
- Lasch, P., et al. MALDI-TOF mass spectrometry compatible inactivation method for highly pathogenic microbial cells and spores. Anal Chem. 80 (6), 2026-2034 (2008).
- Usbeck, J. C., Kern, C. C., Vogel, R. F., Behr, J. Optimization of experimental and modelling parameters for the differentiation of beverage spoiling yeasts by Matrix-Assisted-Laser-Desorption/Ionization Time-of-Flight Mass Spectrometry (MALDI-TOF MS) in response to varying growth conditions. Food Microbiol. 36 (2), 379-387 (2013).
- Del Chierico, F., et al. MALDI-TOF MS proteomic phenotyping of filamentous and other fungi from clinical origin. J Proteomics. 75 (11), 3314-3330 (2012).
- Vitale, R., Roine, E., Bamford, D. H., Corcelli, A. Lipid fingerprints of intact viruses by MALDI-TOF/mass spectrometry. Bba-Mol Cell Biol L. 1831 (4), 872-879 (2013).
- Zhang, L., Borror, C. M., Sandrin, T. R. A designed experiments approach to optimization of automated data acquisition during characterization of bacteria with MALDI-TOF mass spectrometry. Plos One. 9 (3), (2014).
- Christner, M., Rohde, H., Wolters, M., Sobottka, I., Wegscheider, K., Aepfelbacher, M. Rapid identification of bacteria from positive blood culture bottles by use of matrix-assisted laser desorption-ionization time of flight mass spectrometry fingerprinting. J ClinMicrobiol. 48 (5), 1584-1591 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon
Utilizziamo i cookies per migliorare la tua esperienza sul nostro sito web.
Continuando a utilizzare il nostro sito web o cliccando “Continua”, accetti l'utilizzo dei cookies.
