Mappatura quantitativa della deformazione di un aneurisma dell'aorta addominale
Panoramica
Fonte: Hannah L. Cebull1, Arvin H. Soepriatna1, John J. Boyle2 e Craig J. Goergen1
1 Weldon School of Biomedical Engineering, Purdue University, West Lafayette, Indiana
2 Ingegneria meccanica e scienza dei materiali, Washington University di St. Louis, St Louis, Missouri
Il comportamento meccanico dei tessuti molli, come vasi sanguigni, pelle, tendini e altri organi, è fortemente influenzato dalla loro composizione di elastina e collagene, che forniscono elasticità e forza. L'orientamento delle fibre di queste proteine dipende dal tipo di tessuto molle e può variare da una singola direzione preferita a reti intricate, che possono alterarsi nei tessuti malattiati. Pertanto, i tessuti molli spesso si comportano in modo anisotropico a livello cellulare e di organi, creando la necessità di una caratterizzazione tridimensionale. Lo sviluppo di un metodo per stimare in modo affidabile i campi di deformazione all'interno di tessuti o strutture biologiche complesse è importante per caratterizzare e comprendere meccanicamente la malattia. La deformazione rappresenta il modo in cui i tessuti molli si deformano relativamente nel tempo e può essere descritta matematicamente attraverso varie stime.
L'acquisizione dei dati di immagine nel tempo consente di stimare la deformazione e la deformazione. Tuttavia, tutte le modalità di imaging medico contengono una certa quantità di rumore, il che aumenta la difficoltà di stimare con precisione il ceppo in vivo. La tecnica qui descritta supera con successo questi problemi utilizzando un metodo di stima della deformazione diretta (DDE) per calcolare campi di deformazione 3D che variano spazialmente dai dati dell'immagine volumetrica.
Gli attuali metodi di stima della deformazione includono la correlazione delle immagini digitali (DIC) e la correlazione del volume digitale. Sfortunatamente, DIC può solo stimare con precisione la deformazione da un piano 2D, limitando gravemente l'applicazione di questo metodo. Sebbene utili, i metodi 2D come DIC hanno difficoltà a quantificare la deformazione nelle regioni che subiscono la deformazione 3D. Questo perché il movimento fuori piano crea errori di deformazione. La correlazione digitale del volume è un metodo più applicabile che divide i dati del volume iniziale in regioni e trova la regione più simile del volume deformato, riducendo così l'errore fuori piano. Tuttavia, questo metodo si dimostra sensibile al rumore e richiede ipotesi sulle proprietà meccaniche del materiale.
La tecnica qui dimostrata elimina questi problemi utilizzando un metodo DDE, rendendolo così molto utile nell'analisi dei dati di imaging medico. Inoltre, è robusto a ceppo alto o localizzato. Qui descriviamo l'acquisizione di dati a ultrasuoni 4D volumetrici gated, la sua conversione in un formato analizzabile e l'uso di un codice Matlab personalizzato per stimare la deformazione 3D e le corrispondenti deformazioni Green-Lagrange, un parametro che descrive meglio le grandi deformazioni. Il tensore di deformazione green-lagrange è implementato in molti metodi di stima della deformazione 3D perché consente di calcolare F da un Least Squares Fit (LSF) degli spostamenti. L'equazione seguente rappresenta il tensore di deformazione di Green-Lagrange, E, dove F e I rappresentano rispettivamente il gradiente di deformazione e il tensore di identità del secondo ordine.
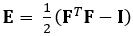 (1)
(1)
Principi
L'ecografia 4D è un volume dinamico che viene acquisito utilizzando un motore a trasduzione lineare collegato a un trasduttore ad ultrasuoni, consentendo l'acquisizione di loop video sequenziali cardiaci e respiratori in una regione di interesse. Questo metodo è utile per visualizzare strutture complesse come il cuore, dove l'ipertrofia o l'infarto causano geometrie uniche, o aneurismi aortici, dove l'espansione e la dissezione asimmetrica dei vasi si verificano spesso in vasi tortuosi. Inoltre, i dati 4D possono fornire informazioni spaziali e temporali ad alta risoluzione, che è anche importante per l'imaging cardiovascolare.
Il metodo DDE applicato ai dati ecografici 4D è superiore ad altri metodi perché utilizza la registrazione delle immagini non rigida. I tensori del gradiente di deformazione sono tradizionalmente stimati dai campi di spostamento seguendo la correlazione del volume digitale. Al contrario, il metodo DDE stima intrinsecamente i tensori del gradiente di deformazione durante la registrazione del volume ottimizzando una funzione di deformazione che viene accuratamente scelta per essere direttamente analoga al tensore di deformazione. La funzione di deformazione dipende sia dalla posizione spaziale che dal parametro di deformazione (p):
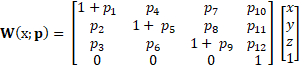 (2)
(2)
I primi tre elementi di questa funzione rappresentano il tensore del gradiente di deformazione, F, che consente di incorporare direttamente il calcolo della deformazione nella funzione di deformazione. Questo metodo di deformazione ha dimostrato di aumentare l'accuratezza e la precisione della stima della deformazione rispetto a tecniche precedenti simili perché consente deformazioni grandi o localizzate che si trovano comunemente nei tessuti molli.
Procedura
1. Configurazione ad ultrasuoni 4D
- Quando si utilizza il software di imaging, utilizzare un laptop in grado di eseguire software di calcolo matematico per automatizzare il processo di acquisizione 4D. Collegare il laptop con questo codice personalizzato al sistema ad ultrasuoni tramite la porta USB. Si noti che il software di imaging ha una funzione di ecografia 4D integrata nel software.
- Dopo aver acceso il sistema a ultrasuoni, impostare l'unità di monitoraggio fisiologico assicurandosi anche che i pulsanti della frequenza cardiaca e della temperatura siano accesi. Inizializzare lo stadio del motore 3D collegato al supporto del trasduttore.
- Utilizzare lo stadio appropriato e il trasduttore ad ultrasuoni per l'acquisizione delle immagini. Assicurarsi che vengano effettuate tutte le connessioni corrette.
- Procedere con l'anestesia e la preparazione dell'animale per l'imaging. Aggiungere unguento oftalmico agli occhi per prevenire l'essiccazione corneale, fissare le zampe agli elettrodi dello stadio e inserire una sonda di temperatura rettale lubrificata. Rimuovere la pelliccia nell'area di interesse utilizzando una crema depilatoria.
- Assicurarsi che la crema depilatoria sia completata rimossa. Quindi, applicare una generosa quantità di gel trasduttore ad ultrasuoni riscaldato all'animale. Questo è particolarmente importante per creare una buona connessione su tutta la regione di interesse per l'imaging 4D.
2. Acquisizione di ultrasuoni 4D
- Inizia un nuovo studio sul sistema a ultrasuoni e apri la finestra di imaging in modalità B (modalità luminosità). Abbassare il trasduttore sull'animale e individuare la regione di interesse utilizzando le manopole degli assi x e y sul palco, assicurandosi che la frequenza respiratoria non diminuisca in modo sostanziale. Monitora questo nella parte inferiore dello schermo.
- Posizionare il trasduttore all'interno del centro della regione di interesse. Da lì, approssimare la distanza necessaria affinché il trasduttore si muova su e giù in modo tale da includere l'intera regione di interesse.
- Immettere le dimensioni approssimative nel codice del software di calcolo, inclusa una dimensione del gradino che in genere è di ~ 0,08 mm per l'imaging dell'aneurisma dell'aorta addominale. Inizia a eseguire il codice dopo aver assicurato che il cuore e le frequenze respiratorie dell'animale siano stabili. Questo è importante per ridurre gli errori durante la ricostruzione delle immagini.
- Dopo aver completato l'acquisizione dell'immagine, esportare i dati come file XML non elaborati.
3. Conversione dei dati ad ultrasuoni 4D
- Inserisci i file XML grezzi in un software in grado di convertire i dati nel formato corretto per l'analisi delle macchie 3D. Qui usiamo Matlab per convertire i file XML in file MAT. Lo script Matlab completo è disponibile qui.
- Per una corretta conversione, sarà necessario inserire anche il numero di fotogrammi, la dimensione del passo e la risoluzione di output desiderata.
- Dopo aver ricampionato la matrice attraverso il piano, importate il nuovo file MAT nel codice di analisi della deformazione 3D.
4.3D Analisi del codice di deformazione
- Iniziate l'analisi regolando correttamente il file MAT importato. Ad esempio, potrebbe essere necessario ridimensionare il volume dell'immagine per ridurre i tempi di calcolo.
- Immettere l'area da analizzare e determinare il modello di mesh appropriato per segmentare i dati dell'immagine come semplici caselle o poligoni scelti manualmente. Potrebbe essere necessario modificare le dimensioni della casella delle regioni e la spaziatura tra i punti centrali per ogni set di dati. I numeri ottimali scelti per le dimensioni della casella saranno intorno all'ordine dei pixel della feature da tracciare, che può essere approssimato osservando il numero di pixel in due dimensioni in una sezione. La spaziatura delle caselle determinerà la risoluzione dei campi di deformazione. Più caselle aumenteranno la risoluzione, ma potrebbero anche aumentare sostanzialmente il tempo di calcolo.
- Inizia a calcolare i giacobini e i gradienti iterativamente all'interno di ciascuna di queste regioni. Al termine del precalcolo, applicare la funzione di deformazione.
- Calcolate il tensore del gradiente di deformazione. Prima calcola la deformazione, quindi calcola gli autovalori e gli autovettori usando il metodo di stima della deformazione diretta.
- Tracciate questi risultati nei piani desiderati utilizzando una tecnica come la mappatura a colori di un piano di taglio per rappresentare il campo di deformazione sulla regione di interesse.
Risultati
Utilizzando la procedura sopra descritta, è stata acquisita l'ecografia 4D di un aneurisma dell'aorta addominale ad antropo indotto da angiotensina II (AAA) di un topo. Più loop video EKV ad asse corto sono stati acquisiti lungo l'aorta e combinati per creare dati 4D, come mostrato nella Figura 1. Questi dati sono stati quindi convertiti in un file MAT utilizzando un codice personalizzato, che è stato quindi analizzato in un codice di calcolo della deformazione 3D utilizzando una funzione di deformazione. Dopo aver ottimizzato i parametri del codice per un set di dati specifico, è stata prodotta una vista rappresentativa ad asse lungo con i corrispondenti valori di deformazione e un grafico di visualizzazione delle sezioni 3D con una mappa dei colori della deformazione sovrapposta (Figura 2). Questa tecnica DDE e i dati di deformazione evidenziano le variazioni spaziali eterogenee della deformazione, in particolare quando è presente un trombo. Questi risultati possono quindi essere correlati con la struttura dei vasi per determinare la relazione tra deformazione in vivo e composizione dell'aneurisma.

Figura 1: I loop di visualizzazione eKV (ECG-gated kilohertz) dell'aorta vengono acquisiti da posizioni di partenza e di fine immesse manualmente, seguendo una dimensione del passo di 0,2 mm.
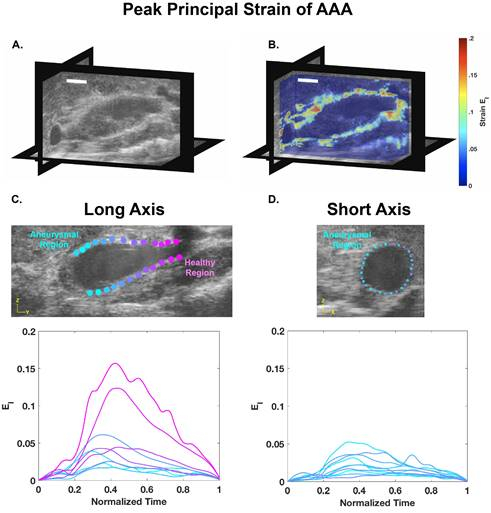
Figura 2: Dati ecografici 4D ad alta frequenza di un aneurisma dell'aorta addominale murino sezionante rappresentato in sistole (A) con principali campi di deformazione stimati e sovrapposti (B) (Scalebar = 5 mm). Viste ad asse lungo e corto che rappresentano sia le regioni aneurismatiche che le regioni sane corrispondenti al ceppo principale su un ciclo cardiaco (sistole: t = 0,4) (C, D). Questi dati mostrano livelli di ceppo relativamente elevati nelle regioni sane e valori di deformazione ridotti all'interno dell'aneurisma di dissezione.
Applicazione e Riepilogo
La caratterizzazione meccanica localizzata in vivo è una parte importante della comprensione della crescita e del rimodellamento dei tessuti biologici. Rispetto agli approcci esistenti, la procedura di quantificazione della deformazione qui descritta utilizza un metodo migliorato per calcolare con precisione la deformazione 3D attraverso la deformazione ottimale dell'immagine non deformata prima della correlazione incrociata. Questo metodo non utilizza alcuna ipotesi materiale per determinare i ceppi all'interno dei volumi di tessuto. Sfortunatamente, la stima della deformazione è affidabile solo fino a una dimensione del kernel di 15x15x15 voxel quando si utilizzano i dati a ultrasuoni, suggerendo che questo approccio DDE potrebbe non rilevare caratteristiche sottili all'interno di un campo di deformazione. Nonostante questa limitazione, rimane uno strumento importante per studiare le risposte meccaniche, diagnosticare la patologia e migliorare i modelli di malattia.
Molte aree di ricerca oltre agli aneurismi aortici possono beneficiare di questo strumento di misurazione della deformazione. Lo sforzo cardiaco può anche essere facilmente quantificato usando questo metodo. Poiché il miocardio subisce una deformazione 3D durante il ciclo cardiaco, quantificare la deformazione in tre dimensioni è parte integrante per caratterizzare in modo affidabile la dinamica di questo tessuto. Dati affidabili sui ceppi sono particolarmente importanti quando si monitora la progressione della malattia in modelli animali.
L'analisi 3D del ceppo può anche essere applicata all'imaging ecografico intestinale. La caratterizzazione meccanica del tessuto intestinale è più comunemente condotta in vitro. Tuttavia, questa non è sempre una vera rappresentazione del comportamento effettivo dell'intestino in vivo a causa degli effetti delle strutture circostanti. Come esempio di traduzione clinica di questo approccio, il calcolo del ceppo da immagini di fibrosi intestinale a causa di una pressione luminale anormale potrebbe fornire una diagnosi precoce di aree problematiche che richiedono un intervento chirurgico.
Oltre alle applicazioni su larga scala, questo metodo può essere applicato anche a livello cellulare utilizzando tecniche di imaging ad alta risoluzione, come la microscopia confocale. Caratterizzare la matrice extracellulare è importante per capire come le cellule comunicano. Sono state condotte molte ricerche sulla caratterizzazione biochimica, ma capire come la comunicazione può essere condotta attraverso risposte meccaniche richiede una comprensione della deformazione e della deformazione. La deformazione di massa non è vantaggiosa perché non c'è modo di determinare l'origine del cambiamento di deformazione. L'applicazione di un approccio DDE ad alta risoluzione potrebbe rivelare direttamente come la matrice extracellulare risponde ai cambiamenti meccanici.
RICONOSCIMENTI
Vorremmo ringraziare John Boyle, Guy Genin e Stavros Thomopoulos per il contributo del codice Matlab personalizzato DDE in grado di stimare direttamente il ceppo Lagrange-Green.
Vai a...
Video da questa raccolta:

Now Playing
Mappatura quantitativa della deformazione di un aneurisma dell'aorta addominale
Biomedical Engineering
4.6K Visualizzazioni

Imaging di campioni biologici con microscopia ottica e confocale
Biomedical Engineering
35.8K Visualizzazioni

Imaging di campioni biologici con microscopio elettronico a scansione (SEM)
Biomedical Engineering
23.8K Visualizzazioni

Biodistribuzione dei vettori di nanofarmaci: applicazioni del microscopio elettronico a scansione (SEM)
Biomedical Engineering
9.3K Visualizzazioni

Imaging a ultrasuoni ad alta frequenza dell'aorta addominale
Biomedical Engineering
14.5K Visualizzazioni

Tomografia fotoacustica per l'immagine di sangue e lipidi nell'aorta infrarenale
Biomedical Engineering
5.7K Visualizzazioni

Imaging a risonanza magnetica cardiaca
Biomedical Engineering
14.8K Visualizzazioni

Simulazioni fluidodinamiche computazionali del flusso sanguigno in un aneurisma cerebrale
Biomedical Engineering
11.8K Visualizzazioni

Imaging a fluorescenza nel vicino-infrarosso di aneurismi dell'aorta addominale
Biomedical Engineering
8.3K Visualizzazioni

Tecniche non invasive di misurazione della pressione sanguigna
Biomedical Engineering
11.9K Visualizzazioni

Acquisizione e analisi di un segnale ECG (elettrocardiogramma)
Biomedical Engineering
105.6K Visualizzazioni

Resistenza alla trazione dei biomateriali riassorbibili
Biomedical Engineering
7.5K Visualizzazioni

Imaging micro-CT di un midollo spinale di topo
Biomedical Engineering
8.0K Visualizzazioni

Visualizzazione della degenerazione dell'articolazione del ginocchio dopo lesione del LCA non invasiva nei ratti
Biomedical Engineering
8.2K Visualizzazioni

Imaging combinato SPECT e CT per la visualizzazione della funzionalità cardiaca
Biomedical Engineering
11.0K Visualizzazioni