Method Article
Analisi proteomica comparativa di interi reni, midollo e tubuli corticali nella patogenesi diabetica del danno renale nei topi
In questo articolo
Riepilogo
Qui, presentiamo un protocollo dettagliato per l'analisi proteomica dell'intero rene, del tubulo corticale isolato e dei proteomi midollari. Lo studio confronta anche i proteomi regionali in un modello murino diabetico e in topi non diabetici.
Abstract
Definire la sequenza degli eventi nella malattia renale è la pietra angolare della pratica clinica nel kit di strumenti del nefrologo. Le analisi proteomiche tissutali sono un approccio significativo per comprendere i processi fisiologici e molecolari fondamentali della fisiopatologia renale. I metodi e i protocolli che presentiamo qui consentiranno la dissezione molecolare del rene in ogni specifica regione di interesse correlata alle sequele della malattia. Per determinare gli effetti della malattia su specifiche regioni e strutture renali con funzioni uniche, gli obiettivi di questo protocollo sono dimostrare tecniche semplificate di compartimentazione renale del topo e di isolamento del tubulo corticale renale in tandem con flussi di lavoro proteomici quantitativi semplificati e privi di marcatura. La combinazione di questi metodi aiuterà nell'identificazione di pattern molecolari perturbati nell'intero rene, nei compartimenti midollari e nelle strutture dei tubuli corticali dei reni, con l'obiettivo finale della proteomica a singola cellula in contesti patologici. L'applicazione di questi metodi in quasi tutti i modelli di malattia sarà utile per delineare i meccanismi della patologia correlata alla disfunzione renale.
Introduzione
La malattia renale cronica (CKD) è una delle principali preoccupazioni della medicina moderna, con oltre 86 miliardi di dollari di spese sanitarie solo negli Stati Uniti. In tutto il mondo, l'incidenza della malattia renale cronica è in aumento con la prevalenza del diabete e delle comorbidità renali associate. Quasi il 14% della popolazione statunitense ha la malattia renale cronica (rapporto annuale USRDS 2024). Inoltre, la nefropatia diabetica è una forma di CKD e la principale causa di malattia renale allo stadio terminale (ESRD) e il 60% dei pazienti con ESRD ha il diabete 1,2,3. Il diabete colpisce tutte le strutture renali e i tipi di cellule dei nefroni, l'unità funzionale dei reni. Come mostrato nella Figura 1, diverse porzioni di nefroni sono contenute nella corteccia renale e nelle regioni del midollo. La maggior parte del rene è composta da tubuli. La disfunzione dei tubuli renali e le lesioni strutturali contribuiscono in modo significativo allo sviluppo della nefropatia diabetica (DN) e questi cambiamenti si correlano bene con il tasso di declino della funzione renale 4,5,6,7,8. All'inizio del diabete, in risposta all'aumento del glucosio e all'assorbimento di sodio associato e alla richiesta di proteine di trasporto di membrana in tutti i segmenti tubuli, i tubuli vanno incontro a ipertrofia. Con l'aumento del danno microvascolare più tardi nel diabete, i tubuli mostrano atrofia e dilatazione, mentre c'è fibrosi ed espansione dell'interstizio9. Precedenti studi del nostro laboratorio hanno trovato proteomi alterati e un'abbondanza di proteine di risposta allo stress nei tubuli corticali di topi diabetici10,11. Il midollo è importante per regolare la concentrazione nelle urine e la disfunzione durante la malattia renale è associata allo stress ossidativo, con il diabete che porta a una diminuzione della tensione di ossigeno in questa regione del rene a causa dell'aumento del consumo di ossigeno dalle attività metaboliche, dell'aumento dell'attività delle proteine di trasporto della membrana e del danno microvascolare 12,13,14.
Comprendere i meccanismi dettagliati di sviluppo e progressione della DN è uno sforzo continuo che richiederà approcci nuovi e integrati che invochino la modellazione della malattia e la profilazione molecolare dei processi di segnalazione, i cambiamenti adattativi nella dinamica delle proteine cellulari e la definizione precisa delle cellule renali e dei componenti tissutali interessati da lesioni in condizioni croniche. La proteomica, la metabolomica e la trascrittomica offrono la possibilità di sondare analiticamente i meccanismi molecolari delle malattie renali. L'omica è un campo relativamente nuovo che utilizza approcci di biologia dei sistemi per ottenere una comprensione più globale dei sistemi biologici. La proteomica è stata un potente strumento omico in nefrologia negli ultimi decenni. La ricerca sui biomarcatori si è espansa negli ultimi 20 anni, come indicato dalla crescita delle pubblicazioni, anche se è necessario un ampio lavoro per implementare pienamente questi risultati nella clinica15. Con le popolazioni di cellule tubulari molto diverse e i rispettivi ruoli funzionali all'interno della corteccia renale e del midollo, l'analisi proteomica di interi reni può mascherare cambiamenti unici associati a strutture specifiche all'interno di queste diverse regioni. Pertanto, gli obiettivi di questo studio sono dimostrare la separazione della corteccia renale e del midollo, nonché la separazione dei tubuli corticali dai glomeruli, seguita da protocolli dettagliati per la preparazione di estratti proteici da strutture isolate per analisi di spettrometria di massa e bioinformatica all'avanguardia. I modelli murini di nefropatia diabetica sono fondamentali per definire i meccanismi di progressione della malattia. Per questo studio, abbiamo utilizzato il topo transgenico OVE26, che sviluppa il diabete di tipo I ad esordio precoce e mostra caratteristiche di DN in fase precoce e tardiva nell'uomo, tra cui 1) un aumento precoce e un successivo declino della velocità di filtrazione glomerulare, 2) ipertrofia renale, 3) ispessimento della membrana basale glomerulare ed espansione mesangiale, 4) grave proteinuria e 5) fibrosi tubulointerstiziale9, 16,17. Topi di due mesi sono stati scelti per dimostrare cambiamenti proteomici nei compartimenti tubulici prima di lesioni strutturali evidenti. Come precedentemente riportato e mostrato nella Figura 2, i topi OVE26 di 2 mesi mostrano un'espansione della matrice mesangiale glomerulare (Figura 2, freccia verde) e una grave proteinuria17, senza alterazioni istologiche evidenti dei tubuli prossimali (Figura 2, freccia gialla) nei giovani topi diabetici. Qui presentiamo un approccio combinato di proteomica quantitativa per la caratterizzazione dell'intero rene, del midollo e dei tubuli corticali per chiarire e illustrare le differenze nel proteoma in ciascun compartimento con diabete.
Protocollo
Gli studi con topi OVE26 e FVB sono stati approvati dalle linee guida dell'Institutional Animal Care and Use Committee (IACUC) dell'Università di Louisville. Femmina transgenica OVE26 (diabetica; Ceppo #:005564) e FVB (non diabetico; ceppo di fondo; Ceppo #:001800) sono stati acquistati da Jackson Laboratories (Bar Harbor, ME). Gli animali sono stati mantenuti con un ciclo luce/buio di 12 ore a 25 °C e hanno avuto libero accesso all'acqua e al cibo. Tutti gli studi sono stati condotti su topi di 2 mesi.
1. Modello animale
- Isolamento renale
- Anestetizzare i topi somministrando per via intraperitoneale 80 mg/kg di ketamina e 12 mg/kg di xilazina.
- Praticare un'incisione addominale sulla linea mediana con piccole forbici chirurgiche e spostare delicatamente l'intestino verso l'esterno e verso il lato del topo utilizzando tamponi per esporre la vena cava.
- Fai un piccolo taglio nella vena cava sopra il rene destro.
- Perfondere il topo attraverso il ventricolo sinistro con 20 mL di soluzione salina fredda tamponata con fosfato (PBS: pH 7,4; 210 mg/L KH2PO4, 9 g/L NaCl, 726 mg/L Na2HPO4·7H2O) utilizzando una pompa peristaltica, alla velocità di 10 mL/min.
NOTA: Questo passaggio non è necessario se l'inclusione di proteine e cellule del sangue nei reni non è un fattore critico nelle analisi a valle. - Rimuovere i reni usando piccole forbici chirurgiche. Rimuovi il grasso in eccesso dall'esterno dei reni. Pesare i reni e metterli in PBS freddo su ghiaccio.
- Rimuovere la capsula renale.
- Mettere i reni su un piatto di vetro o una capsula di Petri posta sul ghiaccio. Fai diversi tagli trasversali attraverso i reni usando una lama di rasoio (Figura 3) per finire con 3-4 fette tra i poli superiore e inferiore dei reni.
- Riservare 1-2 fette centrali e i due poli (fette di rene cerchiate nella Figura 3) da ciascun rene da parte per fungere da campioni di "rene intero". Mettere una porzione in provette da centrifuga da 1,5 mL e aggiungere un tampone di omogeneizzazione ghiacciato (10% di glicerolo, 50 mM di HEPES, 100 mM di KCl, 2 mM di EDTA, 0,1% di NP40, 10 mM di NaF, 0,25 mM di NaVO3, 1x inibitore della proteasi HALTS) a circa 10 μL/mg di campione di tessuto (basato sulla massa stimata delle fette di rene, derivata dal peso acquisito dai reni). Lasciare il tubo sul ghiaccio.
- Per il resto delle fette trasversali centrali, stendere la fetta in piano e tagliare con cura la corteccia (esterna di 1 mm) da ciascuna fetta usando una lama di rasoio o un bisturi (Figura 3, flusso di lavoro schematico). Tenere la corteccia separata dalle regioni del midollo per il protocollo di isolamento dei tubuli corticali (sotto, passaggio 1.3).
- Posizionare una parte del midollo sezionato in provette da centrifuga da 1,5 mL, aggiungere un tampone di omogeneizzazione ghiacciato come al punto 1.1.8 e lasciare la provetta sul ghiaccio.
- Congelare a scatto in azoto liquido tutti i pezzi di rene e midollo interi rimanenti non utilizzati per l'omogeneizzazione e conservare a -80 °C.
- In alternativa, congelare tutti i compartimenti interi del rene e del midollo separati e conservarli come descritto al punto 1.1.11. Rimuoverli dal magazzino in un secondo momento per l'aggiunta del tampone di omogeneizzazione come al punto 1.1.8.
- Isolamento dei tubuli corticali
- Tritare la corteccia sezionata in pezzi di 1-3 mm su una lastra di vetro o una piastra di Petri con una lama di rasoio, formando una pasta.
- Digerire la corteccia tritata in 1 mL di collagenasi di tipo IA (1 mg/mL) per 30 minuti, a 37 °C, in un bagno d'acqua a dondolo.
- Posizionare un colino cellulare da 100 μm sopra una provetta conica da 50 mL su ghiaccio.
- Posizionare la sospensione corticale digerita sul colino cellulare da 100 μm e premere delicatamente attraverso il filtro utilizzando lo stantuffo di una siringa da 10 ml. Lavare la parte superiore del colino con 1 ml di PBS e la parte inferiore del colino con 1 ml di PBS.
- Passare il filtrato nella provetta da 50 mL attraverso un ulteriore filtro da 100 μm, quindi lavare la parte superiore del filtro con 1 mL di PBS.
- Passare il filtrato attraverso un filtro cellulare da 70 μm posizionato su una provetta conica da 50 mL su ghiaccio. I tubuli passeranno attraverso questo filtro nel filtrato (i glomeruli verranno trattenuti sul filtro da 70 μm). Lavare il colino con 1 mL di PBS.
- Centrifugare il filtrato finale a 120 × g per 2 minuti a 4 °C. Scartare l'eccesso di PBS dal pellet del tubulio.
- Controllare la purezza del pellet del tubulo corticale mediante visualizzazione al microscopio, utilizzando un obiettivo 10x. Se più del 10% della frazione contiene glomeruli, risospendere il filtrato con 1 mL di PBS e passare attraverso un filtro cellulare pulito da 70 μm senza alcun lavaggio aggiuntivo del filtro.
- Distribuire la frazione di tubuli arricchiti in provette da centrifuga da 1,5 mL e centrifugare a 2000 × g per 2 minuti a 4 °C. Scartare il surnatante.
- Aggiungere un tampone di omogeneizzazione al pellet del tubulo come per l'intero rene e il midollo (passaggi 1.1.8 e 1.1.10).
- Omogeneizzazione tissutale/estrazione di proteine
- Omogeneizzare ogni tipo di campione in provette da microcentrifuga da 1,5 mL utilizzando un pestello di plastica specifico per provette da 1,5 mL. Se la sospensione omogeneizzata è troppo densa, aggiungere un ulteriore tampone di omogeneizzazione secondo necessità.
- Lasciare la sospensione omogeneizzata sul ghiaccio per 15 minuti, seguita dalla sonicazione per 5 minuti in un sonicatore da bagno (solo interruttore on/off, nessuna impostazione regolabile sull'unità), con acqua nel bagno a 25 °C. Lasciare i campioni in ghiaccio per 10 minuti.
- Omogeneizzare brevemente a vortice (1000-1500 giri/min), quindi triturare 20 volte con una pipetta e lasciare in ghiaccio per altri 15 minuti.
- Triturare i campioni più volte prima della centrifugazione a 13.000 × g per 20 minuti a 4 °C.
- Trasferire gli estratti proteici chiari in provette pulite.
- Aliquotare 30 μl di estratto chiarificato per la stima delle proteine e la preparazione del campione per l'analisi della spettrometria di massa. Conservare il resto dei campioni a -80 °C.
2. Protocollo di digestione della mini colonna di centrifuga Suspension-Trap
- Digestione proteolitica di proteine in frammenti triptici per analisi proteomiche
- Diluire ~40-50 μg di campione (a ~2 μg/μL) in tampone di lisi del campione (10% p/v di sodio dodecil solfato [SDS] in 100 mM di trietilammonio bicarbonato (TEA-BC) pH 8,5, 40 mM di tris (2-carbossietil)fosfina [TCEP]) fino a un volume finale di 46 μL.
- Riduzione e alchilazione delle proteine
- Incubare a 65 °C per 30 minuti (la concentrazione finale di TCEP è di 20 mM).
- Aggiungere 4 μL di iodoacetammide 0,5 M in acqua di grado cromatografia-spettrometria di massa liquida (LC-MS) e incubare a temperatura ambiente (RT) al buio per 30 minuti (la concentrazione finale di iodoacetamide è 40 mM).
- Aggiungere 5 μL di acido fosforico al 12% p/p in acqua di grado LC-MS (diluire 141 μL di acido fosforico all'85% in 859 μL di acqua per una soluzione al 12%).
- In alternativa all'acido fosforico, utilizzare acido trifluoroacetico o acido formico se si esegue l'arricchimento di fosfopeptide18.
- Aggiungere 350 μl di tampone legante la trappola in sospensione (100 mM TEA-BC in metanolo 90% v/v e 10% v/v acqua pH 7,55).
- Aggiungere un campione alla colonna della trappola di sospensione. Centrifugare a 4.000 × g per 30 s a RT in un rotore ad angolo fisso per far passare ogni volume attraverso la colonna.
- Lavare con 4 x 400 μL di tampone legante la sospensione-trappola. Centrifugare a 4.000 x g per 30 s a RT in un rotore ad angolo fisso per far passare ogni lavaggio attraverso la colonna.
NOTA: Per facilitare la rimozione della SDS, ruotare la colonna nel rotore di 180° dopo ogni lavaggio. - Centrifugare a 4.000 g per 1 minuto a RT per rimuovere completamente il tampone legante (previene il gocciolamento durante la digestione).
- Trasferire la colonna di sospensione in una provetta di raccolta pulita.
- Aggiungere 2,5-5 μg di tripsina in 125 μL di 50 mM TEA-BC pH 8,5 in acqua di grado LC-MS (Trypsin Protease, MS-Grade). Il rapporto finale tra tripsina e campione è compreso tra 1:20 e 1:10.
- Incubare a 47 °C per 2 ore (non serrare il tappo; è preferibile un miscelatore termico impostato a 0 giri/min). Praticare un foro nel tappo con un ago può impedire l'accumulo di pressione nella camera superiore del trappola di sospensione (l'aumento della pressione causerà il gocciolamento della soluzione di tripsina attraverso la colonna).
- Aggiungere 80 μl di 50 mM TEA-BC pH 8,5 in acqua di grado LC-MS. Centrifugare a 4.000 g a RT per 1 minuto in un rotore ad angolo fisso per far passare ogni digestato attraverso la colonna.
- Aggiungere 80 μl di acido formico allo 0,2% v/v in acqua di grado LC-MS. Centrifugare a 4.000 g per 1 min a RT in un rotore ad angolo fisso per raccogliere con il digestato dal passaggio 2.11.
- Aggiungere 80 μl di acetonitrile al 50% v/v in acqua di grado LC-MS. Centrifugare a 4.000 g a RT per 1 min in un rotore ad angolo fisso per raccogliere con il digestato dal passaggio 2.11.
- Conservare l'eluato della trappola di sospensione a -80 °C.
- Asciugare il campione in un concentratore a vuoto.
- Conservare il campione essiccato a -80 °C.
3. Pulizia con colonna per equilibrio idrofilo-lipofilo (HLB)
- Produrre le soluzioni A (2% v/v acetonitrile/0,1% v/v acido formico) e B (80% v/v acetonitrile/0,1% v/v acido formico).
- Sciogliere il campione in 500 μl di soluzione A.
- Posizionare la colonna in un collettore a vuoto e applicare una pressione del gas a ~1-2 mL/min.
- Aggiungere 500 μl di soluzione B alla colonna HLB ed evacuare a 1-2 mL/min; Ripeti due volte.
- Aggiungere 750 μL di soluzione A alla colonna HLB ed evacuare a 1-2 mL/min; Ripeti due volte.
- Posizionare una microprovetta pulita da 2 mL nel rack sotto la colonna HLB, caricare il campione in 500 μL di soluzione A e la pressione del gas come sopra; Far passare il flusso nella colonna una seconda volta.
- Posizionare la colonna HLB sopra la provetta di scarico, aggiungere 500 μl di soluzione A ed evacuare come sopra; Ripeti due volte.
- Posizionare la colonna sopra una microprovetta pulita da 2 mL, aggiungere 500 μl di soluzione B ed evacuare come sopra; Ripeti due volte.
- Congelare l'eluato a -80 °C, quindi asciugarlo in un concentratore sottovuoto. Conservare il residuo essiccato a -80 °C.
- Sciogliere il residuo in 20 μl di acetonitrile al 2% v/v di acido formico allo 0,1% v/v prima dell'analisi MS. Utilizzare uno spettrofotometro per stimare la concentrazione di peptidi a un'assorbanza di 205 nm.
4. Analisi di spettrometria di massa
- Cromatografia liquida monodimensionale in fase inversa (LC)-MS/MS
- Iniettare una massa uguale di peptidi (~600 ng) su nano-LC 1-dimensionale e frazionarla su colonne C18 a fase invertita.
- Eluire i peptidi direttamente nell'MS a una tensione di spruzzo di 1,8 kV, con un tubo di trasferimento ionico mantenuto a 250 °C.
- Acquisisci gli spettri in modalità top 20 ionica dipendente dai dati, in cui il frammento MS/MS più intenso viene rimosso dalla coda di analisi per migliorare la profondità della mappatura ionica su ioni a bassa abbondanza.
5. Analisi dei dati e bioinformatica
- Modalità di trattamento dei dati
- Dopo l'assegnazione spettrale e l'identificazione di peptidi e proteine in PEAKS 12.0 (software di analisi dei dati LC-MS/MS), inviare gli elenchi di proteine per l'analisi in MetaboAnalyst 6.0 (piattaforma di analisi dei dati metabolomici) utilizzando l'approccio di analisi statistica a fattore singolo (https://www.metaboanalyst.ca/).
- Utilizza il software di analisi dei dati LC-MS/MS per cercare e filtrare gli spettri utilizzando rigorosi criteri di tasso di falsa scoperta (FDR) (1% di proteine e peptidi) rispetto al database FASTA Mouse Reviewed.
NOTA: I programmi di ricerca alternativi prontamente disponibili includono Maxquant, Proteome Discoverer, tra le altre opzioni ampiamente utilizzate. - Specificare la carbamidometilazione della cisteina come modifica fissa. Specificare l'ossidazione della metionina come modifica variabile.
- Converti gli elenchi in formato CSV (Comma Separated Values) e le etichette di gruppo/esempio assegnate a ogni colonna e riga. Questo formato è flessibile nella piattaforma di analisi dei dati di metabolomica.
- Imputare i valori mancanti utilizzando la regola del valore minimo di 1/5 per le variabili mancanti.
- Filtra i dati per escludere i valori altamente variabili utilizzando l'intervallo interquartile, in cui viene utilizzata una varianza del 40% per il limite.
- Normalizza i valori all'intensità mediana, al log10 trasformato e alla media centrata per il ridimensionamento. Utilizzare altri approcci di normalizzazione, tenendo presente che i risultati possono variare.
- Esegui i test T in modo non abbinato con un valore di taglio del valore p < 0,05. Eseguire più correzioni di test in questa fase.
NOTA: Questo studio non ha eseguito questa correzione per il set di dati rappresentativo ai fini della presentazione dei dati. - Indica i grafici del vulcano per una variazione di 1,5 volte (FC) e p < 0,05.
- Generare mappe di calore ed eseguire analisi discriminanti parziali dei minimi quadrati (PLSDA) e analisi statistiche (variabile di importanza della proiezione [VIP]) dei gruppi campione.
- I potenziali candidati per il follow-up sono abbastanza robusti da eclissare 1,5FC e p-val < 0,05 o VIP > 1,0, come indicato nell'analisi PLSDA.
- Genera diagrammi di Venn per illustrare le differenze nel proteoma dell'intero rene, tubulo e midollo nei topi OVE26.
Risultati
Complessivamente, le identificazioni proteiche totali per ciascun tipo di campione sono state: 1) rene intero (1438), 2) midollo (2145) e tubuli corticali (1859). Dopo l'elaborazione dei dati in MetaboAnalyst 6.0, il filtraggio dei dati e l'imputazione, le identificazioni delle proteine analizzate per ciascun compartimento renale sono state: rene intero (455), midollo (997) e tubuli corticali (896). La Figura 4 mostra i cambiamenti proteomici globali nel rene di topo diabetico OVE26. L'analisi quantitativa senza marcatura (LFQ) consente una copertura del proteoma ad alta profondità, introducendo al contempo una maggiore variabilità nel set di dati totale rispetto alla marcatura ionica reporter (TMT, iTRAQ ecc.). Un approccio per superare la variabilità LFQ consiste nell'aumentare il numero di campioni analizzati per ciascuna condizione. Qui, abbiamo utilizzato un approccio di quantificazione basato sull'intensità degli algoritmi di ricerca PEAKS con un tasso di falsa scoperta (FDR) dell'1% a livello di assegnazione di peptidi e proteine per consentire una robusta generazione di set di dati nel nostro confronto accoppiato di due animali in ciascuna condizione con repliche tecniche incluse. La Figura 4A illustra il log2FC (variazione di piegatura) tracciato rispetto al valore p non aggiustato (valore p < 0,05). In genere, il valore p verrà corretto per i test di confronto multipli quando l'intento è quello di convalidare proteine candidate robuste per il follow-up ortogonale. Qui, mostriamo l'intero confronto del proteoma non aggiustato tra OVE26 e FVB nel grafico del vulcano a scopo di presentazione. Come panoramica iniziale dell'analisi quantitativa, i grafici dei vulcani mostrano un'anteprima istantanea di candidati potenzialmente interessanti con implicazioni biologiche. La Figura 4A mostra il rapporto tra la quantificazione della proteina OVE26 e il ceppo FVB di controllo nei proteomi dell'intero rene. In particolare, GAPDH è fortemente sovraregolato nel rene OVE26 insieme a ALDH6A1, suggerendo che le risposte metaboliche sono montate nei reni diabetici precoci. Nella Figura 4B sono forniti singoli diagrammi a violino per illustrare la variabilità e gli effetti della normalizzazione dei dati utilizzando MetaboAnalyst 6.0. Si noti la robusta sottoregolazione di una metalloendopeptidasi della matrice (MME) e della catepsina A (CTSA) nell'intero rene di topo OVE26. Per illustrare le differenze complessive del proteoma nell'intero rene, il set di dati del proteoma filtrato può essere sottoposto a riduzione dimensionale e analisi multivariate supervisionate (PLSDA, sPLSDA, oPLSDA) per mostrare le differenze a livello di gruppo e per trovare variabili dell'importanza della proiezione (VIP). Per questo, abbiamo eseguito PLSDA sul proteoma renale OVE26 e FVB, come mostrato nella Figura 4C, e i VIP sono mostrati nella Figura 4D. Le analisi PLSDA mostrano una robusta separazione del proteoma OVE26 (verde) rispetto al proteoma renale FVB di controllo (rosso). I punteggi VIP > 1 sono in genere buone variabili candidate, in questo caso le proteine, per ulteriori analisi nell'arricchimento dell'ontologia del percorso utilizzando Gprofiler (https://biit.cs.ut.ee/gprofiler/gost) mostrato nei grafici nelle figure supplementari (Figura supplementare 1). In particolare, molti dei VIP proteici si trovano nelle analisi del grafico del vulcano nella Figura 4A. L'identificazione delle liste candidate per ulteriori analisi dovrebbe includere proteine con grande variazione di fold, proteine statisticamente robuste e importanti coinvolte nella stratificazione dei gruppi (VIP>1). Un'ulteriore illustrazione delle differenze nei proteomi a livello di gruppo e a livello di singola proteina può essere visualizzata in mappe di calore gerarchiche con clustering di gruppi e caratteristiche, come mostrato nella Figura 4E.
A scopo comparativo, abbiamo eseguito un'analisi di proteomica quantitativa LC-MS/MS 1-dimensionale su tubuli corticali isolati (Figura 5) e midollo (Figura 6) da topi OVE26 e FVB per illustrare la capacità di perforare il proteoma di aree specifiche del rene con un'efficace profondità di rilevamento in assenza di componenti di abbondanza più elevati. Infine, la sovrapposizione nel proteoma può essere illustrata da un confronto delle identificazioni significative totali e dalla sovrapposizione delle proteine statisticamente più significative in ciascuna condizione (rene intero, tubuli e midollo) come illustrato in un diagramma di Venn (Figura 7). In particolare, utilizziamo un approccio combinato di t-test e VIP (variabile di importanza della proiezione) da approcci statistici di riduzione dimensionale PLSDA (Figura 4C, D, Figura 5C, D e Figura 6C, D) per determinare potenziali proteine candidate al follow-up. Si noti che i criteri t-stat (T-test) (Figura 7A) erano più suscettibili di identificare un numero maggiore di proteine al di sopra della soglia nell'intero rene e, in misura minore, nel midollo e nei tubuli. L'utilizzo di un'altra soglia statistica (VIP > 1) nella Figura 7B per espandere l'elenco dei candidati ha fornito nuove proteine candidate distribuite in modo più uniforme nelle analisi proteomiche del midollo e del tubulo corticale. Notevole è la mancanza di sovrapposizione in ciascuno dei 3 proteomi, poiché 0 o 2 candidati sono stati condivisi utilizzando entrambi gli approcci. È possibile applicare diversi approcci per consentire la perforazione più profonda nel proteoma renale. L'isolamento e il frazionamento del proteoma a livello del tessuto e del compartimento tissutale è un approccio iniziale che può produrre risultati robusti, come indicato dal confronto del diagramma di Venn (Figura 7) negli approcci t-test e VIP sorting. Tutti i file grezzi e le liste dei picchi saranno depositati in MASSIVE (massive.ucsd.edu) secondo gli standard HUPO e condivisi pubblicamente come invio parziale al momento della pubblicazione.
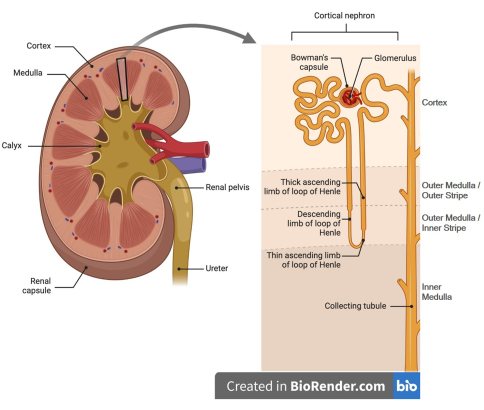
Figura 1: Rendering a fumetti del nefrone renale. (Pannello di sinistra) I reni hanno regioni corteccia (esterna) e midollo (interno). (Pannello di destra) La corteccia renale contiene glomeruli e la rispettiva capsula di Bowman, tubuli contorti prossimali e la maggior parte del suo segmento rettilineo, porzioni di spesse anse ascendenti di Henle, tubuli contorti distali, segmenti corticali dei dotti collettori e capillari peritubulari (non mostrati). Il midollo è costituito da anse discendenti e ascendenti di Henle, dalla porzione midollare della spessa ansa ascendente di Henle e dai dotti collettori, e dai capillari dei vasa recta che circondano questi segmenti tubulari (non mostrati). La figura è stata generata utilizzando Biorender. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Colorazione PAS (Schiff acido periodico) rappresentativa per dimostrare l'istologia in topi FVB (non diabetici) e OVE26 (diabetici) di 2 mesi. I glomeruli (freccia verde) dei topi diabetici mostrano un aumento della colorazione rosa della PAS, dimostrando l'espansione mesangiale, mentre le alterazioni istologiche evidenti del tubulointerstizio non sono ancora evidenti nei giovani topi diabetici. I tubuli prossimali mostrano un lieve/modesto ingrossamento/ipertrofia (freccia gialla). Barre di scala: 30 μm. Ingrandimento originale 100x. Clicca qui per visualizzare una versione più grande di questa figura.
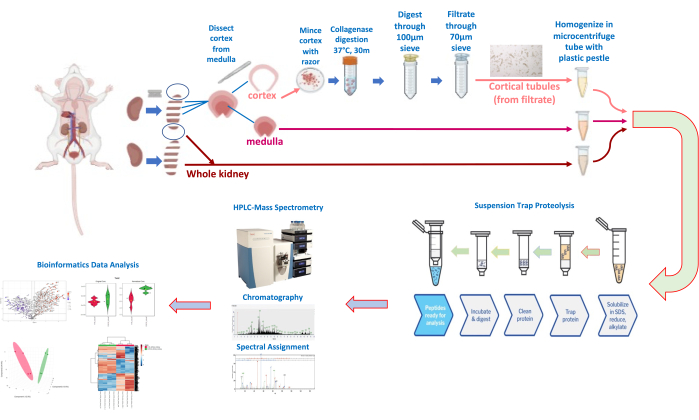
Figura 3: Dissezione renale e isolamento dei tubuli e flusso di lavoro della proteomica quantitativa. Fare clic qui per visualizzare una versione più grande di questa figura.
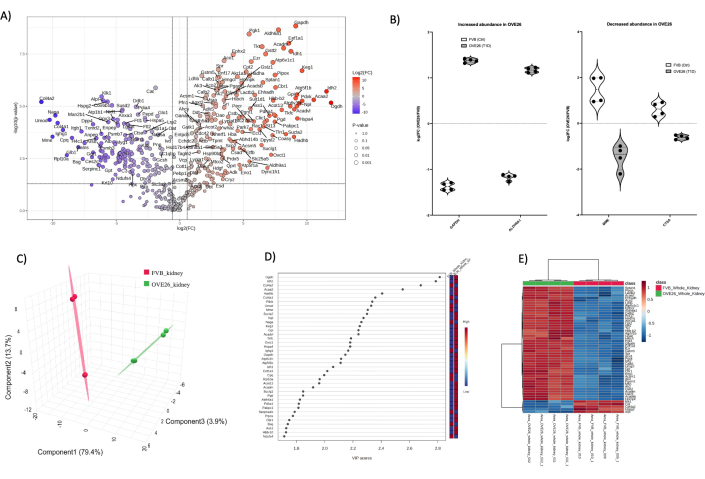
Figura 4: Cambiamenti globali nel proteoma del rene intero di topo diabetico OVE26. (A) Mostra il log2FC (variazione di piega di OVE26/FVB) tracciato rispetto al valore p non aggiustato (-log10 p-value) in un grafico di vulcano con variazione negativa (blu) o positiva (rosso) di 1,5 volte e p < 0,05 cutoff indicati dalle linee verticali e orizzontali, tutte le quantificazioni significative delle proteine sono etichettate nel punto dati. (B) Diagrammi a violino che mostrano la variabilità di ciascuna quantificazione replicata per proteine selezionate GAPDH e ALDH6A1 in aumento in OVE26 e MME e CTSA in diminuzione in OVE26 rene intero. (C) L'approccio della riduzione dimensionale che utilizza l'analisi discriminante dei minimi quadrati parziali mostra la separazione dei gruppi di OVE26 (verde) e FVB (rosso). (D) Le variabili di importanza del grafico di proiezione mostrano il contributo delle singole proteine nelle analisi PLSDA per punteggio VIP. (E) Mappa di calore di clustering gerarchico delle proteine modulate in alto che indica le differenze di gruppo nell'intensità della quantificazione delle proteine. Clicca qui per visualizzare una versione più grande di questa figura.
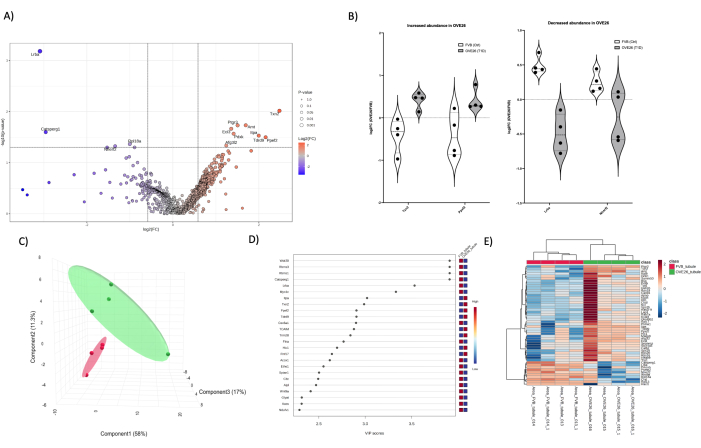
Figura 5: Cambiamenti nel proteoma del tubulo corticale diabetico OVE26. (A) Mostra il log2FC (variazione di piega di OVE26/FVB) tracciato rispetto al valore p non aggiustato (-log10 p-value) in un grafico vulcano con variazione negativa (blu) o positiva (rosso) di 1,5 volte e cutoff p < 0,05 indicati dalle linee verticali e orizzontali, tutte le quantificazioni significative delle proteine sono etichettate nel punto dati. (B) Diagrammi di violino che mostrano la variabilità di ciascuna quantificazione della replicazione per proteine selezionate TXN2 e PPEF2 in aumento in OVE26 e LRBA e NHERF2 in diminuzione nel proteoma tubulare OVE26. (C) L'approccio della riduzione dimensionale che utilizza l'analisi discriminante dei minimi quadrati parziali mostra la separazione dei gruppi di OVE26 (verde) e FVB (rosso). (D) Variabili di importanza del grafico di proiezione mostra il contributo delle singole proteine nelle analisi PLSDA per punteggio VIP. (E) Mappa di calore di clustering gerarchico delle proteine modulate in alto che indica le differenze di gruppo nell'intensità della quantificazione delle proteine. Clicca qui per visualizzare una versione più grande di questa figura.
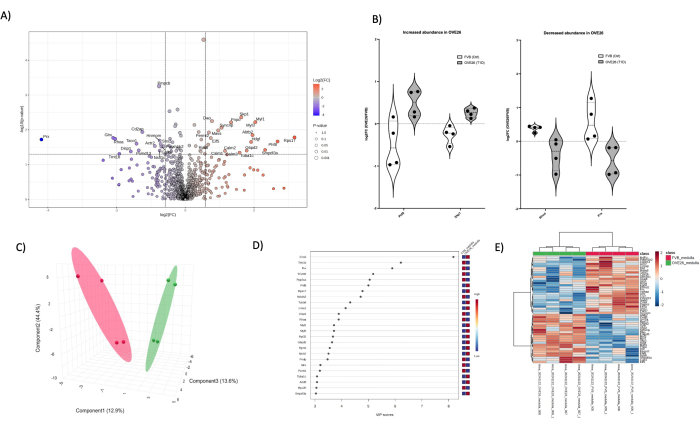
Figura 6: Cambiamenti nel proteoma del midollo diabetico OVE26. (A) Mostra il log2FC (variazione di piega di OVE26/FVB) tracciato rispetto al valore p non aggiustato (-log10 valore p) in un grafico vulcano con variazione negativa (blu) o positiva (rosso) di 1,5 volte e i cutoff p < 0,05 indicati dalle linee verticali e orizzontali, tutte le quantificazioni significative delle proteine sono etichettate nel punto dati. (B) Diagrammi a violino che mostrano la variabilità di ciascuna quantificazione replicata per proteine selezionate PHF8 e SKP1 che aumentano in OVE26 e RHOA e PRX che diminuiscono nel proteoma midollare OVE26. (C) L'approccio della riduzione dimensionale che utilizza l'analisi discriminante dei minimi quadrati parziali mostra la separazione dei gruppi di OVE26 (verde) e FVB (rosso). (D) Variabili di importanza del grafico di proiezione mostra il contributo delle singole proteine nelle analisi PLSDA per punteggio VIP. (E) Mappa di calore di clustering gerarchico delle proteine modulate in alto che indica le differenze di gruppo nell'intensità della quantificazione delle proteine. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 7: Diagramma di Venn delle proteine differenzialmente abbondanti nei proteomi interi del rene, dei tubuli e del midollo osseo. I proteomi renali sono stati confrontati seguendo un test t (p-val < 0,05) e una soglia di variazione della piega di +/- 1,5FC e liste di VIP > 1,0. Clicca qui per visualizzare una versione più grande di questa figura.
Figura 1 supplementare: Analisi delle vie del proteoma dell'intero rene utilizzando Gprofiler generato inviando elenchi di proteine VIP (VIP > 1) nel pannello 4D. Gprofiler è stato implementato per analizzare le differenze di percorso per le proteine con punteggi VIP superiori a 1 dall'analisi PLSDA nella Figura 4. (A) Mostra l'arricchimento grafico dei percorsi con cutoff del valore p per funzioni molecolari (MF), componente cellulare (CC) e processo biologico (BP). (B) Tabella di arricchimento della via dell'ontologia genica per ogni specifico processo, funzione o compartimento cellulare. (C) Grafico specifico dell'arricchimento proteico per le funzioni molecolari del proteoma dell'intero rene. Clicca qui per scaricare questo file.
Discussione
I metodi presentati in questo approccio tecnico sono progettati per illustrare l'analisi comparativa del proteoma di diverse aree del rene. Qui, abbiamo utilizzato metodi per isolare il midollo e i tubuli corticali nei topi diabetici (OVE26) e di controllo (FVB) e abbiamo eseguito analisi LC-MS/MS 1-dimensionali e bioinformatiche per illustrare le differenze di base nel proteoma in ciascuna parte del rene, oltre al proteoma di interi reni.
L'isolamento dei compartimenti renali richiede un'attenta dissezione della corteccia lontano dal midollo. Con l'affettatura trasversale iniziale dei reni, fette sottili consentiranno una dissezione più affidabile della corteccia. La preparazione dei tubuli corticali utilizzando un approccio di setacciatura può portare alla filtrazione dei glomeruli attraverso setacci da 70 μm. È fondamentale controllare la purezza dei tubuli al microscopio per garantire l'arricchimento ma evitare la perdita di massa dei tubuli ad ogni successiva fase di lavaggio. Evitare un'eccessiva pressione della corteccia digerita attraverso le fasi iniziali di setacciatura può ridurre la contaminazione nella preparazione finale dei tubuli.
Gli approcci di intrappolamento in sospensione offrono un ampio grado di flessibilità nelle fasi di solubilizzazione ed estrazione delle proteine senza interferenze ioniche nelle fasi di eluizione della spettrometria di massa. Di fondamentale importanza nella fase 2.10 è il rapporto tra tripsina (1:20 o 1:10 sembrano ottimali per la maggior parte delle applicazioni) e concentrazione di peptidi. Un'efficiente proteolisi delle proteine, ma la minimizzazione delle scissioni non specifiche, produrrà risultati più coerenti. Le fasi di acidificazione successive all'alchilazione nella fase 2.4.1 possono utilizzare acido formico o trifluoroacetico se verrà eseguito l'arricchimento di fosfopeptidi. L'acido fosforico può competere con il legame del fosfopeptide alle resine per cromatografia di affinità metallica.
L'analisi dei percorsi può essere utilizzata per indicare le differenze di percorso e i cambiamenti correlati al danno diabetico nel rene OVE26, come mostrato in un'analisi rappresentativa (Figura supplementare 1). Notevoli differenze nell'analisi dei percorsi indicate dall'annotazione ontologica dimostrano l'eterogeneità del metabolismo nei nefroni renali. L'intero proteoma renale è stato marcatamente arricchito per le proteine coinvolte nei processi metabolici e catabolici degli aminoacidi, nell'ossidazione degli acidi grassi e nei temi generali di gestione dell'energia (Figura 1 supplementare). Il proteoma del tubulo corticale aveva un aumento delle proteine strutturali e dei trasportatori ionici, come previsto in questo compartimento, come mostrato nella Figura 5. Il midollo è arricchito di proteine del metabolismo dei carboidrati (Figura 6) poiché questo sito è arricchito di enzimi glicolitici, e il glucosio è una fonte di carburante nel midollo esterno, rispetto alla corteccia renale, che è il sito della gluconeogenesi e il glucosio è un carburante scarso per la respirazione19,20. Inoltre, l'arricchimento delle vie di segnalazione delle chinasi nel midollo è probabilmente il riflesso dell'osmolalità interstiziale e delle fluttuazioni delle dimensioni cellulari in questa regione21 e dei disturbi metabolici e dello stress ossidativo associati a questa regione durante il diabete22. L'utilizzo dei grafici del vulcano, dei grafici VIP e dei risultati della mappa di calore combinati con un approccio di arricchimento dei percorsi come Gprofiler può aiutare i ricercatori a comprendere meglio gli effetti di lesioni o malattie sui cambiamenti nei diversi compartimenti renali. Ulteriori opzioni software di analisi includono DAVID (https://davidbioinformatics.nih.gov/), Express Analyst (https://www.expressanalyst.ca/ExpressAnalyst/) e PANTHERDB (https://www.pantherdb.org/) per la caratterizzazione di ontologie e percorsi.
In conclusione, il confronto proteomico del proteoma del rene intero totale (Figura 4) e quello dei compartimenti del tubulo corticale (Figura 5) e del midollo (Figura 6) è un approccio utile per illustrare l'eterogeneità negli estratti di rene intero rispetto alle sezioni isolate del rene. Inoltre, i proteomi isolati consentono di perforare più in profondità il proteoma di strutture specifiche in assenza di proteine ad alta abbondanza (tessuto intero), smascherando così le proteine a media abbondanza (compartimenti tissutali) e infine a bassa abbondanza (singola cellula). In definitiva, l'isolamento ideale sarebbe a livello di singola cellula per consentire di misurare la dinamica cellulare e collegarla a una specifica condizione patologica come il diabete o le malattie renali croniche.
Divulgazioni
Gli autori non hanno divulgazioni.
Riconoscimenti
Il lavoro per questo progetto è stato parzialmente sostenuto con fondi per MTB (NIH K01DK080951) e TDC (NephCure-Pediatric Nephrology Research Consortium NKI-2023-04) e il programma per le malattie renali dell'Università di Louisville e il Proteomics Technology Center (TDC, MTB).
Materiali
| Name | Company | Catalog Number | Comments |
| Collagenase type 1A | Millipore Signal | C9891 | |
| Exploris 480 Orbitrap | Thermofisher | https://www.thermofisher.com/order/catalog/product/BRE725539 | MS |
| Falcon Cell Strainer, 100 µm | VWR | 21008-950 | |
| Falcon Cell Strainer, 70 µm | VWR | 21008-952 | |
| Gibco PBS pH 7.4 | Thermo | 1001023 | |
| Halt Protease and Phosphatase Inhibitor Cocktail | VWR | PI78440 | |
| Iodoacetamide | Sigma Aldrich | I1149 | |
| MetaboAnalyst 6.0 | MetaboAnalyst 6.0 | https://www.metaboanalyst.ca/ | metabolomics data analysis platform |
| NanoDrop 2000 | Thermofisher | https://www.thermofisher.com/order/catalog/product/ND-2000 | |
| Oasis HLB column | Waters | 186002034 | |
| PEAKS 12.0 | Bioinformatics Solutions Inc | LC-MS/MS data analysis software | |
| Pestle for 1.5 mL Microtube | Fisher Scientific | NC0782485 | |
| Suspension Trap (S-trap) | Protifi | C02-micro-10 | |
| TCEP | ThermoFisher Scientific | 20490 | |
| TEABC | Sigma Aldrich | T7408 | |
| Trypsin Protease, MS-Grade | ThermoFisher Scientific | 90057 | |
| Ultrasonic Cleaner | Cole-Parmer | Model 0884900 |
Riferimenti
- Ding, X., et al. Epidemiological patterns of chronic kidney disease attributed to type 2 diabetes from 1990-2019. Front Endocrinol. 15, 1383777 (2024).
- Liu, W., et al. Global trends in the burden of chronic kidney disease attributable to type 2 diabetes: an age-period-cohort analysis. Diabetes Obes Metab. 26 (2), 602-610 (2024).
- Xie, D., et al. Global burden and influencing factors of chronic kidney disease due to type 2 diabetes in adults aged 20-59 years, 1990-2019. Sci Rep. 13 (1), 20234 (2023).
- Giunti, S., Barit, D., Cooper, M. E. Mechanisms of diabetic nephropathy: role of hypertension. Hypertension. 48 (4), 519-526 (2006).
- Thomas, M. C., Burns, W. C., Cooper, M. E. Tubular changes in early diabetic nephropathy. Adv Chronic Kidney Dis. 12 (2), 177-186 (2005).
- Thomas, M. C., et al. Diabetic kidney disease. Nat Rev Dis Primers. 1, 15018 (2015).
- Nath, K. A. Tubulointerstitial changes as a major determinant in the progression of renal damage. Am J Kidney Dis. 20 (1), 1-17 (1992).
- Bonventre, J. V. Can we target tubular damage to prevent renal function decline in diabetes. Semin Nephrol. 32 (5), 452-462 (2012).
- Powell, D. W., et al. Renal tubulointerstitial fibrosis in OVE26 type 1 diabetic mice. Nephron Exp Nephrol. 111 (1), e11-e19 (2008).
- Cummins, T. D., et al. Quantitative mass spectrometry of diabetic kidney tubules identifies GRAP as a novel regulator of TGF-beta signaling. Biochim Biophys Acta. (4), 653-661 (2010).
- Barati, M. T., et al. Differential expression of endoplasmic reticulumstress-response proteins in different renal tubule subtypes of OVE26 diabetic mice. Cell Stress Chaperones. 21 (1), 155-166 (2016).
- Zheleznova, N. N., et al. Mitochondrial proteomic analysis reveals deficiencies in oxygen utilization in medullary thick ascending limb of Henle in the Dahl salt-sensitive rat. Physiol Genomics. 44 (17), 829-842 (2012).
- Nordquist, L., Palm, F. Diabetes-induced alterations in renal medullary microcirculation and metabolism. Curr Diabetes Rev. 3 (1), 53-65 (2007).
- dos Santos, E. A., Li, L. -. P., Ji, L., Prasad, P. V. Early changes with diabetes in renal medullary hemodynamics as evaluated by fiberoptic probes and BOLD magnetic resonance imaging. Invest Radiol. 42 (3), 157-162 (2007).
- Cummins, T. D., et al. Quantitative Mass Spectrometry Normalization in UrineBiomarker Analysis in Nephrotic Syndrome. Glomerular Dis. (3), 121-131 (2022).
- Carlson, E. C., Audette, J. L., Klevay, L. M., Nguyen, H., Epstein, P. N. Ultrastructural and functional analyses of nephropathy in calmodulin-induced diabetic transgenic mice. Anat Rec. 247 (1), 9-19 (1997).
- Zheng, S., et al. Development of late-stage diabetic nephropathy in OVE26 diabetic mice. Diabetes. 53 (12), 3248-3257 (2004).
- Wang, F., Veth, T., Kuipers, M., Altelaar, M., Stecker, K. E. Optimized suspension trapping method for phosphoproteomics sample preparation. Anal Chem. 95 (25), 9471-9479 (2023).
- Mather, A., Pollock, C. Glucose handling by the kidney. Kidney Int Suppl. 120, S1-S6 (2011).
- Ross, B. D., Espinal, J., Silva, P. Glucose metabolism in renal tubular function. Kidney Int. 29 (1), 54-67 (1986).
- Roger, F., Martin, P. Y., Rousselot, M., Favre, H., Féraille, E. Cell shrinkage triggers the activation of mitogen-activated protein kinases by hypertonicity in the rat kidney medullary thick ascending limb of the Henle's loop: requirement of p38 kinase for the regulatory volume increase response. J Biol Chem. 274 (48), 34103-34110 (1999).
- Yang, J., Lane, P. H., Pollock, J. S., Carmines, P. K. PKC-dependent superoxide production by the renal medullary thick ascending limb from diabetic rats. Am J Physiol Renal Physiol. 297 (5), F1220-F1228 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon