Method Article
Studio della morte cellulare programmata indotta da nanoparticelle di rame nei batteri
In questo articolo
Riepilogo
Le nanoparticelle di rame agiscono come agenti antimicrobici generando specie reattive dell'ossigeno. Qui vengono presentate le procedure che dimostrano che le nanoparticelle di rame sono efficaci contro tre agenti patogeni clinicamente rilevanti e che alcune vie di morte cellulare programmata sono coinvolte in questo processo battericida.
Abstract
Recentemente, le preoccupazioni per i patogeni multiresistenti ai farmaci e le infezioni incurabili sono aumentate a causa dell'uso eccessivo e improprio di antibiotici. I nanomateriali, come le nanoparticelle metalliche e di ossidi metallici, hanno guadagnato popolarità nel campo biomedico come potenziali nuove strategie per combattere i patogeni multiresistenti. Questo studio ha studiato l'uso di nanoparticelle di rame (CuNP) come battericida contro tre comuni agenti patogeni opportunistici acquisiti in ospedale - Escherichia coli (E. coli), Acinetobacter baumannii (A. baumannii) e Staphylococcus aureus (S. aureus) - che stanno sviluppando sempre più resistenza ai farmaci. Vengono presentati protocolli dettagliati per la sintesi di CuNP di due dimensioni (20 nm e 60 nm) e per la valutazione della loro efficacia battericida attraverso saggi di colonie. I meccanismi d'azione antimicrobica alla base delle CuNP sono stati esplorati valutando i cambiamenti nella produzione di specie reattive dell'ossigeno. Inoltre, sono stati applicati quattro modulatori che inibiscono le funzioni delle proteine umane per studiare il potenziale coinvolgimento delle vie di morte cellulare programmata (PCD) nell'uccisione batterica. Attraverso questo approccio, viene suggerita la potenziale comparsa di ceppi resistenti al rame, basandosi sulla ricerca sulle proteine dell'omeostasi del rame, compresi i regolatori trascrizionali rame-dipendenti. Questi risultati forniscono una metodologia completa per studiare gli effetti battericidi delle CuNP e il loro potenziale ruolo nell'affrontare la resistenza agli antibiotici.
Introduzione
I batteri resistenti ai farmaci sono una seria causa di preoccupazione in medicina. La loro rapida comparsa ha ridotto l'efficacia degli antibiotici convenzionali, con conseguenti complicanze cliniche maggiori. Rappresentano una grave minaccia per la salute pubblica e creano un urgente bisogno di nuovi agenti antimicrobici. Una via di ricerca sono i nanomateriali. I nanomateriali possiedono proprietà fisico-chimiche uniche che consentono loro di interagire con i microbi in modi che ne compromettono la vitalità. Ad esempio, le nanoparticelle d'argento (AgNP) inducono stress ossidativo nei batteri, con conseguente disfunzione proteica, rottura della membrana, danni al DNA e, infine, morte cellulare1. Le nanoparticelle d'oro (AuNP), d'altra parte, sono note per le loro proprietà antimicotiche e possono migliorare l'effetto battericida degli antibiotici fungendo da vettori2.
Inoltre, anche le nanoparticelle di rame (CuNP) hanno attirato una notevole attenzione grazie al loro potente effetto antimicrobico e al basso costo di produzione. Gli studi suggeriscono che le CuNP mostrano un'attività battericida ad ampio spettro attraverso l'interruzione dell'attività enzimatica e la generazione di specie reattive dell'ossigeno (ROS)3. La carica positiva delle CuNP ne facilita la penetrazione nei batteri, migliorandone l'assorbimento cellulare4. Questo meccanismo rende le CuNP un'opzione promettente per il rivestimento superficiale, ad esempio sugli impianti, per prevenire le infezioni3. Una scoperta interessante, tuttavia, è che l'effetto battericida delle CuNP sembra essere dipendente dalle dimensioni. Alcuni studi hanno scoperto che le CuNP più piccole mostrano una maggiore attività antibatterica, probabilmente a causa del loro superiore rapporto superficie/volume5.
La generazione di ROS provoca danni diffusi a cellule e batteri, tra cui perossidazione lipidica, disfunzione proteica, frammentazione del DNA e inibizione della gluconeogenesi/glicogenolisi, ed è coinvolta nella necrosi o morte cellulare programmata (PCD)6,7,8. Studi recenti hanno rivelato che i sistemi PCD esistono nei batteri, con modalità d'azione ed effettori simili a quelli dei sistemi eucariotici9. Le comunità batteriche possono indurre la PCD in risposta allo stress, incluso lo stress ossidativo, attraverso un sistema tossina-antitossina (TA)10. In termini semplici, il sistema tossina-antitossina è costituito da tossine che possono interrompere i processi cellulari essenziali e antitossine che possono formare complessi stabili con le tossine per inibire la loro tossicità in condizioni di crescita normali. La maggior parte dei batteri e degli archei contiene loci TA nei loro genomi, spesso presenti in più copie di DNA extracromosomico e cromosomico. Esistono diversi tipi di sistemi TA, con il tipo II TA (noto come modulo MazE/MazF) di particolare interesse. In condizioni di stress, le antitossine vengono degradate, consentendo alle tossine di inibire i loro bersagli cellulari. In E. coli e S. aureus, la tossina MazF viene attivata in risposta a condizioni di stress come lo stress ossidativo, l'alta temperatura e la carenza di aminoacidi. Di conseguenza, l'espressione dell'antitossina MazE si riduce, rilasciando la tossina MazF10. Gli studi hanno scoperto che MazF consente la sintesi di proteine che consentono a una piccola sottopopolazione di sopravvivere in condizioni avverse, mentre la maggior parte della popolazione subisce la morte cellulare mediata da mazEF. Questa morte cellulare può essere ROS-dipendente, dove i ROS inducono l'inibizione trascrizionale o traduzionale, o ROS-indipendente, dove il danno al DNA innesca le vie di morte11.
Questo studio esplora i meccanismi attraverso i quali le CuNP inducono la morte batterica. Piuttosto che concentrarsi esclusivamente sul sistema TA, quattro modulatori PCD, precedentemente utilizzati nella nostra ricerca 7,12, sono stati impiegati per studiare potenziali percorsi PCD nei batteri.
Esaminando gli effetti battericidi di CuNP di due diverse dimensioni (20 e 60 nm) a concentrazioni variabili e utilizzando metodi come saggi di colonie, rilevamento di ROS e modulatori della PCD (SBI, Z-VAD, NSA e Wortmannin), questa ricerca evidenzia che la PCD non è esclusiva degli organismi multicellulari, ma si verifica anche nelle comunità batteriche sotto stress. Fornendo protocolli dettagliati, questo lavoro mira a consentire ai ricercatori di valutare l'efficacia delle CuNP e i meccanismi battericidi nei propri sistemi. Inoltre, questi risultati fanno progredire la comprensione della PCD batterica e supportano lo sviluppo di terapie a base di CuNP per combattere i batteri resistenti agli antibiotici.
Protocollo
I reagenti e le attrezzature utilizzate in questo studio sono elencati nella Tabella dei materiali.
1. Preparazione della nanoparticella di rame
- Ottenere nanopolveri di rame commerciali (25 nm e 60-80 nm) da una fonte commerciale.
- Utilizzare 1,0 mM di sodio dodecil solfato (SDS) come disperdente per due dimensioni di nanoparticelle da 1 mg/mL.
- Disperdere le nanoparticelle utilizzando un bagno ad ultrasuoni per almeno 30 minuti a temperatura ambiente. Le nanoparticelle completamente disperse sono quindi pronte per l'uso negli esperimenti successivi.
2. Preparazione dei batteri
- Procuratevi E. coli (Migula) Castellani e Chalmers ceppo 25922 e A. baumannii ceppo Bouvet e Grimont dall'American Type Culture Collection. Ottieni S. aureus dal Centro di raccolta e ricerca delle biorisorse.
- Coltura dei batteri in brodo di Luria-Bertani (LB) in condizioni aerobiche a 37 °C.
- Diluire le colture batteriche in terreno LB a una densità ottica a 600 nm (OD600) di circa 0,5.
3. Valutazione della vitalità cellulare
- Saggio delle colonie
- Utilizzare soluzioni stock di CuNP (1 mg/mL) per preparare varie concentrazioni di due dimensioni di CuNP, tra cui 0 μg/mL, 1 μg/mL, 5 μg/mL, 10 μg/mL, 50 μg/mL e 100 μg/mL.
- Dividere le colture batteriche preparate al punto 2.3 in provette da microcentrifuga e centrifugare a 3300 × g per 10 minuti a temperatura ambiente.
- Trattenere i pellet batterici e aggiungere diverse concentrazioni di due dimensioni di CuNP, rispettivamente, con un pipettaggio delicato.
- Trattare i pellet batterici con PBS e alcol al 70% rispettivamente come controlli negativi e positivi.
- Incubare tutti i batteri trattati agitando a 200 giri/min a 37 °C per 24 ore.
- Dopo l'incubazione, lavare tutti i batteri trattati con PBS e distribuirli su piastre di agar LB. Mettere le piastre in un'incubatrice a 37 °C per 24 ore.
- Conta il numero di colonie in ciascun gruppo di trattamento il giorno successivo ed esegui l'analisi statistica. Si consiglia di eseguire questa operazione in triplice copia per garantire l'accuratezza statistica.
- Studio del meccanismo battericida
- Preparare i batteri come descritto al punto 3.1.2 e trattarli con 5 μM di SBI-0206965 (SBI) per 2 ore, 0,5 μM di necrosulfonamide (NSA) per 1 ora, 100 nM di wortmannina (mosto) per 30 minuti o 100 nM di Z-VAD-FMK (Z-VAD) per 30 minuti.
- Co-trattare i batteri con diverse concentrazioni di due dimensioni di soluzioni di CuNP, come descritto nel passaggio 3.1.1, in presenza o assenza di 5 μM di SBI, 0,5 μM di NSA, 100 nM di mosto e 100 nM di Z-VAD.
- Centrifugare i batteri dopo i trattamenti modulatori (passaggio 3.2.1) e rimuovere i surnatanti.
- Risospendere i pellet batterici nelle soluzioni preparate al punto 3.2.2 e incubarli agitandoli a 200 giri/min a 37 °C per 24 ore.
- Trattare i batteri con etanolo al 70% e PBS come controlli positivi e negativi, rispettivamente. Utilizzare una soluzione senza CuNP come controllo in bianco CuNP (0 μg/mL; mock) nelle stesse condizioni di inibitore per ciascun gruppo. Incubare tutti i campioni per altre 24 ore.
- Dopo l'incubazione, aggiungere il reagente di vitalità cellulare alle colture con un rapporto di volume di 1:10. Incubare le colture per altre 2 ore agitando a 37 °C.
- Centrifugare le colture (passaggio 3.1.2) dopo 2 ore di incubazione. Trasferire i surnatanti fluorescenti in piastre a 96 pozzetti. Misura la fluorescenza utilizzando una lunghezza d'onda di eccitazione di 560 nm e una lunghezza d'onda di emissione di 590 nm con un lettore di micropiastre.
- Diluire il surnatante rimanente a 10-5 e 10-4 e distribuirlo su piastre di agar LB per la coltivazione.
- Conta le singole colonie il giorno seguente.
4. Rilevamento di specie reattive dell'ossigeno
- Preparare le colture batteriche come descritto al punto 2.3 e dividerle in provette da microcentrifuga.
- Trattare i batteri con varie condizioni di stress come gruppi di controllo positivi che inducono ROS (dati non mostrati nei risultati). I trattamenti sono descritti nelle fasi 4.2.1-4.2.4.
- Esporre i batteri alla luce UV a 405 nm per 3 ore. Incubare i batteri a 45 °C per 2 ore.
- Successivamente, incubare i batteri a 4 °C per 2 ore.
- Trattare i batteri con il 3% di H2O2 per 30 min.
- Mantenere i batteri a 37 °C in brodo LB come controllo negativo.
- Preparare varie concentrazioni di CuNP come descritto al punto 3.1.1 e trattare i batteri con CuNP da 20 nm o 60 nm a concentrazioni di 1 μg/mL, 5 μg/mL, 10 μg/mL e 100 μg/mL per 24 ore.
- Lavare i batteri incubati due volte con PBS per rimuovere eventuali nanoparticelle rimanenti.
- Preparare il colorante 2′,7′-diclorodiidrofluoresceina diacetato (H2DCFDA) in PBS a una concentrazione finale di 5 μM.
- Risospendere i pellet batterici in 5 μM di H2DCFDA e misurare l'intensità della fluorescenza a 520/30 nm di emissione utilizzando un citometro a flusso.
NOTA: L'intensità della fluorescenza verde FL1 è correlata al livello di ROS nella coltura trattata. Si consiglia di eseguire questa operazione in triplice copia per l'accuratezza statistica.
Risultati
Attività antimicrobiche di CuNP di due dimensioni in tre patogeni
Tre patogeni opportunisti (E. coli, S. aureus e A. baumannii) sono stati utilizzati per testare le attività battericide delle CuNP. I batteri sono stati trattati con 0 μg/mL, 1 μg/mL, 5 μg/mL, 10 μg/mL, 50 μg/mL e 100 μg/mL di CuNP da 20 nm o 60 nm e le attività battericide sono state determinate utilizzando la concentrazione battericida minima (MBC) derivata dalla conta delle colonie. I nostri risultati hanno mostrato effetti battericidi positivi di entrambe le dimensioni di CuNP (Figura 1) per tutti e tre i ceppi. In E. coli, sono state osservate diminuzioni significative del numero di colonie a partire da 1 μg/mL e 5 μg/mL per i trattamenti CuNP a 20 nm e 60 nm, rispettivamente (Figura 1A). Al contrario, sono state mostrate notevoli riduzioni della conta delle colonie di S. aureus in tutte le concentrazioni di CuNP di entrambe le dimensioni (Figura 1B). Per A. baumannii, sono state necessarie concentrazioni più elevate di CuNP (5 μg/mL in CuNP a 20 nm e 10 μg/mL in CuNP a 60 nm) per ottenere una riduzione della conta delle colonie (Figura 1C).
I nostri studi precedenti hanno indicato che le CuNP più piccole (20 nm) mostrano una capacità battericida più straordinaria rispetto a quelle più grandi (60 nm)7,12. In questo caso, sono state mostrate tendenze simili, indicando risultati coerenti. Inoltre, questi risultati suggeriscono che i batteri gram-positivi (in questo caso, S. aureus) sono più suscettibili alle CuNP rispetto ai batteri gram-negativi testati (E. coli e A. baumannii).
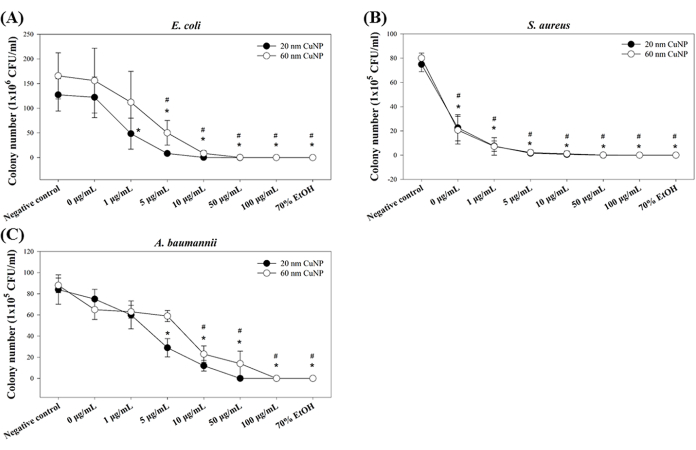
Figura 1: Attività battericida di CuNP di due diverse dimensioni in tre specie batteriche. E. coli (A), S. aureus (B) e A. baumannii (C) sono stati trattati con concentrazioni variabili di CuNP da 20 nm o 60 nm e sono state determinate le conte delle colonie batteriche. I batteri trattati con PBS e alcol al 70% sono serviti rispettivamente come controlli negativi e positivi. I dati sono espressi come media ± deviazione standard (SD) di quattro esperimenti indipendenti, ciascuno eseguito in triplice copia. I valori anomali sono stati identificati attraverso l'ispezione visiva. Il test di Shapiro-Wilk è stato utilizzato per valutare la normalità dei dati. I confronti statistici sono stati eseguiti utilizzando il t-test di Student. Gli asterischi (*) e i segni di cancelletto (#) indicano P < 0,05 per i trattamenti CuNP a 20 nm e 60 nm rispetto al controllo negativo, rispettivamente. Clicca qui per visualizzare una versione più grande di questa figura.
Danno letale ai batteri attraverso la generazione di ROS indotta da CuNP
I meccanismi proposti con cui le CuNP uccidono i batteri variano. Due teorie principali sono in discussione: la sostituzione delle proteine del cluster ferro-zolfo con il rame rilasciato dalle nanoparticelle e la produzione di ROS indotta da CuNP 13,14. La produzione di ROS è stata valutata in tre batteri trattati con CuNP per esplorare i meccanismi (Figura 2). E. coli, S. aureus e A. baumannii sono stati trattati con diverse concentrazioni di CuNP a 20 nm o 60 nm, seguiti dall'applicazione di H2DCFDA per la rilevazione dei ROS. Frazioni elevate di popolazioni fluorescenti verdi sono state misurate mediante citometria a flusso in tutte le concentrazioni di trattamenti CuNP a 20 nm e 60 nm (Figura 2). È interessante notare che le popolazioni di cellule positive nei trattamenti con CuNP a 20 nm erano indipendenti dalla concentrazione (Figura 2A). In particolare, concentrazioni più basse di CuNP a 20 nm hanno causato una maggiore generazione di ROS, mentre concentrazioni più elevate hanno mostrato una frazione inferiore di cellule positive. Tuttavia, questo fenomeno non è stato osservato nelle cellule trattate con CuNP a 60 nm (Figura 2B). Non sono state osservate differenze significative tra i tre batteri trattati con CuNP a 20 nm o 60 nm (Figura 2). Lievi aumenti delle popolazioni positive sono stati rilevati in 0 μg/mL, che possono essere attribuiti ai disperdenti e alla pressione dell'osmosi (Figura 2).
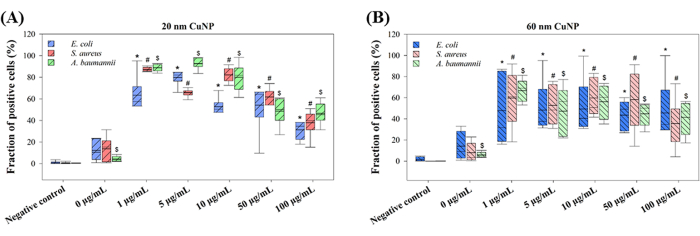
Figura 2: Generazione di ROS dopo il trattamento con CuNP di due diverse dimensioni in tre specie batteriche. E. coli, S. aureus e A. baumannii sono stati esposti a diverse concentrazioni di 20 nm (A) o 60 nm (B) CuNPs. La produzione di ROS è stata valutata utilizzando la colorazione H2DCFDA dopo il trattamento con CuNP e la frazione di cellule fluorescenti positive è stata analizzata mediante citometria a flusso. I dati sono presentati come media ± SD da cinque esperimenti indipendenti, ciascuno eseguito in triplice copia. I valori anomali sono stati identificati attraverso l'ispezione visiva. Il test di Shapiro-Wilk è stato utilizzato per valutare la normalità dei dati. I confronti statistici sono stati condotti utilizzando l'analisi unidirezionale di Kruskal-Wallis. I simboli (*, # e $) indicano P < 0,05 per i trattamenti CuNP a 20 nm e 60 nm rispetto al controllo negativo, rispettivamente. Clicca qui per visualizzare una versione più grande di questa figura.
Quattro modulatori della PCD hanno rivelato i meccanismi di morte batterica delle CuNP
Quattro modulatori della PCD sono stati utilizzati in esperimenti successivi per esplorare ulteriormente i meccanismi di morte cellulare indotti dai trattamenti con CuNP in tre patogeni. Questi modulatori -SBI, Z-VAD, NSA e Wort- erano noti per bloccare le vie di trasduzione del segnale associate all'apoptosi (Z-VAD), all'autofagia (SBI e Wort) e alla necroptosi (NSA) nelle cellule di mammifero15-17. Gli studi hanno suggerito che la PCD non è limitata agli organismi pluricellulari; diverse forme di PCD sono state osservate nelle colonie batteriche in risposta a condizioni difficili, a beneficio dell'intera colonia e facilitando strategie adattative come la trasformazione genetica e la formazione di biofilm10. Sulla base di questa comprensione, abbiamo esaminato se i percorsi correlati alla PCD contribuiscono alla morte cellulare indotta da CuNP nei batteri. I tre batteri, E. coli, S. aureus e A. baumannii, sono stati pretrattati con i quattro modulatori, seguiti da un co-trattamento con due dimensioni di CuNP e i modulatori (Figura 3).
Le vitalità cellulari hanno mostrato diminuzioni dipendenti dalla concentrazione in entrambe le dimensioni dei trattamenti con CuNP in tre batteri (Figura 3), in linea con i risultati presentati nella Figura 1. In E. coli, è stato osservato un aumento della sopravvivenza nel gruppo Z-VAD in trattamento con 1 μg/mL e 5 μg/mL di CuNP a 20 nm, così come nel gruppo NSA in trattamento con 10 μg/mL di CuNP a 20 nm (Figura 3A). Tuttavia, Z-VAD è stato efficace nel salvare la sopravvivenza in E. coli in tutte le concentrazioni di trattamenti con 60 nm-CuNP, ma non a 100 μg/mL (Figura 3B). Diversi risultati di vitalità in S. aureus suggeriscono particolari vie battericide nei trattamenti con CuNP a 20 nm (Figura 3C). L'NSA ha svolto un ruolo critico in tutte le concentrazioni di trattamenti con CuNP a 20 nm, mentre Z-VAD è risultato efficace solo a 1 μg/mL di CuNP a 20 nm. S. aureus non ha mostrato alcuna risposta ai modulatori a nessuna concentrazione, ad eccezione di Z-VAD a 10 μg/mL di trattamenti CuNP a 60 nm (Figura 3D). Tendenze simili nei tassi di sopravvivenza cellulare a quelle osservate in E. coli sono state osservate in A. baumannii trattato con CuNP a 20 nm (Figura 3E). Z-VAD è risultato efficace nel salvataggio a 1 μg/mL, 5 μg/mL e 10 μg/mL nei trattamenti CuNP a 20 nm e 60 nm, mentre la NAS ha aumentato la vitalità solo a 10 μg/mL di CuNP a 20 nm. L'SBI ha mostrato i suoi effetti solo in A. baumannii trattato con 60 nm di CuNP a concentrazioni di 1 μg/mL e 5 μg/mL (Figura 3F).

Figura 3: Effetto dei modulatori della PCD sulla sopravvivenza batterica dopo il trattamento con CuNP di due diverse dimensioni. E. coli (A,B), S. aureus (C,D) e A. baumannii (E,F) sono stati trattati con PBS (controllo negativo) e concentrazioni variabili (0 μg/mL, 1 μg/mL, 5 μg/mL, 10 μg/mL, 50 μg/mL e 100 μg/mL) di CuNP a 20 nm o 60 nm in assenza (simulazione) o presenza dei modulatori della PCD SBI, Z-VAD, NSA e Wort. I batteri trattati con alcol al 70% sono serviti come controllo positivo. La vitalità cellulare è stata valutata utilizzando un test di vitalità disponibile in commercio. I dati sono presentati come media ± SD da almeno tre esperimenti indipendenti, ciascuno eseguito in triplice copia. I valori anomali sono stati identificati attraverso l'ispezione visiva. Il test di Shapiro-Wilk è stato utilizzato per valutare la normalità dei dati. I confronti statistici sono stati condotti utilizzando l'ANOVA e il t-test di Student. Un asterisco (*) indica P < 0,05 per i trattamenti con CuNP rispetto al controllo negativo, mentre una doppia croce (‡) indica P < 0,05 per i confronti tra il mock e altri modulatori ad ogni concentrazione. Clicca qui per visualizzare una versione più grande di questa figura.
Gli effetti sinergici del battericida sono stati osservati anche con i disperdenti e le CuNP (Figura 3). SBI, Wort e NSA hanno migliorato la vitalità batterica in S. aureus quando esposti a SDS in 0 μg/mL di CuNP a 60 nm (Figura 3F). Il mosto ha aumentato i tassi di sopravvivenza in A. baumannii trattati con CuNP a 20 nm e 60 nm in gruppi simulati (Figura 3E, F). L'NSA ha anche aumentato la vitalità di A. baumannii nel trattamento simulato con CuNP a 60 nm (Figura 3F). Al contrario, l'SBI ha ridotto la vitalità in E. coli trattato con il mock di CuNP a 60 nm (Figura 3B), mentre Z-VAD e NSA hanno ridotto i tassi di sopravvivenza in A. baumannii trattato con il mock di CuNP a 20 nm (Figura 3E). Questi risultati suggeriscono che i meccanismi battericidi in questi tre batteri, se trattati con disperdenti, potrebbero differire da quelli che coinvolgono le nanoparticelle.
Il rame è un oligoelemento essenziale per vari processi biologici ed è fondamentale per mantenere l'equilibrio degli ioni rame all'interno dei batteri. I componenti importanti presenti nel sistema di omeostasi del rame nei batteri regolano l'assorbimento, la distribuzione e l'escrezione del rame attraverso le ATPasi esportatrici di rame, le proteine chaperone del rame e i regolatori trascrizionali sensibili al rame (Figura 4). Questi componenti modulano l'espressione delle proteine che legano il rame e le proteine di trasporto in risposta ai livelli intracellulari di rame, garantendo una concentrazione adeguata per i processi essenziali e prevenendo la tossicità18.
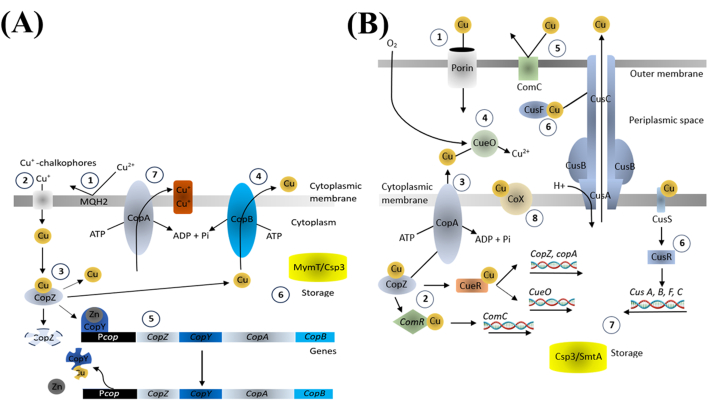
Figura 4: Omeostasi del rame nei batteri gram-positivi (A) e gram-negativi (B). Clicca qui per visualizzare una versione più grande di questa figura.
Ipotizziamo che l'effetto battericida delle CuNP sia strettamente correlato all'interruzione dei meccanismi di omeostasi del rame. Quando i batteri sono sopraffatti da un eccesso di rame, il normale processo di regolazione non riesce a tenere il passo, portando allo stress ossidativo attraverso la generazione di ROS e l'attivazione del sistema PCD batterico. Questo, a sua volta, sottoregola la sintesi proteica importante e promuove la produzione di proteine legate alla morte. L'applicazione dei modulatori dimostra come questi agenti possano migliorare la sopravvivenza batterica, indicando che l'uccisione batterica indotta da CuNP interferisce anche con processi come l'autofagia e la proteolisi, provocando infine la morte batterica. Questi risultati evidenziano l'importanza delle vie della PCD nella morte batterica indotta da CuNP.
Discussione
Questo studio ha studiato gli effetti antimicrobici e i meccanismi delle CuNP a due dimensioni e varie concentrazioni contro E. coli, S. aureus e A. baumannii. Utilizzando i protocolli stabiliti, è stato osservato che gli effetti battericidi indotti da CuNP coinvolgono lo stress ossidativo e la potenziale attivazione della PCD. Tuttavia, l'interazione tra l'omeostasi dei metalli e le risposte allo stress batterico rimane in gran parte inesplorata. Studi precedenti hanno identificato strategie di resistenza batterica, come pompe di efflusso di rame e proteine leganti i metalli, che possono influenzare la suscettibilità alla CuNP19. Inoltre, la potenziale citotossicità degli ioni rame rimane una preoccupazione per le applicazioni terapeutiche, evidenziando la necessità di un'attenta ottimizzazione del dosaggio20. Una comprensione più approfondita dei meccanismi battericidi delle CuNP è essenziale per promuoverne l'uso sicuro ed efficace.
La discussione qui sottolineerà gli aspetti procedurali di questo metodo e le loro implicazioni. Le fasi critiche del protocollo includono la corretta preparazione e dispersione di CuNP, garantendo colture batteriche uniformi a una densità ottica a 600 nm (OD600) di 0,5 per risultati riproducibili e un'accurata risospensione di pellet batterici in soluzioni di CuNP per prevenire l'aggregazione. La corretta sequenza di aggiunta dei modulatori, in particolare in relazione all'esposizione a CuNP, è fondamentale per risultati sperimentali efficaci. I trattamenti di controllo, compresi i controlli negativi (PBS) e positivi (70% di alcol), nonché un controllo in bianco CuNP, sono essenziali per stabilire la vitalità di base e garantire che gli effetti osservati siano direttamente correlati al trattamento con CuNP.
Il metodo include anche misurazioni quantitative della vitalità cellulare, combinando il conteggio delle colonie con il test PrestoBlue per una valutazione accurata dell'attività battericida. Inoltre, la rilevazione dei ROS mediante colorante H2DCFDA e la citometria a flusso è una fase analitica fondamentale per comprendere lo stress ossidativo indotto dalle CuNP, stabilendo che la produzione di ROS è un fattore che contribuisce alla morte cellulare batterica. Le modifiche e la risoluzione dei problemi del metodo si concentrano sulla regolazione delle concentrazioni del modulatore in base alla suscettibilità di ciascun ceppo batterico, sull'ottimizzazione dei tempi di incubazione e sulla garanzia di impostazioni corrette della citometria a flusso per rilevare con precisione i ROS. Tuttavia, lo studio è limitato dal fatto che si concentra solo su tre ceppi batterici e dalla potenziale variabilità degli effetti dei modulatori della PCD tra le diverse specie batteriche. È stato osservato un effetto dipendente dalle dimensioni, con CuNP più piccole che mostrano una maggiore attività battericida, ma è necessaria un'ulteriore esplorazione delle variazioni di dimensioni e concentrazione per comprendere appieno il loro impatto.
Questo metodo fornisce un approccio completo per la valutazione delle CuNP come potenziali agenti antimicrobici. La combinazione di saggi sulle colonie, test di vitalità cellulare, rilevamento dei ROS e studi sui modulatori della PCD offre una comprensione sfaccettata dei meccanismi battericidi CuNP. Questo studio evidenzia il potenziale delle CuNP nella lotta contro i batteri resistenti ai farmaci e presenta opportunità per lo sviluppo di terapie antimicrobiche mirate che manipolano le vie batteriche della PCD. Inoltre, le applicazioni del metodo si estendono allo sviluppo di rivestimenti in biomateriali a base di CuNP per dispositivi medici per prevenire le infezioni. Come strumento fondamentale per la comprensione della PCD batterica, questo protocollo è una risorsa preziosa per far progredire la ricerca sulla biologia batterica e sulle strategie antimicrobiche.
Divulgazioni
L'autore dichiara di non avere alcun conflitto di interessi, finanziario o di altro tipo.
Riconoscimenti
Siamo grati per il supporto del Core Facility Center, Tzu Chi University, Taiwan.
Materiali
| Name | Company | Catalog Number | Comments |
| Acinetobacter baumannii Bouvet and Grimont strain | American Type Culture Collection (ATCC), Manassas, VA, USA | 17978 | Bacteria for CuNP toxocity experiment |
| Bio-Rad iMark Microplate Reader | Bio-Rad Laboratories, Hercules, CA, USA | 168-1130 | Used to measure absorbance in bacterial viability assays. |
| cell-permeant 2’,7’-dichlorodihydrofluorescein diacetate (H2DCFDA) | Sigma-Aldrich, Saint Louis, MO, USA | D6883 | Used for detecting reactive oxygen species (ROS) in treated bacterial cells. |
| Copper nanoparticles (CuNPs) 25 nm | Sigma-Aldrich, St. Louis, MO, USA | 774081 | Used to prepare CuNP stock solution |
| Copper nanoparticles (CuNPs) 60-80 nm | Sigma-Aldrich, St. Louis, MO, USA | 774103 | Used to prepare CuNP stock solution |
| Escherichia coli (Migula) Castellani and Chalmers | American Type Culture Collection (ATCC), Manassas, VA, USA | 25922 | Bacteria for CuNP toxocity experiment |
| Gallios flow cytometer | Beckman Coulter, Brea, CA, USA | Used for flow cytometric analysis in multiple experiments, including reactive oxygen species detection. | |
| LB agar | FocusBio, Miaoli, Taiwan | LBA500 | Used for culturing bacteria |
| Luria-Bertani (LB) broth | Becton, Dickinson and Company, Sparks, MD, USA | 244620 | Used for culturing bacteria |
| Necrosulfonamide (NSA) | Sigma-Aldrich, St. Louis, MO, USA | 480073 | Used as a modulator for pretreatment in bacterial death pathway studies. |
| PrestoBlue Cell Viability Reagent | Invitrogen, Carlsbad, CA, USA | P50200 | Used for assessing cell viability via fluorescence. |
| SBI-0206965 (SBI) | BioVision, Milpitas, CA, USA | 9580 | Used as a modulator for pretreatment in bacterial death pathway studies. |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich, St. Louis, MO, USA | L4509 | Used as a dispersant for copper nanoparticles to reduce aggregation. |
| Staphylococcus aureus | American Type Culture Collection (ATCC), Manassas, VA, USA Bioresource Collection and Research Center (BCRC), Hsinchu, Taiwan | 13567 | Bacteria for CuNP toxocity experiment |
| Varioskan LUX multimode microplate reader | Thermo Fisher Scientific, Waltham, MA, USA | VLBLATGD2 | Used for measuring fluorescence in cell viability assays |
| Wortmannin (Wort) | Abcam, MA, USA | ab120148 | Used as a modulator for pretreatment in bacterial death pathway studies. |
| Z-VAD-FMK (Z-VAD) | Sigma-Aldrich, St. Louis, MO, USA | V116 | Used as a modulator for pretreatment in bacterial death pathway studies. |
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon