Method Article
Untersuchung des durch Kupfer-Nanopartikel induzierten programmierten Zelltods in Bakterien
In diesem Artikel
Zusammenfassung
Kupfer-Nanopartikel wirken als antimikrobielle Wirkstoffe, indem sie reaktive Sauerstoffspezies erzeugen. Hier werden Verfahren vorgestellt, die zeigen, dass Kupfer-Nanopartikel gegen drei klinisch relevante Krankheitserreger wirksam sind und dass bestimmte programmierte Zelltodwege an diesem bakteriziden Prozess beteiligt sind.
Zusammenfassung
In jüngster Zeit hat die Besorgnis über multiresistente Erreger und unheilbare Infektionen aufgrund des übermäßigen und missbräuchlichen Einsatzes von Antibiotika zugenommen. Nanomaterialien wie Metall- und Metalloxid-Nanopartikel haben im biomedizinischen Bereich als potenzielle neue Strategien zur Bekämpfung multiresistenter Krankheitserreger an Popularität gewonnen. In dieser Studie wurde der Einsatz von Kupfer-Nanopartikeln (CuNPs) als Bakterizid gegen drei häufige im Krankenhaus erworbene opportunistische Krankheitserreger - Escherichia coli (E. coli), Acinetobacter baumannii (A. baumannii) und Staphylococcus aureus (S. aureus) - untersucht, die zunehmend Arzneimittelresistenzen entwickeln. Es werden detaillierte Protokolle für die Synthese von CuNPs in zwei Größen (20 nm und 60 nm) und die Bewertung ihrer bakteriziden Wirksamkeit durch Kolonieassays vorgestellt. Die Mechanismen der antimikrobiellen Wirkung, die CuNPs zugrunde liegen, wurden durch die Bewertung von Veränderungen in der Produktion reaktiver Sauerstoffspezies untersucht. Darüber hinaus wurden vier Modulatoren, die die menschlichen Proteinfunktionen hemmen, eingesetzt, um die mögliche Beteiligung des programmierten Zelltods (PCD) an der bakteriellen Abtötung zu untersuchen. Durch diesen Ansatz wird das mögliche Auftreten kupferresistenter Stämme vorgeschlagen, aufbauend auf der Erforschung von Kupfer-Homöostaseproteinen, einschließlich kupferabhängiger Transkriptionsregulatoren. Diese Ergebnisse bieten eine umfassende Methodik zur Untersuchung der bakteriziden Wirkung von CuNPs und ihrer potenziellen Rolle bei der Bekämpfung von Antibiotikaresistenzen.
Einleitung
Arzneimittelresistente Bakterien sind in der Medizin ein ernstzunehmender Grund zur Sorge. Ihr schnelles Auftreten hat die Wirksamkeit herkömmlicher Antibiotika verringert, was zu mehr klinischen Komplikationen führt. Sie stellen eine große Bedrohung für die öffentliche Gesundheit dar und schaffen einen dringenden Bedarf an neuen antimikrobiellen Wirkstoffen. Ein Forschungszweig sind Nanomaterialien. Nanomaterialien besitzen einzigartige physikalisch-chemische Eigenschaften, die es ihnen ermöglichen, mit Mikroben auf eine Weise zu interagieren, die ihre Lebensfähigkeit beeinträchtigt. Zum Beispiel induzieren Silbernanopartikel (AgNPs) oxidativen Stress in Bakterien, was zu Proteindysfunktion, Membranzerstörung, DNA-Schäden und schließlich zum Zelltod führt1. Gold-Nanopartikel (AuNPs) hingegen sind für ihre antimykotischen Eigenschaften bekannt und können die bakterizide Wirkung von Antibiotika verstärken, indem sie als Träger dienen2.
Darüber hinaus haben Kupfer-Nanopartikel (CuNPs) aufgrund ihrer starken antimikrobiellen Wirkung und niedrigen Produktionskosten große Aufmerksamkeit erregt. Studien deuten darauf hin, dass CuNPs durch Störung der enzymatischen Aktivität und die Bildung reaktiver Sauerstoffspezies (ROS) eine bakterizide Breitbandaktivität aufweisen3. Die positive Ladung von CuNPs erleichtert deren Eindringen in die Bakterien und verbessert deren zelluläre Aufnahme4. Dieser Mechanismus macht CuNP zu einer vielversprechenden Option für Oberflächenbeschichtungen, z. B. auf Implantaten, um Infektionen zu verhindern3. Eine interessante Erkenntnis ist jedoch, dass die bakterizide Wirkung von CuNPs größenabhängig zu sein scheint. Einige Studien haben ergeben, dass kleinere CuNPs eine höhere antibakterielle Aktivität aufweisen, wahrscheinlich aufgrund ihres überlegenen Verhältnisses von Oberfläche zu Volumen5.
Die ROS-Erzeugung verursacht weitreichende Schäden an Zellen und Bakterien, einschließlich Lipidperoxidation, Proteindysfunktion, DNA-Fragmentierung und Hemmung der Glukoneogenese/Glykogenolyse, und ist an Nekrose oder programmiertem Zelltod (PCD) beteiligt6,7,8. Jüngste Studien haben gezeigt, dass PCD-Systeme in Bakterien existieren, mit ähnlichen Wirkungsweisen und Effektoren wie in eukaryotischen Systemen9. Bakteriengemeinschaften können PCD als Reaktion auf Stress, einschließlich oxidativen Stress, durch ein Toxin-Antitoxin (TA)-System induzieren10. Vereinfacht ausgedrückt besteht das Toxin-Antitoxin-System aus Toxinen, die essentielle zelluläre Prozesse stören können, und Antitoxinen, die mit den Toxinen stabile Komplexe bilden können, um ihre Toxizität unter normalen Wachstumsbedingungen zu hemmen. Die meisten Bakterien und Archaeen enthalten TA-Loci in ihrem Genom, die oft in mehreren Kopien extrachromosomaler und chromosomaler DNA vorhanden sind. Es gibt verschiedene Arten von TA-Systemen, wobei das Typ II TA (bekannt als MazE/MazF-Modul) von besonderem Interesse ist. Unter Stressbedingungen werden Antitoxine abgebaut, so dass Toxine ihre zellulären Ziele hemmen können. In E. coli und S. aureus wird das Toxin MazF als Reaktion auf Stressbedingungen wie oxidativen Stress, hohe Temperaturen und Aminosäuremangel aktiviert. Infolgedessen wird die Expression des Antitoxins MazE reduziert, wodurch das Toxin MazF10 freigesetzt wird. Studien haben gezeigt, dass MazF die Synthese von Proteinen ermöglicht, die es einer kleinen Subpopulation ermöglichen, unter ungünstigen Bedingungen zu überleben, während der größte Teil der Population einen mazEF-vermittelten Zelltod erfährt. Dieser Zelltod kann entweder ROS-abhängig sein, wobei ROS eine transkriptionelle oder translationale Hemmung induziert, oder ROS-unabhängig, wobei DNA-Schäden die Todeswege auslösen11.
Diese Studie untersucht die Mechanismen, durch die CuNPs den bakteriellen Tod induzieren. Anstatt sich nur auf das TA-System zu konzentrieren, wurden vier PCD-Modulatoren, die zuvor in unserer Forschung verwendet wurden 7,12, eingesetzt, um potenzielle PCD-Signalwege in Bakterien zu untersuchen.
Durch die Untersuchung der bakteriziden Wirkungen von CuNPs in zwei verschiedenen Größen (20 und 60 nm) bei unterschiedlichen Konzentrationen und unter Verwendung von Methoden wie Kolonie-Assays, ROS-Nachweis und PCD-Modulatoren (SBI, Z-VAD, NSA und Wortmannin) unterstreicht diese Forschung, dass PCD nicht nur bei mehrzelligen Organismen auftritt, sondern auch in Bakteriengemeinschaften unter Stress auftritt. Durch die Bereitstellung detaillierter Protokolle soll diese Arbeit die Forscher in die Lage versetzen, die Wirksamkeit von CuNP und die bakteriziden Mechanismen in ihren eigenen Systemen zu bewerten. Darüber hinaus fördern diese Erkenntnisse das Verständnis der bakteriellen PCD und unterstützen die Entwicklung von CuNP-basierten Therapien zur Bekämpfung antibiotikaresistenter Bakterien.
Protokoll
Die in dieser Studie verwendeten Reagenzien und Geräte sind in der Materialtabelle aufgeführt.
1. Herstellung von Kupfer-Nanopartikeln
- Beziehen Sie kommerzielle Kupfer-Nanopulver (25 nm und 60-80 nm) aus einer kommerziellen Quelle.
- Verwenden Sie 1,0 mM Natriumdodecylsulfat (SDS) als Dispergiermittel für zwei Größen von 1 mg/ml-Nanopartikeln.
- Dispergieren Sie die Nanopartikel mit einem Ultraschallbad für mindestens 30 min bei Raumtemperatur. Die vollständig dispergierten Nanopartikel sind dann bereit für den Einsatz in nachfolgenden Experimenten.
2. Vorbereitung der Bakterien
- Besorgen Sie sich E. coli (Migula) Castellani und Chalmers Stamm 25922 und A. baumannii Bouvet und Grimont Stamm aus der American Type Culture Collection. Beziehen Sie S. aureus vom Bioresource Collection and Research Center.
- Die Bakterien werden in Luria-Bertani (LB)-Bouillon unter aeroben Bedingungen bei 37 °C kultiviert.
- Die Bakterienkulturen werden in LB-Medium auf eine optische Dichte bei 600 nm (OD600) von ca. 0,5 verdünnt.
3. Bewertung der Zellviabilität
- Kolonie-Assay
- Verwenden Sie CuNP-Stammlösungen (1 mg/ml), um verschiedene Konzentrationen von CuNPs in zwei Größen herzustellen, darunter 0 μg/ml, 1 μg/ml, 5 μg/ml, 10 μg/ml, 50 μg/ml und 100 μg/ml.
- Die in Schritt 2.3 hergestellten Bakterienkulturen werden in Mikrozentrifugenröhrchen aufgeteilt und bei 3300 × g 10 min bei Raumtemperatur zentrifugiert.
- Halten Sie die Bakterienpellets zurück und fügen Sie durch schonendes Pipettieren unterschiedliche Konzentrationen von CuNPs jeweils in zwei Größen hinzu.
- Behandeln Sie Bakterienpellets mit PBS und 70% Alkohol als Negativ- bzw. Positivkontrolle.
- Inkubieren Sie alle behandelten Bakterien durch Schütteln bei 200 U/min bei 37 °C für 24 h.
- Waschen Sie nach der Inkubation alle behandelten Bakterien mit PBS und verteilen Sie sie auf LB-Agarplatten. Legen Sie die Platten für 24 h in einen 37 °C heißen Inkubator.
- Zählen Sie die Koloniezahlen in jeder Behandlungsgruppe am nächsten Tag und führen Sie eine statistische Analyse durch. Es wird empfohlen, dies aus Gründen der statistischen Genauigkeit in dreifacher Ausfertigung durchzuführen.
- Untersuchung des bakteriziden Mechanismus
- Bereiten Sie die Bakterien wie in Schritt 3.1.2 beschrieben vor und behandeln Sie sie entweder mit 5 μM SBI-0206965 (SBI) für 2 h, 0,5 μM Necrosulfonamid (NSA) für 1 h, 100 nM Wortmannin (Würze) für 30 min oder 100 nM Z-VAD-FMK (Z-VAD) für 30 min.
- Die Bakterien werden mit unterschiedlichen Konzentrationen von CuNP-Lösungen in zwei Größen, wie in Schritt 3.1.1 beschrieben, in Gegenwart oder Abwesenheit von 5 μM SBI, 0,5 μM NSA, 100 nM Würze und 100 nM Z-VAD gleichzeitig behandelt.
- Die Bakterien nach den Modulatorbehandlungen (Schritt 3.2.1) zentrifugieren und die Überstände entfernen.
- Die Bakterienpellets werden in den in Schritt 3.2.2 hergestellten Lösungen resuspendiert und unter Schütteln bei 200 U/min bei 37 °C 24 h lang inkubiert.
- Behandeln Sie die Bakterien mit 70 % Ethanol und PBS als Positiv- bzw. Negativkontrolle. Verwenden Sie eine Lösung ohne CuNPs als CuNP-Blindkontrolle (0 μg/ml; Mock) unter den gleichen Inhibitorbedingungen für jede Gruppe. Inkubieren Sie alle Proben für weitere 24 Stunden.
- Nach der Inkubation geben Sie das Reagenz für die Zellviabilität in einem Volumenverhältnis von 1:10 in die Kulturen. Inkubieren Sie die Kulturen für weitere 2 Stunden unter Schütteln bei 37 °C.
- Die Kulturen werden nach der 2-stündigen Inkubation zentrifugiert (Schritt 3.1.2). Übertragen Sie die fluoreszierenden Überstände auf 96-Well-Platten. Messen Sie die Fluoreszenz mit einer Anregungswellenlänge von 560 nm und einer Emissionswellenlänge von 590 nm mit einem Mikroplatten-Reader.
- Den restlichen Überstand auf 10-5 und 10-4 verdünnen und zur Kultivierung auf LB-Agarplatten verteilen.
- Einzelne Völker am nächsten Tag zählen.
4. Nachweis reaktiver Sauerstoffspezies
- Bereiten Sie Bakterienkulturen wie in Schritt 2.3 beschrieben vor und teilen Sie sie in Mikrozentrifugenröhrchen auf.
- Behandeln Sie die Bakterien mit verschiedenen Stressbedingungen als ROS-induzierende Positivkontrollgruppen (Daten in den Ergebnissen nicht gezeigt). Die Behandlungen sind in den Schritten 4.2.1-4.2.4 beschrieben.
- Setzen Sie die Bakterien 3 h lang 405 nm UV-Licht aus. Inkubieren Sie die Bakterien 2 Stunden lang bei 45 °C.
- Anschließend die Bakterien 2 h lang bei 4 °C inkubieren.
- Bakterien mit 3%H2O2 für 30 min behandeln.
- Halten Sie die Bakterien bei 37 °C in LB-Brühe als Negativkontrolle.
- Bereiten Sie verschiedene Konzentrationen von CuNP vor, wie in Schritt 3.1.1 beschrieben, und behandeln Sie die Bakterien mit 20 nm oder 60 nm CuNP in Konzentrationen von 1 μg/ml, 5 μg/ml, 10 μg/ml und 100 μg/ml für 24 h.
- Waschen Sie die inkubierten Bakterien zweimal mit PBS, um alle verbleibenden Nanopartikel zu entfernen.
- Bereiten Sie den Farbstoff 2′,7′-Dichlordihydrofluoresceindiacetat (H2DCFDA) in PBS in einer Endkonzentration von 5 μM vor.
- Resuspendieren Sie die Bakterienpellets in 5 μM H2DCFDA und messen Sie die Fluoreszenzintensität bei 520/30 nm Emission mit einem Durchflusszytometer.
HINWEIS: Die Intensität der grünen Fluoreszenz von FL1 korreliert mit dem ROS-Spiegel in der behandelten Kultur. Es wird empfohlen, dies aus Gründen der statistischen Genauigkeit in dreifacher Ausfertigung durchzuführen.
Ergebnisse
Antimikrobielle Aktivitäten von CuNPs der zweiten Größe in drei Krankheitserregern
Drei opportunistische Krankheitserreger (E. coli, S. aureus und A. baumannii) wurden verwendet, um die bakterizide Aktivität von CuNPs zu testen. Die Bakterien wurden mit 0 μg/ml, 1 μg/ml, 5 μg/ml, 10 μg/ml, 50 μg/ml und 100 μg/ml von 20 nm oder 60 nm CuNPs behandelt, und die bakteriziden Aktivitäten wurden unter Verwendung der minimalen bakteriziden Konzentration (MBC) bestimmt, die aus den Koloniezahlen abgeleitet wurde. Unsere Ergebnisse zeigten positive bakterizide Effekte beider Größen von CuNPs (Abbildung 1) für alle drei Stämme. Bei E. coli wurde eine signifikante Abnahme der Koloniezahlen ab 1 μg/ml bzw. 5 μg/ml für die 20-nm- bzw. 60-nm-CuNP-Behandlungen beobachtet (Abbildung 1A). Im Gegensatz dazu zeigten sich bemerkenswerte Reduzierungen der Koloniezahlen für S. aureus über alle Konzentrationen beider CuNP-Größen hinweg (Abbildung 1B). Für A. baumannii waren höhere Konzentrationen von CuNPs (5 μg/mL in 20 nm CuNPs und 10 μg/mL in 60 nm CuNPs) erforderlich, um eine Verringerung der Koloniezahlen zu erreichen (Abbildung 1C).
Unsere früheren Studien zeigten, dass kleinere CuNPs (20 nm) im Vergleich zu den größeren (60 nm) eine außergewöhnlichere bakterizide Fähigkeit aufweisen7,12. Hier zeigten sich ähnliche Trends, was auf die konsistenten Ergebnisse hinweist. Darüber hinaus deuten diese Befunde darauf hin, dass grampositive Bakterien (in diesem Fall S. aureus) anfälliger für CuNPs sind als die getesteten gramnegativen Bakterien (E. coli und A. baumannii).
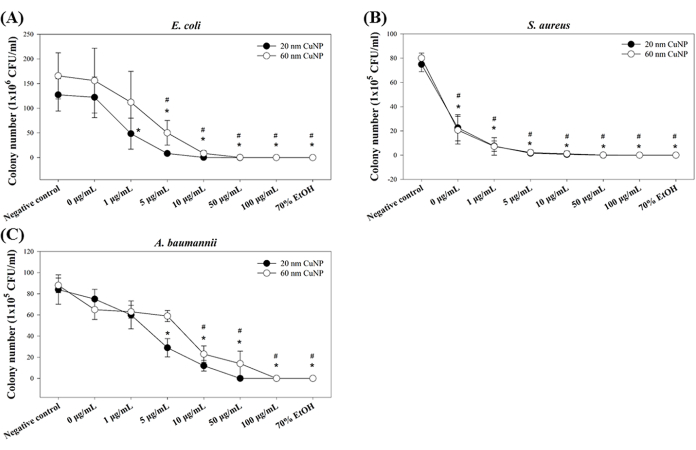
Abbildung 1: Bakterizide Aktivität von CuNPs zweier unterschiedlicher Größe in drei Bakterienarten. E. coli (A), S. aureus (B) und A. baumannii (C) wurden mit unterschiedlichen Konzentrationen von 20 nm oder 60 nm CuNPs behandelt, und die Anzahl der Bakterienkolonien wurde bestimmt. PBS- und 70% alkoholbehandelte Bakterien dienten als Negativ- bzw. Positivkontrollen. Die Daten werden als Mittelwert ± Standardabweichung (SD) aus vier unabhängigen Experimenten ausgedrückt, die jeweils in dreifacher Ausfertigung durchgeführt wurden. Ausreißer wurden durch visuelle Inspektion identifiziert. Der Shapiro-Wilk-Test wurde verwendet, um die Datennormalität zu beurteilen. Statistische Vergleiche wurden mit dem Student's t-Test durchgeführt. Sternchen (*) und Nummernzeichen (#) zeigen P < 0,05 für 20-nm- bzw. 60-nm-CuNP-Behandlungen im Vergleich zur Negativkontrolle an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tödliche Schädigung von Bakterien durch CuNP-induzierte ROS-Erzeugung
Die vorgeschlagenen Mechanismen, mit denen CuNPs Bakterien abtöten, variieren. Zwei Haupttheorien werden diskutiert: der Ersatz von Eisen-Schwefel-Clusterproteinen durch Kupfer, das aus Nanopartikeln freigesetzt wird, und die CuNP-induzierte ROS-Produktion13,14. Die ROS-Produktion wurde in drei CuNP-behandelten Bakterien untersucht, um die Mechanismen zu untersuchen (Abbildung 2). E. coli, S. aureus und A. baumannii wurden mit unterschiedlichen Konzentrationen von 20 nm oder 60 nm CuNPs behandelt, gefolgt von der Anwendung von H2DCFDA zum ROS-Nachweis. Hohe Anteile von grün fluoreszierenden Populationen wurden mittels Durchflusszytometrie in allen Konzentrationen von 20 nm und 60 nm CuNP-Behandlungen gemessen (Abbildung 2). Interessanterweise waren die Populationen positiver Zellen in den 20-nm-CuNP-Behandlungen konzentrationsunabhängig (Abbildung 2A). Bemerkenswert ist, dass niedrigere Konzentrationen von 20 nm CuNPs eine höhere ROS-Erzeugung verursachten, während höhere Konzentrationen einen geringeren Anteil positiver Zellen aufwiesen. Dieses Phänomen wurde jedoch in den mit 60 nm CuNP behandelten Zellen nicht beobachtet (Abbildung 2B). Es wurden keine signifikanten Unterschiede zwischen den drei Bakterien festgestellt, die entweder mit 20 nm oder 60 nm CuNPs behandelt wurden (Abbildung 2). Bei 0 μg/ml wurden leichte Erhöhungen positiver Populationen festgestellt, die auf Dispergiermittel und Osmosedruck zurückzuführen sind (Abbildung 2).
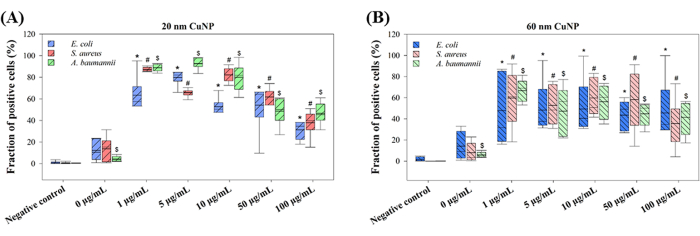
Abbildung 2: ROS-Bildung nach Behandlung mit CuNPs unterschiedlicher Größe bei drei Bakterienarten. E. coli, S. aureus und A. baumannii wurden unterschiedlichen Konzentrationen von 20 nm (A) oder 60 nm (B) CuNPs ausgesetzt. Die ROS-Produktion wurde mittels H2-DCFDA-Färbung nach der CuNP-Behandlung bewertet, und der Anteil fluoreszierender positiver Zellen wurde mittels Durchflusszytometrie analysiert. Die Daten werden als Mittelwert ± SD aus fünf unabhängigen Experimenten dargestellt, die jeweils in dreifacher Ausfertigung durchgeführt wurden. Ausreißer wurden durch visuelle Inspektion identifiziert. Der Shapiro-Wilk-Test wurde verwendet, um die Datennormalität zu beurteilen. Statistische Vergleiche wurden mit Hilfe der Kruskal-Wallis-Einweganalyse durchgeführt. Die Symbole (*, # und $) zeigen P < 0,05 für 20-nm- bzw. 60-nm-CuNP-Behandlungen relativ zur Negativkontrolle an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Vier PCD-Modulatoren entschlüsseln die bakteriellen Abtötungsmechanismen von CuNPs
Vier PCD-Modulatoren wurden in nachfolgenden Experimenten verwendet, um die durch CuNP-Behandlungen induzierten Zelltodmechanismen bei drei Krankheitserregern weiter zu untersuchen. Es ist bekannt, dass diese Modulatoren - SBI, Z-VAD, NSA und Wort - die Signaltransduktionswege blockieren, die mit Apoptose (Z-VAD), Autophagie (SBI und Wort) und Nekroptose (NSA) in Säugetierzellen verbunden sind15-17. Studien haben gezeigt, dass PCD nicht auf mehrzellige Organismen beschränkt ist; Verschiedene Formen von PCD wurden in Bakterienkolonien als Reaktion auf raue Bedingungen beobachtet, die der gesamten Kolonie zugute kamen und Anpassungsstrategien wie genetische Transformation und Biofilmbildung erleichterten10. Aufbauend auf diesem Verständnis untersuchten wir, ob PCD-bezogene Signalwege zum CuNP-induzierten Zelltod in Bakterien beitragen. Die drei Bakterien, E. coli, S. aureus und A. baumannii, wurden mit den vier Modulatoren vorbehandelt, gefolgt von einer Co-Behandlung mit zwei Größen von CuNPs und den Modulatoren (Abbildung 3).
Die Zellviabilität zeigte eine konzentrationsabhängige Abnahme in beiden Größen der CuNP-Behandlungen bei drei Bakterien (Abbildung 3), was mit den in Abbildung 1 dargestellten Ergebnissen übereinstimmt. Bei E. coli wurde ein erhöhtes Überleben in der Z-VAD-Gruppe unter Behandlung mit 1 μg/ml und 5 μg/ml 20 nm CuNP sowie in der NSA-Gruppe unter Behandlung mit 10 μg/ml 20 nm CuNP beobachtet (Abbildung 3A). Z-VAD war jedoch bei allen Konzentrationen von 60 nm-CuNP-Behandlungen wirksam bei der Rettung des Überlebens bei E. coli , jedoch nicht bei 100 μg/ml (Abbildung 3B). Unterschiedliche Viativitätsergebnisse bei S. aureus deuten auf bestimmte bakterizide Signalwege bei den 20-nm-CuNP-Behandlungen hin (Abbildung 3C). NSA spielte bei allen Konzentrationen von 20-nm-CuNP-Behandlungen eine entscheidende Rolle, während Z-VAD nur bei 1 μg/ml 20-nm-CuNPs wirksam war. S. aureus zeigte bei keiner Konzentration ein Ansprechen auf Modulatoren, mit Ausnahme von Z-VAD bei 10 μg/ml von 60 nm CuNP-Behandlungen (Abbildung 3D). Ähnliche Trends bei den Zellüberlebensraten wie bei E. coli wurden bei A. baumannii festgestellt, die mit 20-nm-CuNPs behandelt wurden (Abbildung 3E). Z-VAD war wirksam bei der Rettung bei 1 μg/ml, 5 μg/ml und 10 μg/ml bei 20 nm und 60 nm CuNP-Behandlungen, während NAS die Lebensfähigkeit nur bei 10 μg/ml 20 nm CuNP erhöhte. SBI zeigte seine Wirkung nur bei A. baumannii, die mit 60 nm CuNPs in Konzentrationen von 1 μg/ml und 5 μg/ml behandelt wurden (Abbildung 3F).

Abbildung 3: Einfluss von PCD-Modulatoren auf das bakterielle Überleben nach Behandlung mit CuNPs in zwei verschiedenen Größen. E. coli (A,B), S. aureus (C,D) und A. baumannii (E,F) wurden mit PBS (Negativkontrolle) und unterschiedlichen Konzentrationen (0 μg/ml, 1 μg/ml, 5 μg/ml, 10 μg/ml, 50 μg/ml und 100 μg/ml) von 20 nm oder 60 nm CuNP in Abwesenheit (Schein) oder Vorhandensein der PCD-Modulatoren SBI behandelt. Z-VAD, NSA und Würze. Als Positivkontrolle dienten Bakterien, die mit 70%igem Alkohol behandelt wurden. Die Zellviabilität wurde mit einem kommerziell erhältlichen Viabilitätsassay bewertet. Die Daten werden als Mittelwert ± SD aus mindestens drei unabhängigen Experimenten dargestellt, die jeweils in dreifacher Ausfertigung durchgeführt wurden. Ausreißer wurden durch visuelle Inspektion identifiziert. Der Shapiro-Wilk-Test wurde verwendet, um die Datennormalität zu beurteilen. Statistische Vergleiche wurden mit Hilfe der ANOVA und des Student's t-Tests durchgeführt. Ein Sternchen (*) bedeutet P < 0,05 für CuNP-Behandlungen im Vergleich zur Negativkontrolle, während ein Doppelkreuz (‡) P < 0,05 für Vergleiche zwischen dem Mock und anderen Modulatoren bei jeder Konzentration anzeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Synergieeffekte des Bakterizids wurden auch mit den Dispergiermitteln und CuNPs beobachtet (Abbildung 3). SBI, Würze und NSA verbesserten die bakterielle Lebensfähigkeit von S. aureus, wenn sie SDS in 0 μg/ml von 60 nm CuNPs ausgesetzt wurden (Abbildung 3F). Die Würze erhöhte die Überlebensraten bei A. baumannii, die mit 20 nm und 60 nm CuNPs in Scheingruppen behandelt wurden (Abbildung 3E,F). NSA erhöhte auch die Lebensfähigkeit von A. baumannii in der Scheinbehandlung mit 60 nm CuNPs (Abbildung 3F). Im Gegensatz dazu verringerte SBI die Lebensfähigkeit von E. coli, die mit dem Mock von 60 nm CuNPs behandelt wurden (Abbildung 3B), während Z-VAD und NSA die Überlebensraten bei A. baumannii reduzierten, die mit dem Mock von 20 nm CuNPs behandelt wurden (Abbildung 3E). Diese Ergebnisse deuten darauf hin, dass sich die bakteriziden Mechanismen in diesen drei Bakterien, wenn sie mit Dispergiermitteln behandelt werden, von denen mit Nanopartikeln unterscheiden könnten.
Kupfer ist ein essentielles Spurenelement für verschiedene biologische Prozesse und es ist wichtig, das Gleichgewicht der Kupferionen in Bakterien aufrechtzuerhalten. Die wichtigen Komponenten, die im Kupferhomöostasesystem in Bakterien gefunden werden, regulieren die Aufnahme, Verteilung und Ausscheidung von Kupfer durch kupferexportierende ATPasen, Kupfer-Chaperon-Proteine und kupfer-responsive Transkriptionsregulatoren (Abbildung 4). Diese Komponenten modulieren die Expression von kupferbindenden und -transportierenden Proteinen als Reaktion auf den intrazellulären Kupferspiegel, wodurch eine angemessene Konzentration für essentielle Prozesse sichergestellt und gleichzeitig Toxizität verhindertwird 18.
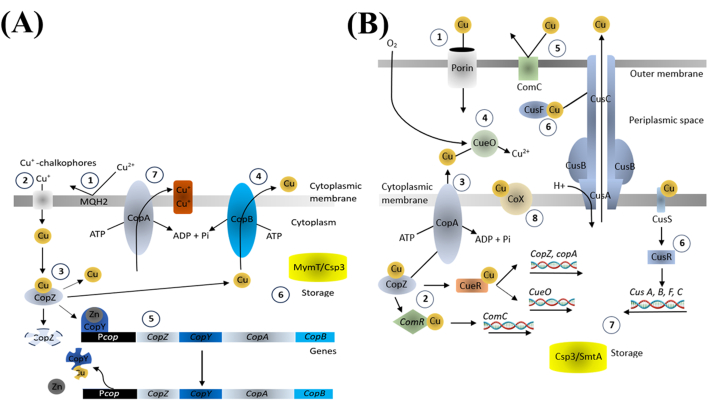
Abbildung 4: Kupferhomöostase in grampositiven (A) und gramnegativen (B) Bakterien. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Wir postulieren, dass die bakterizide Wirkung von CuNPs eng mit der Störung der Kupfer-Homöostase-Mechanismen zusammenhängt. Wenn Bakterien von überschüssigem Kupfer überwältigt werden, kann der normale Regulationsprozess nicht Schritt halten, was zu oxidativem Stress durch die Erzeugung von ROS und die Aktivierung des bakteriellen PCD-Systems führt. Dies wiederum reguliert die wichtige Proteinsynthese herunter und fördert die Produktion von todrelevanten Proteinen. Die Anwendung der Modulatoren zeigt, wie diese Wirkstoffe die Überlebensfähigkeit von Bakterien verbessern könnten, was darauf hindeutet, dass die CuNP-induzierte bakterielle Abtötung auch Prozesse wie Autophagie und Proteolyse stört, was letztendlich zum bakteriellen Tod führt. Diese Ergebnisse unterstreichen die Bedeutung der PCD-Signalwege für den CuNP-induzierten bakteriellen Tod.
Diskussion
In dieser Studie wurden die antimikrobiellen Wirkungen und Mechanismen von CuNPs in zwei Größen und unterschiedlichen Konzentrationen gegen E. coli, S. aureus und A. baumannii untersucht. Unter Verwendung der etablierten Protokolle wurde beobachtet, dass CuNP-induzierte bakterizide Wirkungen oxidativen Stress und eine mögliche PCD-Aktivierung beinhalten. Das Zusammenspiel zwischen Metallhomöostase und bakteriellen Stressreaktionen ist jedoch noch weitgehend unerforscht. Frühere Studien haben bakterielle Resistenzstrategien wie Kupfer-Efflux-Pumpen und metallbindende Proteine identifiziert, die die CuNP-Empfindlichkeit beeinflussen können19. Darüber hinaus bleibt die potenzielle Zytotoxizität von Kupferionen ein Problem für therapeutische Anwendungen, was die Notwendigkeit einer sorgfältigen Dosisoptimierung unterstreicht20. Ein tieferes Verständnis der bakteriziden Mechanismen von CuNPs ist unerlässlich, um ihre sichere und effektive Anwendung voranzutreiben.
In der Diskussion hier werden die prozessualen Aspekte dieser Methode und ihre Implikationen hervorgehoben. Zu den kritischen Schritten des Protokolls gehören die ordnungsgemäße Vorbereitung und Dispersion von CuNP, die Gewährleistung gleichmäßiger Bakterienkulturen mit einer optischen Dichte bei 600 nm (OD600) von 0,5 für reproduzierbare Ergebnisse und die genaue Resuspension von Bakterienpellets in CuNP-Lösungen, um eine Aggregation zu verhindern. Die richtige Reihenfolge der Zugabe von Modulatoren, insbesondere in Bezug auf die CuNP-Exposition, ist entscheidend für effektive experimentelle Ergebnisse. Kontrollbehandlungen, einschließlich negativer (PBS) und positiver (70 % Alkohol) Kontrollen sowie einer CuNP-Blindkontrolle, sind unerlässlich, um die Ausgangsviabilität zu ermitteln und sicherzustellen, dass die beobachteten Wirkungen in direktem Zusammenhang mit der CuNP-Behandlung stehen.
Die Methode umfasst auch quantitative Messungen der Zellviabilität, wobei die Koloniezählung mit dem PrestoBlue-Assay kombiniert wird, um eine genaue Beurteilung der bakteriziden Aktivität zu ermöglichen. Darüber hinaus ist der ROS-Nachweis mittels H2DCFDA-Farbstoff und Durchflusszytometrie ein wichtiger analytischer Schritt zum Verständnis des durch CuNPs induzierten oxidativen Stresses, der die ROS-Produktion als einen Faktor für den bakteriellen Zelltod etabliert. Modifikationen und Fehlerbehebungen der Methode konzentrieren sich auf die Anpassung der Modulatorkonzentrationen basierend auf der Anfälligkeit jedes Bakterienstamms, die Optimierung der Inkubationszeiten und die Sicherstellung der korrekten Durchflusszytometrie-Einstellungen, um ROS genau zu erkennen. Die Studie ist jedoch durch ihren Fokus auf nur drei Bakterienstämme und die potenzielle Variabilität in der Wirkung von PCD-Modulatoren über verschiedene Bakterienarten hinweg eingeschränkt. Es wurde ein größenabhängiger Effekt beobachtet, wobei kleinere CuNPs eine höhere bakterizide Aktivität zeigten, aber weitere Untersuchungen von Größen- und Konzentrationsvariationen sind erforderlich, um ihre Auswirkungen vollständig zu verstehen.
Diese Methode bietet einen umfassenden Ansatz zur Bewertung von CuNPs als potenzielle antimikrobielle Wirkstoffe. Die Kombination von Kolonie-Assays, Zellviabilitätstests, ROS-Detektion und PCD-Modulator-Studien bietet ein facettenreiches Verständnis der bakteriziden Mechanismen von CuNP. Diese Studie unterstreicht das Potenzial von CuNPs bei der Bekämpfung arzneimittelresistenter Bakterien und zeigt Möglichkeiten für die Entwicklung gezielter antimikrobieller Therapien auf, die die bakteriellen PCD-Signalwege manipulieren. Darüber hinaus erstrecken sich die Anwendungen des Verfahrens auf die Entwicklung von CuNP-basierten Biomaterialbeschichtungen für Medizinprodukte zur Vorbeugung von Infektionen. Als grundlegendes Instrument zum Verständnis der bakteriellen PCD ist dieses Protokoll eine wertvolle Ressource für die Förderung der Forschung in der bakteriellen Biologie und antimikrobiellen Strategien.
Offenlegungen
Der Autor erklärt keinen Interessenkonflikt, weder finanziell noch anderweitig.
Danksagungen
Wir sind dankbar für die Unterstützung durch das Core Facility Center der Tzu Chi Universität, Taiwan.
Materialien
| Name | Company | Catalog Number | Comments |
| Acinetobacter baumannii Bouvet and Grimont strain | American Type Culture Collection (ATCC), Manassas, VA, USA | 17978 | Bacteria for CuNP toxocity experiment |
| Bio-Rad iMark Microplate Reader | Bio-Rad Laboratories, Hercules, CA, USA | 168-1130 | Used to measure absorbance in bacterial viability assays. |
| cell-permeant 2’,7’-dichlorodihydrofluorescein diacetate (H2DCFDA) | Sigma-Aldrich, Saint Louis, MO, USA | D6883 | Used for detecting reactive oxygen species (ROS) in treated bacterial cells. |
| Copper nanoparticles (CuNPs) 25 nm | Sigma-Aldrich, St. Louis, MO, USA | 774081 | Used to prepare CuNP stock solution |
| Copper nanoparticles (CuNPs) 60-80 nm | Sigma-Aldrich, St. Louis, MO, USA | 774103 | Used to prepare CuNP stock solution |
| Escherichia coli (Migula) Castellani and Chalmers | American Type Culture Collection (ATCC), Manassas, VA, USA | 25922 | Bacteria for CuNP toxocity experiment |
| Gallios flow cytometer | Beckman Coulter, Brea, CA, USA | Used for flow cytometric analysis in multiple experiments, including reactive oxygen species detection. | |
| LB agar | FocusBio, Miaoli, Taiwan | LBA500 | Used for culturing bacteria |
| Luria-Bertani (LB) broth | Becton, Dickinson and Company, Sparks, MD, USA | 244620 | Used for culturing bacteria |
| Necrosulfonamide (NSA) | Sigma-Aldrich, St. Louis, MO, USA | 480073 | Used as a modulator for pretreatment in bacterial death pathway studies. |
| PrestoBlue Cell Viability Reagent | Invitrogen, Carlsbad, CA, USA | P50200 | Used for assessing cell viability via fluorescence. |
| SBI-0206965 (SBI) | BioVision, Milpitas, CA, USA | 9580 | Used as a modulator for pretreatment in bacterial death pathway studies. |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich, St. Louis, MO, USA | L4509 | Used as a dispersant for copper nanoparticles to reduce aggregation. |
| Staphylococcus aureus | American Type Culture Collection (ATCC), Manassas, VA, USA Bioresource Collection and Research Center (BCRC), Hsinchu, Taiwan | 13567 | Bacteria for CuNP toxocity experiment |
| Varioskan LUX multimode microplate reader | Thermo Fisher Scientific, Waltham, MA, USA | VLBLATGD2 | Used for measuring fluorescence in cell viability assays |
| Wortmannin (Wort) | Abcam, MA, USA | ab120148 | Used as a modulator for pretreatment in bacterial death pathway studies. |
| Z-VAD-FMK (Z-VAD) | Sigma-Aldrich, St. Louis, MO, USA | V116 | Used as a modulator for pretreatment in bacterial death pathway studies. |
Referenzen
- Anees Ahmad, S., et al. Bactericidal activity of silver nanoparticles: A mechanistic review. Mater Sci Energy Technol. 3, 756-769 (2020).
- Zhang, Y., Shareena Dasari, T. P., Deng, H., Yu, H. Antimicrobial activity of gold nanoparticles and ionic gold. J Environ Sci Health C Environ Carcinog Ecotoxicol Rev. 33 (3), 286-327 (2015).
- Solioz, M. Copper and Bacteria: Evolution, Homeostasis and ToxicitySpringerBriefs in Molecular Science. Ch 2, Springer Cham. 11-19 (2018).
- Ma, X., Zhou, S., Xu, X., Du, Q. Copper-containing nanoparticles: Mechanism of antimicrobial effect and application in dentistry-a narrative review. Front Surg. 9, 905892(2022).
- Draviana, H. T., et al. Size and charge effects of metal nanoclusters on antibacterial mechanisms. J Nanobiotechnology. 21 (1), 428(2023).
- Hasanuzzaman, M., et al. Regulation of ROS metabolism in plants under environmental stress: A review of recent experimental evidence. Int J Mol Sci. 21 (22), ijms21228695(2020).
- Lai, M. J., et al. Effect of size and concentration of copper nanoparticles on the antimicrobial activity in Escherichia coli through multiple mechanisms. Nanomaterials (Basel). 12 (21), 12213715(2022).
- Sai, D. L., Lee, J., Nguyen, D. L., Kim, Y. P. Tailoring photosensitive ROS for advanced photodynamic therapy. Exp Mol Med. 53 (4), 495-504 (2021).
- Peeters, S. H., de Jonge, M. I. For the greater good: Programmed cell death in bacterial communities. Microbiol Res. 207, 161-169 (2018).
- Allocati, N., Masulli, M., Di Ilio, C., De Laurenzi, V. Die for the community: An overview of programmed cell death in bacteria. Cell Death Dis. 6 (1), e1609(2015).
- Kolodkin-Gal, I., Sat, B., Keshet, A., Engelberg-Kulka, H. The communication factor EDF and the toxin-antitoxin module mazEF determine the mode of action of antibiotics. PLoS Biol. 6 (12), e319(2008).
- Lai, M. J., Huang, Y. W., Wijaya, J., Liu, B. R. Copper Overview. Fernández, G. D. Ch 4, IntechOpen. (2024).
- Macomber, L., Imlay, J. A. The iron-sulfur clusters of dehydratases are primary intracellular targets of copper toxicity. Proc Natl Acad Sci U S A. 106 (20), 8344-8349 (2009).
- Ramos-Zúñiga, J., Bruna, N., Pérez-Donoso, J. M. Toxicity mechanisms of copper nanoparticles and copper surfaces on bacterial cells and viruses. Int J Mol Sci. 24 (13), ijms241310503(2023).
- Ihara, M., Shichijo, K., Takeshita, S., Kudo, T. Wortmannin, a specific inhibitor of phosphatidylinositol-3-kinase, induces accumulation of DNA double-strand breaks. J Radiat Res. 61 (2), 171-176 (2020).
- Yang, Y. P., et al. Application and interpretation of current autophagy inhibitors and activators. Acta Pharmacol Sin. 34 (5), 625-635 (2013).
- Li, X., et al. The caspase inhibitor Z-VAD-FMK alleviates endotoxic shock via inducing macrophages necroptosis and promoting MDSCs-mediated inhibition of macrophages activation. Front Immunol. 10, 1824(2019).
- Solioz, M. Copper and Bacteria: Evolution, Homeostasis and ToxicitySpringerBriefs in Molecular Science. Chapter 3, Springer. Cham. 21-48 (2018).
- Greene, N. P., Koronakis, V. Bacterial metal resistance: Coping with copper without cooperativity. mBio. 12 (3), e0065321(2021).
- Butsyk, A., et al. Copper nanoparticle loaded electrospun patches for infected wound treatment: from development to in-vivo application. Polymers (Basel). 16 (19), polym16192733(2024).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten