Method Article
Spettrometria di massa a scambio idrogeno/deuterio al millisecondo per lo studio della dinamica strutturale dell'alfa-sinucleina in condizioni fisiologiche
In questo articolo
Riepilogo
L'insieme strutturale dell'alfa-sinucleina monomerica influenza la sua funzione fisiologica e le proprietà fisico-chimiche. Il presente protocollo descrive come eseguire la spettrometria di massa a scambio idrogeno/deuterio al millisecondo e le successive analisi dei dati per determinare le informazioni conformazionali sul monomero di questa proteina intrinsecamente disordinata in condizioni fisiologiche.
Abstract
L'alfa-sinucleina (aSyn) è una proteina intrinsecamente disordinata i cui aggregati fibrillari sono abbondanti nei corpi di Lewy e nei neuriti, che sono i segni distintivi della malattia di Parkinson. Tuttavia, gran parte della sua attività biologica, così come la sua aggregazione, coinvolge centralmente la forma monomerica solubile della proteina. La delucidazione dei meccanismi molecolari di aSyn biologia e fisiopatologia richiede metodi strutturalmente altamente risolti ed è sensibile alle condizioni biologiche. Le sue strutture meta-stabili dispiegate nativamente rendono l'aSyn monomerico intrattabile a molte tecniche di biologia strutturale. Qui, viene descritta l'applicazione di uno di questi approcci: spettrometria di massa a scambio idrogeno/deuterio (HDX-MS) sulla scala temporale al millisecondo per lo studio di proteine con bassa stabilità termodinamica e deboli fattori di protezione, come aSyn. Alla scala temporale del millisecondo, i dati HDX-MS contengono informazioni sull'accessibilità al solvente e sulla struttura legata all'idrogeno di aSyn, che vengono perse a tempi di etichettatura più lunghi, producendo in definitiva una risoluzione strutturale fino al livello di amminoacidi. Pertanto, HDX-MS può fornire informazioni ad alte risoluzioni strutturali e temporali sulla dinamica conformazionale e la termodinamica, le interazioni intra e intermolecolari e l'impatto strutturale di mutazioni o alterazioni delle condizioni ambientali. Sebbene ampiamente applicabile, è dimostrato come acquisire, analizzare e interpretare misurazioni HDX-MS al millisecondo in aSyn monomerico.
Introduzione
Il morbo di Parkinson (PD) è una malattia neurodegenerativa che colpisce milioni di persone in tutto il mondo1. È caratterizzato dalla formazione di inclusioni citoplasmatiche note come corpi di Lewy e neuriti di Lewy nella regione della substantia nigra pars compacta del cervello. Queste inclusioni citoplasmatiche sono state trovate per contenere aggregati della proteina intrinsecamente disordinata aSyn2. Nel PD e in altre sinucleinopatie, aSyn si trasforma da uno stato disordinato solubile in uno stato malato insolubile e altamente strutturato. Nella sua forma nativa, l'aSyn monomerico adotta una vasta gamma di conformazioni stabilizzate da interazioni elettrostatiche a lungo raggio tra i suoi N- e C-termini e interazioni idrofobiche tra la sua regione C-terminus e la componente beta non amiloide (NAC) 3,4,5,6. Eventuali interruzioni in queste interazioni stabilizzanti, come mutazioni, modifiche post-traduzionali e cambiamenti nell'ambiente locale, possono portare al misfolding del monomero, innescando così il processo di aggregazione7.
Mentre esiste una grande quantità di ricerche sulle forme oligomeriche e fibrillari di aSyn 8,9,10,11, c'è un bisogno cruciale di studiare la forma monomerica della proteina e capire meglio quali conformisti sono funzionali (e come) e quali sono inclini ad aggregare 8,9,10,11 . Essendo intrinsecamente disordinato, solo 14 kDa di dimensioni e difficile da cristallizzare, il monomero aSyn non è suscettibile alla maggior parte delle tecniche biologiche strutturali. Tuttavia, una tecnica in grado di misurare la dinamica conformazionale di aSyn monomerico è millisecondo HDX-MS, che ha recentemente generato importanti osservazioni strutturali che sarebbero difficili o impossibili da ottenere altrimenti 12,13,14. Millisecond HDX-MS misura sensibilmente la media dell'insieme conformazionale proteico monitorando lo scambio isotopico a idrogeno ammidico, indicando l'accessibilità al solvente e la partecipazione alla rete di legame a idrogeno di una particolare regione proteica sulla scala temporale del millisecondo. È necessario sottolineare l'aspetto millisecondo dell'HDX-MS poiché, a causa della sua natura meta-stabile e dispiegata nativamente, aSyn presenta una cinetica di scambio idrogeno molto veloce che si manifesta ben al di sotto del limite inferiore dei sistemi HDX-MS convenzionali. Ad esempio, la maggior parte della molecola aSyn ha completamente scambiato idrogeno per deuterio in condizioni intracellulari in meno di 1 s. Diversi laboratori hanno ora costruito strumentazione a miscelazione rapida; in questo caso, viene utilizzato un prototipo di strumento a flusso di spegnimento a miscelazione rapida in grado di eseguire HDX-MS con un tempo morto di 50 ms e una risoluzione temporale di 1 ms15. Mentre il millisecondo HDX-MS è stato recentemente molto importante nello studio di aSyn, è importante per studiare proteine / regioni intrinsecamente disordinate più ampiamente e un gran numero di proteine con loop / regioni che sono solo debolmente stabili. Ad esempio, farmaci peptidici (ad esempio, insulina; GLP-1/glucagone; tirzepatide) e le proteine di fusione peptidica (ad esempio, l'inibitore dell'HIV FN3-L35-T1144) sono i principali formati farmacologici in cui le informazioni strutturali e di stabilità in fase di soluzione possono essere un input critico per le decisioni di sviluppo del farmaco e, tuttavia, la porzione peptidica è spesso solo debolmente stabile e intrattabile da HDX-MS alla scala temporale dei secondi 16,17,18,19,20 . I metodi emergenti HDX-MS con etichettatura nei domini secondi/minuti hanno dimostrato di ricavare informazioni strutturali per i G-quadruplex del DNA, ma dovrebbe essere possibile estenderlo a strutture oligonucleotidiche più diverse mediante l'applicazione di HDX-MS21 al millisecondo.
Gli esperimenti HDX-MS possono essere eseguiti a tre diversi livelli: (1) bottom-up (per cui la proteina etichettata viene digerita proteoliticamente), (2) middle-down (in cui la proteina etichettata viene digerita proteoliticamente e i peptidi risultanti vengono ulteriormente frammentati da tecniche di soft-fragmentation) e (3) top-down (per cui le tecniche di soft-fragmentation frammentano direttamente la proteina etichettata)22 . Pertanto, i dati sub-molecolari HDX-MS ci consentono di localizzare il comportamento di scambio in regioni specifiche di una proteina, rendendo fondamentale avere un'adeguata copertura della sequenza per tali esperimenti. La risoluzione strutturale di qualsiasi esperimento HDX-MS si basa sul numero di peptidi proteolitici o frammenti derivati dalla proteina durante la digestione o la frammentazione morbida, rispettivamente. In ciascuno dei tre tipi di esperimento sopra descritti, il cambiamento nello scambio ammidico in ogni peptide / frammento è mappato sulla struttura primaria della proteina per indicare il comportamento delle regioni localizzate della proteina. Mentre la massima risoluzione strutturale è raggiunta attraverso la frammentazione morbida, la descrizione di questi esperimenti è fuori dallo scopo del presente studio, che si concentra sulla misurazione delle conformazioni monomeriche aSyn. Risultati eccellenti possono essere ottenuti con il flusso di lavoro "bottom-up" comunemente applicato descritto qui.
Qui vengono fornite procedure su (1) come preparare e gestire campioni aSyn e buffer HDX-MS, (2) come eseguire la mappatura peptidica per un esperimento HDX-MS bottom-up, (3) come acquisire dati HDX-MS su aSyn monomerico in condizioni fisiologiche, in particolare nel dominio del tempo millisecondo (utilizzando uno strumento personalizzato; sono stati descritti anche strumenti alternativi per l'etichettatura dei millisecondi), e (4) come elaborare e analizzare i dati HDX-MS. I metodi che utilizzano aSyn monomerico a pH fisiologico (7.40) in due condizioni di soluzione sono esemplificati qui. Sebbene siano di fondamentale utilità nello studio di aSyn, queste procedure possono essere applicate a qualsiasi proteina e non sono limitate alle proteine intrinsecamente disordinate.
Protocollo
1. Espressione proteica e purificazione di aSyn
- Preparare aSyn seguendo un report pubblicato in precedenza9.
- Dializzare in un tampone di conservazione sicuro (ad esempio, Tris, pH 7,2 ).
- Se necessario, concentrare il campione (ad esempio, tubi microcentrifuga con filtro spin utilizzando 3 kDa MWCO, 14.000 x g per circa 10-30 minuti, vedere Tabella dei materiali).
NOTA: Si consiglia di non concentrarsi eccessivamente. L'integrità dell'insieme monomerico non è stata verificata oltre i 25 μM. - Aliquota e conservazione a -80 °C
NOTA: La proteina monomerica aSyn è stabile fino a 1 anno in queste condizioni di conservazione.
2. Preparazione del buffer HDX
NOTA: poiché Tris ha un coefficiente di temperatura elevato, la misurazione del pH deve essere regolata per la temperatura alla quale verrà eseguita la reazione HDX, che è di 20 °C in questo protocollo.
- Preparare il tampone di equilibrio per lo stato A e lo stato B pesando 0,002 mol di Tris in 100 ml di acqua di grado LC-MS. Per lo stato B, aggiungere 29,8 mg di KCl, 14,2 mg di MgCl2, 36,8 g di CaCl2 e 836 mg di NaCl al tampone Tris. Regolare il pH a 7,40 ± 0,05.
NOTA: il tampone di equilibrio deve contenere le condizioni alle quali aSyn deve essere studiato. In questo caso, si tratta di 20 mM Tris a pH 7,4 +/− sali. - Preparare il tampone di etichettatura per lo stato A e lo stato B pesando 0,002 mol di Tris in 100 ml di acqua deuterata. Per lo Stato B, aggiungere 29,8 mg di KCl, 14,2 mg di MgCl2, 36,8 g di CaCl2 e 836 mg di NaCl al tampone di etichettatura Tris. Il pD del tampone di etichettatura corrisponde al pH del tampone di equilibrio. Poiché pH = pD - 0,41, regolare in modo che il pHmetro legga 6,99 ± 0,0523,24.
NOTA: il buffer di etichettatura deve avere gli stessi componenti del buffer di equilibrio, tranne per il fatto che viene preparato utilizzando acqua deuterata. - Preparare il tampone di tempra pesando 0,010 mol di Tris e 0,050 mol di urea e portare a 100 ml con acqua di grado LC-MS. Regolare il pH a 2,50 ± 0,05 a 0,5 °C.
NOTA: prima degli esperimenti HDX è necessario eseguire una schermata del buffer di tempra per identificare il miglior buffer di spegnimento per la proteina di interesse. Diverse concentrazioni e combinazioni di denaturanti (ad esempio, urea e guanidinio cloridrato) e agenti riducenti (ad esempio, tris (2-carbossietil) fosfina) vengono sottoposti a screening, insieme a parametri fisici come il volume e la temperatura di intrappolamento, per dispiegare e digerire efficacemente la proteina estinta. Un tampone di tempra composto da 100 mM Tris e 0,5 M urea a pH 2,50 è ottimale per il presente studio. - Preparare il tampone di lavaggio della colonna di digestione pesando 0,125 mol di guanidinio cloridrato in una bottiglia di vetro Duran. Aggiungere 25 mL di metanolo e 250 μL di acido formico. Portare a 250 ml con acqua di grado LC-MS.
NOTA: per la colonna di pepsina Enzymate BEH (vedere Tabella dei materiali), utilizzare un tampone di lavaggio a colonna di 0,5 M di guanidinio cloridrato, 10% (v/v) metanolo e 0,1% (v/v) di acido formico. - Preparare il lavaggio debole della siringa pipettando 0,5 μL di acido formico in 249,5 mL di acqua di grado LC-MS.
- Preparare il lavaggio forte della siringa mescolando parti uguali di acqua di grado LC-MS, metanolo, acetonitrile e isopropanolo. Aggiungere acido formico a una concentrazione finale del 2% (v/v).
NOTA: per prevenire la contaminazione incrociata tra i vari tamponi e la proteina e per consentire la pulizia della porta di iniezione, è fondamentale che vengano preparate soluzioni di lavaggio delle siringhe e che il percorso del flusso verso la valvola (spesso chiamato "fodera di lavaggio") sia completamente innescato con liquido. L'acido formico è facoltativo per il lavaggio debole della siringa.
3. Procedura di mappatura dei peptidi
- Preparare l'esempio seguendo il passaggio seguente.
- Il filtro ha scongelato il brodo proteico aSyn dal congelatore a -80 °C con filtri a siringa da 0,22 μm. Misurare l'assorbanza della proteina madre filtrata a 280 nm per determinare la concentrazione secondo la legge di Beer-Lambert. Diluire la proteina ad una concentrazione di 5 μM nel tampone di equilibrio (fase 2.1.).
NOTA: La legge di Beer-Lambert: A = εcl, dove A è assorbanza, ε è il coefficiente di estinzione della proteina alla lunghezza d'onda misurata (280 nm qui) con unità M−1cm−1, c è la concentrazione proteica in M, e l è la lunghezza del percorso in cm. Per aSyn26 wild-type, ε = 5960 M−1cm−1.
- Il filtro ha scongelato il brodo proteico aSyn dal congelatore a -80 °C con filtri a siringa da 0,22 μm. Misurare l'assorbanza della proteina madre filtrata a 280 nm per determinare la concentrazione secondo la legge di Beer-Lambert. Diluire la proteina ad una concentrazione di 5 μM nel tampone di equilibrio (fase 2.1.).
- Impostare il metodo di cromatografia liquida.
- Creare un file di ingresso con un tempo di caricamento/trapping di 3 minuti ad una pressione di 7000-9000 psi, seguito da un gradiente dal 5% di acetonitrile al 40% di acetonitrile in 7 minuti, seguito da ripetute fasi di lavaggio del 5%-95% acetonitrile-acqua per 10 min.
- Assicurarsi che lo spray di blocco (ad esempio, encefalina di leucina, vedere Tabella dei materiali) scorra a 2000 psi e sia collegato alla sonda di spruzzatura di blocco della sorgente dello spettrometro di massa.
- Impostare i metodi di spettrometria di massa MSE .
NOTA: MSE è un metodo di acquisizione indipendente dai dati a banda larga senza isolamento di massa precursore. Pertanto, tutti gli ioni all'interno dell'intervallo m/z selezionato sono ulteriormente frammentati utilizzando la dissociazione indotta da collisione (CID)27.- Nel file di metodo MS, scegli MSE Continuum e imposta un tempo di acquisizione compreso tra 2-10 minuti, sorgente elettrospray e modalità di risoluzione positiva. Acquisisci MSE oltre 50-2000 Da, scansionando ogni 0,3 s.
- Per la funzione 1 (bassa energia), impostare la trappola e trasferire le energie di collisione a 4 V. Per la funzione 2 (alta energia), impostare l'energia di collisione di trasferimento della rampa in modo che sia costante a 4 V e l'energia di collisione della trappola sia come indicato nella Tabella 1 per ciascun livello di energia di mappatura.
NOTA: È possibile utilizzare anche i metodi MSE di mobilità ionica. Metodi di mappatura alternativi (ad esempio, acquisizione dipendente dai dati o DDA) possono essere utilizzati a discrezione dell'utente.
- Impostare il robot autocampionatore (vedere Tabella dei materiali).
- Aggiungere 50 μL della proteina da 5 μM a un flaconcino di recupero totale. Posizionare il flaconcino in una posizione di campionamento nella camera destra HDX. Assicurarsi che questa camera sia a 0,5 °C.
- Aggiungere un flaconcino di reagente del tampone di equilibrio e due flaconcini di reagente del tampone di etichettatura alle posizioni del reagente uno, due e tre nella camera sinistra HDX. Assicurarsi che questa camera sia a 20 °C impostando il regolatore di temperatura Peltier (vedere Tabella dei materiali). Aggiungere un flaconcino di tampone di tempra alla posizione del reagente uno nella camera destra HDX.
- Aggiungere otto flaconcini di recupero totale nelle posizioni di reazione della camera sinistra HDX e otto flaconcini di recupero massimo nelle posizioni di reazione della camera destra HDX.
NOTA: Per garantire la completa pulizia e la massima riproducibilità dei volumi erogati, si consiglia di eseguire un lavaggio a siringa e un lavaggio primario sulle siringhe autocampionatore. Ad esempio, eseguire una sequenza di (1) lavaggio debole, (2) lavaggio forte, (3) lavaggio debole prima di iniziare gli esperimenti di mappatura. Questa sequenza può essere ripetuta ampiamente e si consiglia di farlo fino a 20 volte o fino a quando la siringa non è completamente bagnata. - Impostare un elenco di esempio con i metodi LC e MS appropriati nel software di pianificazione e avviare la pianificazione.
NOTA: Per il presente studio, Chronos viene utilizzato come software di pianificazione (vedere Tabella dei materiali).
- Elaborare i dati di mappatura.
- Identificare i peptidi dai file dell'esperimento di mappatura utilizzando un software appropriato (vedere Tabella dei materiali).
- Importare i dati di identificazione dei peptidi in DynamX (vedere Tabella dei materiali) utilizzando i seguenti parametri di soglia peptidica: intensità minima = 5000, lunghezza minima della sequenza = 0, lunghezza massima della sequenza = 40, prodotti minimi = 1, prodotti minimi per amminoacido = 0,25, prodotti consecutivi minimi = 2, intensità minima della somma per i prodotti = 0, punteggio minimo = 0 e errore MH+ massimo (ppm) = 0.
NOTA: in alternativa, derivare le impostazioni ottimizzate in base a un flusso di lavoro consigliato28. - Seleziona Dati dal menu e fai clic su Importa risultati PLGS. Fare clic su Aggiungi per scegliere i file di dati pertinenti per l'assegnazione spettrale. Quando tutti sono stati aggiunti, fare clic su Avanti e inserire le impostazioni del filtro sopra. Quindi, fare clic su Fine.
- Curare manualmente le assegnazioni isotopiche per ottenere la mappa finale di copertura peptidica di aSyn in DynamX.
4. Studio di scambio idrogeno/deuterio al millisecondo
- Pulire lo strumento prototipo FastHDX (vedere Tabella dei materiali) prima di iniziare gli esperimenti HDX.
- Aprire la GUI del software HDX compatibile e consentire l'inizializzazione del sistema.
- Immettere la temperatura della camera campione a 20 °C e la camera di spegnimento a 0,5 °C. Fare clic su Imposta per applicare le nuove temperature.
- Installare i tubi della centrifuga con acqua di grado LC-MS in tutte le prese.
- Nella scheda Titrator Plumbing Delivery , controllare sia le siringhe sinistra che quelle destra e fare clic su Prime per rimuovere eventuali bolle d'aria latenti nei tubi. Ripeti fino a quando tutte le bolle sono sparite.
- Nella scheda Macro , selezionare tutte le caselle relative alle siringhe. Fare clic su Calibra posizione iniziale delle siringhe. Fare clic su Wash Syringe Load Loop. Fare clic su Lava tutti i volumi del ciclo di miscelazione.
- Ripetere il passaggio 4.1.5. 1x di più.
- Se nelle siringhe tampone compaiono bolle, degassare scollegando la siringa ed espellendo la bolla verticalmente. Sostituire la siringa e ricalibrare in posizione zero.
- Configurare lo strumento prototipo FastHDX per esperimenti HDX.
- Aggiungere 500 uL di aSyn filtrato da 5 μM in un flaconcino di recupero totale e posizionare all'interno di un frigorifero da tavolo per evitare oligomerizzazione e aggregazione indotte dalla temperatura.
- Aggiungere 50 ml di tamponi di equilibrio, etichettatura e tempra a ciascun buffer (2. Fasi di preparazione del buffer HDX 1-3) ingresso nelle camere sinistra e destra.
- Aggiungere 50 mL di tampone di lavaggio della colonna (2. Fase di preparazione del tampone HDX 4) all'ingresso di lavaggio della pepsina.
- Per innescare le linee di lavaggio delle proteine e delle colonne 1x, controllare le siringhe sinistra e destra nella scheda Consegna idraulica del titolatore e fare clic su Prime una volta.
NOTA: Qualsiasi clic successivo su Prime causerà ulteriori ripetizioni del processo primo, con conseguente consumo di grandi quantità di campione proteico. Si consiglia di evitare questo, anche quando c'è un ritardo nel software dopo che è stato tentato un clic sul pulsante. - Ripetere i passaggi 4.1.5. 1x.
- Nella scheda Flusso di spegnimento manuale , immettere le impostazioni richieste, spiegate nei passaggi 4.2.7.-4.2.10.
- Per un esperimento di percorso temporale, immettere i tempi in millisecondi utilizzando il pulsante Punti simbolici . Se sono necessarie repliche, aggiungere lo stesso punto temporale più volte, ad esempio, per un triplice di 50 ms, è necessario inserire 50 50 50. L'elenco di esempio nel software dello spettrometro di massa (MassLynx è usato qui, vedi Tabella dei materiali) deve corrispondere esattamente a quei punti temporali.
NOTA: i nomi dei file dell'elenco dei campioni dello spettrometro di massa e/o il testo di esempio devono essere utilizzati per garantire una registrazione permanente dei tempi di etichettatura HDX corrispondenti a quelli immessi nella GUI del software FastHDX. I tempi di etichettatura per ogni esecuzione del campione non verranno memorizzati da nessun'altra parte. - Impostare Tempo trappola (min) come lunghezza del tempo di abbondanza. Qui, sono le 3.00.
- Impostare Wait for HPLC (mins) = (tempo di trappola + tempo di esecuzione + 1,5 min).
NOTA: ad esempio, per un esperimento con 3 min di trapping e 17 min di gradiente, questo sarà di 21,50 min. - Fare clic sulla casella Esegui vuoto solo se si eseguono esperimenti vuoti tra le esecuzioni di esempio. In caso affermativo, assicurarsi una voce (ad esempio, una riga valida nell'elenco di esempio) nel software per l'esecuzione vuota dopo ogni esecuzione di esempio.
- Una volta che l'elenco di esempio è pronto sul software, evidenziare le voci appropriate e avviare l'esecuzione nel software facendo clic sul pulsante Riproduci e FastHDX nel software.
NOTA: a causa dello scrambling di idrogeno/ deuterio, i metodi solo MS o le tecniche di frammentazione morbida (dissociazione a trasferimento di elettroni, dissociazione a cattura elettronica e fotodissociazione ultravioletta) possono essere utilizzati solo29. Per aSyn a un pH fisiologico di 7,40, i punti temporali che vanno da 50 ms a 300 s sono più applicabili in quanto coprono l'intera curva di assorbimento del deuterio8.
5. Trattamento dei dati
- Caricare il file dei peptidi assegnati spettralmente dagli esperimenti di mappatura dei peptidi. Aprire il menu File e fare clic su Apri nel software DynamX (vedere Tabella dei materiali).
- Importare i file raw nel software di misurazione della massa preferito (ad esempio, DynamX, HDExaminer, ecc.). Apri il menu "Dati" e fai clic su MS Files. Fare clic su Nuovo stato per creare stati per ogni condizione proteica studiata.
- Fare clic su Nuova esposizione per aggiungere ogni timepoint HDX. Fare clic su Nuovo RAW per importare i file .raw. Trascinare ogni file .raw nella posizione corretta. Fare clic su OK al termine.
- Assegna automaticamente prima gli isotopi (questo è automatico dopo il passaggio 5.2. sopra), quindi cura manualmente l'assegnazione isotopica per garantire un'elevata qualità dei dati.
- Esportare i dati del cluster in un file .csv con colonne nel seguente ordine: nome della proteina, numero di inizio della sequenza, numero di fine sequenza, sequenza, modifica, frammento, assorbimento massimo possibile, massa delle specie monoisotopiche, nome dello stato, tempo di esposizione, nome del file, carica, tempo di ritenzione, intensità e centroide.
- Aprire il menu Dati nel software di misurazione di massa e fare clic su Esporta dati cluster.
6. Analisi dei dati
- Caricare i dati del cluster esportati nel software di analisi HDX preferito. Qui, HDfleX viene utilizzato30 (vedi Tabella dei materiali).
- Adatta i dati sperimentali per tutti i peptidi e gli stati, selezionando i metodi di correzione dello scambio posteriore appropriati per ottenere costanti di velocità osservate per la reazione HDX.
- Calcolare una soglia di significatività globale con il metodo preferito (HDfleX supporta diverse opzioni per questo) ed eseguire test di significatività ibrida per determinare le differenze significative tra gli stati rispetto a31,32.
NOTA: se la differenza osservata è maggiore della soglia globale e il valore p è inferiore al livello di confidenza scelto (ad esempio, 95%), la differenza è considerata significativa.
Risultati
A causa della sua natura intrinsecamente disordinata, è difficile catturare gli intricati cambiamenti strutturali in aSyn a pH fisiologico. HDX-MS monitora lo scambio isotopico a idrogeno ammidico della spina dorsale, sondando le dinamiche e le interazioni conformazionali delle proteine. È una delle poche tecniche per acquisire queste informazioni ad alte risoluzioni strutturali e temporali. Questo protocollo è ampiamente applicabile a una vasta gamma di proteine e condizioni tampone, e questo è esemplificato dalla misurazione della cinetica di scambio di aSyn in due diverse condizioni di soluzione: Stato A e Stato B8, come definito nei passaggi 2.1.-2.2.
In primo luogo, è stato eseguito un esperimento di mappatura su aSyn ed è stata ottenuta una mappa di copertura peptidica, come mostrato nella Figura 1. La mappa copre il 100% della sequenza proteica e ha una ridondanza media di 3,79. Il valore di copertura del 100% indica che tutti gli amminoacidi nella proteina sono stati trovati nei digest proteici e consentirà un'analisi completa del comportamento di scambio di aSyn. Il valore di ridondanza indica il numero di peptidi sovrapposti. Un valore di ridondanza più elevato aumenta la risoluzione strutturale della mappa finale, dato l'appiattimento sottrattivo dei dati per i peptidi32 sovrapposti.
Utilizzando il prototipo dello strumento a flusso di spegnimento a miscelazione rapida (vedere Tabella dei materiali), sono stati raccolti dati HDX-MS di alta qualità su scala temporale di millisecondi su aSyn a pH 7,4 nello stato A e nello stato B (Figura 2). A seguito di un'assegnazione isotopica in DynamX, sono state ottenute curve di assorbimento del deuterio "grezze", come mostrato nella Figura 3A. Mostra curve di assorbimento per tre peptidi selezionati in ciascun dominio proteico. Viene visualizzata l'incorporazione del deuterio nel tempo. L'asse x è sulla scala temporale del millisecondo, che si allinea con la cinetica molto veloce di aSyn in condizioni fisiologiche. La regione ombreggiata rossa mostra i dati tipicamente ottenuti dagli strumenti HDX convenzionali, con misurazioni iniziali da 30 s. È importante sottolineare che questo non può essere ulteriormente ridotto dalla manipolazione del pH per la cosiddetta "espansione della finestra temporale"; tale approccio non è valido per lo studio di proteine/regioni intrinsecamente disordinate, in quanto lo spostamento del pH perturberà l'insieme conformazionale del polipeptide debolmente stabile. Come si può vedere qui, la maggior parte di aSyn è completamente scambiata da 1 s (Figura 3C). Ciò dimostra l'importanza delle misurazioni HDX al millisecondo per aSyn monomerico quando viene catturata la curva di assorbimento cinetica completa per la reazione di scambio, che produce la misurazione più accurata delle conformazioni monomeriche.
HDfleX ha eseguito la correzione del back-exchange utilizzando l'incorporazione del deuterio plateau. I punti dati sono stati successivamente montati secondo l'equazione 1, fornendo una costante di velocità osservata, kobs, indicativa dell'accessibilità al solvente e del coinvolgimento del legame idrogeno di quel particolare peptide (Figura 3B).
 Equazione 1
Equazione 1
dove Dt è l'incorporazione di deuterio al tempo t, nExp è il numero di fasi esponenziali, N è il numero massimo di idrogeni labili, kobs è la costante del tasso di cambio osservata e β è un fattore di allungamento30,33.
Dopo il montaggio della curva, l'area di assorbimento sotto la curva montata può essere calcolata integrando la funzione adattata che descrive la curva di assorbimento all'interno della finestra temporale sperimentale. Sono state eseguite analisi di significatività statistica tra l'area di assorbimento dei due stati. In primo luogo, una soglia di significatività globale per l'area di assorbimento è stata calcolata in HDfleX con un livello di confidenza del 95%. Sono stati quindi generati grafici di differenza dell'area di assorbimento, che mostrano la differenza tra lo stato A e lo stato B a due livelli di risoluzione strutturale: risoluzione peptidica (Figura 4A) e risoluzione degli amminoacidi (Figura 4B). Il grafico della differenza di risoluzione peptidica mostra la differenza nell'area di assorbimento tra lo Stato A e lo Stato B per ogni singolo peptide, mentre il grafico della differenza di risoluzione degli amminoacidi mostra la differenza nell'area di assorbimento tra lo Stato A e lo Stato B appiattita attraverso l'intera sequenza di amminoacidi di aSyn30,34. Entrambi i grafici indicano un maggiore assorbimento complessivo di deuterio in tutto il monomero aSyn nello Stato B rispetto allo Stato A. Questo risultato può essere giustificato esaminando i grafici di assorbimento del deuterio nella Figura 3, dove la curva di assorbimento dello Stato B è sempre al di sopra della curva di assorbimento dello Stato A. Inoltre, si può vedere che l'entità della differenza dell'area di assorbimento è molto più alta al C-terminus. Ancora una volta, questo può essere giustificato risalendo alle curve di assorbimento originali, dove i peptidi C-terminali (peptidi 124-140 mostrati in Figura 3) mostrano un divario molto più grande tra le curve di assorbimento rispetto al resto della proteina. In conclusione, le condizioni di soluzione nello stato B causano un aumento dell'esposizione al solvente o una diminuzione della partecipazione alla rete di legame idrogeno in tutta la proteina, ma soprattutto al C-terminus.

Figura 1: Mappa della copertura peptidica di aSyn wild-type con un totale di 30 peptidi e copertura di sequenza al 100%. I tre domini di aSyn sono evidenziati come segue: N-terminus (blu), regione della componente beta non amiloide (giallo) e C-terminus (rosso). Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Flusso di lavoro per un esperimento HDX-MS al millisecondo su aSyn. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Esempi di grafici di assorbimento da tre peptidi selezionati nei tre domini di aSyn per lo stato A (giallo) e lo stato B (blu). (A) Grafico di assorbimento corretto non adattato e non back-exchange. (B) Piazzole di assorbimento corrette montate e di scambio posteriore. La regione ombreggiata rossa rappresenta i dati ottenibili dai sistemi HDX-MS convenzionali, in genere a partire da 30 s. Le barre di errore corrispondono alla deviazione standard delle tre repliche. (C) Grafico della mappa di calore della percentuale di assorbimento del deuterio attraverso la sequenza di amminoacidi per punto temporale. La barra dei colori rappresenta la percentuale di assorbimento del deuterio. Fare clic qui per visualizzare una versione più grande di questa figura.
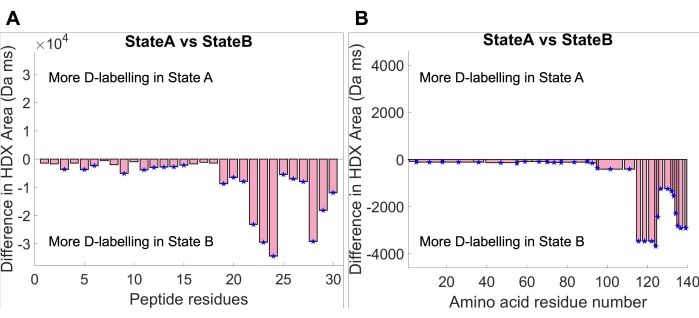
Figura 4: Grafici di differenza dell'area di assorbimento al tempo massimo di plateau della curva (14.084 ms). (A) I grafici di risoluzione dei residui peptidici mostrano la differenza dell'area di assorbimento di ciascun peptide tra lo Stato A e lo Stato B. (B) Grafico della risoluzione degli amminoacidi che mostra la differenza dell'area di assorbimento tra lo Stato A e lo Stato B appiattita attraverso la sequenza di amminoacidi. Fare clic qui per visualizzare una versione più grande di questa figura.
| Mappatura del livello di energia | Tensione di rampa (V) |
| Basso | 20-40 |
| Medio | 25-45 |
| Alto | 30-50 |
| Molto alto | 35-55 |
Tabella 1: Mappatura dei livelli di energia e delle corrispondenti tensioni di rampa di trasferimento.
Discussione
Nel presente articolo vengono descritte le seguenti procedure: (1) esecuzione di esperimenti di mappatura peptidica su aSyn monomerico per ottenere la massima copertura di sequenza, (2) acquisizione di dati HDX-MS millisecondi su aSyn monomerico in condizioni fisiologiche e (3) esecuzione di analisi dei dati e interpretazione dei dati HDX-MS risultanti. Le procedure fornite sono generalmente semplici da eseguire, ogni esperimento di etichettatura dura in genere solo circa 8 ore per tre repliche e otto punti temporali e l'esperimento di mappatura dura solo circa 2 ore. Data la strumentazione completamente automatizzata utilizzata qui, è possibile acquisire un set di dati completo in 1 giorno. Tuttavia, quando si maneggiano campioni e si preparano tamponi, è necessario prestare attenzione per garantire che le misurazioni derivino da proteine (o regioni proteiche) debolmente stabili nello stato desiderato. È importante sottolineare che uno studio precedente ha dimostrato che diverse condizioni di conservazione, come il congelamento e la liofilizzazione, hanno portato a diversi conformatori monomeri aSyn e che è importante caratterizzare il potenziale impatto della gestione del campione sull'insieme conformazionale monomero aSyn10. In effetti, HDX-MS è una misura altamente sensibile di tali perturbazioni conformazionali, con un intervallo dinamico da microsecondi ad almeno mesi. Inoltre, se si studia rigorosamente solo il monomero aSyn, si consiglia vivamente di rimuovere oligomeri e fibrille indesiderati che potrebbero essersi formati nel campione al momento della conservazione o della manipolazione. Inoltre, i buffer HDX-MS devono essere strettamente controllati entro 0,05 del pH o pD desiderato, poiché eventuali discrepanze influenzeranno in modo significativo il tasso di scambio intrinseco e porteranno a errori indesiderati. È anche importante notare che i confronti tra le condizioni di soluzione per qualsiasi proteina che differisce per pH, temperatura o composizione salina altereranno la velocità intrinseca. Pertanto, questi dati richiederanno ulteriori correzioni, come l'applicazione di un fattore di regolazione del pH35 o di un fattore di correzione empirico 8,30.
In termini di strumentazione, non esistono sistemi disponibili in commercio che consentano l'acquisizione di dati HDX-MS al millisecondo. Diversi gruppi di ricerca hanno sviluppato i propri sistemi, dai sistemi di flusso di spegnimento 13,15,36 ai chip microfluidici 37,38,39 per catturare la cinetica di scambio rapido di alcune proteine. Un altro metodo che è stato utilizzato per ottenere dati HDX-MS su scala temporale di millisecondi è noto come metodo di espansione del tempo 40,41, in cui il pH dei tamponi viene ridotto per rallentare la cinetica di scambio. Tuttavia, questo metodo non si applica ad aSyn (o a qualsiasi caratteristica proteica debolmente stabile) in quanto (1) l'abbassamento del pH modifica drasticamente la densità di carica della proteina e aumenta il tasso di aggregazione 8,42 e (2) i conformisti aSyn sono solo meta-stabili e sono suscettibili di essere perturbati da queste alterazioni del pH. Per questi motivi, si raccomanda di mantenere un pH costante nei tamponi HDX-MS quando si studiano le conformazioni monomeriche aSyn, a meno che non siano fisiologicamente rilevanti, e di utilizzare uno strumento di etichettatura al millisecondo.
La maggior parte del monomero aSyn scambia completamente entro 1 s e, al massimo, ci vogliono circa 15 s per scambiare completamente con il deuterio (Figura 3) ad un pH fisiologicamente rilevante di 7,4 (che riflette le condizioni citosoliche intracellulari alla presinapsi). Utilizzando i sistemi HDX-MS convenzionali, partire da 30 s non è appropriato in quanto i dati HDX-MS corrisponderebbero al plateau della reazione di scambio, che non fornisce alcuna informazione conformazionale utile. Tuttavia, il limite inferiore di misura dello strumento HDX al millisecondo (corrispondente a un "tempo morto" di 50 ms) consente il monitoraggio della reazione di scambio dal completamento di circa il 25% per il monomero aSyn a pH 7,4. Questo ci ha permesso di catturare la maggior parte della curva di assorbimento cinetico. L'adattamento della curva di assorbimento del deuterio all'equazione 1 fornisce importanti informazioni cinetiche; corrisponde ad una stima della costante di velocità osservata, kobs. Sebbene non sia trattato qui, è possibile effettuare esperimenti di cinetica di aggregazione ed esaminare le morfologie delle fibrille di aSyn nelle stesse condizioni di soluzione degli esperimenti HDX-MS poiché HDX-MS è altamente tollerante di una vasta gamma di buffer8. Così, ad esempio, i kobs dell'esperimento HDX-MS possono essere correlati con i risultati degli esperimenti di aggregazione per ottenere informazioni su quali conformazioni sono più inclini a determinati comportamenti di aggregazione e morfologie di fibrille.
Per il caso semplice di esperimenti differenziali HDX-MS, in cui devono essere confrontate due o più condizioni o varianti proteiche, l'area sotto la curva di assorbimento adattata può essere integrata per ciascuno stato e confrontata tra loro. In questo studio, le aree di assorbimento per lo Stato A e lo Stato B sono state confrontate a due diversi livelli di risoluzione strutturale: risoluzione peptidica e risoluzione degli amminoacidi, entrambi con punti di forza e sfide distinti. Ad esempio, i dati sulla risoluzione peptidica riflettono più da vicino i dati spettrali grezzi e hanno subito il minimo processo. Tuttavia, i dati di risoluzione degli amminoacidi "appiattiti" consentono di combinare sia le informazioni peptidiche che quelle di frammentazione morbida in un singolo output piuttosto che separare le uscite non fondenti e, in definitiva, presentare i dati alla massima risoluzione strutturale. Una limitazione del rilevamento della spettrometria di massa dell'etichettatura HDX è la sfida di ottenere la risoluzione degli amminoacidi. Mentre le tecniche di "soft-fragmentation", come la dissociazione a trasferimento di elettroni (ETD), la dissociazione a cattura elettronica (ECD) e la fotodissociazione ultravioletta (UVPD), si sono dimostrate efficaci nel generare risoluzioni più elevate, rimangono impegnative, imprevedibili e inefficienti 30,43,44,45,46,47,48.
Rispetto ad altre tecniche strutturali, il millisecondo HDX-MS ha il vantaggio unico di catturare la dinamica conformazionale di aSyn monomerico ad alte risoluzioni strutturali e temporali. Poiché la cinetica a scambio rapido del monomero non è più un fattore limitante, ulteriori studi possono essere eseguiti su aSyn monomerico con diverse mutazioni, modificazioni post-traduzionali, componenti e concentrazioni saline e partner leganti a pH fisiologico. Correlare i risultati hdx-MS con studi funzionali, come la cinetica di aggregazione e le morfologie delle fibrille, può fornire informazioni sui conformisti che promuovono la normale funzione cellulare o sono soggetti a malattie. In definitiva, si prevede che tale HDX-MS al millisecondo possa essere cruciale per scoprire farmaci mirati che stabilizzano specifici conformisti fisiologicamente tollerati.
Divulgazioni
Gli autori non dichiarano interessi concorrenti.
Riconoscimenti
NS è finanziato dalla borsa di studio Diamond Jubilee del Consiglio universitario. JJP è supportato da una UKRI Future Leaders Fellowship [Numero di sovvenzione: MR/T02223X/1].
Materiali
| Name | Company | Catalog Number | Comments |
| 1 × 100 mm ACQUITY BEH 1.7 μm C18 column | Waters Corporation | 186002346 | Analytical column |
| Acetonitrile HPLC grade >99.9% HiPerSolv | VWR | 20060.420 | For LC mobile phases |
| CaCl2 | Sigma Aldrich | C5670 | Salt for HDX buffers |
| Chronos | Axel Semrau (Purchased from Waters Corporation) | 667006090 | Scheduling software to enable multiple HDX-MS sample injections automatically. Alternative software is available from other vendors e.g. HDXDirector or LEAP Shell |
| Deuterium chloride | Goss Scientific (Cambridge Isotope Laboratories) | DLM-2-50 | For HDX labelling buffers |
| Deuterium oxide (99.9% D2O) | Goss Scientific (Cambridge Isotope Laboratories) | DLM-4 | Deuterated water |
| DynamX 3.0 | Waters Corporation | 176016027 | Isotopic assignment and deuterium incorporation calculation |
| Enzymate BEH Pepsin Column | Waters Corporation | 186007233 | Pepsin digestion column |
| Formic Acid, 99.0% LC/MS Grade | Fisher Scientific | 10596814 | For LC mobile phases |
| Guanidinium hydrochloride | Sigma Aldrich | RDD001-500G | Chaotrope/Denaturant |
| HDfleX | University of Exeter | N/A | https://ore.exeter.ac.uk/repository/handle/10871/127982 |
| KCl | Sigma Aldrich | P3911 | Salt for HDX buffers |
| LEAP HDX-2 CTC PAL sampling robot | Waters Corporation | 725000637 | Autosampler robot |
| Leucine enkephalin | Waters Corporation | 186006013 | For mass spectrometry lockspray calibration. |
| MassLynx | Waters Corporation | 667004007 | Software controlling inlet methods and mass spectrometer |
| Maximum recovery vials | Waters Corporation | 600000670CV | 100 pack including caps - used for quench tray in LEAP HDX-2 |
| MgCl2 | Sigma Aldrich | M8266 | Salt for HDX buffers |
| Millipore 0.22 µm syringe filters | Millipore | N9CA7069B | Syringe filters |
| ms2min | Applied Photophysics Ltd | N/A | fast-mix quench-flow millisecond hdx instrument |
| NaCl | Sigma Aldrich | S9888 | Salt for HDX buffers |
| Peltier temperature controller | LEAP Technologies Inc. | HP115-COOL/D | Peltier controller to set precise temperature of chambers in the LEAP robot. |
| ProteinLynx Global Server 3.0 | Waters Corporation | 715001030 | Peptide identification software. Alternative software is available from other vendors. |
| Reagent pot caps | Waters Corporation | 186004632 | 100 pack |
| Reagent pots for LEAP HDX-2 | Waters Corporation | 186001420 | 100 pack excluding caps - used for buffers in LEAP HDX-2 |
| Sodium deuteroxide (99.5% in D2O) | Goss Scientific (Cambridge Isotope Laboratories) | DLM-57 | For HDX labelling buffers |
| Spin filter microcentrifuge tubes (3 kDa MWCO) | Amicon (Merck Sigma Aldrich) | UFC5003 | Micro centrifuge tubes to concentrate protein. This facilitates buffer exchange and accurate sample loading for HDX-MS experiments. |
| Synapt G2-Si mass spectrometer | Waters Corporation | 176850035 | Mass spectrometer |
| Total recovery vials | Waters Corporation | 600000671CV | 100 pack including caps - used for labelling tray in LEAP HDX-2 |
| Tris-HCl | Sigma Aldrich | T3253-250G | Buffer |
| Trizma base | Sigma Aldrich | T60040-B2005 | Buffer |
| Urea | Sigma Aldrich | U5378-1KG | Chaotrope/Denaturant |
| VanGuard 2.1 x 5 mm ACQUITY BEH C18 column | Waters Corporation | 186004623 | Trap desalting column |
Riferimenti
- Dorsey, E. R., et al. regional, and national burden of Parkinson's disease, 1990-2016: A systematic analysis for the Global Burden of Disease Study 2016. The Lancet Neurology. 17 (11), 939-953 (2018).
- Breydo, L., Wu, J. W., Uversky, V. N. α-Synuclein misfolding and Parkinson's disease. Biochimica et Biophysica Acta (BBA): Molecular Basis of Disease. 1822 (2), 261-285 (2012).
- Dedmon, M. M., Lindorff-Larsen, K., Christodoulou, J., Vendruscolo, M., Dobson, C. M. Mapping long-range interactions in α-synuclein using spin-label NMR and ensemble molecular dynamics simulations. Journal of the American Chemical Society. 127 (2), 476-477 (2005).
- Esteban-Martín, S., Silvestre-Ryan, J., Bertoncini, C. W., Salvatella, X. Identification of fibril-like tertiary contacts in soluble monomeric α-synuclein. Biophysical Journal. 105 (5), 1192-1198 (2013).
- McClendon, S., Rospigliosi, C. C., Eliezer, D. Charge neutralization and collapse of the C-terminal tail of alpha-synuclein at low pH. Protein Science. 18 (7), 1531-1540 (2009).
- Ranjan, P., Kumar, A. Perturbation in long-range contacts modulates the kinetics of amyloid formation in α-synuclein familial mutants. ACS Chemical Neuroscience. 8 (10), 2235-2246 (2017).
- Villar-Piqué, A., da Fonseca, T. L., Outeiro, T. F. Structure, function and toxicity of alpha-synuclein: the Bermuda triangle in synucleinopathies. Journal of Neurochemistry. 139, Suppl 1 240-255 (2015).
- Seetaloo, N., Zacharopoulou, M., Stephens, A. D., Schierle, G. S. K., Phillips, J. J. Local structural dynamics of alpha-synuclein correlate with aggregation in different physiological conditions. bioRxiv. , (2022).
- Stephens, A. D., et al. Extent of N-terminus exposure of monomeric alpha-synuclein determines its aggregation propensity. Nature Communications. 11 (1), 2820(2020).
- Stephens, A. D., et al. Different structural conformers of monomeric α-synuclein identified after lyophilizing and freezing. Analytical Chemistry. 90 (11), 6975-6983 (2018).
- Lautenschläger, J., et al. C-terminal calcium binding of α-synuclein modulates synaptic vesicle interaction. Nature Communications. 9 (1), 712(2018).
- Oganesyan, I., Lento, C., Tandon, A., Wilson, D. J. Conformational dynamics of α-synuclein during the interaction with phospholipid nanodiscs by millisecond hydrogen-deuterium exchange mass spectrometry. Journal of the American Society for Mass Spectrometry. 32 (5), 1169-1179 (2021).
- Keppel, T. R., Weis, D. D. Analysis of disordered proteins using a simple apparatus for millisecond quench-flow H/D exchange. Analytical Chemistry. 85 (10), 5161-5168 (2013).
- Al-Naqshabandi, M. A., Weis, D. D. Quantifying protection in disordered proteins using millisecond hydrogen exchange-mass spectrometry and peptic reference peptides. Biochemistry. 56 (31), 4064-4072 (2017).
- Kish, M., et al. Allosteric regulation of glycogen phosphorylase solution phase structural dynamics at high spatial resolution. bioRxiv. , (2019).
- El-Amine, M., et al. Mechanisms of tolerance induction by a gene-transferred peptide-IgG fusion protein expressed in B lineage cells. Journal of Immunology. 165 (10), 5631-5636 (2000).
- Kishimoto, S., et al. Site-specific chemical conjugation of antibodies by using affinity peptide for the development of therapeutic antibody format. Bioconjugate Chemistry. 30 (3), 698-702 (2019).
- Xu, W., et al. A protein-based, long-acting HIV-1 fusion inhibitor with an improved pharmacokinetic profile. Pharmaceuticals. 15 (4), 424(2022).
- Frías, J. P., et al. Tirzepatide versus semaglutide once weekly in patients with type 2 diabetes. The New England Journal of Medicine. 385 (6), 503-515 (2021).
- Gerstein, H. C., et al. Cardiovascular and renal outcomes with efpeglenatide in type 2 diabetes. The New England Journal of Medicine. 385 (10), 896-907 (2021).
- Largy, E., Gabelica, V. Native hydrogen/deuterium exchange mass spectrometry of structured DNA oligonucleotides. Analytical Chemistry. 92 (6), 4402-4410 (2020).
- Marcsisin, S. R., Engen, J. R. Hydrogen exchange mass spectrometry: What is it and what can it tell us. Analytical and Bioanalytical Chemistry. 397 (3), 967-972 (2010).
- Glasoe, P. K., Long, F. A. Use of glass electrodes to measure acidities in deuterium oxide. Journal of Physical Chemistry. 64 (1), 188-190 (1960).
- Krȩzel, A., Bal, W. A formula for correlating pKa values determined in D2O and H2O. Journal of Inorganic Biochemistry. 98 (1), 161-166 (2004).
- Mayerhöfer, T. G., Pahlow, S., Popp, J. The Bouguer-Beer-Lambert law: Shining light on the obscure. ChemPhysChem. 21 (18), 2029-2046 (2020).
- Gasteiger, E., et al. The Proteomics Protocols Handbook. , Springer. New York. 571-607 (2005).
- Bateman, R. H., et al. A novel precursor ion discovery method on a hybrid quadrupole orthogonal acceleration time-of-flight (Q-TOF) mass spectrometer for studying protein phosphorylation. Journal of the American Society for Mass Spectrometry. 13 (7), 792-803 (2002).
- Sørensen, L., Salbo, R. Optimized workflow for selecting peptides for HDX-MS data analyses. Journal of the American Society for Mass Spectrometry. 29 (11), 2278-2281 (2018).
- Demmers, J. A. A., Rijkers, D. T. S., Haverkamp, J., Killian, J. A., Heck, A. J. R. Factors affecting gas-phase deuterium scrambling in peptide ions and their implications for protein structure determination. Journal of the American Chemical Society. 124 (37), 11191-11198 (2002).
- Seetaloo, N., Kish, M., Phillips, J. J. HDfleX: Software for flexible high structural resolution of hydrogen/deuterium-exchange mass spectrometry data. Analytical Chemistry. 94 (11), 4557-4564 (2022).
- Hageman, T. S., Weis, D. D. Reliable identification of significant differences in differential hydrogen exchange-mass spectrometry measurements using a hybrid significance testing approach. Analytical Chemistry. 91 (13), 8008-8016 (2019).
- Hageman, T. S., Weis, D. D. A structural variant approach for establishing a detection limit in differential hydrogen exchange-mass spectrometry measurements. Analytical Chemistry. 91 (13), 8017-8024 (2019).
- Chetty, P. S., et al. Helical structure and stability in human apolipoprotein A-I by hydrogen exchange and mass spectrometry. Proceedings of the National Academy of Sciences of the United States of America. 106 (45), 19005-19010 (2009).
- Keppel, T. R., Weis, D. D. Mapping residual structure in intrinsically disordered proteins at residue resolution using millisecond hydrogen/deuterium exchange and residue averaging. Journal of the American Society for Mass Spectrometry. 26 (4), 547-554 (2015).
- Li, J., Rodnin, M. V., Ladokhin, A. S., Gross, M. L. Hydrogen-deuterium exchange and mass spectrometry reveal the pH-dependent conformational changes of diphtheria toxin T domain. Biochemistry. 53 (43), 6849-6856 (2014).
- Roder, H., Elöve, G. A., Englander, S. W. Structural characterization of folding intermediates in cytochrome c by H-exchange labelling and proton NMR. Nature. 335 (6192), 700-704 (1988).
- Rob, T., et al. Measuring dynamics in weakly structured regions of proteins using microfluidics-enabled subsecond H/D exchange mass spectrometry. Analytical Chemistry. 84 (8), 3771-3779 (2012).
- Rob, T., Gill, P. K., Golemi-Kotra, D., Wilson, D. J. An electrospray ms-coupled microfluidic device for sub-second hydrogen/deuterium exchange pulse-labelling reveals allosteric effects in enzyme inhibition. Lab on a Chip. 13 (13), 2528-2532 (2013).
- Svejdal, R. R., Dickinson, E. R., Sticker, D., Kutter, J. P., Rand, K. D. Thiol-ene microfluidic chip for performing hydrogen/deuterium exchange of proteins at subsecond time scales. Analytical Chemistry. 91 (2), 1309-1317 (2018).
- Goswami, D., et al. Time window expansion for HDX analysis of an intrinsically disordered protein. Journal of The American Society for Mass Spectrometry. 24 (10), 1584-1592 (2013).
- Coales, S. J., E, S. Y., Lee, J. E., Ma, A., Morrow, J. A., Hamuro, Y. Expansion of time window for mass spectrometric measurement of amide hydrogen/deuterium exchange reactions. Rapid Communications in Mass Spectrometry. 24 (24), 3585-3592 (2010).
- Hoyer, W., et al. Dependence of alpha-synuclein aggregate morphology on solution conditions. Journal of Molecular Biology. 322 (2), 383-393 (2002).
- Rand, K. D., Pringle, S. D., Morris, M., Engen, J. R., Brown, J. M. ETD in a traveling wave ion guide at tuned Z-spray ion source conditions allows for site-specific hydrogen/deuterium exchange measurements. Journal of the American Society for Mass Spectrometry. 22 (10), 1784-1793 (2011).
- Kan, Z. Y., Ye, X., Skinner, J. J., Mayne, L., Englander, S. W. ExMS2: An integrated solution for hydrogen-deuterium exchange mass spectrometry data analysis. Analytical Chemistry. 91 (11), 7474-7481 (2019).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Characterizing short-lived protein folding intermediates by top-down hydrogen exchange mass spectrometry. Analytical Chemistry. 82 (20), 8591-8597 (2010).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Hydrogen/deuterium exchange mass spectrometry with top-down electron capture dissociation for characterizing structural transitions of a 17 kDa protein. Journal of the American Chemical Society. 131 (35), 12801-12808 (2009).
- Mistarz, U. H., et al. Photodissociation mass spectrometry accurately localizes sites of backbone deuteration in peptides. Analytical Chemistry. 90 (2), 1077-1080 (2017).
- Phillips, J. J., et al. Rate of asparagine deamidation in a monoclonal antibody correlating with hydrogen exchange rate at adjacent downstream residues. Analytical Chemistry. 89 (4), 2361-2368 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon