Method Article
Millisekunden-Wasserstoff/Deuterium-Austausch-Massenspektrometrie zur Untersuchung der Alpha-Synuclein-Strukturdynamik unter physiologischen Bedingungen
In diesem Artikel
Zusammenfassung
Das strukturelle Ensemble des monomeren Alpha-Synucleins beeinflusst seine physiologische Funktion und seine physikalisch-chemischen Eigenschaften. Das vorliegende Protokoll beschreibt, wie eine Millisekunden-Wasserstoff/Deuterium-Austausch-Massenspektrometrie und anschließende Datenanalysen durchgeführt werden können, um Konformationsinformationen über das Monomer dieses intrinsisch ungeordneten Proteins unter physiologischen Bedingungen zu bestimmen.
Zusammenfassung
Alpha-Synuclein (aSyn) ist ein intrinsisch ungeordnetes Protein, dessen fibrilläre Aggregate in Lewy-Körpern und Neuriten, den Kennzeichen der Parkinson-Krankheit, reichlich vorhanden sind. Ein Großteil seiner biologischen Aktivität sowie seine Aggregation betreffen jedoch zentral die lösliche Monomerform des Proteins. Die Aufklärung der molekularen Mechanismen der aSyn-Biologie und Pathophysiologie erfordert strukturell hochaufgelöste Methoden und ist empfindlich gegenüber biologischen Bedingungen. Seine nativ entfalteten, metastabilen Strukturen machen monomere aSyn für viele strukturbiologische Techniken unlösbar. Hier wird die Anwendung eines solchen Ansatzes beschrieben: Wasserstoff/Deuterium-Austausch-Massenspektrometrie (HDX-MS) auf der Millisekunden-Zeitskala zur Untersuchung von Proteinen mit geringer thermodynamischer Stabilität und schwachen Schutzfaktoren, wie z.B. aSyn. Auf der Millisekunden-Zeitskala enthalten HDX-MS-Daten Informationen über die Zugänglichkeit von Lösungsmitteln und die wasserstoffgebundene Struktur von aSyn, die bei längeren Markierungszeiten verloren gehen, was letztendlich zu einer strukturellen Auflösung bis zum Aminosäureniveau führt. Daher kann HDX-MS bei hohen strukturellen und zeitlichen Auflösungen Informationen über Konformationsdynamik und Thermodynamik, intra- und intermolekulare Wechselwirkungen und die strukturellen Auswirkungen von Mutationen oder Veränderungen der Umweltbedingungen liefern. Obwohl breit anwendbar, wird gezeigt, wie Millisekunden-HDX-MS-Messungen in monomeren aSyn erfasst, analysiert und interpretiert werden können.
Einleitung
Die Parkinson-Krankheit (PD) ist eine neurodegenerative Erkrankung, von der weltweit Millionen von Menschen betroffensind 1. Es ist gekennzeichnet durch die Bildung von zytoplasmatischen Einschlüssen, die als Lewy-Körper und Lewy-Neuriten in der Substantia nigra pars compacta-Region des Gehirns bekannt sind. Es wurde festgestellt, dass diese zytoplasmatischen Einschlüsse Aggregate des intrinsisch ungeordneten Proteins aSyn2 enthalten. Bei PD und anderen Synucleinopathien wandelt sich aSyn von einem löslichen ungeordneten Zustand in einen unlöslichen, hochstrukturierten Krankheitszustand um. In seiner nativen Form nimmt das monomere aSyn eine breite Palette von Konformationen an, die durch langreichweitige elektrostatische Wechselwirkungen zwischen seinen N- und C-Termini und hydrophobe Wechselwirkungen zwischen seinem C-Terminus und der Nicht-Amyloid-Beta-Komponente (NAC) Region 3,4,5,6 stabilisiert werden. Alle Störungen in diesen stabilisierenden Interaktionen, wie Mutationen, posttranslationale Modifikationen und Veränderungen in der lokalen Umgebung, können zur Fehlfaltung des Monomers führen und so den Prozess der Aggregation auslösen7.
Während eine große Menge an Forschung über die oligomeren und fibrillären Formen von aSyn 8,9,10,11 existiert, besteht ein entscheidender Bedarf, die monomere Form des Proteins zu untersuchen und besser zu verstehen, welche Konformer funktionell sind (und wie) und welche anfällig für Aggregatesind 8,9,10,11 . Da das aSyn-Monomer intrinsisch ungeordnet ist, nur 14 kDa groß ist und schwer zu kristallisieren ist, ist es für die meisten strukturbiologischen Techniken nicht zugänglich. Eine Technik, die in der Lage ist, die Konformationsdynamik des monomeren aSyn zu messen, ist jedoch die Millisekunde HDX-MS, die in letzter Zeit wichtige strukturelle Beobachtungen hervorgebracht hat, die sonst schwierig oder unmöglich zu erhaltenwären 12,13,14. Die Millisekunde HDX-MS misst sensitiv den Durchschnitt des Proteinkonformationsensembles, indem sie den Isotopenaustausch an Amidwasserstoffen überwacht und die Zugänglichkeit von Lösungsmitteln und die Beteiligung des Wasserstoffbrückennetzwerks einer bestimmten Proteinregion auf der Millisekunden-Zeitskala anzeigt. Es ist notwendig, den Millisekundenaspekt des HDX-MS hervorzuheben, da aSyn aufgrund seiner nativ entfalteten, metastabilen Natur eine sehr schnelle Wasserstoffaustauschkinetik aufweist, die sich deutlich unter der unteren Grenze herkömmlicher HDX-MS-Systeme manifestiert. Zum Beispiel hat der größte Teil des aSyn-Moleküls Wasserstoff unter intrazellulären Bedingungen in weniger als 1 s vollständig gegen Deuterium ausgetauscht. Mehrere Labore haben inzwischen Schnellmischgeräte gebaut; In diesem Fall wird ein Prototyp eines schnell mischenden Quench-Flow-Instruments verwendet, das HDX-MS mit einer Totzeit von 50 ms und einer zeitlichen Auflösung von 1 ms ausführen kann15. Während Millisekunden-HDX-MS in letzter Zeit bei der Untersuchung von aSyn akut wichtig war, ist es wertvoll für die Untersuchung von intrinsisch ungeordneten Proteinen / Regionen im weiteren Sinne und einer großen Anzahl von Proteinen mit Schleifen / Regionen, die nur schwach stabil sind. Zum Beispiel Peptidarzneimittel (z. B. Insulin; GLP-1/Glucagon; Tirzepatid) und Peptidfusionsproteine (z. B. der HIV-Inhibitor FN3-L35-T1144) sind wichtige Arzneimittelformate, in denen Struktur- und Stabilitätsinformationen der Lösungsphase ein kritischer Input für Entscheidungen zur Arzneimittelentwicklung sein können, und dennoch ist die Peptideinheit durch HDX-MS auf der Sekundenskala 16,17,18,19,20 oft nur schwach stabil und hartnäckig. . Es hat sich gezeigt, dass emergente HDX-MS-Methoden mit Markierung in den Sekunden/Minuten-Domänen strukturelle Informationen für DNA-G-Quadruplexe ableiten, aber es sollte möglich sein, diese durch die Anwendung von Millisekunden-HDX-MS21 auf vielfältigere Oligonukleotidstrukturen auszudehnen.
HDX-MS-Experimente können auf drei verschiedenen Ebenen durchgeführt werden: (1) bottom-up (wobei das markierte Protein proteolytisch verdaut wird), (2) Middle-Down (wobei das markierte Protein proteolytisch verdaut wird und die resultierenden Peptide durch Soft-Fragmentierungstechniken weiter fragmentiert werden) und (3) Top-Down (wobei Soft-Fragmentierungstechniken das markierte Protein direkt fragmentieren)22 . Submolekulare HDX-MS-Daten ermöglichen es uns daher, das Austauschverhalten auf bestimmte Regionen eines Proteins zu lokalisieren, was es für solche Experimente entscheidend macht, eine ausreichende Sequenzabdeckung zu haben. Die strukturelle Auflösung eines HDX-MS-Experiments hängt von der Anzahl der proteolytischen Peptide oder Fragmente ab, die bei der Verdauung bzw. Weichfragmentierung aus dem Protein stammen. In jedem der drei oben beschriebenen Experimenttypen wird die Änderung des Amidaustauschs an jedem Peptid / Fragment auf die Primärstruktur des Proteins zurückgebildet, um das Verhalten lokalisierter Regionen des Proteins anzuzeigen. Während die höchste strukturelle Auflösung durch Soft-Fragmentierung erreicht wird, liegt die Beschreibung dieser Experimente außerhalb des Rahmens der aktuellen Studie, die sich auf die Messung von aSyn-Monomer-Konformationen konzentriert. Hervorragende Ergebnisse können mit dem hier beschriebenen häufig angewendeten "Bottom-up"-Workflow erzielt werden.
Hier werden Verfahren zur Verfügung gestellt, (1) wie man aSyn-Proben und HDX-MS-Puffer vorbereitet und handhabt, (2) wie man ein Peptid-Mapping für ein Bottom-up-HDX-MS-Experiment durchführt, (3) wie man HDX-MS-Daten auf monomerem aSyn unter physiologischen Bedingungen, insbesondere im Millisekunden-Zeitbereich, erfasst (mit einem speziell angefertigten Instrument; alternative Instrumente zur Millisekundenmarkierung wurden ebenfalls beschrieben), und (4) wie die HDX-MS-Daten verarbeitet und analysiert werden. Methoden zur Verwendung von monomerem aSyn bei physiologischem pH-Wert (7,40) in zwei Lösungsbedingungen werden hier veranschaulicht. Obwohl diese Verfahren für die Untersuchung von aSyn von entscheidender Bedeutung sind, können sie auf jedes Protein angewendet werden und sind nicht auf intrinsisch ungeordnete Proteine beschränkt.
Protokoll
1. Proteinexpression und Reinigung von aSyn
- Bereiten Sie aSyn nach einem zuvor veröffentlichten Berichtvor 9.
- Dialysieren Sie in einen sicheren Speicherpuffer (z. B. Tris, pH 7,2 ).
- Konzentrieren Sie bei Bedarf die Probe (z. B. Spinfilter-Mikrozentrifugenröhrchen mit 3 kDa MWCO, 14.000 x g für ca. 10-30 min, siehe Materialtabelle).
HINWEIS: Es wird empfohlen, sich nicht übermäßig zu konzentrieren. Die Integrität des Monomer-Ensembles wurde nicht über 25 μM hinaus verifiziert. - Aliquot und bei −80 °C lagern
HINWEIS: Das aSyn-Monomerprotein ist unter diesen Lagerbedingungen bis zu 1 Jahr stabil.
2. HDX-Puffervorbereitung
HINWEIS: Da Tris einen hohen Temperaturkoeffizienten hat, muss die pH-Messung für die Temperatur angepasst werden, bei der die HDX-Reaktion durchgeführt wird, die in diesem Protokoll 20 ° C beträgt.
- Bereiten Sie den Gleichgewichtspuffer für den Zustand A und den Zustand B vor, indem Sie 0,002 mol Tris in 100 ml Wasser der LC-MS-Klasse wiegen. Für den Zustand B werden 29,8 mg KCl, 14,2 mg MgCl2, 36,8 gCaCl2 und 836 mg NaCl zum Tris-Puffer gegeben. Stellen Sie den pH-Wert auf 7,40 ± 0,05 ein.
HINWEIS: Der Gleichgewichtspuffer muss die Bedingungen enthalten, unter denen aSyn untersucht werden soll. In diesem Fall sind es 20 mM Tris bei pH 7,4 +/− Salzen. - Bereiten Sie den Markierungspuffer für den Zustand A und den Zustand B vor, indem Sie 0,002 mol Tris in 100 ml deuteriertes Wasser wiegen. Für den Zustand B werden 29,8 mg KCl, 14,2 mg MgCl 2, 36,8 g CaCl2 und 836 mg NaCl zum Tris-Markierungspuffer gegeben. Der pD des Markierungspuffers entspricht dem pH-Wert des Gleichgewichtspuffers. Da pH = pD - 0,41, so einstellen, dass das pH-Messgerät 6,99 ± 0,0523,24 anzeigt.
HINWEIS: Der Beschriftungspuffer muss die gleichen Komponenten wie der Gleichgewichtspuffer haben, mit der Ausnahme, dass er mit deuteriertem Wasser hergestellt wird. - Bereiten Sie den Abschreckpuffer vor, indem Sie 0,010 mol Tris und 0,050 mol Harnstoff wiegen und mit Wasser der LC-MS-Klasse bis zu 100 ml herstellen. Stellen Sie den pH-Wert auf 2,50 ± 0,05 bei 0,5 °C ein.
HINWEIS: Vor HDX-Experimenten muss ein Quench-Puffer-Screen durchgeführt werden, um den besten Quench-Puffer für das interessierende Protein zu identifizieren. Verschiedene Konzentrationen und Kombinationen von Denaturierungsmitteln (z. B. Harnstoff und Guanidiniumhydrochlorid) und Reduktionsmitteln (z. B. Tris(2-Carboxyethyl)phosphin) werden zusammen mit physikalischen Parametern wie Fangvolumen und Temperatur untersucht, um das abgeschreckte Protein effektiv zu entfalten und zu verdauen. Ein Quenchpuffer bestehend aus 100 mM Tris und 0,5 M Harnstoff bei pH 2,50 ist für die vorliegende Studie optimal. - Bereiten Sie den Waschpuffer der Verdauungskolonne vor, indem Sie 0,125 mol Guanidiniumhydrochlorid in eine Duran-Glasflasche wiegen. 25 ml Methanol und 250 μL Ameisensäure hinzufügen. Machen Sie bis zu 250 ml mit LC-MS-Wasserqualität.
HINWEIS: Verwenden Sie für die Enzymate BEH Pepsin-Säule (siehe Materialtabelle) einen Säulenwaschpuffer von 0,5 M Guanidiniumhydrochlorid, 10% (v/v) Methanol und 0,1% (v/v) Ameisensäure. - Bereiten Sie die Spritzenschwachwäsche vor, indem Sie 0,5 μl Ameisensäure in 249,5 ml LC-MS-Wasser pipettieren.
- Bereiten Sie die starke Spritzenwäsche vor, indem Sie gleiche Teile von LC-MS-Wasser, Methanol, Acetonitril und Isopropanol mischen. Ameisensäure zu einer endgültigen Konzentration von 2% (v/v) hinzufügen.
HINWEIS: Um eine Kreuzkontamination zwischen den verschiedenen Puffern und dem Protein zu verhindern und die Reinigung des Injektionsports zu ermöglichen, ist es wichtig, dass Spritzenwaschlösungen hergestellt werden und dass der Durchflussweg zum Ventil (oft als "Waschliner" bezeichnet) vollständig mit Flüssigkeit grundiert wird. Die Ameisensäure ist optional für die Spritze Schwachwäsche.
3. Verfahren zur Peptidkartierung
- Bereiten Sie das Beispiel nach dem folgenden Schritt vor.
- Filtern Sie aufgetaute aSyn-Proteinvorräte aus dem −80 °C Gefrierschrank mit 0,22 μm Spritzenfiltern. Messen Sie die Absorption des gefilterten Stammproteins bei 280 nm, um die Konzentration nach dem Beer-Lambert-Gesetz zu bestimmen. Das Protein wird im Gleichgewichtspuffer auf eine Konzentration von 5 μM verdünnt (Schritt 2.1.).
HINWEIS: Das Beer-Lambert-Gesetz: A = εcl, wobei A die Absorption ist, ε der Extinktionskoeffizient des Proteins bei der gemessenen Wellenlänge (280 nm hier) mit den Einheiten M−1 cm−1, c die Proteinkonzentration in M und l die Weglänge in cm ist. Für den Wildtyp aSyn26 ist ε = 5960 M−1 cm−1.
- Filtern Sie aufgetaute aSyn-Proteinvorräte aus dem −80 °C Gefrierschrank mit 0,22 μm Spritzenfiltern. Messen Sie die Absorption des gefilterten Stammproteins bei 280 nm, um die Konzentration nach dem Beer-Lambert-Gesetz zu bestimmen. Das Protein wird im Gleichgewichtspuffer auf eine Konzentration von 5 μM verdünnt (Schritt 2.1.).
- Richten Sie die Flüssigkeitschromatographie-Methode ein.
- Erstellen Sie eine Einlassdatei mit einer Lade- / Fangzeit von 3 min bei einem Druck von 7000-9000 psi, gefolgt von einem Gradienten von 5% Acetonitril bis 40% Acetonitril in 7 min, gefolgt von wiederholten Waschschritten von 5% -95% Acetonitril-Wasser für 10 min.
- Stellen Sie sicher, dass Lock-Spray (z. B. Leucin-Enkephalin, siehe Materialtabelle) mit 2000 psi fließt und mit der Source-Lock-Spray-Sonde des Massenspektrometers verbunden ist.
- Richten Sie die Massenspektrometrie-MSE-Methoden ein.
HINWEIS: MSE ist ein breitbandiges datenunabhängiges Erfassungsverfahren ohne Massenisolierung des Vorläufers. Daher werden alle Ionen innerhalb des ausgewählten m/z-Bereichs mittels kollisionsinduzierter Dissoziation (CID)27 weiter fragmentiert.- Wählen Sie in der MS-Methodendatei MSE Continuum und richten Sie eine Erfassungszeit zwischen 2-10 min, Elektrosprayquelle und positivem Auflösungsmodus ein. Erwerben Sie MSE über 50-2000 Da, scannen Sie alle 0,3 s.
- Stellen Sie für Funktion 1 (niedrige Energie) die Falle auf und übertragen Sie die Kollisionsenergien auf 4 V. Stellen Sie für Funktion 2 (hohe Energie) die Kollisionsenergie der Rampenübertragung so ein, dass sie konstant bei 4 V ist, und die Fallenkollisionsenergie, wie in Tabelle 1 für jede Abbildungsenergiestufe angegeben.
HINWEIS: Ionenmobilität MSE-Methoden können ebenfalls verwendet werden. Alternative Mapping-Methoden (z.B. datenabhängige Erfassung oder DDA) können nach Belieben des Nutzers eingesetzt werden.
- Richten Sie den Autosampler-Roboter ein (siehe Materialtabelle).
- Geben Sie 50 μL des 5 μM-Proteins zu einer vollständigen Durchstechflasche für die Erholung. Stellen Sie die Durchstechflasche in einer Probenposition in der rechten HDX-Kammer auf. Stellen Sie sicher, dass diese Kammer bei 0,5 ° C liegt.
- Fügen Sie ein Reagenzfläschchen mit Gleichgewichtspuffer und zwei Reagenzfläschchen mit Markierungspuffer zu den Reagenzienpositionen eins, zwei und drei in der linken HDX-Kammer hinzu. Stellen Sie sicher, dass diese Kammer bei 20 °C steht, indem Sie den Peltier-Temperaturregler einstellen (siehe Materialtabelle). Fügen Sie ein Reagenzfläschchen mit Abschreckpuffer hinzu, um das Reagenz in der rechten HDX-Kammer zu positionieren.
- Fügen Sie acht Gesamtrückgewinnungsfläschchen in den Reaktionspositionen der linken HDX-Kammer und acht Durchstechflaschen mit maximaler Rückgewinnung in den Reaktionspositionen der rechten HDX-Kammer hinzu.
HINWEIS: Um eine vollständige Reinigung und maximale Reproduzierbarkeit der abgegebenen Volumina zu gewährleisten, wird empfohlen, eine Spritzenwäsche und eine Prime-Wäsche an den Autosampler-Spritzen durchzuführen. Führen Sie beispielsweise eine Sequenz von (1) schwacher Wäsche, (2) starker Wäsche, (3) schwacher Wäsche aus, bevor Sie mit den Mapping-Experimenten beginnen. Diese Sequenz kann ausgiebig wiederholt werden, und es wird empfohlen, dies bis zu 20x oder bis zur vollständigen Benetzung der Spritze zu tun. - Richten Sie in der Planungssoftware eine Beispielliste mit entsprechenden LC- und MS-Methoden ein und starten Sie den Zeitplan.
HINWEIS: Für die vorliegende Studie wird Chronos als Planungssoftware verwendet (siehe Materialverzeichnis).
- Verarbeiten Sie die Mapping-Daten.
- Identifizieren Sie Peptide aus den Mapping-Experimentdateien mit entsprechender Software (siehe Materialverzeichnis).
- Importieren Sie Peptididentifikationsdaten in DynamX (siehe Materialverzeichnis) mit den folgenden Peptidschwellenwertparametern: minimale Intensität = 5000, minimale Sequenzlänge = 0, maximale Sequenzlänge = 40, minimale Produkte = 1, minimale Produkte pro Aminosäure = 0,25, minimale aufeinanderfolgende Produkte = 2, minimale Summenintensität für Produkte = 0, minimale Punktzahl = 0 und maximaler MH+ Fehler (ppm) = 0.
HINWEIS: Alternativ können Sie die optimierten Einstellungen nach einem empfohlenen Workflow28 ableiten. - Wählen Sie Daten aus dem Menü und klicken Sie auf PLGS-Ergebnisse importieren. Klicken Sie auf Hinzufügen , um die relevanten Datendateien für die Spektralzuordnung auszuwählen. Wenn alle hinzugefügt wurden, klicken Sie auf Weiter und geben Sie die obigen Filtereinstellungen ein. Klicken Sie dann auf Fertig stellen.
- Kuratieren Sie Isotopenzuweisungen manuell, um die endgültige Peptidabdeckungskarte von aSyn in DynamX zu erhalten.
4. Millisekunden-Wasserstoff/Deuterium-Austauschstudie
- Reinigen Sie das FastHDX-Prototyp-Instrument (siehe Materialtabelle), bevor Sie mit HDX-Experimenten beginnen.
- Öffnen Sie die kompatible grafische Benutzeroberfläche der HDX-Software und lassen Sie das System initialisieren.
- Geben Sie die Temperatur der Probenkammer als 20 °C und die Quenchkammer als 0,5 °C ein. Klicken Sie auf Einstellen , um die neuen Temperaturen anzuwenden.
- Stellen Sie die Zentrifugenröhrchen an allen Einlässen mit LC-MS-Wasser auf.
- Überprüfen Sie auf der Registerkarte Titrator Plumbing Delivery sowohl die linke als auch die rechte Spritze und klicken Sie auf Prime , um latente Luftblasen in den Schläuchen zu entfernen. Wiederholen Sie den Vorgang, bis alle Blasen verschwunden sind.
- Aktivieren Sie auf der Registerkarte Makros alle Kontrollkästchen für die Spritzen. Klicken Sie auf Spritzen Home Position kalibrieren. Klicken Sie auf Spritzenladeschleife waschen. Klicken Sie auf Alle Mischschleifenvolumina waschen.
- Wiederholen Sie Schritt 4.1.5. 1x mehr.
- Wenn Blasen in den Pufferspritzen auftreten, entgasen Sie, indem Sie die Spritze trennen und die Blase vertikal auswerfen. Ersetzen Sie die Spritze und kalibrieren Sie sie auf die Nullposition neu.
- Richten Sie das FastHDX-Prototyp-Instrument für HDX-Experimente ein.
- Geben Sie 500 uL gefiltertes 5 μM aSyn in eine Durchstechflasche mit Gesamtrückgewinnung und legen Sie sie in einen Tischkühlschrank, um eine temperaturbedingte Oligomerisierung und Aggregation zu verhindern.
- Fügen Sie jedem Puffer 50 ml Gleichgewichts-, Beschriftungs- und Abschreckpuffer hinzu (2. HDX-Puffervorbereitungsschritte 1-3) Einlass in der linken und rechten Kammer.
- Fügen Sie 50 ml Säulenwaschpuffer hinzu (2. HDX-Puffervorbereitungsschritt 4) zum Pepsin-Wascheinlass.
- Um die Protein- und Säulenwaschlinien 1x zu grundieren, überprüfen Sie sowohl die linken als auch die rechten Spritzen auf der Registerkarte Titrator Plumbing Delivery und klicken Sie einmal auf Prime.
HINWEIS: Jeder nachfolgende Klick auf Prime führt zu zusätzlichen Wiederholungen des Prime-Prozesses, was zum Verbrauch großer Mengen an Proteinproben führt. Es wird empfohlen, dies zu vermeiden, auch wenn es zu einer Verzögerung in der Software kommt, nachdem versucht wurde, auf eine Schaltfläche zu klicken. - Wiederholen Sie die Schritte 4.1.5. 1x.
- Geben Sie auf der Registerkarte Manueller Abschreckfluss die erforderlichen Einstellungen ein, die in den Schritten 4.2.7.-4.2.10 erläutert werden.
- Geben Sie für ein Zeitverlaufsexperiment die Zeiten in Millisekunden mit der Schaltfläche Symbolische Punkte ein. Wenn Replikate erforderlich sind, fügen Sie denselben Zeitpunkt mehrmals hinzu, z. B. für eine Verdreifachung von 50 ms müssen 50 50 50 eingegeben werden. Die Beispielliste in der Massenspektrometer-Software (MassLynx wird hier verwendet, siehe Materialtabelle) muss genau mit diesen Zeitpunkten übereinstimmen.
HINWEIS: Die Dateinamen der Massenspektrometer-Probenliste und/oder der Probentext sollten verwendet werden, um eine dauerhafte Aufzeichnung der HDX-Kennzeichnungszeiten zu gewährleisten, die den in der FastHDX-Software-GUI eingegebenen Zeiten entsprechen. Die Beschriftungszeiten für jeden Samplelauf werden nirgendwo anders gespeichert. - Legen Sie die Fangzeit (Minuten) auf die Länge der Fangzeit fest. Hier ist es 3.00 Uhr.
- Legen Sie Wait for HPLC (mins) = (Trap-Zeit + Laufzeit + 1,5 min) fest.
HINWEIS: Für ein Experiment mit 3 min Trapping und 17 min Gradienten sind dies beispielsweise 21,50 min. - Klicken Sie nur dann auf das Feld Leer ausführen, wenn leere Experimente zwischen Beispielläufen ausgeführt werden. Wenn ja, stellen Sie nach jedem Beispiellauf einen Eintrag (d. h. eine gültige Zeile in der Beispielliste) in der Software für den leeren Lauf sicher.
- Sobald die Beispielliste auf der Software fertig ist, markieren Sie die entsprechenden Einträge und starten Sie den Lauf in der Software, indem Sie auf die Schaltfläche Play und FastHDX in der Software klicken.
HINWEIS: Aufgrund des Wasserstoff/Deuterium-Scramblings können nur MS-only-Methoden oder Soft-Fragmentierungstechniken (Elektronentransferdissoziation, Elektroneneinfang-Dissoziation und ultraviolette Photodissoziation) nur29 verwendet werden. Für aSyn bei einem physiologischen pH-Wert von 7,40 sind Zeitpunkte im Bereich von 50 ms bis 300 s am besten geeignet, da diese die gesamte Deuterium-Aufnahmekurve8 abdecken.
5. Datenverarbeitung
- Laden Sie die Datei der spektral zugewiesenen Peptide aus den Peptid-Mapping-Experimenten. Öffnen Sie das Menü Datei und klicken Sie in der DynamX-Software auf Öffnen (siehe Materialverzeichnis).
- Importieren Sie Rohdateien in die bevorzugte Massenmesssoftware (z. B. DynamX, HDExaminer usw.). Öffnen Sie das Menü "Daten" und klicken Sie auf MS Files. Klicken Sie auf Neuer Zustand , um Zustände für jeden untersuchten Proteinzustand zu erstellen.
- Klicken Sie auf Neue Belichtung , um jeden HDX-Zeitpunkt hinzuzufügen. Klicken Sie auf Neues RAW , um die .raw Dateien zu importieren. Ziehen Sie jede .raw Datei an die richtige Position. Klicken Sie auf OK, wenn Sie fertig sind.
- Weisen Sie zuerst automatisch Isotope zu (dies geschieht automatisch nach Schritt 5.2. oben), und kuratieren Sie dann manuell die Isotopenzuweisung, um eine hohe Datenqualität sicherzustellen.
- Exportieren Sie die Clusterdaten in eine .csv Datei mit Spalten in der folgenden Reihenfolge: Proteinname, Sequenzstartnummer, Sequenzendnummer, Sequenz, Modifikation, Fragment, maximal mögliche Aufnahme, Masse der monoisotopischen Spezies, Zustandsname, Expositionszeit, Dateiname, Ladung, Retentionszeit, Intensität und Schwerpunkt.
- Öffnen Sie das Menü Daten in der Massenmesssoftware und klicken Sie auf Clusterdaten exportieren.
6. Datenanalyse
- Laden Sie die exportierten Clusterdaten in die bevorzugte HDX-Analysesoftware. Hier wird HDfleX30 verwendet (siehe Tabelle der Materialien).
- Passen Sie die experimentellen Daten für alle Peptide und Zustände an und wählen Sie die geeigneten Back-Exchange-Korrekturmethoden aus, um beobachtete Ratenkonstanten für die HDX-Reaktion zu erhalten.
- Berechnen Sie einen globalen Signifikanzschwellenwert nach der bevorzugten Methode (HDfleX unterstützt mehrere Optionen dafür) und führen Sie hybride Signifikanztests durch, um die signifikanten Unterschiede zwischen den Staaten im Vergleich zu31,32 zu bestimmen.
HINWEIS: Wenn die beobachtete Differenz größer als der globale Schwellenwert ist und der p-Wert kleiner als das gewählte Konfidenzniveau ist (z. B. 95%), wird die Differenz als signifikant angesehen.
Ergebnisse
Aufgrund seiner intrinsisch ungeordneten Natur ist es schwierig, die komplizierten strukturellen Veränderungen in aSyn bei physiologischem pH-Wert zu erfassen. HDX-MS überwacht den Isotopenaustausch an Backbone-Amid-Wasserstoffen und untersucht die Proteinkonformationsdynamik und -wechselwirkungen. Es ist eine der wenigen Techniken, diese Informationen mit hohen strukturellen und zeitlichen Auflösungen zu erfassen. Dieses Protokoll ist allgemein auf eine breite Palette von Proteinen und Pufferbedingungen anwendbar, und dies wird durch die Messung der Austauschkinetik von aSyn in zwei verschiedenen Lösungsbedingungen veranschaulicht: Zustand A und Zustand B8, wie in den Schritten 2.1.-2.2 definiert.
Zuerst wurde ein Mapping-Experiment auf aSyn durchgeführt und eine Peptidabdeckungskarte erhalten, wie in Abbildung 1 gezeigt. Die Karte deckt 100% der Proteinsequenz ab und hat eine durchschnittliche Redundanz von 3,79. Der 100%-Abdeckungswert zeigt an, dass alle Aminosäuren im Protein in den Proteinverdauungen gefunden wurden und ermöglicht eine umfassende Analyse des Austauschverhaltens von aSyn. Der Redundanzwert gibt die Anzahl der überlappenden Peptide an. Ein höherer Redundanzwert erhöht die strukturelle Auflösung der endgültigen Abbildung bei subtraktiver Abflachung der Daten für überlappendePeptide 32.
Unter Verwendung des Prototyps des schnell mischenden Quench-Flow-Instruments (siehe Tabelle der Materialien) wurden qualitativ hochwertige HDX-MS-Daten auf Millisekunden-Zeitskala auf aSyn bei pH 7,4 in Zustand A und Zustand B gesammelt (Abbildung 2). Nach einer Isotopenzuordnung in DynamX wurden "rohe" Deuterium-Aufnahmekurven erhalten, wie in Abbildung 3A gezeigt. Es zeigt Aufnahmekurven für drei Peptide, die in jeder Proteindomäne ausgewählt wurden. Die Deuteriuminkorporation im Laufe der Zeit wird angezeigt. Die x-Achse liegt auf der Millisekunden-Zeitskala, die sich mit der sehr schnellen Kinetik von aSyn unter physiologischen Bedingungen ausrichtet. Der rot schattierte Bereich zeigt die Daten, die typischerweise von herkömmlichen HDX-Geräten mit Startmessungen ab 30 s gewonnen werden. Wichtig ist, dass dies durch pH-Manipulation für die sogenannte "Zeitfenstererweiterung" nicht weiter reduziert werden kann; Dieser Ansatz ist für die Untersuchung von intrinsisch ungeordneten Proteinen / Regionen ungültig, da die pH-Verschiebung das Konformationsensemble des schwach stabilen Polypeptids stört. Wie hier zu sehen ist, wird der größte Teil von aSyn vollständig um 1 s ausgetauscht (Abbildung 3C). Dies zeigt die Bedeutung von Millisekunden-HDX-Messungen für monomere aSyn, da die vollständige kinetische Aufnahmekurve für die Austauschreaktion erfasst wird, was die genaueste Messung der Monomerkonformationen ergibt.
HDfleX führte eine Back-Exchange-Korrektur unter Verwendung der Plateau-Deuterium-Einarbeitung durch. Die Datenpunkte wurden anschließend gemäß Gleichung 1 angepasst, was eine beobachtete Geschwindigkeitskonstante, kobs, liefert, die auf die Zugänglichkeit des Lösungsmittels und die Beteiligung dieses bestimmten Peptids an der Wasserstoffbindung hinweist (Abbildung 3B).
 Gleichung 1
Gleichung 1
wobei D t der Deuteriumeinbau zum Zeitpunkt t, nExp die Anzahl der exponentiellen Phasen, N die maximale Anzahl labiler Wasserstoffe, kobs die beobachtete Wechselkurskonstante und β ein Dehnungsfaktor30,33 ist.
Nach der Kurvenanpassung kann der Aufnahmebereich unter der angepassten Kurve berechnet werden, indem die angepasste Funktion, die die Aufnahmekurve beschreibt, in das experimentelle Zeitfenster integriert wird. Es wurden statistische Signifikanzanalysen zwischen dem Aufnahmegebiet der beiden Staaten durchgeführt. Zunächst wurde in HDfleX eine globale Signifikanzschwelle für den Aufnahmebereich mit einem Konfidenzniveau von 95% berechnet. Anschließend wurden Aufnahmeflächendifferenzdiagramme generiert, die die Differenz zwischen Zustand A und Zustand B auf zwei Ebenen der strukturellen Auflösung zeigten: Peptidauflösung (Abbildung 4A) und Aminosäureauflösung (Abbildung 4B). Das Diagramm der Peptidauflösungsdifferenz zeigt den Unterschied in der Aufnahmefläche zwischen Zustand A und Zustand B für jedes einzelne Peptid, während das Diagramm der Aminosäureauflösungsdifferenz den Unterschied in der Aufnahmefläche zwischen Zustand A und Zustand B über die gesamte Aminosäuresequenz von aSyn30,34 abflacht. Beide Diagramme zeigen eine insgesamt größere Deuteriumaufnahme im gesamten aSyn-Monomer im Zustand B im Vergleich zu Zustand A. Dieser Befund kann durch die Untersuchung der Deuteriumaufnahmediagramme in Abbildung 3 gerechtfertigt werden, wobei die Aufnahmekurve des Zustands B immer über der Aufnahmekurve des Zustands A liegt. Darüber hinaus kann gesehen werden, dass das Ausmaß der Aufnahmeflächendifferenz am C-Terminus viel höher ist. Auch dies kann gerechtfertigt werden, indem auf die ursprünglichen Aufnahmekurven zurückgegriffen wird, wo die C-terminalen Peptide (Peptide 124-140 in Abbildung 3) eine viel größere Lücke zwischen den Aufnahmekurven aufweisen als der Rest des Proteins. Zusammenfassend lässt sich sagen, dass die Lösungsmittelbedingungen im Zustand B eine Zunahme der Lösungsmittelexposition oder eine Abnahme der Beteiligung des Wasserstoffbrückenbindungsnetzwerks im gesamten Protein, aber mehr noch am C-Terminus verursachen.

Abbildung 1: Peptidabdeckungskarte des Wildtyps aSyn mit insgesamt 30 Peptiden und 100% Sequenzabdeckung. Die drei Domänen von aSyn werden wie folgt hervorgehoben: N-Terminus (blau), Nicht-Amyloid-Betakomponentenregion (gelb) und C-Terminus (rot). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 2: Workflow für ein Millisekunden-HDX-MS-Experiment auf aSyn. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Beispielaufnahmediagramme aus drei Peptiden, die in den drei Domänen von aSyn für den Zustand A (gelb) und den Zustand B (blau) ausgewählt wurden. (A) Nicht eingebautes und nicht rücktauschkorrigiertes Aufnahmediagramm. (B) Einbau- und rücktauschkorrigierte Aufnahmeflächen. Der rot schattierte Bereich stellt Daten dar, die von herkömmlichen HDX-MS-Systemen abgerufen werden können, typischerweise ab 30 s. Fehlerbalken entsprechen der Standardabweichung der drei Replikate. (C) Heatmap-Diagramm der prozentualen Deuteriumaufnahme über die Aminosäuresequenz pro Zeitpunkt. Der Farbbalken stellt den Prozentsatz der Deuteriumaufnahme dar. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
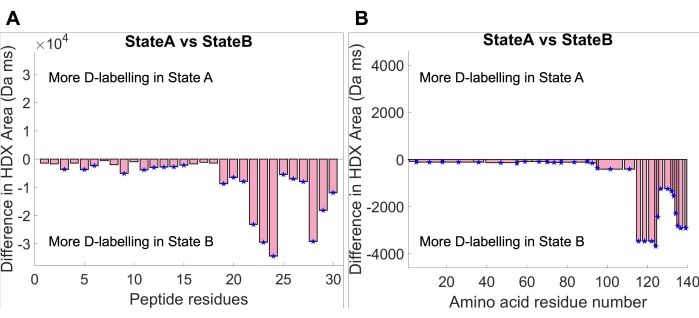
Abbildung 4: Aufnahmebereichsdifferenzdiagramme bei maximaler Kurvenplateauzeit (14.084 ms). (A) Diagramme zur Auflösung von Peptidresten zeigen die Aufnahmebereichsdifferenz jedes Peptids zwischen Zustand A und Zustand B. (B) Diagramm mit Aminosäureauflösung, das die Aufnahmebereichsdifferenz zwischen Zustand A und Zustand B über die Aminosäuresequenz abgeflacht zeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
| Abbildung des Energieniveaus | Rampenspannung (V) |
| Niedrig | 20-40 |
| Mittel | 25-45 |
| Hoch | 30-50 |
| Sehr hoch | 35-55 |
Tabelle 1: Abbildung von Energieniveaus und entsprechenden Übertragungsrampenspannungen.
Diskussion
Im vorliegenden Artikel werden die folgenden Verfahren beschrieben: (1) Durchführung von Peptid-Mapping-Experimenten an monomerem aSyn, um die höchste Sequenzabdeckung zu erhalten, (2) Erfassung von Millisekunden-HDX-MS-Daten auf monomerem aSyn unter physiologischen Bedingungen und (3) Durchführung von Datenanalysen und -interpretationen der resultierenden HDX-MS-Daten. Die bereitgestellten Verfahren sind im Allgemeinen einfach auszuführen, jedes Kennzeichnungsexperiment dauert in der Regel nur etwa 8 Stunden für drei Replikate und acht Zeitpunkte, und das Mapping-Experiment dauert nur etwa 2 Stunden. Aufgrund der hier verwendeten vollautomatischen Instrumentierung kann ein vollständiger Datensatz in 1 Tag erfasst werden. Bei der Handhabung von Proben und der Vorbereitung von Puffern muss jedoch darauf geachtet werden, dass Messungen aus schwach stabilen Proteinen (oder Proteinregionen) im gewünschten Zustand abgeleitet werden. Wichtig ist, dass eine frühere Studie zeigte, dass unterschiedliche Lagerbedingungen, wie Einfrieren und Lyophilisieren, zu unterschiedlichen aSyn-Monomerkonformisten führten und dass es wichtig ist, die potenziellen Auswirkungen der Probenhandhabung auf das aSyn-Monomer-Konformationsensemble10 zu charakterisieren. Tatsächlich ist HDX-MS ein hochempfindliches Maß für solche Konformationsstörungen mit einem Dynamikbereich von Mikrosekunden bis mindestens Monaten. Wenn nur das aSyn-Monomer streng untersucht wird, wird außerdem dringend empfohlen, unerwünschte Oligomere und Fibrillen zu entfernen, die sich bei der Lagerung oder Handhabung in der Probe gebildet haben könnten. Darüber hinaus müssen die HDX-MS-Puffer innerhalb von 0,05 des gewünschten pH-Wertes oder pD streng kontrolliert werden, da Diskrepanzen die intrinsische Wechselkursrate erheblich beeinflussen und zu unerwünschten Fehlern führen. Es ist auch wichtig zu beachten, dass Vergleiche zwischen den Lösungsbedingungen für jedes Protein, das sich in pH-Wert, Temperatur oder Salzzusammensetzung unterscheidet, die intrinsische Rate verändern. Daher erfordern diese Daten weitere Korrekturen, wie z.B. die Anwendung eines pH-Einstellfaktors 35 oder eines empirischen Korrekturfaktors 8,30.
In Bezug auf die Instrumentierung gibt es keine kommerziell erhältlichen Systeme, die die Erfassung von Millisekunden-HDX-MS-Daten ermöglichen. Mehrere Forschungsgruppen haben ihre eigenen Systeme entwickelt, von Quench-Flow-Systemen13,15,36 bis hin zu mikrofluidischen Chips37,38,39, um die schnelle Austauschkinetik bestimmter Proteine zu erfassen. Eine weitere Methode, die verwendet wurde, um Millisekunden-Zeitskala-HDX-MS-Daten zu erhalten, ist die Zeitexpansionsmethode40,41, bei der der pH-Wert der Puffer reduziert wird, um die Austauschkinetik zu verlangsamen. Diese Methode gilt jedoch nicht für aSyn (oder für schwach stabile Proteinmerkmale), da (1) die Senkung des pH-Wertes die Ladungsdichte des Proteins drastisch verändert und die Aggregationsrateum 8,42 erhöht und (2) die aSyn-Konformer nur metastabil sind und wahrscheinlich durch diese pH-Änderungen gestört werden. Aus diesen Gründen wird empfohlen, bei der Untersuchung monomerer aSyn-Konformationen, sofern sie nicht physiologisch relevant sind, einen konstanten pH-Wert in den HDX-MS-Puffern beizubehalten und ein Millisekunden-Markierungsinstrument zu verwenden.
Der größte Teil des aSyn-Monomers tauscht sich innerhalb von 1 s vollständig aus, und höchstens 15 s dauert der vollständige Austausch mit Deuterium (Abbildung 3) bei einem physiologisch relevanten pH-Wert von 7,4 (was die intrazellulären zytosolischen Bedingungen an der Präsynapse widerspiegelt). Bei herkömmlichen HDX-MS-Systemen ist ein Start bei 30 s nicht geeignet, da die HDX-MS-Daten dem Plateau der Austauschreaktion entsprechen würden, das keine nützlichen Konformationsinformationen liefert. Die untere Messgrenze des Millisekunden-HDX-Instruments (entsprechend einer "Totzeit" von 50 ms) ermöglicht jedoch die Überwachung der Austauschreaktion ab ~25% Fertigstellung für das aSyn-Monomer bei pH 7,4. Dies ermöglichte es uns, den Großteil der kinetischen Aufnahmekurve zu erfassen. Die Anpassung der Deuterium-Aufnahmekurve an Gleichung 1 liefert wichtige kinetische Informationen; Sie entspricht einer Schätzung der beobachteten Ratenkonstante K Obs. Obwohl hier nicht behandelt, ist es möglich, Aggregationskinetikexperimente durchzuführen und Fibrillenmorphologien von aSyn unter den gleichen Lösungsbedingungen wie die HDX-MS-Experimente zu untersuchen, da HDX-MS eine hohe Toleranz gegenüber einer Vielzahl von Puffernaufweist 8. So können beispielsweise die kobs aus dem HDX-MS-Experiment mit den Ergebnissen aus den Aggregationsexperimenten korreliert werden, um Erkenntnisse darüber zu gewinnen, welche Konformationen am anfälligsten für bestimmte Aggregationsverhalten und Fibrillenmorphologien sind.
Für den einfachen Fall von differentiellen HDX-MS-Experimenten, bei denen zwei oder mehr Bedingungen oder Proteinvarianten verglichen werden sollen, kann der Bereich unter der angepassten Aufnahmekurve für jeden Zustand integriert und miteinander verglichen werden. In dieser Studie wurden die Aufnahmebereiche für Zustand A und Zustand B auf zwei verschiedenen Ebenen der strukturellen Auflösung verglichen: Peptidauflösung und Aminosäureauflösung, die beide unterschiedliche Stärken und Herausforderungen aufweisen. Zum Beispiel spiegeln die Peptidauflösungsdaten die rohen Spektraldaten genauer wider und haben die geringste Verarbeitung erfahren. Die "abgeflachten" Aminosäure-Auflösungsdaten ermöglichen es jedoch, sowohl Peptid- als auch Soft-Fragmentierungsinformationen in einem einzigen Output zu kombinieren, anstatt separate nicht zusammenführbare Outputs zu trennen und die Daten letztendlich mit der höchsten strukturellen Auflösung darzustellen. Eine Einschränkung der massenspektrometrieren Detektion der HDX-Markierung ist die Herausforderung, eine Aminosäureauflösung zu erhalten. Während sich "Soft-Fragmentation" -Techniken wie Elektronentransferdissoziation (ETD), Elektroneneinfangdissoziation (ECD) und ultraviolette Photodissoziation (UVPD) als wirksam bei der Erzeugung höherer Auflösungen erwiesen haben, bleiben sie herausfordernd, unvorhersehbar und ineffizient 30,43,44,45,46,47,48.
Im Vergleich zu anderen Strukturtechniken hat Millisekunden-HDX-MS den einzigartigen Vorteil, die Konformationsdynamik monomerer aSyn bei hohen strukturellen und zeitlichen Auflösungen zu erfassen. Da die schnelle Austauschkinetik des Monomers kein limitierender Faktor mehr ist, können weitere Studien an monomerem aSyn mit verschiedenen Mutationen, posttranslationalen Modifikationen, Salzkomponenten und -konzentrationen sowie Bindungspartnern bei physiologischem pH-Wert durchgeführt werden. Die Korrelation der HDX-MS-Ergebnisse mit funktionellen Studien wie Aggregationskinetik und Fibrillenmorphologien kann Einblicke in Konformatoren geben, die entweder die normale Zellfunktion fördern oder krankheitsanfällig sind. Letztendlich wird erwartet, dass eine solche Millisekunden-HDX-MS entscheidend für die Entdeckung gezielter Medikamente sein könnte, die bestimmte physiologisch verträgliche Konformer stabilisieren.
Offenlegungen
Die Autoren erklären keine konkurrierenden Interessen.
Danksagungen
NS wird durch das Diamond Jubilee Scholarship des Universitätsrats finanziert. JJP wird durch ein UKRI Future Leaders Fellowship [Fördernummer: MR/T02223X/1] unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 1 × 100 mm ACQUITY BEH 1.7 μm C18 column | Waters Corporation | 186002346 | Analytical column |
| Acetonitrile HPLC grade >99.9% HiPerSolv | VWR | 20060.420 | For LC mobile phases |
| CaCl2 | Sigma Aldrich | C5670 | Salt for HDX buffers |
| Chronos | Axel Semrau (Purchased from Waters Corporation) | 667006090 | Scheduling software to enable multiple HDX-MS sample injections automatically. Alternative software is available from other vendors e.g. HDXDirector or LEAP Shell |
| Deuterium chloride | Goss Scientific (Cambridge Isotope Laboratories) | DLM-2-50 | For HDX labelling buffers |
| Deuterium oxide (99.9% D2O) | Goss Scientific (Cambridge Isotope Laboratories) | DLM-4 | Deuterated water |
| DynamX 3.0 | Waters Corporation | 176016027 | Isotopic assignment and deuterium incorporation calculation |
| Enzymate BEH Pepsin Column | Waters Corporation | 186007233 | Pepsin digestion column |
| Formic Acid, 99.0% LC/MS Grade | Fisher Scientific | 10596814 | For LC mobile phases |
| Guanidinium hydrochloride | Sigma Aldrich | RDD001-500G | Chaotrope/Denaturant |
| HDfleX | University of Exeter | N/A | https://ore.exeter.ac.uk/repository/handle/10871/127982 |
| KCl | Sigma Aldrich | P3911 | Salt for HDX buffers |
| LEAP HDX-2 CTC PAL sampling robot | Waters Corporation | 725000637 | Autosampler robot |
| Leucine enkephalin | Waters Corporation | 186006013 | For mass spectrometry lockspray calibration. |
| MassLynx | Waters Corporation | 667004007 | Software controlling inlet methods and mass spectrometer |
| Maximum recovery vials | Waters Corporation | 600000670CV | 100 pack including caps - used for quench tray in LEAP HDX-2 |
| MgCl2 | Sigma Aldrich | M8266 | Salt for HDX buffers |
| Millipore 0.22 µm syringe filters | Millipore | N9CA7069B | Syringe filters |
| ms2min | Applied Photophysics Ltd | N/A | fast-mix quench-flow millisecond hdx instrument |
| NaCl | Sigma Aldrich | S9888 | Salt for HDX buffers |
| Peltier temperature controller | LEAP Technologies Inc. | HP115-COOL/D | Peltier controller to set precise temperature of chambers in the LEAP robot. |
| ProteinLynx Global Server 3.0 | Waters Corporation | 715001030 | Peptide identification software. Alternative software is available from other vendors. |
| Reagent pot caps | Waters Corporation | 186004632 | 100 pack |
| Reagent pots for LEAP HDX-2 | Waters Corporation | 186001420 | 100 pack excluding caps - used for buffers in LEAP HDX-2 |
| Sodium deuteroxide (99.5% in D2O) | Goss Scientific (Cambridge Isotope Laboratories) | DLM-57 | For HDX labelling buffers |
| Spin filter microcentrifuge tubes (3 kDa MWCO) | Amicon (Merck Sigma Aldrich) | UFC5003 | Micro centrifuge tubes to concentrate protein. This facilitates buffer exchange and accurate sample loading for HDX-MS experiments. |
| Synapt G2-Si mass spectrometer | Waters Corporation | 176850035 | Mass spectrometer |
| Total recovery vials | Waters Corporation | 600000671CV | 100 pack including caps - used for labelling tray in LEAP HDX-2 |
| Tris-HCl | Sigma Aldrich | T3253-250G | Buffer |
| Trizma base | Sigma Aldrich | T60040-B2005 | Buffer |
| Urea | Sigma Aldrich | U5378-1KG | Chaotrope/Denaturant |
| VanGuard 2.1 x 5 mm ACQUITY BEH C18 column | Waters Corporation | 186004623 | Trap desalting column |
Referenzen
- Dorsey, E. R., et al. regional, and national burden of Parkinson's disease, 1990-2016: A systematic analysis for the Global Burden of Disease Study 2016. The Lancet Neurology. 17 (11), 939-953 (2018).
- Breydo, L., Wu, J. W., Uversky, V. N. α-Synuclein misfolding and Parkinson's disease. Biochimica et Biophysica Acta (BBA): Molecular Basis of Disease. 1822 (2), 261-285 (2012).
- Dedmon, M. M., Lindorff-Larsen, K., Christodoulou, J., Vendruscolo, M., Dobson, C. M. Mapping long-range interactions in α-synuclein using spin-label NMR and ensemble molecular dynamics simulations. Journal of the American Chemical Society. 127 (2), 476-477 (2005).
- Esteban-Martín, S., Silvestre-Ryan, J., Bertoncini, C. W., Salvatella, X. Identification of fibril-like tertiary contacts in soluble monomeric α-synuclein. Biophysical Journal. 105 (5), 1192-1198 (2013).
- McClendon, S., Rospigliosi, C. C., Eliezer, D. Charge neutralization and collapse of the C-terminal tail of alpha-synuclein at low pH. Protein Science. 18 (7), 1531-1540 (2009).
- Ranjan, P., Kumar, A. Perturbation in long-range contacts modulates the kinetics of amyloid formation in α-synuclein familial mutants. ACS Chemical Neuroscience. 8 (10), 2235-2246 (2017).
- Villar-Piqué, A., da Fonseca, T. L., Outeiro, T. F. Structure, function and toxicity of alpha-synuclein: the Bermuda triangle in synucleinopathies. Journal of Neurochemistry. 139, Suppl 1 240-255 (2015).
- Seetaloo, N., Zacharopoulou, M., Stephens, A. D., Schierle, G. S. K., Phillips, J. J. Local structural dynamics of alpha-synuclein correlate with aggregation in different physiological conditions. bioRxiv. , (2022).
- Stephens, A. D., et al. Extent of N-terminus exposure of monomeric alpha-synuclein determines its aggregation propensity. Nature Communications. 11 (1), 2820(2020).
- Stephens, A. D., et al. Different structural conformers of monomeric α-synuclein identified after lyophilizing and freezing. Analytical Chemistry. 90 (11), 6975-6983 (2018).
- Lautenschläger, J., et al. C-terminal calcium binding of α-synuclein modulates synaptic vesicle interaction. Nature Communications. 9 (1), 712(2018).
- Oganesyan, I., Lento, C., Tandon, A., Wilson, D. J. Conformational dynamics of α-synuclein during the interaction with phospholipid nanodiscs by millisecond hydrogen-deuterium exchange mass spectrometry. Journal of the American Society for Mass Spectrometry. 32 (5), 1169-1179 (2021).
- Keppel, T. R., Weis, D. D. Analysis of disordered proteins using a simple apparatus for millisecond quench-flow H/D exchange. Analytical Chemistry. 85 (10), 5161-5168 (2013).
- Al-Naqshabandi, M. A., Weis, D. D. Quantifying protection in disordered proteins using millisecond hydrogen exchange-mass spectrometry and peptic reference peptides. Biochemistry. 56 (31), 4064-4072 (2017).
- Kish, M., et al. Allosteric regulation of glycogen phosphorylase solution phase structural dynamics at high spatial resolution. bioRxiv. , (2019).
- El-Amine, M., et al. Mechanisms of tolerance induction by a gene-transferred peptide-IgG fusion protein expressed in B lineage cells. Journal of Immunology. 165 (10), 5631-5636 (2000).
- Kishimoto, S., et al. Site-specific chemical conjugation of antibodies by using affinity peptide for the development of therapeutic antibody format. Bioconjugate Chemistry. 30 (3), 698-702 (2019).
- Xu, W., et al. A protein-based, long-acting HIV-1 fusion inhibitor with an improved pharmacokinetic profile. Pharmaceuticals. 15 (4), 424(2022).
- Frías, J. P., et al. Tirzepatide versus semaglutide once weekly in patients with type 2 diabetes. The New England Journal of Medicine. 385 (6), 503-515 (2021).
- Gerstein, H. C., et al. Cardiovascular and renal outcomes with efpeglenatide in type 2 diabetes. The New England Journal of Medicine. 385 (10), 896-907 (2021).
- Largy, E., Gabelica, V. Native hydrogen/deuterium exchange mass spectrometry of structured DNA oligonucleotides. Analytical Chemistry. 92 (6), 4402-4410 (2020).
- Marcsisin, S. R., Engen, J. R. Hydrogen exchange mass spectrometry: What is it and what can it tell us. Analytical and Bioanalytical Chemistry. 397 (3), 967-972 (2010).
- Glasoe, P. K., Long, F. A. Use of glass electrodes to measure acidities in deuterium oxide. Journal of Physical Chemistry. 64 (1), 188-190 (1960).
- Krȩzel, A., Bal, W. A formula for correlating pKa values determined in D2O and H2O. Journal of Inorganic Biochemistry. 98 (1), 161-166 (2004).
- Mayerhöfer, T. G., Pahlow, S., Popp, J. The Bouguer-Beer-Lambert law: Shining light on the obscure. ChemPhysChem. 21 (18), 2029-2046 (2020).
- Gasteiger, E., et al. The Proteomics Protocols Handbook. , Springer. New York. 571-607 (2005).
- Bateman, R. H., et al. A novel precursor ion discovery method on a hybrid quadrupole orthogonal acceleration time-of-flight (Q-TOF) mass spectrometer for studying protein phosphorylation. Journal of the American Society for Mass Spectrometry. 13 (7), 792-803 (2002).
- Sørensen, L., Salbo, R. Optimized workflow for selecting peptides for HDX-MS data analyses. Journal of the American Society for Mass Spectrometry. 29 (11), 2278-2281 (2018).
- Demmers, J. A. A., Rijkers, D. T. S., Haverkamp, J., Killian, J. A., Heck, A. J. R. Factors affecting gas-phase deuterium scrambling in peptide ions and their implications for protein structure determination. Journal of the American Chemical Society. 124 (37), 11191-11198 (2002).
- Seetaloo, N., Kish, M., Phillips, J. J. HDfleX: Software for flexible high structural resolution of hydrogen/deuterium-exchange mass spectrometry data. Analytical Chemistry. 94 (11), 4557-4564 (2022).
- Hageman, T. S., Weis, D. D. Reliable identification of significant differences in differential hydrogen exchange-mass spectrometry measurements using a hybrid significance testing approach. Analytical Chemistry. 91 (13), 8008-8016 (2019).
- Hageman, T. S., Weis, D. D. A structural variant approach for establishing a detection limit in differential hydrogen exchange-mass spectrometry measurements. Analytical Chemistry. 91 (13), 8017-8024 (2019).
- Chetty, P. S., et al. Helical structure and stability in human apolipoprotein A-I by hydrogen exchange and mass spectrometry. Proceedings of the National Academy of Sciences of the United States of America. 106 (45), 19005-19010 (2009).
- Keppel, T. R., Weis, D. D. Mapping residual structure in intrinsically disordered proteins at residue resolution using millisecond hydrogen/deuterium exchange and residue averaging. Journal of the American Society for Mass Spectrometry. 26 (4), 547-554 (2015).
- Li, J., Rodnin, M. V., Ladokhin, A. S., Gross, M. L. Hydrogen-deuterium exchange and mass spectrometry reveal the pH-dependent conformational changes of diphtheria toxin T domain. Biochemistry. 53 (43), 6849-6856 (2014).
- Roder, H., Elöve, G. A., Englander, S. W. Structural characterization of folding intermediates in cytochrome c by H-exchange labelling and proton NMR. Nature. 335 (6192), 700-704 (1988).
- Rob, T., et al. Measuring dynamics in weakly structured regions of proteins using microfluidics-enabled subsecond H/D exchange mass spectrometry. Analytical Chemistry. 84 (8), 3771-3779 (2012).
- Rob, T., Gill, P. K., Golemi-Kotra, D., Wilson, D. J. An electrospray ms-coupled microfluidic device for sub-second hydrogen/deuterium exchange pulse-labelling reveals allosteric effects in enzyme inhibition. Lab on a Chip. 13 (13), 2528-2532 (2013).
- Svejdal, R. R., Dickinson, E. R., Sticker, D., Kutter, J. P., Rand, K. D. Thiol-ene microfluidic chip for performing hydrogen/deuterium exchange of proteins at subsecond time scales. Analytical Chemistry. 91 (2), 1309-1317 (2018).
- Goswami, D., et al. Time window expansion for HDX analysis of an intrinsically disordered protein. Journal of The American Society for Mass Spectrometry. 24 (10), 1584-1592 (2013).
- Coales, S. J., E, S. Y., Lee, J. E., Ma, A., Morrow, J. A., Hamuro, Y. Expansion of time window for mass spectrometric measurement of amide hydrogen/deuterium exchange reactions. Rapid Communications in Mass Spectrometry. 24 (24), 3585-3592 (2010).
- Hoyer, W., et al. Dependence of alpha-synuclein aggregate morphology on solution conditions. Journal of Molecular Biology. 322 (2), 383-393 (2002).
- Rand, K. D., Pringle, S. D., Morris, M., Engen, J. R., Brown, J. M. ETD in a traveling wave ion guide at tuned Z-spray ion source conditions allows for site-specific hydrogen/deuterium exchange measurements. Journal of the American Society for Mass Spectrometry. 22 (10), 1784-1793 (2011).
- Kan, Z. Y., Ye, X., Skinner, J. J., Mayne, L., Englander, S. W. ExMS2: An integrated solution for hydrogen-deuterium exchange mass spectrometry data analysis. Analytical Chemistry. 91 (11), 7474-7481 (2019).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Characterizing short-lived protein folding intermediates by top-down hydrogen exchange mass spectrometry. Analytical Chemistry. 82 (20), 8591-8597 (2010).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Hydrogen/deuterium exchange mass spectrometry with top-down electron capture dissociation for characterizing structural transitions of a 17 kDa protein. Journal of the American Chemical Society. 131 (35), 12801-12808 (2009).
- Mistarz, U. H., et al. Photodissociation mass spectrometry accurately localizes sites of backbone deuteration in peptides. Analytical Chemistry. 90 (2), 1077-1080 (2017).
- Phillips, J. J., et al. Rate of asparagine deamidation in a monoclonal antibody correlating with hydrogen exchange rate at adjacent downstream residues. Analytical Chemistry. 89 (4), 2361-2368 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten