Method Article
Risonanza magnetica nucleare Pure Shift: un nuovo strumento per la metabolomica delle piante
In questo articolo
Riepilogo
Questo articolo presenta l'uso di PSYCHE e SAPPHIRE-PSYCHE nella profilazione metabolica delle piante e include procedure dettagliate per la preparazione del campione e la registrazione ottimale degli spettri NMR Pure Shift. Vengono discussi esempi attraverso i quali il guadagno in risoluzione ottenuto dal disaccoppiamento omonucleare consente una comprensione più completa del sistema.
Abstract
La Risonanza Magnetica Nucleare (NMR) è uno degli strumenti più potenti utilizzati in metabolomica. Si pone come un metodo altamente accurato e riproducibile che non solo fornisce dati quantitativi, ma consente anche l'identificazione strutturale dei metaboliti presenti in miscele complesse.
La profilazione metabolica mediante NMR 1H si è dimostrata utile nello studio di vari tipi di scenari vegetali, che includono la valutazione delle condizioni delle colture, i trattamenti del raccolto e post-raccolto, la fenotipizzazione metabolica, le vie metaboliche, la regolazione genica, l'identificazione di biomarcatori, la chemiotassonomia, il controllo di qualità, la denominazione di origine, tra gli altri. Tuttavia, la sovrapposizione del segnale del gran numero di risonanze con molteplicità di accoppiamento J espanse complica l'analisi degli spettri e la sua interpretazione e rappresenta una limitazione per il classico profilo NMR 1H.
Nell'ultimo decennio, sono state sviluppate nuove tecniche di disaccoppiamento omonucleare a banda larga NMR attraverso le quali i segnali multipli collassano in singole linee di risonanza - comunemente chiamate metodi Pure Shift - per superare il problema della risoluzione degli spettri inerente aglispettri classici NMR 1 H.
Qui viene presentato un protocollo passo-passo della preparazione dell'estratto vegetale e la procedura per registrare gli spettri ottimali di Pure Shift PSYCHE e SAPPHIRE-PSYCHE in tre diverse matrici vegetali: foglie di pianta di vaniglia , tuberi di patata (S. tuberosum) e uva spina del Capo (P. peruviana). Viene discusso l'effetto dell'aumento della risoluzione nell'identificazione metabolica, nell'analisi di correlazione e nelle analisi multivariate, rispetto agli spettri classici.
Introduzione
L'insieme completo dei metaboliti che compongono un organismo - substrati, intermedi e prodotti finali dei processi biologici - è stato coniato nel 1998 con il termine metaboloma. E' noto che il metaboloma è strettamente correlato al fenotipo, ed è di particolare interesse nelle piante in quanto riflette l'interazione diretta tra il genotipo e l'ambiente 1,2. Pertanto, la caratterizzazione del profilo metabolomico è diventata di fondamentale importanza nelle piante. Attraverso l'identificazione e la quantificazione di biomarcatori (metaboliti chiave) e modelli metabolici, è stato possibile discriminare tra specie, cultivar, stadi di sviluppo, malattie patogene o condizioni ambientali (cambiamenti giornalieri e stagionali, suoli, stress idrico, stress meccanico, raccolti e trattamenti post-raccolta), tra gli altri, 3,4,5.
La spettrometria di massa (MS) e la spettroscopia di risonanza magnetica nucleare (NMR) sono le piattaforme analitiche più utilizzate per questo scopo. Contrariamente alle metodologie MS, la risonanza magnetica nucleare è una tecnica altamente riproducibile, non distorta, quantitativa, accurata e non distruttiva che richiede una preparazione minima del campione, il che la rende adatta per gli studi di metabolomica. Tuttavia, rispetto ai metodi MS, la bassa sensibilità intrinseca è un limite. Negli ultimi anni e attraverso l'uso di magneti ad alto campo, sonde criogeniche, dispositivi a micro-bobina e metodi di polarizzazione nucleare dinamica (DNP), la sensibilità della risonanza magnetica nucleare è stata notevolmente migliorata. Nel caso di quest'ultimo approccio, ad esempio, il guadagno di sensibilità era di due o tre ordini di grandezza 6,7. Ad oggi, quasi il 20% degli studi di metabolomica pubblicati sono basati sulla risonanza magnetica nucleare e il numero è in aumento,7.
Anche se la risonanza magnetica namica protonica è l'esperimento più popolare e sensibile per l'impronta digitale metabolomica NMR, presenta alcuni inconvenienti. In primo luogo, tutti i segnali NMR di 1ora rilevati nel campione sono distribuiti in una piccola finestra corrispondente alla finestra di spostamento chimico del protone, il che si traduce in spettri affollati. In secondo luogo, l'accoppiamento scalare omonucleare divide i segnali in più componenti (molteplicità del segnale), diffondendo il segnale del protone su una gamma di frequenze più ampia, complicando ulteriormente la lettura degli spettri aumentando l'affollamento e la sovrapposizione del segnale. Inoltre, la metabolomica NMR viene impiegata nell'analisi di miscele solitamente contenenti da 50 a 300 molecole a una concentrazione osservabile NMR, generando spettri complessi composti da 200 a 2000 picchi.
La NMR del protone di disaccoppiamento omonucleare, nota anche come Pure Shift, è un metodo che induce il collasso di un segnale multipletto in un singolo picco. Si pone come uno strumento eccellente per aumentare la risoluzione del segnale negli spettri affollati 8,9,10 e quindi rappresenta uno strumento conveniente per la metabolomica delle piante11.
Nell'ultimo decennio sono emerse nuove sequenze di impulsi Pure Shift, che aumentano sia la sensibilità che le prestazioni di disaccoppiamento. Anche la loro gamma di applicazioni si è ampliata, dalla delucidazione della struttura molecolare12,13, alla fluxomica14, all'assegnazione di miscele 15,16,17, alle misure di diffusione traslazionale 18, alla discriminazione enantiomerica19, alla distribuzione unitaria in copolimeri20, tra le altre.
Storicamente, gli esperimenti Broadband Pure Shift soffrono di bassa sensibilità e metodi di lavorazione complicati, limitando il loro campo di applicazione nella valutazione degli estratti biologici8. Nel 2014, Foroozandeh et al. hanno pubblicato un nuovo esperimento Pure Shift, PSYCHE (Pure Shift Yielded by Chirp Excitation), basato sulla sequenza di impulsi anti-z-COSY che ha prodotto un eccellente disaccoppiamento omonucleare e un miglioramento dei valori di sensibilità21. Tuttavia, poiché PSYCHE è un esperimento di interferogramma 2D in cui vengono acquisiti blocchi di dati nel dominio del tempo, soffre di artefatti periodici della banda laterale che derivano da distorsioni di modulazione dell'accoppiamento J ai bordi del pezzo. Nelle miscele complesse, questi artefatti producono segnali più grandi di quelli associati ai metaboliti presenti a concentrazioni molto basse, ostacolando l'analisi11. Esistono due metodi per rimuovere questi artefatti: TSE-PHYCHE22 e una modifica più recente dell'esperimento PSYCHE chiamata SAPPHIRE-PSYCHE (Sideband Averaging by Periodic PHase Incrementation of Residual J Evolution)23.
Nel 2019, abbiamo dimostrato per la prima volta11 che il metodo SAPPHIRE-PSYCHE Pure Shift, che rimuove gli artefatti quasi senza penalità di sensibilità23, potrebbe essere impiegato per l'analisi di miscele biologiche complesse, come gli estratti dei frutti di Physalis peruviana, comunemente noti come uva spina del Capo11. Abbiamo dimostrato che questi metodi aumentano le prestazioni delle analisi dei dati metabolomici come l'assegnazione metabolica, l'analisi di correlazione e l'analisi dei coefficienti multivariati11. Da allora, sono stati affrontati diversi studi di metabolomica Pure Shift su diverse matrici biologiche, come i coralli molli24, le piante di iperico25, il miele26,27, il tè27, l'olio di menta piperita26 e le noci28, dimostrando la sua importanza come nuovo strumento per l'analisi metabolomica. Paradossalmente, la stragrande maggioranza di questi studi ha utilizzato la sequenza di impulsi PSYCHE standard e facile da implementare, disponibile in qualsiasi libreria di spettrometri, invece della sequenza di impulsi SAPPHIRE-PSYCHE, che ha dimostrato di funzionare meglio. Tuttavia, richiede una migliore comprensione della sequenza di impulsi per una corretta configurazione.
Questo articolo ha lo scopo di aiutare i nuovi utenti ad applicare i metodi Pure Shift nello studio delle piante, in particolare, foglie di Vanilla sp (V. planifolia e V. pompona)29, tuberi di patata (S. tuberosum)30 e uva spina del Capo (P. peruviana)31. La preparazione del campione, l'impostazione sperimentale NMR, l'acquisizione e l'analisi dei dati sono descritti in dettaglio. Inoltre, il protocollo include note chiave per aiutare i ricercatori, nuovi nel campo, a impostare correttamente gli esperimenti PSYCHE e SAPPHIRE-PSYCHE nella profilazione metabolomica delle piante.
Protocollo
1. Preparazione del campione
- Uva spina del Capo
- Mettere 100-200 g di frutta fresca in un vaso frullatore. Mantenere a 4 °C per 30 minuti, quindi omogeneizzare in un frullatore da laboratorio.
- Trasferisci immediatamente il succo in provette di plastica da 50 ml, congelale in azoto liquido e liofilizzale a secco per 4 o 5 giorni.
- Macinare il materiale liofilizzato in una polvere fine utilizzando un macinino elettrico.
NOTA: La manipolazione del materiale secco deve essere eseguita rapidamente perché la polvere è altamente igroscopica. - Pesare 1 g di materiale macinato e aggiungere 10 ml di acqua ultrapura. Vortice per 1 min.
- Sonicare per 20 minuti a 10 °C, quindi centrifugare a 23.000 × g per 20 minuti a 10 °C.
- Recuperare il surnatante e filtrarlo attraverso un filtro per siringa da 13 mm in politetrafluoroetilene (PTFE) da 0,45 μm.
- Liofilizzare 1 mL dell'estratto filtrato fino a secchezza e quindi risospendere il solido ottenuto in 0,9 mL di tampone ossalato di sodio 200 mM pH 4. Vortice.
- Liofilizzare il campione risultante fino a portarlo a secco e scioglierlo in 0,9 mL di ossido di deuterio contenente 5 mM di sale sodico acido 3-(trimetilsilil)propionico-2,2,3,3-d4 (TMSP-d4).
- Riempire la provetta NMR con 0,6 mL di campione utilizzando una micropipetta.
- Foglie di vaniglia
- Raccogli le foglie, puliscile con carta velina umida e congelale intere in azoto liquido.
- Rompere le foglie in piccoli pezzi e liofilizzare per 4 giorni fino a quando non si asciugano.
- Macinare la sostanza secca fino a ridurla in polvere fine utilizzando un macinino elettrico.
- Pesare 50 mg di materiale macinato e aggiungere 0,75 mL di tampone fosfato pH 6,0 in ossido di deuterio contenente lo 0,1% di TMSP (p/p) e 0,75 mL di metanolo-d4. Vortice per 1 min.
- Sonicare per 20 minuti a 25 °C.
- Centrifugare a 13.000 × g per 10 min a 25 °C.
- Recuperare il surnatante (~1,3-1,4 mL) e filtrarlo attraverso un filtro per siringa da 13 mm in PTFE da 0,45 μm.
- Riempire la provetta NMR con 0,6 mL del campione filtrato utilizzando una micropipetta.
- Tuberi di patata
- Sbucciare e affettare da 4 a 8 tuberi. Immediatamente, mettere circa 125 g di materiale in sacchetti stand-up e congelarli in azoto liquido.
NOTA: Per evitare l'ossidazione durante la manipolazione, mantenere la patata umida. - Liofilizzare per 4-6 giorni fino a completa secchezza.
- Macinare la sostanza secca fino a ridurla in polvere fine utilizzando un macinino elettrico.
- Pesare 160 mg di tubero macinato e aggiungere 1,6 ml di acqua deionizzata. Vortice per 1 min.
- Sonicare per 45 minuti a 10 °C.
- Centrifugare a 23.000 x g per 20 min a 10 °C.
- Recuperare il surnatante (~1,5 -1,6 mL) e farlo evaporare a secco in un concentratore centrifugo sottovuoto refrigerato per 16 ore, a 10 °C.
- Aggiungere al solido ottenuto (20-25 mg) 0,9 mL di tampone ossalato di sodio 100 mM pH 4, vorticare ed evaporare per 16 ore a 10 °C.
- Sciogliere il solido ottenuto in 0,9 mL di ossido di deuterio contenente 3 mM di TMSP.
- Centrifugare a 23.000 x g per 5 minuti a 10 °C e filtrare il surnatante direttamente nella provetta NMR attraverso un filtro per siringa da 13 mm in PTFE da 0,45 μm.
NOTA: In questo caso, è stata eseguita una filtrazione diretta nella provetta NMR per ridurre le fasi di preparazione di oltre 1000 campioni.
- Sbucciare e affettare da 4 a 8 tuberi. Immediatamente, mettere circa 125 g di materiale in sacchetti stand-up e congelarli in azoto liquido.
2. Acquisizione ed elaborazione dei dati NMR
- Configurazione iniziale NMR
- Trasferire i campioni allo spettrometro NMR.
- Sintonizza e abbina la testa della sonda.
- Bloccare e spessorare il campione.
- Calibrare l'impulso duro a 90°. Calibrare l'impulso a 90° utilizzando una qualsiasi delle procedure standard.
- Esegui uno spettro NMR protonico 1D standard.
- Esperimento PSYCHE
- Selezionare la sequenza di impulsi reset_psyche_1d dalla libreria Bruker Topspin (Figura S1). Utilizzare i seguenti parametri standard: larghezza spettrale di 5 kHz (SW2), almeno 1 o 2 secondi di ritardo di recupero del rilassamento (D1), 16 scansioni fittizie (DS), 64 o 128 punti dati complessi per blocco (L31) e 64 o 128 scansioni (NS) (Figura S1).
NOTA: L31 è il numero di punti digitali complessi acquisiti in ogni blocco Pure Shift, che è meglio impostare su una potenza di 2. 21 - Impostare l'eccitazione dell'angolo di inversione dell'impulso CHIRP desiderata (CNST61) e 10 kHz per la larghezza di banda dell'impulso CHIRP (CNST60) (Figura S2).
NOTA: L'esperimento PSYCHE si basa su uno schema anti-z-COY; di conseguenza, l'angolo di inversione dell'impulso CHIRP deve essere piccolo per evitare artefatti di riaccoppiamento (Figura 1). L'intensità assoluta aumenta con l'angolo di inversione dell'eccitazione. Anche gli artefatti periodici vengono migliorati, diffondendosi nello spettro e aumentando il "rumore" (Figura 1). Il "rumore" diventa una combinazione di rumore standard e artefatti di chuncking. Un buon compromesso tra sensibilità e bassi artefatti di riaccoppiamento è impostare CNST61 = 20°. Ore 19,22 - Impostare la lunghezza dell'impulso forte (P1) sul valore precedentemente calibrato e la lunghezza dell'impulso della forma PSYCHE su 30 ms (P49) (Figura S2).
NOTA: È molto importante calibrare il valore dell'impulso duro poiché la potenza dell'impulso di forma verrà calcolata automaticamente da questo valore. - Scegliere l'impulso di forma Crp_psyche,20 (SPNAM 37) per l'elemento PSYCHE (Figura S2).
- Impostare l'intensità del gradiente del campo impulsivo applicato durante l'elemento PSYCHE (GPZ0). Scegliere RECT.1 per l'impulso di forma a gradiente (GPNAM 0) (Figura S2).
NOTA: Un debole gradiente di campo magnetico viene applicato durante l'elemento PSYCHE, normalmente, tra l'1% e il 4% della forza massima del gradiente, a seconda della sonda. - Impostare il numero di blocchi da acquisire per ricostruire il Pure Shift FID (TD1) (Figura S3).
NOTA: PSYCHE viene acquisito come un esperimento pseudo-2D dove TD1 è il numero di blocchi di interferogramma Pure Shift. La risoluzione dello spettro dipende dalla dimensione della finestra spettrale (SW1) e dal numero totale di punti acquisiti, che è TD1*2*L31. In genere, 16 o 32 blocchi con 64 o 128 punti complessi per blocco forniranno una risoluzione digitale sufficiente. Poiché PSYCHE viene registrato in modo interferogramma, un numero maggiore di blocchi aumenta la risoluzione digitale, ma anche il tempo totale di acquisizione19. Gli accoppiamenti J omonucleari si evolvono durante ogni blocco, risultando in un modello di modulazione J oscillante21,23. Dopo la trasformata di Fourier, questo genera artefatti periodici della banda laterale che dipendono dalla lunghezza del blocco (Figura 2). Per ridurre gli artefatti, la durata del blocco deve essere breve, in genere inferiore a 16 ms (durata del blocco = 2*in0: Figura S1). Se la durata del blocco è elevata, ridurre L31. - Elabora i dati con il programma AU Proc_reset di Bruker e la trasformata di Fourier.
NOTA: Si consiglia di trasformare lo spettro utilizzando il riempimento zero e un'apodizzazione a campana sinusoidale (Figura S4).
- Selezionare la sequenza di impulsi reset_psyche_1d dalla libreria Bruker Topspin (Figura S1). Utilizzare i seguenti parametri standard: larghezza spettrale di 5 kHz (SW2), almeno 1 o 2 secondi di ritardo di recupero del rilassamento (D1), 16 scansioni fittizie (DS), 64 o 128 punti dati complessi per blocco (L31) e 64 o 128 scansioni (NS) (Figura S1).
- Esperimento SAPPHIRE-PSYCHE
- Selezionare la sequenza di impulsi SAPPHIRE-PSYCHE e impostare i parametri della sequenza di impulsi. I parametri standard sarebbero i seguenti: 5 kHz di larghezza spettrale (SW3), almeno 1 o 2 secondi di ritardo di rilassamento (D1), 16 scansioni fittizie (DS), 8 o 16 scansioni per incremento (NS) e da D2 a 14 ms (Figura S5).
NOTA: Questa sequenza non è nel repertorio di Bruker, tuttavia, la sequenza e i programmi di elaborazione possono essere ottenuti dal sito web del Manchester NMR Methodology Group, (https://www.nmr.chemistry.manchester.ac.uk/?q=node/426)23. Il ritardo D2 assicura che il rilassamento T2 rimanga costante ad ogni incremento di modulazione J. D2 deve essere maggiore di 1/4*SW1+p16+2*d16. 23 - Impostare l'eccitazione dell'angolo di inversione dell'impulso CHIRP desiderata (CNST20) e 10 kHz per la larghezza di banda dell'impulso CHIRP (CNST21) (Figura S6).
NOTA: Come nel normale esperimento PSYCHE, l'angolo di inversione dell'impulso CHIRP deve essere corto per evitare artefatti di riaccoppiamento. CNST20 = 20° è un buon compromesso tra sensibilità e bassi artefatti di riaccoppiamento 21,23,25. - Impostare la lunghezza dell'impulso forte (P1) sul valore precedentemente calibrato e la lunghezza dell'impulso della forma PSYCHE su 30 ms (P40) (Figura S6).
NOTA: È importante calibrare il valore dell'impulso duro poiché la potenza dell'impulso di forma verrà calcolata automaticamente da esso. - Scegliere l'impulso di forma PSYCHE_Saltire_10kHz_30m per l'elemento PSYCHE (Figura S6).
- Impostare l'intensità del gradiente del campo impulsivo applicato durante l'elemento PSYCHE (GPZ10). Scegliere RECT.1 per l'impulso di forma del gradiente (GPNAM 10) (Figura S7).
NOTA: Durante l'elemento PSYCHE viene applicato un debole gradiente di campo magnetico, normalmente compreso tra l'1% e il 4% dell'intensità massima del gradiente, valore che dipende dalla sonda. - Impostare il numero di incrementi di modulazione SAPPHIRE J in F2 (TD2) (Figura S7).
NOTA: normalmente 8 incrementi assicurano un'ottima soppressione degli artefatti della banda laterale (Figure 2 e 3). Il numero totale di scansioni del FID finale di Pure Shift è NS*TD2. 23 - Impostare le finestre spettrali F1 e F2 (SW1 e SW2) (Figura S5).
NOTA: SW2=SW3/(2*TD2) e SW3/SW1 = TD2*N, dove TD2 e N sono interi pari23. L'esperimento SAPPHIRE-PSYCHE viene acquisito come pseudo 3D dove F2 codifica la modulazione di fase dell'artefatto di accoppiamento J e F1 l'acquisizione dell'interferogramma Pure Shift20. Poiché SAPPHIRE-PSYCHE rimuove le bande laterali di modulazione J , la durata del blocco Pure Shift dell'interferogramma potrebbe essere più lunga del normale PSYCHE (durata del blocco Pure Shift = 1/SW1), tipicamente compresa tra 20 e 40 ms (Figura 2). Tuttavia, l'acquisizione di dati a blocchi più lunghi porta a evoluzioni più elevate dell'accoppiamento J, che richiederebbero più incrementi di modulazione di fase dell'accoppiamento J per rimuovere le bande laterali più forti raggiunte23. - Impostare il numero di blocchi Pure Shift (TD1) (Figura S7).
NOTA: Poiché SAPPHIRE-PSYCHE deve compensare la modulazione di fase dell'accoppiamento J del primo blocco, è necessario acquisire un blocco aggiuntivo. Tipicamente, 17 (16+1) o 33 (32+1) blocchi forniscono una risoluzione digitale sufficiente23. - Elabora i dati eseguendo i programmi pm_pshift e pm_fidadd AU seguiti dalla trasformata di Fourier23.
NOTA: Si consiglia di trasformare lo spettro utilizzando il riempimento zero e un'apodizzazione a campana sinusoidale (Figura S4).
- Selezionare la sequenza di impulsi SAPPHIRE-PSYCHE e impostare i parametri della sequenza di impulsi. I parametri standard sarebbero i seguenti: 5 kHz di larghezza spettrale (SW3), almeno 1 o 2 secondi di ritardo di rilassamento (D1), 16 scansioni fittizie (DS), 8 o 16 scansioni per incremento (NS) e da D2 a 14 ms (Figura S5).
Risultati
Analisi dello spettro NMR
Gli esperimenti PSYCHE aumentano la risoluzione degli spettri facendo collassare le risonanze accoppiate in singoletti21, che a loro volta riducono la sovrapposizione e facilitano l'assegnazione e l'analisi dei dati. Pure Shift NMR può essere applicato agli estratti vegetali. Qui dimostriamo il suo utilizzo in tre diverse matrici: foglie di vaniglia, tuberi di patata e frutti di Physalis peruviana . L'aumento della risoluzione ottenuto negli spettri di questi estratti vegetali è chiaro dalle figure S8-S11.
Al fine di valutare l'effetto di queste sequenze di impulsi nella risoluzione del segnale NMR, la larghezza della linea in unità di frequenza al 10% dell'altezza massima, W10, sono state calcolate per diversi tipi di risonanze, tutte esemplificate in spettri ottenuti con estratti di Vanilla sp - Pure Shift versus classic 1H NMR - Figura 4. Complessivamente, l'espansione delle risonanze accoppiate ha raggiunto valori di W10 da 1 a 60 Hz, mentre i picchi di singoletto variavano tra 1 e 10 Hz: il gruppo metilico dell'acido acetico (1,97 ppm in Figura 4) ha raggiunto un valore di W10 di 2,0 Hz nella classica 1H-NMR e di 2,1 Hz nello spettro SAPPHIRE-PSYCHE (S). La risonanza anomerica del saccarosio (a 5,40 ppm), un doppietto con un accoppiamento J di 3,9 Hz, si estende su una regione equivalente a un W10 di 6,5 Hz, con ogni singolo picco che rappresenta 2,6 Hz di W10 (Figura 4). Questo valore era superiore a quello del picco collassato ottenuto nello spettro SAPPHIRE-PSYCHE, W10 = 1,9 Hz (Figura 4). Nel caso dell'acido malico Hβ' (a 2,55 ppm), un doppietto di doppietti con J = 7,8 Hz, 15,6 Hz, si estende oltre i 29,4 Hz (Figura 4), con valori calcolati di W10 per ogni singolo picco compresi tra 4,7 e 4,9 Hz. Questo segnale multiplet è collassato da Pure Shift in una singola linea (a 2,55 ppm), espandendosi su un valore W10 di 4,7 Hz (Figura 4).
La molteplicità del segnale diventa più complessa negli idrogeni altamente accoppiati dove i picchi costitutivi del multipletto non sono più facilmente distinguibili, formando un segnale quasi continuo. Questo è il caso degli idrogeni dell'acido omocitrico, Hγ (a 2,24 ppm), Hδ (a 2,02 ppm) e Hδ' (a 1,91 ppm), Figura 4. I multipletti Hγ, Hδ e Hδ' con W10=38,3 Hz, W10=41,5 Hz e W10=34,8 Hz, rispettivamente, sono collassati a singoletti di W10=7,8 Hz (Hγ, 2,24 ppm), W10=8,0 Hz (Hδ, 2,02 ppm) e W10=6,2 Hz (Hδ', 1,91 ppm), Figura 4. Questa maggiore risoluzione ha permesso una migliore discriminazione di altri segnali sovrapposti corrispondenti all'acido malico (Hβ, 2,77 ppm Hβ', 2,55 ppm), all'acido omocitrico (Hα, 2,80 ppm e Hα', 2,63 ppm) e all'acido omocitrico lattone (Hα', 2,84 ppm e Hδ, 2,65 ppm) (Figura 4).
Il miglioramento della risoluzione negli spettri omonucleari disaccoppiati SAPPHIRE-PSYCHE, che consente una migliore discriminazione tra i segnali, è ulteriormente dimostrato in due regioni altamente compromesse, 4,85 - 5,08 ppm e 7,05 - 7,31 ppm, dove è stato possibile identificare importanti metaboliti coinvolti nella via metabolica della fragranza di vaniglia: glucoside A (vedi struttura in Figura 5), CH-2',6' (a 7,28 ppm), CH-2", 6" (a 7,18 ppm), CH-3',5' (a 7,09 ppm), CH-3",5" (a 7,07 ppm), CH2-7' (a 4,97 ppm e 5,05 ppm), CH2-7" (a 4,89 ppm), CH-Glc (a 4,96 ppm) e CH'-Glc (a 4,93 ppm), glucoside B (vedi struttura in Figura 4), CH-2',6' (a 7,29 ppm), CH-2",6" (a 7,23 ppm), CH-3', 5' (a 7,09 ppm), CH-3",5" (a 7,07 ppm), CH2-7' (a 4,99 ppm), CH2-7" (a 4,91 ppm), alcol idrossibenzilico (7,23 ppm) e glucoside dell'alcol idrossibenzilico (7,10 e 4,51 ppm), Figura 4.
Gli stessi risultati sono stati osservati con gli estratti di uva spina del Capo e nell'analisi del tubero di patata. In questi due casi, diverse regioni affollate su 1H-NMR regolare, PSYCHE e SAPPHIRE-PSYCHE sono state ampliate per i confronti (Figure 6 e 7).
È chiaro che gli esperimenti PSYCHE e SAPPHIRE-PSYCHE hanno chiaramente migliorato la risoluzione del segnale degli estratti di uva spina del Capo per saccarosio (4,04 ppm), β-fruttosio (4,02 ppm e 3,99 ppm), β-glucosio (3,24 ppm), prolina (2,34 ppm, 2,07 ppm e 2,00 ppm), acido glutammico (2,16 ppm) e glutammina (2,13 ppm) (Figura 6), come abbiamo mostrato nel precedente lavoro11, così come nel caso dello S. tuberosum estratti: GABA (1,91 ppm, 2,33 ppm e 3,02 ppm), acido piroglutammico (2,04 ppm, 2,41 ppm e 2,51 ppm), prolina (2,00 ppm, 2,07 ppm, 3,33 ppm e 3,42 ppm), glutammina (2,14 ppm e 2,46 ppm), valina (2,27 ppm), acido citrico (2,64 ppm e 2,76 ppm) e β-glucosio (3,25 ppm e 3,47 ppm), Figura 7.
La sequenza PSYCHE è relativamente facile da usare ed è stata implementata con successo in un'ampia gamma di applicazioni9. Per ottenere lo spettro disaccoppiato, la sequenza di impulsi acquisisce piccoli pezzi di FID rifocalizzando l'accoppiamento J al centro di ciascun blocco. Tuttavia, durante ogni blocco si verifica una piccola evoluzione dell'accoppiamento J e genera gli artefatti periodici delle bande laterali che normalmente rappresentano meno del 5% del loro picco genitore21. La presenza di questi artefatti di modulazione J , inerenti agli esperimenti PSYCHE21, è evidente nelle zone espanse della Figura 6 . Nell'analisi dei composti puri questi artefatti possono essere trascurati. Nei campioni biologici, questo potrebbe non essere il caso, perché, come mostrato nella Figura 6 per la prolina (4,13 ppm, 3,41 ppm e 3,33 ppm), l'asparagina (3,97 ppm), il mio-inositolo (3,27 ppm), il GABA (3,03 ppm) e l'acido malico (2,67 ppm) e nella Figura 6 per l'acido piroglutammico (2,04 ppm, 2,41 ppm e 2,51 ppm) e la valina (2,27 ppm), I metaboliti a concentrazioni più elevate generano artefatti grandi quanto alcuni dei segnali appartenenti a composti presenti a basse concentrazioni, compromettendo in questo modo l'accuratezza del profilo metabolico.
L'esperimento SAPPHIRE-PSYCHE è una modifica della normale sequenza PSYCHE in cui questi artefatti periodici vengono rimossi mediante modulazione di fase sistematica, ottenuta spostando il punto di rifocalizzazione J 23. Di conseguenza, l'esperimento SAPPHIRE-PSYCHE consente di garantire uno spettro di Pure Shift molto più pulito, come mostrato nella Figura 6 per la prolina (4,13 ppm, 3,41 ppm e 3,33 ppm), l'asparagina (3,97 ppm), il mio-inositolo (3,27 ppm), il GABA (3,03 ppm) e l'acido malico (2,67 ppm), nel caso dell'uva spina del Capo11, e nella Figura 7 per l'acido piroglutammico (2,04 ppm, 2,41 ppm e 2,51 ppm) e valina (2,27 ppm), nel caso delle patate.
Un altro artefatto di cui soffrono tutti gli esperimenti di Pure Shift sono quelli generati dall'effetto di accoppiamento forte. Alcuni importanti metaboliti primari come l'acido citrico (2,64 ppm e 2,76 ppm) e la glutammina (2,14 ppm e 2,46 ppm), mostrano forti artefatti di accoppiamento, come mostrato nell'estratto di tubero di patata (Figura 7). Ad oggi, non esiste una sequenza di impulsi che possa eliminare convenientemente questo problema; tuttavia, SAPPHIRE-PSYCHE si comporta meglio del normale PSYCHE 8,9,23.
Analisi della matrice di correlazione
Uno dei principali vantaggi della spettroscopia NMR è che le concentrazioni relative di metaboliti in una miscela sono direttamente proporzionali all'intensità dei loro segnali. Quindi, una matrice di correlazione a coppie di metaboliti può essere ottenuta direttamente dalla matrice di correlazione spettrale32.
La matrice di correlazione 1H-NMR, comunemente nota come STOCSY32 (Statistical Total Correlation Spectroscopy), è solitamente rappresentata come uno spettro pseudo-2D, dove ogni picco incrociato è un coefficiente di correlazione tra due segnali (Figura 8)11. STOCSY mostra un'elevata correlazione tra segnali che appartengono alla stessa molecola, ma anche con segnali provenienti da molecole che appartengono alla stessa via metabolica 11,32. Pertanto, i modelli di correlazione forniscono informazioni sullo stato fisiologico del sistema e possono quindi essere impiegati come impronta digitale dello stadio fisiologico33.
La principale limitazione di STOCSY è la sovrapposizione del segnale, che diminuisce la correlazione a coppie 11,32. Inoltre, le correlazioni di molteplicità dell'accoppiamento J portano a modelli altamente complessi, complicando ulteriormente l'analisi11,32. L'uso di SAPPHIRE-PSYCHE STOCSY, mostrato in Figura 8, migliora i valori di correlazione perché riduce le correlazioni di molteplicità dell'accoppiamento J in un unico picco, riducendo così la sovrapposizionedi 11.
Diverse regioni sono state ampliate nella Figura 8. L'acido malico mostra pattern multipli complessi J associati a Hα (4,41 ppm), Hβ (2,82 ppm) e Hβ' (2,67 ppm); in Pure Shift STOCSY questi segnali collassano in singoli picchi di correlazione (Figura 9A)11. Gli stessi risultati si osservano per il β-glucosio, dove le correlazioni intermolecolari e intramolecolari (α-glucosio, α-fruttosio e β-fruttosio) sono meglio rappresentate da SAPPHIRE-PSYCHE (Figura 9B)11. Alcuni aminoacidi mostrano anche forti correlazioni intramolecolari negli estratti di uva spina del Capo. Tuttavia, la loro identificazione nel normale 1H-NMR STOCSY è compromessa dalla sovrapposizione nelle regioni affollate. Con Pure Shift STOCSY, le correlazioni intermolecolari e intramolecolari tra prolina, alanina, glutammina e acido glutammico sono meglio rappresentate (Figure 9C e 9D)11.
Analisi multivariata
L'analisi multivariata è uno dei principali strumenti impiegati nell'affrontare i dati metabolomici34,35. Mentre la discriminazione del campione mediante PCA (analisi delle componenti principali) o PLS-DA (analisi discriminante dei minimi quadrati parziali) potrebbe essere facilmente ottenuta attraverso spettri regolari 1H-NMR, l'interpretazione del carico è meglio affrontata attraverso i dati di puro spostamento 11,24,25,27,28.
Nella Figura 10, mostriamo il grafico del punteggio PLS-DA ottenuto utilizzando SAPPHIRE-PSYCHE (Figura 10A) e spettri di protoni regolari (Figura 10B) nella discriminazione di sei ecotipi di estratti di P. peruviana 11. Anche se ci sono alcuni studi che sostengono migliori prestazioni di discriminazione quando si utilizzano i dati Pure Shift24,25, i nostri risultati mostrano che le prestazioni sono state a malapena influenzate dal disaccoppiamento omonucleare11. Nel caso dell'analisi dei dati di carico, come mostrato nelle Figure 10C e 10D, l'aumento della risoluzione raggiunto con i dati SAPPHIRE-PSYCHE ha semplificato l'analisi e ha permesso una migliore identificazione dei metaboliti specifici responsabili della discriminazione di P. peruviana, vale a dire, α-glucosio, β-glucosio, α-fruttosio, β-fruttosio, saccarosio, acido citrico e alanina11. Questo guadagno di risoluzione è stato fondamentale anche quando si combina l'analisi dei coefficienti PLS con la correlazione STOCSY, Figure 10C e 10D11. La forte correlazione tra α-glucosio (il vettore STOCSY a 5,23 ppm è codificato a colori sul primo componente PLS) e β-glucosio, α-fruttosio e β-fruttosio - tutti metaboliti che evolvono dalla stessa via metabolica - e la sua anti-correlazione rispetto al saccarosio, sono evidenti11. Con la normale analisi STOCSY, l'ampia sovrapposizione non ha permesso una chiara rappresentazione tra i coefficienti di correlazione e ha prodotto la perdita di questa particolare informazione sulla via metabolica (Figura 10)11.
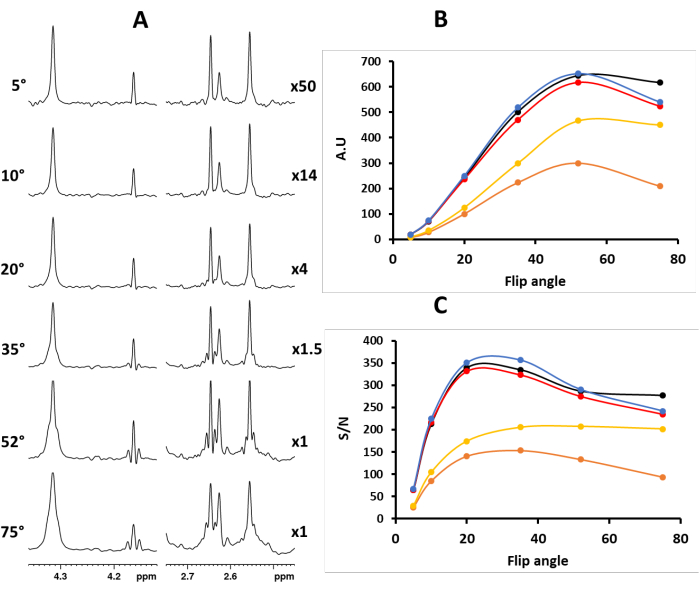
Figura 1. (A) Spettri PSYCHE di estratti di Vanilla planifolia utilizzando diversi angoli di inversione dell'impulso CHIRP per l'elemento PSYCHE: valori dell'angolo di inversione a sinistra; a destra, fattore di moltiplicazione dell'intensità. I grafici (B) e (C) mostrano l'intensità e il rapporto segnale/rumore di cinque picchi disaccoppiati (nero - 4,31 ppm, arancione - 4,16 ppm, rosso - 2,65 ppm, giallo - 2,63 ppm e blu 2,56 ppm) in funzione dell'angolo di inversione dell'impulso PSYCHE CHIRP, rispettivamente. Il rapporto segnale/rumore (S/N) (più precisamente, rapporto segnale/rumore + artefatto) è stato calcolato utilizzando il segnale di massima intensità in contrapposizione a quello del rumore, valore calcolato su un intervallo di 2 ppm: da 7,75 ppm a 9,75 ppm (Figura S12). Clicca qui per visualizzare una versione più grande di questa figura.
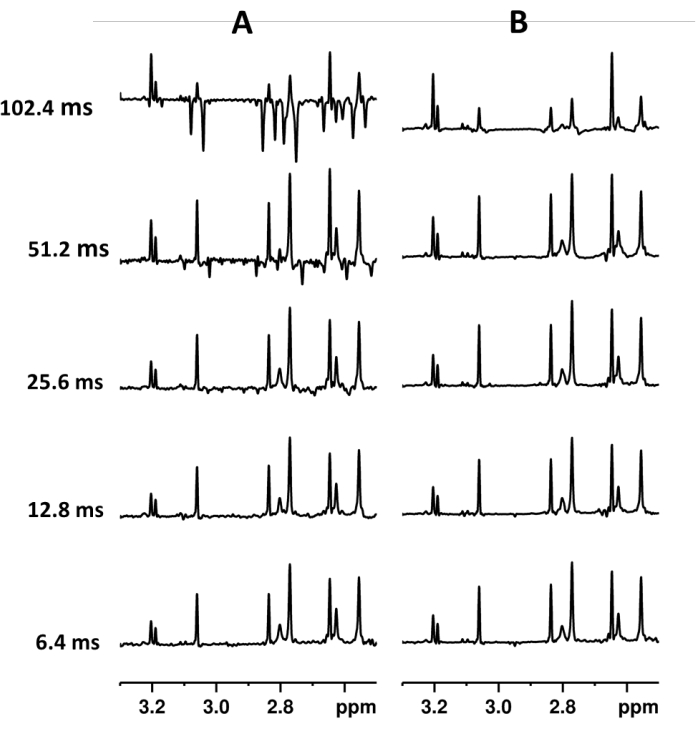
Figura 2. Spettri PSYCHE (A) e SAPPHIRE-PSYCHE (B) di estratti di Vanilla planifolia con diversa durata del blocco Pure Shift dell'interferogramma. Gli esperimenti sono stati acquisiti in modo da mantenere la stessa risoluzione e sensibilità digitale. Parametro PSYCHE (A) dal basso verso l'alto: 128 scansioni, 64 blocchi di interferogramma, lunghezza del blocco 6,4 ms, tempo totale di acquisizione 6h07min; 128 scansioni, 32 blocchi di interferogramma, lunghezza del blocco 12,8 ms, tempo totale di acquisizione 3h04min; 128 scansioni, 16 blocchi di interferogramma, lunghezza del blocco 25,6 ms, tempo totale di acquisizione 1h32min; 128 scansioni, 8 blocchi di interferogramma, lunghezza del blocco 51,2 ms, tempo totale di acquisizione 46min; 128 scansioni, 4 blocchi di interferogramma, lunghezza del blocco 102,4 ms, tempo di acquisizione totale 24min. Parametro SAPPHIRE-PSYCHE (B) dal basso verso l'alto: 16 scansioni, incrementi di modulazione di 8 J , 65 blocchi di interferogramma, lunghezza del blocco 6,4 ms, tempo di acquisizione totale 6h19min; 16 scansioni, 8 incrementi di modulazione J , 33 blocchi di interferogramma, lunghezza del blocco 12,8 ms, tempo totale di acquisizione 3h13min; 16 scansioni, incrementi di modulazione 8 J , 17 blocchi di interferogramma, lunghezza del blocco 25,6 ms, tempo totale di acquisizione 1h40min; 16 scansioni, 8 incrementi di modulazione J , 9 blocchi di interferogramma, lunghezza del blocco 51,2 ms, tempo totale di acquisizione 53 minuti; 16 scansioni, 8 incrementi di modulazione J , 5 blocchi di interferogramma, lunghezza del blocco 102,4 ms, tempo di acquisizione totale 30 minuti. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3. (A) spettri di estratti di Vanilla planifolia con lunghezza del pezzo di 102,4 ms: in alto, 128 scansioni PSYCHE, 4 incrementi di Pure Shift; parte superiore centrale, 4 scansioni, 4 incrementi di Pure Shift, 32 incrementi di SAPPHIRE; fondo centrale, 8 scansioni, 4 incrementi di Pure Shift, 16 incrementi di SAPPHIRE; in basso, 16 scansioni, 4 incrementi di Pure Shift, 16 incrementi di SAPPHIRE. (B) spettri di estratti di Vanilla planifolia con lunghezza del pezzo di 51,2 ms: in alto, PSYCHE 128 scansioni, 8 incrementi di Pure Shift; parte superiore centrale, 4 scansioni, 9 incrementi di Pure Shift, 32 incrementi di SAPPHIRE; fondo centrale, 8 scansioni, 9 incrementi di Pure Shift, 16 incrementi di SAPPHIRE; in basso, 16 scansioni, 9 incrementi di Pure Shift, 16 incrementi di SAPPHIRE. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4. Regioni di espansione selezionate degli spettri 1H NMR (1H) e SAPPHIRE-PSYCHE (S) V . planifolia (1.85 - 2.9 ppm) e V. pompona (4.85 - 7.31 ppm), che mostrano l'assegnazione dei segnali. Clicca qui per visualizzare una versione più grande di questa figura.
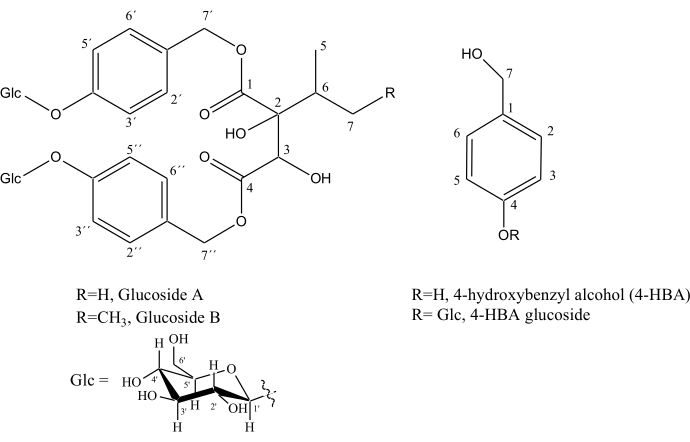
Figura 5. Strutture dei precursori delle fragranze alla vaniglia: alcool 4-idrossibenzilico (4-HBA), 4-HBA glucoside, glucoside A e glucoside B. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6. Regioni di espansione selezionate di spettri 1H NMR (1H), PSYCHE (P) e SAPPHIRE (S) di un estratto acquoso di uva spina del Capo (regione andina peruviana28 di Bambamarca I) che mostrano assegnazioni di segnale (ristampato con il permesso di Lopez et al.11 ). Clicca qui per visualizzare una versione più grande di questa figura.
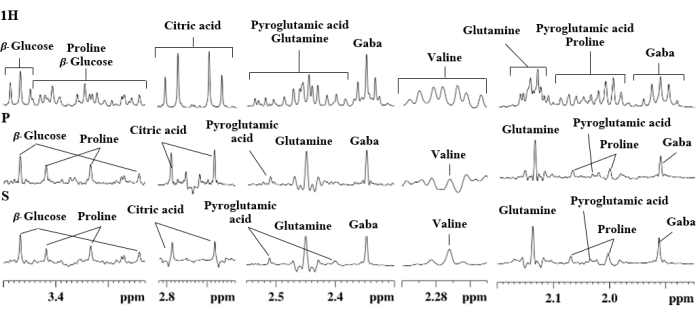
Figura 7. Regioni di espansione selezionate degli spettri 1H NMR (1H), PSYCHE (P) e ZAFFIRO (S) di un estratto acquoso di patata che mostrano l'assegnazione del segnale. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 8. Regioni espanse selezionate (3,20 ppm-4,30 ppm) di spettri NMR STOCSY bidimensionali ottenuti con i dati di sei estratti di uva spina del Capo che mostrano valori di correlazione (r2) superiori a 0,85: (A) 1 H NMR STOCSY regolare e(B) SAPPHIRE-PSYCHE STOCSY. Ristampato con il permesso di Lopez et al.11 Clicca qui per visualizzare una versione più grande di questa figura.
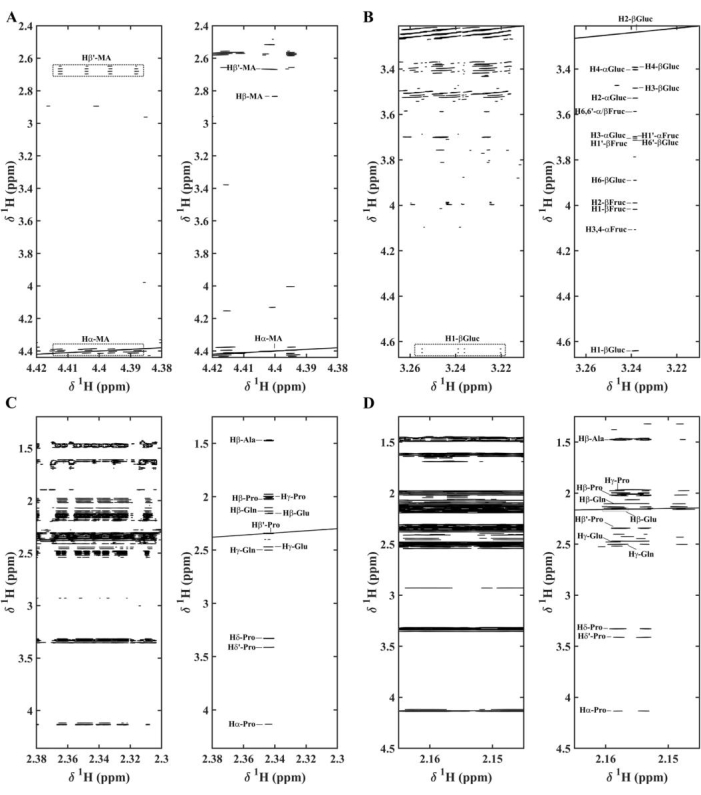
Figura 9. Rappresentazioni STOCSY degli spettri NMR di sei diversi estratti di uva spina del Capo che mostrano correlazioni (r2), a sinistra senza omodisaccoppiamento e a destra con omodisaccoppiamento per le regioni: (A) 4,38-4,42 ppm e 2,40-4,42 ppm con r2 superiore a 0,80 per il segnale dell'acido malico (MA) (Hα-MA); (B) 3,21-3,27 ppm e 3,21-4,67 ppm con r2 superiore a 0,85 per il segnale β-glucosio (H2-β-Gluc); (C) 2,30-2,38 ppm e 1,25-4,36 con r2 superiore a 0,93 per il segnale della prolina (Pro) (Hβ'-Pro); (D) 2,15-2,17 ppm e 1,25-4,5 ppm con r2 superiore a 0,90 per il segnale dell'acido glutammico (Glu) (Hβ-Glu); α-glucosio, α-fruttosio, β-fruttosio sono simboleggiati rispettivamente come α-Gluc, α-Fruc e β-Frutto. Ristampato con il permesso di Lopez et al.11 Clicca qui per visualizzare una versione più grande di questa figura.

Figura 10. Punteggi PLS grafici di estratti di uva spina del Capo coltivati in sei diverse regioni andine11,28 (San Marcos: cerchi rossi, Celendin III: triangoli marroni, Bambamarca I: stelle blu, Celendin I: triangoli gialli, Bambamarca II: quadrati verdi, Celendin II: diamanti magenta) sulla base di (A) esperimenti NMR classici 1 H e (B) Zaffiro-Psiche. Le ellissi T2 di Hotelling sono state impostate su un livello di confidenza del 95%. Combinazione di carichi PLS1 e 1D STOCSY per la correlazione α-glucosio utilizzando il segnale STOCSY a 5,23 ppm come picco del driver. I coefficienti di determinazione (r2) sono stati codificati a colori e proiettati sui coefficienti del primo componente PLS: (A) STOCSY 1D ottenuto con i dati SAPPHIRE-PSYCHE (in alto) e la sua espansione 3,15-4,17 ppm (in basso); (B) STOCSY 1D ottenuto con dati NMR 1H (in alto) e la sua espansione 3.15-4.17 ppm (in basso); α-glucosio, β-glucosio, α-fruttosio, β-fruttosio e saccarosio sono simboleggiati rispettivamente come α-G, β-G, α-F, β-F e S. L'analisi PLS-DA e STOCSY è stata eseguita utilizzando MATLAB versione R2018a. (Ristampato con il permesso di Lopez et al.11) Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
L'identificazione strutturale e la quantificazione dei metaboliti sono questioni chiave nella caratterizzazione del metaboloma, dati che, sottoposti ad analisi multivariate, permettono di comprendere meglio il sistema biologico oggetto di studio. La preparazione dei campioni e l'acquisizione dei dati sono aspetti critici che devono essere ottimizzati per fornire risultati affidabili.
In questo articolo, descriviamo e illustriamo la preparazione del campione per l'analisi NMR di tre diverse matrici vegetali. Come per qualsiasi procedura di estrazione, la quantità di solvente per grammo di materiale e le proprietà fisiche del solvente selezionato determineranno la composizione chimica dell'estratto finale e la concentrazione dei metaboliti estratti. Nel caso della profilazione metabolomica NMR, anche il pH, la riproducibilità tra estrazioni indipendenti del campione e la quantità finale di estratto nella provetta NMR sono aspetti che richiedono un'ottimizzazione. L'importanza della riproducibilità in metabolomica è quella di evitare l'introduzione di varianza non correlata, che potrebbe portare a risultati inaffidabili. In base alla nostra esperienza, le condizioni di estrazione ottimali sono state raggiunte con il materiale vegetale secco e macinato. Nel caso dell'uva spina del Capo, il prodotto secco era molto difficile da maneggiare (altamente igroscopico), quindi le bacche fresche sono state prima omogeneizzate, prima della liofillizzazione.
Nel caso dell'acquisizione di spettri, la configurazione sperimentale Pure Shift è di particolare importanza, poiché parametri errati possono portare a suddividere e riaccoppiare gli artefatti. La teoria alla base dei principi degli esperimenti di Pure Shift, ampiamente esaminata altrove 8,9,10, è importante per capire come configurare correttamente la sequenza di impulsi e implementarla come un esperimento di routine.
In breve, la maggior parte degli esperimenti di Pure Shift si basano sulla rifocalizzazione dell'evoluzione dell'accoppiamento J durante la registrazione dello spostamento chimico. Ciò è tipicamente ottenuto da un elemento di rifocalizzazione dell'accoppiamento J che inverte selettivamente gli spin "passivi", mentre gli spin "attivi" rimangono inalterati. PSYCHE e SAPPHIRE-PSYCHE si basano su un esperimento anti-z-COSY in cui gli spin "passivi" sono statisticamente invertiti.
L'elemento PSYCHE, che consiste in due impulsi a basso angolo di inversione a frequenza spazzata in presenza di un debole gradiente di campo pulsato, induce la media spaziotemporale di frequenza, selezionando i termini COSY anti-diagonali e sopprimendo i termini quantistici e di correlazione incrociata pari a zero. Di conseguenza, per evitare gli artefatti di riaccoppiamento intrinseci, l'angolo di inversione dell'impulso CHIRP deve essere corto (Figura 1). In genere, un angolo di rotazione di 20° è un buon compromesso tra sensibilità e prestazioni di disaccoppiamento (Figura 1). Pertanto, la calibrazione degli impulsi è fondamentale per la qualità e la sensibilità dello spettro.
Gli studi metabolomici sono solitamente associati alla registrazione degli spettri su un gran numero di campioni, il che implica che la calibrazione degli impulsi deve essere rapida o automatica. Nella nostra esperienza, se i campioni vengono preparati esattamente nello stesso modo, la variabilità della lunghezza dell'impulso duro tra i campioni è inferiore a ± 0,2 μs. Normalmente calibriamo tra i 6 e i 12 campioni e poi utilizziamo il valore medio raggiunto per impostare l'intero gruppo di campioni. Nel caso in cui la variabilità della lunghezza dell'impulso da campione a campione fosse maggiore, la calibrazione automatica di ciascun campione deve essere eseguita utilizzando il programma di automazione pulsata Topspin sullo spettrometro Bruker.
Il secondo parametro importante da considerare è la lunghezza dei blocchi registrati durante l'acquisizione dell'interferogramma a gradini21,23. L'acquisizione dell'interferogramma consiste nel registrare il FID per piccoli blocchi, con il punto di rifocalizzazione dell'evoluzione dell'accoppiamento J sempre coincidente con il centro del pezzo acquisito. Il FID disaccoppiato viene costruito concatenando ogni blocco successivo 8,9,10. Per garantire che l'acquisizione del blocco non tronchi l'evoluzione dello spostamento chimico, l'inizio di ogni registrazione di dati deve corrispondere esattamente alla fine del blocco precedente.
Sebbene questa procedura ci permetta di ottenere uno spettro omodisaccoppiato, la piccola evoluzione dell'accoppiamento J durante ogni blocco genera artefatti periodici di bande laterali direttamente dipendenti dalla lunghezza del blocco. D'altra parte, la risoluzione digitale spettrale dipende dalla finestra spettrale e dalla durata totale del FID disaccoppiato, che, a sua volta, dipende dalla lunghezza del blocco e dal numero di blocchi registrati. Pertanto, per ridurre gli artefatti periodici senza sacrificare la risoluzione, la durata del blocco deve essere breve e il numero totale di blocchi registrati deve essere elevato. Queste condizioni, tuttavia, aumenteranno notevolmente il tempo totale di acquisizione sperimentale senza aumentare la sensibilità (Figura 2). Tipicamente, un esperimento PSYCHE acquisito con 16-32 blocchi di durata compresa tra 10 e 16 ms fornisce una risoluzione digitale sufficiente in un tempo sperimentale ragionevole (da 30 minuti a 5 ore) (Figura 2).
Nel caso di SAPPHIRE-PSYCHE, un esperimento che viene acquisito come pseudo 3D, una delle dimensioni indirette codifica per l'acquisizione dell'interferogramma Pure Shift e l'altra per la modulazione di fase degli artefatti periodici attraverso lo spostamento sistematico del punto di rifocalizzazione J in ogni pezzo.
Poiché gli artefatti periodici sono fortemente soppressi da SAPPHIRE, le lunghezze dei blocchi potrebbero essere più lunghe; tuttavia, blocchi molto lunghi influenzano fortemente l'intensità dei segnali (Figure 2 e 3). In SAPPHIRE, gli incrementi di modulazione J contribuiscono alla sensibilità dello spettro, pertanto il numero totale di scansioni FID disaccoppiate risultante è uguale a TD2 * NS (Figure 2 e 3)23. In generale, otto incrementi di modulazione J assicurano un'eccellente soppressione periodica degli artefatti e più di otto incrementi hanno un effetto molto limitato sulla qualità dello spettro, anche se vengono utilizzate lunghe lunghezze di chunk (Figura 3)23. Gli incrementi Pure Shift di 33 o 17 con durate comprese tra 20 e 40 ms garantiscono una buona risoluzione digitale spettrale.
Una limitazione di entrambe queste sequenze di impulsi Pure Shift, PSYCHE e SAPPHIRE-PSYCHE, è la quantificazione mediante integrazione standard assoluta di metaboliti interni. Nella normale 1H-NMR, l'intensità integrata è direttamente proporzionale alla concentrazione di ciascun metabolita. In PSYCHE non è più così, perché una serie di fenomeni distorcono i segnali e influenzano l'integrazione. Ad esempio, il valore integrale totale diminuisce a causa del rilassamento T2 durante la selezione dello spin della sequenza di impulsi. Inoltre, l'evoluzione troncata dell'accoppiamento J durante l'acquisizione del chunk che genera artefatti di bande laterali, sconvolge la forma lorentziana del segnale. Quindi, l'integrale è ora composto da aree sotto il picco principale e sotto tutte le bande laterali, complicando l'integrazione del segnale 8,9,21,23. La frequenza e l'ampiezza delle bande laterali sono direttamente correlate alla lunghezza del pezzo ma anche a proprietà intrinsecamente molecolari come il rilassamento e la grandezza e la molteplicità dell'accoppiamento J: grandezze di accoppiamento J più elevate e molteplicità più elevate, portano a segnali più distorti. Nel caso di SAPPHIRE, anche se questo esperimento NMR rimuove efficacemente gli artefatti della banda laterale, le intensità del segnale sono compromesse dall'evoluzione troncata dell'accoppiamento J. La somma di ogni incremento modulato J genera un FID disaccoppiato medio in cui la diminuzione del segnale è direttamente correlata alla lunghezza del blocco e all'ampiezza e alla molteplicità dell'accoppiamento J 23. Inoltre, gli angoli di inversione dell'impulso CHIRP generano artefatti di riaccoppiamento che influenzano anche ciascun segnale in modo diverso, complicando ulteriormente la quantificazione21. L'entità dell'effetto di queste sequenze di impulsi nell'analisi quantitativa è stata valutata nel nostro precedente studio sull'uva spina del Capo, che ha prodotto errori compresi tra il 10% e il 30%11.
Infine, possiamo concludere che Pure Shift è un nuovo eccellente strumento per la metabolomica delle piante, poiché aumenta drasticamente la risoluzione dello spettro, consentendo un'analisi della matrice di correlazione più fine e una migliore interpretazione delle analisi multivariate 11,24,25,27,28.
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
Questo studio è stato finanziato dal Consejo Nacional de Ciencia, Tecnología e Innovación Tecnológica (CONCYTEC) - Programa Atracción de Investigadores Cienciactiva - Contract # 008-2017-FONDECYT.
Materiali
| Name | Company | Catalog Number | Comments |
| 77500 Series Freezone 4.5 Liter benchtop | Labconco | 77500 | |
| Bruker Avance III 500 MHz equiped with a 5 mm TCI Z-gradient cryogenic probe | Bruker Corporation | ||
| Centrivap Refrigerated Centrifugal Concentrators Labconco 7310000 Series | Labconco | 7310000 | |
| Deuterium oxide | Sigma-Aldrich | 151882 | |
| Grinder machine MKM6003 | Bosch | MKM6003 | |
| Licuadora Blender 8011S model Hgb2wts3 | Waring | Hgb2wts3 | |
| Methanol-d4 | Sigma-Aldrich | 151947 |
Riferimenti
- Hall, R., Beale, M., Fiehn, O., Hardy, N., Sumner, L., Bino, R. Plant metabolomics: the missing link in functional genomics strategies. The Plant Cell. 14 (7), 1437-1440 (2002).
- Fiehn, O. Metabolomics-the link between genotypes and phenotypes. Plant Molecular Biology. 48 (1-2), 155-171 (2002).
- Schauer, N., Fernie, A. R. Plant metabolomics: towards biological function and mechanism. Trends in Plant Science. 11 (10), 508-516 (2006).
- Kim, H. K., Choi, Y. H., Verpoorte, R. NMR-based plant metabolomics: where do we stand, where do we go. Trends in Biotechnology. 29 (6), 267-275 (2011).
- Kumar, R., Bohra, A., Pandey, A. K., Pandey, M. K., Kumar, A. Metabolomics for Plant Improvement: Status and Prospects. Frontiers in Plant Science. 8, (2017).
- Dumez, J. -. N., et al. Hyperpolarized NMR of plant and cancer cell extracts at natural abundance. Analyst. 140 (17), 5860-5863 (2015).
- Emwas, A. -. H., et al. NMR Spectroscopy for Metabolomics Research. Metabolites. 9 (7), (2019).
- Zangger, K. Pure shift NMR. Progress in Nuclear Magnetic Resonance Spectroscopy. 86-87, 1-20 (2015).
- Foroozandeh, M., Morris, G. A., Nilsson, M. PSYCHE Pure Shift NMR Spectroscopy. Chemistry - A European Journal. 24 (53), 13988-14000 (2018).
- Castañar, L. Pure shift NMR: Past, present, and future. Magnetic Resonance in Chemistry. 56 (10), 874-875 (2018).
- Lopez, J. M., Cabrera, R., Maruenda, H. Ultra-Clean Pure Shift 1 H-NMR applied to metabolomics profiling. Scientific Reports. 9 (1), 1-8 (2019).
- Marcó, N., Gil, R. R., Parella, T. Isotropic/Anisotropic NMR Editing by Resolution-Enhanced NMR Spectroscopy. Chemphyschem: A European Journal of Chemical Physics and Physical Chemistry. 19 (9), 1024-1029 (2018).
- Kaltschnee, L., et al. Extraction of distance restraints from pure shift NOE experiments. Journal of Magnetic Resonance. 271, 99-109 (2016).
- Sinnaeve, D., et al. Improved Isotopic Profiling by Pure Shift Heteronuclear 2D J-Resolved NMR Spectroscopy. Analytical Chemistry. 90 (6), 4025-4031 (2018).
- Timári, I., et al. Real-Time Pure Shift HSQC NMR for Untargeted Metabolomics. Analytical Chemistry. 91 (3), 2304-2311 (2019).
- Zhao, Q., et al. Combination of pure shift NMR and chemical shift selective filters for analysis of Fischer-Tropsch waste-water. Analytica Chimica Acta. 1110, 131-140 (2020).
- Zhao, Q., et al. Pure Shift NMR: Application of 1D PSYCHE and 1D TOCSY-PSYCHE Techniques for Directly Analyzing the Mixtures from Biomass-Derived Platform Compound Hydrogenation/Hydrogenolysis. ACS Sustainable Chemistry & Engineering. 9 (6), 2456-2464 (2021).
- Foroozandeh, M., et al. Ultrahigh-Resolution Diffusion-Ordered Spectroscopy. Angewandte Chemie International Edition. 55 (50), 15579-15582 (2016).
- Castañar, L., Pérez-Trujillo, M., Nolis, P., Monteagudo, E., Virgili, A., Parella, T. Enantiodifferentiation through Frequency-Selective Pure-Shift 1H Nuclear Magnetic Resonance Spectroscopy. ChemPhysChem. 15 (5), 854-857 (2014).
- Lopez, J. M., Sánchez, L. F., Nakamatsu, J., Maruenda, H. Study of the Acetylation Pattern of Chitosan by Pure Shift NMR. Analytical Chemistry. , (2020).
- Foroozandeh, M., Adams, R. W., Meharry, N. J., Jeannerat, D., Nilsson, M., Morris, G. A. Ultrahigh-Resolution NMR Spectroscopy. Angewandte Chemie International Edition. 53 (27), 6990-6992 (2014).
- Foroozandeh, M., Adams, R. W., Kiraly, P., Nilsson, M., Morris, G. A. Measuring couplings in crowded NMR spectra: pure shift NMR with multiplet analysis. Chemical Communications. 51 (84), 15410-15413 (2015).
- Moutzouri, P., et al. Ultraclean pure shift NMR. Chemical Communications. 53 (73), 10188-10191 (2017).
- Santacruz, L., Hurtado, D. X., Doohan, R., Thomas, O. P., Puyana, M., Tello, E. Metabolomic study of soft corals from the Colombian Caribbean: PSYCHE and 1 H-NMR comparative analysis. Scientific Reports. 10 (1), 5417 (2020).
- Stark, P., Zab, C., Porzel, A., Franke, K., Rizzo, P., Wessjohann, L. A. PSYCHE-A Valuable Experiment in Plant NMR-Metabolomics. Molecules. 25 (21), 5125 (2020).
- Kakita, V. M. R., Rachineni, K., Hosur, R. V. Ultraclean Pure Shift NMR Spectroscopy with Adiabatic Composite Refocusing Pulses: Application to Metabolite Samples. ChemistrySelect. 4 (34), 9893-9896 (2019).
- Bo, Y., et al. High-resolution pure shift NMR spectroscopy offers better metabolite discrimination in food quality analysis. Food Research International. 125, 108574 (2019).
- Watermann, S., Schmitt, C., Schneider, T., Hackl, T. Comparison of Regular, Pure Shift, and Fast 2D NMR Experiments for Determination of the Geographical Origin of Walnuts. Metabolites. 11 (1), 39 (2021).
- Leyva-Zegarra, V., et al. NMR-based leaf metabolic profiling of V. planifolia and three endemic Vanilla species from the Peruvian Amazon. Food Chemistry. , 129365 (2021).
- Toubiana, D., et al. Morphological and metabolic profiling of a tropical-adapted potato association panel subjected to water recovery treatment reveals new insights into plant vigor. The Plant Journal. 103 (6), 2193-2210 (2020).
- Maruenda, H., Cabrera, R., Cañari-Chumpitaz, C., Lopez, J. M., Toubiana, D. NMR-based metabolic study of fruits of Physalis peruviana L. grown in eight different Peruvian ecosystems. Food Chemistry. 262, 94-101 (2018).
- Cloarec, O., et al. Statistical total correlation spectroscopy: an exploratory approach for latent biomarker identification from metabolic 1H NMR data sets. Analytical Chemistry. 77 (5), 1282-1289 (2005).
- Steuer, R. Review: on the analysis and interpretation of correlations in metabolomic data. Briefings in Bioinformatics. 7 (2), 151-158 (2006).
- Trygg, J., Holmes, E., Lundstedt, T. Chemometrics in Metabonomics. Journal of Proteome Research. 6 (2), 469-479 (2007).
- Worley, B., Powers, R. Multivariate Analysis in Metabolomics. Current Metabolomics. 1 (1), 92-107 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon