Method Article
Reine Shift-Kernspinresonanz: ein neues Werkzeug für die pflanzliche Metabolomik
In diesem Artikel
Zusammenfassung
Dieser Artikel stellt die Verwendung von PSYCHE und SAPPHIRE-PSYCHE bei der metabolischen Profilerstellung von Pflanzen vor und enthält detaillierte Verfahren zur Probenvorbereitung und optimalen Aufzeichnung von Pure Shift NMR-Spektren. Es werden Beispiele diskutiert, anhand derer der durch die homonukleare Entkopplung erzielte Auflösungsgewinn ein umfassenderes Verständnis des Systems ermöglicht.
Zusammenfassung
Die Kernspinresonanz (NMR) ist eines der leistungsfähigsten Werkzeuge, die in der Metabolomik eingesetzt werden. Es handelt sich um eine hochgenaue und reproduzierbare Methode, die nicht nur quantitative Daten liefert, sondern auch die strukturelle Identifizierung der in komplexen Gemischen vorhandenen Metaboliten ermöglicht.
Die Erstellung von metabolischen Profilen durch 1-H-NMR hat sich bei der Untersuchung verschiedener Arten von Pflanzenszenarien als nützlich erwiesen, darunter die Bewertung von Erntebedingungen, Ernte- und Nacherntebehandlungen, metabolische Phänotypisierung, Stoffwechselwege, Genregulation, Identifizierung von Biomarkern, Chemotaxonomie, Qualitätskontrolle, Herkunftsbezeichnung und andere. Die Signalüberlappung der großen Anzahl von Resonanzen mit erweiterten J-Kopplungs-Multiplizitäten erschwert jedoch die Spektrenanalyse und deren Interpretation und stellt eine Einschränkung für die klassische 1-H-NMR-Profilierungdar.
In den letzten zehn Jahren wurden neuartige NMR-Breitband-homonukleare Entkopplungstechniken entwickelt, bei denen Multiplett-Signale in einzelne Resonanzlinien kollabieren - allgemein als Pure-Shift-Methoden bezeichnet - um das Problem der Spektrenauflösung zu überwinden, das klassischen 1-H-NMR-Spektren innewohnt.
Hier wird ein Schritt-für-Schritt-Protokoll der Pflanzenextraktherstellung und das Verfahren zur Aufzeichnung optimaler Pure Shift PSYCHE- und SAPPHIRE-PSYCHE-Spektren in drei verschiedenen Pflanzenmatrices - Vanilleblättern , Kartoffelknollen (S. tuberosum) und Kapstachelbeeren (P. peruviana) - vorgestellt. Der Effekt des Auflösungsgewinns bei der metabolischen Identifizierung, Korrelationsanalyse und multivariaten Analyse im Vergleich zu klassischen Spektren wird diskutiert.
Einleitung
Die Gesamtheit der Metaboliten, aus denen ein Organismus besteht - Substrate, Zwischenprodukte und Endprodukte biologischer Prozesse - wurde 1998 mit dem Begriff Metabolom geprägt. Es ist allgemein bekannt, dass das Metabolom eng mit dem Phänotyp verwandt ist, und es ist für Pflanzen von besonderem Interesse, da es die direkte Wechselwirkung zwischen dem Genotyp und der Umwelt widerspiegelt 1,2. Daher ist die Charakterisierung des metabolomischen Profils bei Pflanzen von größter Bedeutung geworden. Durch die Identifizierung und Quantifizierung von Biomarkern (Schlüsselmetaboliten) und Stoffwechselmustern war die Unterscheidung zwischen Arten, Sorten, Entwicklungsstadien, pathogenen Krankheiten oder Umweltbedingungen (tägliche und saisonale Veränderungen, Böden, Wasserstress, mechanischer Stress, Ernte- und Nacherntebehandlungen) möglich 3,4,5.
Die Massenspektrometrie (MS) und die Kernspinresonanzspektroskopie (NMR) sind die am weitesten verbreiteten analytischen Plattformen für diesen Zweck. Im Gegensatz zu MS-Methoden ist die NMR eine hochgradig reproduzierbare, unverzerrte, quantitative, genaue und zerstörungsfreie Technik, die nur eine minimale Probenvorbereitung erfordert und daher für Metabolomik-Studien geeignet ist. Im Vergleich zu MS-Methoden stellt jedoch die inhärent geringe Sensitivität eine Einschränkung dar. In den letzten Jahren und durch den Einsatz von Hochfeldmagneten, kryogenen Sonden, Mikrospulen und Methoden der dynamischen Kernpolarisation (DNP) wurde die Empfindlichkeit der NMR stark verbessert. Im Falle des letztgenannten Ansatzes lag der Sensitivitätsgewinn beispielsweise in der Größenordnung vonzwei bis drei Größenordnungen 6,7. Bis heute sind fast 20% der veröffentlichten Metabolomik-Studien NMR-basiert, Tendenzsteigend 7.
Obwohl die Protonen-NMR das beliebteste und empfindlichste Experiment für das NMR-Metabolomik-Fingerabdruck ist, hat es einige Nachteile. Zunächst werden alle inder Probe detektierten 1H-NMR-Signale in einem kleinen Fenster verteilt, das dem chemischen Verschiebungsfenster der Protonen entspricht, was zu überfüllten Spektren führt. Zweitens spaltet die homonukleare skalare Kopplung die Signale in mehrere Komponenten auf (Signalvielfalt), wodurch das Protonensignal über einen breiteren Frequenzbereich verteilt wird, was die Spektrenlesung durch zunehmende Überfüllung und Signalüberlappung weiter erschwert. Darüber hinaus wird die NMR-Metabolomik bei der Analyse von Gemischen eingesetzt, die in der Regel 50 bis 300 Moleküle bei einer beobachtbaren NMR-Konzentration enthalten, wodurch komplexe Spektren aus 200 bis 2000 Peaks erzeugt werden.
Die homonukleare Entkopplungsprotonen-NMR, auch bekannt als Pure Shift, ist eine Methode, die den Kollaps eines Multiplett-Signals in einen einzigen Peak induziert. Es stellt ein hervorragendes Werkzeug zur Erhöhung der Signalauflösung in überfüllten Spektren 8,9,10 dar und stellt daher ein geeignetes Werkzeug für die Metabolomik von Pflanzendar 11.
In den letzten zehn Jahren sind neue Pure Shift-Pulssequenzen entstanden, die sowohl die Empfindlichkeit als auch die Entkopplungsleistung erhöhen. Auch ihr Anwendungsspektrum hat sich erweitert, unter anderem von der Molekularstrukturaufklärung12,13 über die Fluxomik14, die Mischungszuordnung 15,16,17, die translationale Diffusionsmessung18, die enantiomere Diskriminierung 19 bis hin zur Einheitenverteilung in Copolymeren20.
In der Vergangenheit litten Breitband-Pure-Shift-Experimente unter geringer Sensitivität und komplizierten Verarbeitungsmethoden, was ihren Anwendungsbereich bei der Bewertung biologischer Extrakte einschränkte8. Im Jahr 2014 veröffentlichten Foroozandeh et al. ein neues Pure-Shift-Experiment, PSYCHE (Pure Shift Yielded by Chirp Excitation), das auf einer anti-z-COSY-Pulssequenz basierte, die eine hervorragende homonukleäre Entkopplung und verbesserte Empfindlichkeitswerte ergab21. Da es sich bei PSYCHE jedoch um ein 2D-Interferogramm-Experiment handelt, bei dem Blöcke von Zeitbereichsdaten erfasst werden, leidet es unter periodischen Seitenbandartefakten, die aus J-Kopplungsmodulationsverzerrungen an den Rändern des Chunks resultieren. In komplexen Gemischen liefern diese Artefakte Signale, die größer sind als diejenigen, die mit Metaboliten in sehr niedrigen Konzentrationen verbunden sind, was die Analyse behindert11. Es gibt zwei Methoden, um diese Artefakte zu entfernen - TSE-PHYCHE22 und eine neuere Modifikation des PSYCHE-Experiments namens SAPPHIRE-PSYCHE (Sideband Averaging by Periodic PHase Incrementation of Residual J Evolution)23.
Im Jahr 2019 haben wir zum ersten Malgezeigt 11, dass die SAPPHIRE-PSYCHE Pure Shift-Methode, die Artefakte fast ohne Sensitivitätseinbußen23 entfernt, für die Analyse komplexer biologischer Gemische eingesetzt werden kann, wie z. B. Extrakte aus den Früchten von Physalis peruviana, allgemein bekannt als Kapstachelbeeren11. Wir zeigten, dass diese Methoden die Leistung von Metabolomik-Datenanalysen wie der metabolischen Zuordnung, der Korrelationsanalyse und der multivariaten Koeffizientenanalyse11 erhöhen. Seitdem wurden mehrere Pure Shift-Metabolomik-Studien an verschiedenen biologischen Matrices wie Weichkorallen24, Hypericum-Pflanzen25, Honig26,27, Tee27, Pfefferminzöl26 und Walnüssen28 durchgeführt, was seine Bedeutung als neues Werkzeug für die Metabolomik-Analyse zeigt. Paradoxerweise wurde bei der überwiegenden Mehrheit dieser Studien die standardmäßige und einfach zu implementierende PSYCHE-Pulssequenz verwendet, die in jeder Spektrometerbibliothek verfügbar ist, anstelle der SAPPHIRE-PSYCHE-Pulssequenz, die sich als leistungsfähiger erwiesen hat. Für die richtige Einrichtung ist jedoch ein besseres Verständnis der Impulssequenz erforderlich.
Diese Arbeit soll neuen Anwendern helfen, Pure-Shift-Methoden bei der Untersuchung von Pflanzen anzuwenden, insbesondere von Blättern von Vanilla sp (V. planifolia und V. pompona)29, Kartoffelknollen (S. tuberosum)30 und Kapstachelbeeren (P. peruviana)31. Die Probenvorbereitung, der Aufbau des NMR-Experiments, die Datenerfassung und die Datenanalyse werden ausführlich beschrieben. Darüber hinaus enthält das Protokoll wichtige Hinweise, die Forschern, die neu auf diesem Gebiet sind, helfen sollen, PSYCHE- und SAPPHIRE-PSYCHE-Experimente zur Erstellung von metabolomischen Profilen von Pflanzen richtig einzurichten.
Protokoll
1. Vorbereitung der Probe
- Kapstachelbeeren
- 100-200 g frische Früchte in eine Mixervase geben. 30 min bei 4 °C halten und dann in einem Labormixer homogenisieren.
- Den Saft sofort in 50-ml-Plastikröhrchen umfüllen, in flüssigem Stickstoff einfrieren und 4 bis 5 Tage lang bis zum Trocknen lyophilisieren.
- Mahlen Sie das lyophilisierte Material mit einer elektrischen Mühle zu einem feinen Pulver.
HINWEIS: Die Handhabung des trockenen Materials muss schnell erfolgen, da das Pulver stark hygroskopisch ist. - Wiegen Sie 1 g des Mahlguts ab und fügen Sie 10 mL Reinstwasser hinzu. 1 Min. Vortexen.
- 20 min bei 10 °C beschallen und anschließend 20 min bei 10 °C bei 23.000 × g zentrifugieren.
- Den Überstand zurückgewinnen und durch einen 13 mm 0,45 μm Spritzenvorsatzfilter aus Polytetrafluorethylen (PTFE) filtrieren.
- 1 ml des gefilterten Extrakts wird bis zur Trockenheit lyophilisiert und der erhaltene Feststoff wird dann in 0,9 ml 200 mM Natriumoxalatpuffer pH 4 resuspendiert. Wirbel.
- Die resultierende Probe wird bis zur Trockenheit lyophilisiert und in 0,9 ml Deuteriumoxid gelöst, das 5 mM 3-(Trimethylsilyl)propionsäure-2,2,3,3-d4-saures Natriumsalz (TMSP-d4) enthält.
- Füllen Sie das NMR-Röhrchen mit 0,6 ml der Probe mit einer Mikropipette.
- Vanilleblätter
- Sammeln Sie die Blätter, reinigen Sie sie mit feuchtem Seidenpapier und frieren Sie sie im Ganzen in flüssigem Stickstoff ein.
- Die Blätter in kleine Stücke brechen und 4 Tage bis zum Trocknen lyophilisieren.
- Mahlen Sie die Trockenmasse mit einer elektrischen Mühle zu einem feinen Pulver.
- Wiegen Sie 50 mg gemahlenes Material und fügen Sie 0,75 ml Phosphatpuffer pH 6,0 in Deuteriumoxid mit 0,1 % TMSP (w/w) und 0,75 mL Methanol-d4 hinzu. 1 Min. Vortexen.
- 20 min bei 25 °C beschallen.
- Zentrifugieren Sie bei 13.000 × g für 10 min bei 25 °C.
- Gewinnen Sie den Überstand (~1,3-1,4 mL) zurück und filtrieren Sie ihn durch einen 13 mm PTFE 0,45 μm Spritzenvorsatzfilter.
- Füllen Sie das NMR-Röhrchen mit 0,6 mL der gefilterten Probe mit einer Mikropipette.
- Kartoffelknollen
- 4 bis 8 Knollen schälen und in Scheiben schneiden. Legen Sie sofort ca. 125 g Material in Standbodenbeutel und frieren Sie diese in flüssigem Stickstoff ein.
HINWEIS: Um Oxidation während der Handhabung zu vermeiden, halten Sie die Kartoffel feucht. - 4 bis 6 Tage bis zur vollständigen Trockenheit lyophilisieren.
- Mahlen Sie die Trockenmasse mit einer elektrischen Mühle zu einem feinen Pulver.
- Wiegen Sie 160 mg gemahlene Knolle und fügen Sie 1,6 ml entionisiertes Wasser hinzu. 1 Min. Vortexen.
- 45 min bei 10 °C beschallen.
- Zentrifugieren Sie bei 23.000 x g für 20 min bei 10 °C.
- Den Überstand (~1,5 -1,6 mL) zurückgewinnen und in einem gekühlten Zentrifugal-Vakuumkonzentrator 16 Stunden lang bei 10 °C bis zur Trockenheit verdampfen.
- Zu dem erhaltenen Feststoff (20-25 mg) werden 0,9 ml 100 mM Natriumoxalatpuffer pH 4 gegeben, vortexen und 16 Stunden bei 10 °C verdampfen.
- Der erhaltene Feststoff wird in 0,9 ml Deuteriumoxid gelöst, das 3 mM TMSP enthält.
- Zentrifugieren Sie bei 23.000 x g für 5 min bei 10 °C und filtrieren Sie den Überstand durch einen 13 mm PTFE 0,45 μm Spritzenvorsatz direkt in das NMR-Röhrchen.
HINWEIS: In diesem Fall wurde eine direkte Filtration in das NMR-Röhrchen durchgeführt, um die Schritte bei der Vorbereitung von mehr als 1000 Proben zu reduzieren.
- 4 bis 8 Knollen schälen und in Scheiben schneiden. Legen Sie sofort ca. 125 g Material in Standbodenbeutel und frieren Sie diese in flüssigem Stickstoff ein.
2. NMR-Datenerfassung und -verarbeitung
- NMR-Ersteinrichtung
- Übertragen Sie die Proben auf das NMR-Spektrometer.
- Stimmen Sie den Sondenkopf ab und passen Sie ihn an.
- Sperren und unterlegen Sie die Probe.
- Kalibrieren Sie den 90° harten Impuls. Kalibrieren Sie den 90°-Impuls mit einem der Standardverfahren.
- Führen Sie ein standardmäßiges 1D-Protonen-NMR-Spektrum durch.
- PSYCHE-Experiment
- Wählen Sie die reset_psyche_1d Pulssequenz aus der Bruker Topspin-Bibliothek aus (Abbildung S1). Verwenden Sie die folgenden Standardparameter: 5 kHz spektrale Breite (SW2), mindestens 1 oder 2 Sekunden Relaxationswiederherstellungsverzögerung (D1), 16 Dummy-Scans (DS), 64 oder 128 komplexe Datenpunkte pro Block (L31) und 64 oder 128 Scans (NS) (Abbildung S1).
HINWEIS: L31 ist die Anzahl der komplexen digitalen Punkte, die in jedem Pure Shift-Block erfasst werden, am besten auf eine Potenz von 2 einstellen. 21 - Stellen Sie die gewünschte CHIRP-Impuls-Flip-Winkel-Anregung (CNST61) und 10 kHz für die CHIRP-Impulsbandbreite (CNST60) ein (Abbildung S2).
HINWEIS: Das PSYCHE-Experiment basiert auf einem Anti-z-COSY-Schema; daher muss der CHIRP-Puls-Flip-Winkel klein sein, um Rekopplungsartefakte zu vermeiden (Abbildung 1). Die absolute Intensität nimmt mit dem Anregungs-Flip-Winkel zu. Die periodischen Artefakte werden ebenfalls verstärkt, indem sie sich in das Spektrum ausbreiten und das "Rauschen" erhöhen (Abbildung 1). Das "Rauschen" wird zu einer Kombination aus Standardgeräuschen und Chuncking-Artefakten. Ein guter Kompromiss zwischen Empfindlichkeit und geringen Rekopplungsartefakten besteht darin, CNST61 = 20° festzulegen. 19,22 kg - Stellen Sie die Länge des harten Impulses (P1) auf den zuvor kalibrierten Wert und die Länge des PSYCHE-Formimpulses auf 30 ms (P49) ein (Abbildung S2).
HINWEIS: Es ist sehr wichtig, den harten Impulswert zu kalibrieren, da die Formimpulsleistungen automatisch aus diesem Wert berechnet werden. - Wählen Sie den Formimpuls Crp_psyche.20 (SPNAM 37) für das PSYCHE-Element (Abbildung S2).
- Stellen Sie die Stärke des Pulsfeldgradienten ein, der während des PSYCHE-Elements (GPZ0) angewendet wird. Wählen Sie RECT.1 für den Gradientenformpuls (GPNAM 0) (Abbildung S2).
HINWEIS: Während des PSYCHE-Elements wird ein schwacher Magnetfeldgradient angelegt, der normalerweise je nach Sonde zwischen 1 % und 4 % der maximalen Stärke des Gradienten liegt. - Legen Sie die Anzahl der Blöcke fest, die erfasst werden sollen, um die reine Verschiebungs-FID (TD1) zu rekonstruieren (Abbildung S3).
HINWEIS: PSYCHE wird als Pseudo-2D-Experiment erfasst, wobei TD1 die Anzahl der Reinen Shift-Interferogrammblöcke ist. Die Spektralauflösung hängt von der Größe des Spektralfensters (SW1) und der Gesamtzahl der erfassten Punkte ab, die TD1*2*L31 beträgt. In der Regel bieten 16 oder 32 Blöcke mit 64 oder 128 komplexen Punkten pro Block eine ausreichende digitale Auflösung. Da PSYCHE in einer Interferogramm-Art aufgezeichnet wird, erhöht eine höhere Anzahl von Blöcken die digitale Auflösung, aber auch die Gesamterfassungszeit19. Homonukleäre J-Kopplungen entwickeln sich während jedes Blocks, was zu einem oszillierenden J-Modulationsmuster führt21,23. Nach der Fourier-Transformation werden periodische Seitenbandartefakte generiert, die von der Länge des Blocks abhängen (Abbildung 2). Um Artefakte zu reduzieren, muss die Dauer des Blocks kurz sein, in der Regel weniger als 16 ms (Blockdauer = 2*in0: Abbildung S1). Wenn die Blockdauer hoch ist, reduzieren Sie L31. - Verarbeiten Sie die Daten mit dem Proc_reset AU-Programm von Bruker und der Fourier-Transformation.
HINWEIS: Wir empfehlen, das Spektrum mit Nullfüllung und einer Sinusglocken-Apodisierung zu transformieren (Abbildung S4).
- Wählen Sie die reset_psyche_1d Pulssequenz aus der Bruker Topspin-Bibliothek aus (Abbildung S1). Verwenden Sie die folgenden Standardparameter: 5 kHz spektrale Breite (SW2), mindestens 1 oder 2 Sekunden Relaxationswiederherstellungsverzögerung (D1), 16 Dummy-Scans (DS), 64 oder 128 komplexe Datenpunkte pro Block (L31) und 64 oder 128 Scans (NS) (Abbildung S1).
- SAPPHIRE-PSYCHE Experiment
- Wählen Sie die SAPPHIRE-PSYCHE-Pulsfolge aus und stellen Sie die Parameter der Pulssequenz ein. Standardparameter wären die folgenden: 5 kHz spektrale Breite (SW3), mindestens 1 oder 2 Sekunden Relaxationsverzögerung (D1), 16 Dummy-Scans (DS), 8 oder 16 Scans pro Inkrement (NS) und D2 bis 14 ms (Abbildung S5).
HINWEIS: Diese Sequenz gehört nicht zu Brukers Repertoire, die Sequenz und die Verarbeitungsprogramme können jedoch von der Website der Manchester NMR Methodology Group, (https://www.nmr.chemistry.manchester.ac.uk/?q=node/426)23, bezogen werden. Das D2-Delay stellt sicher, dass die T2-Relaxation mit jedem J-Modulationsinkrement konstant bleibt. D2 muss größer als 1/4*SW1+p16+2*d16 sein. 23 - Stellen Sie die gewünschte CHIRP-Impuls-Flip-Winkel-Anregung (CNST20) und 10 kHz für die CHIRP-Impulsbandbreite (CNST21) ein (Abbildung S6).
HINWEIS: Wie im regulären PSYCHE-Experiment muss der CHIRP-Puls-Flip-Winkel kurz sein, um Rekopplungsartefakte zu vermeiden. CNST20 = 20° ist ein guter Kompromiss zwischen Sensitivität und geringen Rekopplungsartefakten 21,23,25. - Stellen Sie die harte Pulslänge (P1) auf den zuvor kalibrierten Wert und die PSYCHE-Shape-Pulslänge auf 30 ms (P40) ein (Abbildung S6).
HINWEIS: Es ist wichtig, den harten Impulswert zu kalibrieren, da die Formimpulsleistungen automatisch daraus berechnet werden. - Wählen Sie den PSYCHE_Saltire_10kHz_30m Formimpuls für das Element PSYCHE (Abbildung S6).
- Stellen Sie die Stärke des Impulsfeldgradienten ein, der während des PSYCHE-Elements (GPZ10) angewendet wird. Wählen Sie RECT.1 für den Gradientenformpuls (GPNAM 10) (Abbildung S7).
HINWEIS: Während des PSYCHE-Elements wird ein schwacher Magnetfeldgradient angelegt, normalerweise zwischen 1 % und 4 % der maximalen Stärke des Gradienten, ein Wert, der von der Sonde abhängt. - Stellen Sie die Anzahl der SAPPHIRE J-Modulationsinkremente in F2 (TD2) ein (Abbildung S7).
HINWEIS: Normalerweise sorgen 8 Inkremente für eine hervorragende Unterdrückung von Seitenbandartefakten (Abbildung 2 und 3). Die Gesamtzahl der Scans der endgültigen Pure Shift FID beträgt NS*TD2. 23 - Stellen Sie die Spektralfenster F1 und F2 (SW1 und SW2) ein (Abbildung S5).
HINWEIS: SW2=SW3/(2*TD2) und SW3/SW1 = TD2*N, wobei TD2 und N gerade ganze Zahlen23 sind. Das SAPPHIRE-PSYCHE-Experiment wird als Pseudo-3D aufgenommen, wobei F2 die Phasenmodulation des J-Kopplungsartefakts und F1 die Pure Shift-Interferogrammaufnahme20 kodiert. Da SAPPHIRE-PSYCHE J-Modulationsseitenbänder entfernt, kann die Interferogramm-Pure-Shift-Blockdauer länger sein als die reguläre PSYCHE (Pure Shift-Blockdauer = 1/SW1), typischerweise zwischen 20 und 40 ms (Abbildung 2). Eine längere Datenerfassung führt jedoch zu höheren J-Kopplungsentwicklungen, was mehr Inkremente der J-Kopplungsphasenmodulation erfordern würde, um die erreichten stärkeren Seitenbänder zu entfernen23. - Legen Sie die Anzahl der Pure Shift-Blöcke (TD1) fest (Abbildung S7).
HINWEIS: Da SAPPHIRE-PSYCHE die J-Kopplungsphasenmodulation des ersten Blocks kompensieren muss, muss ein zusätzlicher Block erworben werden. In der Regel ergeben 17 (16+1) oder 33 (32+1) Blöcke eine ausreichende digitale Auflösung23. - Verarbeiten Sie die Daten, die die pm_pshift und die pm_fidadd AU-Programme ausführen, gefolgt von der Fourier-Transformation23.
HINWEIS: Wir empfehlen, das Spektrum mit Nullfüllung und einer Sinusglocken-Apodisierung zu transformieren (Abbildung S4).
- Wählen Sie die SAPPHIRE-PSYCHE-Pulsfolge aus und stellen Sie die Parameter der Pulssequenz ein. Standardparameter wären die folgenden: 5 kHz spektrale Breite (SW3), mindestens 1 oder 2 Sekunden Relaxationsverzögerung (D1), 16 Dummy-Scans (DS), 8 oder 16 Scans pro Inkrement (NS) und D2 bis 14 ms (Abbildung S5).
Ergebnisse
NMR-Spektrum-Analyse
PSYCHE-Experimente erhöhen die Spektrenauflösung, indem sie gekoppelte Resonanzen in Singulden21 kollabieren, was wiederum die Überlappung reduziert und die Zuordnung und Datenanalyse erleichtert. Pure Shift NMR kann auf Pflanzenextrakte angewendet werden. Hier zeigen wir die Verwendung in drei verschiedenen Matrices: Vanilleblättern, Kartoffelknollen und Physalis peruviana-Früchten . Die in den Spektren dieser Pflanzenextrakte erzielte Auflösungsverbesserung ist aus den Abbildungen S8-S11 ersichtlich.
Um den Einfluss dieser Pulssequenzen auf die Auflösung des NMR-Signals zu bewerten, wurde die Breite der Linie in Frequenzeinheiten bei 10 % der maximalen Höhe W10 für verschiedene Arten von Resonanzen berechnet, die alle in Spektren dargestellt sind, die mit Vanilla sp-Extrakten erhalten wurden - Reine Verschiebung gegenüber klassischer 1H-NMR - Abbildung 4. Insgesamt erreichte die Ausdehnung der gekoppelten Resonanzen W10-Werte von 1 bis 60 Hz, während die Singulett-Peaks zwischen 1 und 10 Hz schwankten: Die Methylgruppe der Essigsäure (1,97 ppm in Abbildung 4) erreichte einen W10-Wert von 2,0 Hz im klassischen 1H-NMR und 2,1 Hz im SAPPHIRE-PSYCHE (S)-Spektrum. Die anomere Resonanz von Saccharose (bei 5,40 ppm), einem Dublett mit einer J-Kopplung von 3,9 Hz, erstreckt sich über einen Bereich, der einem W10 von 6,5 Hz entspricht, wobei jeder einzelne Peak 2,6 Hz von W10 ausmacht (Abbildung 4). Dieser Wert war höher als der des kollabierten Peaks im SAPPHIRE-PSYCHE-Spektrum, W10 = 1,9 Hz (Abbildung 4). Im Falle der Apfelsäure Hβ' (bei 2,55 ppm) erstreckt sich ein Dublett von Dubletten mit J = 7,8 Hz, 15,6 Hz, über 29,4 Hz (Abbildung 4), mit berechneten W10-Werten für jeden einzelnen Peak zwischen 4,7 und 4,9 Hz. Dieses Multiplett-Signal kollabierte durch Pure Shift zu einer einzigen Zeile (bei 2,55 ppm) und expandierte über einen W10-Wert von 4,7 Hz (Abbildung 4).
Die Signalvielfalt wird in hochgekoppelten Wasserstoffatomen komplexer, da die konstitutiven Peaks des Multipletts nicht mehr leicht zu unterscheiden sind und ein nahezu kontinuierliches Signal bilden. Dies ist der Fall für die homozitronischen Säurewasserstoffe Hγ (bei 2,24 ppm), Hδ (bei 2,02 ppm) und Hδ' (bei 1,91 ppm), Abbildung 4. Die Hγ-, Hδ- und Hδ'-Multipletts mit W10=38,3 Hz, W10=41,5 Hz bzw. W10=34,8 Hz kollabierten zu Unterlingen von W10=7,8 Hz (Hγ, 2,24 ppm), W10=8,0 Hz (Hδ, 2,02 ppm) und W10=6,2 Hz (Hδ', 1,91 ppm), Abbildung 4. Diese verbesserte Auflösung ermöglichte eine bessere Unterscheidung anderer überlappender Signale, die Apfelsäure (Hβ, 2,77 ppm Hβ', 2,55 ppm), Homozitronensäure (Hα, 2,80 ppm und Hα', 2,63 ppm) und Homozitronensäurelacton (Hα', 2,84 ppm und Hδ, 2,65 ppm) entsprechen (Abbildung 4).
Die Verbesserung der Auflösung in den homonuklearen entkoppelten Spektren von SAPPHIRE-PSYCHE, die eine bessere Unterscheidung zwischen den Signalen ermöglicht, wird auch in zwei stark kompromittierten Regionen, 4,85 - 5,08 ppm und 7,05 - 7,31 ppm, demonstriert, in denen wichtige Metaboliten identifiziert werden konnten, die am Stoffwechselweg des Vanilleduftes beteiligt sind: Glucosid A (siehe Struktur in Abbildung 5), CH-2',6' (bei 7,28 ppm), CH-2", 6" (bei 7,18 ppm), CH-3',5' (bei 7,09 ppm), CH-3",5" (bei 7,07 ppm), CH2-7' (bei 4,97 ppm und 5,05 ppm), CH2-7'' (bei 4,89 ppm), CH-Glc (bei 4,96 ppm) und CH'-Glc (bei 4,93 ppm), Glucosid B (siehe Struktur in Abbildung 4), CH-2',6' (bei 7,29 ppm), CH-2",6" (bei 7,23 ppm), CH-3', 5' (bei 7,09 ppm), CH-3",5" (bei 7,07 ppm), CH2-7' (bei 4,99 ppm), CH2-7'' (bei 4,91 ppm), Hydroxybenzylalkohol (7,23 ppm) und Hydroxybenzylalkoholglucosid (7,10 und 4,51 ppm), Abbildung 4.
Die gleichen Ergebnisse wurden bei Kapstachelbeerextrakten und bei der Kartoffelknollenanalyse beobachtet. In diesen beiden Fällen wurden mehrere überfüllte Regionen auf regulärem 1H-NMR, PSYCHE und SAPPHIRE-PSYCHE für Vergleiche erweitert (Abbildungen 6 und 7).
Es ist klar, dass die Experimente von PSYCHE und SAPPHIRE-PSYCHE die Signalauflösung von Kapstachelbeerextrakten für Saccharose (4,04 ppm), β-Fruktose (4,02 ppm und 3,99 ppm), β-Glucose (3,24 ppm), Prolin (2,34 ppm, 2,07 ppm und 2,00 ppm), Glutaminsäure (2,16 ppm) und Glutamin (2,13 ppm) deutlich verbesserten, wie wir in früheren Arbeiten11 gezeigt haben, sowie im Fall von S. tuberosum Extrakte: GABA (1,91 ppm, 2,33 ppm und 3,02 ppm), Pyroglutaminsäure (2,04 ppm, 2,41 ppm und 2,51 ppm), Prolin (2,00 ppm, 2,07 ppm, 3,33 ppm und 3,42 ppm), Glutamin (2,14 ppm und 2,46 ppm), Valin (2,27 ppm), Zitronensäure (2,64 ppm und 2,76 ppm) und β-Glukose (3,25 ppm und 3,47 ppm), Abbildung 7.
Die PSYCHE-Sequenz ist relativ einfach zu bedienen und wurde in einer Vielzahl von Anwendungen erfolgreich eingesetzt9. Um das entkoppelte Spektrum zu erreichen, nimmt die Pulssequenz kleine FID-Brocken auf, die die J-Kopplung in der Mitte jedes Blocks neu fokussieren. Während jedes Blocks findet jedoch eine kleine J-Kopplungsentwicklung statt, die die periodischen Seitenbandartefakte erzeugt, die normalerweise weniger als 5 % ihres übergeordneten Peaks21 ausmachen. Das Vorhandensein dieser J-Modulationsartefakte, die den PSYCHE-Experimenten21 inhärent sind, ist in den erweiterten Zonen in Abbildung 6 offensichtlich. Bei der Analyse von reinen Verbindungen können diese Artefakte vernachlässigt werden. In biologischen Proben ist dies möglicherweise nicht der Fall, denn wie in Abbildung 6 für Prolin (4,13 ppm, 3,41 ppm und 3,33 ppm), Asparagin (3,97 ppm), Myo-Inositol (3,27 ppm), GABA (3,03 ppm) und Apfelsäure (2,67 ppm) und in Abbildung 6 für Pyroglutaminsäure (2,04 ppm, 2,41 ppm und 2,51 ppm) und Valin (2,27 ppm) gezeigt, Metaboliten in höheren Konzentrationen erzeugen Artefakte, die so groß sind wie einige der Signale, die zu Verbindungen gehören, die in niedrigen Konzentrationen vorhanden sind, und beeinträchtigen auf diese Weise die Genauigkeit der metabolischen Profilerstellung.
Das SAPPHIRE-PSYCHE-Experiment ist eine Modifikation der regulären PSYCHE-Sequenz, bei der diese periodischen Artefakte durch systematische Phasenmodulation entfernt werden, die durch Verschiebung des J-Refokussierungspunktes23 erreicht wird. Folglich ermöglicht das SAPPHIRE-PSYCHE-Experiment, ein viel saubereres Pure-Shift-Spektrum zu gewährleisten, wie in Abbildung 6 für Prolin (4,13 ppm, 3,41 ppm und 3,33 ppm), Asparagin (3,97 ppm), Myo-Inositol (3,27 ppm), GABA (3,03 ppm) und Apfelsäure (2,67 ppm) im Fall von Kapstachelbeeren11 und in Abbildung 7 für Pyroglutaminsäure (2,04 ppm, 2,41 ppm und 2,51 ppm) und Valin (2,27 ppm) bei Kartoffeln.
Ein weiteres Artefakt, unter dem alle Pure Shift-Experimente leiden, sind diejenigen, die durch den starken Kopplungseffekt erzeugt werden. Einige wichtige Primärmetaboliten wie Zitronensäure (2,64 ppm und 2,76 ppm) und Glutamin (2,14 ppm und 2,46 ppm) weisen starke Kopplungsartefakte auf, wie im Kartoffelknollenextrakt gezeigt wird (Abbildung 7). Bis heute gibt es keine Impulssequenz, mit der dieses Problem bequem behoben werden kann. jedoch schneidet SAPPHIRE-PSYCHE besser ab als reguläres PSYCHE 8,9,23.
Analyse der Korrelationsmatrix
Einer der Hauptvorteile der NMR-Spektroskopie besteht darin, dass die relativen Konzentrationen der Metaboliten in einem Gemisch direkt proportional zur Intensität ihrer Signale sind. Dann kann eine paarweise Korrelationsmatrix des Metaboliten direkt aus der Spektrenkorrelationsmatrix32 erhalten werden.
Die 1-H-NMR-Korrelationsmatrix, allgemein bekannt als STOCSY32 (Statistical Total Correlation Spectroscopy), wird in der Regel als Pseudo-2D-Spektrum dargestellt, wobei jeder Querpeak ein Korrelationskoeffizient zwischen zwei Signalen ist (Abbildung 8)11. STOCSY weist eine hohe Korrelation zwischen Signalen auf, die zum selben Molekül gehören, aber auch mit Signalen von Molekülen, die zum selben Stoffwechselweg gehören11,32. Korrelationsmuster liefern daher Informationen über den physiologischen Zustand des Systems und können daher als physiologischer Stadien-Fingerabdruck verwendet werden33.
Die Haupteinschränkung von STOCSY ist die Signalüberlappung, die die paarweise Korrelation verringert11,32. Darüber hinaus führen J-Kopplungs-Multiplizitätskorrelationen zu hochkomplexen Mustern, was die Analyse weiter erschwert11,32. Die Verwendung von SAPPHIRE-PSYCHE STOCSY, das in Abbildung 8 gezeigt ist, verbessert die Korrelationswerte, da es die J-Kopplungs-Multiplizitätskorrelationen in einem einzigen Peak reduziert und dadurch die Überlappung11 reduziert.
In Abbildung 8 wurden mehrere Regionen erweitert. Apfelsäure zeigt komplexe J-Multiplett-Muster, die mit Hα (4,41 ppm), Hβ (2,82 ppm) und Hβ' (2,67 ppm) assoziiert sind; in Pure Shift STOCSY kollabieren diese Signale zu einzelnen Korrelationspeaks (Abbildung 9A)11. Die gleichen Ergebnisse werden für β-Glucose beobachtet, wobei intermolekulare und intramolekulare (α-Glucose, α-Fructose und β-Fructose) Korrelationen in SAPPHIRE-PSYCHE besser dargestellt werden (Abbildung 9B)11. Einige Aminosäuren weisen auch starke intramolekulare Korrelationen in Kapstachelbeerextrakten auf. Ihre Identifizierung in regulären 1H-NMR-STOCSY wird jedoch durch die Überlappung in dicht besiedelten Bereichen beeinträchtigt. Mit Pure Shift STOCSY werden intermolekulare und intramolekulare Korrelationen von Prolin, Alanin, Glutamin und Glutaminsäure besser dargestellt (Abbildungen 9C und 9D)11.
Multivariate Analyse
Die multivariate Analyse ist eines der wichtigsten Instrumente zur Verarbeitung von Metabolomik-Daten34,35. Während die Probenunterscheidung durch PCA (Principal component analysis) oder PLS-DA (Partial least squares-diskriminant analysis) leicht durch reguläre 1H-NMR-Spektren erreicht werden könnte, wird die Interpretation der Belastung durch reine Shift-Daten 11,24,25,27,28 besser berücksichtigt.
In Abbildung 10 zeigen wir das PLS-DA-Score-Diagramm, das mit SAPPHIRE-PSYCHE (Abbildung 10A) und regulären Protonenspektren (Abbildung 10B) in der Unterscheidung von sechs Ökotypen von P. peruviana-Extrakten erhalten wurde 11. Auch wenn es einige Studien gibt, die eine bessere Diskriminationsleistung bei Verwendung von Pure Shift-Daten behaupten24,25, zeigen unsere Ergebnisse, dass die Leistung durch die homonukleäre Entkopplung kaum beeinflusst wurde11. Im Falle der Analyse der Belastungsdaten, wie in den Abbildungen 10C und 10D gezeigt, vereinfachte die erhöhte Auflösung, die mit den SAPPHIRE-PSYCHE-Daten erreicht wurde, die Analyse und ermöglichte eine bessere Identifizierung der spezifischen Metaboliten, die für die Diskriminierung von P. peruviana verantwortlich sind, nämlich α-Glucose, β-Glucose, α-Fructose, β-Fructose, Saccharose, Zitronensäure und Alanin11. Dieser Auflösungsgewinn war auch bei der Kombination der PLS-Koeffizientenanalyse mit der STOCSY-Korrelation von entscheidender Bedeutung, Abbildungen 10C und 10D11. Die starke Korrelation zwischen α-Glucose (der STOCSY-Vektor bei 5,23 ppm ist auf der ersten PLS-Komponente farbcodiert) und β-Glucose, α-Fructose und β-Fructose - alles Metaboliten, die sich aus demselben Stoffwechselweg entwickeln - und ihre Antikorrelation in Bezug auf Saccharose sindoffensichtlich 11. Bei normaler STOCSY-Analyse erlaubte die umfangreiche Überlappung keine eindeutige Abbildung zwischen den Korrelationskoeffizienten und führte zum Verlust dieser speziellen Information über den Stoffwechselweg (Abbildung 10)11.
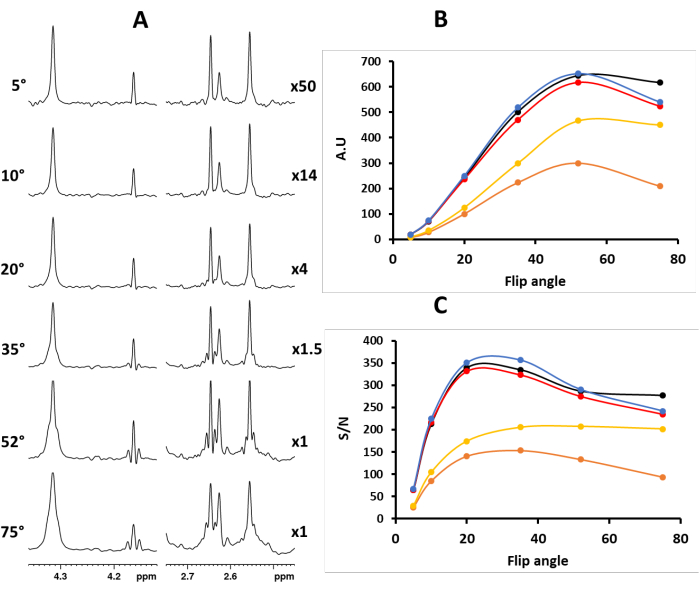
Abbildung 1. (A) PSYCHE-Spektren von Vanilla planifolia-Extrakten unter Verwendung verschiedener CHIRP-Puls-Flip-Winkel für das PSYCHE-Element: links, Flip-Winkel-Werte; Rechts, Intensitätsmultiplikationsfaktor. Die Diagramme (B) und (C) zeigen die Intensitäten und das Signal-Rausch-Verhältnis von fünf entkoppelten Peaks (schwarz - 4,31 ppm, orange - 4,16 ppm, rot - 2,65 ppm, gelb - 2,63 ppm und blau 2,56 ppm) als Funktion des Flip-Winkels des PSYCHE CHIRP-Pulses. Das Signal-Rausch-Verhältnis (S/N) (genauer gesagt Signal-Rausch + Artefaktverhältnis) wurde berechnet, indem das Signal der maximalen Intensität mit dem des Rauschens verglichen wurde, ein Wert, der über einen Bereich von 2 ppm berechnet wurde: von 7,75 ppm bis 9,75 ppm (Abbildung S12). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
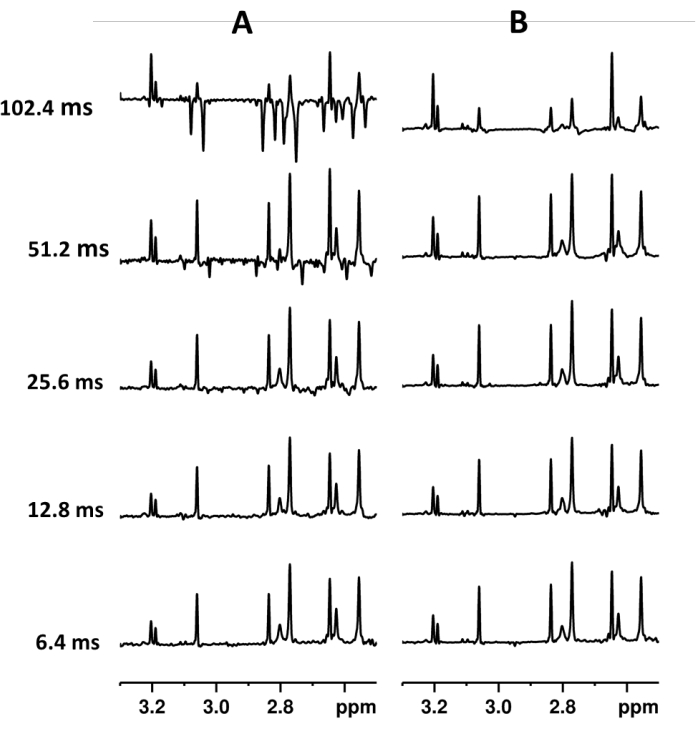
Abbildung 2. PSYCHE (A) und SAPPHIRE-PSYCHE (B) Spektren von Vanilla planifolia Extrakten mit unterschiedlicher Interferogramm Pure Shift Blockdauer. Die Experimente wurden aufgenommen, um die gleiche digitale Auflösung und Empfindlichkeit beizubehalten. PSYCHE (A) Parameter von unten nach oben: 128 Scans, 64 Interferogrammblöcke, 6,4 ms Blocklänge, Gesamtaufnahmezeit 6h07min; 128 Scans, 32 Interferogramm-Blöcke, 12,8 ms Blocklänge, Gesamtaufnahmezeit 3h04min; 128 Scans, 16 Interferogramm-Blöcke, 25,6 ms Blocklänge, Gesamtaufnahmezeit 1h32min; 128 Scans, 8 Interferogramm-Blöcke, 51,2 ms Blocklänge, Gesamtaufnahmezeit 46min; 128 Scans, 4 Interferogramm-Blöcke, 102,4 ms Blocklänge, Gesamtaufnahmezeit 24min. SAPPHIRE-PSYCHE (B) Parameter von unten nach oben: 16 Scans, 8 J-Modulationsschritte, 65 Interferogramm-Blöcke, 6,4 ms Blocklänge, Gesamtaufnahmezeit 6h19min; 16 Scans, 8 J-Modulationsschritte, 33 Interferogramm-Blöcke, 12,8 ms Blocklänge, Gesamtaufnahmezeit 3h13min; 16 Scans, 8 J-Modulationsschritte, 17 Interferogramm-Blöcke, 25,6 ms Blocklänge, Gesamtaufnahmezeit 1h40min; 16 Scans, 8 J-Modulationsschritte, 9 Interferogramm-Blöcke, 51,2 ms Blocklänge, Gesamtaufnahmezeit 53min; 16 Scans, 8 J-Modulationsschritte, 5 Interferogramm-Blöcke, 102,4 ms Blocklänge, Gesamtaufnahmezeit 30 Minuten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3. (A) Spektren von Vanilla planifolia-Extrakten mit 102,4 ms Stücklänge: oben, PSYCHE 128 Scans, 4 Pure Shift-Inkremente; Mitte oben, 4 Scans, 4 Pure Shift-Inkremente, 32 SAPPHIRE-Inkremente; Mitte unten, 8 Scans, 4 Pure Shift-Inkremente, 16 SAPPHIRE-Inkremente; unten, 16 Scans, 4 Pure Shift-Inkremente, 16 SAPPHIRE-Inkremente. (B) Spektren von Vanilla planifolia-Extrakten mit 51,2 ms Stücklänge: oben, PSYCHE 128 Scans, 8 Pure Shift-Inkremente; Mitte oben, 4 Scans, 9 Pure Shift-Schritte, 32 SAPPHIRE-Schritte; Mitte unten, 8 Scans, 9 Pure Shift-Inkremente, 16 SAPPHIRE-Inkremente; unten, 16 Scans, 9 Pure Shift-Inkremente, 16 SAPPHIRE-Inkremente. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4. Ausgewählte Expansionsbereiche der 1H NMR (1H) und SAPPHIRE-PSYCHE (S) Spektren V. planifolia (1,85 - 2,9 ppm) und V. pompona (4,85 - 7,31 ppm) zeigen Signalzuordnungen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
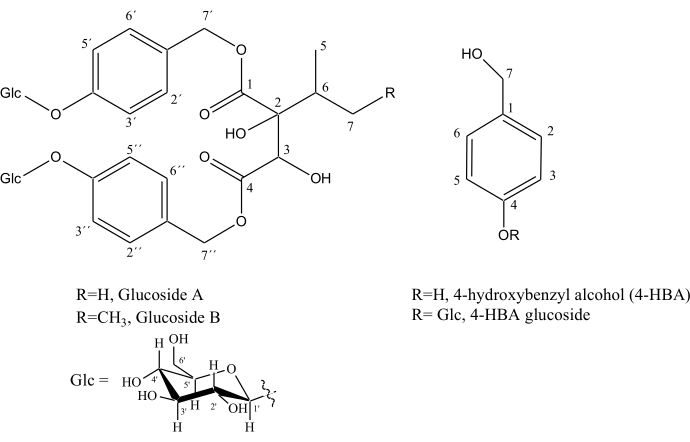
Abbildung 5. Strukturen der Vanilleduftvorläufer: 4-Hydroxybenzylalkohol (4-HBA), 4-HBA-Glucosid, Glucosid A und Glucosid B. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6. Ausgewählte Expansionsregionen von 1H NMR (1H), PSYCHE (P) und SAPPHIRE (S) Spektren eines wässrigen Extrakts aus der Kapstachelbeere (Bambamarca I Peruanische Andenregion28) zeigen Signalzuordnungen (Nachdruck mit Genehmigung von Lopez et al.11 ). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
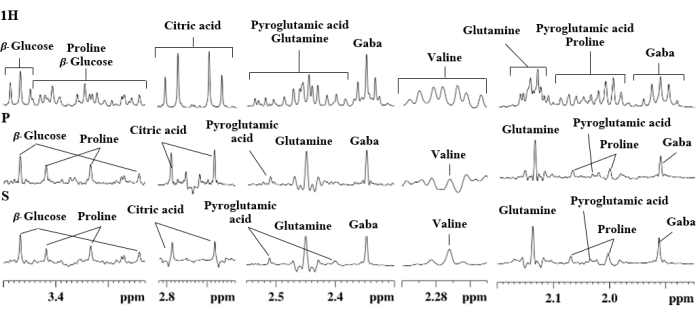
Abbildung 7. Ausgewählte Expansionsbereiche von 1H NMR (1H), PSYCHE (P) und SAPPHIRE (S) Spektren eines wässrigen Kartoffelextrakts zeigen die Signalzuordnung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 8. Ausgewählte erweiterte Regionen (3,20 ppm-4,30 ppm) von zweidimensionalen STOCSY-NMR-Spektren, die mit Daten von sechs Kapstachelbeerextrakten erhalten wurden, die Korrelationswerte (r2) über 0,85 zeigten: (A) regulärer 1-H-NMR-STOCSY und (B) SAPHIR-PSYCHE-STOCSY. Nachdruck mit Genehmigung von Lopez et al.11 Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
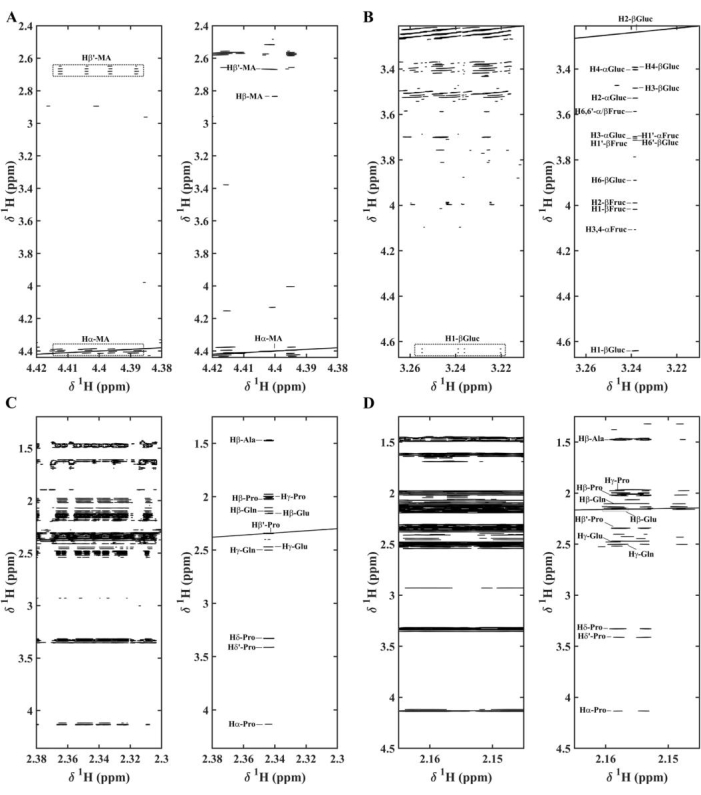
Abbildung 9. STOCSY-Darstellungen von NMR-Spektren von sechs verschiedenen Kapstachelbeerextrakten zeigen Korrelationen (r2), links ohne Homödekopplung und rechts mit Homodierkupplung für Regionen: (A) 4,38-4,42 ppm und 2,40-4,42 ppm mit r2 über 0,80 für das Apfelsäure (MA)-Signal (Hα-MA); (B) 3,21-3,27 ppm und 3,21-4,67 ppm mit r2 über 0,85 für das β-Glucose-Signal (H2-β-Gluc); (C) 2,30-2,38 ppm und 1,25-4,36 mit r2 über 0,93 für Prolin (Pro)-Signal (Hβ'-Pro); (D) 2,15-2,17 ppm und 1,25-4,5 ppm mit r2 über 0,90 für das Glutaminsäure (Glu)-Signal (Hβ-Glu); α-Glukose, α-Fruktose β-Fruktose werden als α-Gluc, α-Fruc bzw. β-Fruc symbolisiert. Nachdruck mit Genehmigung von Lopez et al.11 Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 10. PLS bewertet die Darstellung von Kapstachelbeerextrakten, die in sechs verschiedenen Andenregionen angebaut wurden11,28 (San Marcos: rote Kreise, Celendin III: braune Dreiecke, Bambamarca I: blaue Sterne, Celendin I: gelbe Dreiecke, Bambamarca II: grüne Quadrate, Celendin II: magentafarbene Diamanten) basierend auf (A) klassischer 1H-NMRund (B) Saphir-Psyche-Experimenten. DieT2-Ellipsen von Hotelling wurden auf ein Konfidenzniveau von 95 % festgelegt. Kombination von PLS1-Ladungen und 1D-STOCSY für die α-Glukose-Korrelation unter Verwendung des STOCSY-Signals bei 5,23 ppm als Treiberpeak. Der Determinationskoeffizient (r2) wurde farbcodiert und auf die Koeffizienten der ersten PLS-Komponente projiziert: (A) 1D STOCSY, erhalten mit SAPPHIRE-PSYCHE-Daten (oben) und seiner Expansion 3,15-4,17 ppm (unten); (B) 1D-STOCSY, erhalten mit 1-H-NMR-Daten (oben) und deren Ausdehnung 3,15-4,17 ppm (unten); α-Glukose, β-Glukose, α-Fruktose, β-Fruktose und Saccharose werden als α-G, β-G, α-F, β-F bzw. S symbolisiert. Die PLS-DA- und STOCSY-Analyse wurde mit MATLAB Version R2018a durchgeführt. (Nachdruck mit Genehmigung von Lopez et al.11) Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die Identifizierung und Quantifizierung der Metabolitenstruktur sind Schlüsselfragen bei der Charakterisierung des Metaboloms, Daten, die bei multivariablen Analysen ein besseres Verständnis des untersuchten biologischen Systems ermöglichen. Die Probenvorbereitung und die Datenerfassung sind kritische Aspekte, die optimiert werden müssen, um zuverlässige Ergebnisse zu liefern.
In diesem Artikel beschreiben und veranschaulichen wir die Probenvorbereitung für die NMR-Analyse von drei verschiedenen Pflanzenmatrizen. Wie bei jedem Extraktionsverfahren bestimmen die Menge des Lösungsmittels pro Gramm Material und die physikalischen Eigenschaften des ausgewählten Lösungsmittels die chemische Zusammensetzung des endgültigen Extrakts und die Konzentration der extrahierten Metaboliten. Im Falle der NMR-Metabolomisierung sind der pH-Wert, die Reproduzierbarkeit zwischen unabhängigen Probenextraktionen und die endgültige Extraktmenge im NMR-Röhrchen ebenfalls Aspekte, die optimiert werden müssen. Die Bedeutung der Reproduzierbarkeit in der Metabolomik besteht darin, die Einführung unkorrelierter Varianz zu vermeiden, die zu unzuverlässigen Ergebnissen führen könnte. Nach unserer Erfahrung wurden mit dem trockenen und zerkleinerten Pflanzenmaterial optimale Extraktionsbedingungen erreicht. Im Fall von Kapstachelbeeren war das Trockenprodukt sehr schwer zu handhaben (stark hygroskopisch), so dass die frischen Beeren zuerst homogenisiert wurden, bevor sie lyophilisiert wurden.
Bei der Spektrenerfassung ist der Versuchsaufbau von Pure Shift von besonderer Bedeutung, da falsche Parameter zu Chunking und Recoupling-Artefakten führen können. Die Theorie hinter den Prinzipien von Pure-Shift-Experimenten, die an anderer Stelle ausführlich besprochen wurde 8,9,10, ist wichtig, um zu verstehen, wie die Pulssequenz korrekt konfiguriert und als Routineexperiment implementiert wird.
Kurz gesagt, die meisten Pure-Shift-Experimente basieren auf der Neufokussierung der J-Kopplungsevolution während der Aufzeichnung chemischer Shifts. Dies wird typischerweise durch ein J-Kopplungs-Refokussierungselement erreicht, das selektiv "passive" Spins invertiert, während die "aktiven" Spins unbeeinflusst bleiben. PSYCHE und SAPPHIRE-PSYCHE basieren auf einem Anti-z-COSY-Experiment, bei dem "passive" Spins statistisch invertiert sind.
Das PSYCHE-Element, das aus zwei Sweep-Frequenz-Pulsen mit niedrigem Flip-Winkel in Gegenwart eines schwachen gepulsten Feldgradienten besteht, induziert die räumlich-zeitliche Mittelung der Frequenz, indem es die antidiagonalen COSY-Terme auswählt und gleichzeitig Null-Quanten- und Kreuzkorrelationsterme unterdrückt. Um die inhärenten Rekopplungsartefakte zu vermeiden, muss der CHIRP-Impuls-Flip-Winkel daher kurz sein (Abbildung 1). In der Regel ist ein Schwenkwinkel von 20° ein guter Kompromiss zwischen Empfindlichkeit und Entkopplungsleistung (Abbildung 1). Daher ist die Pulskalibrierung entscheidend für die Qualität und Empfindlichkeit des Spektrums.
Metabolomische Studien sind in der Regel mit der Aufzeichnung von Spektren an einer großen Anzahl von Proben verbunden, was bedeutet, dass die Pulskalibrierung schnell oder automatisch erfolgen muss. Unserer Erfahrung nach beträgt die Variabilität der harten Pulslänge zwischen den Proben weniger als ± 0,2 μs, wenn die Proben auf genau die gleiche Weise vorbereitet werden. In der Regel kalibrieren wir zwischen 6 und 12 Proben und verwenden dann den erreichten Durchschnittswert, um die gesamte Probengruppe aufzubauen. Für den Fall, dass die Variabilität der Pulslänge von Probe zu Probe höher wäre, sollte die automatische Kalibrierung jeder Probe mit dem Pulsautomatisierungsprogramm Topspin auf dem Bruker-Spektrometer durchgeführt werden.
Der zweite wichtige Parameter, der zu berücksichtigen ist, ist die Länge der aufgezeichneten Blöcke während der schrittweisen Interferogrammaufnahme21,23. Die Interferogrammerfassung besteht darin, die FID in kleinen Brocken aufzuzeichnen, wobei der Refokussierungspunkt der J-Kopplungsentwicklung immer mit dem Zentrum des erfassten Brockens zusammenfällt. Die entkoppelte FID wird durch Verkettung jedes aufeinanderfolgenden Chunks 8,9,10 gebildet. Um sicherzustellen, dass die Blockerfassung die Entwicklung der chemischen Verschiebung nicht abschneidet, muss der Beginn jeder Datenaufzeichnung genau mit dem Ende des vorherigen Blocks übereinstimmen.
Obwohl dieses Verfahren es uns ermöglicht, ein homodegekoppeltes Spektrum zu erhalten, erzeugt die kleine J-Kopplungsentwicklung während jedes Blocks periodische Seitenbandartefakte, die direkt von der Chunklänge abhängen. Auf der anderen Seite hängt die spektrale digitale Auflösung vom Spektralfenster und von der Gesamtdauer des entkoppelten FID ab, die wiederum von der Blocklänge und der Anzahl der aufgezeichneten Blöcke abhängt. Um periodische Artefakte zu reduzieren, ohne die Auflösung zu beeinträchtigen, sollte daher die Blockdauer kurz sein, und die Gesamtzahl der aufgezeichneten Blöcke sollte hoch sein. Diese Bedingungen erhöhen jedoch die gesamte experimentelle Erfassungszeit, ohne die Empfindlichkeit zu erhöhen (Abbildung 2). Typischerweise liefert ein PSYCHE-Experiment, das mit 16 bis 32 Blöcken von 10 bis 16 ms Dauer aufgenommen wurde, eine ausreichende digitale Auflösung in einer angemessenen experimentellen Zeit (30 min bis 5 h) (Abbildung 2).
Im Fall von SAPPHIRE-PSYCHE, einem Experiment, das als Pseudo-3D aufgenommen wird, kodiert eine der indirekten Dimensionen für die Aufnahme des Pure Shift-Interferogramms und die andere für die Phasenmodulation der periodischen Artefakte durch die systematische Verschiebung des J-Refokussierungspunkts in jedem Block.
Da periodische Artefakte durch SAPPHIRE stark unterdrückt werden, könnten die Blocklängen länger sein; Sehr lange Blöcke beeinflussen jedoch stark die Intensität der Signale (Abbildungen 2 und 3). In SAPPHIRE tragen die J-Modulationsinkremente zur Spektralempfindlichkeit bei, daher ist die resultierende entkoppelte FID-Gesamtzahl der Scans gleich TD2 * NS (Abbildungen 2 und 3)23. Im Allgemeinen gewährleisten acht J-Modulationsschritte eine hervorragende periodische Artefaktunterdrückung, und mehr als acht Inkremente haben nur einen sehr geringen Einfluss auf die Qualität des Spektrums, selbst wenn lange Chunklängen verwendet werden (Abbildung 3)23. Pure Shift-Inkremente von 33 oder 17 ms mit Dauern zwischen 20 und 40 ms sorgen für eine gute spektrale digitale Auflösung.
Eine Einschränkung dieser beiden Pure Shift-Pulssequenzen, PSYCHE und SAPPHIRE-PSYCHE, ist die Quantifizierung durch absolute Metaboliten-interne Standardintegration. Bei der regulären 1H-NMR ist die integrierte Intensität direkt proportional zur Konzentration jedes Metaboliten. In PSYCHE ist dies nicht mehr der Fall, da eine Reihe von Phänomenen die Signale verzerren und die Integration beeinflussen. Zum Beispiel nimmt der Gesamtintegralwert aufgrund der T2-Relaxation während der Spin-Auswahl der Pulssequenz ab. Auch die verkürzte J-Kopplungsentwicklung während der Chunk-Erfassung, die Seitenbandartefakte erzeugt, stört die Lorentzsche Form des Signals. Daher besteht das Integral nun aus Bereichen unter dem Hauptpeak und unter allen Seitenbändern, was die Signalintegration 8,9,21,23 erschwert. Die Häufigkeit und Größe der Seitenbänder stehen in direktem Zusammenhang mit der Stücklänge, aber auch mit intrinsisch molekularen Eigenschaften wie Relaxation und der J-Kopplungsgröße und -Multiplizität: Höhere J-Kopplungsgrößen und höhere Multiplizitäten führen zu stärker verzerrten Signalen. Im Fall von SAPPHIRE werden die Signalintensitäten durch die verkürzte J-Kopplungsentwicklung beeinträchtigt, obwohl dieses NMR-Experiment die Seitenbandartefakte effizient entfernt. Die Summe jedes J-modulierten Inkrements erzeugt einen gemittelten entkoppelten FID, bei dem die Signalverminderung in direktem Zusammenhang mit der Chunklänge und der J-Kopplungsgröße und -multiplizität23 steht. Darüber hinaus erzeugen die CHIRP-Puls-Flip-Winkel ein Rekopplungsartefakt, das jedes Signal ebenfalls unterschiedlich beeinflusst, was die Quantifizierung weiter erschwert21. Das Ausmaß der Wirkung dieser Pulssequenzen in der quantitativen Analyse wurde in unserer früheren Studie an der Kapstachelbeere bewertet, die Fehler von etwa 10 % bis 30 % ergab11.
Abschließend können wir den Schluss ziehen, dass Pure Shift ein hervorragendes neues Werkzeug für die pflanzliche Metabolomik ist, da es die Spektralauflösung drastisch erhöht und eine feinere Korrelationsmatrixanalyse und eine bessere Interpretation von multivariaten Analysen ermöglicht 11,24,25,27,28.
Offenlegungen
Die Autoren haben keine Interessenkonflikte anzugeben.
Danksagungen
Diese Studie wurde vom Consejo Nacional de Ciencia, Tecnología e Innovación Tecnológica (CONCYTEC) - Programa Atracción de Investigadores Cienciactiva - Contract # 008-2017-FONDECYT finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| 77500 Series Freezone 4.5 Liter benchtop | Labconco | 77500 | |
| Bruker Avance III 500 MHz equiped with a 5 mm TCI Z-gradient cryogenic probe | Bruker Corporation | ||
| Centrivap Refrigerated Centrifugal Concentrators Labconco 7310000 Series | Labconco | 7310000 | |
| Deuterium oxide | Sigma-Aldrich | 151882 | |
| Grinder machine MKM6003 | Bosch | MKM6003 | |
| Licuadora Blender 8011S model Hgb2wts3 | Waring | Hgb2wts3 | |
| Methanol-d4 | Sigma-Aldrich | 151947 |
Referenzen
- Hall, R., Beale, M., Fiehn, O., Hardy, N., Sumner, L., Bino, R. Plant metabolomics: the missing link in functional genomics strategies. The Plant Cell. 14 (7), 1437-1440 (2002).
- Fiehn, O. Metabolomics-the link between genotypes and phenotypes. Plant Molecular Biology. 48 (1-2), 155-171 (2002).
- Schauer, N., Fernie, A. R. Plant metabolomics: towards biological function and mechanism. Trends in Plant Science. 11 (10), 508-516 (2006).
- Kim, H. K., Choi, Y. H., Verpoorte, R. NMR-based plant metabolomics: where do we stand, where do we go. Trends in Biotechnology. 29 (6), 267-275 (2011).
- Kumar, R., Bohra, A., Pandey, A. K., Pandey, M. K., Kumar, A. Metabolomics for Plant Improvement: Status and Prospects. Frontiers in Plant Science. 8, (2017).
- Dumez, J. -. N., et al. Hyperpolarized NMR of plant and cancer cell extracts at natural abundance. Analyst. 140 (17), 5860-5863 (2015).
- Emwas, A. -. H., et al. NMR Spectroscopy for Metabolomics Research. Metabolites. 9 (7), (2019).
- Zangger, K. Pure shift NMR. Progress in Nuclear Magnetic Resonance Spectroscopy. 86-87, 1-20 (2015).
- Foroozandeh, M., Morris, G. A., Nilsson, M. PSYCHE Pure Shift NMR Spectroscopy. Chemistry - A European Journal. 24 (53), 13988-14000 (2018).
- Castañar, L. Pure shift NMR: Past, present, and future. Magnetic Resonance in Chemistry. 56 (10), 874-875 (2018).
- Lopez, J. M., Cabrera, R., Maruenda, H. Ultra-Clean Pure Shift 1 H-NMR applied to metabolomics profiling. Scientific Reports. 9 (1), 1-8 (2019).
- Marcó, N., Gil, R. R., Parella, T. Isotropic/Anisotropic NMR Editing by Resolution-Enhanced NMR Spectroscopy. Chemphyschem: A European Journal of Chemical Physics and Physical Chemistry. 19 (9), 1024-1029 (2018).
- Kaltschnee, L., et al. Extraction of distance restraints from pure shift NOE experiments. Journal of Magnetic Resonance. 271, 99-109 (2016).
- Sinnaeve, D., et al. Improved Isotopic Profiling by Pure Shift Heteronuclear 2D J-Resolved NMR Spectroscopy. Analytical Chemistry. 90 (6), 4025-4031 (2018).
- Timári, I., et al. Real-Time Pure Shift HSQC NMR for Untargeted Metabolomics. Analytical Chemistry. 91 (3), 2304-2311 (2019).
- Zhao, Q., et al. Combination of pure shift NMR and chemical shift selective filters for analysis of Fischer-Tropsch waste-water. Analytica Chimica Acta. 1110, 131-140 (2020).
- Zhao, Q., et al. Pure Shift NMR: Application of 1D PSYCHE and 1D TOCSY-PSYCHE Techniques for Directly Analyzing the Mixtures from Biomass-Derived Platform Compound Hydrogenation/Hydrogenolysis. ACS Sustainable Chemistry & Engineering. 9 (6), 2456-2464 (2021).
- Foroozandeh, M., et al. Ultrahigh-Resolution Diffusion-Ordered Spectroscopy. Angewandte Chemie International Edition. 55 (50), 15579-15582 (2016).
- Castañar, L., Pérez-Trujillo, M., Nolis, P., Monteagudo, E., Virgili, A., Parella, T. Enantiodifferentiation through Frequency-Selective Pure-Shift 1H Nuclear Magnetic Resonance Spectroscopy. ChemPhysChem. 15 (5), 854-857 (2014).
- Lopez, J. M., Sánchez, L. F., Nakamatsu, J., Maruenda, H. Study of the Acetylation Pattern of Chitosan by Pure Shift NMR. Analytical Chemistry. , (2020).
- Foroozandeh, M., Adams, R. W., Meharry, N. J., Jeannerat, D., Nilsson, M., Morris, G. A. Ultrahigh-Resolution NMR Spectroscopy. Angewandte Chemie International Edition. 53 (27), 6990-6992 (2014).
- Foroozandeh, M., Adams, R. W., Kiraly, P., Nilsson, M., Morris, G. A. Measuring couplings in crowded NMR spectra: pure shift NMR with multiplet analysis. Chemical Communications. 51 (84), 15410-15413 (2015).
- Moutzouri, P., et al. Ultraclean pure shift NMR. Chemical Communications. 53 (73), 10188-10191 (2017).
- Santacruz, L., Hurtado, D. X., Doohan, R., Thomas, O. P., Puyana, M., Tello, E. Metabolomic study of soft corals from the Colombian Caribbean: PSYCHE and 1 H-NMR comparative analysis. Scientific Reports. 10 (1), 5417 (2020).
- Stark, P., Zab, C., Porzel, A., Franke, K., Rizzo, P., Wessjohann, L. A. PSYCHE-A Valuable Experiment in Plant NMR-Metabolomics. Molecules. 25 (21), 5125 (2020).
- Kakita, V. M. R., Rachineni, K., Hosur, R. V. Ultraclean Pure Shift NMR Spectroscopy with Adiabatic Composite Refocusing Pulses: Application to Metabolite Samples. ChemistrySelect. 4 (34), 9893-9896 (2019).
- Bo, Y., et al. High-resolution pure shift NMR spectroscopy offers better metabolite discrimination in food quality analysis. Food Research International. 125, 108574 (2019).
- Watermann, S., Schmitt, C., Schneider, T., Hackl, T. Comparison of Regular, Pure Shift, and Fast 2D NMR Experiments for Determination of the Geographical Origin of Walnuts. Metabolites. 11 (1), 39 (2021).
- Leyva-Zegarra, V., et al. NMR-based leaf metabolic profiling of V. planifolia and three endemic Vanilla species from the Peruvian Amazon. Food Chemistry. , 129365 (2021).
- Toubiana, D., et al. Morphological and metabolic profiling of a tropical-adapted potato association panel subjected to water recovery treatment reveals new insights into plant vigor. The Plant Journal. 103 (6), 2193-2210 (2020).
- Maruenda, H., Cabrera, R., Cañari-Chumpitaz, C., Lopez, J. M., Toubiana, D. NMR-based metabolic study of fruits of Physalis peruviana L. grown in eight different Peruvian ecosystems. Food Chemistry. 262, 94-101 (2018).
- Cloarec, O., et al. Statistical total correlation spectroscopy: an exploratory approach for latent biomarker identification from metabolic 1H NMR data sets. Analytical Chemistry. 77 (5), 1282-1289 (2005).
- Steuer, R. Review: on the analysis and interpretation of correlations in metabolomic data. Briefings in Bioinformatics. 7 (2), 151-158 (2006).
- Trygg, J., Holmes, E., Lundstedt, T. Chemometrics in Metabonomics. Journal of Proteome Research. 6 (2), 469-479 (2007).
- Worley, B., Powers, R. Multivariate Analysis in Metabolomics. Current Metabolomics. 1 (1), 92-107 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten