Method Article
Xenopus laevis Preparazione dell'estratto di uova e metodi di imaging dal vivo per la visualizzazione dell'organizzazione citoplasmatica dinamica
In questo articolo
Riepilogo
Descriviamo un metodo per la preparazione e l'imaging dal vivo di estratti citoplasmatici non diluiti da uova di Xenopus laevis .
Abstract
Tradizionalmente utilizzati per saggi biochimici di massa, gli estratti di uova di Xenopus laevis sono emersi come un potente strumento basato sull'imaging per lo studio di fenomeni citoplasmatici, come la citochinesi, la formazione del fuso mitotico e l'assemblaggio del nucleo. Basandosi sui primi metodi che riprendevano estratti fissi campionati in punti temporali sparsi, i recenti approcci hanno ottenuto estratti live utilizzando la microscopia time-lapse, rivelando caratteristiche più dinamiche con una risoluzione temporale migliorata. Questi metodi di solito richiedono sofisticati trattamenti superficiali del vaso di imaging. Qui introduciamo un metodo alternativo per l'imaging dal vivo di estratti di uova che non richiedono alcun trattamento chimico superficiale. È semplice da implementare e utilizza materiali di consumo di laboratorio prodotti in serie per l'imaging. Descriviamo un sistema che può essere utilizzato sia per la microscopia a campo largo che per quella confocale. È progettato per l'imaging di estratti in un campo 2-dimensionale (2D), ma può essere facilmente esteso all'imaging in 3D. È adatto per studiare la formazione di modelli spaziali all'interno del citoplasma. Con dati rappresentativi, dimostriamo la tipica organizzazione dinamica di microtubuli, nuclei e mitocondri in estratti interfasici preparati con questo metodo. Questi dati di immagine possono fornire informazioni quantitative sulla dinamica citoplasmatica e sull'organizzazione spaziale.
Introduzione
Il citoplasma costituisce il volume principale di una cellula e ha un'organizzazione distinta. Gli ingredienti del citoplasma eucariotico possono auto-assemblarsi in una vasta gamma di strutture spaziali, come gli astri dei microtubuli e l'apparato di Golgi, che a loro volta sono disposti dinamicamente e girati a seconda dell'identità della cellula e dello stato fisiologico. Comprendere l'organizzazione spaziale del citoplasma e il suo legame con le funzioni cellulari è quindi importante per capire come funziona la cellula. Gli estratti di uova di Xenopus laevis sono stati tradizionalmente utilizzati per saggi biochimici di massa 1,2,3,4,5,6,7,8, ma recenti lavori li stabiliscono come un potente sistema di imaging dal vivo per studi meccanicistici delle strutture citoplasmatiche e delle loro funzioni cellulari 9,10,11, 12,13,14,15,16,17,18. Questi estratti non diluiti preservano molte strutture e funzioni del citoplasma, consentendo manipolazioni dirette di contenuti citoplasmatici non ottenibili nei modelli cellulari convenzionali19,20. Ciò li rende ideali per caratterizzare i fenomeni citoplasmatici e sezionare le loro basi meccanicistiche.
I metodi esistenti per l'imaging degli estratti richiedono modifiche chimiche della superficie o la fabbricazione di dispositivi microfluidici. Un metodo basato su coprivetrini richiede la passivazione al glicole polietilenico (PEG) dei vetrini di vetro21. Un metodo basato su microemulsioni richiede la deposizione da vapore di tricloro(1H,1H,2H,2H-perfluoroottil)silano su superfici vetrate22,23. I sistemi basati sulla microfluidica consentono un controllo preciso del volume, della geometria e della composizione delle goccioline di estratto, ma richiedono strutture di microfabbricazione specializzate11,12,24.
Qui introduciamo un metodo alternativo per l'imaging degli estratti di uova che è facile da implementare e utilizza materiali prontamente disponibili e a basso costo. Ciò include la preparazione di una camera di imaging con un vetrino e un coprivetrino rivestito con nastro fluorurato di etilene propilene (FEP). La camera può essere utilizzata per l'imaging di estratti con una varietà di sistemi di microscopia, tra cui stereoscopi e microscopi verticali e invertiti. Questo metodo non richiede alcun trattamento chimico delle superfici, pur ottenendo una chiarezza ottica simile ottenuta con i metodi esistenti a base di vetro discussi sopra. È progettato per visualizzare uno strato di estratti con uno spessore uniforme su un campo 2D e può essere facilmente esteso per visualizzare un volume 3D di estratti. È adatto per l'imaging time-lapse del comportamento citoplasmatico collettivo su un ampio campo visivo.
Abbiamo utilizzato estratti di uova interfasici per dimostrare il nostro metodo di imaging. La preparazione dell'estratto segue il protocollo di Deming e Kornbluth19. In breve, le uova naturalmente arrestate in metafase della meiosi II vengono schiacciate da una rotazione a bassa velocità. Questo spin rilascia il citoplasma dall'arresto meiotico e consente all'estratto di procedere in interfase. Normalmente, la citocalasina B viene aggiunta prima dello spin di schiacciamento per inibire la formazione di F-actina. Tuttavia, può essere omesso se si desidera F-actina. La cicloeximide viene anche aggiunta prima dello spin di schiacciamento per impedire all'estratto interfase di entrare nella mitosi successiva. Gli estratti vengono successivamente collocati nelle suddette camere di imaging e posti su un microscopio. Infine, le immagini vengono registrate nel tempo a intervalli definiti da una fotocamera collegata al microscopio, producendo serie di immagini time-lapse che catturano il comportamento dinamico dell'estratto in un campo 2D.
Protocollo
Tutti i metodi qui descritti sono stati approvati dall'Institutional Animal Care and Use Committee (IACUC) della Stanford University.
1. Preparazione di diapositive e copertine
- Applicare uno strato di nastro adesivo fluorurato di etilene propilene (FEP) su un vetrino con un applicatore a rullo. Tagliare il nastro adesivo eccessivo sui bordi con una lama di rasoio pulita. Preparare i coprivetrini rivestiti con nastro FEP nello stesso modo (Figura 1A).
- Applicare un distanziatore di immagini adesivo fronte-retro sul lato rivestito con nastro FEP del vetrino. Lasciare la fodera protettiva sulla parte superiore non sbucciata (Figura 1A).
NOTA: Le diapositive e le copertine devono essere preparate prima dell'esperimento. Possono essere utilizzati immediatamente o conservati in scatole per evitare l'accumulo di polvere sulle superfici. Il pozzo nel distanziatore di imaging è profondo 120 μm e ha un diametro di 9 mm.
2. Preparazione e imaging dal vivo di estratti di uova interfasiche
NOTA: Il seguente protocollo è adattato da Deming e Kornbluth19, Murray20 e Smythe e Newport25 con modifiche. Tutti i passaggi devono essere eseguiti a temperatura ambiente, se non diversamente specificato.
- Da tre a dieci giorni prima della raccolta delle uova, iniettare rane mature di Xenopus laevis per via sottocutanea nel sacco linfatico dorsale con 100 UI di gonadotropina sierica di cavalla gravida (PMSG).
- Da sedici a diciotto ore prima della raccolta delle uova pianificata, iniettare le rane dal punto 2.1 con 500 UI di gonadotropina corionica umana (hCG). Lasciare le rane a 18 °C nel tampone per la deposizione delle uova (100 mM NaCl, 2 mM KCl, 1 mM MgSO 4·7H 2 O, 2,5mM CaCl 2·2H2 O, 0,5 mM HEPES, 0,1 mM EDTA, preparare come soluzione madre 20x a pH7,4 e diluire con acqua pulita del serbatoio di rana a 1x prima dell'uso) fino alla raccolta delle uova.
- Il giorno dell'esperimento, raccogli le uova in una grande piastra di Petri di vetro e valuta la qualità delle uova. Scartare tutte le uova che sembrano palline bianche gonfie o che appaiono in una stringa (Figura 1B). Esaminare le uova sotto uno stereoscopio, mantenere le uova con aspetto normale (Figura 1C) e scartare quelle con pigmento irregolare o screziato (Figura 1D).
NOTA: Questo protocollo funziona con uova raccolte da una singola rana, che in genere depone 25 ml di uova entro 16 ore dopo l'iniezione di hCG. Di solito, un totale di 3-6 rane sono indotte da hCG e la rana con la più alta qualità dell'uovo viene scelta per l'esperimento di preparazione dell'estratto. - Trasferire le uova in un becher di vetro da 400 ml e rimuovere il più possibile il tampone per la deposizione delle uova mediante decantazione.
- Incubare le uova in 100 ml di soluzione dejellying appena preparata (2% p/v di L-cisteina in acqua, regolare a pH 8,0 con NaOH) e ruotarle delicatamente periodicamente. Dopo circa 3 minuti, versare la soluzione e aggiungere 100 ml di soluzione fresca di dejelly. Continuare l'incubazione fino a quando le uova sono ben imballate (senza spazio tra le uova), ma evitare di lasciare le uova nella soluzione di dejellying per più di un totale di 5 minuti.
- Rimuovere il più possibile la soluzione di degelatinaggio mediante decantazione e lavare le uova in tampone MMR 0,25x (25 mM NaCl, 0,5 mM KCl, 0,25 mM MgCl 2, 0,5 mM CaCl2, 0,025 mM EDTA, 1,25 mM HEPES, preparato come soluzione madre 10x, regolato a pH 7,8 con NaOH e diluito in acqua Milli-Q prima dell'uso) aggiungendo il tampone, Ruotando le uova e poi versando il tampone. Ripetere alcune volte fino a quando non viene utilizzato un totale di 1 L del tampone per i lavaggi.
- Lavare le uova alcune volte con un totale di 400 ml di tampone di lisi delle uova (250 mM di saccarosio, 10 mM HEPES, 50 mM KCl, 2,5 mM MgCl2, 1 mM DTT, reso fresco e regolato a pH 7,7 con KOH). Rimuovere le uova con aspetto anomalo usando una pipetta Pasteur tra i lavaggi.
NOTA: Le uova con aspetto anormale si riferiscono a quelle che sembrano palline bianche gonfie (Figura 1B), hanno pigmentazione chiazzata (Figura 1D), si stanno deteriorando con una regione bianca in crescita (Figura 1E) o mostrano un'area pigmentata scura e contratta nell'emisfero animale (Figura 1F). - Utilizzando una pipetta di trasferimento con la punta ben aperta, trasferire le uova in una provetta da centrifuga a fondo tondo da 17 mL contenente 1 mL di tampone per lisi delle uova. Girare il tubo in una centrifuga clinica a 400 x g per 15 secondi per confezionare le uova.
- Rimuovere il più possibile il tampone di lisi delle uova dalla parte superiore delle uova confezionate utilizzando una pipetta Pasteur.
NOTA: È importante rimuovere quanto più tampone possibile dalle uova confezionate, al fine di ridurre al minimo la diluizione dell'estratto di uova. A volte è necessario rimuovere alcune uova sciolte insieme al tampone residuo per raggiungere questo obiettivo. - Determinare il volume approssimativo delle uova confezionate, quindi aggiungere 5 μg/mL di aprotinina, 5 μg/mL di leupeptina, 5 μg/mL di citocalasina B e 50 μg/mL di cicloeximide direttamente sopra le uova confezionate.
NOTA: Aprotinina e leupeptina sono inibitori della proteasi. La citocalasina B inibisce la polimerizzazione dell'actina, impedendo all'estratto di contrarsi e gelificare26. La cicloeximide inibisce la sintesi proteica, mantenendo così l'estratto nell'interfase del ciclo cellulare. - Schiacciare le uova centrifugando il tubo a 12.000 x g, 4 °C, per 15 minuti, in un rotore oscillante.
NOTA: Alla fine della centrifugazione, le uova dovrebbero essersi rotte e il lisato separato in tre strati principali: uno strato lipidico giallo in alto, l'estratto citoplasmatico (chiamato anche estratto grezzo) nel mezzo e uno strato denso scuro contenente i granuli di pigmento nella parte inferiore (Figura 1G). - Collegare un ago da 18 gauge a una siringa. Con la punta dell'ago smussata rivolta verso l'alto, forare il tubo dal lato nella parte inferiore dello strato citoplasmatico e recuperare l'estratto disegnando lentamente.
NOTA: Prelevare lentamente l'estratto citoplasmatico per evitare l'inclusione di contenuto contaminante dallo strato lipidico giallo. - Trasferire l'estratto citoplasmatico recuperato in una nuova provetta da microcentrifuga e tenerlo in ghiaccio. Utilizzare l'estratto entro 1 ora.
- Quando è pronto per l'immagine, integrare l'estratto con i reagenti desiderati e le sonde di imaging a fluorescenza.
NOTA: Le sonde di imaging a fluorescenza etichettano strutture citoplasmatiche specifiche in modo che possano essere visualizzate da un microscopio a fluorescenza. - Rimuovere il rivestimento protettivo superiore dal distanziatore di imaging sul vetrino preparato al punto 1.2 e depositare circa 7 μL di estratto al centro del pozzetto. Applicare immediatamente il coprislip rivestito con nastro FEP con il lato FEP rivolto verso l'estratto per sigillare il pozzetto. Procedere rapidamente all'imaging (Figura 1H,I).
- Impostare il vetrino su un microscopio invertito o verticale con un palco motorizzato e una fotocamera digitale. Immagina gli estratti nelle posizioni spaziali e negli intervalli di tempo desiderati sia nei canali a campo chiaro che in quelli a fluorescenza.
NOTA: in genere, per l'imaging viene utilizzato un obiettivo 5x. Il palco motorizzato consente l'acquisizione automatica delle immagini in più posizioni spaziali definite. La microscopia a campo chiaro visualizza le strutture citoplasmatiche con diversi gradi di trasparenza. La microscopia a fluorescenza visualizza le strutture citoplasmatiche specificamente etichettate dalle sonde di imaging a fluorescenza aggiunte al punto 2.14. La telecamera registra immagini time-lapse di queste strutture, catturando così le dinamiche dell'organizzazione citoplasmatica.
Risultati
Gli estratti di uovo di Xenopus laevis possono essere utilizzati per studiare l'auto-organizzazione del citoplasma durante l'interfase. La Figura 2A mostra i risultati di un esperimento riuscito. Abbiamo integrato estratti interfasici con nuclei di spermatozoi demembranati di Xenopus laevis 19 ad una concentrazione di 27 nuclei / μL e 0,38 μM purificati GST-GFP-NLS 27,28,29,30 (proteina di fusione costituita da glutatione-S-transferasi, proteina fluorescente verde e una sequenza di localizzazione nucleare) per consentire la ricostituzione e la visualizzazione dei nuclei interfase. Abbiamo anche aggiunto 1 μM di tubulina marcata con fluorescenza per visualizzare i microtubuli e 500 nM MitoTracker Red CMXRos per visualizzare i mitocondri. Pochi istanti dopo che l'estratto è stato inserito nella camera di imaging, il citoplasma è apparso disorganizzato. Nel corso dei successivi 30 minuti a temperatura ambiente, il citoplasma ha iniziato ad auto-organizzarsi in compartimenti simili a cellule. Gli astri dei microtubuli sono cresciuti dai centrosomi introdotti con i nuclei degli spermatozoi e hanno formato zone di confine impoverite dai microtubuli quando si sono incontrati con i microtubuli degli astri vicini. Proteina GST-GFP-NLS traslocata nei nuclei interfase rotondi auto-assemblati dai nuclei di spermatozoi demembranati aggiunti. Le aree impoverite di componenti citoplasmatiche di diffusione della luce erano visibili sia nei canali del campo luminoso che nei mitocondri (Figura 2A, 20 min e 35 min). Anche i mitocondri si sono impoveriti dai bordi stabiliti dai microtubuli e si sono arricchiti in compartimenti isolati che si sono allineati con i compartimenti dei microtubuli. Entro 60 minuti a temperatura ambiente, un modello spaziale costituito da compartimenti simili a cellule dovrebbe essere ben stabilito, con microtubuli che formano una struttura vuota simile a una corona e mitocondri chiaramente suddivisi in ciascun compartimento (Figura 2A, 53 min).
La Figura 2B confronta le prestazioni degli estratti nelle camere di imaging con e senza nastri FEP su vetro. Abbiamo integrato estratti interfasici con nuclei spermatici demembranati di Xenopus laevis 19 ad una concentrazione di 27 nuclei / μL e 0,35 μM GST-mCherry-NLS27,28,29,30 (proteina di fusione costituita da glutatione-S-transferasi, proteina fluorescente mCherry e una sequenza di localizzazione nucleare) per consentire la ricostituzione e la visualizzazione dei nuclei interfase. Abbiamo anche aggiunto 1 μM di tubulina marcata con fluorescenza per visualizzare i microtubuli. Le differenze dinamiche sono diventate evidenti di circa 20 minuti a temperatura ambiente. Nella camera realizzata con vetro nastrato FEP, l'estratto si auto-organizza in normali modelli simili a cellule (Figura 2B, immagini nelle righe 1 e 3). Tuttavia, nella camera in cui le superfici di vetro non erano coperte dal nastro FEP (non passivato), l'estratto mostrava schemi anomali di campo luminoso e microtubuli che diventavano sempre più interrotti nel tempo (Figura 2B, immagini nelle righe 2 e 4). Non sono state osservate differenze significative nell'importazione nucleare della proteina GST-mCherry-NLS (Figura 2B, immagini nelle righe 5 e 6).

Figura 1: Schemi e foto relativi alla procedura sperimentale. (A) Schema per la preparazione di vetrini e vetrini rivestiti di nastro FEP. B) uova di Xenopus laevis depositate in uova ovaiole, con esempi di uova di scarsa qualità indicate da frecce. Frecce gialle, esempi di uova che sembrano palline bianche e gonfie. Frecce rosse, esempi di uova che appaiono in una stringa. C) Esempi di uova di Xenopus laevis dall'aspetto normale. D) Esempi di uova di scarsa qualità con pigmento irregolare o chiazzato. (E) Un uovo deteriorato con una regione bianca in crescita. (F) Un uovo che mostra un'area pigmentata scura e contratta, probabilmente a causa dell'attivazione partenogenetica. (G) Gli strati formati dalle uova di Xenopus laevis rotte dopo la centrifugazione di 12.000 x g nella fase 2.11. (H) Schemi per la preparazione della camera di imaging dell'estratto al punto 2.15. (I) Una foto di una camera di imaging preparata con un estratto di uova all'interno. (C) e (D) condividono la stessa barra della scala nella parte inferiore di (D). (E) e (F) condividono la stessa barra di scala nella parte inferiore di (F). Le barre della scala in (B) (C) (D) (E) (F) e (I) sono approssimative. Fare clic qui per visualizzare una versione ingrandita di questa figura.
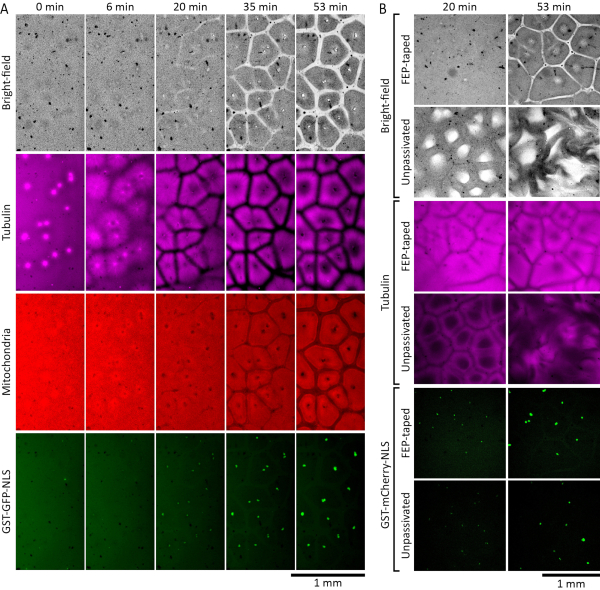
Figura 2: Gli estratti di uova arrestati interfase si auto-organizzano in compartimenti simili a cellule . (A) Montaggio time-lapse della formazione di pattern auto-organizzati in uno strato sottile (120 μm) di estratto di uovo di Xenopus laevis interfase-arrestato. L'estratto è stato integrato con 27 nuclei/μL di nuclei spermatici demembranati di Xenopus laevis per consentire la ricostituzione dei nuclei interfase. I microtubuli sono stati visualizzati dalla tubulina marcata con HiLyte 647 (mostrata in magenta), dai mitocondri da MitoTracker Red CMXRos (mostrata in rosso) e dai nuclei da GST-GFP-NLS (mostrata in verde). (B) Formazione di pattern auto-organizzati in estratti di uova di Xenopus laevis interphase-arrested collocati in camere con e senza superfici di vetro ricoperte di nastro FEP. Gli estratti sono stati integrati con 27 nuclei/μL di nuclei spermatici di Xenopus laevis demembranati per consentire la ricostituzione dei nuclei interfase. I microtubuli sono stati visualizzati dalla tubulina marcata con HiLyte 488 (mostrata in magenta) e i nuclei da GST-mCherry-NLS (mostrati in verde). Fare clic qui per visualizzare una versione ingrandita di questa figura.
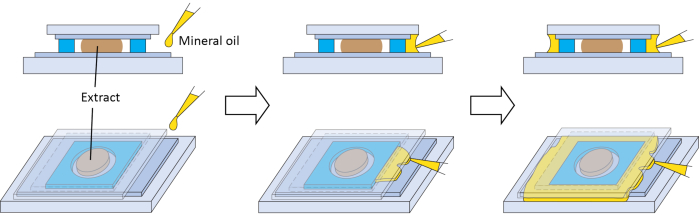
Figura 3: Guarnizione secondaria opzionale per la camera di imaging. Schemi per la preparazione di una guarnizione secondaria opzionale con olio minerale per evitare che l'estratto entri in contatto prolungato con l'aria. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Discussione
Gli estratti di uova di Xenopus laevis sono emersi come un potente sistema modello per studi basati sull'imaging di varie strutture subcellulari 10,14,15,16,17,18,21,31,32,33,34,35,36 e organizzazione citoplasmatica nel suo complesso scala cellulare9. Qui abbiamo descritto un metodo di imaging dal vivo adatto per visualizzare l'organizzazione citoplasmatica dinamica in 2D. L'efficacia del metodo è dimostrata da risultati rappresentativi.
Diversi passaggi sono fondamentali per il successo del metodo. La qualità delle uova è importante per gli estratti, quindi i passaggi 2.3 e 2.7 sono fondamentali. Nella nostra esperienza, l'emisfero animale delle uova di alta qualità ha una pigmentazione uniforme, un punto bianco distinto al centro (indicativo della rottura della vescicola germinale, GVBD) e un bordo chiaro con l'emisfero vegetale (Figura 1C). Gli estratti ottenuti da uova di alta qualità hanno prestazioni migliori nelle nostre condizioni di imaging. Se più del 5% delle uova ha pigmento screziato (Figura 1D), ha un'area pigmentata scura e contratta (Figura 1F), assomiglia a palline bianche gonfie (Figura 1B), appare in una stringa (Figura 1B) o mostra segni di deterioramento dopo i lavaggi nei passaggi 2.6 e 2.7 (Figura 1E), allora l'intero lotto di uova deve essere scartato. Gli estratti mantengono una notevole attività biologica perché sono essenzialmente citoplasma non diluito. Pertanto, il passaggio che mira a ridurre al minimo la diluizione dell'estratto (passo 2.9) è importante per il successo dell'esperimento. Per l'imaging, gli estratti vengono gestiti in volumi molto piccoli ed evaporano rapidamente se a contatto prolungato con l'aria. Ciò influenzerà negativamente la loro attività. Pertanto, al punto 2.15, dopo che l'estratto è stato depositato, deve essere sigillato con il lato rivestito di nastro FEP del vetrino di copertura il più presto possibile. La sigillatura può essere confermata visivamente dal cambiamento di texture nel sito di contatto tra l'adesivo sul distanziatore e il coprivetrino. Il distanziatore dovrebbe essere in grado di creare una tenuta completa se applicato correttamente. Tuttavia, se si desidera una guarnizione aggiuntiva, l'olio minerale può essere erogato tra la sporgenza del vetrino e il vetrino. L'olio può formare un sigillo aggiuntivo attorno al distanziatore per azione capillare (Figura 3). La passivazione delle superfici vetrate può ridurre l'adsorbimento non specifico delle molecole, ed è importante per gli astri di microtubuli interfase negli estratti21,37. L'applicazione del nastro FEP su una superficie di vetro qui descritta sembra offrire vantaggi simili, come suggerito dall'assemblaggio di normali astri di microtubuli interfase (Figura 2, 6 min). Pertanto, anche il passaggio 1.1 è fondamentale.
Abbiamo dimostrato l'applicazione di un metodo di imaging utilizzando estratti di uova interfasiche arrestate seguendo il protocollo di Deming e Kornbluth19. Per impostazione predefinita, il protocollo integra gli estratti con l'inibitore della polimerizzazione dell'actina citocalasina B per prevenire la gelificazione-contrazione negli estratti dopo un'incubazione prolungata a temperatura ambiente26. Per consentire la polimerizzazione dell'actina e osservare la dinamica dell'actina, si può tralasciare la citocalasina B nel passo 2.10 del protocollo9. Una modifica che abbiamo apportato al protocollo di Deming e Kornbluth è che non integriamo gli estratti con un mix energetico per rigenerare l'ATP19. Questo perché nelle nostre mani, negli estratti integrati con questo mix rigenerante ATP19 e nuclei spermatici, i microtubuli occasionalmente formano un reticolo reticolato che interferisce con la formazione del pattern citoplasmatico. Pertanto, il protocollo non include il passaggio che aggiunge il mix energetico agli estratti19.
Il protocollo di estrazione interfase si basa sulla frantumazione delle uova in tampone privo di EGTA per liberarle dall'arresto meiotico (arresto CSF)37. Gli estratti successivamente progrediscono in interfase e sono mantenuti in interfase dal cicloeximide. Esistono altri metodi consolidati per preparare estratti interfase. Alcuni metodi preparano prima estratti che mantengono l'arresto meiotico schiacciando le uova nel tampone di lisi con EGTA38, e quindi rilasciano l'arresto aggiungendo calcio, guidando l'estratto nell'interfase 21,37,39. Gli estratti possono essere successivamente tenuti in interfase mediante aggiunta di inibitori della sintesi proteica come la cicloeximide37,39. Altri metodi partenogeneticamente attivano le uova con ionoforo di calcio (A23187) o scosse elettriche per liberarle dall'arresto meiotico, prima di schiacciare le uova in assenza di EGTA20,28 (recensito in Field et al.37). Questi estratti possono entrare in interfase ma in genere non rimarranno lì in quanto sono in grado di subire più cicli cellulari. Allo stesso modo, sono stati sviluppati metodi consolidati ottimizzati per la preparazione di estratti con citoscheletro di actina intatto 10,37,39. Il metodo di imaging può essere adatto per questi tipi di estratti, ma non lo abbiamo testato con loro.
Ai fini dell'imaging dell'organizzazione interna del citoplasma, il sistema di imaging qui presentato è relativamente facile da configurare, richiedendo solo l'applicazione di un nastro FEP alle superfici vetrate. Consente l'assemblaggio di strutture citoscheletriche in estratti9 riportati in sistemi di imaging più sofisticati in cui le superfici vetrate sono passivate con trattamento poli-L-lisina-g-polietilenglicole (PLL-g-PEG) o rivestite con doppi strati lipidici supportati10,21. Il metodo consente allo strato estratto di formarsi con uno spessore definito (determinato dalla profondità del distanziatore, che è di 120 μm per il sistema mostrato in Figura 1A, 1H, 1I e Figura 2). Possiamo regolare lo spessore impilando distanziali aggiuntivi. Abbiamo impilato fino a 6 distanziatori di questo tipo (720 μm di spessore) e i compartimenti si sono formati normalmente. Questa flessibilità consente applicazioni future come l'imaging degli estratti in 3D utilizzando la microscopia confocale o a foglio leggero.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo J. Kamenz, Y. Chen e W. Y. C. Huang per i commenti sul manoscritto. Questo lavoro è stato supportato da sovvenzioni del National Institutes of Health (R01 GM110564, P50 GM107615 e R35 GM131792) assegnate a James E. Ferrell, Jr.
Materiali
| Name | Company | Catalog Number | Comments |
| 17 ml centrifuge tube | Beckman Coulter | 337986 | |
| 22x22 mm square #1 cover glass | Corning | 284522 | |
| Aprotinin | MilliporeSigma | 10236624001 | Protease inhibitor |
| Cycloheximide | MilliporeSigma | 01810 | Protein synthesis inhibitor |
| Cytochalasin B | MilliporeSigma | C6762 | Actin polymerization inhibitor |
| Female Xenopus laevis frogs | Nasco | LM00535MX | |
| Fluorescent HiLyte 488 labeled tubulin protein | Cytoskeleton, Inc. | TL488M-A | For visualizing the microtubule cytoskeleton |
| Fluorescent HiLyte 647 labeled tubulin protein | Cytoskeleton, Inc. | TL670M-A | For visualizing the microtubule cytoskeleton |
| Fluorinated ethylene propylene (FEP) optically clear tape | CS Hyde company | 23-FEP-2-5 | |
| Glass Pasteur pipette | Fisher Scientific | 13-678-20C | |
| Human chorionic gonadotropin (hCG) | MilliporeSigma | CG10 | |
| Imaging spacer | Electron Microscopy Sciences | 70327-8S | |
| Leupeptin | MilliporeSigma | 11017101001 | Protease inhibitor |
| Microscope slides | Fisher Scientific | 12-518-100B | |
| Mineral oil | MilliporeSigma | 330760 | |
| MitoTracker Red CMXRos | Thermo Fisher Scientific | M7512 | For visualizing mitochondria |
| Pregnant mare serum gonadotropin (PMSG) | BioVendor | RP1782725000 | |
| Roller applicator | Amazon | B07HMBJSP8 | For applying the FEP tape to the glass slides and coverslips |
| Single-edged razor blades | Fisher Scientific | 12-640 | For removing excessive FEP tape |
| Transfer pipette | Fisher Scientific | 13-711-7M |
Riferimenti
- Murray, A. W., Kirschner, M. W. Cyclin synthesis drives the early embryonic cell cycle. Nature. 339 (6222), 275-280 (1989).
- Dunphy, W. G., Brizuela, L., Beach, D., Newport, J. The Xenopus cdc2 protein is a component of MPF, a cytoplasmic regulator of mitosis. Cell. 54 (3), 423-431 (1988).
- Minshull, J., Golsteyn, R., Hill, C. S., Hunt, T. The A- and B-type cyclin associated cdc2 kinases in Xenopus turn on and off at different times in the cell cycle. EMBO J. 9 (9), 2865-2875 (1990).
- Wu, J. Q., et al. PP1-mediated dephosphorylation of phosphoproteins at mitotic exit is controlled by inhibitor-1 and PP1 phosphorylation. Nature Cell Biology. 11 (5), 644-651 (2009).
- Pomerening, J. R., Sontag, E. D., Ferrell, J. E. Building a cell cycle oscillator: hysteresis and bistability in the activation of Cdc2. Nature Cell Biology. 5 (4), 346-351 (2003).
- Mochida, S., Maslen, S. L., Skehel, M., Hunt, T. Greatwall phosphorylates an inhibitor of protein phosphatase 2A that is essential for mitosis. Science. 330 (6011), 1670-1673 (2010).
- Blow, J. J., Laskey, R. A. Initiation of DNA replication in nuclei and purified DNA by a cell-free extract of Xenopus eggs. Cell. 47 (4), 577-587 (1986).
- Lohka, M. J., Masui, Y. Formation in vitro of sperm pronuclei and mitotic chromosomes induced by amphibian ooplasmic components. Science. 220 (4598), 719-721 (1983).
- Cheng, X., Ferrell, J. E. Spontaneous emergence of cell-like organization in Xenopus egg extracts. Science. 366 (6465), 631-637 (2019).
- Nguyen, P. A., et al. Spatial organization of cytokinesis signaling reconstituted in a cell-free system. Science. 346 (6206), 244-247 (2014).
- Good, M. C., Vahey, M. D., Skandarajah, A., Fletcher, D. A., Heald, R. Cytoplasmic volume modulates spindle size during embryogenesis. Science. 342 (6160), 856-860 (2013).
- Hazel, J., et al. Changes in cytoplasmic volume are sufficient to drive spindle scaling. Science. 342 (6160), 853-856 (2013).
- Brownlee, C., Heald, R. Importin alpha Partitioning to the Plasma Membrane Regulates Intracellular Scaling. Cell. 176 (4), 805-815 (2019).
- Nguyen, P. A., Field, C. M., Mitchison, T. J. Prc1E and Kif4A control microtubule organization within and between large Xenopus egg asters. Molecular Biology of the Cell. 29 (3), 304-316 (2018).
- Desai, A., Maddox, P. S., Mitchison, T. J., Salmon, E. D. Anaphase A chromosome movement and poleward spindle microtubule flux occur At similar rates in Xenopus extract spindles. Journal of Cell Biology. 141 (3), 703-713 (1998).
- Mitchison, T. J., et al. Roles of polymerization dynamics, opposed motors, and a tensile element in governing the length of Xenopus extract meiotic spindles. Molecular Biology of the Cell. 16 (6), 3064-3076 (2005).
- Mitchison, T. J., et al. Bipolarization and poleward flux correlate during Xenopus extract spindle assembly. Molecular Biology of the Cell. 15 (12), 5603-5615 (2004).
- Murray, A. W., Desai, A. B., Salmon, E. D. Real time observation of anaphase in vitro. Proceedings of the National Academy of Sciences of the United States of America. 93 (22), 12327-12332 (1996).
- Deming, P., Kornbluth, S. Study of apoptosis in vitro using the Xenopus egg extract reconstitution system. Methods in Molecular Biology. 322, 379-393 (2006).
- Murray, A. W. Cell cycle extracts. Methods in Cell Biology. 36, 581-605 (1991).
- Field, C. M., Mitchison, T. J. Assembly of Spindles and Asters in Xenopus Egg Extracts. Cold Spring Harbor Protocols. 2018 (6), (2018).
- Guan, Y., et al. A robust and tunable mitotic oscillator in artificial cells. Elife. 7, (2018).
- Guan, Y., Wang, S., Jin, M., Xu, H., Yang, Q. Reconstitution of Cell-cycle Oscillations in Microemulsions of Cell-free Xenopus Egg Extracts. Journal of Visualized Experiments. (139), e58240(2018).
- Oakey, J., Gatlin, J. C. Microfluidic Encapsulation of Demembranated Sperm Nuclei in Xenopus Egg Extracts. Cold Spring Harbor Protocols. 2018 (8), (2018).
- Smythe, C., Newport, J. W. Systems for the study of nuclear assembly, DNA replication, and nuclear breakdown in Xenopus laevis egg extracts. Methods in Cell Biology. 35, 449-468 (1991).
- Field, C. M., et al. Actin behavior in bulk cytoplasm is cell cycle regulated in early vertebrate embryos. Journal of Cell Science. 124, Pt 12 2086-2095 (2011).
- Chang, J. B., Ferrell, J. E. Mitotic trigger waves and the spatial coordination of the Xenopus cell cycle. Nature. 500 (7464), 603-607 (2013).
- Chang, J. B., Ferrell, J. E. Robustly Cycling Xenopus laevis Cell-Free Extracts in Teflon Chambers. Cold Spring Harbor Protocols. 2018 (8), (2018).
- Chatterjee, S., Javier, M., Stochaj, U. In vivo analysis of nuclear protein traffic in mammalian cells. Experimental Cell Research. 236 (1), 346-350 (1997).
- Mochida, S., Hunt, T. Calcineurin is required to release Xenopus egg extracts from meiotic M phase. Nature. 449 (7160), 336-340 (2007).
- Heald, R., et al. Self-organization of microtubules into bipolar spindles around artificial chromosomes in Xenopus egg extracts. Nature. 382 (6590), 420-425 (1996).
- Helmke, K. J., Heald, R. TPX2 levels modulate meiotic spindle size and architecture in Xenopus egg extracts. Journal of Cell Biology. 206 (3), 385-393 (2014).
- Sawin, K. E., Mitchison, T. J. Mitotic spindle assembly by two different pathways in vitro. Journal of Cell Biology. 112 (5), 925-940 (1991).
- Belmont, L. D., Hyman, A. A., Sawin, K. E., Mitchison, T. J. Real-time visualization of cell cycle-dependent changes in microtubule dynamics in cytoplasmic extracts. Cell. 62 (3), 579-589 (1990).
- Krauss, S. W., Lee, G., Chasis, J. A., Mohandas, N., Heald, R. Two protein 4.1 domains essential for mitotic spindle and aster microtubule dynamics and organization in vitro. Journal of Biological Chemistry. 279 (26), 27591-27598 (2004).
- Wang, S., Romano, F. B., Field, C. M., Mitchison, T. J., Rapoport, T. A. Multiple mechanisms determine ER network morphology during the cell cycle in Xenopus egg extracts. Journal of Cell Biology. 203 (5), 801-814 (2013).
- Field, C. M., Nguyen, P. A., Ishihara, K., Groen, A. C., Mitchison, T. J. Xenopus egg cytoplasm with intact actin. Methods in Enzymology. 540, 399-415 (2014).
- Good, M. C., Heald, R. Preparation of Cellular Extracts from Xenopus Eggs and Embryos. Cold Spring Harbor Protocols. 2018 (6), (2018).
- Field, C. M., Pelletier, J. F., Mitchison, T. J. Xenopus extract approaches to studying microtubule organization and signaling in cytokinesis. Methods in Cell Biology. 137, 395-435 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon