Method Article
Produzione cell-free di proteoliposomi per l'analisi funzionale e lo sviluppo di anticorpi mirati alle proteine di membrana
In questo articolo
Riepilogo
Questo protocollo descrive un metodo efficiente privo di cellule per la produzione di proteoliosomi di alta qualità mediante metodo di dialisi a doppio strato utilizzando un sistema privo di cellule di grano e liposomi. Questo metodo fornisce mezzi adeguati per l'analisi funzionale delle proteine di membrana, lo screening dei bersagli farmacologici e lo sviluppo di anticorpi.
Abstract
Le proteine di membrana svolgono ruoli essenziali in una varietà di processi cellulari e svolgono funzioni vitali. Le proteine di membrana sono importanti dal punto di vista medico nella scoperta di farmaci perché sono gli obiettivi di oltre la metà di tutti i farmaci. Un ostacolo alla conduzione di studi biochimici, biofisici e strutturali sulle proteine di membrana e sullo sviluppo di anticorpi è stata la difficoltà di produrre grandi quantità di proteine di membrana di alta qualità con corretta conformazione e attività. Qui descriviamo un "metodo di dialisi a doppio strato" che utilizza un sistema privo di cellule di germe di grano, liposomi e coppette di dialisi per sintetizzare efficacemente le proteine di membrana e preparare proteoliposomi purificati in breve tempo con un alto tasso di successo. Le proteine di membrana possono essere prodotte tanto quanto in diversi milligrammi, come GPCR, canali ionici, trasportatori e tetraspanine. Questo metodo privo di cellule contribuisce a ridurre i tempi, i costi e gli sforzi per la preparazione di proteoliposomi di alta qualità e fornisce mezzi adeguati per l'analisi funzionale delle proteine di membrana, lo screening di bersagli farmacologici e lo sviluppo di anticorpi.
Introduzione
Le proteine di membrana sono uno dei bersagli farmacologici più importanti nella diagnosi e nella terapia. Infatti, metà dei piccoli farmaci composti bersaglio sono proteine di membrana, come i recettori accoppiati a proteine G (GPCR) e i canali ionici1. Nel corso degli anni, i ricercatori hanno lavorato su studi biochimici, biofisici e strutturali delle proteine di membrana per chiarire la loro struttura e funzione 2,3. Anche lo sviluppo di anticorpi monoclonali contro le proteine di membrana viene eseguito attivamente al fine di accelerare gli studi funzionali e strutturali e sviluppare applicazioni terapeutiche e diagnostiche 4,5,6,7,8,9. Tutti questi studi richiedono una grande quantità di proteine di membrana di alta qualità10. Ad esempio, sono necessari diversi milligrammi di proteine di membrana purificate con conformazione naturale per lo sviluppo di anticorpi. Una quantità molto maggiore di proteine di membrana altamente purificate è necessaria per la cristallografia a raggi X. Tuttavia, la produzione di massa di proteine di membrana rimane un collo di bottiglia nella ricerca sulle proteine di membrana11. Le proteine di membrana hanno strutture complicate con una o più eliche transmembrana e svolgono un ruolo importante nell'omeostasi cellulare. La sovraespressione eterologa delle proteine di membrana porta a molteplici ostacoli come l'aggregazione di proteine di membrana che si accumulano ad alte concentrazioni locali o il disturbo delle vie del segnale cellulare. Anche se l'espressione ha successo, anche le fasi successive della preparazione del campione incontrano difficoltà. Ad esempio, la preparazione del proteoliposoma, richiede competenze di alto livello ed esperienze professionali nella solubilizzazione, purificazione e stabilizzazione delle proteine di membrana, e costa molto sforzo e tempo anche12,13.
D'altra parte, alcune tecnologie avanzate sono emerse negli ultimi decenni per produrre proteine senza l'uso di cellule viventi 14,15,16,17,18. La tecnologia di sintesi proteica priva di cellule ricostituisce la reazione di traduzione in una provetta. Poiché non ci sono limitazioni che il sistema di espressione cellulare ha, i sistemi privi di cellule hanno il potenziale per sintetizzare una varietà di proteine che sono difficili da esprimere o mostrano tossicità nelle cellule. L'estratto cellulare purificato o il macchinario traslazionale ricostituito viene mescolato con mRNA modello, amminoacidi e fonti di energia e le proteine ricombinanti vengono sintetizzate in breve tempo. Per quanto riguarda la sintesi proteica di membrana, alcuni tipi di scaffold composti da lipidi o anfifili, come liposomi, bicelle, nanodischi o copolimeri vengono aggiunti alla reazione cell-free 19,20,21,22,23,24. Le proteine di membrana sintetizzate interagiscono con gli scaffold e possono essere stabilizzate in acqua. Le proteine di membrana sintetizzate prive di cellule sono ampiamente utilizzate negli studi funzionali e nella produzione di anticorpi 25,26,27,28,29,30,31.
In questo protocollo, descriviamo un metodo efficiente di produzione di proteoliposomi senza cellule utilizzando il sistema privo di cellule di grano e liposomi. Il sistema di sintesi proteica senza cellule di grano è un potente sistema di traduzione in vitro che utilizza l'estratto del germe di grano 15,32,33. Il germe di grano contiene una grande quantità di macchinari traslazionali e pochi inibitori della traduzione. Il meccanismo traslazionale nel grano, un membro degli eucarioti, è adatto per tradurre le proteine eucariotiche e la sua efficienza di traduzione è difficilmente influenzata dall'uso del codone del modello mRNA. Utilizzando il sistema privo di cellule di grano, abbiamo sintetizzato una varietà di proteine tra cui protein chinasi 34,35, ubiquitina ligasi36, fattori di trascrizione37 e proteine di membrana con alti tassi di successo. Per la produzione di proteine di membrana, aggiungiamo liposomi di vescicole lipidiche nella miscela di traduzione come scaffold19,38. I domini idrofobici delle proteine di membrana interagiscono con il doppio strato lipidico e sono spontaneamente integrati con il liposoma. La centrifugazione a gradiente di densità viene utilizzata per separare rigorosamente il proteoliosoma dalle proteine endogene del grano, anche se una centrifugazione comune della miscela della reazione di traduzione è sufficiente per una semplice purificazione del proteoliposomia20. Molti tipi di proteine integrali di membrana sono stati sintetizzati utilizzando il sistema privo di cellule di grano e applicati per varie ricerche e sviluppi 25,38,39,40,41,42,43,44. Inoltre, abbiamo sviluppato il "metodo di dialisi a doppio strato" per la produzione su larga scala45,46. In questo metodo, il dispositivo di dialisi a coppa viene immerso nel tampone di alimentazione del substrato e nella tazza si formano due strati di miscela di reazione di traslazione e tampone di alimentazione del substrato, come mostrato nella Figura 1. La fornitura continua di substrati e la rimozione del sottoprodotto possono essere condotte in modo efficiente sia nella parte superiore che in quella inferiore della miscela di reazione per lungo tempo, il che porta a un'eccellente efficacia di traduzione (Figura 2A e Figura 2B)45.
Protocollo
1. Preparazione del plasmide di espressione pEU
NOTA: il plasmide di espressione pEU deve includere il codone iniziale, il frame di lettura aperto della proteina di membrana bersaglio e il codone di arresto nel frammento (vedere Figura 1). Aggiungere sequenze di tag di rilevamento/purificazione nella posizione appropriata quando necessario. Per la subclonazione è applicabile la digestione enzimatica di restrizione o la clonazione senza soluzione di continuità. Qui descriviamo un protocollo che utilizza un metodo di clonazione senza soluzione di continuità.
- Preparare inserire un frammento di DNA.
- Amplificare il gene di interesse mediante PCR utilizzando il modello di cDNA, primer 1 e primer 2. Il primer 1 e il primer 2 contengono rispettivamente sovrapposizioni di 15 bp per una clonazione senza soluzione di continuità (vedere la tabella dei materiali).
NOTA: Non includere sequenze da elaborare e rimuovere da proteine mature nelle cellule (ad esempio, sequenza di segnali). L'elaborazione delle proteine sintetizzate non viene eseguita nel sistema privo di cellule di grano. Non aggiungere la sequenza Kozak. Il vettore pEU-E01-MCS ha un potenziatore di traduzione E01. - Aggiungere 1/25 di volume dell'enzima di restrizione DpnI al prodotto PCR per rimuovere il DNA plasmidico modello. Incubare per 30 minuti a 37 °C.
- Utilizzare un kit di purificazione PCR per purificare il prodotto PCR e regolare la concentrazione a 20-50 ng / μL.
- Amplificare il gene di interesse mediante PCR utilizzando il modello di cDNA, primer 1 e primer 2. Il primer 1 e il primer 2 contengono rispettivamente sovrapposizioni di 15 bp per una clonazione senza soluzione di continuità (vedere la tabella dei materiali).
- Linearizzare il vettore pEU-E01-MCS.
- Condurre la PCR inversa utilizzando pEU-E01-MCS, primer 3 e primer 4.
- Aggiungere 1/25 di volume dell'enzima di restrizione DpnI al prodotto PCR inverso. Incubare per 30 minuti a 37 °C.
- Utilizzare un kit di purificazione PCR per purificare il prodotto PCR secondo la raccomandazione del produttore. Regolare la concentrazione a 20–50 ng/μL.
- Mescolare 2 μL di frammento di DNA inserito, 2 μL di vettore linearizzato e 4 μL di miscela enzimatica di clonazione 2x senza soluzione di continuità.
- Trasforma Escherichia coli ceppo JM109 con il prodotto di clonazione senza soluzione di continuità. Utilizzando uno spargitore, distribuire la sospensione batterica su una piastra di agar di ampicillina LB.
NOTA: il vettore pEU ha un marcatore di resistenza all'ampicillina. - Confermare la sequenza del plasmide di espressione costruita utilizzando primer 5 e primer 6 dal lato 5' e 3' di MCS nel plasmide pEU, rispettivamente.
- Amplificare e purificare i plasmidi di espressione.
- Coltura del ceppo di E. coli trasformato plasmidico JM109 in 150 mL di mezzo di ampicillina LB a 37 °C e agitazione di 125 colpi al minuto durante la notte.
- Estrarre e purificare i plasmidi utilizzando il kit midi di preparazione del plasmide disponibile in commercio. Sciogliere i plasmidi in 500 μL di tampone TE.
ATTENZIONE: Non utilizzare un mini kit di preparazione per l'estrazione del plasmide. Non fornisce sufficiente qualità e quantità di plasmide. - Aggiungere 500 μL di fenolo/cloroformio/alcool isoamilico (25:24:1). Mescolare energicamente per 5 minuti e centrifugare per 5 minuti a 17.800 x g e temperatura ambiente. Trasferire la soluzione plasmidica superiore in un nuovo tubo.
ATTENZIONE: Indossare guanti monouso per proteggere la pelle da fenolo e cloroformio.
NOTA: Al fine di rimuovere la contaminazione della RNasi dal kit di estrazione del plasmide, purificare i plasmidi utilizzando la purificazione fenolo-cloroformio. - Aggiungere 500 μL di cloroformio per rimuovere completamente il fenolo. Mescolare energicamente per 5 minuti e centrifugare per 5 minuti a 17.800 x g e temperatura ambiente. Trasferire la soluzione plasmidica superiore in un nuovo tubo.
- Aggiungere 2,5 volume di etanolo e 1/8 di volume di acetato di ammonio 7,5 M e conservare a -30 °C per 1 ora.
- Centrifugare a 17.800 x g a 4 °C per 10 min. Lavare il pellet con 500 μL di etanolo al 70%. Rimuovere accuratamente il surnatante e lasciare asciugare il pellet per 5 minuti.
- Sciogliere completamente i plasmidi di espressione in 100 μL di acqua ultrapura. Misurare la concentrazione di plasmidi con assorbanza a 260 nm. Regolare la concentrazione a 1 mg/ml.
2. Trascrizione in vitro
ATTENZIONE: Utilizzare tubi e punte di plastica privi di DNasi e nucleasi nelle fasi di trascrizione e traduzione. Evitare l'autoclave di articoli di plastica per prevenire la contaminazione.
- Raccogliere acqua ultrapura in un nuovo tubo di plastica.
ATTENZIONE: Non utilizzare acqua trattata con DEPC perché il DEPC residuo inibisce fortemente la reazione. Utilizzare acqua ultrapura appena purificata per la trascrizione e la traduzione. - Preparare la miscela di reazione di trascrizione miscelando 115,2 μL di acqua ultrapura, 40 μL di Tampone Trascrizionale LM, 20 μL di miscela NTP, 2,4 μL di 80 U/μL di inibitore della RNasi, 2,4 μL di 80 U/μL SP6 polimerasi e 20 μL di plasmidi di espressione pEU da 1 mg/ml. Mescolare delicatamente i reagenti capovolgendo. Esegui un giro veloce.
- Incubare la reazione di trascrizione a 37 °C per 6 ore.
- Mescolare delicatamente la reazione capovolgendo e ruotare rapidamente verso il basso. Utilizzarlo immediatamente per la traduzione, altrimenti congelare e conservare a -80 °C.
- Confermare il prodotto di trascrizione mediante elettroforesi.
- Mescolare 100 ml di 1x tampone TAE e 1 g di agarosio. Riscaldare la sospensione in un forno a microonde per preparare il gel TAE di agarosio all'1%.
- Prelevare 1 μL di reazione di trascrizione e mescolare con 3 μL di acqua e 4 μL di colorante di carico 2x.
NOTA: La denaturazione dell'RNA non è richiesta. - Caricare 4 μL della miscela e 2 μL del marcatore della scala del DNA sul gel TAE dell'agarosio.
- Elettroforesi a 100 V per 20 min.
- Colorare il gel in bromuro di etidio per 30 min. Controllare il modello di banda della scala di mRNA utilizzando il transilluminatore UV e l'imager del gel.
NOTA: Quando si osserva una banda spalmata inferiore a 500 bp, si sospetta una degradazione dell'mRNA.
3. Preparazione dei materiali per la traduzione
- Preparare il buffer di traduzione.
- Mescolare 27 ml di acqua ultrapura appena preparata e 0,75 ml di ciascuna soluzione madre 40x per S1, S2, S3 e S4, rispettivamente in un tubo da 50 ml.
NOTA: modulare la quantità di materiali in base alla quantità finale richiesta di 1x buffer di traduzione.
ATTENZIONE: non conservare o ricongelare il buffer di conversione 1x in eccesso dopo l'uso.
- Mescolare 27 ml di acqua ultrapura appena preparata e 0,75 ml di ciascuna soluzione madre 40x per S1, S2, S3 e S4, rispettivamente in un tubo da 50 ml.
- Preparare la soluzione madre di creatinchinasi. Sciogliere la creatinchinasi liofilizzata in acqua ultrapura fino a una concentrazione finale di 20 mg/ml. Erogare la soluzione in piccole quantità (da 10 a 50 μL ciascuna) in provette PCR a 8 strisce da 0,2 ml. Congelare i tubi in azoto liquido e conservare a -80 °C.
ATTENZIONE: Non ricongelare la soluzione di creatinchinasi dopo lo scongelamento. - Lavare le coppette di dialisi (dimensioni 0,1 ml) per rimuovere il glicerolo dalla membrana di dialisi.
NOTA: Ci sono diverse coppe di dialisi di dimensioni diverse. Tazze di piccole dimensioni (0,1 ml) sono utilizzate rispettivamente per test su piccola scala (sezione 5.4) e tazze di grandi dimensioni (2 ml) per la produzione su larga scala (sezione 5.5). La fase di lavaggio della membrana di dialisi di coppette di grandi dimensioni è evitabile.- Mettere 1 mL di acqua ultrapura in un nuovo tubo da 1,5 mL. Inserire una coppa di dialisi di piccole dimensioni (0,1 ml) nel tubo. Aggiungere 0,5 ml di acqua ultrapura nella tazza.
- Incubare per più di 30 minuti a temperatura ambiente.
4. Preparazione dei liposomi
NOTA: Qui descriviamo due protocolli per la preparazione dei liposomi. Uno utilizza liposomi liofilizzati pronti all'uso (sezione 4.1), mentre l'altro produce liposomi idratando un sottile film lipidico (paragrafo 4.2).
- Preparare liposomi usando liposomi liofilizzati.
NOTA: Un modo più semplice per produrre proteoliposomi è quello di utilizzare liposoma Asolectin disponibile in commercio. L'asolectina è un tipo di lipide naturale estratto dalla soia.- Aprire il flaconcino contenente 10 mg di liposomi di asolectina liofilizzati (vedere la tabella dei materiali) e aggiungere 200 μL di tampone di traduzione (paragrafo 3.1) sul fondo del flaconcino. Sigillare il flaconcino e incubare per 10 min.
- Mescolare energicamente mettendo il flaconcino sul miscelatore a vortice per 1 minuto.
- Inserire il flaconcino in un tubo da 50 ml. Centrifugare il tubo centrifugando a 500 x g per 1 minuto.
- Utilizzando una pipetta, trasferire la sospensione liposomica di asolectina (50 mg lipidi/ml) in una nuova provetta da 1,5 ml. Utilizzare immediatamente il liposoma per la traduzione, altrimenti congelare in azoto liquido e conservare a -80 °C.
- Preparare i liposomi idratando un sottile film lipidico.
- Se un lipide viene venduto in polvere, sciogliere in cloroformio o solvente organico appropriato a 10-100 mg / mL concentrazione.
NOTA: Un sottile film lipidico può essere preparato utilizzando lipidi anfifilici purificati e/o sintetizzati. Il metodo di purificazione dell'asolectina è precedentemente descritto38. I lipidi funzionalmente modificati, come i lipidi biotinilati, i lipidi fluorescenti e i lipidi adiuvanti, possono essere aggiunti ai lipidi basali per produrre liposomi funzionali. - Trasferire la soluzione lipidica contenente 50 mg di lipidi in un matraccio di evaporazione.
- Utilizzando un evaporatore rotante, far evaporare il solvente e distribuire uniformemente il lipide sulla parete del fondo del pallone per formare un sottile film di lipidi.
- Mettere il pallone in un essiccatore sottovuoto e lasciarlo sotto pressione negativa per una notte per rimuovere completamente il solvente.
- Aggiungere 1 mL di tampone di traslazione al matraccio di evaporazione. Ruotare il matraccio per distribuire il tampone sul sottile film lipidico. Incubare per 5 minuti per idratare il film.
- Sonicare il pallone con un omogeneizzatore ad ultrasuoni o un pulitore ad ultrasuoni. Cambiare di tanto in tanto l'angolazione del matraccio per consentire alla soluzione di toccare completamente la pellicola. Assicurarsi che il sottile film lipidico venga staccato dal fondo ed emulsionato in modo completo e omogeneo.
NOTA: La micrografia elettronica dei lipidi biotinilati contenenti liposomi è mostrata nella Figura 1. - Trasferire la sospensione liposomica (50 mg lipidi/ml) in un nuovo tubo da 1,5 ml. Se non deve essere utilizzato immediatamente, congelare i liposomi in azoto liquido e conservare a -80 °C.
- Se un lipide viene venduto in polvere, sciogliere in cloroformio o solvente organico appropriato a 10-100 mg / mL concentrazione.
5. Traduzione in vitro
- Scongelare rapidamente l'estratto di germe di grano facendo galleggiare i tubi sull'acqua a temperatura ambiente per alcuni minuti. Dopo lo scongelamento, mescolare immediatamente delicatamente capovolgendo i tubi, girare verso il basso e raffreddare sul ghiaccio fino all'uso.
NOTA: Congelare l'estratto di germe di grano in azoto liquido dopo l'uso e conservare a -80 °C. Resiste a diversi cicli di congelamento/scongelamento. - Scongelare 20 mg/mL di soluzione madre di creatinchinasi. Miscelare 5 μL di soluzione madre e 45 μL di tampone di traduzione per preparare 2 mg/mL di soluzione di lavoro di creatinchinasi.
ATTENZIONE: Il ricongelamento della creatinchinasi non è raccomandato. - Scongelare liposomi o mRNA quando necessario.
- Condurre una traduzione proteica su piccola scala.
- Rimuovere l'acqua sia dal tubo che dalla coppa di dialisi (0,1 ml), come preparato al punto 3.3.2.
- Iniettare 1 mL e 300 μL di tampone di traslazione rispettivamente nel tubo e nella coppa di dialisi.
ATTENZIONE: Nel caso in cui il fondo della coppa di dialisi non raggiunga la superficie del tampone di traslazione nel tubo da 1,5 ml, iniettare altri 50-100 μL di tampone nel tubo. - Preparare la miscela della reazione di traduzione miscelando 15,6 μL di tampone di traduzione, 2,4 μL di 2 mg/ml di creatina chinasi, 12 μL di 50 mg/ml di liposomi, 15 μL di estratto di germe di grano e 15 μL di mRNA. Mescolare delicatamente capovolgendo i tubi e ruotare verso il basso.
- Aspirare 60 μL della miscela della reazione di traslazione utilizzando una pipetta da 200 μL.
- Inserire la punta della pipetta nella superficie inferiore del tampone di traslazione nella coppa di dialisi. Pipettare la miscela di reazione lentamente e delicatamente. Coprire la coppa di dialisi con un coperchio per evitare l'evaporazione.
NOTA: La miscela di reazione affonda naturalmente sul fondo della tazza e forma un doppio strato. Non disturbare il doppio strato mescolando o agitando la tazza.
- Eseguire una traduzione su larga scala (Figura 1).
- Versare 22 mL di tampone di traslazione in una provetta da 25 mL. Inserire una coppa di dialisi di grandi dimensioni (2 ml) nel tubo e aggiungere 2 ml di tampone di traslazione nella tazza.
- Preparare una miscela di reazione di traduzione mescolando 130 μL di tampone di traduzione, 20 μL di 2 mg/mL di creatina chinasi, 100 μL di 50 mg/ml di liposoma, 125 μL di estratto di germe di grano e 125 μL di mRNA. Mescolare delicatamente mediante pipettaggio.
- Aspirare tutta la miscela di reazione di traslazione (500 μL) utilizzando una pipetta da 1.000 μL. Iniettare la miscela della reazione di traslazione nella coppa di dialisi nello stesso modo descritto al punto 5.4.5. Coprire la coppa di dialisi con un coperchio per evitare l'evaporazione.
- Incubare le reazioni a 15 °C per 24 ore.
- Mescolare bene la reazione nella coppa di dialisi mediante pipettaggio. Trasferire la sospensione grezza di proteoliosoma in un nuovo tubo.
NOTA: Si consiglia un tubo piatto da 1,5 ml per raccogliere il proteoliposoma dalla traduzione su piccola scala. Dopo la centrifugazione, i liposomi formano pellet compatto e facilmente visibile nell'angolo inferiore del tubo.
6. Purificazione dei proteoliposomi
- Centrifugare la provetta contenente la sospensione di proteoliosoma grezzo a 17.800 x g a 4 °C per 10 minuti.
- Rimuovere il surnatante. Sospendere il pellet proteoliosoma in PBS (piccola scala: 1 mL, grande scala: 10 ml) mediante pipettaggio.
- Ripetere la centrifugazione e il lavaggio dei proteoliposomi per altri due cerchi.
- Dopo il lavaggio, aggiungere una piccola quantità di PBS e risospendere bene il pellet di proteoliposomia mediante pipettaggio. Misurare il volume della sospensione utilizzando una micro pipetta. Aggiungere PBS per regolare il volume a 60 μL (piccola scala) o 500 μL (grande scala). Trasferire la sospensione in un nuovo tubo da 1,5 ml.
- Trasferire 10 μL di sospensione di proteoliposomia in una nuova provetta PCR per SDS-PAGE. Dividere il resto dei campioni in porzioni più piccole da utilizzare quando necessario. Congelare in azoto liquido e conservare a -80 °C.
7. Colorazione SDS-PAGE e CBB
- Aggiungere 70 μL di acqua e 40 μL di 3x tampone campione SDS-PAGE a 10 μL di sospensione di proteoliposomi.
ATTENZIONE: Non far bollire il campione SDS-PAGE, poiché le proteine di membrana si aggregano e difficilmente penetrano nel gel di acrilammide in elettroforesi. Inoltre, aggiungere abbastanza agente riducente al tampone campione SDS-PAGE (ad esempio, 2-mercaptoetanolo al 3% di concentrazione finale) per prevenire l'ossidazione. - Impostare un gel SDS-PAGE con gradiente del 5%–20% in una camera di elettroforesi. Carico 3 μL, 6 μL, 12 μL di campioni di proteoliposomi, 2 μL di marcatore di dimensione proteica e serie standard BSA.
- Elettroforesi a 52 mA, 400 V per 30 min.
- Macchiare il gel con colorante CBB per 1 ora. Decolorare in acqua calda e scansionare l'immagine del gel.
- Utilizzando il software NIH Image J (https://imagej.nih.gov/ij/), quantificare l'intensità della banda della proteina di membrana in ciascuna corsia. Stimare la quantità di proteine di membrana sintetizzate con la serie standard BSA.
Risultati
Utilizzando questo protocollo, i proteoliposomi parzialmente purificati possono essere ottenuti in breve tempo. I risultati rappresentativi sono mostrati nella Figura 2A. Venticinque GPCR di Classe A, B e C sono stati sintetizzati con successo utilizzando il metodo della dialisi a doppio strato (piccola scala) e parzialmente purificati mediante centrifugazione e lavaggio tampone. Sebbene la quantità di proteine sintetizzate vari a seconda del tipo di proteina, da 50 a 400 μg di proteine di membrana di solito possono essere sintetizzate per reazione quando vengono utilizzate coppe di dialisi di grandi dimensioni. Diversi milligrammi di proteine di membrana possono essere facilmente prodotti aumentando il numero di reazioni, grazie all'elevata scalabilità del sistema privo di cellule di grano. Un pre-test con una piccola coppa di dialisi è sufficiente per determinare l'efficacia di produzione della proteina bersaglio nel metodo di dialisi a doppio strato. In base alla produttività ottenuta, è possibile stimare la quantità della proteina bersaglio da produrre utilizzando grandi coppette di dialisi.
Questo protocollo è adatto per l'espressione di proteine di membrana, in particolare per quelle con eliche transmembrana multiple. Nella maggior parte dei casi, le proteine di membrana con tre o più eliche transmembrana sono facilmente incorporate nei proteoliposomi dopo la sintesi (Figura 2B), il che rende una buona produttività dei proteoliposomi. Le proteine a elica a singola transmembrana sono solitamente sintetizzate senza problemi; Tuttavia, difficilmente si integrano nei liposomi a causa della piccola regione idrofobica. Per quanto riguarda le proteine con due eliche transmembrana, il fatto che siano ancorate o meno ai liposomi dipende dal modo in cui le loro eliche transmembrana sono esposte.
I proteoliposomi sintetizzati vengono raccolti mediante semplice centrifugazione e parzialmente purificati con un tampone di lavaggio, che accorcia notevolmente il processo di purificazione delle proteine di membrana. Sebbene sia le membrane biologiche che le proteine di membrana siano state rimosse in precedenza dagli estratti di germe di grano, piccole quantità di proteine del grano sono talvolta co-precipitate legandosi ai liposomi o alle proteine di membrana sintetizzate (Figura 2A). Tali contaminanti proteici sono difficili da rimuovere con una semplice centrifugazione e lavaggio tampone. Quando è richiesta una proteina di membrana altamente purificata, è necessario solubilizzare i proteoliposomi parzialmente purificati con un tensioattivo e purificarli mediante cromatografia su colonna.

Figura 1: Schema di produzione di proteoliposomi privi di cellule. SP6, sequenza promotore SP6; E01, E01 sequenza di potenziamento della traduzione; Ampr, gene di resistenza all'ampicillina; DTT, ditiotreitolo. La micrografia elettronica mostra la marcatura immunogold del liposoma contenente lipidi biotinilati. Bar, 0,2 μm. Questa immagine al micrografo elettronico proviene dalla Figura 1D in Takeda et al., 201545. Fare clic qui per visualizzare una versione ingrandita di questa figura.
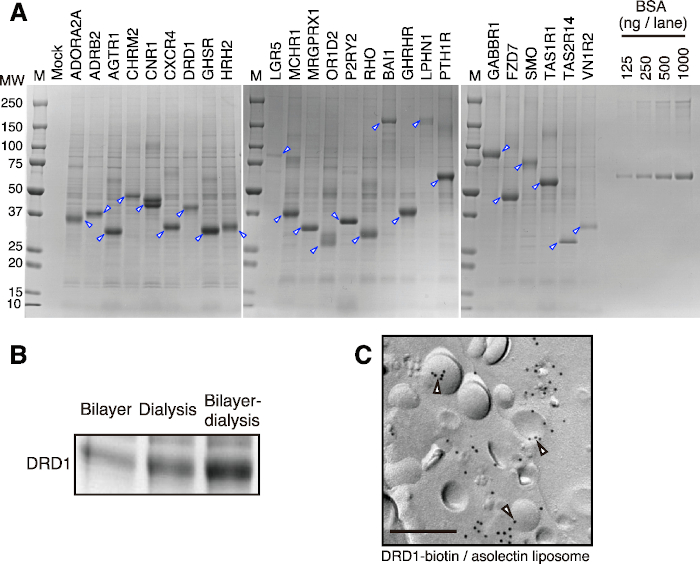
Figura 2: Risultati rappresentativi della produzione di proteoliposomi mediante metodo di dialisi a doppio strato. (A) Immagine SDS-PAGE di GPCR sintetizzati senza cellule. Venticinque GPCR selezionati sono stati sintetizzati con il metodo della dialisi a doppio strato. I proteoliposomi sono stati parzialmente purificati e applicati alla colorazione SDS-PAGE e CBB. Le punte delle frecce indicano i GPCR target. (B) Confronto delle produzioni di proteine di membrana tra diversi metodi di traduzione. La proteina del recettore D1 della dopamina (DRD1) è stata sintetizzata con ciascun metodo nello stesso rapporto di estratto di germe di grano, liposomi e mRNA, rispettivamente. Il proteoliosoma DRD1 è stato parzialmente purificato mediante centrifugazione e sottoposto a colorazione SDS-PAGE e CBB. (C) Marcatura immunogold del complesso DRD1-biotina/liposoma. DRD1 è stato biotinilato enzimaticamente dalla BirA biotina ligasi. Bar, 0,2 μm. Le punte di freccia vuote indicano DRD1-biotina sui liposomi. Questa figura è stata modificata dalla Figura 1 in Takeda et al., 201545. Fare clic qui per visualizzare una versione ingrandita di questa figura.
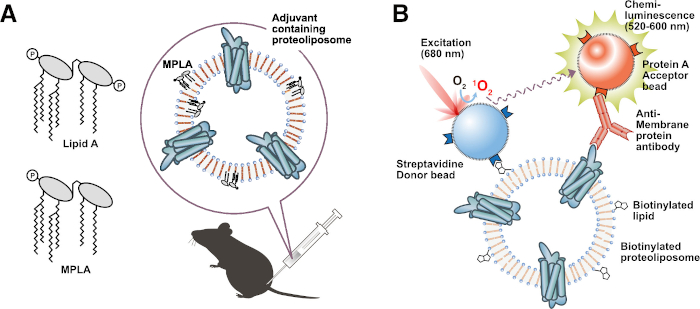
Figura 3: Applicazione dei proteoliposomi funzionali. (A) Immunizzazione del proteoliposomia adiuvante contenente lipidi. (B) Saggio di interazione basato su liposomi biotinilati (BiLIA). L'interazione tra la proteina di membrana e l'anticorpo della proteina anti-membrana è stata rilevata da AlphaScreen. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Discussione
Il protocollo presentato fornisce un metodo per produrre proteine di membrana ad un alto tasso di successo. Questo protocollo è semplice, altamente riproducibile e facile da scalare. Ha anche il potenziale per ridurre i tempi e i costi degli esperimenti che consumano una grande quantità di proteine di membrana. Il metodo di dialisi a doppio strato migliora la produttività di 4-10 volte rispetto al metodo a doppio strato o al metodo di dialisi (Figura 2B)45. In un caso estremo, la resa di un canale ionico e di un trasportatore è aumentata rispettivamente di 30 e 20 volte con il metodo di dialisi a doppio strato rispetto a quello con il metodo a doppio strato (dati non mostrati). L'alta produttività di questo protocollo è un vantaggio nella produzione di antigene per l'immunizzazione. I proteoliposomi sono spesso usati come antigeni immunizzanti per lo sviluppo di anticorpi proteici anti-membrana. Le proteine di membrana altamente concentrate e purificate incorporate nel proteoliposomia stimolano efficacemente la risposta immunitaria e inducono anticorpi41,47. Utilizzando questo metodo di dialisi a doppio strato, i proteoliposomi che trasportano diversi milligrammi di proteine di membrana a scopo di immunizzazione possono essere facilmente preparati in pochi giorni. Infatti, il nostro gruppo ha sintetizzato GPCR, canali ionici e claudine utilizzando questo protocollo e immunizzato topi con i prodotti per ottenere anticorpi monoclonali contro di loro31,41,45. Alcuni degli anticorpi monoclonali ottenuti sono stati verificati come anticorpi funzionali, come anticorpi ad alta affinità, anticorpi sensibili alla conformazione, anticorpi applicabili alla citometria a flusso e anticorpi inibitori, il che indica che questo protocollo è in grado di produrre proteine di membrana con conformazioni funzionalmente corrette.
Un altro vantaggio interessante di questo protocollo è quello di consentire la produzione di proteoliposomi a cui vengono assegnate funzioni specifiche utilizzando lipidi modificati, come lipidi biotinilati, lipidi fluorescenti o lipidi adiuvanti. I proteoliposomi preparati con funzioni specifiche sono utili e applicabili a una vasta gamma di esperimenti. Ad esempio, i proteoliposomi contenenti lipidi adiuvanti, come il lipide A48 o il monofosforil lipide A (MPLA)49, rendono convenienti antigeni immunizzanti, perché possono essere somministrati direttamente per immunizzare i topi senza emulsione. I lipidi adiuvanti stimolano efficacemente la risposta immunitaria negli animali ospiti, inducendo anticorpi contro le proteine di membrana bersaglio (Figura 3A). Infatti, abbiamo indotto con successo anticorpi applicabili alla citometria a flusso immunizzando topi con proteoliposa31 contenente MPLA. Inoltre, i proteoliposomi preparati da lipidi biotinilati sono sonde ideali per i saggi di screening. Abbiamo sviluppato un metodo di screening ad alto rendimento per selezionare anticorpi proteici anti-membrana utilizzando proteoliposomi biotinilati e AlphaScreen (metodo BiLIA) (Figura 3B)45. Sandwich ELISA è anche in grado di essere facilmente costruito utilizzando proteoliposomi biotinilati e piastre rivestite di streptavidina.
Infine, ci sono due importanti avvertenze che dovrebbero essere affrontate quando si utilizza questo metodo. In primo luogo, la formazione di legami disolfuro può essere insufficiente a causa delle alte concentrazioni di DTT nel tampone di traduzione, che possono influenzare la struttura di alcuni tipi di proteine di membrana15. Sebbene i legami disolfuro siano in grado di formarsi dopo che il riducente è stato rimosso durante il processo di purificazione, è possibile che si formino in tipi diversi piuttosto che in quelli naturali. L'altro è che le proteine di membrana non sono glicosilate. Gli enzimi necessari per la glicosilazione sono teoricamente assenti nel sistema privo di cellule perché durante il processo in cui viene prodotto l'estratto di germe di grano, le biomembrane, tra cui Golgi e ER, sono state rimosse. Poiché la mancanza di legami disolfuro e glicosilazione può causare diverse conformazioni, è necessario prestare un'attenta considerazione e valutazione al disegno sperimentale, in particolare quando le modifiche post-traduzionali sono critiche per l'espressione funzionale delle proteine secondo gli scopi sperimentali.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questa ricerca è stata supportata dal Platform Project for Supporting Drug Discovery and Life Science Research (Basis for Supporting Innovative Drug Discovery and Life Science Research (BINDS)) di AMED con il numero di sovvenzione JP20am0101077. Questo lavoro è stato anche parzialmente supportato dal JSPS KAKENHI Grant Number 20K05709.
Materiali
| Name | Company | Catalog Number | Comments |
| ×3 SDS-PAGE sample buffer | Containing 10% 2-mercaptoethanol | ||
| 5-20% gradient SDS-PAGE gel | ATTO | E-D520L | |
| 70% ethanol | Diluted ethanol by ultrapure water. | ||
| Agarose | Takara Bio | ||
| Ammonium acetate | Nakalai tesque | 02406-95 | As this reagent is deliquescent, dissolve all of it in water once opened and store it at -30°C. |
| Ampicillin Sodium | Nakalai tesque | 02739-74 | |
| Asolectin Liposome, lyophilized | CellFree Sciences | CFS-PLE-ASL | A vial contains 10 mg of lyophilized liposomes. |
| BSA standard | 1000 ng, 500 ng, 250 ng, 125 ng BSA / 10 µL ×1 SDS-PAGE sample buffer | ||
| CBB gel stain | |||
| cDNA clone of interest | Plasmid harboring cDNA clone or synthetic DNA fragment | ||
| Chloroform | Nakalai tesque | 08402-84 | |
| Cooled incubator | Temperature ranging from 0 to 40 °C or wider. | ||
| Creatine kinase | Roche Diagnostics | 04524977190 | |
| Dialysis cup (0.1 mL) | Thermo Fisher Scientific | 69570 | Slide-A-Lyzer MINI Dialysis Device, 10K MWCO, 0.1 mL |
| Dialysis cup (2 mL) | Thermo Fisher Scientific | 88404 | Slide-A-Lyzer MINI Dialysis Device, 10K MWCO, 2 mL |
| DNA ladder marker | Thermo Fisher Scientific | SM0311 | GeneRuler 1 kb DNA Ladder |
| DpnI | Thermo Fisher Scientific | FD1703 | FastDigest DpnI |
| E. coli strain JM109 | |||
| Electrophoresis chamber | ATTO | ||
| Ethanol (99.5%) | Nakalai tesque | 14713-95 | |
| Ethidium bromide | |||
| Evaporation flask, 100 mL | |||
| Gel imager | |||
| Gel scanner | We use document scanner and LED immuninator as a substitute. | ||
| LB broth | |||
| Lipids of interest | Avanti Polar Lipids | ||
| Micro centrifuge | TOMY | MX-307 | |
| NTP mix | CellFree Sciences | CFS-TSC-NTP | Mixture of ATP, GTP, CTP, UTP, at 25 mM each |
| Nuclease-free 25 mL tube | IWAKI | 362-025-MYP | |
| Nucrease-free plastic tubes | Watson bio labs | Do not autoclave. Use them separately from other experiments. | |
| Nucrease-free tips | Watson bio labs | Do not autoclave. Use them separately from other experiments. | |
| PBS buffer | |||
| PCR purification kit | MACHEREY-NAGEL | 740609 | NucleoSpin Gel and PCR Clean-up |
| pEU-E01-MCS vector | CellFree Sciences | CFS-11 | |
| Phenol/chloroform/isoamyl alcohol (25:24:1) | Nippon Gene | 311-90151 | |
| Plasmid prep Midi kit | MACHEREY-NAGEL | 740410 | NucleoBond Xtra Midi |
| Primer 1 | Thermo Fisher Scientific | Custom oligo synthesis | 5’-CCAAGATATCACTAGnnnnnnnnnnnnnnnnnnnnnnnn-3’ Gene specific primer, forward. Upper case shows overlap sequence to be added for seamless cloning. Lower case nnnn…. (20-30 bp) shows gene specific sequence. |
| Primer 2 | Thermo Fisher Scientific | Custom oligo synthesis | 5'-CCATGGGACGTCGACnnnnnnnnnnnnnnnnnnnnnnnn-3’ Gene specific primer, reverse. Upper case shows overlap sequence to be added for seamless cloning. Lower case nnnn…. (20-30 bp) shows gene specific sequence. |
| Primer 3 | Thermo Fisher Scientific | Custom oligo synthesis | 5'-GTCGACGTCCCATGGTTTTGTATAGAAT-3' Forward primer for vector linearization. Underline works as overlap in seamless cloning. |
| Primer 4 | Thermo Fisher Scientific | Custom oligo synthesis | 5'-CTAGTGATATCTTGGTGATGTAGATAGGTG-3' Reverse primer for vector linearization. Underline works as overlap in seamless cloning. |
| Primer 5 | Thermo Fisher Scientific | Custom oligo synthesis | 5’-CAGTAAGCCAGATGCTACAC-3’ Sequencing primer, forward |
| Primer 6 | Thermo Fisher Scientific | Custom oligo synthesis | 5’- CCTGCGCTGGGAAGATAAAC-3’ Sequencing primer, reverse |
| Protein size marker | Bio-Rad | 1610394 | Precision Plus Protein Standard |
| Rotary evaporator | |||
| seamless cloning enzyme mixture | New England BioLabs | E2611L | Gibson Assembly Master Mix Other seamless cloning reagents are also avairable. |
| SP6 RNA Polymerase & RNase Inhibitor | CellFree Sciences | CFS-TSC-ENZ | |
| Submarine Electrophoresis system | |||
| TAE buffer | |||
| Transcription Buffer LM | CellFree Sciences | CFS-TSC-5TB-LM | |
| Translation buffer | CellFree Sciences | CFS-SUB-SGC | SUB-AMIX SGC (×40) stock solution (S1, S2, S3, S4). Prepare ×1 translation buffer before use by mixing stock S1, S2, S3, S4 stock and ultrapure water. |
| Ultrapure water | We recommend to prepare ultrapure water by using ultrapure water production system every time you do experiment. Do not autoclave. We preparaed ultrapure water by using Milli-Q Reference and Elix10 system. Commercially available nuclease-free water (not DEPC-treated water) can be used as a substitute. Take care of contamination after open the bottle. | ||
| Ultrasonic homogenizer | Branson | SONIFIER model 450D-Advanced | Ultrasonic cleaner can be used as a substitute. |
| UV transilluminator | |||
| Vacuum desiccator | |||
| Wheat germ extract | CellFree Sciences | CFS-WGE-7240 | WEPRO7240 |
Riferimenti
- Santos, R., et al. A comprehensive map of molecular drug targets. Nature Reviews. Drug Discovery. 16 (1), 19-34 (2017).
- Gusach, A., et al. Beyond structure: emerging approaches to study GPCR dynamics. Current Opinion in Structural Biology. 63, 18-25 (2020).
- Congreve, M., de Graaf, C., Swain, N. A., Tate, C. G. Impact of GPCR Structures on Drug Discovery. Cell. 181 (1), 81-91 (2020).
- Wilkinson, T. C. I. Discovery of functional monoclonal antibodies targeting G-protein-coupled receptors and ion channels. Biochemical Society Transactions. 44 (3), 831-837 (2016).
- Hino, T., Iwata, S., Murata, T. Generation of functional antibodies for mammalian membrane protein crystallography. Current Opinion in Structural Biology. 23 (4), 563-568 (2013).
- Webb, D. R., Handel, T. M., Kretz-Rommel, A., Stevens, R. C. Opportunities for functional selectivity in GPCR antibodies. Biochemical Pharmacology. 85 (2), 147-152 (2013).
- Douthwaite, J. A., Finch, D. K., Mustelin, T., Wilkinson, T. C. I. Development of therapeutic antibodies to G-protein coupled receptors and ion channels: Opportunities, challenges and their therapeutic potential in respiratory diseases. Pharmacology and Therapeutics. 169, 113-123 (2016).
- Hashimoto, Y., Yagi, K., Kondoh, M. Current progress in a second-generation claudin binder, anti-claudin antibody, for clinical applications. Drug Discovery Today. 21 (10), 1711-1718 (2016).
- Hutchings, C. J., Colussi, P., Clark, T. G. Ion channels as therapeutic antibody targets. mAbs. 11 (2), 265-296 (2019).
- Errey, J. C., Fiez-Vandal, C. Production of membrane proteins in industry: The example of GPCRs. Protein Expression and Purification. 169, 105569 (2020).
- Pandey, A., Shin, K., Patterson, R. E., Liu, X. Q., Rainey, J. K. Current strategies for protein production and purification enabling membrane protein structural biology. Biochemistry and Cell Biology. 94 (6), 507-527 (2016).
- Wiseman, D. N., et al. Expression and purification of recombinant G protein-coupled receptors: a review. Protein Expression and Purification. 167, 105524 (2020).
- Jeffery, C. J. Expression, Solubilization, and Purification of Bacterial Membrane Proteins. Current Protocols in Protein Science. 83 (1), 1-15 (2016).
- Spirin, A. S., Baranov, V. I., Ryabova, L. A., Ovodov, S. Y., Alakhov, Y. B. A continuous cell-free translation system capable of producing polypeptides in high yield. Science. 242 (4882), 1162-1164 (1988).
- Takai, K., Sawasaki, T., Endo, Y. Practical cell-free protein synthesis system using purified wheat embryos. Nature Protocols. 5 (2), 227-238 (2010).
- Endo, Y., Sawasaki, T. Cell-free expression systems for eukaryotic protein production. Current Opinion in Biotechnology. 17 (4), 373-380 (2006).
- Shimizu, Y., Kuruma, Y., Ying, B. W., Umekage, S., Ueda, T. Cell-free translation systems for protein engineering. The FEBS Journal. 273 (18), 4133-4140 (2006).
- Klammt, C., et al. Cell-free expression as an emerging technique for the large scale production of integral membrane protein. The FEBS Journal. 273 (18), 4141-4153 (2006).
- Nozawa, A., et al. A cell-free translation and proteoliposome reconstitution system for functional analysis of plant solute transporters. Plant and Cell Physiology. 48 (12), 1815-1820 (2007).
- Nozawa, A., et al. Production and partial purification of membrane proteins using a liposome-supplemented wheat cell-free translation system. BMC Biotechnology. 11 (1), 35-45 (2011).
- Henrich, E., Hein, C., Dotsch, V., Bernhard, F. Membrane protein production in Escherichia coli cell-free lysates. FEBS Letters. 589 (15), 1713-1722 (2015).
- Henrich, E., Peetz, O., et al. Analyzing native membrane protein assembly in nanodiscs by combined non-covalent mass spectrometry and synthetic biology. eLife. 6, 243 (2017).
- Shelby, M. L., He, W., Dang, A. T., Kuhl, T. L., Coleman, M. A. cell-free co-translational approaches for producing mammalian receptors: expanding the cell-free expression toolbox using nanolipoproteins. Frontiers in Pharmacology. 10, 744 (2019).
- Jacobs, M. L., Boyd, M. A., Kamat, N. P. Diblock copolymers enhance folding of a mechanosensitive membrane protein during cell-free expression. Proceedings of the National Academy of Sciences of the United States of America. 116 (10), 4031-4036 (2019).
- Sackin, H., Nanazashvili, M., Makino, S. I. Direct injection of cell-free Kir1.1 protein into Xenopus oocytes replicates single-channel currents derived from Kir1.1 mRNA. Channels. 9 (4), 196-199 (2015).
- Zemella, A., Richter, T., Thoring, L., Kubick, S. A combined cell-free protein synthesis and fluorescence-based approach to investigate GPCR binding properties. Methods in Molecular Biology. 1947 (10), 57-77 (2019).
- Vaish, A., Guo, S., Murray, R. M., Grandsard, P. J., Chen, Q. On-chip membrane protein cell-free expression enables development of a direct binding assay: A curious case of potassium channel KcsA-Kv1.3. Analytical Biochemistry. 556, 70-77 (2018).
- Suzuki, Y., et al. Functional G-Protein-Coupled Receptor (GPCR) synthesis: the pharmacological analysis of Human Histamine H1 Receptor (HRH1) synthesized by a wheat germ cell-free protein synthesis system combined with asolectin glycerosomes. Frontiers in Pharmacology. 9, 38 (2018).
- Cortes, S., Barette, C., Beroud, R., De Waard, M., Schaack, B. Functional characterization of cell-free expressed Kv1.3 channel using a voltage-sensitive fluorescent dye. Protein Expression and Purification. 145, 94-99 (2018).
- Woznicka-Misaila, A., Juillan-Binard, C., Baud, D., Pebay-Peyroula, E., Ravaud, S. Cell-free production, purification and characterization of human mitochondrial ADP/ATP carriers. Protein Expression and Purification. 144, 46-54 (2018).
- Hashimoto, Y., et al. Engineered membrane protein antigens successfully induce antibodies against extracellular regions of claudin-5. Scientific Reports. 8 (1), 8383 (2018).
- Sawasaki, T., et al. A bilayer cell-free protein synthesis system for high-throughput screening of gene products. FEBS Letters. 514 (1), 102-105 (2002).
- Goshima, N., et al. Human protein factory for converting the transcriptome into an in vitro-expressed proteome. Nature Methods. 5 (12), 1011-1017 (2008).
- Nemoto, K., Takemori, N., Seki, M., Shinozaki, K., Sawasaki, T. Members of the plant CRK superfamily are capable of trans- and autophosphorylation of tyrosine residues. The Journal of Biological Chemistry. 290 (27), 16665-16677 (2015).
- Takeda, H., et al. Comparative analysis of human src-family kinase substrate specificity in vitro. Journal of Proteome Research. 9 (11), 5982-5993 (2010).
- Takahashi, H., et al. Establishment of a wheat cell-free synthesized protein array containing 250 human and mouse E3 ubiquitin ligases to identify novel interaction between E3 ligases and substrate proteins. PLoS One. 11 (6), 0156718 (2016).
- Nozawa, A., et al. Construction of a protein library of Arabidopsis transcription factors using a wheat cell-free protein production system and its application for DNA binding analysis. Bioscience, Biotechnology, and Biochemistry. 73 (7), 1661-1664 (2009).
- Goren, M. A., Nozawa, A., Makino, S. I., Wrobel, R. L., Fox, B. G. Cell-free translation of integral membrane proteins into unilamelar liposomes. Methods in Enzymology. 463, 647-673 (2009).
- Nozawa, A., et al. Production and partial purification of membrane proteins using a liposome-supplemented wheat cell-free translation system. BMC Biotechnology. 11 (1), 35 (2011).
- Renauld, S., et al. Functional reconstitution of cell-free synthesized purified Kv channels. Biochimica Et Biophysica Acta. Biomembranes. 1859 (12), 2373-2380 (2017).
- Liu, S., et al. Efficiency and Safety of CRAC Inhibitors in Human Rheumatoid Arthritis Xenograft Models. Journal of Immunology. 199 (5), 1584-1595 (2017).
- Jarecki, B. W., Makino, S. I., Beebe, E. T., Fox, B. G., Chanda, B. Function of shaker potassium channels produced by cell-free translation upon injection into xenopus oocytes. Scientific Reports. 3, 1-7 (2013).
- David, G., et al. Phosphorylation and alternative translation on wheat germ cell-free protein synthesis of the DHBV large envelope protein. Frontiers in Molecular Biosciences. 6, 138 (2019).
- Jirasko, V., et al. Proton-detected solid-state NMR of the cell-free synthesized α-helical transmembrane protein NS4B from hepatitis C virus. Chembiochem: A European Journal of Chemical Biology. 21 (10), 1453-1460 (2020).
- Takeda, H., et al. Production of monoclonal antibodies against GPCR using cell-free synthesized GPCR antigen and biotinylated liposome-based interaction assay. Scientific Reports. 5, 11333 (2015).
- Zhou, W., Takeda, H. Production of immunizing antigen proteoliposome using cell-free protein synthesis system. Methods in Molecular Biology. 1868, 49-67 (2018).
- Hutchings, C. J., Koglin, M., Marshall, F. H. Therapeutic antibodies directed at G protein-coupled receptors. mAbs. 2 (6), 594-606 (2010).
- Raetz, C. R. H., et al. Discovery of new biosynthetic pathways: the lipid A story. Journal of Lipid Research. 50, 103-108 (2009).
- Baldridge, J. R., Crane, R. T. Monophosphoryl lipid A (MPL) formulations for the next generation of vaccines. Methods. 19 (1), 103-107 (1999).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon