Method Article
Creazione di sistemi di dimerizzazione delle proteine altamente specifici indotta chimicamente mediante la selezione stepwise di phage di una biblioteca combinatoria di anticorpi a dominio singolo
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
La creazione di sistemi di dimerizzazione delle proteine indotta chimicamente con l'affinità e la specificità desiderate per ogni piccolo ligando molecolare avrebbe molte applicazioni biologiche di rilevamento e attuazione. Qui, descriviamo un metodo efficiente e generalizzabile per l'ingegneria de novo di sistemi di dimerizzazione chimicamente indotti attraverso la selezione graduale di una libreria di anticorpi combinatori combinatori e di foragi.
Abstract
Gli eventi di dimerizzazione delle proteine che si verificano solo in presenza di un ligando di piccole molecole consentono lo sviluppo di biosensori di piccole molecole per la dissezione e la manipolazione dei percorsi biologici. Attualmente, ne esistono solo un numero limitato di sistemi di dimerizzazione chimicamente indotti (CID) e l'ingegneria di nuovi sistemi con la sensibilità e la selettività desiderate per specifici ligandi di piccole molecole rimane una sfida nel campo dell'ingegneria delle proteine. Qui descriviamo un metodo di screening ad alta produttività, combinatorial binders-enabled selezione di CID (COMBINES-CID), per l'ingegneria de novo di sistemi CID applicabili a una grande varietà di ligandi. Questo metodo utilizza la selezione in due passaggi di una libreria nanobody combinatorial visualizzata da fago per ottenere 1) "leganti di ancoraggio" che prima si legano a un ligando di interesse e poi 2) "leganti di dimerizzazione" che si legano solo ai complessi binder-ligando di ancoraggio. Per selezionare i leganti di ancoraggio, una libreria combinatoria di oltre 109 nanocorpi randomizzati di regioni (CDR) viene sottoposta a screening con un legamento biotinylato e gli colpi vengono convalidati con il legamento senza etichetta tramite interferometria biostrato (BLI). Per ottenere leganti di dimerizzazione, la libreria nanobody viene sottoposta a screening con complessi di legatrice-ligando di ancoraggio come obiettivi per lo screening positivo e i leganti di ancoraggio non associati per lo screening negativo. COMBINES-CID è ampiamente applicabile per selezionare leganti CID con altri immunoglobulina, non immunoglobulina, o scaffold progettati computazionalmente per creare biosensori per il rilevamento in vitro e in vivo di farmaci, metaboliti, molecole di segnalazione, ecc.
Introduzione
I sistemi CID, in cui due proteine si dimerdano solo in presenza di un ligando di piccole molecole (Figura 1), offrono strumenti versatili per sezionare e manipolare metaboliche, segnalazioni e altre vie biologiche1. Hanno dimostrato il potenziale nell'attuazione biologica, come l'attivazione delle cellule T controllate da farmaci2 e l'apoptosi3,4, per migliorare la sicurezza e l'efficacia della terapia con cellule T adottiva. Inoltre, forniscono una nuova metodologia per il rilevamento in vivo o in vitro di bersagli di piccole molecole. Ad esempio, le proteine CID possono essere geneticamente fuse con sistemi di segnalazione di fluorescenza (ad esempio, il trasferimento di energia di risonanza di fluorescenza (FRET)5 e proteine fluorescenti permutate in modo circolare)6 per misurazioni in vivo in tempo reale, o servono come reagenti di affinità per gli immunosbenatori simili a assaggi di assaggio (ELISALISA).
Nonostante le loro ampie applicazioni, la creazione di nuovi sistemi CID che possono essere controllati da un determinato ligando di piccole molecole ha grandi sfide. Metodi di ingegneria dei leganti proteici stabiliti, tra cui l'immunizzazione animale7, la selezione in vitro8,9e il disegno delle proteine computazionali10 possono generare proteine leganti che funzionano tramite interazioni binarie proteina-ligando. Tuttavia, questi metodi hanno difficoltà a creare un complesso CID ternario indotto dal ligando. Alcuni metodi creano CID collegando chimicamente due ligando che si legano in modo indipendente alle stesse proteine11,12, 13,14,15,16 o si basano sulla selezione di proteine leganti come anticorpi che colpiscono piccoli complessi molecolari-proteine preesistenti17,18, e quindi hanno una scelta limitata di ligandi.
Recentemente abbiamo sviluppato un metodo combinatorial binders-enabled di CID (COMBINES-CID) per l'ingegneria de novo dei sistemi CID19. Questo metodo può ottenere l'elevata specificità della dimerizzazione indotta dal ligando (ad esempio, una costante di dissociazione del legante di ancoraggio, KD (senza ligando)/KD (con ligando) > 1.000). La specificità della dimerizzazione si ottiene utilizzando leganti di ancoraggio con siti di legame flessibili che possono introdurre cambiamenti conformazionali al momento della legatura del ligando, fornendo una base per la selezione di leganti conformazionali selettivi che riconoscono solo leganti legati al ligando. Abbiamo dimostrato una prova di principio creando eterodimeri indotta da cannabidiolo (CBD) di nanocorpi, un frammento di anticorpi funzionale da 12-15 kDa da camelid che comprende un ponteggio universale e tre anelli CDR flessibili (Figura 2)20, che possono formare una tasca legante adattabile con epitopi di piccole molecole21,22. In particolare, la selezione invitro di una biblioteca proteica combinatoria dovrebbe essere conveniente e generalizzabile per l'ingegneria del CID, poiché la stessa libreria di alta qualità può essere applicata a diversi ligandi.
In questo protocollo e video, ci concentriamo sulla descrizione della selezione in vitro in due fasi e della convalida dell'ancora (Figura 3A) e dei leganti di dimerizzazione (Figura 3B) esaminando la biblioteca combinatoria di nanobody con una diversità superiore a 109 utilizzando il CBD come obiettivo, ma il protocollo dovrebbe essere applicabile ad altre librerie proteiche o a bersagli di piccole molecole. Lo screening dei leganti CID richiede solitamente 6-10 settimane (Figura 4).
Protocollo
1. Costruzione della biblioteca
- Utilizzare una libreria di anticorpi combinatori sintetici a dominio singolo con una varietà di 1,23–7,14 x 109, come descritto in precedenza19. Anche se questo protocollo non include la costruzione di librerie, può essere applicato ad altre librerie di leganti combinatori.
2. Biotinylazione dell'obiettivo di ligando o del ligando
- Biotinylate il ligando selezionato, ad esempio, CBD e tetraidrocannabinolo (THC)19, attraverso varie strategie di sintesi chimica, a seconda dei siti di biotinylazione adatti di un bersaglio.
3. Screening del legante di ancoraggio
- Inizio della selezione
- Inizia ogni round di selezione inoculando una singola colonia di cellule TG1, appena coltivata in 6 mL di 2YT a 37 gradi centigradi e 250 giri al minuto (rpm) a un assorbimento di 600 nm (OD600)di 0,5 dollari. Incubare le cellule sul ghiaccio per l'uso al punto 3.5.1.
- Selezione negativa con perline di streptavidina legate alla biotina
- Preparare le "perle di selezione negativa" lavando 300 -L di perline magnetiche rivestite di streptavidin utilizzando un rack di separazione magnetica, 3x con 0,05% di salina con tampone Tween (PBST, 1 x PBS con 0,05% vol/Vol Tween 20%) e 2x con 1 x PBS.
- Risospendere le perline con 1 mL dell'1% di caseina in 1 x PBS (pH - 7,4) e saturare le perline aggiungendo 5 volte la capacità di legame riportata utilizzando biotina. Incubare a temperatura ambiente (RT) su un rotatore per 1 h.
- Lavare le perline 5x utilizzando 0,05% PBST e 3x utilizzando 1 x PBS, per un totale di otto lavaggi.
- Aggiungete 1013 particelle di fagi nell'1% di caseina/1% di BSA in 1 x PBS (pH - 7,4) e incubate a RT su un rotatore per 1 h.
- Dopo l'incubazione, raccogliere il super-an viene utilizzato nel passaggio 3.3.6.
- Selezione positiva con perline di streptavidina leganti biotinylate
- Preparare le "perline di selezione positiva" utilizzando 1/2 il volume delle perline utilizzate per le "perline di selezione negativa" seguendo i passaggi 3.2.1.
- Sospendere nuovamente le perline con 1 mL 1% caseina in 1 PBS, pH 7.4 e saturare le perline aggiungendo 5 volte la capacità di legame completa calcolata in base al manuale utilizzando il legamento biotinyato di scelta. Incubare a RT su un rotatore per 1 h.
- Lavare le perline 5x utilizzando 0,05% PBST e 3x utilizzando 1 x PBS, per un totale di otto lavaggi.
- Bloccare le perline con 1 mL dell'1% di caseina/1% BSA in 1 x PBS (pH - 7,4) e incubare a RT su un rotatore per 1 h per evitare il legame non specifico tra i fagi e le perline magnetiche rivestite di streptavidin.
- Lavare le perle magnetiche rivestite di streptavidin 3x utilizzando 0,05% PBST e una volta utilizzando 1 x PBS, per un totale di quattro lavaggi.
- Risospendere le perle magnetiche rivestite di streptavidin utilizzando i fagi non legati presi dal passo 3.2.5 e incubare a RT su un rotatore per 1 h.
- Estrarre il supernatante senza disturbare le perline magnetiche. Salvare i fagi non associati come input, da utilizzare nel passaggio 3.5.1.Save the unbound phages as input, to be used in step 3.5.1.
- Lavare le perline 10x utilizzando 0,05% PBST e 5x utilizzando 1 x PBS. Tra ogni tre fumi, trasferiscili in un nuovo tubo per evitare fagi non specificamente legati alle pareti del tubo.
- Elution of phage-displayed nanobodies
- I fagi legati in modo competitivo sommando 450 -L del ligando non biotinylato,utilizzando una concentrazione nella gamma micromolare (ad es., 10-50 M) e incubando a RT su un rotatore per 30 min. La concentrazione di ligando selezionata per l'elusione competitiva dei fagi legati dipende dal KD desiderato del "legante di ancoraggio". Le concentrazioni di ligando possono essere relativamente elevate nei turni di selezione iniziali e quindi diminuite nei turni successivi.
- Raccogliere supernatali e salvare i fagi eluiti come output, da utilizzare nel passaggio 3.5.2.
- Titrazione di ingresso/uscita e infezione
- Per la titolazione di input, preparare 10x diluizioni seriali in 1 x PBS fino a 109-fold con il fago di ingresso dal passaggio 3.3.7. Utilizzare le diluizioni seriali da 107–109 per fare infezioni trasferendo un fagiolo di ingresso di 10 l da ciascuna diluizione a 70 celle TG1 (OD600 di 0,5 usd). Incubare a 37s per 45 min, placcare le cellule TG1 infette su tre piatti da 2YT-agar da 90 mm contenenti 100 anttadina e il 2% (wt/vol) di glucosio, e incubare la notte a 37 . Dalle piastre notturne, l'input di fago può essere calcolato come segue:

- Per l'infezione e la titolazione in uscita, trasferire i fagi eluiti dal passo 3.4.2 a 3 mL di cellule TG1 (OD600 di 0,5 dollari). Incubare in un bagno d'acqua a 37 gradi centigradi per 45 min. Quindi preparate le diluizioni seriali 10x in 2YT fino a 103volte, placcate ogni diluizione su piatti da 90 mm 2YT-agar e incubate la notte a 37 gradi centigradi. Dalle piastre notturne, l'uscita del fago può essere calcolata come segue:

- Dividere le restanti cellule TG1 infette su tre piastre da 2YT-agar da 150 mm contenenti 100 ampicillina e il 2% (wt/vol) di glucosio. Incubare le piastre durante la notte a 37 .
- Per la titolazione di input, preparare 10x diluizioni seriali in 1 x PBS fino a 109-fold con il fago di ingresso dal passaggio 3.3.7. Utilizzare le diluizioni seriali da 107–109 per fare infezioni trasferendo un fagiolo di ingresso di 10 l da ciascuna diluizione a 70 celle TG1 (OD600 di 0,5 usd). Incubare a 37s per 45 min, placcare le cellule TG1 infette su tre piatti da 2YT-agar da 90 mm contenenti 100 anttadina e il 2% (wt/vol) di glucosio, e incubare la notte a 37 . Dalle piastre notturne, l'input di fago può essere calcolato come segue:
- Amplificazione e recupero della libreria per ulteriori cicli di selezione
- Aggiungere 3 mL di 2YT per piastra, raschiare con un raschietto sterile e raccogliere tutte le cellule in un tubo conico da 50 mL. Mescolare le cellule raccolte con glicerolo sterile (20% wt/vol concentrazione finale). Misurare l'OD600 della miscela e fare 3-5 aliquote. Conservare a -80 gradi centigradi per lo stoccaggio a lungo termine.
- Per il salvataggio dei fagi, diluire la miscela batterica TG1 contenente faghe con un composto da fagomici utilizzando 25 mL di supporti 2YT integrati con il 2% di glucosio e 100 ampillina a un OD600 di 0,1. Cellule di coltura a 37 e 250 rpm a un OD600 di 0,5 USD.
- Superinettare le cellule aggiungendo il fagiolo di aiuto CM13 a 5 x 109 pfu/mL e incubare a 37 e 250 rpm per 45 min. Il fago di aiuto CM13 fornisce le proteine del mantello di fago necessarie per l'assemblaggio di particelle di fagi complete.
- Centrifugare la coltura a 8.000 x g per 10 min per rimuovere il glucosio. Risospendere le cellule utilizzando un supporto da 50 mL di 2YT integrato con 100 ampicillina g/mL e 50 g/mL di kanamicina e incubare a 25 e 250 rpm durante la notte.
- Centrifugare le cellule della coltura notturna a 9.000 x g, 4 gradi centigradi per 30 min. Trasferire supernatali in un nuovo tubo e fagi precipitati nel supernatante utilizzando 1/5 volume peg/ NaCl soluzione (20% wt / vol polietilene glicole-6.000 e 2.5 M NaCl). Mescolare delicatamente e mettere sul ghiaccio per 1 h.
- Raccogliere le particelle di fago mediante centrifugazione utilizzando 12.000 g a 4 gradi centigradi per 30 min. Risospendere i pellet utilizzando 1 mL di 1 x PBS e trasferire la sospensione in un tubo di microcentrifuga. Centrifugare il tubo a 20.000 x g e 4 gradi centigradi per 10 min per rimuovere i batteri residui.
- Trasferire il supernatante in un nuovo tubo di microcentrifuga senza disturbare il pellet batterico. Utilizzare una diluizione 1:100 per misurare l'assorbimento a 269 nm e 320 nm. Il numero totale di fagi può essere calcolato utilizzando la seguente formula23:

- Conservare la biblioteca di fagi a 4 gradi centigradi per uso a breve termine o con il 25% di glicerolo a -80 gradi centigradi per lo stoccaggio a lungo termine.
- Ripetere i round di selezione (passaggi da 3,1 a 3,6) per 3-6 turni o fino a quando non viene osservato l'arricchimento desiderato (fare riferimento alla sezione Risultati). Piastra e scegliere singoli cloni (sezione 4) al fine di caratterizzare la loro affinità e specificità al ligando (sezioni 5-7).
4. Isolamento di un singolo clone
- Per isolare singoli cloni da una sottolibreria arricchita, preparare le diluizioni seriali 10 x delle cellule TG1 infettate da fagi (passaggio 3.5.2). Piatto diluizioni seriali su 90 mm 2YT-agar-agar piatti contenenti 100 g/mL ampicillina e 2% (wt/vol) glucosio e incubazione a 37 gradi durante la notte.
- Dalle piastre notturne, scegliete le colonie singole in 250 gradi di supporto 2YT integrati con 100 anflussi per pozzo in sterili piastre di pozzi profondi e crescono a 37 gradi durante la notte.
- Dalle colture notturne, inoculari 10 gradi l in 500 -L di nuovi supporti 2YT integrati con 100 ampicillina di g/mL.
- Far crescere le cellule fino a un OD600 di 0,5 dollari, aggiungere il fago di supporto CM13 a 5 x 109 pfu/mL e incubare a 37 e 250 rpm per 45 min.
- Aggiungete 500 l di supporti 2YT integrati con 100 ampicillin a 10g/mL e 50 g/mL di kanamycin. Incubare a 25 e 250 rpm durante la notte.
- Centrifugare le piastre del pozzo profondo dalle colture durante la notte a 3.000 x g per 10 min. Raccogliere il supernatante contenente le particelle di fago senza disturbare il pellet cellulare.
- Le particelle di fago possono essere utilizzate per ELISA per determinare la specificità dei cloni selezionati al ligando. La biotina o un omologo strutturale dell'obiettivo possono essere utilizzati come controllo negativo.
5. Convalida del legante di ancoraggio da parte di ELISA
- Cappotto 96 pozze ELISA utilizzando 100 g l di 5 g/mL streptavidin nel tampone di rivestimento (100 mM di carbonato di tampone, pH - 8,6) a 4 gradi durante la notte.
- Lavare le piastre ELISA 3x utilizzando 0,05% PBST e aggiungere 100 -L di 1 bersaglio biotinylato di 1 M ai pozzedi bersaglio. Aggiungete 100 l di biotina o omologio bersaglio ai pozzetti di controllo. Incubare a RT per 1 h.
- Piastre lacerate 5x utilizzando 0,05% PBST e bloccano l'attacco non specifico aggiungendo 300 - L dell'1% di caseina in 1 x PBS. Incubare a RT per 1 h.
- Lavare le piastre ELISA 3x utilizzando 0,05%-PBST e aggiungere il supernatante di fago purificato. Incubare per 1 h a RT.
- Lavare le piastre ELISA 10x utilizzando 0,05% PBST e aggiungere 100 -L rafano perossidasi (HRP)-M13 anticorpo proteico principale del mantello (1:10.000 diluizione con 1 x PBS con 1% di caseina). Incubare a RT per 1 h.
- Lavare le piastre ELISA 3 volte utilizzando 0,05% PBST e aggiungere il substrato TMB (Tetramethylbenzidinine) di 100.L. Incubare per 10 min o fino a quando non si osserva un cambiamento di colore visibile. Fermare la reazione aggiungendo 100 luna di 1 M HCl. Leggere la piastra a 450 nm su uno spettrofotometro.
- Per l'espressione e la purificazione delle proteine, scegliere i cloni che mostrano un'elevata affinità e specificità per il target (vedere Discussione).
6. Espressione proteica, purificazione e biotinylazione
- Come riportato in precedenza19, i cloni secondari selezionavano i cloni della sezione 5 ed esprimono come nanocorpi con etichetta Avi e His.
- Esprimere i nanocorpi selezionati nel periplasma delle cellule E. coli WK6 (tipicamente nella coltura 1 L), rilasciare per shock osmotico e purificare utilizzando una colonna nichel-NTA (vedi Tabella dei materiali).
- Scambio di buffer con una colonna di salanding (1 x PBS con 5% glicerolo; vedere Tabella dei materiali).
- Nanocorpi biotinylate utilizzando un kit commerciale (vedi Tabella dei materiali)per un ulteriore utilizzo.
7. Caratterizzazione del legante di ancoraggio di BLI
- Analizzare l'affinità di legame e la cinetica di leganti di ancoraggio selezionati immobilizzando 200 leganti di ancoraggio biotinylati nM su biosensori streptavidina (vedi Tabella dei materiali) con buffer di analisi di legame (1 x PBS (pH - 7,4), 0.05% Tween 20, 0.2% BSA, 3% metanolo).
- Calcolare le costanti di dissociazione (KD) delle interazioni ancora binder-ligando mediante analisi dello stato costante utilizzando il software di analisi dei dati (vedere Tabella dei materiali). I valori KD ottenuti variano in genere da micromolare a singola e a due cifre.
8. Screening del legante di dimerizzazione
NOTA: Lo screening biopanning di "leganti di dimerizzazione" è simile a quello dei leganti di ancoraggio, ad eccezione di due passaggi critici: 1) I leganti di dimerazione vengono selezionati utilizzando un legante di ancoraggio biotinylato selezionato e il complesso di legatrice-ligando di ancoraggio per e le selezioni positive, rispettivamente. 2) Durante la fase di eluizione, 100 mM triethylamine viene utilizzato per eluire i fagi selezionati positivamente che erano legati solo al legante di ancoraggio --ligand omodello. La soluzione di trimethylamine da 100 mM (pH - 11,5) viene utilizzata per elutizzare cloni positivi interrompendo le interazioni proteiche.
- Inizio della selezione
- Inizia ogni round di selezione inoculando una singola colonia di cellule TG1, appena coltivata su un supporto minimo, in 6 mL 2YT a 37 e 250 rpm a un OD600 di 0,5 dollari. Incubano cellule sul ghiaccio.
- Rimozione di nanocorpi selezionati negativamente
- Preparare il "tubo di sottrazione" utilizzando 400 l una di perline magnetiche rivestite di streptavidin e seguire il passaggio 3.2. Tuttavia, invece di saturare con la biotina, aggiungere 5 volte la capacità di legame completa calcolata utilizzando il legante di ancoraggio biotinylato selezionato e salvare i fagi non associati da utilizzare nel passaggio 8.3.3.
- Selezione di nanocorpi selezionati positivamente
- Preparare il "tubo di cattura" utilizzando 1/2 il volume di perline magnetiche rivestite di streptavidin utilizzate per il "tubo di sottrazione" e seguendo i passaggi da 3.3.2 a 3.3.3. Tuttavia, invece di saturare con il ligando biotinylato, aggiungere cinque volte la capacità di legame completa calcolata utilizzando il legante di ancoraggio biotinylatoselezionato .
- Per formare il complesso di legatrice-ligando di ancoraggio per la selezione del legante di dimerizzazione positiva, aggiungere una concentrazione abbastanza alta di ligando non biotinylato. Questo permetterà alla maggior parte dei leganti di ancoraggio legati allo streptavidino di formare il complesso legato al ligando.
- Seguire i passaggi da 3.3.3 a 3.3.8, utilizzando i fagi non legati tratti dal "tubo di sottrazione".
- Elutizzazione di nanocorpi selezionati positivamente
- Elutare i fagi legati al complesso legante-ligando di ancoraggio aggiungendo 450 luna di 100 mM di triethylamine, e incubando a RT su un rotatore per 10 min.
- Raccogliere i fagi concorrenziali e seguire i passaggi da 3.4.1 a 3.4.2.
- Ulteriori cicli di selezione del raccoglitore di dimerizzazione
- Seguire i passaggi 3.5 e 3.6 per amplificare e recuperare la libreria per eseguire ulteriori cicli di selezione. Ripetere i round di selezione per 3-6 turni o fino a quando non viene osservato l'arricchimento desiderato. Piastra e scegliere singoli cloni (fare riferimento alla sezione 4) al fine di caratterizzare la loro affinità e specificità per il bersaglio.
9. Caratterizzazione del legante di dissorizzazione di ELISA
- Seguire i passaggi nella sezione 4 per isolare i singoli cloni per la caratterizzazione tramite ELISA.
- Per verificare l'affinità dei candidati leganti di dimerizzazione al complesso anchor binder-ligand, rivestire la piastra bersaglio ELISA utilizzando 100 nL di 100 nM legante di ancoraggio biotinylato. Dopo l'incubazione per 1 h, aggiungere 1 MM del bersaglio legante per formare il complesso di legante-ligando di ancoraggio.
- La piastra di controllo deve essere rivestita utilizzando il legante di ancoraggio biotinylato da solo per schermare cloni che possono anche legarsi al legante di ancoraggio libero. Aggiungere 100 luna di 100 nM legante di ancoraggio biotinylato e incubare a RT per 1 h.
- Seguire le sezioni 5.3–5.7.
10. Caratterizzazione del legante di dimerizzazione da parte di BLI
- L'affinità di legame e la cinetica dei leganti di dimerizzazione per il legante di ancoraggio-complesso ligando possono essere analizzati immobilizzando leganti di dimerizzazione biotinyati su biosensori di streptavidina (SA) con il buffer di analisi di legame e quindi analizzati con 1 legante di ancoraggio m pre-equilibrato con diluzioni del ligando. Il KD, kon, e koff delle interazioni può essere calcolato utilizzando il nostro metodo segnalato19.
Risultati
Descriviamo la selezione in vitro in due fasi e la convalida dei leganti di ancoraggio e dimerizzazione esaminando la biblioteca combinatoria nanobody con una diversità superiore a 109 utilizzando CBD come obiettivo. È importante valutare l'arricchimento del fago biopanning durante i successivi cicli di selezione sia per i leganti di ancoraggio che per quelli di dimerizzazione. I risultati tipici dell'arricchimento dopo 4-6 cicli di selezione, come illustrato nella Figura 5, sono una buona indicazione che esiste un rapporto elevato di potenziali riscontri nelle sottolibrerie, pertanto potrebbero non essere necessari ulteriori cicli di selezione.
ELISA single-clone è adatto per l'analisi dell'affinità di legame relativa e selettività dei leganti di ancoraggio e dimerizzazione. Figura 6A è un risultato di selezione del legante di ancoraggio rappresentativo dopo sei cicli di biopanning. I cloni che mostrano alta (ad esempio, #87) o bassa (ad esempio, #27) la selettività del ligando può essere confrontata. I cloni ad alta selettività devono essere scelti come candidati del legante di ancoraggio. Allo stesso modo, la figura 6B mostra i risultati della selezione del legante di dimerazione dopo quattro cicli di biopanning. In genere abbiamo osservato cloni che formavano un eterodimero con il legante di ancoraggio immobilizzato solo con il ligando (ad esempio, #49) o senza (ad esempio, #80). Il primo, che mostra la specificità della dimerizzazione, dovrebbe essere selezionato per un'ulteriore convalida.
Legante di ancoraggio ELISA si basa sull'uso del bersaglio biotinylato. Pertanto, è necessario utilizzare BLI per confermare ulteriormente l'associazione all'obiettivo non etichettato. BLI permette anche la caratterizzazione della cinetica legante. I risultati BLI dei leganti di ancoraggio e dimerizzazione sono riportati rispettivamente nella figura 7A e 7B. I pannelli di sinistra mostrano l'attacco dipendente dalla concentrazione, suggerendo che sono adatti per la costruzione di un sistema CID. I pannelli di destra mostrano i controlli negativi. Il KD calcolato delle interazioni di ancoratura e dimerizzazione del legante in presenza del ligando variava tipicamente dal nanomolare a due cifre al micromolare a due cifre. Essi possono variare a seconda del ligando e la biblioteca combinatoria di scelta.
La cromatografia ad esclusione di dimensioni analitiche (SEC) è stata eseguita per confermare la formazione di eterodimeri tra l'ancora e i leganti di dimerizzazione. Un picco di dimerizzazione è stato osservato quando i leganti di ancoraggio e dimerizzazione e CBD sono stati mescolati (Figura 8A, linea rossa). Al contrario, non è stato rilevato alcun picco di dimerizzazione in assenza di CBD (Figura 8A, linea blu) o quando ogni legante è stato caricato nella colonna da solo (Figura 8B). Il crosslinking chimico è stato utilizzato per stabilizzare i complessi CID e i nanocorpi interconnessi hanno dimensioni leggermente aumentate corrispondenti ai picchi eluiti in precedenza.
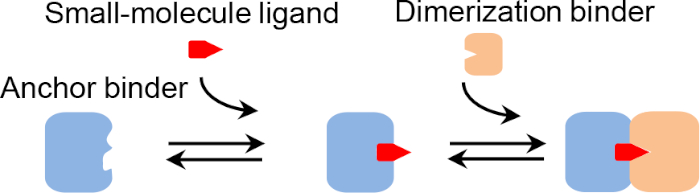
Figura 1: Meccanismo di dimerizzazione delle proteine indotta chimicamente. Fare clic qui per visualizzare una versione più grande di questa figura.
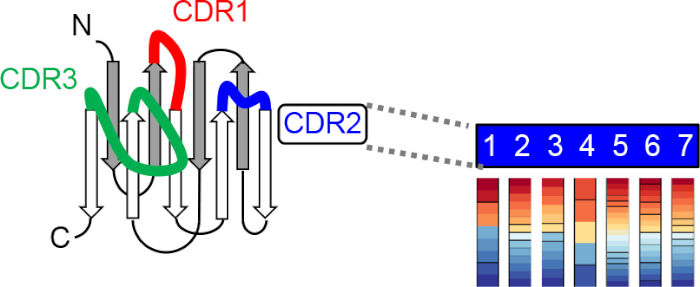
Figura 2: Schematico della generazione di una libreria combinatoria nanobody sintetica. La biblioteca è costruita utilizzando uno scaffold nanobody universale e incorporando distribuzioni progettate di aminoacidi in ogni posizione di randomizzazione in tre regioni che determinano la complementarità (CDR) da una tecnologia Trinucleotide Mutagenesis (TRIM) 24. Fare clic qui per visualizzare una versione più grande di questa figura.
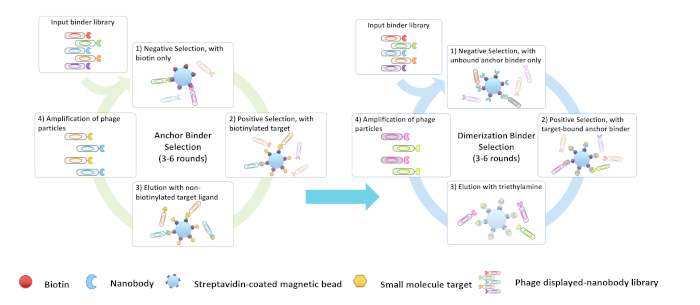
Figura 3: Diagramma di flusso dello screening dell'ancora e della (B) leghere di dimerizzazione (A). Fare clic qui per visualizzare una versione più grande di questa figura.
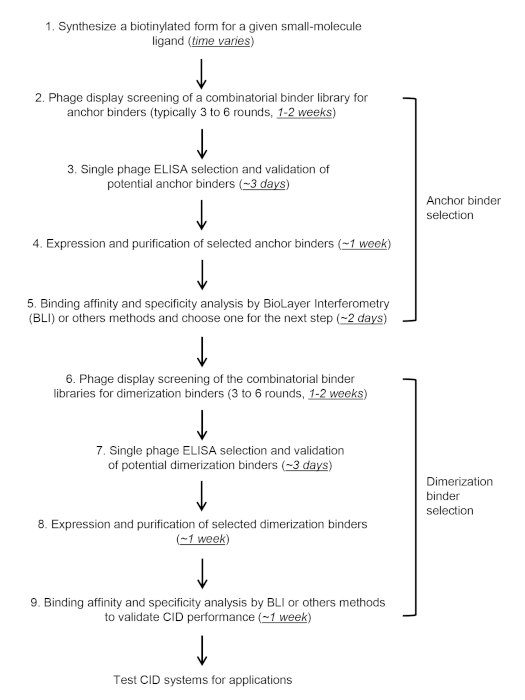
Figura 4: Sequenza temporale di COMBINES-CID. Fare clic qui per visualizzare una versione più grande di questa figura.
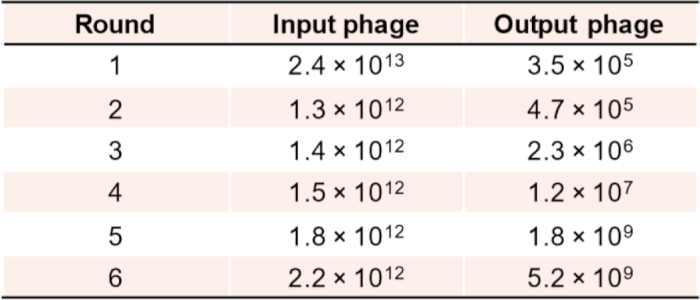
Figura 5: Arricchimento dei titoli di fago seguendo ogni ciclo di biopanning per la selezione del legante di ancoraggio. Fare clic qui per visualizzare una versione più grande di questa figura.
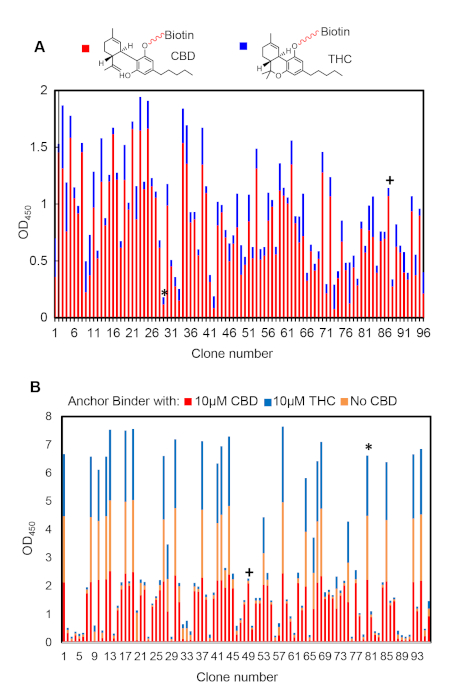
Figura 6: Risultati ELISA rappresentativi che mostrano cloni positivi e negativi. (A) Raccoglitore di ancoraggio ELISA deriva da 96 cloni scelti casualmente dopo sei turni di selezione. (B) Raccoglitore di dimerazione ELISA risultati di 96 cloni scelti casualmente dopo quattro turni di selezione. Fare clic qui per visualizzare una versione più grande di questa figura.
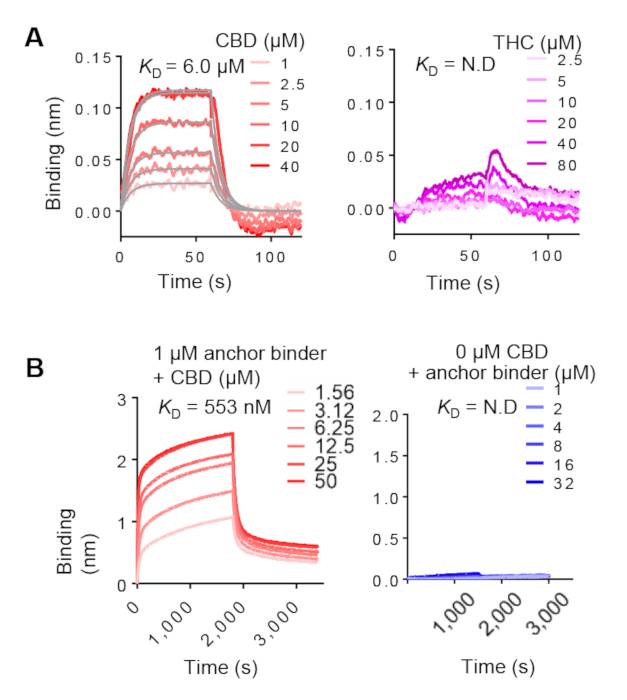
Figura 7: Analisi cinetica legante di ancoraggio e dimerizzazione da parte di BLI. (A) Analisi del legante di ancoraggio con CBD senza etichetta (a sinistra) e THC (a destra). Legante di ancoraggio biotinylato è stato immobilizzato su biosensori Super Streptavidin (SSA) tiitrated con diverse concentrazioni di CBD. I dati misurati per l'attacco CBD (curve rosse) sono stati montati globalmente (linee grigie). (B) Sinistra, analisi BLI di un legante di dimerizzazione a biosensore SA legato al legante di ancoraggio preequilibrato con diverse concentrazioni di CBD. Giusto, la concentrazione del legante di ancoraggio è stata titonata e legata al legante di dimerizzazione immobilizzato in assenza di CBD. Fare clic qui per visualizzare una versione più grande di questa figura.
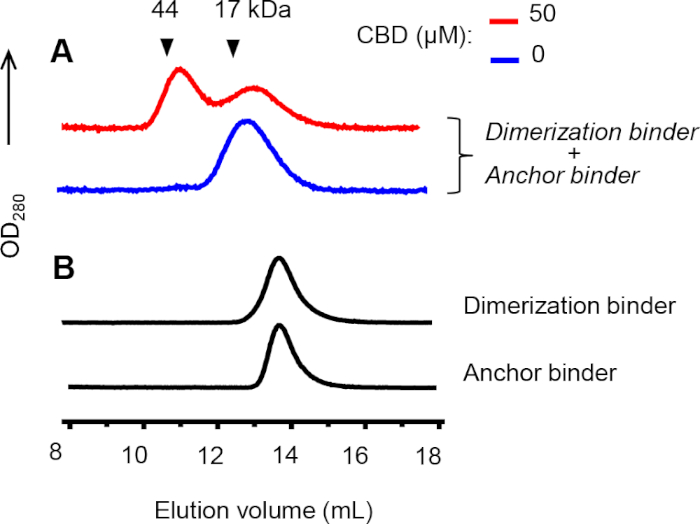
Figura 8: Analisi SEC dell'eterodimerizzazione tra i leganti di ancoraggio e dimerizzazione. (A) La dimerizzazione e i leganti di ancoraggio (5 m ciascuno) in presenza o assenza di CBD sono stati incrociati da 100 m bis-N-succinimidyl-(pentatilelene glicole) ester per 30 min a RT prima dell'analisi. I volumi di eluzione degli standard proteici sono contrassegnati da triangoli. (B) I leganti di ancoraggio e di dimerizzazione non collegati (30 m ciascuno) sono stati iniettati separatamente. I cromatogrammi in A e B sono mostrati in diverse scale Y. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
È fondamentale scegliere le giuste concentrazioni di librerie di faghi di input per diversi cicli di biopanning. In genere siamo partiti da una libreria di input di 10-1013 particelle di fago con una diversità >109, consentendo la presentazione di 100-1.000 copie di ogni clone di fago nel saggio pull-down. Se la concentrazione di fago in un saggio vincolante è troppo alta o bassa, la probabilità di legame non specifico o perdita di cloni positivi aumenterà. La selezione del legante di ancoraggio o di dimerizzazione consiste normalmente di tre o sei cicli di biopanning, e il numero di fagi di uscita di solito inizia da 104 dollari e aumenta a 10–109dollari. È adatto per scegliere singoli cloni per la convalida ELISA dopo aver osservato tale arricchimento. Ulteriori cicli di biopanning potrebbero diminuire le possibilità di identificare cloni positivi adatti a bassa abbondanza.
È importante impostare controlli negativi adatti e selezioni per migliorare il successo delle selezioni. Ad esempio, l'uso di analoghi strutturali di bersagli ligando faciliterà la selezione della specificità del ligando. Nel nostro lavoro, un analogo molto simile, THC, è stato utilizzato come controllo per CBD nella convalida ELISA e BLI di ancora e dimerizzazione leganti19. Nella selezione del legante di dimerizzazione, se i leganti di ancoraggio hanno un'affinità di legatura legamento ligand relativamente bassa, i leganti di ancoraggio liberi e legati al ligando possono essere presentati come obiettivi nella selezione positiva. Pertanto, è importante rimuovere accuratamente i leganti che si legano ai leganti di ancoraggio liberi durante la selezione negativa. Questo può essere ottenuto eseguendo più giri della sottrazione con leganti di ancoraggio liberi.
Una limitazione del nostro protocollo è che le molecole bersaglio devono essere biotinylate per la selezione del legante di ancoraggio e solo uno o pochi leganti di ancoraggio possono essere utilizzati per la selezione della dimerizzazione. L'uso di obiettivi biotinylati può arricchire i leganti che si legano in parte al linker tra biotina e bersagli. Pertanto, è importante convalidare gli hit utilizzando destinazioni senza etichetta da BLI o altre tecniche. La scelta di un singolo o di alcuni leganti di ancoraggio per la selezione del legante di dimerazione può ridurre la possibilità di identificare sistemi CID con sensibilità e specificità adeguate. Pertanto, la capacità di multiplexing della selezione attende un ulteriore miglioramento accoppiandosi ad altre tecniche, ad esempio il sequenziamento a interazione singola molecolare (SMI-Seq) che consente uno screening dell'interazione proteina-proteina "libreria per libreria"25.
Divulgazioni
Un brevetto provvisorio relativo a questo lavoro è stato depositato dall'Università di Washington.
Riconoscimenti
Questo lavoro è stato sostenuto dall'University of Washington Innovation Award (a L.G.), una sovvenzione degli U.S. National Institutes of Health (1R35GM128918 a L.G.), e da un fondo di avvio dell'Università di Washington (a L.G.). H.J. è stato sostenuto da una borsa di studio universitaria della Washington Research Foundation. K.W. è stato sostenuto da una borsa di studio universitaria dell'Università di Washington Institute for Protein Design.
Materiali
| Name | Company | Catalog Number | Comments |
| 1-Step Ultra TMB ELISA substrate solution | Thermo Fisher Scientific | 34029 | |
| Agar | Thermo Fisher Scientific | BP1423-2 | |
| Amicon Ultra-15 Centrifugal Filter unit (3 kDa cutoff) | Millipore | UFC900324 | |
| Ampicillin | Thermo Fisher Scientific | BP1760-25 | |
| Bio-Rad Protein Assay Kit II | Bio-Rad | 5000002 | |
| BirA biotin-protein ligase standard reaction kit | Avidity | BirA500 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A2153-50G | |
| Casein | Sigma-Aldrich | C7078-1KG | |
| CM13 Helper phage | Antibody Design Labs | PH020L | |
| D-(+)-Glucose monohydrate | Alfa Aesar | A11090 | |
| Dynabeads M-280 Streptavidin | Thermo Fisher Scientific | 11205D | |
| DynaMag-2 Magnet | Thermo Fisher Scientific | 12321D | |
| EDTA | Thermo Fisher Scientific | BP120-1 | |
| Fast DNA Ladder | New England Biolabs | N3238S | |
| FastDigest BglI | Thermo Fisher Scientific | FD0074 | |
| Glycerol | Thermo Fisher Scientific | BP229-1 | |
| HiLoad 16/600 Superdex 200 pg | GE Healthcare | 28989335 | |
| HiPrep 26/10 Desalting Column | GE Healthcare | 17508701 | |
| HisTrap-FF-1ml | GE Healthcare | 11000458 | |
| Imidazole | Alfa Aesar | 161-0718 | |
| IPTG | Thermo Fisher Scientific | 34060 | |
| Kanamycin | Thermo Fisher Scientific | BP906-5 | |
| M13 Major Coat Protein Antibody | Santa Cruz Biotechnology | sc-53004 | |
| NaCl | Sigma-Aldrich | S3014-500G | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Fisher Scientific | ND-2000 | |
| Nunc 96-Well Polypropylene DeepWell Storage Plates | Thermo Fisher Scientific | 260251 | |
| Nunc MaxiSorp | Thermo Fisher Scientific | 44-2404-21 | |
| Octet RED96 | ForteBio | N/A | |
| pADL-23c Phagemid Vector | Antibody Design Labs | PD0111 | |
| PEG-6000 | Sigma-Aldrich | 81260-1KG | |
| Platinum SuperFi DNA Polymerase | Invitrogen | 12351010 | |
| PureLink PCR Purification Kit | Thermo Fisher Scientific | K310001 | |
| QIAprep Spin M13 Kit | Qiagen | 27704 | |
| Recovery Medium | Lucigen | 80026-1 | |
| SpectraMax Plus 384 | Molecular Devices | N/A | |
| Sucrose | Sigma-Aldrich | S0389-1KG | |
| Super Streptavidin (SSA) Biosensors | ForteBio | 18-5057 | |
| Superdex 75 increase 10/300 GL Column | GE Healthcare | 28-9909-44 | |
| T4 DNA Ligase | Thermo Fisher Scientific | 15224-025 | |
| TG1 Electrocompetent Cells | Lucigen | 60502-1 | |
| Triethylamine | Sigma-Aldrich | 471283-100mL | |
| Trizma Base | Sigma-Aldrich | T1503 | |
| Tryptone | Thermo Fisher Scientific | BP9726-5 | |
| Tween 20 | Thermo Fisher Scientific | BP337-500 | |
| Yeast Extract | Thermo Fisher Scientific | BP1422-2 | |
| Zeba Spin Desalting Column | Thermo Fisher Scientific | 89882 |
Riferimenti
- Stanton, B. Z., Chory, E. J., Crabtree, G. R. Chemically induced proximity in biology and medicine. Science. 359 (6380), (2018).
- Wu, C. Y., Roybal, K. T., Puchner, E. M., Onuffer, J., Lim, W. A. Remote control of therapeutic T cells through a small molecule-gated chimeric receptor. Science. 350 (6258), (2015).
- Straathof, K. C., et al. An inducible caspase 9 safety switch for T-cell therapy. Blood. 105 (11), 4247-4254 (2005).
- Di Stasi, A., et al. Inducible apoptosis as a safety switch for adoptive cell therapy. The New England Journal of Medicine. 365 (18), 1673-1683 (2011).
- Mank, M., et al. A FRET-based calcium biosensor with fast signal kinetics and high fluorescence change. Biophysical Journal. 90 (5), 1790-1796 (2006).
- Nagai, T., Sawano, A., Park, E. S., Miyawaki, A. Circularly permuted green fluorescent proteins engineered to sense Ca2+. Proceedings of the National Academy of Sciences of the United States of America. 98 (6), 3197-3202 (2001).
- Hunter, M. M., Margolies, M. N., Ju, A., Haber, E. High-affinity monoclonal antibodies to the cardiac glycoside, digoxin. Journal of Immunology. 129 (3), 1165-1172 (1982).
- Bradbury, A. R. M., Sidhu, S., Dubel, S., McCafferty, J. Beyond natural antibodies: the power of in vitro display technologies. Nature Biotechnology. 29 (3), 245-254 (2011).
- Chen, G., et al. Isolation of high-affinity ligand-binding proteins by periplasmic expression with cytometric screening (PECS). Nature. Biotechnology. 19 (6), 537-542 (2001).
- Tinberg, C. E., et al. Computational design of ligand-binding proteins with high affinity and selectivity. Nature. 501 (7466), 212-216 (2013).
- Spencer, D. M., Wandless, T. J., Schreiber, S. L., Crabtree, G. R. Controlling signal transduction with synthetic ligands. Science. 262 (5136), 1019-1024 (1993).
- Ho, S. N., Biggar, S. R., Spencer, D. M., Schreiber, S. L., Crabtree, G. R. Dimeric ligands define a role for transcriptional activation domains in reinitiation. Nature. 382 (6594), 822-826 (1996).
- Belshaw, P. J., Ho, S. N., Crabtree, G. R., Schreiber, S. L. Controlling protein association and subcellular localization with a synthetic ligand that induces heterodimerization of proteins. Proceedings of the National Academy of Sciences of the United States of America. 93 (10), 4604-4607 (1996).
- Farrar, M. A., AlberolaIla, J., Perlmutter, R. M. Activation of the Raf-1 kinase cascade by coumermycin-induced dimerization. Nature. 383 (6596), 178-181 (1996).
- Erhart, D., et al. Chemical Development of Intracellular Protein Heterodimerizers. Chemistry & Biology. 20 (4), 549-557 (2013).
- Ballister, E. R., Aonbangkhen, C., Mayo, A. M., Lampson, M. A., Chenoweth, D. M. Localized light-induced protein dimerization in living cells using a photocaged dimerizer. Nature Communications. 17 (5), 5475 (2014).
- Hill, Z. B., Martinko, A. J., Nguyen, D. P., Wells, J. A. Human antibody-based chemically induced dimerizers for cell therapeutic applications. Nature Chemical Biology. 14 (2), 112-117 (2018).
- Foight, G. W., et al. Multi-input chemical control of protein dimerization for programming graded cellular responses. Nature Biotechnology. 37 (10), 1209-1216 (2019).
- Kang, S., et al. COMBINES-CID: An efficient method for de novo engineering of highly specific chemically induced protein dimerization systems. Journal of the American Chemical Society. 141 (28), 10948-10952 (2019).
- Muyldermans, S. Nanobodies: natural single-domain antibodies. Annual Review of Biochemistry. 82, 775-797 (2013).
- Fanning, S. W., Horn, J. R. An anti-hapten camelid antibody reveals a cryptic binding site with significant energetic contributions from a nonhypervariable loop. Protein Science. 20 (7), 1196-1207 (2011).
- Zavrtanik, U., Luken, J., Loris, R., Lah, J., Hadzi, S. Structural basis of epitope recognition by heavy-chain camelid antibodies. Journal of Molecular Biology. 430 (21), 4369-4386 (2018).
- Denhardt, D. T., Dressler, D., Ray, D. S. . The Single-Stranded DNA Phages. , 605-625 (1978).
- Virnekas, B., et al. Trinucleotide phosphoramidites: ideal reagents for the synthesis of mixed oligonucleotides for random mutagenesis. Nucleic Acids Research. 22 (25), 5600-5607 (1994).
- Gu, L., et al. Multiplex single-molecule interaction profiling of DNA-barcoded proteins. Nature. 515 (7528), 554-557 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon