Method Article
Creación de sistemas de dimerización de proteínas altamente específicos inducidos químicamente por la selección de fagos escalonados de una biblioteca combinatoria de anticuerpos de un solo dominio
* Estos autores han contribuido por igual
En este artículo
Resumen
La creación de sistemas de dimerización de proteínas inducidos químicamente con la afinidad y especificidad deseadas para cualquier ligando de moléculas pequeñas dado tendría muchas aplicaciones de detección y accionamiento biológico. Aquí, describimos un método eficiente y generalizable para la ingeniería de novo de sistemas de dimerización inducidos químicamente a través de la selección escalonada de una biblioteca combinatoria de anticuerpos de un solo dominio visualizada por fago.
Resumen
Los eventos de dimerización de proteínas que se producen sólo en presencia de un ligando de moléculas pequeñas permiten el desarrollo de biosensores de moléculas pequeñas para la disección y manipulación de vías biológicas. Actualmente, sólo existe un número limitado de sistemas de dimerización inducida químicamente (CID) y la ingeniería de otros nuevos con sensibilidad y selectividad deseadas para ligandos específicos de moléculas pequeñas sigue siendo un desafío en el campo de la ingeniería de proteínas. Aquí describimos un método de cribado de alto rendimiento, combin binders-enabled selección de CID (COMBINES-CID), para la ingeniería de novo de sistemas CID aplicables a una gran variedad de ligandos. Este método utiliza la selección en dos pasos de una biblioteca combinatoria nanobody mostrada por fago para obtener 1) "aglutinantes de anclaje" que primero se unen a un ligando de interés y luego 2) "aglutinantes de dimerización" que solo se unen a los complejos de anglutinante-ligando. Para seleccionar aglutinantes de anclaje, una biblioteca combinatoria de más de 109 nanocuerpos aleatorizados de región determinante de complementariedad (CDR) se examinan con un ligando biotinilado y los hits se validan con el ligando sin etiquetar mediante interferometría de capa biológica (BLI). Para obtener aglutinantes de dimerización, la biblioteca de nanobody se analiza con complejos de anclarios de anclaje como objetivos para el cribado positivo y los aglutinantes de anclaje no enlazados para el cribado negativo. COMBINES-CID es ampliamente aplicable a aglutinantes de CID seleccionados con otras inmunoglobulinas, no inmunoglobulina o andamios diseñados computacionalmente para crear biosensores para la detección in vitro e in vivo de fármacos, metabolitos, moléculas de señalización, etc.
Introducción
Los sistemas CID, en los que dos proteínas dimerize sólo en presencia de un ligando de moléculas pequeñas(Figura 1),ofrecen herramientas versátiles para disecar y manipular vías metabólicas, de señalización y otras vías biológicas1. Han demostrado el potencial en el accionamiento biológico, como la activación de células T controlada por fármacos2 y la apoptosis3,4,para mejorar la seguridad y eficacia de la terapia adoptiva de células T. Además, proporcionan una nueva metodología para la detección in vivo o in vitro de objetivos de moléculas pequeñas. Por ejemplo, las proteínas CID pueden fusionarse genéticamente con sistemas de reporteros de fluorescencia (por ejemplo, transferencia de energía por resonancia de fluorescencia (FRET)5 y proteínas fluorescentes permutadas circularmente)6 para mediciones in vivo en tiempo real, o servir como reactivos de afinidad para ensayos similares a inmunoabsorbentes ligados a enzimas sándwich (ELISA).
A pesar de sus amplias aplicaciones, la creación de nuevos sistemas CID que pueden ser controlados por un ligando de moléculas pequeñas determinado tiene grandes desafíos. Los métodos establecidos de ingeniería de aglutinantes de proteínas, incluida la inmunización animal7, la selección in vitro8,9,y el diseño de proteínas computacionales10 pueden generar proteínas de unión de ligando que funcionan a través de interacciones binarias proteína-ligand. Sin embargo, estos métodos tienen dificultades para crear un complejo de CID ternario inducido por ligando. Algunos métodos crean CID mediante la vinculación química de dos ligandos que se unen de forma independiente a las mismas o diferentes proteínas11,12,13,14,15,16 o dependen de la selección de proteínas aglutinantes como anticuerpos dirigidos a pequeños complejos de moléculas-proteínas preexistentes17,18, y por lo tanto tienen una opción limitada de ligandos.
Recientemente desarrollamos un método de ingeniería denovo de los sistemas CID19. Este método puede obtener la alta especificidad de la dimerización inducida por ligando (por ejemplo, una constante de disociación de aglutinante de anclaje-dmerización, KD (sin ligando) /KD (con ligando) > 1.000). La especificidad de la dimerización se logra utilizando aglutinantes de anclaje con sitios de unión flexibles que pueden introducir cambios de conformación sobre la unión de ligandos, proporcionando una base para la selección de aglutinantes selectivos de conformación que sólo reconocen aglutinantes de anclaje ligandos. Demostramos una prueba de principio mediante la creación de heterodímeros inducidos por cannabidiol (CBD) de nanocuerpos, un fragmento de anticuerpo funcional de 12-15 kDa de camión que comprende un andamio universal y tres bucles CDR flexibles(Figura 2)20,que pueden formar un bolsillo de unión con tamaños adaptables para epítopos de moléculas pequeñas21,22. En particular, la selección in vitro de una biblioteca de proteínas combinatorias debe ser rentable y generalizable para la ingeniería CID, ya que la misma biblioteca de alta calidad se puede aplicar a diferentes ligandos.
En este protocolo y vídeo, nos centramos en describir la selección y validación in vitro en dos pasos del anclaje(Figura 3A)y los aglutinantes de dimerización(Figura 3B)mediante la selección de la biblioteca combinatorial de nanobody con una diversidad superior a 109 utilizando el CBD como objetivo, pero el protocolo debe ser aplicable a otras bibliotecas de proteínas o objetivos de moléculas pequeñas. El cribado de aglutinantes CID suele tardar de 6 a 10 semanas(Figura 4).
Protocolo
1. Construcción de bibliotecas
- Utilice una biblioteca de anticuerpos combinatoria sintética de un solo dominio con una diversidad de 1,23 a 7,14 x 109, como se ha descrito anteriormente19. Aunque este protocolo no incluye la construcción de bibliotecas, se puede aplicar a otras bibliotecas de carpetas combinatorias.
2. Biotinylación de ligando objetivo o ligando
- Biotinylate el ligando seleccionado, por ejemplo, CBD y tetrahidrocannabinol (THC)19, a través de diversas estrategias de síntesis química, dependiendo de los sitios de biotinylación adecuados de un objetivo.
3. Detección de aglutinante de anclaje
- Inicio de la selección
- Comience cada ronda de selección inoculando una sola colonia de células TG1, recién cultivada en 6 ml de 2YT a 37 oC y 250 revoluciones por minuto (rpm) a una absorbancia de 600 nm (OD600) de 0,5 euros. Incubar las células sobre hielo para su uso en el paso 3.5.1.
- Selección negativa con perlas de estreptavidina ligadas a la biotina
- Preparar las "perlas de selección negativas" lavando 300 l de perlas magnéticas recubiertas de estreptavidina utilizando un bastidor de separación magnética, 3x con 0.05% de solución salina con fosfato con tampón De weno (PBST, 1 x PBS con 0.05% vol/vol Tween 20%) y 2x con 1 x PBS.
- Resuspenda las perlas con 1 ml de caseína del 1% en 1 x PBS (pH a 7,4) y satura las perlas añadiendo 5 veces la capacidad de unión reportada utilizando biotina. Incubar a temperatura ambiente (RT) en un rotador durante 1 h.
- Lave las perlas 5x usando 0.05% PBST y 3x usando 1 x PBS, para un total de ocho lavados.
- Añadir 1013 partículas de fago en 1% de caseína/1% de BSA en 1 x PBS (pH a 7,4) e incubar a RT en un rotador durante 1 h.
- Después de la incubación, recoja el sobrenadante que se utilizará en el paso 3.3.6.
- Selección positiva con perlas de estreptavidina ligadas biotiniadas
- Prepare las "cuentas de selección positiva" usando 1/2 el volumen de las cuentas utilizadas para las "cuentas de selección negativa" siguiendo los pasos 3.2.1.
- Vuelva a suspender las perlas con 1 ml de caseína de 1 ml en 1 PBS, pH 7,4 y saturar las perlas añadiendo 5 veces la capacidad de unión completa calculada sobre la base del manual utilizando el ligando biotintilado de elección. Incubar a RT en un rotador durante 1 h.
- Lave las perlas 5x usando 0.05% PBST y 3x usando 1 x PBS, para un total de ocho lavados.
- Bloquear las perlas con 1 ml de 1% de caseína/1% de BSA en 1 x PBS (pH a 7,4) e incubar a RT en un rotador durante 1 h para evitar la unión inespecífica entre los fagas y las perlas magnéticas recubiertas de estreptavidina.
- Lave las perlas magnéticas recubiertas de estreptavidina 3 veces usando 0.05% PBST y una vez usando 1 x PBS, para un total de cuatro lavados.
- Resuspenda las perlas magnéticas recubiertas de estreptavidina utilizando los fagos no atados tomados del paso 3.2.5 e incubar a RT en un rotador durante 1 h.
- Extraiga el sobrenadante sin perturbar las cuentas magnéticas. Guarde los fagos no enlazados como entrada, que se utilizarán en el paso 3.5.1.
- Lave las perlas 10x usando 0.05% PBST y 5x usando 1 x PBS. Entre cada tres lavados los transfieren a un tubo nuevo para evitar los fagos no específicamente unidos a las paredes del tubo.
- Elución de nanocuerpos expuestos en fago
- Eluir los fagos unidos competitivamente añadiendo 450 l del ligando no biotinilado,utilizando una concentración en el rango micromolar (por ejemplo, 10-50 m) e incubando a RT en un rotador durante 30 minutos. La concentración de ligando seleccionada para la elución competitiva de fagos enlazados depende de la KD deseada del "aglutinante ancla". Las concentraciones de ligando pueden ser relativamente altas en las rondas de selección iniciales y luego disminuir en rondas posteriores.
- Recoger sobrenadante y guardar los fagos elustres como salida, para ser utilizado en el paso 3.5.2.
- Valoraciones de entrada/salida e infección
- Para la valoración de entrada, prepare diluciones seriales de 10x en 1 x PBS hasta 109veces con el fago de entrada del paso 3.3.7. Utilice las 107–109 diluciones en serie para hacer infecciones transfiriendo un fago de entrada de 10 l de cada dilución a células TG1 de 70 l (OD600 de 0,5). Incubar a 37 oC durante 45 minutos, placar las células TG1 infectadas en tres platos de agar 2YT de 90 mm que contengan 100 g/ml de ampicilina y 2% (wt/vol) glucosa, e incubar durante la noche a 37 oC. A partir de las placas de la noche, la entrada de fago se puede calcular de la siguiente manera:

- Para la infección de salida y la valoración, transfiera los fagos elustres del paso 3.4.2 a 3 mL de las células TG1 (OD600 de 0,5). Incubar en un baño de agua a 37oC durante 45 min. A continuación, prepare diluciones en serie 10x en 2YT hasta 103veces, placa cada dilución en 90 mm 2YT-agar platos, e incubar durante la noche a 37 oC. A partir de las placas de la noche, la salida de fago se puede calcular de la siguiente manera:

- Divida las células TG1 infectadas restantes en tres placas de agar 2YT de 150 mm que contengan 100 g/ml de ampicilina y un 2% (wt/vol) de glucosa. Incubar placas durante la noche a 37oC.
- Para la valoración de entrada, prepare diluciones seriales de 10x en 1 x PBS hasta 109veces con el fago de entrada del paso 3.3.7. Utilice las 107–109 diluciones en serie para hacer infecciones transfiriendo un fago de entrada de 10 l de cada dilución a células TG1 de 70 l (OD600 de 0,5). Incubar a 37 oC durante 45 minutos, placar las células TG1 infectadas en tres platos de agar 2YT de 90 mm que contengan 100 g/ml de ampicilina y 2% (wt/vol) glucosa, e incubar durante la noche a 37 oC. A partir de las placas de la noche, la entrada de fago se puede calcular de la siguiente manera:
- Amplificación y recuperación de la biblioteca para nuevas rondas de selección
- Añadir 3 ml de 2YT por placa, raspar con un rascador de celda estéril y recoger todas las células en un tubo cónico de 50 ml. Mezclar las células recolectadas con glicerol estéril (20% wt/vol concentración final). Mida la OD600 de la mezcla y haga 3-5 alícuotas en stock. Conservar a -80 oC para un almacenamiento a largo plazo.
- Para el rescate de fagos, diluir la mezcla bacteriana TG1 que contiene fagomi utilizando 25 ml de medios 2YT complementados con 2% de glucosa y 100 g/ml de ampicilina a una DO600 de 0,1 euros. Células de cultivo a 37 oC y 250 rpm a una OD600 de 0,5.
- Superinfectar las células mediante la adición de FAgo auxiliar CM13 a 5 x 109 pfu/ml e incubar a 37 oC y 250 rpm durante 45 min. El fago auxiliar CM13 proporciona las proteínas de capa de fago necesarias para el montaje de partículas completas de fago.
- Centrifugar el cultivo a 8.000 x g durante 10 min para eliminar la glucosa. Resuspenda las células utilizando 50 ml de medios 2YT suplementados con 100 g/ml de ampicilina y 50 g/ml de kanamicina e incubar a 25 oC y 250 rpm durante la noche.
- Centrifugar las células del cultivo nocturno a 9.000 x g, 4 oC durante 30 min. Transfiera el sobrenadante a un tubo nuevo y precipite los fagos en el sobrenadante utilizando la solución de 1/5 volumen PEG/NaCl (20% wt/vol polietilenglicol-6,000 y 2,5 M NaCl). Mezclar suavemente y colocar sobre hielo durante 1 h.
- Recoger las partículas de fago por centrifugación utilizando 12.000 x g a 4 oC durante 30 min. Resuspender los pellets usando 1 ml de 1 x PBS, y transferir la suspensión a un tubo de microcentrífuga. Centrifugar el tubo a 20.000 x g y 4 oC durante 10 min para eliminar las bacterias residuales.
- Transfiera el sobrenadante a un nuevo tubo de microcentrífuga sin alterar el pellet bacteriano. Utilice una dilución de 1:100 para medir la absorción a 269 nm y 320 nm. El número total de fagos se puede calcular utilizando la siguiente fórmula23:

- Almacene la biblioteca de fagos a 4 oC para uso a corto plazo o con un 25% de glicerol a -80 oC para un almacenamiento a largo plazo.
- Repita las rondas de selección (pasos 3.1–3.6) durante 3-6 rondas o hasta que se observe el enriquecimiento deseado (consulte la sección Resultados). Placa y recoger clones individuales (sección 4) con el fin de caracterizar su afinidad y especificidad con el ligando (secciones 5–7).
4. Aislamiento de clon único
- Para aislar clones individuales de una subbiblioteca enriquecida, prepare diluciones en serie 10 veces de las células TG1 infectadas por fago (paso 3.5.2). Diluciones en serie de placas en platos de agar de 90 mm 2YT que contienen 100 g/ml de ampicilina y 2% (wt/vol) de glucosa e incuban a 37 oC durante la noche.
- A partir de las placas de perno, recoger colonias individuales en 250 l de medios 2YT complementados con 100 g/ml de ampicilina por pozo en placas estériles de pozo profundo y crecer a 37 oC durante la noche.
- A partir de los cultivos nocturnos, inocular 10 l en 500 ml de medios frescos 2YT complementados con 100 g/ml de ampicilina.
- Cultivar las células a un OD600 de 0,5, añadir el fago auxiliar CM13 a 5 x 109 pfu/ml e incubar a 37 oC y 250 rpm durante 45 min.
- Añadir 500 s de medios 2YT suplementados con 100 g/ml de ampicilina y 50 g/ml de kanamicina. Incubar a 25oC y 250 rpm durante la noche.
- Centrifugar las placas de pozo profundo de los cultivos nocturnos a 3.000 x g durante 10 min. Recoger el sobrenadante que contiene las partículas de fago sin perturbar el pellet celular.
- Las partículas de fago se pueden utilizar para ELISA para determinar la especificidad de los clones seleccionados al ligando. La biotina o un homólogo estructural del objetivo se puede utilizar como un control negativo.
5. Validación del aglutinante de anclaje por ELISA
- Recubrir 96 placas ELISA de pozo sin 100 ml de estreptavidina de 5 g/ml en tampón de recubrimiento (100 mM de tampón de carbonato, pH a 8,6) a 4 oC durante la noche.
- Lave las placas ELISA 3 veces con 0.05% PBST y agregue 100 sL de objetivo biotintilado de 1 M a los pocillos objetivo. Añadir 100 s de biotina de 1 m o seleccionar el homólogo a los pozos de control. Incubar a RT durante 1 h.
- Lave las placas 5 veces con 0.05% PBST y bloquee la unión inespecífica añadiendo 300 sl de 1% de caseína en 1 x PBS. Incubar a RT durante 1 h.
- Lave las placas ELISA 3x usando 0.05%-PBST y agregue el sobrenadante de fago purificado. Incubar durante 1 h a RT.
- Lavar las placas ELISA 10veces usando 0.05% PBST y añadir 100 l de peroxidasa de rábano picante (HRP)-M13 anticuerpo de proteína de capa principal (1:10,000 dilución con 1 x PBS con 1% de caseína). Incubar a RT durante 1 h.
- Lavar las placas ELISA 3x usando 0.05% PBST y añadir 100 éL de sustrato de tetrametilbenzidina (TMB). Incubar durante 10 min o hasta que se observe un cambio de color visible. Detenga la reacción añadiendo 100 ml de 1 M De HCl. Lea la placa a 450 nm en un espectrofotómetro.
- Para la expresión y purificación de proteínas, elija los clones que muestren alta afinidad y especificidad para el objetivo (ver Discusión).
6. Expresión de proteínas, purificación y biotinylación
- Como se informó anteriormente19, subclone seleccionó clones de la sección 5 y expresar como C-terminal Avi-tagged y His-tagged nanobodies.
- Expresar nanocuerpos seleccionados en el periplasma de las células de E. coli WK6 (normalmente en cultivo de 1 L), liberar por choque osmótico y purificar utilizando una columna de níquel-NTA (ver Tabla de Materiales).
- Búfer de intercambio con una columna de desalación (1 x PBS con 5% de glicerol; véase Tabla de materiales).
- Biotinylate nanocuerpos utilizando un kit comercial (ver Tabla de Materiales)para su uso posterior.
7. Caracterización del aglutinante de anclaje por BLI
- Analizar la afinidad de unión y la cinética de los aglutinantes de anclaje seleccionados mediante la inmovilización de aglutinantes de anclaje biotinylados de 200 nM en biosensores de estreptavidina (ver Tabla de Materiales)con tampón de ensayo de unión (1 x PBS (pH a 7,4), 0,05% Tween 20, 0,2% BSA, 3% de metanol).
- Calcular las constantes de disociación (KD) de las interacciones de anglutinante-ligand mediante análisis de estado estacionario mediante software de análisis de datos (véase Tabla de materiales). Los valores KD obtenidos suelen oscilar entre micromolarde de uno a dos dígitos.
8. Detección de aglutinantes de dimerización
NOTA: El cribado de biopausa de los "aglutinantes de dimerización" es similar al de los aglutinantes de anclaje, excepto por dos pasos críticos: 1) Los aglutinantes de dimerización se seleccionan utilizando un aglutinante de anclaje biotinilado seleccionado y el complejo de aglutinantes de anclaje para el complejo negativo de aglutinante para el y selecciones positivas, respectivamente. 2) Durante el paso de elución, la trietilamina de 100 mM se utiliza para eluir fagos seleccionados positivamente que sólo estaban unidos al aglutinante de anclaje--ligand complejo objetivo. La solución de trimetilamina de 100 mM (pH a 11,5) se utiliza para eluir clones positivos al interrumpir las interacciones proteicas.
- Inicio de la selección
- Comience cada ronda de selección inoculando una sola colonia celular TG1, recién cultivada en un medio mínimo, en 6 mL 2YT a 37 oC y 250 rpm a una OD600 de 0,5 euros. Incubar células sobre hielo.
- Eliminación de nanocuerpos seleccionados negativamente
- Preparar el "tubo de resta" utilizando 400 s de perlas magnéticas recubiertas de estreptavidina y siga el paso 3.2. Sin embargo, en lugar de saturar con biotina, agregue 5 veces la capacidad de unión completa calculada utilizando el aglutinante de anclaje biotintilado seleccionado y guarde los fagos no unidos que se utilizarán en el paso 8.3.3.
- Selección de nanocuerpos seleccionados positivamente
- Preparar el "tubo de captura" utilizando 1/2 el volumen de perlas magnéticas recubiertas de estreptavidina utilizadas para el "tubo de resta" y siguiendo los pasos 3.3.2 a 3.3.3. Sin embargo, en lugar de saturar con el ligando biotinilado, agregue cinco veces la capacidad de unión completa calculada utilizando el aglutinante de anclaje biotiniladoseleccionado.
- Para formar el complejo de anglutinantes de anclaje para la selección de aglutinantes de dimerización positiva, agregue una concentración suficientemente alta de ligando no biotinilado. Esto permitirá que la mayoría del aglutinante de anclaje ligado a la estreptavidina forme el complejo ligando al ligando.
- Siga los pasos 3.3.3 a 3.3.8, utilizando los fagos no unidos tomados del "tubo de resta".
- Elución de nanocuerpos seleccionados positivamente
- Eluir los fagos unidos al complejo de anglutinantes de anclaje añadiendo 450 éL de trietilamina de 100 mM, e incubando a RT en un rotador durante 10 min.
- Recoge los fagos esumiantes competitivos y sigue los pasos 3.4.1 a 3.4.2.
- Otras rondas de selección de aglutinantes de dimerización
- Siga los pasos 3.5 y 3.6 para amplificar y recuperar la biblioteca con el fin de realizar más rondas de selección. Repita las rondas de selección durante 3-6 rondas o hasta que se observe el enriquecimiento deseado. Placa y selección de clones individuales (consulte la sección 4) para caracterizar su afinidad y especificidad con el objetivo.
9. Caracterización del aglutinante de dimerización por ELISA
- Siga los pasos de la sección 4 para aislar clones individuales para su caracterización a través de ELISA.
- Para probar la afinidad de los candidatos de aglutinante de dimerización al complejo de aglutinantes de anclaje, cubra la placa diana ELISA utilizando 100 ml de aglutinante de anclaje biotinilado de 100 nM. Después de la incubación durante 1 h, agregue 1 m del objetivo de ligando para formar el complejo de anclarón-ligand de anclaje.
- La placa de control debe ser recubierta usando el aglutinante de anclaje biotinylated solo para analizar clones que también pueden unirse al aglutinante de anclaje libre. Añadir 100 l de aglutinante de anclaje biotinylado de 100 nM e incubar a RT durante 1 h.
- Siga las secciones 5.3–5.7.
10. Caracterización del aglutinante de dimerización por BLI
- La afinidad de unión y la cinética de los aglutinantes de dimerización para el aglutinante de anclaje- complejo de ligando se puede analizar mediante la inmovilización de aglutinantes de dimerización biotinilados en biosensores de estreptavidina (SA) con el búfer de ensayo de unión y luego se ensayo con 1 Aglutinante de anclaje preequilibrado con diluciones en serie del ligando. El KD, kon, y kfuera de las interacciones se puede calcular utilizando nuestro método reportado19.
Resultados
Describimos la selección in vitro en dos pasos y la validación de aglutinantes de anclaje y dimerización mediante la selección de la biblioteca combinatorial nanobody con una diversidad superior a 109 utilizando CBD como objetivo. Evaluar el enriquecimiento de la biopanning del fago durante las sucesivas rondas de selección para aglutinantes de anclaje y de dimerización es importante. Los resultados de enriquecimiento típicos después de 4-6 rondas de selección como se muestra en la Figura 5 son una buena indicación de que hay una alta proporción de impactos potenciales en las subbibliotecas, por lo que es posible que no sean necesarias nuevas rondas de selección.
ELISA de un solo clon es adecuado para analizar la afinidad de enlace relativa y la selectividad de los aglutinantes de anclaje y dimerización. La Figura 6A es un resultado representativo de selección de aglutinante de anclaje después de seis rondas de biopanación. Se pueden comparar clones que muestren alta (p. ej., #87) o baja (p. ej., #27) de ligando). Los clones de alta selectividad deben elegirse como candidatos de aglutinante de anclaje. Del mismo modo, la Figura 6B muestra los resultados de la selección del aglutinante de dimerización después de cuatro rondas de biopanción. Normalmente observamos clones que formaban un heterodimer con el aglutinante de anclaje inmovilizado sólo con el ligando (por ejemplo, #49) o sin (por ejemplo, #80). El primero, que muestra la especificidad de la dmerización, debe seleccionarse para su posterior validación.
El aglutinante de anclaje ELISA se basa en el uso del objetivo biotinilado. Por lo tanto, necesitamos utilizar BLI para confirmar aún más el enlace al objetivo no etiquetado. BLI también permite la caracterización de la cinética de unión. Los resultados representativos de BLI de los aglutinantes de anclaje y dimerización se muestran en las figuras 7A y 7B,respectivamente. Los paneles izquierdos muestran la unión dependiente de la concentración de ligando, lo que sugiere que son adecuados para la construcción de un sistema CID. Los paneles derecho muestran los controles negativos. La KD calculada de las interacciones de ancla y aglutinante de dimerización en presencia del ligando típicamente varió de nanomolar de dos dígitos a micromolar de dos dígitos. Pueden variar dependiendo del ligando y la biblioteca combinatoria de elección.
Se realizó cromatografía analítica de exclusión de tamaño (SEC) para confirmar la formación de heterodímeros entre el anclaje y los aglutinantes de dimerización. Se observó un pico de dimerización cuando se mezclaron los aglutinantes de anclaje y dimerización y CBD(Figura 8A,línea roja). Por el contrario, no se detectó ningún pico de dimerización en ausencia de CBD(Figura 8A,línea azul) o cuando cada aglutinante se cargaba solo en la columna(Figura 8B). El reticulado químico se utilizó para estabilizar los complejos CID, y los nanocuerpos reticulados tienen tamaños ligeramente mayores correspondientes a picos eluyed anteriores.
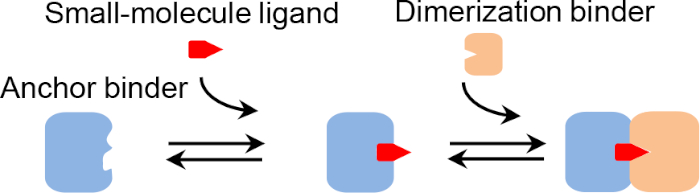
Figura 1: Mecanismo de dimerización de proteínas inducida químicamente. Haga clic aquí para ver una versión más grande de esta figura.
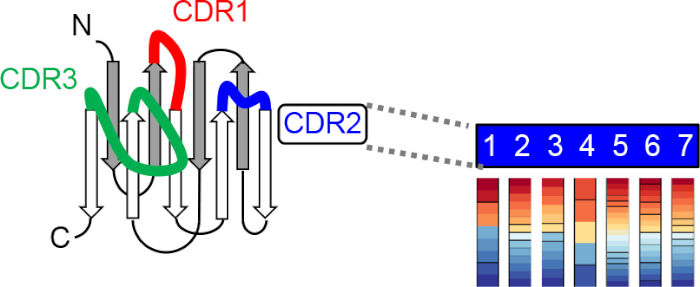
Figura 2: Esquema de la generación de una biblioteca combinatoria sintética nanobody. La biblioteca se construye utilizando un andamio nanobody universal e incorporando distribuciones diseñadas de aminoácidos a cada posición de aleatorización en tres regiones determinantes de complementariedad (CDR) mediante una tecnología trinucleótide Mutagenesis (TRIM) 24. Haga clic aquí para ver una versión más grande de esta figura.
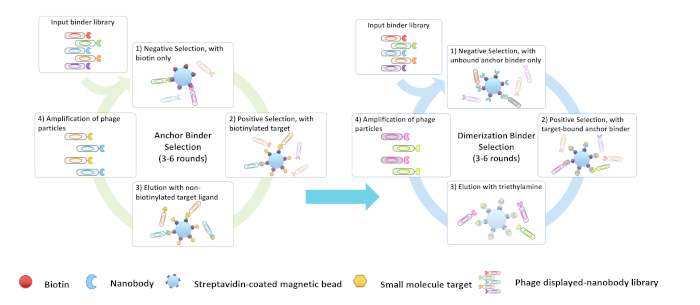
Figura 3: Diagrama de flujo de (A) anclaje y (B) cribado de aglutinante de dimerización. Haga clic aquí para ver una versión más grande de esta figura.
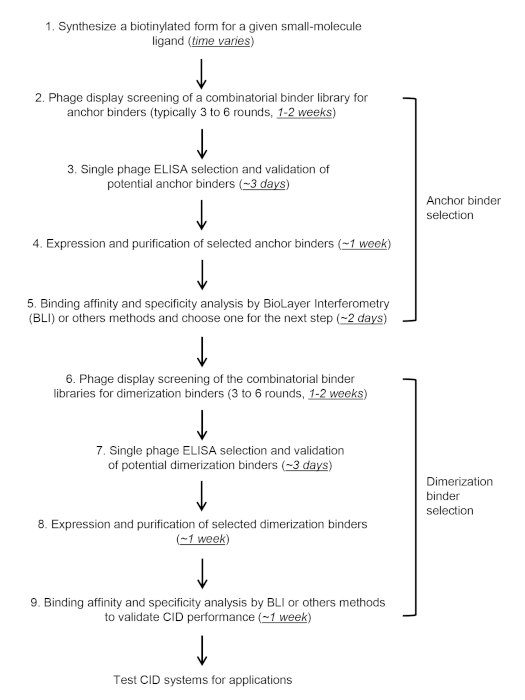
Figura 4: Línea de tiempo de COMBINES-CID. Haga clic aquí para ver una versión más grande de esta figura.
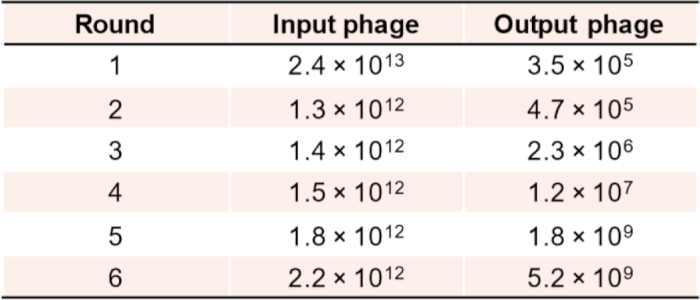
Figura 5: Enriquecimiento de los lanzadores de fagos después de cada ronda de biopanning para la selección de aglutinante de anclaje. Haga clic aquí para ver una versión más grande de esta figura.
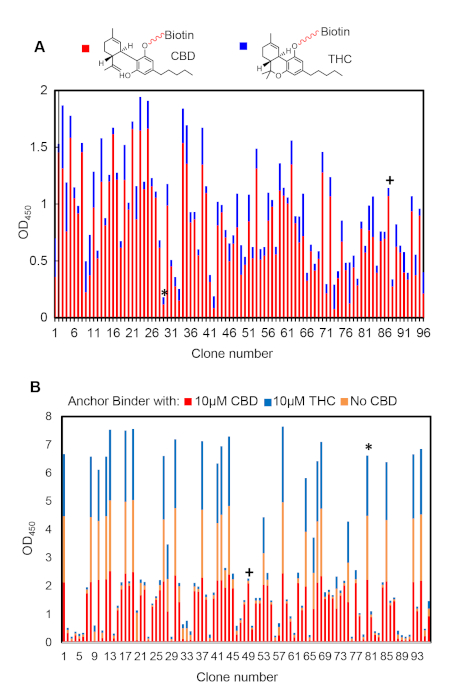
Figura 6: Resultados representativos de ELISA que muestran clones positivos (+) y negativos (*) . (A) El anclarón ELISA resulta de 96 clones seleccionados al azar después de seis rondas de selección. (B) Resultados el ELISA de carpeta de dimerización de 96 clones seleccionados al azar después de cuatro rondas de selección. Haga clic aquí para ver una versión más grande de esta figura.
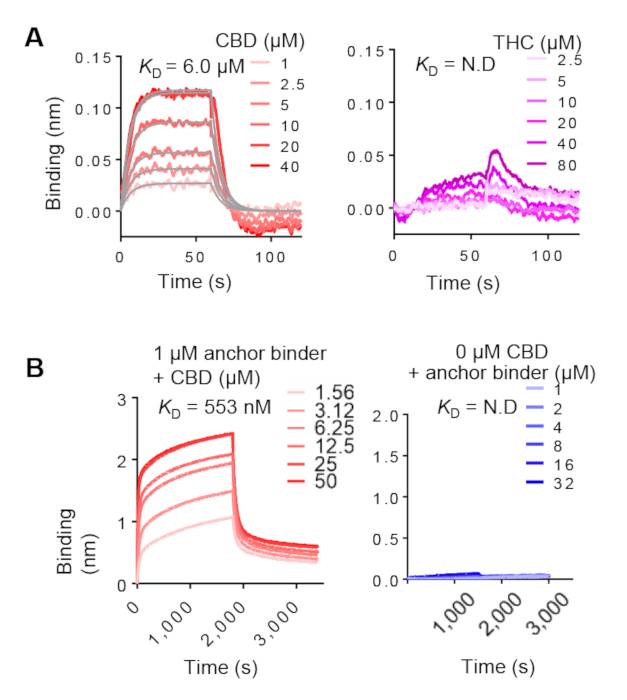
Figura 7: Análisis cinético de anclar y dimerización de BLI. (A) Análisis del aglutinante de anclaje con CBD sin etiquetar (izquierda) y THC (derecha). El aglutinante de anclaje biotinylated se inmovilizó en biosensores Super Streptavidin (SSA) valorados con diferentes concentraciones de CBD. Los datos medidos para la unión al CDB (curvas rojas) se instalaron globalmente (líneas grises). (B) Izquierda, análisis BLI de una unión de aglutinante de dimerización biosensor-inmovilizada SA al aglutinante de anclaje preequilibrado con diferentes concentraciones de CBD. Correcto, la concentración del aglutinante de anclaje fue valorada y unida al aglutinante de dimerización inmovilizada en ausencia de CBD. Haga clic aquí para ver una versión más grande de esta figura.
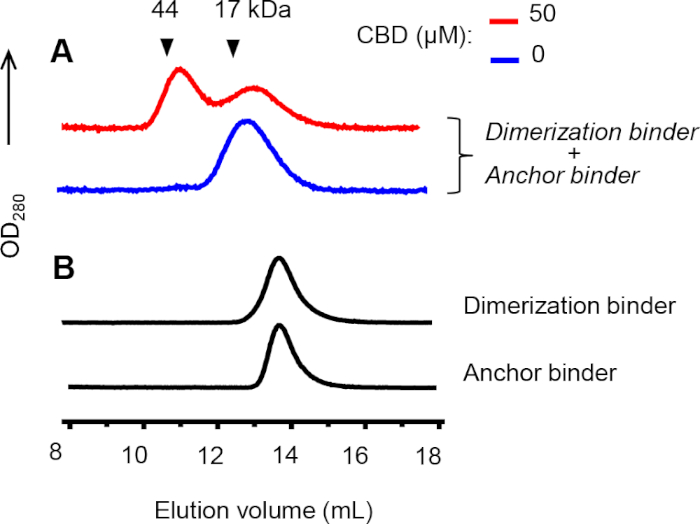
Figura 8: Análisis SEC de la heterodimerización entre el ancla y los aglutinantes de dimerización. (A) Los aglutinantes de dimerización y anclaje (5 m cada uno) en presencia o ausencia de CBD fueron reticulados por 100 éM bis-N-succinimidil-pentametilo) éster durante 30 minutos a RT antes del análisis. Los volúmenes de elución de los estándares proteicos están marcados por triángulos. (B) Los aglutinantes de anclaje y dimerización no reticulados (30 m cada uno) se inyectaron por separado. Los cromatogramas en A y B se muestran en diferentes escalas Y. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Es fundamental elegir las concentraciones correctas de las bibliotecas de fagos de entrada para diferentes rondas de bioparnning. Por lo general, comenzamos a partir de una biblioteca de entrada de 10a 12–10 13 partículas de fago con una diversidad >109,lo que permite que se presenten en el ensayo desplegable las copias de cada clon de fago. Si la concentración de fago en un ensayo de unión es demasiado alta o baja, la probabilidad de unión inespecífica o pérdida de clones positivos aumentará. La selección del aglutinante de anclaje o dimerización normalmente consta de tres a seis rondas de bioparnning, y los recuentos de fagos de salida generalmente comienzan a partir de 104 y aumentan a 108–109. Es adecuado elegir clones individuales para la validación de ELISA después de observar dicho enriquecimiento. Las rondas adicionales de biopanning podrían disminuir las posibilidades de identificar clones positivos adecuados de baja abundancia.
Es importante configurar controles y selecciones negativos adecuados para mejorar el éxito de las selecciones. Por ejemplo, el uso de análogos estructurales de objetivos de ligando facilitará la selección de la especificidad del ligando. En nuestro trabajo, un análogo muy similar, THC, se utilizó como control para el CBD en la validación ELISA y BLI de aglutinantes de ancla y dimerización19. En la selección de aglutinantes de dimerización, si los aglutinantes de anclaje tienen una afinidad de unión de ligando relativamente baja, los aglutinantes de anclaje libres y ligandos se pueden presentar como objetivos en la selección positiva. Por lo tanto, es importante eliminar a fondo los aglutinantes que se unen a los aglutinantes de anclaje libre durante la selección negativa. Esto se puede lograr mediante la realización de múltiples rondas de la resta con aglutinantes de anclaje libre.
Una limitación de nuestro protocolo es que las moléculas objetivo necesitan ser biotinyladas para la selección de aglutinantes de anclaje y sólo uno o unos pocos aglutinantes de anclaje se pueden utilizar para la selección de dimerización. El uso de objetivos biotinylados puede enriquecer aglutinantes que se unen en parte al vinculador entre la biotina y los objetivos. Por lo tanto, es importante validar los hits utilizando objetivos sin etiquetar por BLI u otras técnicas. La elección de uno o unos pocos aglutinantes de anclaje para la selección de aglutinantes de dimerización puede disminuir la posibilidad de identificar sistemas CID con una sensibilidad y especificidad adecuadas. Por lo tanto, la capacidad de multiplexación de la selección espera una mejora adicional mediante el acoplamiento a otras técnicas, por ejemplo, la secuenciación de interacción molecular única (SMI-Seq) que permite un cribado de interacción proteína-proteína "biblioteca por biblioteca"25.
Divulgaciones
La Universidad de Washington ha presentado una patente provisional relacionada con esta obra.
Agradecimientos
Este trabajo fue apoyado por el Premio a la Innovación de la Universidad de Washington (a L.G.), una subvención de los Institutos Nacionales de Salud de los Estados Unidos (1R35GM128918 a L.G.), y un fondo de inicio de la Universidad de Washington (a L.G.). H.J. fue apoyado por una beca de pregrado de la Washington Research Foundation. K.W. fue apoyado por una beca de pregrado del Instituto de Diseño de Proteínas de la Universidad de Washington.
Materiales
| Name | Company | Catalog Number | Comments |
| 1-Step Ultra TMB ELISA substrate solution | Thermo Fisher Scientific | 34029 | |
| Agar | Thermo Fisher Scientific | BP1423-2 | |
| Amicon Ultra-15 Centrifugal Filter unit (3 kDa cutoff) | Millipore | UFC900324 | |
| Ampicillin | Thermo Fisher Scientific | BP1760-25 | |
| Bio-Rad Protein Assay Kit II | Bio-Rad | 5000002 | |
| BirA biotin-protein ligase standard reaction kit | Avidity | BirA500 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A2153-50G | |
| Casein | Sigma-Aldrich | C7078-1KG | |
| CM13 Helper phage | Antibody Design Labs | PH020L | |
| D-(+)-Glucose monohydrate | Alfa Aesar | A11090 | |
| Dynabeads M-280 Streptavidin | Thermo Fisher Scientific | 11205D | |
| DynaMag-2 Magnet | Thermo Fisher Scientific | 12321D | |
| EDTA | Thermo Fisher Scientific | BP120-1 | |
| Fast DNA Ladder | New England Biolabs | N3238S | |
| FastDigest BglI | Thermo Fisher Scientific | FD0074 | |
| Glycerol | Thermo Fisher Scientific | BP229-1 | |
| HiLoad 16/600 Superdex 200 pg | GE Healthcare | 28989335 | |
| HiPrep 26/10 Desalting Column | GE Healthcare | 17508701 | |
| HisTrap-FF-1ml | GE Healthcare | 11000458 | |
| Imidazole | Alfa Aesar | 161-0718 | |
| IPTG | Thermo Fisher Scientific | 34060 | |
| Kanamycin | Thermo Fisher Scientific | BP906-5 | |
| M13 Major Coat Protein Antibody | Santa Cruz Biotechnology | sc-53004 | |
| NaCl | Sigma-Aldrich | S3014-500G | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Fisher Scientific | ND-2000 | |
| Nunc 96-Well Polypropylene DeepWell Storage Plates | Thermo Fisher Scientific | 260251 | |
| Nunc MaxiSorp | Thermo Fisher Scientific | 44-2404-21 | |
| Octet RED96 | ForteBio | N/A | |
| pADL-23c Phagemid Vector | Antibody Design Labs | PD0111 | |
| PEG-6000 | Sigma-Aldrich | 81260-1KG | |
| Platinum SuperFi DNA Polymerase | Invitrogen | 12351010 | |
| PureLink PCR Purification Kit | Thermo Fisher Scientific | K310001 | |
| QIAprep Spin M13 Kit | Qiagen | 27704 | |
| Recovery Medium | Lucigen | 80026-1 | |
| SpectraMax Plus 384 | Molecular Devices | N/A | |
| Sucrose | Sigma-Aldrich | S0389-1KG | |
| Super Streptavidin (SSA) Biosensors | ForteBio | 18-5057 | |
| Superdex 75 increase 10/300 GL Column | GE Healthcare | 28-9909-44 | |
| T4 DNA Ligase | Thermo Fisher Scientific | 15224-025 | |
| TG1 Electrocompetent Cells | Lucigen | 60502-1 | |
| Triethylamine | Sigma-Aldrich | 471283-100mL | |
| Trizma Base | Sigma-Aldrich | T1503 | |
| Tryptone | Thermo Fisher Scientific | BP9726-5 | |
| Tween 20 | Thermo Fisher Scientific | BP337-500 | |
| Yeast Extract | Thermo Fisher Scientific | BP1422-2 | |
| Zeba Spin Desalting Column | Thermo Fisher Scientific | 89882 |
Referencias
- Stanton, B. Z., Chory, E. J., Crabtree, G. R. Chemically induced proximity in biology and medicine. Science. 359 (6380), (2018).
- Wu, C. Y., Roybal, K. T., Puchner, E. M., Onuffer, J., Lim, W. A. Remote control of therapeutic T cells through a small molecule-gated chimeric receptor. Science. 350 (6258), (2015).
- Straathof, K. C., et al. An inducible caspase 9 safety switch for T-cell therapy. Blood. 105 (11), 4247-4254 (2005).
- Di Stasi, A., et al. Inducible apoptosis as a safety switch for adoptive cell therapy. The New England Journal of Medicine. 365 (18), 1673-1683 (2011).
- Mank, M., et al. A FRET-based calcium biosensor with fast signal kinetics and high fluorescence change. Biophysical Journal. 90 (5), 1790-1796 (2006).
- Nagai, T., Sawano, A., Park, E. S., Miyawaki, A. Circularly permuted green fluorescent proteins engineered to sense Ca2+. Proceedings of the National Academy of Sciences of the United States of America. 98 (6), 3197-3202 (2001).
- Hunter, M. M., Margolies, M. N., Ju, A., Haber, E. High-affinity monoclonal antibodies to the cardiac glycoside, digoxin. Journal of Immunology. 129 (3), 1165-1172 (1982).
- Bradbury, A. R. M., Sidhu, S., Dubel, S., McCafferty, J. Beyond natural antibodies: the power of in vitro display technologies. Nature Biotechnology. 29 (3), 245-254 (2011).
- Chen, G., et al. Isolation of high-affinity ligand-binding proteins by periplasmic expression with cytometric screening (PECS). Nature. Biotechnology. 19 (6), 537-542 (2001).
- Tinberg, C. E., et al. Computational design of ligand-binding proteins with high affinity and selectivity. Nature. 501 (7466), 212-216 (2013).
- Spencer, D. M., Wandless, T. J., Schreiber, S. L., Crabtree, G. R. Controlling signal transduction with synthetic ligands. Science. 262 (5136), 1019-1024 (1993).
- Ho, S. N., Biggar, S. R., Spencer, D. M., Schreiber, S. L., Crabtree, G. R. Dimeric ligands define a role for transcriptional activation domains in reinitiation. Nature. 382 (6594), 822-826 (1996).
- Belshaw, P. J., Ho, S. N., Crabtree, G. R., Schreiber, S. L. Controlling protein association and subcellular localization with a synthetic ligand that induces heterodimerization of proteins. Proceedings of the National Academy of Sciences of the United States of America. 93 (10), 4604-4607 (1996).
- Farrar, M. A., AlberolaIla, J., Perlmutter, R. M. Activation of the Raf-1 kinase cascade by coumermycin-induced dimerization. Nature. 383 (6596), 178-181 (1996).
- Erhart, D., et al. Chemical Development of Intracellular Protein Heterodimerizers. Chemistry & Biology. 20 (4), 549-557 (2013).
- Ballister, E. R., Aonbangkhen, C., Mayo, A. M., Lampson, M. A., Chenoweth, D. M. Localized light-induced protein dimerization in living cells using a photocaged dimerizer. Nature Communications. 17 (5), 5475(2014).
- Hill, Z. B., Martinko, A. J., Nguyen, D. P., Wells, J. A. Human antibody-based chemically induced dimerizers for cell therapeutic applications. Nature Chemical Biology. 14 (2), 112-117 (2018).
- Foight, G. W., et al. Multi-input chemical control of protein dimerization for programming graded cellular responses. Nature Biotechnology. 37 (10), 1209-1216 (2019).
- Kang, S., et al. COMBINES-CID: An efficient method for de novo engineering of highly specific chemically induced protein dimerization systems. Journal of the American Chemical Society. 141 (28), 10948-10952 (2019).
- Muyldermans, S. Nanobodies: natural single-domain antibodies. Annual Review of Biochemistry. 82, 775-797 (2013).
- Fanning, S. W., Horn, J. R. An anti-hapten camelid antibody reveals a cryptic binding site with significant energetic contributions from a nonhypervariable loop. Protein Science. 20 (7), 1196-1207 (2011).
- Zavrtanik, U., Luken, J., Loris, R., Lah, J., Hadzi, S. Structural basis of epitope recognition by heavy-chain camelid antibodies. Journal of Molecular Biology. 430 (21), 4369-4386 (2018).
- Denhardt, D. T., Dressler, D., Ray, D. S. The Single-Stranded DNA Phages. , 605-625 (1978).
- Virnekas, B., et al. Trinucleotide phosphoramidites: ideal reagents for the synthesis of mixed oligonucleotides for random mutagenesis. Nucleic Acids Research. 22 (25), 5600-5607 (1994).
- Gu, L., et al. Multiplex single-molecule interaction profiling of DNA-barcoded proteins. Nature. 515 (7528), 554-557 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados