Method Article
Erstellen hochspezifischer chemisch induzierter Proteindimerisierungssysteme durch schrittweise Phage-Auswahl einer kombinatorischen Single-Domain-Antikörperbibliothek
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Die Schaffung chemisch induzierter Proteindimerisierungssysteme mit gewünschter Affinität und Spezifität für einen bestimmten kleinen Molekülligand hätte viele biologische Sensor- und Betätigungsanwendungen. Hier beschreiben wir eine effiziente, verallgemeinerbare Methode zur de novo Engineering von chemisch induzierten Dimerisierungssystemen durch die schrittweise Auswahl einer phagenangezeigten kombinatorischen Ein-Domänen-Antikörperbibliothek.
Zusammenfassung
Protein-Dimerisierungsereignisse, die nur in Gegenwart eines kleinmolekularen Ligands auftreten, ermöglichen die Entwicklung von kleinmolekularen Biosensoren zur Zerlegung und Manipulation biologischer Bahnen. Derzeit gibt es nur eine begrenzte Anzahl chemisch induzierter Dimerisierungssysteme (CID) und neue Systeme mit gewünschter Empfindlichkeit und Selektivität für bestimmte kleinmolekulare Liganden zu erschaffen, bleibt eine Herausforderung im Bereich der Proteintechnik. Wir beschreiben hier ein Verfahren zum Screening mit hohem Durchsatz, kombinatorial binders-enabled sElection von CID (COMBINES-CID), für die de novo Engineering von CID-Systemen, die auf eine Vielzahl von Liganden anwendbar sind. Diese Methode verwendet die zweistufige Auswahl einer phagenangezeigten kombinatorischen Nanokörperbibliothek, um 1) "Ankerbinder" zu erhalten, die zuerst an einen Voninteresse gebundenen Liganden und dann an 2) "Dimerisierungsbinder" binden, die nur an Ankerbinder-Ligand-Komplexe binden. Zur Auswahl von Ankerbindern wird eine kombinatorische Bibliothek mit über 109 komplementär-bestimmenden Nanokörpern (CDR)-randomisierten Nanokörpern mit einem biotinylierten Liganden abgeschirmt und Treffer mit dem unbeschrifteten Liganden durch Bioschichtinterferometrie (BLI) validiert. Um Dimerisierungsbindemittel zu erhalten, wird die Nanokörperbibliothek mit Ankerbinder-Ligand-Komplexen als Ziel für positives Screening und den ungebundenen Ankerbindern für negatives Screening abgeschirmt. COMBINES-CID ist allgemein anwendbar auf ausgewählte CID-Bindemittel mit anderen Immunglobulin,Non-Immunoglobulin oder computerisch konstruierten Gerüsten, um Biosensoren für den In-vitro- und In-vivo-Nachweis von Medikamenten, Metaboliten, Signalmolekülen usw. zu erstellen.
Einleitung
CID-Systeme, bei denen zwei Proteine nur in Gegenwart eines kleinmolekularen Liganden dimerisieren (Abbildung 1), bieten vielseitige Werkzeuge zum Sezieren und Manipulieren von Stoffwechsel-, Signal- und anderen biologischen Wegen1. Sie haben das Potenzial in der biologischen Betätigung, wie die medikamentöse T-Zell-Aktivierung2 und Apoptose3,4, zur Verbesserung der Sicherheit und Wirksamkeit der Adoptiv-T-Zelltherapie gezeigt. Darüber hinaus bieten sie eine neue Methodik für den In-vivo- oder In-vitro-Nachweis von Kleinmolekülzielen. Beispielsweise können CID-Proteine genetisch mit Fluoreszenzreportersystemen (z. B. Fluoreszenzresonanz-Energietransfer (FRET)5 und kreisförmig permutierten fluoreszierenden Proteinen)6 für Echtzeit-In-vivo-Messungen verschmolzen werden oder als Affinitätsreagenzien für Sandwich-Enzym-linked Immunosorbent Assay (ELISA)-ähnliche Assays dienen.
Trotz ihrer breiten Anwendungen stellt die Schaffung neuer CID-Systeme, die von einem bestimmten kleinmolekularen Liganden gesteuert werden können, große Herausforderungen. Etablierte Proteinbinder-Engineering-Methoden einschließlich Tierimmunisierung7, In-vitro-Auswahl8,9und computergestütztes Proteindesign10 können Liganden-Bindungsproteine erzeugen, die über binäre Protein-Liganden-Wechselwirkungen funktionieren. Diese Methoden haben jedoch Schwierigkeiten, einen Liganden-induzierten ternären CID-Komplex zu schaffen. Einige Methoden erzeugen CID, indem sie zwei Liganden chemisch verbinden, die unabhängig voneinander an die gleichen oder unterschiedlichen Proteinebinden 11,12,13,14,15,16 oder sich auf die Auswahl von Bindemittelproteinen wie Antikörpern konzentrieren, die auf bereits bestehende kleine Molekül-Protein-Komplexe abzielen17,18, und somit eine begrenzte Auswahl an Liganden haben.
Wir haben vor kurzem eine combinatorial binders-enabled sWahl der CID (COMBINES-CID) Methode für de novo Engineering von CID-Systemenentwickelt 19. Diese Methode kann die hohe Spezifität der Liganden-induzierten Dimerisierung erhalten (z. B. eine Anker-Dimerisierungsbinder-Dissoziationskonstante, KD (ohne Ligand)/KD (mit Ligand) > 1.000). Die Dimerisierungsssspezifität wird mit Ankerbindern mit flexiblen Bindestellen erreicht, die konforme Veränderungen bei der Ligandenbindung mit sich bringen können, was eine Grundlage für die Auswahl von konform selektiven Bindemitteln bietet, die nur ligaandgebundene Ankerbinder erkennen. Wir demonstrierten einen Proof-of-Prinzip durch die Schaffung von Cannabidiol (CBD)-induzierten Heterodimeren von Nanokörpern, einem 12–15 kDa-Funktionsantikörperfragment aus Kamelid, das ein universelles Gerüst und drei flexible CDR-Schleifen (Abbildung 2)20enthält, die eine Bindungstasche mit anpassungsfähigen Größen für kleinmolekulare Epitope21,22bilden können. Insbesondere sollte die In-vitro-Auswahl einer kombinatorischen Proteinbibliothek kostengünstig und verallgemeinerbar für CID-Engineering sein, da die gleiche hochwertige Bibliothek auf verschiedene Liganden angewendet werden kann.
In diesem Protokoll und Video konzentrieren wir uns auf die Beschreibung der zweistufigen In-vitro-Auswahl und Validierung von Anker- (Abbildung 3A) und Dimerisierungsbindern (Abbildung 3B) durch Screening der kombinatorischen Nanokörperbibliothek mit einer Vielfalt von mehr als 109 unter Verwendung von CBD als Ziel, aber das Protokoll sollte auf andere Proteinbibliotheken oder Kleinmolekülziele anwendbar sein. Das Screening von CID-Bindemitteln dauert in der Regel 6 bis 10 Wochen (Abbildung 4).
Protokoll
1. Bibliotheksbau
- Verwenden Sie eine synthetische kombinatorische Single-Domain-Antikörperbibliothek mit einer Vielfalt von 1,23–7,14 x 109, wie zuvor beschrieben19. Dieses Protokoll enthält zwar keine Bibliothekserstellung, kann aber auch auf andere kombinatorische Binderbibliotheken angewendet werden.
2. Biotinylierung von Ligandenziel oder Liganden
- Biotinylatieren Sie den ausgewählten Liganden, z.B. CBD und Tetrahydrocannabinol (THC)19, über verschiedene chemische Synthesestrategien, abhängig von den geeigneten Biotinylierungsstellen eines Ziels.
3. Ankerbinder-Screening
- Beginn der Auswahl
- Beginnen Sie jede Auswahlrunde, indem Sie eine einzelne TG1-Zellkolonie impfen, die in 6 ml 2YT bei 37 °C und 250 Umdrehungen pro Minute (Rpm) frisch angebaut wird, bis zu einer Absorption von 600 nm (OD600) von 0,5 USD. Inkubieren Sie die Zellen auf Eis für den Einsatz in Schritt 3.5.1.
- Negative Selektion mit biotingebundenen Streptavidinperlen
- Bereiten Sie die "negativen Selektionsperlen" vor, indem Sie 300 L streptavidin-beschichtete Magnetperlen mit einem magnetischen Trennträger, 3x mit 0,05% phosphatgepufferter Saline mit Tween-Puffer (PBST, 1 x PBS mit 0,05% vol/vol Tween 20%) waschen. und 2x mit 1 x PBS.
- Setzen Sie die Perlen mit 1 ml 1% Kasein in 1 x PBS (pH = 7,4) aus und sättigen Sie die Perlen, indem Sie die gemeldete Bindungskapazität mit Biotinum das 5-fache sättigen. Bei Raumtemperatur (RT) auf einem Rotator für 1 h inkubieren.
- Waschen Sie die Perlen 5x mit 0,05% PBST und 3x mit 1 x PBS, für insgesamt acht Waschungen.
- Fügen Sie 1013 Phagenpartikel in 1% Kasein/1% BSA in 1 x PBS (pH = 7,4) hinzu und brüten bei RT auf einem Rotator für 1 h.
- Sammeln Sie nach der Inkubation den Instand, der in Schritt 3.3.6 verwendet werden soll.
- Positive Auswahl mit biotinylierten Liganden-gebundenen Streptavidinperlen
- Bereiten Sie die "positiven Auswahlperlen" mit 1/2 des Volumens der Perlen vor, die für die "negativen Auswahlperlen" verwendet werden, und folgen Sie den Schritten 3.2.1.
- Die Perlen mit 1 ml 1% Kasein in 1 °C, pH 7,4 wieder aufhängen und die Perlen sättigen, indem Sie die auf der Grundlage des Handbuchs berechnete volle Bindungskapazität mit dem biotinylierten Liganden ihrer Wahl hinzufügen. Inkubieren Sie bei RT auf einem Rotator für 1 h.
- Waschen Sie die Perlen 5x mit 0,05% PBST und 3x mit 1 x PBS, für insgesamt acht Waschungen.
- Die Perlen mit 1 ml 1% Kasein/1% BSA in 1 x PBS (pH = 7,4) blockieren und bei RT auf einem Rotator für 1 h brüten, um eine unspezifische Bindung zwischen den Phagen und den streptavidin-beschichteten Magnetperlen zu verhindern.
- Waschen Sie die mit Streptavidin beschichteten Magnetperlen 3x mit 0,05% PBST und einmal mit 1 x PBS für insgesamt vier Waschungen.
- Setzen Sie die mit Streptavidin beschichteten Magnetperlen mit den ungebundenen Phagen aus Schritt 3.2.5 wieder auf und brüten Sie bei RT auf einem Rotator für 1 h.
- Extrahieren Sie den Überstand, ohne die magnetischen Perlen zu stören. Speichern Sie die ungebundenen Phagen als Eingabe, die in Schritt 3.5.1 verwendet werden soll.
- Waschen Sie die Perlen 10x mit 0,05% PBST und 5x mit 1 x PBS. Zwischen durch alle drei Wärer werden sie in ein neues Rohr überführen, um Phagen zu vermeiden, die nicht spezifisch an die Rohrwände gebunden sind.
- Elution von phagenangezeigten Nanokörpern
- Wettbewerbsfähige gebundene Phagen durch Zugabe von 450 l des nicht-biotinylierten Liganden,unter Verwendung einer Konzentration im Mikromolarbereich (z. B. 10–50 m) und Inkubation bei RT auf einem Rotator für 30 min. Die gewählte Ligandenkonzentration für die wettbewerbsfähige Elution von gebundenen Phagen ist abhängig vom gewünschten KD des "Ankerbinders". Die Ligandkonzentrationen können in den ersten Auswahlrunden relativ hoch sein und dann in späteren Runden abgenommen haben.
- Sammeln Sie Überstand und speichern Sie die eluierten Phagen als Ausgabe, in Schritt 3.5.2 verwendet werden.
- Input/Output Titrationen und Infektionen
- Für die Eingangstitration 10x serielle Verdünnungen in 1 x PBS bis zu 109-fachmit dem Eingangsphagen ab Schritt 3.3.7 vorbereiten. Verwenden Sie die 107-109 seriellen Verdünnungen, um Infektionen zu begehen, indem Sie 10 l Eingangsphagen von jeder Verdünnung auf 70 -L TG1-Zellen übertragen (OD600 von 0,5). Bei 37 °C für 45 min aufbuchen, die infizierten TG1-Zellen auf drei 90 mm 2YT-Agar-Gerichte mit 100 g/ml Ampicillin und 2% (wt/vol) Glukose aufsieben und über Nacht bei 37 °C inkubieren. Aus den Nachttafeln lässt sich der Phageneingang wie folgt berechnen:

- Übertragen Sie bei Ausgangsinfektionen und Titration die eluierten Phagen von Schritt 3,4,2 auf 3 ml TG1-Zellen (OD600 von 0,5). Inkubieren Sie in einem Wasserbad bei 37 °C für 45 min. Dann 10x serielle Verdünnungen in 2YT bis 103-fachzubereiten, jede Verdünnung auf 90 mm 2YT-Agar-Gerichte verdünnen und bei 37 °C über Nacht bebrüten. Aus den Nachtplatten lässt sich die Phagenleistung wie folgt berechnen:

- Teilen Sie die verbleibenden infizierten TG1-Zellen auf drei 150 mm 2YT-Agar-Platten, die 100 g/ml Ampicillin und 2% (wt/vol) Glucose enthalten. Teller über Nacht bei 37 °C inkubieren.
- Für die Eingangstitration 10x serielle Verdünnungen in 1 x PBS bis zu 109-fachmit dem Eingangsphagen ab Schritt 3.3.7 vorbereiten. Verwenden Sie die 107-109 seriellen Verdünnungen, um Infektionen zu begehen, indem Sie 10 l Eingangsphagen von jeder Verdünnung auf 70 -L TG1-Zellen übertragen (OD600 von 0,5). Bei 37 °C für 45 min aufbuchen, die infizierten TG1-Zellen auf drei 90 mm 2YT-Agar-Gerichte mit 100 g/ml Ampicillin und 2% (wt/vol) Glukose aufsieben und über Nacht bei 37 °C inkubieren. Aus den Nachttafeln lässt sich der Phageneingang wie folgt berechnen:
- Bibliotheksverstärkung und -wiederherstellung für weitere Auswahlrunden
- 3 ml 2YT pro Platte hinzufügen, mit einem sterilen Zellschaber kratzen und alle Zellen in einem 50 ml konischen Rohr sammeln. Mischen Sie die gesammelten Zellen mit sterilem Glycerin (20% wt/vol Endkonzentration). Messen Sie die OD600 der Mischung und machen Sie 3-5 Stock Aliquots. Bei -80 °C für langzeitbereite Lagerung lagern.
- Bei der Phagenrettung das phagenmidhaltige TG1-Bakteriengemisch mit 25 ml 2YT-Medien, ergänzt mit 2% Glukose und 100 g/ml Ampicillin, auf eine OD600 von 0,1 verdünnen. Kulturzellen bei 37 °C und 250 Rpm bis zu einem OD600 von 0,5 .
- Überinfizieren Sie die Zellen durch Zugabe von CM13-Helferphagen bei 5 x 109 pfu/mL und inkubieren Sie bei 37 °C und 250 U/min für 45 min. Der CM13 Helferphagen liefert benötigte Phagenschichtproteine für die Montage kompletter Phagenpartikel.
- Zentrifugieren Sie die Kultur bei 8.000 x g für 10 min, um die Glukose zu entfernen. Setzen Sie die Zellen mit 50 ml 2YT-Medien, die mit 100 g/ml Ampicillin und 50 g/ml Kanamycin ergänzt werden, wieder ab und brüten Sie über Nacht bei 25 °C und 250 Umdrehungen pro Minute.
- Zentrifugieren Sie die Zellen aus der Nachtkultur bei 9.000 x g, 4 °C für 30 min. Übertragen Sie den Überstand in ein neues Rohr und die Niederschlagsphagen im Überstand mit 1/5 Volumen PEG/NaCl Lösung (20% gew/vol Polyethylenglykol-6.000 und 2,5 M NaCl). Sanft mischen und 1 h auf Eis legen.
- Sammeln Sie Phagenpartikel durch Zentrifugation mit 12.000 x g bei 4 °C für 30 min. Setzen Sie die Pellets mit 1 ml 1 x PBS ab und übertragen Sie die Suspension in ein Mikrozentrifugenrohr. Zentrifugieren Sie das Rohr bei 20.000 x g und 4 °C für 10 min, um Restbakterien zu entfernen.
- Übertragen Sie den Überstand auf ein neues Mikrozentrifugenrohr, ohne das bakterielle Pellet zu stören. Verwenden Sie eine Verdünnung von 1:100, um die Absorption bei 269 nm und 320 nm zu messen. Die Gesamtzahl der Phagen kann nach folgender Formel23berechnet werden:

- Phagenbibliothek bei 4 °C für den kurzfristigen Gebrauch oder mit 25% Glycerin bei -80 °C für die Langzeitlagerung lagern.
- Wiederholen Sie Auswahlrunden (Schritte 3.1–3.6) für 3–6 Runden oder bis die gewünschte Anreicherung beobachtet wird (siehe Ergebnisabschnitt). Platten und Pick-Einzelklone (Abschnitt 4), um ihre Affinität und Spezifität zum Liganden zu charakterisieren (Abschnitte 5–7).
4. Single-Klon-Isolation
- Um einzelne Klone aus einer angereicherten Unterbibliothek zu isolieren, bereiten Sie 10x serielle Verdünnungen der phageninfizierten TG1-Zellen vor (Schritt 3.5.2). Platte serielle Verdünnungen auf 90 mm 2YT-Agar-Schalen mit 100 g/ml Ampicillin und 2% (wt/vol) Glukose und inkubieren bei 37 °C über Nacht.
- Von den Nachtplatten aus pflücken Sie einzelne Kolonien in 250 L 2YT-Medien, ergänzt mit 100 g/ml Ampicillin pro Brunnen in sterilen Tiefbrunnenplatten und wachsen bei 37 °C über Nacht.
- Aus den Nachtkulturen impfen Sie 10 l in 500 l frische 2YT-Medien, die mit 100 g/ml Ampicillin ergänzt werden.
- Wachsen Sie Zellen zu einem OD600 von 0,5, fügen Sie CM13 Helfer-Phagen bei 5 x 109 pfu/mL und inkubieren bei 37 °C und 250 U/min für 45 min.
- Fügen Sie 500 l 2YT-Medien hinzu, die mit 100 g/ml Ampicillin und 50 g/ml Kanamycin ergänzt werden. Bei 25 °C und 250 Umdrehungen von 150 M/min über Nacht inkubieren.
- Zentrifugieren Sie die Tiefbrunnenplatten aus den Nachtkulturen bei 3.000 x g für 10 min. Sammeln Sie den Überstand, der die Phagenpartikel enthält, ohne das Zellpellet zu stören.
- Phage-Partikel können für ELISA verwendet werden, um die Spezifität der ausgewählten Klone für den Liganden zu bestimmen. Biotin oder ein struktureller Homolog des Ziels kann als Negativkontrolle verwendet werden.
5. Ankerbindervalidierung durch ELISA
- 96 Well-ELISA-Platten mit 100 l von 5 g/ml Streptavidin im Beschichtungspuffer (100 mM Karbonatpuffer, pH = 8,6) bei 4 °C über Nacht schichten.
- Waschen Sie die ELISA-Platten 3x mit 0,05% PBST und fügen Sie 100 l von 1 'M biotinyliertes Ziel zu den Zielbrunnen hinzu. Fügen Sie den Kontrollbrunnen 100 L mit 1 M Biotin oder Zielhomolog hinzu. Bei RT für 1 h inkubieren.
- Waschplatten 5x mit 0,05% PBST und blocken unspezifische Bindung durch Zugabe von 300 l von 1% Casein in 1 x PBS. Bei RT für 1 h inkubieren.
- Waschen Sie die ELISA-Platten 3x mit 0,05%-PBST und fügen Sie den gereinigten Phagenüberstand hinzu. 1 h bei RT inkubieren.
- Waschen Sie die ELISA-Platten 10x mit 0,05% PBST und fügen Sie 100 L Meerrettichperoxidase (HRP)-M13 Hauptschichtproteinantikörper (1:10.000 Verdünnung mit 1 x PBS mit 1% Casein) hinzu. Bei RT für 1 h inkubieren.
- Waschen Sie die ELISA-Platten 3x mit 0,05% PBST und fügen Sie 100 L Tetramethylbenzidin (TMB) Substrat hinzu. Inkubieren Sie 10 min oder bis eine sichtbare Farbänderung beobachtet wird. Beenden Sie die Reaktion, indem Sie 100 l von 1 M HCl hinzufügen. Lesen Sie die Platte bei 450 nm auf einem Spektralphotometer.
- Wählen Sie für die Proteinexpression und -reinigung die Klone aus, die eine hohe Affinität und Spezifität für das Ziel aufweisen (siehe Diskussion).
6. Proteinexpression, Reinigung und Biotinylierung
- Wie bereits berichtet19, subclone ausgewählte Klone aus Abschnitt 5 und Express als C-terminal Avi-tagged und His-tagged Nanobodies.
- Ausgewählte Nanokörper im Periplasma von E. coli WK6-Zellen (typischerweise in 1 L-Kultur) exprimieren, durch osmotischen Schock freisetzen und mit einer Nickel-NTA-Säule reinigen (siehe Materialtabelle).
- Exchange-Puffer mit einer Entsalzungssäule (1 x PBS mit 5% Glycerin; siehe Materialtabelle).
- Biotinylat-Nanokörper mit einem kommerziellen Kit (siehe Materialtabelle) zur weiteren Verwendung.
7. Ankerbindercharakterisierung durch BLI
- Analysieren Sie die Bindungsaffinität und Kinetik ausgewählter Ankerbinder, indem Sie 200 nM biotinylierte Ankerbindemittel auf Streptavidin-Biosensoren (siehe Materialtabelle)mit Bindungstestpuffer (1 x PBS (pH = 7,4), 0,05% Tween 20, 0,2% BSA, 3% Methanol) immobilisieren.
- Berechnen Sie Dissoziationskonstanten (KD) von Ankerbinder-Ligand-Wechselwirkungen durch stationäre Analyse mit Datenanalysesoftware (siehe Materialtabelle). Die erhaltenen KD-Werte reichen in der Regel von ein- bis zweistelligen Mikromolaren.
8. Dimerisierungsbinder-Screening
ANMERKUNG: Das Biopanning-Screening von "Dimerisierungsbindern" ähnelt dem von Ankerbindern, mit Ausnahme von zwei kritischen Schritten: 1) Dimerisierungsbinder werden mit einem ausgewählten biotinylierten Ankerbinder und dem Ankerbinder-Ligand-Komplex für die negative und positive Auswahlen. 2) Während des Elutionsschritts wird 100 mM Triethylamin verwendet, um positiv ausgewählte Phagen zu entkernen, die nur an den Ankerbinder gebunden waren - Ligandenzielkomplex. Die 100 mM Trimethylamin-Lösung (pH = 11,5) wird verwendet, um positive Klone zu entschärten, indem die Proteinwechselwirkungen gestört werden.
- Beginn der Auswahl
- Beginnen Sie jede Auswahlrunde, indem Sie eine einzelne TG1-Zellkolonie, frisch auf einem minimalen Medium angebaut, in 6 ml 2YT bei 37 °C und 250 Rpm bis zu einem OD600 von 0,5 impfen. Inkubieren Sie Zellen auf Eis.
- Entfernung negativ ausgewählter Nanokörper
- Bereiten Sie das "Subtraktionsrohr" vor, indem Sie 400 L streptavidinbeschichtete Magnetperlen verwenden und Schritt 3.2 folgen. Anstatt jedoch mit Biotin zu sättigen, fügen Sie die berechnete volle Bindungskapazität mit dem ausgewählten biotinylierten Ankerbinder hinzu und speichern Sie die ungebundenen Phagen, die in Schritt 8.3.3 verwendet werden sollen.
- Auswahl positiv ausgewählter Nanokörper
- Bereiten Sie das "Aufnahmerohr" vor, indem Sie 1/2 das Volumen der mit Streptavidin beschichteten Magnetperlen für das "Subtraktionsrohr" und die folgenden Schritte 3.3.2 bis 3.3.3 verwenden. Anstatt jedoch mit dem biotinylierten Liganden zu sättigen, fügen Sie mit dem ausgewählten biotinylierten Ankerbinderdas Fünffache der berechneten vollen Bindungskapazität hinzu.
- Um den Ankerbinder-Ligand-Komplex für die positive Dimerisierungsbinderauswahl zu bilden, fügen Sie eine ausreichend hohe Konzentration von nicht-biotinyliertem Ligandenhinzu. Dadurch können die meisten streptavidingebundenen Ankerbinder den Ligandengebundenen Komplex bilden.
- Befolgen Sie die Schritte 3.3.3 bis 3.3.8, indem Sie die ungebundenen Phagen aus dem "Subtraktionsrohr" verwenden.
- Elution positiv ausgewählter Nanokörper
- Elute die Phagen an den Ankerbinder-Ligand-Komplex gebunden durch Zugabe von 450 l von 100 mM Triethylamin, und Inkubation bei RT auf einem Rotator für 10 min.
- Sammeln Sie die wettbewerbsfähig eluierten Phagen und folgen Sie den Schritten 3.4.1 bis 3.4.2.
- Weitere Runden der Dimerisierungsbinderauswahl
- Befolgen Sie die Schritte 3.5 und 3.6, um die Bibliothek zu erweitern und wiederherzustellen, um weitere Auswahlrunden durchzuführen. Wiederholen Sie Auswahlrunden für 3–6 Runden oder bis die gewünschte Anreicherung beobachtet wird. Platten und Kommissionieren einzelner Klone (siehe Abschnitt 4), um ihre Affinität und Spezifität zum Ziel zu charakterisieren.
9. Dimerisierungsbindercharakterisierung durch ELISA
- Führen Sie die Schritte in Abschnitt 4 aus, um einzelne Klone für die Charakterisierung über ELISA zu isolieren.
- Um die Affinität von Dimerisierungsbinder-Kandidaten zum Ankerbinder-Ligand-Komplex zu testen, beschichten Sie die ELISA-Zielplatte mit 100 l von 100 nM biotinylierten Ankerbindern. Nach der Inkubation für 1 h, fügen Sie 1 M des Ligandenziels hinzu, um den Ankerbinder-Ligand-Komplex zu bilden.
- Die Steuerplatte sollte allein mit dem biotinylierten Ankerbinder beschichtet werden, um Klone abzuschirmen, die auch an das freie Ankerbinder binden können. 100 l mit 100 nM biotinyliertem Ankerbinder hinzufügen und bei RT für 1 h brüten.
- Folgen Sie den Abschnitten 5.3–5.7.
10. Dimerisierungsbindercharakterisierung durch BLI
- Die Bindungsaffinität und Kinetik von Dimerisierungsbindern für den Ankerbinder-Ligand-Komplex kann analysiert werden, indem biotinylierte Dimerisationsbinder auf Streptavidin (SA) Biosensoren mit dem Bindungstestpuffer immobilisiert und dann mit 1 'M Ankerbinder vordausbagriert mit seriellen Dilutionen des Liganden untersucht werden. Die KD, kaufund kaus den Wechselwirkungen können mit unserer gemeldeten Methode19berechnet werden.
Ergebnisse
Wir beschreiben die zweistufige In-vitro-Auswahl und Validierung von Anker- und Dimerisierungsbindern, indem wir die kombinatorische Nanokörperbibliothek mit einer Vielfalt von mehr als 109 unter Verwendung von CBD als Ziel screeningen. Die Beurteilung der Anreicherung der Phagenbiopanning während der aufeinanderfolgenden Selektionsrunden für Anker- und Dimerisierungsbindemittel ist wichtig. Typische Anreicherungsergebnisse nach 4:6 Auswahlrunden, wie in Abbildung 5 dargestellt, sind ein gutes Indiz dafür, dass es ein hohes Verhältnis potenzieller Treffer in den Unterbibliotheken gibt, so dass weitere Auswahlrunden möglicherweise nicht erforderlich sind.
Single-Clone-ELISA eignet sich zur Analyse der relativen Bindungsaffinität und Selektivität von Anker- und Dimerisierungsbindern. Abbildung 6A ist ein repräsentatives Ankerbinderauswahlergebnis nach sechs Runden Biopanning. Klone mit hoher (z.B. #87) oder niedriger (z.B. #27) Ligandenselektivität können verglichen werden. Hochselektivitätsklone sollten als Ankerbinder-Kandidaten ausgewählt werden. Ebenso zeigt Abbildung 6B die Ergebnisse der Dimerisierungsbinderauswahl nach vier Runden Biopanning. Typischerweise beobachteten wir Klone, die einen Heterodimer mit dem immobilisierten Ankerbinder nur mit dem Liganden bildeten (z.B. #49) oder ohne (z.B. #80). Erstere, die Dimerisierungsssspezifität zeigen, sollten für die weitere Validierung ausgewählt werden.
Ankerbinder ELISA setzt auf die Verwendung des biotinylierten Targets. Daher müssen wir BLI verwenden, um die Bindung an das nicht gekennzeichnete Ziel weiter zu bestätigen. BLI ermöglicht auch die Charakterisierung der Bindungskinetik. Repräsentative BLI-Ergebnisse von Anker- und Dimerisierungsbindern sind in Abbildung 7A bzw. 7Bdargestellt. Die linken Paneele zeigen die ligaden konzentrationsabhängige Bindung, was darauf hindeutet, dass sie für den Bau eines CID-Systems geeignet sind. Die rechten Bedienfelder zeigen die Negativkontrollen an. Die berechneten KD von Anker- und Dimerisierungsbinderwechselwirkungen in Gegenwart des Liganden reichten typischerweise von zweistelligen Nanomolaren bis zu zweistelligen Mikromolaren. Sie können je nach Liganden und der kombinatorischen Bibliothek ihrer Wahl variieren.
Zur Bestätigung der Heterodimerbildung zwischen Anker- und Dimerisierungsbindern wurde eine analytische Größenausschlusschromatographie (SEC) durchgeführt. Ein Dimerisierungsgipfel wurde beobachtet, wenn Anker- und Dimerisierungsbinder und CBD gemischt wurden(Abbildung 8A, rote Linie). Im Gegensatz dazu wurde kein Dimerisierungsspitzeninnungspeak in Abwesenheit von CBD(Abbildung 8A, blaue Linie) oder wenn jeder Bindemittel allein auf die Säule geladen wurde (Abbildung 8B). Die chemische Vernetzung wurde verwendet, um CID-Komplexe zu stabilisieren, und vernetzte Nanokörper haben leicht erhöhte Größen, die früheren eluierten Spitzen entsprechen.
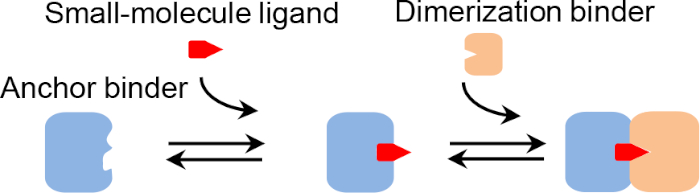
Abbildung 1: Mechanismus der chemisch induzierten Proteindimerisierung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
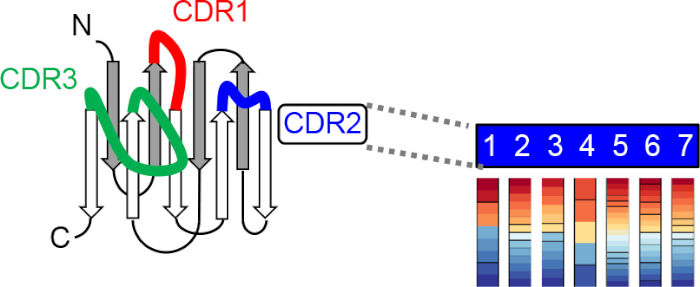
Abbildung 2: Schemat der Erzeugung einer synthetischen Nanokörper-Kombinatorbibliothek. Die Bibliothek wird unter Verwendung eines universellen Nanokörpergerüsts und unter Einbeziehung von designierten Verteilungen von Aminosäuren zu jeder Randomisierungsposition in drei komplementäritätsbestimmenden Regionen (CDRs) durch eine Trinukleotid-Mutagenese-Technologie (TRIM) erstellt 24. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
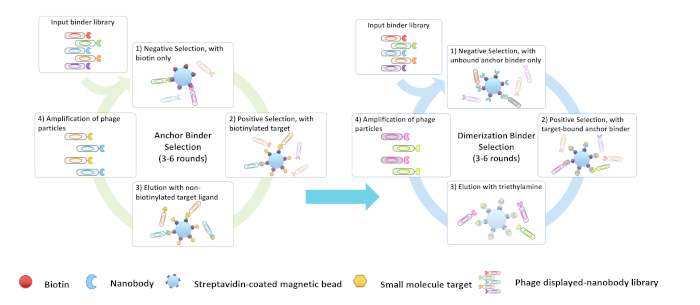
Abbildung 3: Flussdiagramm (A) Anker und (B) Dimerisierungsbindersiebung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
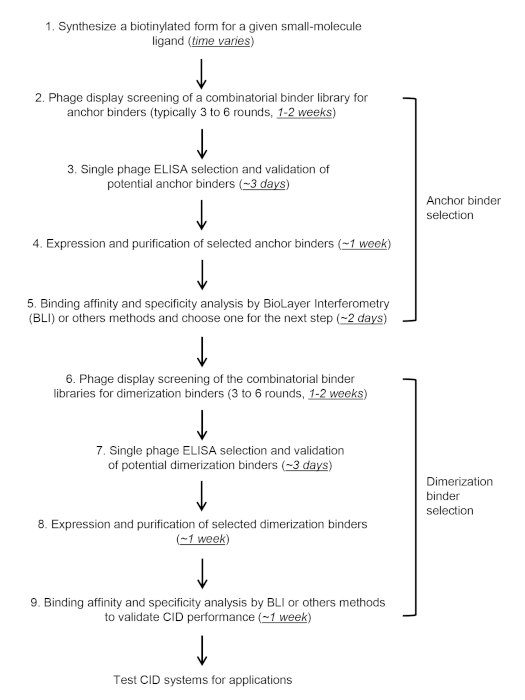
Abbildung 4: Zeitleiste von COMBINES-CID. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
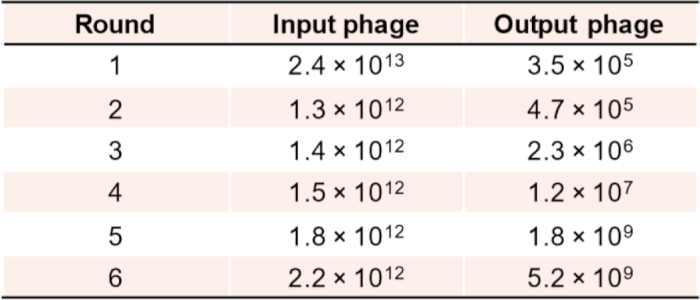
Abbildung 5: Anreicherung von Phagenitern nach jeder Biopanning-Runde für die Ankerbinderauswahl. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
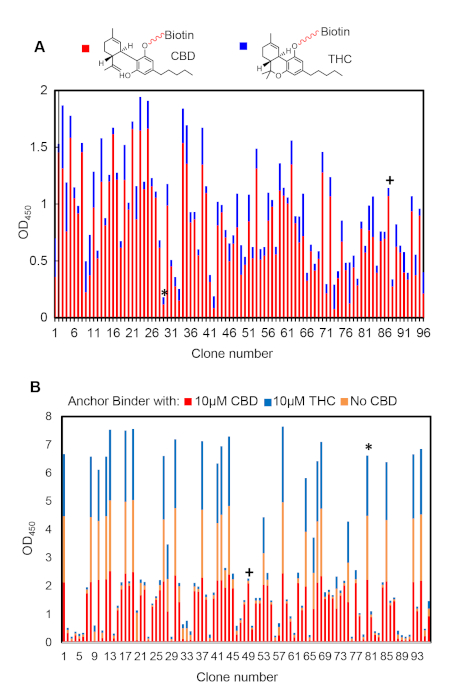
Abbildung 6: Repräsentative ELISA-Ergebnisse mit positiven (+) und negativen (*) Klonen. (A) Ankerbinder ELISA resultiert aus 96 zufällig ausgewählten Klonen nach sechs Auswahlrunden. (B) Dimerisierungsbinder ELISA Ergebnisse von 96 zufällig ausgewählten Klonen nach vier Runden der Auswahl. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
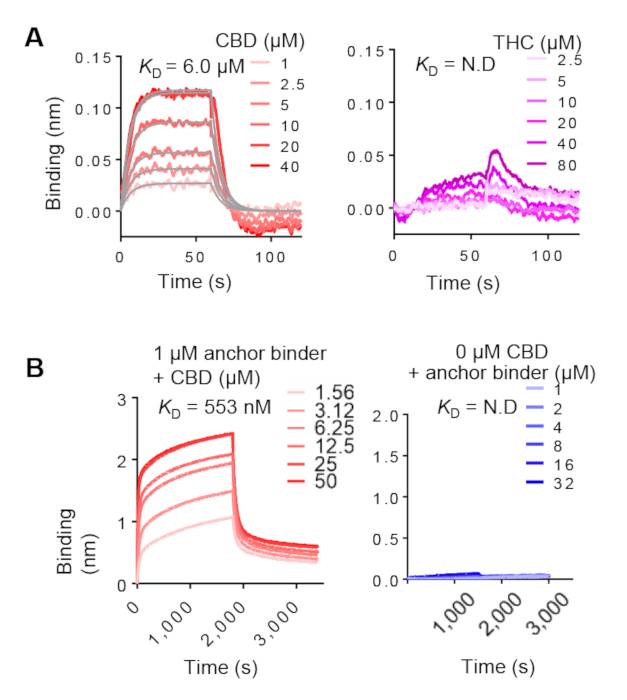
Abbildung 7: Bindungsanalyse des Anker- und Dimerisierungsbinders durch BLI. (A) Analyse des Ankerbinders mit unbeschrifteter CBD (links) und THC (rechts). Biotinylierte ankerbinder wurde auf Super Streptavidin (SSA) Biosensoren mit verschiedenen Konzentrationen von CBD titriert immobilisiert. Die Messwerte für die CBD-Bindung (rote Kurven) wurden global angepasst (graue Linien). (B) Links, BLI-Analyse eines SA Biosensor-immobilisierten Dimerisierungsbinderbindung an den Ankerbinder mit verschiedenen Konzentrationen von CBD vorlagert. Richtig, die Ankerbinderkonzentration wurde titriert und in Abwesenheit von CBD an das immobilisierte Dimerisierungsbinder gebunden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
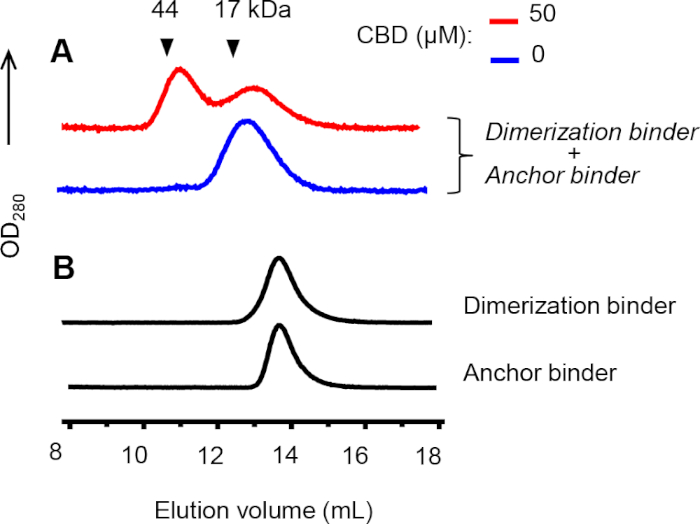
Abbildung 8: SEC-Analyse der Heterodimerisierung zwischen Anker- und Dimerisierungsbindern. (A) Die Dimerisierungs- und Ankerbinder (je 5 M) in Gegenwart oder Abwesenheit von CBD wurden vor der Analyse 30 min bei RT mit 100 m bis- N-Succinimidyl-(Pentaethylenglykol) Ester für 30 min bei RT vernetzt. Die Elutionsmengen von Proteinstandards sind durch Dreiecke gekennzeichnet. (B) Nicht vernetzte Anker- und Dimerisierungsbindemittel (je 30 M) wurden getrennt injiziert. Chromatogramme in A und B werden in verschiedenen Y-Skalen dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Es ist wichtig, die richtigen Konzentrationen von Eingangs-Phagenbibliotheken für verschiedene Runden der Biopanning zu wählen. Wir begannen in der Regel mit einer Eingabebibliothek von 1012–1013 Phagenpartikeln mit einer Diversität >109,so dass 100–1.000 Kopien jedes Phagenklons im Pull-Down-Test präsentiert werden konnten. Wenn die Phagenkonzentration in einem Bindungstest zu hoch oder zu niedrig ist, steigt die Wahrscheinlichkeit einer unspezifischen Bindung oder des Verlusts positiver Klone. Die Anker- oder Dimerisierungsbinderauswahl besteht normalerweise aus drei bis sechs Runden Biopanning, und die Anzahl der Ausgangsphagen beginnt in der Regel bei 104 und steigt auf 108–109. Es ist geeignet, einzelne Klone für die ELISA-Validierung zu wählen, nachdem sie eine solche Anreicherung beobachtet haben. Zusätzliche Runden der Biopannierung könnten die Chancen verringern, geeignete positive Klone mit geringer Häufigkeit zu identifizieren.
Es ist wichtig, geeignete Negativkontrollen und Auswahlen einzurichten, um den Erfolg der Auswahl zu verbessern. Zum Beispiel wird die Verwendung von strukturellen Analoga von Ligandenzielen die Auswahl der Ligandenspezifität erleichtern. In unserer Arbeit wurde ein sehr ähnliches Analog, THC, als Steuerung für CBD in der ELISA und BLI Validierung von Anker- und Dimerisierungsbindern19verwendet. Wenn Ankerbinder bei der Dimerisierungsbinderauswahl eine relativ geringe Ligandenbindungsaffinität haben, können sowohl freie als auch ligaandgebundene Ankerbinder als Ziele in der positiven Auswahl dargestellt werden. Daher ist es wichtig, Bindemittel, die während der negativen Selektion an freie Ankerbindemittel binden, gründlich zu entfernen. Dies kann erreicht werden, indem mehrere Runden der Subtraktion mit freien Ankerbindern ausgeführt werden.
Eine Einschränkung unseres Protokolls ist, dass Zielmoleküle für die Ankerbinderauswahl biotinyliert werden müssen und nur ein oder wenige Ankerbindemittel für die Dimerisierungsauswahl verwendet werden können. Die Verwendung von biotinylierten Targets kann Bindemittel anreichern, die teilweise an den Linker zwischen Biotin und Targets binden. Daher ist es wichtig, Treffer mit nicht beschrifteten Zielen durch BLI oder andere Techniken zu validieren. Die Wahl eines einzelnen oder einiger Ankerbinder für die Dimerisierungsbindemittelauswahl kann die Wahrscheinlichkeit verringern, CID-Systeme mit geeigneter Empfindlichkeit und Spezifität zu identifizieren. So wartet die Multiplexing-Fähigkeit der Selektion durch Kopplung an andere Techniken weiter auf eine weitere Verbesserung – z. B. die Single-Molecular-Interaction-Sequenzierung (SMI-Seq), die ein "Library-by-Library"-Protein-Protein-Interaktionsscreeningermöglicht 25.
Offenlegungen
Die University of Washington hat ein vorläufiges Patent für diese Arbeit eingereicht.
Danksagungen
Diese Arbeit wurde durch den University of Washington Innovation Award (an L.G.), ein Stipendium der U.S. National Institutes of Health (1R35GM128918 to L.G.) und einen Startup-Fonds der University of Washington (zu L.G.) unterstützt. H.J. wurde von einem Stipendium der Washington Research Foundation unterstützt. K.W. wurde von einem Stipendium des University of Washington Institute for Protein Design unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 1-Step Ultra TMB ELISA substrate solution | Thermo Fisher Scientific | 34029 | |
| Agar | Thermo Fisher Scientific | BP1423-2 | |
| Amicon Ultra-15 Centrifugal Filter unit (3 kDa cutoff) | Millipore | UFC900324 | |
| Ampicillin | Thermo Fisher Scientific | BP1760-25 | |
| Bio-Rad Protein Assay Kit II | Bio-Rad | 5000002 | |
| BirA biotin-protein ligase standard reaction kit | Avidity | BirA500 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A2153-50G | |
| Casein | Sigma-Aldrich | C7078-1KG | |
| CM13 Helper phage | Antibody Design Labs | PH020L | |
| D-(+)-Glucose monohydrate | Alfa Aesar | A11090 | |
| Dynabeads M-280 Streptavidin | Thermo Fisher Scientific | 11205D | |
| DynaMag-2 Magnet | Thermo Fisher Scientific | 12321D | |
| EDTA | Thermo Fisher Scientific | BP120-1 | |
| Fast DNA Ladder | New England Biolabs | N3238S | |
| FastDigest BglI | Thermo Fisher Scientific | FD0074 | |
| Glycerol | Thermo Fisher Scientific | BP229-1 | |
| HiLoad 16/600 Superdex 200 pg | GE Healthcare | 28989335 | |
| HiPrep 26/10 Desalting Column | GE Healthcare | 17508701 | |
| HisTrap-FF-1ml | GE Healthcare | 11000458 | |
| Imidazole | Alfa Aesar | 161-0718 | |
| IPTG | Thermo Fisher Scientific | 34060 | |
| Kanamycin | Thermo Fisher Scientific | BP906-5 | |
| M13 Major Coat Protein Antibody | Santa Cruz Biotechnology | sc-53004 | |
| NaCl | Sigma-Aldrich | S3014-500G | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Fisher Scientific | ND-2000 | |
| Nunc 96-Well Polypropylene DeepWell Storage Plates | Thermo Fisher Scientific | 260251 | |
| Nunc MaxiSorp | Thermo Fisher Scientific | 44-2404-21 | |
| Octet RED96 | ForteBio | N/A | |
| pADL-23c Phagemid Vector | Antibody Design Labs | PD0111 | |
| PEG-6000 | Sigma-Aldrich | 81260-1KG | |
| Platinum SuperFi DNA Polymerase | Invitrogen | 12351010 | |
| PureLink PCR Purification Kit | Thermo Fisher Scientific | K310001 | |
| QIAprep Spin M13 Kit | Qiagen | 27704 | |
| Recovery Medium | Lucigen | 80026-1 | |
| SpectraMax Plus 384 | Molecular Devices | N/A | |
| Sucrose | Sigma-Aldrich | S0389-1KG | |
| Super Streptavidin (SSA) Biosensors | ForteBio | 18-5057 | |
| Superdex 75 increase 10/300 GL Column | GE Healthcare | 28-9909-44 | |
| T4 DNA Ligase | Thermo Fisher Scientific | 15224-025 | |
| TG1 Electrocompetent Cells | Lucigen | 60502-1 | |
| Triethylamine | Sigma-Aldrich | 471283-100mL | |
| Trizma Base | Sigma-Aldrich | T1503 | |
| Tryptone | Thermo Fisher Scientific | BP9726-5 | |
| Tween 20 | Thermo Fisher Scientific | BP337-500 | |
| Yeast Extract | Thermo Fisher Scientific | BP1422-2 | |
| Zeba Spin Desalting Column | Thermo Fisher Scientific | 89882 |
Referenzen
- Stanton, B. Z., Chory, E. J., Crabtree, G. R. Chemically induced proximity in biology and medicine. Science. 359 (6380), (2018).
- Wu, C. Y., Roybal, K. T., Puchner, E. M., Onuffer, J., Lim, W. A. Remote control of therapeutic T cells through a small molecule-gated chimeric receptor. Science. 350 (6258), (2015).
- Straathof, K. C., et al. An inducible caspase 9 safety switch for T-cell therapy. Blood. 105 (11), 4247-4254 (2005).
- Di Stasi, A., et al. Inducible apoptosis as a safety switch for adoptive cell therapy. The New England Journal of Medicine. 365 (18), 1673-1683 (2011).
- Mank, M., et al. A FRET-based calcium biosensor with fast signal kinetics and high fluorescence change. Biophysical Journal. 90 (5), 1790-1796 (2006).
- Nagai, T., Sawano, A., Park, E. S., Miyawaki, A. Circularly permuted green fluorescent proteins engineered to sense Ca2+. Proceedings of the National Academy of Sciences of the United States of America. 98 (6), 3197-3202 (2001).
- Hunter, M. M., Margolies, M. N., Ju, A., Haber, E. High-affinity monoclonal antibodies to the cardiac glycoside, digoxin. Journal of Immunology. 129 (3), 1165-1172 (1982).
- Bradbury, A. R. M., Sidhu, S., Dubel, S., McCafferty, J. Beyond natural antibodies: the power of in vitro display technologies. Nature Biotechnology. 29 (3), 245-254 (2011).
- Chen, G., et al. Isolation of high-affinity ligand-binding proteins by periplasmic expression with cytometric screening (PECS). Nature. Biotechnology. 19 (6), 537-542 (2001).
- Tinberg, C. E., et al. Computational design of ligand-binding proteins with high affinity and selectivity. Nature. 501 (7466), 212-216 (2013).
- Spencer, D. M., Wandless, T. J., Schreiber, S. L., Crabtree, G. R. Controlling signal transduction with synthetic ligands. Science. 262 (5136), 1019-1024 (1993).
- Ho, S. N., Biggar, S. R., Spencer, D. M., Schreiber, S. L., Crabtree, G. R. Dimeric ligands define a role for transcriptional activation domains in reinitiation. Nature. 382 (6594), 822-826 (1996).
- Belshaw, P. J., Ho, S. N., Crabtree, G. R., Schreiber, S. L. Controlling protein association and subcellular localization with a synthetic ligand that induces heterodimerization of proteins. Proceedings of the National Academy of Sciences of the United States of America. 93 (10), 4604-4607 (1996).
- Farrar, M. A., AlberolaIla, J., Perlmutter, R. M. Activation of the Raf-1 kinase cascade by coumermycin-induced dimerization. Nature. 383 (6596), 178-181 (1996).
- Erhart, D., et al. Chemical Development of Intracellular Protein Heterodimerizers. Chemistry & Biology. 20 (4), 549-557 (2013).
- Ballister, E. R., Aonbangkhen, C., Mayo, A. M., Lampson, M. A., Chenoweth, D. M. Localized light-induced protein dimerization in living cells using a photocaged dimerizer. Nature Communications. 17 (5), 5475 (2014).
- Hill, Z. B., Martinko, A. J., Nguyen, D. P., Wells, J. A. Human antibody-based chemically induced dimerizers for cell therapeutic applications. Nature Chemical Biology. 14 (2), 112-117 (2018).
- Foight, G. W., et al. Multi-input chemical control of protein dimerization for programming graded cellular responses. Nature Biotechnology. 37 (10), 1209-1216 (2019).
- Kang, S., et al. COMBINES-CID: An efficient method for de novo engineering of highly specific chemically induced protein dimerization systems. Journal of the American Chemical Society. 141 (28), 10948-10952 (2019).
- Muyldermans, S. Nanobodies: natural single-domain antibodies. Annual Review of Biochemistry. 82, 775-797 (2013).
- Fanning, S. W., Horn, J. R. An anti-hapten camelid antibody reveals a cryptic binding site with significant energetic contributions from a nonhypervariable loop. Protein Science. 20 (7), 1196-1207 (2011).
- Zavrtanik, U., Luken, J., Loris, R., Lah, J., Hadzi, S. Structural basis of epitope recognition by heavy-chain camelid antibodies. Journal of Molecular Biology. 430 (21), 4369-4386 (2018).
- Denhardt, D. T., Dressler, D., Ray, D. S. . The Single-Stranded DNA Phages. , 605-625 (1978).
- Virnekas, B., et al. Trinucleotide phosphoramidites: ideal reagents for the synthesis of mixed oligonucleotides for random mutagenesis. Nucleic Acids Research. 22 (25), 5600-5607 (1994).
- Gu, L., et al. Multiplex single-molecule interaction profiling of DNA-barcoded proteins. Nature. 515 (7528), 554-557 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten