Method Article
Test delle terapie mirate nel cancro utilizzando l'analisi dell'alterazione strutturale del DNA e gli Xenogrini derivati dal paziente
In questo articolo
Riepilogo
Qui presentiamo un protocollo per testare l'efficacia delle terapie mirate selezionate in base alla trucco genomico di un tumore. Il protocollo descrive l'identificazione e la convalida dei riarrangiamenti strutturali del DNA, l'innesto dei tumori dei pazienti nei topi e il test delle risposte ai farmaci corrispondenti.
Abstract
Vi presentiamo un approccio integrativo per testare l'efficacia delle terapie mirate che combina il sequenziamento tecnolo-gies di prossima generazione, le analisi terapeutiche degli obiettivi e il monitoraggio della risposta ai farmaci utilizzando xenografifti derivati dal paziente (PDX). Questa strategia è stata convalidata utilizzando tumori ovarici come esempio. Il protocollo di sequenziamento della coppia di accoppiamento di nuova generazione (MPseq) è stato utilizzato per identificare le alterazioni strutturali e seguito dall'analisi delle alterazioni potenzialmente indirizzabili. I tumori umani coltivati in topi immunocompromessi sono stati trattati con farmaci selezionati sulla base delle analisi genomiche. I risultati hanno dimostrato una buona correlazione tra le risposte previste e le risposte osservate nel modello PDX. L'approccio presentato può essere utilizzato per testare l'efficacia dei trattamenti combinati e aiutare il trattamento personalizzato per i pazienti con cancro ricorrente, in particolare nei casi in cui la terapia standard fallisce e c'è la necessità di utilizzare farmaci fuori etichetta.
Introduzione
Gli xenografi (PDX) derivati dal paziente, generati dall'impianto di pezzi di tumore del paziente in topi immunodeficienti, sono emersi come un potente modello preclinico per aiutare la cura personalizzata anti-cancro. I modelli PDX sono stati sviluppati con successo per una varietà di neoplasie umane. Questi includono cancri al seno e alle ovaie, melanoma maligno, cancro colorettale, adenocarcinoma pancreatico e cancro del polmone non a piccole cellule1,2,2,4,5.5 Il tessuto tumorale può essere impiantato ortotopicamente o eterotopicamente. Il primo, considerato più preciso ma tecnicamente difficile, comporta il trapianto direttamente nell'organo di origine tumorale. Si ritiene che questi tipi di modelli imitano con precisione l'istologia del tumore originale a causa del microambiente "naturale" per il tumore6,7. Ad esempio, il trapianto ortotopico nella borsa dell'ovaio del topo ha portato alla diffusione del tumore nella cavità peritoneale e alla produzione di asciti, tipici del cancro ovarico8. Allo stesso modo, l'iniezione di tumori al seno nel toracico invece della ghiandola mammaria addominale ha influenzato il tasso di successo e il comportamento della PDX9. Tuttavia, i modelli ortotopici richiedono sofisticati sistemi di imaging per monitorare la crescita del tumore. L'impianto eterotopico del tumore solido viene in genere eseguito impiantando tessuto nel fianco sottocutaneo di un topo che consente un monitoraggio più facile della crescita del tumore ed è meno costoso e richiede tempo7. Tuttavia, i tumori cresciuti sottocutaneamente raramente metastatizzano a differenza di quanto osservato nel caso dell'impianto ortotopico10.
Il tasso di successo di innesto ha dimostrato di variare e dipendono notevolmente dal tipo di tumore. Tumori più aggressivi e campioni di tessuto contenenti una percentuale più alta di cellule tumorali sono stati segnalati per avere migliori tassi di successo12,13. Coerentemente con questo, i tumori derivati da siti metastatici hanno dimostrato di innestare a frequenze del 50-80%, mentre quelli dei siti primari innevano frequenze fino al 14%12. Al contrario, tessuto contenente cellule necrotiche e meno cellule tumorali vitali innevamale male. La crescita del tumore può anche essere promossa dall'aggiunta di proteine della matrice della membrana seminterrato nel mix di tessuto al momento dell'iniezione nei topi14 senza compromettere le proprietà del tumore originale. Le dimensioni e il numero di pezzi di tessuto destinati all'impianto sono stati trovati anche per influenzare il tasso di successo dell'innesto. Sono stati segnalati maggiori consumi tumorali per l'impianto nella capsula sub-renale rispetto all'impianto sottocutaneo dovuto alla capacità della capsula sub-renale di mantenere lo stroma tumorale originale e fornire anche le cellule stromali ospiti15.
La maggior parte degli studi utilizza topi immunodeficienti NOD/SCID, che mancano di cellule killer naturali16 e hanno dimostrato di aumentare l'innesto tumorale, la crescita e la metastasi rispetto ad altri ceppi14. Tuttavia, è necessario un ulteriore monitoraggio in quanto possono sviluppare linfomi timici già a 3-4 mesi di età13. Nei trapianti di tumore ovarico coltivati in topi SCID, la disoccupazione delle cellule B è stata inibita con successo da rituximab, impedendo lo sviluppo di linfomi ma senza influenzare l'innesto dei tumori ovarici17.
Più recentemente, NSG (NOD. Cg-Prkdcha scritto il 2rg Il2rgTm1Wjl/SzJ) topi, portando una mutazione nulla nel gene che codifica la catena gamma del recettore interleucolo 218,è diventato un ceppo frequentemente utilizzato per la generazione di modelli PDX. I tumori da modelli PDX consolidati passaggiano alle generazioni future di topi sono segnalati per mantenere le proprietà istologiche e molecolari per 3 a 6 generazioni19,20. Numerosi studi hanno dimostrato che gli esiti del trattamento nei modelli PDX imitano quelli dei loro pazienti corrispondenti2,3,4,21,22,23.23 Il tasso di risposta alla chemioterapia nei modelli PDX per il cancro del polmone non piccolo e carcinomi colorettali era simile a quello degli studi clinici per gli stessi farmaci24,25. Studi condotti in modelli PDX, sviluppati per pazienti arruolati in studi clinici, hanno dimostrato risposte a farmaci testati simili a quelli osservati clinicamente nei pazienti corrispondenti2,3,4.
Le analisi genomiche ad alto valore di throughput di un tumore del paziente in combinazione con i modelli PDX forniscono un potente strumento per studiare le correlazioni tra specifiche alterazioni genomiche e una risposta terapeutica. Queste sono state descritte in alcune pubblicazioni26,27. Ad esempio, le risposte terapeutiche all'inibitore EGFR cetuximab in una serie di modelli pdX colorettale che trasportano amplificazione EGFR, hanno parallelo le risposte cliniche acetuximab nei pazienti28.
Ci sono alcune sfide associate allo sviluppo e all'applicazione di modelli PDX. Tra questi c'è l'eterogeneità tumorale29,30 che può compromettere l'accuratezza dell'interpretazione della risposta al trattamento come un clone a cella singola con maggiore capacità proliferviva all'interno di una PDX può superare gli altri31, con conseguente perdita di eterogeneità. Inoltre, quando vengono utilizzate biopsie a singolo tumore per sviluppare la PDX, alcune delle popolazioni cellulari potrebbero essere perse e non saranno rappresentate nell'innesto finale. Più campioni dello stesso tumore sono raccomandati per l'impianto per risolvere questo problema. Anche se i tumori PDX tendono a contenere tutti i tipi di cellule del tumore donatore originale, queste cellule vengono gradualmente sostituite da quelle di origine murina3. L'interazione tra murine stroma e cellule tumorali umane nei modelli PDX non è ben compresa. Tuttavia, le cellule stromali hanno dimostrato di ricapitolare il microambiente tumorale33.
Nonostante queste limitazioni, i modelli PDX rimangono tra gli strumenti più preziosi per la ricerca traslazionale e la medicina personalizzata per la selezione delle terapie per i pazienti. Le principali applicazioni dei PDX includono la scoperta di biomarcatori e i test antidroga. I modelli PDX sono utilizzati con successo anche per studiare i meccanismi di resistenza ai farmaci e identificare le strategie per superare la resistenza ai farmaci34,35. L'approccio descritto nel presente manoscritto consente al ricercatore di identificare potenziali bersagli terapeutici nei tumori umani e di valutare l'efficacia dei farmaci corrispondenti invivo, nei topi che ospitano tumori innestati inizialmente caratterizzati genomicamente. Il protocollo utilizza tumori ovarici engrafted intraperitoneally ma è applicabile a qualsiasi tipo di tumore sufficientemente aggressivo per crescere nei topi2,3,12.
Protocollo
Tessuti freschi da pazienti consenzienti con cancro ovarico sono stati raccolti al momento della chirurgia di debulking secondo un protocollo approvato dal Mayo Clinic Institutional Review Board (IRB). Tutte le procedure e i trattamenti per gli animali utilizzati in questo protocollo sono stati approvati dal Comitato per la cura e l'uso degli animali della Mayo Clinic (IACUC) e hanno seguito le linee guida per la cura degli animali.
1. Accoppiamento di sequenziamento e analisi delle coppie
NOTA: per il sequenziamento della coppia di accoppiamenti (MPseq) è necessario utilizzare tessuto congelato fresco o flash. Il materiale incorporato della paraffina non è adatto perché contiene DNA frammentato.
- Isolare il DNA dal tessuto tumorale congelato. Utilizzare campioni umani originali ottenuti da materiale chirurgico o biopsia36.
- Utilizzare 1000 ng di DNA per creare librerie MPseq e sequenza come 2 campioni per corsia sul sequencer di prossima generazione (vedere Tabella dei materiali)36.
- Analizzare i dati utilizzando una serie di algoritmi per rilevare grandi aberrazioni cromosomiche (eliminazioni, inserimenti, amplificazioni, inversioni e traslocazioni) come descritto in precedenza36,37.
2. Selezione di bersagli terapeutici
- Utilizzare lo strumento Panda ad accesso aperto (Pathway e Annotazione) o uno strumento analogo per identificare le alterazioni di destinazione (http://bioinformaticstools.mayo.edu/Panda).
- Fare un elenco di geni che sono identificati da MSeq come alterati, come un semplice file delimitato da tabulazioni, utilizzando simboli genici accettati standard.
NOTA: l'esempio incluso presenta l'analisi delle amplificazioni e dei guadagni. - Aggiungere un segno di addiimazione nella riga di intestazione dell'elenco per assicurarsi che l'intestazione della tabella venga trasferita alla vista a livello di percorso del software.
- Caricare il file facendo clic sulla scheda di navigazione Carica gruppo di annotazioni.
- Assegnare una singola icona dal menu per rappresentare i dati sottostanti facendo clic sull'icona desiderata e quindi facendo clic sulla scheda Finalizza.
- Dopo aver caricato i file di annotazione, identificare una colonna che visualizza il numero di geni annotati per percorso. Questa è l'ultima colonna a destra.
- Utilizzare Filtro Pathway in alto a sinistra nella finestra principale per visualizzare le vie contenenti geni di interesse.
- Identificare le vie con più geni annotati di quanto ci si aspetta per caso. Utilizzare la funzione che si trova nella scheda Enrichment.
- Selezionare un database per visualizzare i geni potenzialmente drogabili da Preset Annotation controllando un'icona appropriata a sinistra della finestra principale (ad esempio, DGIdb, PharmGKB).
- Selezionare il percorso per la visualizzazione facendo clic sul nome visualizzato nella pagina Visualizzatore percorso.
NOTA: le icone che rappresentano ogni gruppo di annotazioni vengono visualizzate accanto al gene associato. Clicca sul gene di interesse per aprire la pagina web GeneCards corrispondente. - Selezionare le vie che hanno mostrato i geni annotati di interesse (cioè, alterati in un determinato tumore) e "hit" per potenziali farmaci per ulteriori analisi.
- Fare un elenco di geni che sono identificati da MSeq come alterati, come un semplice file delimitato da tabulazioni, utilizzando simboli genici accettati standard.
- Utilizzare un database contenente farmaci approvati per l'applicazione clinica (https://clinicaltrials.gov/) per fare riferimento incrociato agli obiettivi identificati.
- Assegna priorità alle modifiche mirabili per ulteriori test nei modelli PDX eseguendo una revisione della letteratura (ad esempio PubMed) per confermare la rilevanza per la biologia di un particolare tipo di tumore.
3. Convalida dei riarrangiamenti genomici mediante sequenziamento pcR e Sanger
- Progettare i primer utilizzando letture di sequenziamento ottenute dai dati MPseq.
- Selezionare un incrocio di interesse per la convalida (cioè potenziale obiettivo terapeutico) in base alle analisi di MPseq.
- Progetta primer direzionalmente in modo che l'amplificatore contenga la giunzione. Progettare 2 primer su ciascun lato della giunzione, per un totale di 4, per aumentare le probabilità di amplificare la giunzione.
NOTA: assegnare un nome ai primer in base alla custodia e alla posizione del cromosoma. - Utilizzare i parametri PCR standard per la progettazione del primer e un software di scelta. Selezionare la temperatura di fusione (60-62 gradi centigradi) e il contenuto di GC (40-60%). Assicurarsi che la sequenza primer non forma dimeri primer, palindromi o tornanti.
- Confermare che la sequenza di primer manca di omologia ad altre aree del genoma umano controllandola utilizzando BLAT (http://genome.ucsc.edu/cgi-bin/hgBlat?command=start).
- Eseguire una PCR per amplificare la giunzione di interesse.
- Diluire i primer con acqua a 10 mM e combinare 10 mL di ogni primer in modo che ogni primer in avanti sia abbinato a ogni primer inverso in un mix di primer.
NOTA: le sequenze per i primer per l'esempio selezionato sono mostrate nella Tabella 1. - Etichettare i tubi a striscia 0,2 mL come: C1, T1, C2, T2, C3, T3, C4 e T4 dove C-controllo DNA genomico umano (commerciale), DNA tumorale, isolato dal paziente o tumore PDX, e il numero indica il mix primer.
- Aggiungere 1 mL di ogni miscela di primer nei suoi tubi etichettati.
- Preparare il mastermix Taq combinando i reagenti elencati nella tabella 2, lasciando l'enzima nel congelatore fino a quando necessario, aggiungendolo al mix alla fine.
- Fare 2 miscele master, uno per ogni modello di DNA, controllare il DNA umano e il DNA tumorale. Aggiungere 24 mL di ogni mix master Taq al rispettivo tubo a strisce. Il volume di reazione totale è 25 mL. Vortice molto brevemente e poi girare il tubo striscia verso il basso.
- Eseguire una PCR in un termociclore. Utilizzare i parametri riportati nella tabella 3. Regolare la temperatura di annealing in modo che sia di almeno 1 gradi centigradi più fredda della temperatura di fusione dei primer.
- Conservare il prodotto PCR completato a -20 gradi centigradi (a lungo termine) o refrigerato a 4 gradi centigradi (a breve termine) fino a quando necessario.
- Eseguire l'elettroforesi a 1-5 V/cm per visualizzare il prodotto PCR utilizzando un gel agarose dell'1,5%. Lasciare 2 aliquote mL del prodotto da utilizzare per il sequenziamento di Sanger.
- Diluire i primer con acqua a 10 mM e combinare 10 mL di ogni primer in modo che ogni primer in avanti sia abbinato a ogni primer inverso in un mix di primer.
- Eseguire la sequenza Sanger per confermare la giunzione e identificare il punto di interruzione esatto38.
- Utilizzare il prodotto PCR se la PCR ha generato un singolo prodotto (banda). In alternativa, tagliare la banda di gel, purificare e presentare per il sequenziamento Sanger insieme al primer utilizzato per l'amplificazione.
NOTA: I tumori per i quali sono state eseguite analisi genomiche vengono quindi utilizzati per l'impianto nei topi.
- Utilizzare il prodotto PCR se la PCR ha generato un singolo prodotto (banda). In alternativa, tagliare la banda di gel, purificare e presentare per il sequenziamento Sanger insieme al primer utilizzato per l'amplificazione.
4. Innesto e manutenzione del tumore
- Impostare i preparativi per innestare i tumori nei modelli murini PDX. Selezionare per tumori di innesto per i quali le analisi genomiche saranno o sono state eseguite.
- Assicurarsi che al momento dell'inizio dello sviluppo di qualsiasi modello PDX, compresi laboratori dedicati e strutture animali, personale tecnico qualificato e procedure operative standard dettagliate, sia necessario un'infrastruttura di supporto.
- Garantire il trasporto rapido e l'elaborazione dei campioni come velocità è fondamentale per la vitalità cellulare e l'innesto di successo.
- Utilizzare un ambiente sterile per ridurre la contaminazione batterica e fungina per la lavorazione e l'incisa dei campioni.
- Maneggiare i campioni umani con cautela, in conformità con le politiche istituzionali relative ai materiali potenzialmente biopericolosi, in quanto possono ospitare agenti patogeni trasmessi dal sangue.
- Preparare il tessuto (0,5-0,7 cm3 dimensioni) per l'innesto mettendo il campione chirurgico in un tubo pre-raffreddato da 50 mL con 20 mL di supporti per coltura tissutale.
NOTA: Il tessuto tumorale può essere fresco o recuperato da materiale crioconservato precedentemente5. - Confermare il contenuto tumorale nel campione consultando un patologo.
- Collocare il tessuto tumorale in un piatto contenente 10-15 mL di PBS freddo, o mezzi di coltura tissutale come RPMI 1640 o DMEM contenenti antibiotici (1% penicillina e streptomia).
- Identificare e isolare il materiale tumorale vitale dal tessuto normale e necrotico adiacente con l'aiuto di un patologo. Utilizzare pinze sterili e bisturi per rimuovere il materiale necrotico sottolineato da un patologo.
NOTA: Il tumore può essere impiantato intraperitonelmente o sottocutaneamente nei topi. Seguire il passaggio 4.2 per eseguire un impianto intraperitunole o andare al passaggio 4.3 per eseguire un trapianto sottocutaneo. In questo studio, terapie mirate selezionate sulla base di analisi genomiche sono state testate in una serie di modelli PDX per il cancro ovarico sieroco di alto grado con impianto intraperitoneale.
- Preparare il tessuto per l'impianto intraperitoneale (IP) mininando il tessuto con pinze sterili e un bisturi sul ghiaccio per fare pezzi di circa 1-1,5 mm3 di dimensioni e mescolando con mezzo di coltura fredda. Iniettare 0,3–0,5 mL di liquami tumorali utilizzando un ago calibro 16-17.
NOTA: Tutte le procedure chirurgiche vengono eseguite utilizzando tecniche asettiche. Per ridurre la probabilità di infezione sono stati utilizzati guanti sterili, strumenti sterili, forniture e materiali impiantati.- Mescolare i pezzi 1:1 con mezzo di coltura ghiacciata e iniettare 100 mL intraperitonealmente in almeno tre topi SCID femminili.
- Tagliare il tessuto tumorale per l'innesto sottocutaneo usando pinze sterili, bisturi o forbici chirurgiche in piccoli frammenti, di circa 2 x 2 x 2 mm, e trasferire i tessuti frammentati in una parabola Petri pre-raffreddata sul ghiaccio.
- Aggiungere la matrice fredda della membrana seminterrato nel piatto con il tessuto frammentato (circa 200 mL per 10 pezzi di tessuto), mescolare bene, e lasciare che i frammenti di tessuto immergere nella matrice fredda della membrana seminterrato per 10 min.
- Anestetizza 5 topi NOD/SCID femminili per prepararli all'innesto.
- Iniettare ogni topo per via intraperitamente con ketamina (150 mg/kg) e xylazina (10 mg/kg).
- Verificare che il mouse sia correttamente anetizzato pizzicando la punta della coda con pinze atraumatiche.
- Rimuovere delicatamente il mouse completamente anestesizzato dalla camera e mettere sul mouse un cono naso che ha ingresso da vaporizzatore e l'uscita dal sistema di scavenging gas di scarico.
- Mettere unguento veterinario sugli occhi del topo per evitare la secchezza durante l'anestesia. Preparare l'area in cui verrà eseguito l'intervento chirurgico. Utilizzare la tecnica asettica per eseguire l'intervento chirurgico.
- Assicurarsi che la superficie su cui l'intervento chirurgico sta per prendere è non poroso, sigillato ed è disinfettato prima dell'intervento chirurgico.
- Avviare la chirurgia con strumenti sterili (per autoclave, gas o sterilizzazione chimica).
- Utilizzare i guanti per gestire gli strumenti e mantenere la sterilità delle punte dello strumento per tutta la procedura immergendoli in etanolo tra passaggi chirurgici.
- Sterilizzare il sito chirurgico applicando 3 scrub alternati di iodio e alcol. Utilizzare forbici chirurgiche sterili e pinze per fare una pelle di incisione verticale di 5-10 mm su entrambi i fianchi di un topo.
- Inserire pinze dritte delicatamente nello spazio sottocutaneo per creare una tasca abbastanza grande da poter posizionare un frammento tumorale sotto il cuscinetto a grasso.
- Utilizzare le pinze dritte sterili per inserire frammenti tumorali nella tasca precedentemente preparata in ciascuno dei 5 topi.
NOTA: Mettere 3-4 pezzi di tessuto tumorale in una tasca. - Chiudere le incisioni cutanee utilizzando la colla del tessuto.
- Intraperitonealmente iniettare ogni topo con 100 mL di Rituximab dopo l'impianto per inibire la proliferazione dei linfociti.
- Mettere il mouse in una gabbia sotto una lampada termica per circa 20 minuti fino a quando non viene recuperato dall'anestesia. Monitorare i segni vitali del topo e garantire la sua sufficiente idratazione.
- Riportare il topo alla compagnia di altri topi dopo che si è ripreso dall'anestesia e ha iniziato ad avere cibo e acqua. Fornire assistenza postoperatoria e monitoraggio secondo le linee guida istituzionali. Controllare i segni di dolore e angoscia ogni giorno per 3 giorni consecutivi dopo l'intervento chirurgico.
NOTA: I criteri per il dolore o l'angoscia includono necrosi o ulcerazione, perdita di peso / punteggio condizione del corpo, segni comportamentali come il livello di attività, funzione motoria e postura. - Controllare regolarmente i topi per la formazione del tumore bisettimanale fino a quando i tumori raggiungono la dimensione di 0,5 cm di diametro come misurato da una pinza.
- Valutare il punteggio di integrità di ogni mouse come derivato dall'aspetto, dal comportamento e dalla condizione del corpo39. Utilizzare punteggi di 6 come criteri per i topi moribondi che devono essere sacrificati dall'inalazione di biossido di carbonio.
5. Test delle risposte a obiettivi identificati genomicamente nei modelli PDX
- Iniziare i trattamenti mirati selezionati quando i tumori sono palpabili e raggiungono 0,5 cm3 come misurato da una scansione ecografica.
- Prima di eseguire una ecografia dell'addome rimuovere la pelliccia addominale del topo e applicare lubrificante sterile gelatina.
- Utilizzare una macchina ad ultrasuoni con un trasduttore per ottenere immagini con il tumore posizionato nella sezione trasversale. Effettuare 3 misurazioni per sessione per ogni animale e calcolare in media il valore per una valutazione più accurata delle dimensioni del tumore.
- Analizzare le immagini utilizzando il software disponibile40.
- Somministrare la chemioterapia costituita da una miscela di carboplatina a 51 mg/kg e paclitaxel a 15 mg/kg intraperitonealmente (IP) una volta alla settimana per una durata totale del trattamento di 4-6 settimane. Assicurarsi che il volume totale per l'iniezione non superi 0,2 mL.
- Effettua una soluzione MK-220641 stock in 30% ciclodextrin (ad esempio, Captisol) e consegna tramite gavage orale a 120 mg/kg al giorno per 4 settimane consecutive.
- Preparare il mouse per il gavage orale tenendolo pizzicando la pelle della schiena e appuntandolo indietro, in modo che la testa e gli arti del mouse siano immobilizzati.
- Inserire la sonda gavage lungo la parte posteriore della gola del mouse fino a quando la sonda raggiunge l'esofago. Assicurarsi che la sonda non sia inserita troppo lontano, poiché i polmoni del topo possono perforare, causando la morte.
- Fare una soluzione di stock MK-866942 in etanolo a 25 mg/mL. Diluire in un veicolo contenente 5.2% Tween 80, 5.2% PEG400 in acqua sterile per iniezioni IP a 10 mg/kg per 5 giorni ogni due settimane, con una durata totale del trattamento di 4 settimane.
NOTA: Il volume iniettato nei topi deve essere 50-120 mL, a seconda del peso dell'animale. - Utilizzare 7-8 topi per gruppo di trattamento per avere abbastanza energia statistica per rilevare le differenze43,44.
- Valutare il peso corporeo e le condizioni generali dei topi in terapia ogni giorno. Trattenere i farmaci se il peso di un animale scende del 20% o più dal loro peso iniziale.
- Valutare la dimensione del tumore settimanalmente utilizzando la scansione a ultrasuoni. Assicurarsi che l'individuo che esegue il monitoraggio della crescita del tumore è accecato al trattamento per garantire il punteggio imparziale delle risposte.
NOTA: I laboratori più piccoli possono assumere 2 persone diverse per somministrare il trattamento e il monitoraggio degli ultrasuoni.
Risultati
I tessuti provenienti da tumori ovarici resezionati al momento degli interventi chirurgici di debulking sono stati raccolti in conformità con la guida Dell'IRB e utilizzati per 1) caratterizzazione genomica e 2) innesto di topi immunocompromessi (Figura 1). Il protocollo di sequenziamentoMate-pair 36,37 è stato utilizzato per identificare le alterazioni strutturali nel DNA, tra cui perdite, guadagni e amplificazioni. Un grafico genoma rappresentativo che illustra un paesaggio di cambiamenti genomici in un tumore (designato come OC101) è mostrato nella Figura 2. Tipico per tumori di sottotipo serosomi di alto grado, sono stati trovati più guadagni (linee blu) e delezioni (linee rosse), che indicano alti livelli di instabilità genomica, così come le perdite cromosomiche e guadagni, indicativi di aneuploidia, sono stati osservati. In media 300-700 alterazioni totali sono identificati in tumori di sottotipo siero di alto grado45. Le analisi successive hanno rivelato alcune alterazioni del DNA potenzialmente indirizzabili con farmaci clinicamente rilevanti. L'alterazione più classifica per l'intervento terapeutico nel tumore OC101 è stata un'amplificazione al cromosoma 17 che coinvolge l'ERBB2(Figura 2 e Figura 3A). ERBB2 è un gene che codifica per il recettore HER2 che è noto sulla dimerizzazione con EGFR, HER3 o HER4 per attivare i percorsi di segnalazione RAS/ERK e PI3K/AKT e promuovere la crescita cellulare, la migrazione cellulare e l'invasione. Gli inibitori HER2 (ad es. anticorpi monoclonali pertuzumab e trastuzumab) sono efficaci nel trattamento di pazienti affetti da cancro al seno quando i tumori esprimono la proteina HER2. La terapia anti-HER2 per il cancro ovarico, tuttavia, non è approvata dalla FDA.
Il confronto tra il profilo genomico del tumore dei donatori (Figura 1) e quello di un corrispondente modello PDX (non mostrato) ha rivelato una sorprendente somiglianza, coerente con tutti gli studi precedenti che riportano la vicinanza molecolare dei tumori originali ai loro derivati della PDX.
Per convalidare i risultati di MPseq a livello di DNA, sono stati progettati diversi gruppi di primer specifici per i bordi della regione amplificata contenente il gene ERBB2, e la PCR è stata effettuata utilizzando DNA isolato dal tumore originale e da un tumore propagato per diverse generazioni nei topi. Un'immagine gel rappresentativa dei prodotti amplificati utilizzando due diversi set di primer è mostrata nella Figura 3B. Nessuna banda è stata rilevata quando il normale DNA genomico raggruppato (designato come C), che non conteneva amplificazione del locus ERBB, è stato amplificato. La purificazione dei prodotti dal gel e dal sequenziamento di Sanger (non mostrato) ha ulteriormente confermato l'alterazione prevista da MPseq. Un'ulteriore convalida è stata condotta esaminando l'espressione della proteina HER2 nel tumore PDX corrispondente utilizzando l'immunoblotting. L'analisi ha rivelato un alto livello di proteina HER2 (il risultato non è mostrato, coerente con l'amplificazione osservata del gene ERBB2.
Nel DNA di un tumore ovarico diverso (designato T14) sono stati osservati numerosi guadagni regionali. Tra questi, i geni AKT2 e RICTOR (Figura 3C). Entrambi erano di grande interesse da una prospettiva terapeutica come inibitori di AKT2 e mTOR, che RICTOR associa con, sono disponibili e attualmente in studi clinici. Poiché non c'erano leghe accoppiate che si estendevano dalla vicinanza di entrambi i geni, non era possibile una semplice convalida PCR del guadagno rilevato da MPseq. Pertanto, abbiamo testato il livello di espressione delle proteine corrispondenti mediante immunoblotting. Alti livelli di AKT e RICTOR sono stati osservati (Figura 3D) suggerendo che il trattamento con farmaci mirati è giustificato.
Per testare la sensibilità di questo tumore agli inibitori di AKT e mTOR, i topi PDX con tumore T14 impiantato intraperitonealmente sono stati espansi e randomizzati per ricevere solo la chemioterapia (carboplatina/paclitaxel) o una combinazione di chemioterapia con inibitore pan-AKT MK-220641 o mTOR inibitore MK-866942. La chemioterapia è stata somministrata ai topi nel braccio combinato per 2 settimane prima dell'aggiunta della terapia mirata (Figura 4). Le misurazioni ad ultrasuoni sono state effettuate settimanalmente per monitorare la regressione/crescita del tumore.
Ogni braccio di trattamento conteneva non meno di 7 topi. Questo numero è stato sufficiente per osservare le differenze nelle risposte tra i gruppi, mantenendo i costi dello studio a un livello notevolmente inferiore. Meno topi (da tre a quattro) possono essere utilizzati nel gruppo di controllo non trattato, come variazioni individuali nel tasso di crescita del tumore sono trascurabili e la crescita è normalizzata a una dimensione del tumore a cui inizia il trattamento in altre braccia.
Non è stata osservata alcuna differenza tra gruppi trattati con chemio e non trattati nelle prime 3 settimane di osservazione (non mostrato). Una significativa riduzione del carico tumorale (58% mediana) è stata osservata nel gruppo trattato con chemioterapia entro la fine della settimana 6. Un ulteriore beneficio rispetto alla sola chemioterapia è stato osservato in gruppi che hanno ricevuto una combinazione di chemioterapia con terapie mirate. La differenza è diventata evidente rispettivamente alla settimana 4 e 3 per MK-8669 (Figura 4A) e MK-2206 (Figura 4B).
Gli animali sono stati eutanasia e il tessuto tumorale è stato raccolto per l'analisi molecolare della risposta al trattamento alla fine dello studio di trattamento alla settimana 7. A tal fine, le quantità di chinasi S6 totale e fosforato (Figura 5A,B), AKT e mTOR (Figura 5C,D) sono state determinate utilizzando l'immunoblotting. La proteina ribosomica S6 chinasi è un messaggero a valle del percorso AKT-mTOR, noto per essere up-regolato e fosforillato dopo la stimolazione dell'asse AKT-mTOR da fattori di crescita per promuovere la sopravvivenza cellulare e la crescita. Il confronto dei livelli di queste proteine in tumori PDX non trattati o trattati con chemioterapia PDX a topi che hanno ricevuto aKT o inibitori mTOR ha mostrato una notevole diminuzione negli ultimi due (Figura 5), che indica l'efficacia della terapia mirata a livello molecolare. Le regolazioni al reggimento di trattamento, per quanto riguarda la tempistica di applicazione per i farmaci combinati e la durata della terapia, dovrebbero essere effettuate e testate per ottenere risposte migliori.
| Nome Primer | Sequenza | Primer Mix | Primer combinati | ||
| OC101 17a-17b R1 | CTGCTCTGAATAGACACTAGATAAATCATC | 1 | OC101 F1 e R1 | ||
| OC101 17a-17b R2 | GCTCAAGACAGTAACACCTAGTGATCTGC | 2 | OC101 F1 e R2 | ||
| OC101 17b-17a F1 | GGATTACGGGAACGTGCTACCTG | 3 | OC101 F2 e R1 | ||
| OC101 17b-17a F2 | AATGCCCTAGCAGTTCTATCCACTG | 4 | OC101 F2 e R2 | ||
Tabella 1: Primer utilizzati per la convalida dell'alterazione OC101chromosome 17.
| Reagente | Quantità da aggiungere per 1 reazione (L) | Quantità da aggiungere per 4 reazioni (L). [Moltiplica per 4.3 per fare abbastanza per ogni mix di primer (4)] |
| Acqua priva di nucle | 20.45 | 89.94 |
| Buffer 10x (Facile A) | 2.5 | 10.75 |
| dNTPs 10 mM | 0.5 | 2.15 |
| DNA del modello (concentrazione >10 ng/L) | 0.3 | 1.29 |
| Polimerasi Taq | 0.25 | 1.08 |
Tabella 2: Configurazione PCR per convalidare un nodo.
| Temp (C) | Ore | |
| 94 | 5 min | |
| 94 | 40 s | 35 cicli |
| 59 | 40 s | |
| 72 | 2 min | |
| 72 | 5 min | |
| 4 | Tenere |
Tabella 3: Condizioni di ciclismo per PCR per convalidare un incrocio.
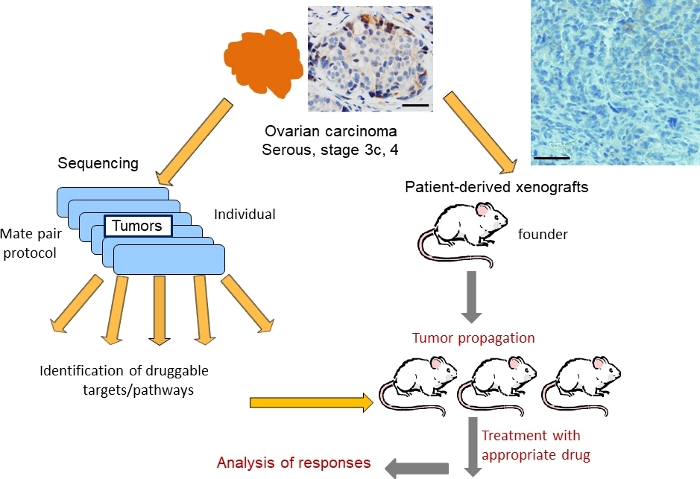
Figura 1: Rappresentazione schematica della strategia per i test di terapia a guida genomica utilizzando modelli PDX per il carcinoma ovarico sero. La colorazione H&E del tumore ovarico è mostrata (in alto). Barra di scala 100 mm. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Caratterizzazione genomica dei tumori ovarici utilizzando MPseq. Grafico genomico che mostra il paesaggio delle alterazioni strutturali e dei cambiamenti del numero di copie rilevato da MPseq. L'asse X si estende per la lunghezza del cromosoma con il numero di posizione del cromosoma mostrato. Ogni cromosoma è indicato sull'asse Y destro e sinistro. L'altezza delle tracce orizzontali per ogni cromosoma indica il numero di letture rilevate per le finestre delle coppie di basi di 30k. I numeri di copia del DNA sono indicati dal colore, con il grigio che rappresenta il normale stato di copia 2N, il rosso corrispondente alle eliminazioni e i guadagni blu-to. Le linee nere di collegamento corrispondono a riarrangiamenti cromosomici. Sono raffigurate alterazioni presso il locus ERBB2. Fare clic qui per visualizzare una versione più grande di questa figura.
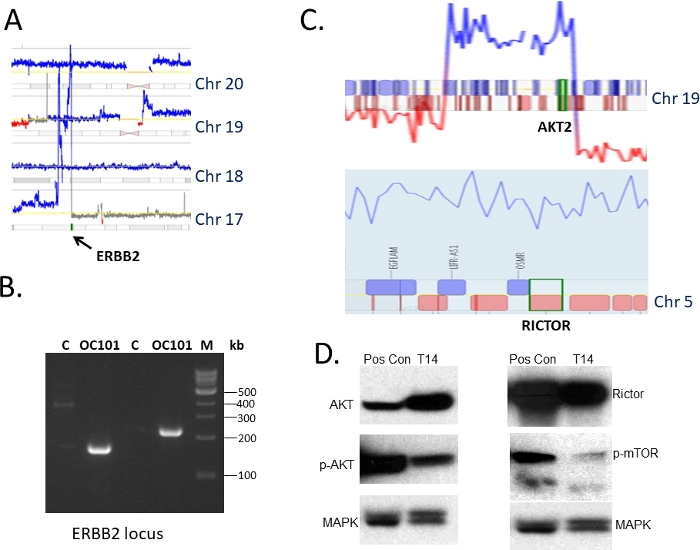
Figura 3: Selezione delle modifiche indirizzabili e della relativa convalida. (A) Un segmento ravvicinato della trama del genoma mostrato in Fig.1 che illustra un'amplificazione del gene ERBB2 sul cromosoma 17 (in blu). I numeri dei cromosomi sono indicati. (B) Analisi di convalida dei punti di interruzione nel locus ERBB2 identificata da MPseq mediante amplificazione PCR. C è un controllo del DNA genomico in pool, OC101 è DNA dal tumore del paziente, M è una scala del DNA. (C) Un primo piano di segmenti del terreno genoma per un altro tumore ovarico che mostra guadagni (indicati dalla linea blu) al locus AKT (in alto) e al gene RICTOR (in basso). I cromosomi sono come indicato. (D) Analisi di convalida dell'espressione delle proteine della via AKT/mTOR mediante immunoblotting utilizzando il tessuto tumorale di PDX (T14), sono state utilizzate alterazioni genomiche per le quali sono rappresentate in C. 30 mg di proteine totali e anticorpi specifici per AKT, RICTOR, p-mTOR. MAPK fungeva da controllo di carico. Pos con è un tumore indipendente usato come controllo positivo. Fare clic qui per visualizzare una versione più grande di questa figura.
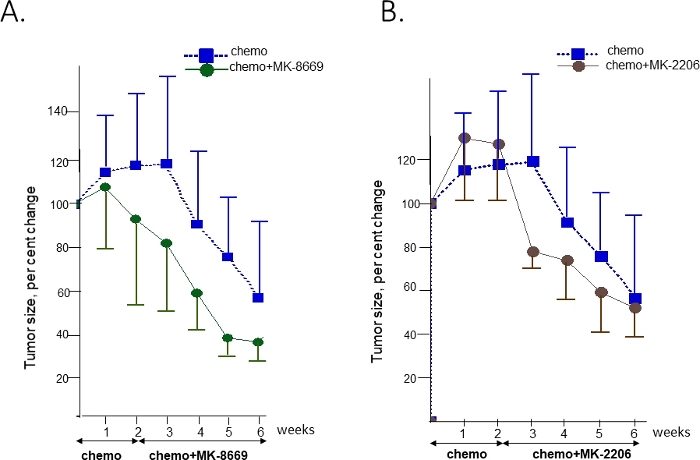
Figura 4: Il confronto delle risposte alla chemioterapia da solo e combinato con la terapia mirata nel tumore che ospita guadagni ai geni AKT2 e RICTOR con i farmaci corrispondenti. Risposte al trattamento a una combinazione di chemioterapia e farmaco anti-mTOR MK-8669 (A) o inibitore di pan-AKT MK2206 (B) contro chemioterapia da solo. Il tempo di somministrazione per ogni trattamento e la durata sono indicati dalle frecce. I volumi sono espressi come percentuale del volume iniziale all'inizio del trattamento come media s/- SD. Chemio è chemioterapia. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: Confronto dei cambiamenti molecolari suscitati da ogni trattamento come determinato dall'analisi dell'immunoblotting. (A) Vengono mostrati i livelli di S6 e fosfo-S6, l'effettuatore a valle del percorso AKT-mTOR. Sono stati utilizzati 30 mg di proteine totali e anticorpi specifici per S6 e p-S6. GAPDH è stato utilizzato come controllo del carico. La quantificazione dei livelli proteici normalizzati al livelloCGAPDH è indicata nei livellidimTOR, p-mTOR, AKT e p-AKT rilevati dall'immunoblotting. GAPDH è stato utilizzato come controllo del carico. (D) Quantificazione dei livelli proteici normalizzati a livello di GAPDH. NT non è trattato, la chemio è chemioterapia. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Descriviamo l'approccio e i protocolli che abbiamo usato per condurre una "sperimentazione clinica" nei modelli PDX che sfrutta le caratteristiche molecolari del tumore ottenute dalla profilazione genomica per determinare la migliore scelta di farmaci per il test. Più piattaforme di sequenziamento sono attualmente utilizzate per la caratterizzazione genomica dei tumori primari, tra cui il sequenziamento dell'intero genoma, gli RNA aseq e i pannelli genici personalizzati. Per il carcinoma ovarico siero di alta qualità, MPseq per identificare alterazioni strutturali, riarrangiamenti del DNA e cambiamenti del numero di copie, è particolarmente utile a causa dell'elevato grado di instabilità genomica osservato in questo tipo di tumore. Il secondo vantaggio della piattaforma MPseq è che copre l'intero genoma, ma costa significativamente meno rispetto ad altre tecnologie di sequenziamento complete. MPseq, tuttavia, non è adatto per il rilevamento della mutazione puntiforme in quanto la copertura di base non è sufficiente, raggiungendo solo 8-10x. Uno dei limiti dell'uso di MPseq da solo per la caratterizzazione genomica di un tumore è la presenza di complessi riarrangiamenti cromosomici raggruppati, la cui analisi non prevede l'espressione di geni impattati di interesse. Una giunzione rilevata da MPseq prevede di creare un gene di fusione putativa che convalida nel DNA da PCR non può essere espressa a causa di uno spostamento del telaio o di piccole eliminazioni e inserimenti nella regione promotrice. Allo stesso modo, i guadagni cromosomici e le amplificazioni per potenziali bersagli terapeutici devono essere attentamente valutati e convalidati a livello di RNA o proteina per garantire l'espressione del gene di interesse.
Anche se, la composizione molecolare del tumore è ampiamente conservata dopo la propagazione nei topi per diverse generazioni, i cambiamenti nei livelli di espressione dei geni chiave possono verificarsi nel tempo riflettendo l'evoluzione del tumore, la selezione clonale o la risposta adattiva all'ambiente murino. Pertanto, la convalida di un'alterazione destinata all'intervento terapeutico sia nel tumore del donatore originale che nel tumore della PDX è fondamentale. Per il tumore isolato dai modelli PDX sia l'RNA che la proteina possono essere utilizzati per interrogare il livello di espressione in quanto il materiale è abbondante. Uno dei due può essere scelto per controllare l'espressione nel tumore originale, a seconda della disponibilità e del tipo del tessuto immagazzinato, e la disponibilità di anticorpi per il rilevamento delle proteine corrispondente. Il modello PDX è particolarmente prezioso nei test terapie combinate come l'impostazione in vivo consente il monitoraggio degli effetti negativi, così come il dosaggio o regolazioni di durata nel regime di trattamento.
La scelta tra l'innesto ortotopico e quello sottocutaneo può essere effettuata a seconda delle domande specifiche affrontate nello studio. Tuttavia, è importante tenere a mente che la sensibilità dei tumori xenotrapita alle terapie può essere modulata dal sito di impianto46. D'altra parte, nessuna prova è stata ancora riportata sulla scoperta di farmaci che mostrano una risposta terapeutica in modelli ortotopici, ma assenti nella PDX9 impiantatosottocutaneamente .
Divulgazioni
Gli autori dichiarano di non avere alcun conflitto di interessi.
Riconoscimenti
Ringraziamo i membri del Mayo Clinic Center for Individualized Medicine (CIM) Dr. Lin Yang e Faye R. Harris, MS, per l'aiuto nello svolgimento di esperimenti. Questo lavoro è stato sostenuto da Don.
Materiali
| Name | Company | Catalog Number | Comments |
| 3M Vetbond | 3M, Co. | 1469SB | |
| anti-AKT antibody | Cell Signaling Technologies, Inc. | 9272 | |
| Anti-GAPDH antibody(G-9) | Santa Cruz Biotech. Inc. | sc-365062 | |
| Anti-MAPK antibody | Cell Signaling Technologies, Inc. | 9926 | |
| Anti-phospho-AKT antibody | Cell Signaling Technologies, Inc. | 9271 | |
| Anti-mTOR antibody | Cell Signaling Technologies, Inc. | 2972 | |
| Anti-Phospho-mTOR antibody | Cell Signaling Technologies, Inc. | 2971 | |
| Anti-Phospho-S6 antibody | Cell Signaling Technologies, Inc. | 4858 | |
| Anti-Rictor antibody | Cell Signaling Technologies, Inc. | 2114 | |
| Anti-S6 antibody | Cell Signaling Technologies, Inc. | 2217 | |
| Captisol | ChemScene, Inc. | cs-0731 | |
| Carboplatin | NOVAPLUS, Inc. | 61703-360-18 | |
| DMEM | Mediatech, Inc. | 10-013-CV | |
| Easy-A Hi-Fi PCR Cloning Enzyme | Agilent, Inc. | 600404-51 | |
| Lubricant | Cardinal Healthcare | 82-280 | |
| Matrigel | Corning, Inc. | 356234 | |
| McCoy's media | Mediatech, Inc. | 10-050-CV | |
| MK-2206 | ApexBio, Inc. | A3010 | |
| MK-8669 | ARIAD Pharmaceuticals, Inc. | AP23573 | |
| Nair Sensitive Skin | Church & Dwight Co. | Nair Hair Remover Shower Power Sensitive | |
| NOD/SCID mice | Charles River, Inc. | NOD.CB17-Prkdcscid/NCrCrl | |
| Paclitaxel | NOVAPLUS, Inc. | 55390-304-05 | |
| PEG400 | Millipore Sigma, Inc. | 88440-250ML-F | |
| Perjeta | Genetech, Co. | Pertuzumab | |
| Rituximab | Genetech, Co. | Rituxan | |
| RPMI1640 | Mediatech, Inc. | 10-040-CV | |
| SCID mice | Harlan Laboratories, Inc. | C.B.-17/IcrHsd-PrkdcscidLystbg | |
| SLAx 13-6MHz linear transducer | FUJIFILM SonoSite, Inc | HFL38xp | |
| SonoSite S-series Ultrasound machine | FUJIFILM SonoSite, Inc | SonoSite SII | |
| Tween 80 | Millipore Sigma, Inc. | P4780-100ML |
Riferimenti
- Tentler, J. J., et al. Patient-derived tumour xenografts as models for oncology drug development. Nature Reviews Clinical Oncology. 9, 338-350 (2012).
- Marangoni, E., et al. A new model of patient tumor-derived breast cancer xenografts for preclinical assays. Clinical Cancer Research. 13, 3989-3998 (2007).
- Zhang, X., et al. A renewable tissue resource of phenotypically stable, biologically and ethnically diverse, patient-derived human breast cancer xenograft models. Cancer Research. 73, 4885-4897 (2013).
- Hidalgo, M., et al. Patient-derived xenograft models: an emerging platform for translational cancer research. Cancer Discovery. 4, 998-1013 (2014).
- Weroha, S. J., et al. Tumorgrafts as in vivo surrogates for women with ovarian cancer. Clinical Cancer Research. 20, 1288-1297 (2014).
- Rubio-Viqueira, B., et al. Optimizing the development of targeted agents in pancreatic cancer: tumor fine-needle aspiration biopsy as a platform for novel prospective ex vivo drug sensitivity assays. Molecular Cancer Therapeutics. 6, 1079-1088 (2007).
- Rubio-Viqueira, B., Hidalgo, M. Direct in vivo xenograft tumor model for predicting chemotherapeutic drug response in cancer patients. Clinical Pharmacology and Therapeutics. 85, 217-221 (2009).
- Ricci, F., et al. Patient-derived ovarian tumor xenografts recapitulate human clinicopathology and genetic alterations. Cancer Research. 74, 6980-6990 (2014).
- Fleming, J. M., et al. Local regulation of human breast xenograft models. Journal of Cellular Physiology. 224, 795-806 (2010).
- Hoffman, R. M. Patient-derived orthotopic xenografts: better mimic of metastasis than subcutaneous xenografts. Nature Reviews Cancer. 15, 451-452 (2015).
- Jung, J., Seol, H. S., Chang, S. The Generation and Application of Patient-Derived Xenograft Model for Cancer Research. Cancer Research and Treatment. 50, 1-10 (2018).
- Sivanand, S., et al. A validated tumorgraft model reveals activity of dovitinib against renal cell carcinoma. Science Translational Medicine. 4, 137-152 (2012).
- Pavía-Jiménez, A., Tcheuyap, V. T., Brugarolas, J. Establishing a human renal cell carcinoma tumorgraft platform for preclinical drug testing. Nature Protocols. 9, 1848-1859 (2014).
- Fridman, R., Benton, G., Aranoutova, I., Kleinman, H. K., Bonfil, R. D. Increased initiation and growth of tumor cell lines, cancer stem cells and biopsy material in mice using basement membrane matrix protein (Cultrex or Matrigel) co-injection. Nature Protocols. 7, 1138-1144 (2012).
- Cutz, J. C., et al. Establishment in severe combined immunodeficiency mice of subrenal capsule xenografts and transplantable tumor lines from a variety of primary human lung cancers: potential models for studying tumor progression-related changes. Clinical Cancer Research. 12, 4043-4054 (2006).
- Siolas, D., Hannon, G. J. Patient-derived tumor xenografts: transforming clinical samples into mouse models. Cancer Research. 73, 5315-5319 (2013).
- Butler, K. A., et al. Prevention of Human Lymphoproliferative Tumor Formation in Ovarian Cancer Patient-Derived Xenografts. Neoplasia. 19, 628-636 (2017).
- Cao, X., et al. Defective lymphoid development in mice lacking expression of the common cytokine receptor gamma chain. Immunity. 2, 223-238 (1995).
- Dobbin, Z. C., et al. Using heterogeneity of the patient-derived xenograft model to identify the chemoresistant population in ovarian cancer. Oncotarget. 5, 8750-8764 (2014).
- Choi, Y. Y., et al. Establishment and characterisation of patient-derived xenografts as paraclinical models for gastric cancer. Scientific Reports. 6, 22172(2016).
- Malaney, P., Nicosia, S. V., Davé, V. One mouse, one patient paradigm: New avatars of personalized cancer therapy. Cancer Letters. 344, 1-12 (2014).
- Rosfjord, E., Lucas, J., Li, G., Gerber, H. P. Advances in patient-derived tumor xenografts: from target identification to predicting clinical response rates in oncology. Biochemical Pharmacology. 91, 135-143 (2014).
- Braekeveldt, N., Bexell, D. Patient-derived xenografts as preclinical neuroblastoma models. Cell and Tissue Research. 372, 233-243 (2018).
- 'Perez-Soler, R., et al. Response and determinants of sensitivity to paclitaxel in human non-small cell lung cancer tumors heterotransplanted in nude mice. Clinical Cancer Research. 6, 4932-4938 (2000).
- Fichtner, I., et al. Anticancer drug response and expression of molecular markers in early-passage xenotransplanted colon carcinomas. European Journal of Cancer. 40, 298-307 (2004).
- Gao, H., et al. High-throughput screening using patient-derived tumor xenografts to predict clinical trial drug response. Nature Medicine. 21, 1318-1325 (2015).
- Izumchenko, E., et al. Patient-derived xenografts effectively capture responses to oncology therapy in a heterogeneous cohort of patients with solid tumors. Annals of Oncology. 28, 2595-2605 (2017).
- Bertotti, A., et al. A molecularly annotated platform of patient-derived xenografts ("xenopatients") identifies HER2 as an effective therapeutic target in cetuximab-resistant colorectal cancer. Cancer Discovery. 1, 508-523 (2011).
- Mengelbier, L. H., et al. Intratumoral genome diversity parallels progression and predicts outcome in pediatric cancer. Nature Communications. 27, 6125(2015).
- McGranahan, N., Swanton, C. Clonal Heterogeneity and Tumor Evolution: Past, Present, and the Future. Cell. 168, 613-628 (2017).
- Marusyk, A., et al. Non-cell-autonomous driving of tumour growth supports sub-clonal heterogeneity. Nature. 514, 54-58 (2014).
- Braekeveldt, N., et al. Neuroblastoma patient-derived orthotopic xenografts reflect the microenvironmental hallmarks of aggressive patient tumours. Cancer Letters. 375, 384-389 (2016).
- DeRose, Y. S., et al. Tumor grafts derived from women with breast cancer authentically reflect tumor pathology, growth, metastasis and disease outcomes. Nature Medicine. 17, 1514-1520 (2011).
- Das Thakur, M., et al. Modelling vemurafenib resistance in melanoma reveals a strategy to forestall drug resistance. Nature. 494, 251-255 (2013).
- Girotti, M. R., et al. Application of Sequencing, Liquid Biopsies, and Patient-Derived Xenografts for Personalized Medicine in Melanoma. Cancer Discovery. 6, 286-299 (2016).
- Murphy, S. J., et al. Mate pair sequencing of whole-genome-amplified DNA following laser capture microdissection of prostate cancer. DNA Research. 19, 395-406 (2012).
- Smadbeck, J. B., et al. Copy number variant analysis using genome-wide mate-pair sequencing. Genes Chromosomes and Cancer. 57, 459-470 (2018).
- Kovtun, I. V., et al. Lineage relationship of Gleason patterns in Gleason score 7 prostate cancer. Cancer Research. 73, 3275-3284 (2013).
- Paster, E. V., Villines, K. A., Hickman, D. L. Endpoints for mouse abdominal tumor models: refinement of current criteria. Comparative Medicine. 59, 234-241 (2009).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature Methods. 9, 671-675 (2012).
- Cheng, Y., et al. MK-2206, a novel allosteric inhibitor of Akt, synergizes with gefitinib against malignant glioma via modulating both autophagy and apoptosis. Molecular Cancer Therapeutics. 11, 154-164 (2012).
- Rivera, V. M., et al. Ridaforolimus (AP23573; MK-8669), a potent mTOR inhibitor, has broad antitumor activity and can be optimally administered using intermittent dosing regimens. Molecular Cancer Therapeutics. 10, 1059-1071 (2011).
- Heitjan, D. F., Manni, A., Santen, R. J. Statistical analysis of in vivo tumor growth experiments. Cancer Research. 53, 6042-6050 (1993).
- Vargas, R., et al. Case study: patient-derived clear cell adenocarcinoma xenograft model longitudinally predicts treatment response. NPJ Precision Oncology. 2, 14(2018).
- Harris, F. R., et al. Targeting HER2 in patient-derived xenograft ovarian cancer models sensitizes tumors to chemotherapy. Molecular Oncology. 13, 132-152 (2019).
- Fidler, I. J., et al. Modulation of tumor cell response to chemotherapy by the organ environment. Cancer and Metastasis Reviews. 13, 209-222 (1994).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon