Method Article
Purificazione di Human S100A12 e delle sue Oligomeri indotte dallo ione per la stimolazione cellulare immunitaria
In questo articolo
Riepilogo
Questo protocollo descrive un metodo di purificazione per la proteina ricombinante di legame del calcio senza tag S100A12 e i suoi oligomeri indotti dagli ioni per i saggi di stimolazione monocite umana.
Abstract
In questo protocollo, descriviamo un metodo per purificare la proteina che lega il calcio umano S100A12 e i suoi oligomeri indotti dagli oligomeri di Escherichia coli per la stimolazione delle cellule immunitarie. Questo protocollo si basa su una strategia di cromatografia in due fasi, che comprende la pre-purificazione delle proteine su una colonna di cromatografia di scambio di anioni e un successivo passo di lucidatura su una colonna di interazione idrofobica. Questa strategia produce proteine S100A12 ad alta purezza e resa a costi gestibili. Per i test funzionali sulle cellule immunitarie l'eventuale contaminazione da endotossina residua richiede un attento monitoraggio e ulteriori passaggi di pulizia per ottenere proteine prive di endotossina. La maggior parte delle contaminazioni da endotossina può essere esclusa dalla cromatografia di scambio di anioni. Per esaurire le contaminazioni residue, questo protocollo descrive una fase di rimozione con filtri centrifughi. A seconda della forza io-resistenza disponibile S100A12 può organizzare in diversi omomultimer. Per studiare il rapporto tra struttura e funzione, questo protocollo descrive ulteriormente il trattamento io-io della proteina S100A12 seguito dal crosslinking chimico per stabilizzare gli oligomeri S100A12 e la loro successiva separazione per dimensione-esclusione Cromatografia. Infine, descriviamo un saggio basato sulle cellule che conferma l'attività biologica della proteina purificata e conferma la preparazione senza LPS.
Introduzione
S100A12 è una proteina legante di calcio che è prevalentemente prodotta da granulociti umani. La proteina è sovraespressa durante l'infiammazione (sistemica) e i suoi livelli di siero, in particolare nelle malattie (auto)infiammatorie come l'artrite idiopatica giovanile sistemica (sJIA), la febbre mediterranea familiare (FMF) o la malattia di Kawasaki (KD) l'attività della malattia e la risposta alla terapia. A seconda dei recettori di riconoscimento dei modelli (PRR), come i recettori a pedaggio (TLR), il sistema immunitario innato può essere attivato da modelli molecolari associati a i patogeni (PPR) come lipopolioaccharides (LPS) o modelli molecolari associati al danno (DAMP; denominato anche "alarmins"). I DAMP sono molecole endogene come proteine cellulari, lipidi o acidi nucleici1. Le funzioni DAMP sono ben descritte per i membri della famiglia di proteine calgranulina, S100A8/A9 e S100A122, che sono anche segnalati per operare come peptides antimicrobico cheonali metallico divalenteleoni 3,4, 5,6. A seconda della forza iologica disponibile S100A12 può, come altri membri della famiglia S100, organizzare in diversi omomultime e fino a poco tempo fa l'impatto di S100A12-oligomerisation sull'interazione PRR, in particolare TLR4, era sconosciuto.
La forma monomerica della proteina (92 aminoacidi, 10,2 kDa) è costituita da due strutture eliche-loop-eliche a mano eF collegate da un linker flessibile. La mano EF -terminale Ccontiene il classico motivo di rilegatura Ca2,mentre la mano EF -terminale Npresenta una struttura a ciclo esteso specifica della proteina S100 ('pseudo-EF-hand') e rivela una ridotta affinità Ca2. Il legame di Ca2 xda parte di S100A12 può indurre un importante cambiamento conformazionale nel capolinea Cdelle proteine, che si traduce in un'esposizione di una macchia idrofobica su ciascun monomero e forma l'interfaccia di dimerizzazione. Così, in condizioni fisiologiche, la più piccola struttura quaternaria formata da S100A12 è un dimero non covalente (circa 21 kDa) in cui i singoli monomeri sono in orientamento antiparallelo. Una volta disposto come dimero, S100A12 viene segnalato al sequestro di n2 e ad altri ioni metallici divalenti, ad esempio Cu2, con alta affinità7. Questi ioni sono coordinati all'interfaccia dimero S100A12 da aminoacidi H15 e D25 di una sottounità e H85, nonché H89 dell'anti-paralleling di altre sottounità8,9,10. Mentre gli studi precedenti propongono che l'S100A12 caricato dan.2o 2 può indurre l'organizzazione della proteina in omo-tetrameri (44 kDa) e per provocare un aumento dell'affinità Ca2,12, recente titolazione metallica Gli studi6 suggeriscono che il legame Di Ca2 diS100A12 per aumentare l'affinità della proteina conn. 2. Una volta che le lancette EF S100A12 sono completamente occupate da Ca2o, si pensa che ca2 o più si leghi tra i dimeri, innescando la formazione di esamere (circa 63 kDa). L'architettura della struttura trimestrale hexamica è chiaramente diversa da quella del tetramero. Si propone che l'interfaccia tetramer è interrotta per dare origine a nuove interfacce dimer-dimer che beneficiano la formazione di esagomeri10. S100A12 è quasi esclusivamente espresso da granulociti umani dove costituisce circa il 5% di tutte le proteine citosoliche13. Nella sua funzione DAMP S100A12 è stato storicamente descritto come agonista del recettore multi-ligando per prodotti finali di glicazione avanzata (RAGE), poi chiamato extracellulare proteina legante RAGE di recente identificata (EN-RAGE)14. Anche se in precedenza abbiamo segnalato biochimica S100A12-legatura sia rage e TLR415, abbiamo recentemente dimostrato monociti umani per rispondere alla stimolazione S100A12 in modo tramite TLR416. Ciò richiede la disposizione di S100A12 nella suastrutturatrimestrale hexameric indotta da Ca2/ n2.
Qui descriviamo una procedura di purificazione per l'uomo ricombinante S100A12 e le sue oligomeri indotte dagli ioni per le stimolazioni delle cellule immunitarie16,17. Questo si basa su una strategia di cromatografia in due fasi, che inizialmente include una colonna di scambio di anioni per isolare e concentrare la proteina e rimuovere le contaminazioni sfuse (ad esempio, endotossine/lipopolysaccharides)18. Le resine di cromatografia dello scambio io-scie separano le proteine sulla base di diverse cariche nette di superficie. Per le proteine acide come S100A12 (punto isoelettrico di 5.81), un sistema di tampone con un pH di 8,5 e una forte resina di scambio di anion porta ad una buona separazione. Le proteine legate sono state eluite con un gradiente tampone ad alto contenuto di sale. Con un aumento degli ioni negativi di forza ionica nel tampone di eluizione competere con le proteine per le cariche sulla superficie della resina. Le proteine si eludono individualmente a seconda della loro carica netta e in conseguenza di ciò, i tamponi descritti qui permettono di isolare e concentrare la proteina S100A12 sovraespressa. A causa di gruppi caricati negativamente nei lipopolioaccharide, queste molecole si legano anche alle resine di scambio di anioni. Tuttavia, la loro maggiore carica netta si traduce in una successiva eluizione nel gradiente ad alto sale applicato. La seconda fase della procedura di purificazione è stata introdotta a i fini della lucidatura. Questo fa uso della capacità di legame del calcio di S100A12 e rimuove le impurità rimanenti su una colonna di interazione idrofobica. Il legame di calcio di S100A12 porta a un cambiamento conformazionale e ad un'esposizione di macchie idrofobiche sulla superficie della proteina. A tale condizione, S100A12 interagisce con la superficie idrofobica della resina. Al momento del chelante di calcio da parte di EDTA, questa interazione è invertita. In presenza di ioni, in particolare calcio e zinco, S100A12 si organizza in oligomeri ommerici. Per studiare le relazioni struttura-funzione dei diversi oligomeri, abbiamo stabilizzato la cromatoria dimericica, tetramerica e hexamcumso S100A12 con un crosslinker chimico e abbiamo separato i complessi su una colonna cromatografica di dimensioni-esclusione. Infine, per analizzare la funzionalità e l'attività biologica della proteina purificata e dei suoi oligomeri indotti dagli ioni, è possibile confrontare il rilascio di citochina del monocito stimolato S100A12 e del monocito stimolato LPS.
Finora sono stati descritti vari metodi per purificare S100A12. Jackson et al.19, per esempio, ha pubblicato un protocollo con purificazione tramite una colonna di scambio di anion e una successiva cromatografia di esclusione di dimensioni. La purificazione in una colonna di esclusione delle dimensioni porta a buoni risultati, ma, ad esempio a causa dei volumi di carico limitati, è meno flessibile in termini di scalabilità. Un approccio diverso, pubblicato da Kiss et al.20, descrive la purificazione delle proteine marcate tramite la colonna di affinità Ni2 come primo passaggio di purificazione, seguito da scissione enzimatica per rimuovere il tag e ulteriori passaggi di purificazione. In contrasto con gli studi foriciti19,20, la proteina prodotta come descritto in questo protocollo è determinata per esperimenti sulle cellule immunitarie. Pertanto, la contaminazione da endotossina residua dalla coltura batterica è una sfida. Anche se finora sono stati descritti diversi approcci per la rimozione dell'endotossina, non esiste un metodo uniforme che funzioni altrettanto bene per una determinata soluzione proteica21,22.
In sintesi, il nostro protocollo combina i vantaggi di un'espressione senza tag in un sistema batterico con un'efficiente rimozione dell'endotossina e un alto rendimento di proteine pure.
Protocollo
NOT: Si prega di fare riferimento alla tabella supplementare 1 per la preparazione di buffer e soluzioni di stock.
1. Espressione proteica in E. coli
-
Clonazione
- Clona l'uomo s100A12 (ncBI Reference Sequence: NP_005612.1) in un vettore di espressione batterica pET11b. Per esprimere la proteina, trasformare il costrutto in E. coli BL21(DE3).
-
cultura
- Preparare una coltura di avviamento inoculando una singola colonia in 5 mL di mezzo di crescita (brodo LB con 100 ampicillina g/mL) in un tubo rotondo da 14 mL. Incubare pernottamento a 37 gradi centigradi con agitazione a 220 giri/min. Trasferire 2/4 mL di coltura overnight in 400 mL di mezzo di crescita in un flacone di Erlenmayer 2 L e incubare la coltura a 37 gradi centigradi con agitazione a 220 rpm.
NOT: La densità iniziale della coltura principale deve essere densità ottica a 600 nm (OD600) - 0,1. - Monitorare l'OD600 durante la crescita. Indurre l'espressione proteica con l'aggiunta di 1 M isopropile--D-Thiogalactopyranosid (IPTG) ad una concentrazione finale di 1 mM aOD 600 - 0,5-0,6. Incubare a 37 e 220 giri/min per ulteriori 4 h.
NOT: In generale, un OD600 di 0,6 sarà raggiunto dopo 1,5,2,5 h a 37 gradi centigradi. - Preparare 50 mL di sonicazione buffer sciogliendo 50 mM MM Tris, 50 mM NaCl e 1 mM di acido tetraacetico (EDTA) in 40 mL di acqua deionizzata. Regolare il pH con HCl a 8,0 e fare fino a 50 mL. Aggiungere l'inibitore della proteasi (1 compressa per soluzione da 50 mL) e far eclimare il tampone a 4 gradi centigradi.
- Trasferire la coltura batterica in bottiglie di centrifuga e raccogliere le cellule a 3.200 x g per 30 min a 4 gradi centigradi. Scartare il supernatante e risospendere il pellet in 25 mL di cuscinetto di sonicazione a freddo ghiaccio. D'ora in poi tenere le cellule sul ghiaccio.
NOTA: Le celle risospese possono essere conservate a -20 gradi centigradi per breve termine e a -80 gradi centigradi a lungo termine.
- Preparare una coltura di avviamento inoculando una singola colonia in 5 mL di mezzo di crescita (brodo LB con 100 ampicillina g/mL) in un tubo rotondo da 14 mL. Incubare pernottamento a 37 gradi centigradi con agitazione a 220 giri/min. Trasferire 2/4 mL di coltura overnight in 400 mL di mezzo di crescita in un flacone di Erlenmayer 2 L e incubare la coltura a 37 gradi centigradi con agitazione a 220 rpm.
-
Sonicazione/lisi
- Sonicare le cellule per 6 cicli di 30 s sul ghiaccio. Dopo ogni ciclo, riposare le cellule per 30-60 s per proteggere le cellule dal surriscaldamento.
- Trasferire la sospensione cellulare in un tubo di centrifugazione ad alta velocità pre-raffreddato da 50 mL e centrifugare in un rotore ad angolo fisso a 15.000 x g per 30 min a 4 gradi centigradi. Decant il lisato eliminato che contiene le proteine citosoliche solubili in un tubo fresco da 50 mL e scarti il pellet.
2. Purificazione delle proteine
- Cromatografia per lo scambio di anioni
- dialisi f
- Preparare il buffer A per lo scambio di anion -exchange (AIEX) sciogliendo 20 mM Tris, 1 mM EDTA e 1 mM di glicole etilene(2-aminoethylether)-N,N'N'N',N'-tetraacetica (EGTA) in acqua deionizzata e il pH a 8,5 con HCl. Per la dialisi preparare 2 x 5 L e per la cromatografia 2x 1 L del buffer A A IEX.
NOT: Il volume di dialisato deve essere di circa 100 volte il volume del campione. Tutti i buffer utilizzati per la cromatografia devono essere filtrati (0,45 m o più piccoli) e degassed (ad esempio, dal bagno ad ultrasuoni o dalla degassamento del vuoto). - Tagliare i tubi di dialisi (peso molecolare cut-off [MWCO]: 3.5 kDa) in una lunghezza appropriata con spazio aggiuntivo per l'aria per garantire la galleggiabilità del campione sopra la barra di stirazione rotante.
NOT: Glicerolo conserva la membrana e deve essere rimosso prima dell'uso. - Per ridurre la viscosità della soluzione proteica cancellata dal passaggio 1.3.2, diluire la soluzione con 25 mL di tampone A A per facilitare la successiva applicazione alla colonna di cromatografia. Fissare la prima chiusura sul tubo, caricare il campione nella membrana e fissare la seconda chiusura ad almeno 1 cm dall'estremità superiore del tubo.
- Mettere il contenitore 5 L con tampone A A su una piastra di agitazione, aggiungere una barra di agitazione e la membrana riempita con soluzione proteica. Regolare la velocità per ruotare il campione evitando interferenze con la barra di agitazione rotante. Dialyze per 12-24 h a 4 gradi centigradi, quindi sostituite il tampone di dialisate (tampone A A di AIEX) con una nuova preparazione pre-raffreddata e continuate per almeno 4 ore aggiuntive. Trasferire la soluzione proteica dialisata in un tubo da 50 mL e filtrare attraverso un'unità di filtro di 0,45 m.
NOT: Stoccaggio possibile.
- Preparare il buffer A per lo scambio di anion -exchange (AIEX) sciogliendo 20 mM Tris, 1 mM EDTA e 1 mM di glicole etilene(2-aminoethylether)-N,N'N'N',N'-tetraacetica (EGTA) in acqua deionizzata e il pH a 8,5 con HCl. Per la dialisi preparare 2 x 5 L e per la cromatografia 2x 1 L del buffer A A IEX.
- Cromatografia
- Avviare il sistema di cromatografia liquida (FPLC) con manutenzione generale, collegare i buffer di colonna AIEX A e AIEX B (tamponi AIEX A con 1 M NaCl) e la colonna contenente resina anion-exchange. Fare riferimento alla Tabella 1 per i parametri cromatografici generali.
NOTA: le apparecchiature Buffer, colonna e FPLC devono essere eclabili alla stessa temperatura prima di iniziare la corsa (fare riferimento ai parametri cromatografici nella tabella 1, tabella 2, tabella 3, tabella 4e tabella 5). - Evitare la colonna con il tampone A IIEX, caricare successivamente il campione sulla colonna ed eluire le proteine con un gradiente lineare compreso tra 0% e 100% di cuscinetto ad alto sale (AIEX B). Fare riferimento alla tabella 2 per un protocollo del metodo dettagliato.
- Raccogliere 2 frazioni mL durante l'eluizione e analizzare 10 l una di ogni frazione su un'elettroforesi da solfato di solfato di sodio al solfato di sodio polyacrilio (SDS-PAGE) macchiata di Coomassie. Mettere in comune le frazioni contenenti la proteina S100A12 per la dialisi.
NOT: Il peso molecolare di S100A12 è 10,575 Da.
- dialisi f
- Cromatografia di interazione idrofobica (HIC) dipendente dal calcio
- dialisi f
- Diallyze la soluzione proteica contro 20 mM Tris, 140 mM NaCl, pH 7.5 seguendo la procedura descritta nella sezione 2.1.1.
- Cromatografia
- Preparare 1 L di tampone cromatografia HIC A sciogliendo 20 mM Tris, 140 mM NaCl e 25 mMCaCl2 in acqua deionizzata e regolare il pH a 7.5. Per il buffer HIC B, dissolvere 20 mM Tris, 140 mM NaCl e 50 mM EDTA. Regolare il pH a 7.0 e filtrare e degas i buffer. Aggiungete CaCl2 al campione a una concentrazione finale di 25 m equilibratm e filtrate fino a 0,45 m e campionate a 4 gradi (temperatura di colonna).
- Avviare il sistema di cromatografia liquida con manutenzione generale, collegare i buffer di colonna HIC A e B e la colonna. Fare riferimento alla tabella 3 per ulteriori parametri cromatografici.
- Equilibrate la colonna, caricate il campione ed estendete il blocco "lavare il campione non legato" fino a quando il segnale UV non raggiunge nuovamente il livello di base. Quindi iniziare l'eluzione con un chelante di calcio contenente tampone (EDTA). Fare riferimento alla tabella 4 per un protocollo del metodo dettagliato.
NOT: Esperimenti precedenti hanno dimostrato che un eccesso di calcio sembra essere utile per legare S100A12 alla resina di cromatografia. - Raccogliere frazioni di picco di 2 mL e analizzare 10 l di ogni frazione su un SDS-PAGE color Coomassie. Pool di frazioni S100A12 pure e diallyze contro la salina tampone di epes (HBS; 20 mM Hepes, 140 mM NaCl, pH 7.0) come descritto nella sezione 2.1.1.
NOTA: Coefficiente di estinzione di monomerico S100A12 è 2980 M-1 cm-1.
- dialisi f
3. Rilevamento e rimozione dell'endotossina
-
Rilevamento di endotossina
- Per determinare la contaminazione da endotossina, misurare le concentrazioni di proteine diluite dal punto 2.2.2.4. (ad esempio, 1:10 e 1:100 in HBS) utilizzando un saggio di qualità dell'immunosorbent (ELISA) e/o enzimana (ELISA) basato su un saggio di rilevamento dell'endotossina (Tabella dei materiali). Eseguire questo saggio seguendo il protocollo del produttore.
NOT: Utilizzare soluzioni HBS appena preparate disciolte in acqua ultrapura deionizzata per evitare (nuova) contaminazione da endotossina dal tampone.
- Per determinare la contaminazione da endotossina, misurare le concentrazioni di proteine diluite dal punto 2.2.2.4. (ad esempio, 1:10 e 1:100 in HBS) utilizzando un saggio di qualità dell'immunosorbent (ELISA) e/o enzimana (ELISA) basato su un saggio di rilevamento dell'endotossina (Tabella dei materiali). Eseguire questo saggio seguendo il protocollo del produttore.
-
Rimozione dell'endotossina e concentrazione di proteine
- Caricare 15 mL di campione su un 50 kDafugal filter unit e centrifugare a 3.200 x g e 10 C per circa 10 min. Trasferire il flow-through in un vaso fresco (sul ghiaccio) e ricaricare e centrifugare il tubo filtrante 50 kDa tutte le volte che è necessario. Lavare la membrana del filtro due volte con HBS per recuperare quanta più proteine possibile dopo ogni passaggio.
- Concentrate il flusso-attraverso S100A12 utilizzando un filtro centrifuga di 3 kDafugal fino a quando il volume non viene ridotto a un quinto fino a un decimo del volume di carico iniziale (centrifugazione a 3.200 x g, 10 gradi centigradi per circa 30 min). Riempire il filtro tutte le volte che è necessario, risciacquare la membrana e trasferire la soluzione concentrata in un nuovo tubo dopo ogni ricarica. Eliminare il flusso attraverso. Filtrare di nuovo attraverso 50 kDa come descritto sopra.
NOT: Durante questa procedura, la perdita di proteine è notevole (fino al 50%), ma la preparazione rimanente delle proteine è completamente esaurita da LPS. Questo metodo produce circa 10-15 mg di proteine dalla coltura 400 mL. - Regola la soluzione proteica a 1 mg/mL con HBS privo di endotossina e misura il contenuto di LPS come descritto al punto 3.1.1. Nel caso in cui la soluzione proteica non sia ancora testata come l'uso di LPS (<0.1 EU/mL), eliminare le contaminazioni residue utilizzando una resina di rimozione dell'endotossina.
NOT: Con una concentrazione proteica di 1 mg/mL, la contaminazione di 0,1 EU/mL LPS equivale a circa 0,01 pg Proteina LPS/g.
4. Separazione chimica crosslink e oligomera
-
Intercollegamento chimico
- Preparare soluzioni di stock altamente pure (endotossina-free) di 1 M CaCl2 e 100 mM snCl2 in acqua diionizzata ultrapura (Tabella dei materiali). Utilizzare questo buffer, appena fatto, per il passaggio successivo.
- Incubare 10 mL di S100A12 depurazione endotossina (concentrazione 1 mg/mL in HBS) per 30 min a temperatura ambiente (RT) con 25 mM CaCl2 per dimerico/tetramerico, o 25 mM CaCl2 e 1 mM snCl2 per l'esameerico/tetrame S100A12 Oligomeri.
- Preparare il crosslinker sciogliendo 8 mg di BS3 in 500 -L di acqua endotossina senza endotossina direttamente prima dell'uso (8 mg di crosslinker per una soluzione proteica iospiatoda da 10 mL equivale a una concentrazione finale di 1,4 mM). Mescolare il crosslinker e il campionamento con il pipettaggio e incubare per ulteriori 30 min a RT. Quench la reazione aggiungendo 1 M Tris-HCl, pH 7,5 ad una concentrazione finale di 50 mM e filtrare fino a 0,45m.
-
Cromatografia ad esclusione di dimensioni
- Eri il campione incrociato a 12-15 gradi (temperatura di colonna) e inizia il sistema di cromatografia liquida con manutenzione generale. Connettere il buffer di colonna (HBS) e la colonna size-exclusion. Per informazioni dettagliate, fare riferimento alla tabella 5.
- Egli la colonna in HBS, caricare il campione e raccogliere le frazioni di picco (1-2 mL) durante la corsa. Analizzate queste frazioni su un gradiente di 4,20% SDS-PAGE e raggruppate frazioni con le bande principali del complesso proteico desiderato.
NOT: L'idrolisi dei reagenti dell'esterinazione NHS come BS3 in soluzioni acquose si traduce in una forte assorbimento a 280 nm. Il crosslinker non legato (peso molecolare: 572 g/mol) si eluisce alla fine della corsa e si traduce in un forte picco. - Concentrare le soluzioni utilizzando unità filtro centrifughe con MWC di 10 kDa (dimero), 30 kDa (tetramer) o 50 kDa (hexamer). Determinare la contaminazione da endotossina come descritto nella sezione 3.1. Se necessario, rimuovere gli LPS rimanenti con una resina di rimozione dell'endotossina seguendo le raccomandazioni del produttore (Tabella dei materiali).
5. Test funzionali sui monociti
- Preparazione di monociti
- Isolare i monociti dai cappotti di buffy umani mediante la centrifugazione del gradiente di densità e il successivo arricchimento dei monociti utilizzando un kit magnetico di separazione del tallone (Tabella dei materiali).
NOT: Questo protocollo si tradurrà in circa 5-7 x 107 monociti (un cappotto di bufala) con una purezza di 83-95%. Poiché il numero, ma anche la reattività delle cellule dipende fortemente dal donatore, il protocollo potrebbe dover essere aumentato (a seconda del numero di cellule richiesto). - Per la centrifugazione a densità, equilibrate la soluzione di separazione (densità 1,077 g/mL) a RT e trasferite 20 mL in tubi centrifugati da 50 mL (2 tubi per cappotto di bufala). Diluire il sangue dal cappotto di bufalo umano con la soluzione di sale tampone di Hank (HBSS) per un volume totale di 60 mL e strato 30 mL di questa miscela con attenzione sulla parte superiore del mezzo di separazione. Centrifuga a 550 x g per 35 min a RT. Disattivare il freno centrifuga.
- Dopo la centrifugazione, le cellule del sangue periferiche mononucleari (PBMC) si trovano direttamente sopra il mezzo di separazione. Trasferire queste cellule in un tubo di centrifuga fresco da 50 mL, fino a 50 mL con HBSS e centrifugare a 170 x g per 10 m. Aspirata il supernatante e rispendere il pellet cellulare in un piccolo volume di HBSS tramite pipettaggio.
- Riempire il tubo fino a 50 mL e centrifugare a 290 x g per 10 min. Aspirate il supernatante di nuovo, rispendere le cellule in HBSS (50 mL) e centrifuga a 170 x g per 10 min. Contare le cellule e risospende nel buffer di separazione delle celle (Tabella dei materiali. /c9>) a una concentrazione di 5 x 107 celle/mL.
NOT: Invece di HBSS, la salina (PBS) con buffer fosfato può essere utilizzata per lavare le cellule. - Per l'isolamento dei monociti dai PBMC, utilizzare un kit di isolamento cellulare magnetico negativo e seguire il protocollo del produttore. Contare i monociti e risospendere in mezzo monocito (RPMI 1640, 15% siero di vitello fetale inattivato dal calore [FCS], 4 mM L-glutamina, 100 U/mL penicillina/streptomicina) ad una concentrazione di 2 x 106 cellule/mL.
- Ai monociti della coltura, i piatti di coltura del mantello (ad esempio, 100 mm) con pellicola idrofobica e permeabile al gas, adatti alle celle di sospensione (Tabella dei materiali). Sterilizzare le piastre utilizzando la luce UV per circa 30 min. Trasferire le cellule in queste piastre di coltura e lasciarle riposare per tutta la notte a 37 e 5% CO2.
NOT: Utilizzare 15-25 mL di sospensioni cellulari per piatto rivestito.
- Isolare i monociti dai cappotti di buffy umani mediante la centrifugazione del gradiente di densità e il successivo arricchimento dei monociti utilizzando un kit magnetico di separazione del tallone (Tabella dei materiali).
- Stimolazione monociita
- Stimolazione con S100A12 (wildtype)
NOT: Per distinguere l'S100A12 non trattato (prodotto finale dalla sezione 2.2.2) dalla proteina crosslinked, S100A12 è indicato come "wildtype".- Trasferire le cellule riposate in un tubo centrifugato da 50 mL e centrifugare a 350 x g per 10 min. Aspirate il supernatante e risospendere il pellet cellulare in mezzo di stimolazione (RPMI 1640, 5% FCS inattivato dal calore, 4 mM L-glutamine, 100 U/mL penicillina/ streptomicina) ad una concentrazione di 2 x 106 cellule/mL.
- Per la stimolazione, utilizzare 24 piastre di sospensione ben e aggiungere 250 l di sospensione cellulare per pozzo (0,5 x 106 cellule/ pozzo). Aggiungete 50 polimyxinB ai pozzi desiderati, seguiti da LPS in concentrazioni diverse (25, 50, 100 e 200 pg/mL) o dal tipo selvaggio S100A12 (10, 20, 40, 60 g/mL). Inoltre, applicare la proteina non trattata o denaturata al calore (99 , 10 min) in diverse concentrazioni alle cellule.
NOT: Un breve trattamento termico denatura la proteina S100A12 ma non ha alcun effetto sull'LPS. - Incubare piastre per 4 h a 37 e 5% DI CO2. Raccogliere le cellule trasferendo la sospensione cellulare di ogni pozzo a tubi di reazione 1,5 mL. Centrifuga a 500 x g per 10 min. Trasferire i supernatanti in tubi freschi e misurare il rilascio di TNF, in diverse diluizioni (ad esempio, 1:2, 1:5, 1:10) con un kit tNF-ELISA umano seguendo le raccomandazioni del produttore.
- Stimolazione con oligomeri S100A12
- Preparare e semiare monociti in 24 piastre di sospensione ben come descritto sopra. Stimolare le cellule aggiungendo oligomeri S100A12 dal punto 4.2.3. in diverse concentrazioni molare (125 nM, 250 nM, 500 nM, 1000 nM).
NOT: Per confrontare le capacità dei diversi oligomeri per stimolare i monociti, gli oligomeri sono stati applicati alle cellule in concentrazioni molare comparabili. - Incubare per 4 h a 37 e 5% di CO2,raccogliere le cellule e misurare il rilascio di TNF, come descritto sopra.
- Preparare e semiare monociti in 24 piastre di sospensione ben come descritto sopra. Stimolare le cellule aggiungendo oligomeri S100A12 dal punto 4.2.3. in diverse concentrazioni molare (125 nM, 250 nM, 500 nM, 1000 nM).
- Stimolazione con S100A12 (wildtype)
Risultati
In seguito alla pre-purificazione sulla colonna AIEX (Figura 1A-C) e al successivo HIC dipendente dal calcio (Figura 2A,B), sono state ottenute proteine altamente pure (Figura 2C). Inoltre, le misurazioni dell'endotossina hanno rivelato un successo della rimozione degli LPS. Il contenuto LPS seguito all'AIEX è stato misurato in una diluizione 1:10 superiore al limite di rilevamento degli saggi, vale a dire superiore a 500 EU/mL. Dopo la prima filtrazione attraverso un'unità di filtro da 50 kDa, il contenuto di LPS è stato ridotto a 1 EU/mL. Dopo la concentrazione con un'unità filtrante da 3 kDa e una filtrazione aggiuntiva attraverso 50 kDa, la contaminazione misurata da LPS è stata di 0,08 EU/mL.
Come ulteriore controllo, i monociti umani sono stati stimolati con la proteina wildtype prodotta (Figura 3A,B). Il trattamento della polimysina B abroga il rilascio di TNF da monociti stimolati da LPS, che non possono essere osservati con S100A12. D'altra parte, il trattamento termico di LPS e S100A12 abolisce la capacità della proteina di stimolare le cellule, mentre questo non influisce sulla risposta cellulare alla stimolazione LPS.
L'esposizione proteica a diversi ioni comporta la disposizione di diversi oligomeri S100A12 (Figura 4A). Il collegamento chimico permette di catturare complessi definiti come dimer, tetrameri e argher, nonché stati di transizione (ad esempio, "trimer", banda a circa 30 kDa). Per indurre uno spostamento pronunciato dell'equilibrio oligomero prima del crosslinking, è stato applicato un eccesso di ioni (Figura 4B).
Gli oligomeri isolati in concentrazioni molare uguali(Figura 5A-C) sono stati poi utilizzati per la stimolazione monocitica per confrontare le capacità di segnalazione tramite PRR. Moncyte-stimolazione con hexameric S100A12 ha provocato un rilascio pronunciato di TNF (Figura 6 ). Il rilascio di citochine residua potrebbe essere rilevato da cellule stimolate con tetrameria S100A12, mentre il trattamento con proteina dimeric non induce il rilascio di TNF.

Figura 1: Risultati della cromatografia dello scambio di anioni. (A) Un cromatogramma con assorbimento a 280 nm (A280) e la percentuale di buffer di eluizione B (linea tratteggiata). I blocchi di metodi sono indicati con A - lavare il campione non legato, B , gradiente lineare con buffer di eluizione (buffer B), C e lavarsi con il buffer B e D - ri-bilanciamento nel buffer A. (B) Concentrarsi sui picchi rilevanti con i numeri dei tubi di frazione in rosso. (C) Le frazioni selezionate sono state analizzate su SDS-PAGE macchiato di Coomassie al 15%. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Risultati della cromatografia di interazione idrofobica. (A) Un cromatogramma con assorbimento a 280 nm (A280) e la percentuale di buffer di eluizione B (linea tratteggiata). I blocchi di metodi sono indicati con A - lavare il campione non legato, B eluizione con il buffer B e C - ri-equilibration nel buffer A. (B) Focus sui picchi pertinenti con i numeri dei tubi di frazione in rosso. (C) Frazioni analizzate su SDS-PAGE macchiate di Coomassie. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: I monociti umani primari sono stati stimolati a concentrazioni indicate. LPS (A) o S100A12 (wildtype, B) sono stati lasciati non trattati o denaturati a caldo (99 o C, 10 min). Entrambe le condizioni sono state testate in presenza e assenza di polimysina B. Fare clic qui per visualizzare una versione più grande di questa figura.
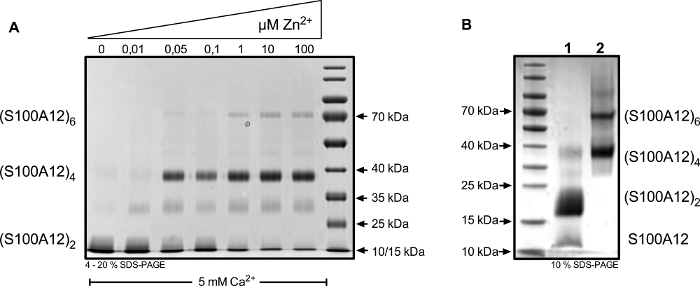
Figura 4: La proteina S100A12 è stata collegata con BS3 dopo l'incubazione nel buffer HBS contenente 5 mM Ca2 e ha indicato le concentrazioni di n2. (A) L'aumento delle concentrazioni din. 2 o n induce la disposizione di S100A12 in tetrameri e hexamers dopo la separazione su 4-20% di Coomassie-colorato SDS-PAGE. (B) Risultato rappresentativo di oligomeri interconnessi con condizioni utilizzate per la separazione in una colonna di esclusione delle dimensioni. S100A12 è stato collegato in presenza di 25 mM Ca2 o 25 mM Ca2 e 1 mM .n2 (corsia 2). (S100A12) 2 - dimero; (S100A12) 4 - tetramero; (S100A12) 6 - esagono. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: gli oligomeri S100A12 sono stati separati in una colonna di esclusione delle dimensioni. (A) Cromatogramma di separazione esamera/tetramero dopo il collegamento incrociato nel buffer HBS con 25 mM CaCl2 e 1 mM snCl2. (B) Cromatogramma per la separazione tetramer/dimero nel buffer HBS con 25 mM CaCl2. (C) Esempio di oligomeri raggruppati e concentrati dopo la separazione su un gradiente SDS-PAGE macchiato di Coomassie. Corsia 1 - dimero; corsia 2 - tetramer; corsia 3 - esameratore. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 6: Stimolazione dei monociti con oligomeri S100A12 purificati. Il rilascio della TNF dopo l'incubazione di 4 h è stato quantificato da ELISA. I dati mostrano il valore medio di due esperimenti indipendenti. Fare clic qui per visualizzare una versione più grande di questa figura.
| Volume letto (CV) | 75 mL |
| monitor | Assorbimento a 280 nM |
| Pressione Max | 3 bar |
| Buffer di colonna A | 20 mM Tris-HCl, 1 mM EDTA, 1 mM EGTA, pH 8.5 |
| Buffer di colonna B | 20 mM Tris-HCl, 1 mM EDTA, 1 mM EGTA, 1 M NaCl, pH 8.5 |
| Volume campione | variabile |
| Portata | 1/2 mL/min |
| temperatura | 4 gradi centigradi |
Tabella 1: Informazioni dettagliate sui parametri applicati della cromatografia di scambio di anioni.
| isolato | volume | buffer m inv | negozio |
| Equilibratura | Volumi di colonne (CV) | un | spreco |
| Carico campione | non pertinente | un | spreco |
| Estrarre il campione non associato | 1 CV | un | Presa ad alto volume |
| Sfumatura -Elusione | 0 -100 % Buffer B in 1 CV | Da A a B | Collezionista di frazioni |
| Lavaggio: buffer B | 1 CV | B | spreco |
| Ri-Equilibration | 2 CV | un | spreco |
Tabella 2: Informazioni dettagliate sul metodo usato della cromatografia di scambio di anioni.
| Volume letto (CV) | 125 mL |
| monitor | Assorbimento a 280 nM |
| Pressione Max | 4 barre |
| Buffer di colonna A | 20 mM Tris, 140 mM NaCl, 25 mM CaCl2, pH 7,5 |
| Buffer di colonna B | 20 mM Tris, 140 mM NaCl, 50 mM EDTA, pH 7,5 |
| Volume campione | variabile |
| Portata | 1/2 mL/min |
| temperatura | 4 gradi centigradi |
Tabella 3: Informazioni dettagliate sui parametri applicati della cromatografia dell'interazione idrofobica.
| isolato | volume | buffer m inv | negozio |
| Equilibratura | Volumi di colonne (CV) | un | spreco |
| Carico campione | non pertinente | un | spreco |
| Estrarre il campione non associato | 1/2 CV | un | Presa ad alto volume |
| Eluizione | Buffer B al 100% | B | Collezionista di frazioni |
| Lavaggio: buffer B | 1 CV | B | spreco |
| Ri-Equilibration | 2 CV | un | spreco |
Tabella 4: Informazioni dettagliate sul metodo usato della cromatografia di interazione idrofobica.
| Volume letto (CV) | 320 mL |
| monitor | Absorbance presso 280 nm |
| Pressione Max | 3 bar |
| Buffer di colonna A | 20 mM di epes, 140 mM NaCl, pH 7.2 |
| Volume campione | Fino a 13 mL |
| Portata | 1/1,5 mL/min |
| temperatura | 12/15 gradi centigradi |
Tabella 5: Informazioni dettagliate sui parametri applicati della cromatografia dimensione-esclusione.
Tabella supplementare 1: Preparazione di buffer e soluzioni azionarie. Fare clic qui per scaricare questo file.
Discussione
In questo protocollo, descriviamo l'espressione batterica priva di tag dell'uomo S100A12 e la sua purificazione, nonché la separazione in diversi oligomeri indotti dagli ioni per la stimolazione delle cellule immunitarie. Rispetto alla letteratura pubblicata sulla purificazione della proteina S100A128,23,24, l'uso dell'alto CaCl2 (25 mM) nella cromatografia di interazione idrofobica è a nostra conoscenza unica. Diversi protocolli che applicano concentrazioni da 1 a 5 mM producono proteine pure, ma abbiamo osservato una resa diverse volte maggiore seguendo il nostro approccio utilizzando 25 mM CaCl2 invece. Ciò potrebbe essere spiegato da una gerarchia di interazione proteica con il materiale della colonna:S100A12 può legarsi direttamente al materiale della colonna, ma l'eccesso di Ca2' può anche facilitare il legame indiretto dei dimeri S100A12 alla proteina già associata a colonne 8. Così, alte concentrazioni di Ca2 o più possono ingrandire la superficie disponibile per la purificazione S100A12. L'eludio (utilizzando un gradiente lineare) di S100A12 da HIC come uno precoce (indirettamente legato S100A12) e un picco molto tardivo (proteina associata al materiale colonna) possono supportare questa speculazione (dati non mostrati).
Per la produzione di S100A12 ricombinante (così come altre proteine) ad alte rese e costi gestibili, l'espressione proteica in E. coli è ancora il metodo di scelta. Tuttavia, l'inevitabile contaminazione con endotossine batteriche rimane un problema, quando le proteine sono determinate per gli esperimenti di coltura cellulare, in particolare negli studi che coinvolgono cellule immunitarie innate. Per quanto riguarda la nostra esperienza, anche le proteine esplicitamente dichiarate in commercio per l'uso della coltura cellulare possono contenere contaminazioni da endotossina fino a 1 proteina dell'UE/g, che può distorcere significativamente i saggi. Pertanto, una rimozione completa delle endotossine è obbligatoria. I momomeri di endotossina in soluzione variano da pesi molecolari da 10 a 20 kDa, ma possono formare micelle e strutture con pesi molecolari più elevati. La formazione di strutture molto grandi è, ad esempio, promossa attraverso ioni bivalenti21,25.
Secondo il nostro protocollo, verifichiamo la produzione senza endotossina della proteina S100A12 combinando misurazioni di endotossina ad alta sensibilità con analisi di stimolazione monocitica. Consideriamo tale combinazione particolarmente significativa come a) la contaminazione da endotossina di basso livello può essere difficile da valutare a seconda della sensibilità dell'analisi e b) l'uso della polimysina B come inibitore LPS sui monociti può comportare difficile interpretazione dei dati dovuti agli effetti esclusivi di polimyssina B sulle cellule26,27. Polimyssina B così come altri peptidi cationici sono segnalati per legare LPS tramite lipidico caricato negativamente A28. Poiché la superficie esposta a solvente di S100A12 contiene anche grandi patch caricate negativamente, la riduzione osservata del rilascio di TNF z da monociti umani stimolati da S100A12 in presenza di polimyxina B(Figura 3B) può essere dovuta a un) rilegatura diretta non specifica di polimyxin B a S100A12 e/o b) effetti diretti della polimysina B sulle cellule stimolate26,27. A causa delle limitazioni note del rilevamento sia della contaminazione da endotossina a basso livello che degli effetti non specifici della polimyssina B, il nostro protocollo contiene ulteriormente una fase di inattivazione del calore per distinguere chiaramente tra la segnalazione TLR4 mediata da LPS e proteina.
L'uso di S100A12 senza LPS per la generazione e la purificazione di oligomeri definiti indotti da ioni è fondamentale e si dovrebbe prestare particolare attenzione alla loro successiva purificazione per evitare l'eventuale reintroduzione dell'endotossina tramite tamponi o materiale colonnae quindi ulteriore esaurimento di LPS che richiede proteine tramite resine di rimozione dell'endotossina.
La rilevanza dell'oligomerizzazione per la funzione biologica delle proteine può essere valutata con diversi mezzi. Nel caso di S100A12, abbiamo utilizzato la risonanza del plasmone superficiale e gli scambi mirati di amminoacidi nei siti di legame agli ioni e, per definire con maggiore precisione la proteina-complessa, in grado di legare e segnalare attraverso il TLR4, abbiamo impiegato il crosslink chimico di Ca2 -pulsed ricombinante S100A1216. Il crosslinking chimico di S100A12 in diverse condizioni ioniche blocca uno stato momentaneo che include diverse forme oligomeriche in transizione. Dagli esperimenti sulla titolazione degli ioni, abbiamo definito le condizioni in cui gli oligomeri dimerici, tetramerici o hexameric potevano essere determinati come oligomeri predominanti16. Inoltre, esperimenti precedenti hanno dimostrato che un eccesso di ioni è vantaggioso per un crosslinking stabile e comparabile e per la successiva purificazione, anche se l'oligomerizzazione può anche essere indotta a concentrazioni di ioni significativamente più basse. Tuttavia, purificando questi oligomeri per cromatografia di dimensioni-esclusione si traduce in una buona, ma non assoluta separazione. Tuttavia, l'arricchimento selettivo degli oligomeri consente analisi a valle affidabili.
In sintesi, questo protocollo fornisce un metodo per la purificazione dell'Uomo S100A12 senza LPS o delle proteine leganti di calcio correlate. Per fissare i cambiamenti conformazionali indotti dagli ioni, il crosslink chimico e la successiva separazione complessa per cromatografia di esclusione di dimensioni è uno strumento utile per comprendere la rilevanza dell'oligomerizzazione delle proteine per i processi biologici a valle.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo studio è stato sostenuto da sovvenzioni del programma di ricerca medica innovativo intramurale della facoltà di medicina dell'Università Muenster (da KE121201 a C.K.) e della Fondazione tedesca per la ricerca (DFG, Fo354/3-1 a D.F.).
Materiali
| Name | Company | Catalog Number | Comments |
| pET11b vector | Novagen | ||
| BL21(DE3) competent E. coli | New England Biolabs | C2527 | |
| 100x Non-essential amino acids | Merck | K 0293 | |
| 25% HCl | Carl Roth | X897.1 | |
| 4−20% Mini-PROTEAN TGX Protein Gels | BioRad | 4561093 | |
| Ampicillin sodium salt | Carl Roth | HP62.1 | |
| BS3 (bis(sulfosuccinimidyl)suberate) - 50 mg | ThermoFisher Scientific | 21580 | |
| Calciumchlorid Dihydrat | Carl Roth | 5239.1 | |
| Coomassie Briliant Blue R250 Destaining Solution | BioRad | 1610438 | |
| Coomassie Briliant Blue R250 Staining Solution | BioRad | 1610436 | |
| EasySep Human Monocyte Enrichment Kit | Stemcell | 19059 | Magnetic negative cell isolation kit |
| EDTA disodium salt dihydrate | Carl Roth | 8043.1 | |
| EGTA | Carl Roth | 3054.3 | |
| EndoLISA | Hyglos | 609033 | Endotoxin detection assay |
| Endotoxin-Free Ultra Pure Water | Sigma-Aldrich | TMS-011-A | Ultrapure water for preparation of endotoxin-free buffers |
| EndoTrap red | Hyglos | 321063 | Endotoxin removal resin |
| FBS (heat-inactivated) | Gibco | 10270 | |
| HBSS, no calcium, no magnesium | ThermoFisher Scientific | 14175053 | |
| Hepes | Carl Roth | 9105 | |
| Hepes (high quality, endotoxin testet) | Sigma-Aldrich | H4034 | |
| hTNF-alpha - OptEia ELISA Set | BD | 555212 | |
| IPTG (isopropyl-ß-D-thiogalactopyranosid) | Carl Roth | CN08.1 | |
| L-Glutamine (200 mM) | Merck | K 0282 | |
| LB-Medium | Carl Roth | X968.1 | |
| Lipopolysaccharides from E. coli O55:B5 | Merck | L6529 | |
| Pancoll, human | PAN Biotech | P04-60500 | Separation solution (density gradient centrifugation) |
| Penicillin/Streptomycin (10.000 U/mL) | Merck | A 2212 | |
| Phenyl Sepharose High Performance | GE Healthcare | 17-1082-01 | Resin for hydrophobic interaction chromatography |
| Polymyxin B | Invivogen | tlrl-pmb | |
| Protease inhibitor tablets | Roche | 11873580001 | |
| Q Sepharose Fast Flow | GE Healthcare | 17-0510-01 | Resin for anion-exchange chromatography |
| RoboSep buffer | Stemcell | 20104 | Cell separation buffer (section 5.1.4) |
| RPMI 1640 Medium | Merck | F 1215 | |
| Sodium chloride (NaCl) | Carl Roth | 3957.2 | |
| Sodium hydroxide | Carl Roth | P031.1 | |
| Tris Base | Carl Roth | 4855.3 | |
| Zinc chloride | Carl Roth | T887 | |
| Labware | |||
| 0,45 µm syringe filter | Merck | SLHA033SS | |
| 14 mL roundbottom tubes | BD | 352059 | |
| 2 L Erlenmyer flask | Carl Roth | LY98.1 | |
| 24 well suspension plates | Greiner | 662102 | |
| 5 L measuring beaker | Carl Roth | CKN3.1 | |
| 50 mL conical centrifuge tubes | Corning | 430829 | |
| 50 mL high-speed centrifuge tubes | Eppendorf | 3,01,22,178 | |
| Amicon Ultra-15 Centrifugal Filter Unit MWCO 3 kDa | Merck | UFC900324 | |
| Amicon Ultra-15 Centrifugal Filter Unit MWCO 50 kDa | Merck | UFC905024 | |
| Culture dish (100 mm) | Sarstedt | 83.3902 | |
| Dialysis Tubing Closures | Spectrum | 132738 | |
| EasySep magnet 'The Big Easy` | Stemcell | 18001 | |
| Fraction collector tubes 5 mL | Greiner | 115101 | |
| Lumox film, 25 µm, 305 mm x 40 m | Sarstedt | 94,60,77,316 | Film for monocyte culture plates |
| Spectra/Por Dialysis Membrane (3.5 kDa) | Spectrum | 132724 | |
| Steritop filter unit | Merck | SCGPT01RE | |
| Equipment | |||
| 37 °C Incubator (with shaking) | New Brunswick Scientific | Innova 42 | |
| ÄKTA purifier UPC 10 | GE Healthcare | FPLC System | |
| Fraction collector | GE Healthcare | Frac-920 | |
| Centrifuge (with rotor A-4-81) | Eppendorf | 5810R | |
| Fixed angle rotor | Eppendorf | F-34-6-38 | |
| Mini Protean Tetra Cell | BioRad | 1658000EDU | |
| NanoPhotometer | Implen | P330 | |
| Sonicator | Brandelin | UW2070 | |
| Fluorescence reader | Tecan | infinite M200PRO | |
| pH meter | Knick | 765 |
Riferimenti
- Liston, A., Masters, S. L. Homeostasis-altering molecular processes as mechanisms of inflammasome activation. Nature Reviews Immunology. 17 (3), 208-214 (2017).
- Kessel, C., Holzinger, D., Foell, D. Phagocyte-derived S100 proteins in autoinflammation: putative role in pathogenesis and usefulness as biomarkers. Clinical Immunology. 147 (3), 229-241 (2013).
- Baker, T. M., Nakashige, T. G., Nolan, E. M., Neidig, M. L. Magnetic circular dichroism studies of iron(ii) binding to human calprotectin. Chemical Science. 8 (2), 1369-1377 (2017).
- Nakashige, T. G., Zhang, B., Krebs, C., Nolan, E. M. Human calprotectin is an iron-sequestering host-defense protein. Nature Chemical Biology. 11 (10), 765-771 (2015).
- Nakashige, T. G., Zygiel, E. M., Drennan, C. L., Nolan, E. M. Nickel Sequestration by the Host-Defense Protein Human Calprotectin. Journal of the American Chemical Society. 139 (26), 8828-8836 (2017).
- Cunden, L. S., Gaillard, A., Nolan, E. M. Calcium Ions Tune the Zinc-Sequestering Properties and Antimicrobial Activity of Human S100A12. Chemical Science. 7 (2), 1338-1348 (2016).
- Moroz, O. V., et al. Structure of the human S100A12-copper complex: implications for host-parasite defence. Acta Crystallographica Section D, Biological Crystallography. 59 (Pt 5), 859-867 (2003).
- Moroz, O. V., Blagova, E. V., Wilkinson, A. J., Wilson, K. S., Bronstein, I. B. The crystal structures of human S100A12 in apo form and in complex with zinc: new insights into S100A12 oligomerisation. Journal of Molecular Biology. 391 (3), 536-551 (2009).
- Korndorfer, I. P., Brueckner, F., Skerra, A. The crystal structure of the human (S100A8/S100A9)2 heterotetramer, calprotectin, illustrates how conformational changes of interacting alpha-helices can determine specific association of two EF-hand proteins. Journal of Molecular Biology. 370 (5), 887-898 (2007).
- Moroz, O. V., et al. Both Ca2+ and Zn2+ are essential for S100A12 protein oligomerization and function. BMC Biochemistry. 10, 11 (2009).
- Baudier, J., Glasser, N., Gerard, D. Ions binding to S100 proteins. I. Calcium- and zinc-binding properties of bovine brain S100 alpha alpha, S100a (alpha beta), and S100b (beta beta) protein: Zn2+ regulates Ca2+ binding on S100b protein. Journal of Biological Chemistry. 261 (18), 8192-8203 (1986).
- Dell’Angelica, E. C., Schleicher, C. H., Santome, J. A. Primary structure and binding properties of calgranulin C, a novel S100-like calcium-binding protein from pig granulocytes. Journal of Biological Chemistry. 269 (46), 28929-28936 (1994).
- Vogl, T., et al. S100A12 is expressed exclusively by granulocytes and acts independently from MRP8 and MRP14. Journal of Biological Chemistry. 274 (36), 25291-25296 (1999).
- Hofmann, M. A., et al. RAGE mediates a novel proinflammatory axis: a central cell surface receptor for S100/calgranulin polypeptides. Cell. 97 (7), 889-901 (1999).
- Foell, D., et al. Proinflammatory S100A12 Can Activate Human Monocytes via Toll-like Receptor 4. American Journal of Respiratory and Critical Care Medicine. 187 (12), 1324-1334 (2013).
- Kessel, C., et al. Calcium and zinc tune autoinflammatory Toll-like receptor 4 signaling by S100A12. Journal of Allergy and Clinical Immunology. 142 (4), 1370-1373 (2018).
- Armaroli, G., et al. Monocyte-Derived Interleukin-1beta As the Driver of S100A12-Induced Sterile Inflammatory Activation of Human Coronary Artery Endothelial Cells: Implications for the Pathogenesis of Kawasaki Disease. Arthritis & Rheumatology. 71 (5), 792-804 (2019).
- GE Healthcare. . Strategies for Protein Purification. Handbook. , (2010).
- Jackson, E., Little, S., Franklin, D. S., Gaddy, J. A., Damo, S. M. Expression, Purification, and Antimicrobial Activity of S100A12. Journal of Visualized Experiments. (123), (2017).
- Kiss, B., Ecsedi, P., Simon, M., Nyitray, L. Isolation and Characterization of S100 Protein-Protein Complexes. Methods in Molecular Biology. 1929, 325-338 (2019).
- Magalhaes, P. O., et al. Methods of endotoxin removal from biological preparations: a review. Journal of Pharmacy and Pharmaceutical Sciences. 10 (3), 388-404 (2007).
- Petsch, D., Anspach, F. B. Endotoxin removal from protein solutions. Journal of Biotechnology. 76 (2-3), 97-119 (2000).
- Heilmann, R. M., Suchodolski, J. S., Steiner, J. M. Purification and partial characterization of canine S100A12. Biochimie. 92 (12), 1914-1922 (2010).
- Hung, K. W., Hsu, C. C., Yu, C. Solution structure of human Ca2+-bound S100A12. Journal of Biomolecular NMR. 57 (3), 313-318 (2013).
- Endotoxin Removal. . Application Note - Sartorius Stedim Biotech. , (2010).
- Hogasen, A. K. M., Abrahamsen, T. G. Polymyxin-B Stimulates Production of Complement Components and Cytokines in Human Monocytes. Antimicrobial Agents and Chemotherapy. 39 (2), 529-532 (1995).
- Valentinis, B., et al. Direct effects of polymyxin B on human dendritic cells maturation - The role of I kappa B-alpha/NF-kappa B and ERK1/2 pathways and adhesion. Journal of Biological Chemistry. 280 (14), 14264-14271 (2005).
- Teuber, M., Miller, I. R. Selective Binding of Polymyxin-B to Negatively Charged Lipid Monolayers. Biochimica Et Biophysica Acta. 467 (3), 280-289 (1977).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon