Method Article
Valutazione del rischio di cancro colorettale e della prevalenza da parte del rilevamento dell'integrità del DNA delle feci
In questo articolo
Erratum Notice
Riepilogo
Il kit di diagnostica FL-DNA è un metodo che consente di risparmiare tempo e di utilizzare per determinare la probabilità affidabile della presenza di lesioni del cancro colorettale.
Abstract
Al giorno d'oggi, il DNA delle feci può essere isolato e analizzato con diversi metodi. I lunghi frammenti di DNA nelle feci possono essere rilevati da un test qPCR, che fornisce una probabilità affidabile della presenza di lesioni colorettali pre-neoplastiche o neoplastiche. Questo metodo, chiamato DNA lungo a fluorescenza (FL-DNA), è una procedura veloce e non invasiva che è un miglioramento del sistema di prevenzione primario. Questo metodo si basa sulla valutazione dell'integrità del DNA fecale mediante amplificazione quantitativa di specifici obiettivi del DNA genomico. In particolare, la valutazione di frammenti di DNA più lunghi di 200 bp consente di rilevare pazienti con lesioni colorettali con specificità molto elevata. Tuttavia, questo sistema e tutti i test del DNA delle feci attualmente disponibili presentano alcuni problemi generali che devono essere affrontati (ad esempio, la frequenza con cui devono essere effettuati i test e il numero ottimale di campioni di feci raccolti in ogni momento per ogni individuo). Tuttavia, il principale vantaggio di FL-DNA è la possibilità di usarlo in associazione con un test attualmente utilizzato nel programma di screening CRC, noto come esame del sangue occulto fecale a base immunochimica (iFOBT). Infatti, entrambe le prove possono essere eseguite sullo stesso campione, riducendo i costi e ottenendo una migliore previsione dell'eventuale presenza di lesioni colorettali.
Introduzione
Il cancro colorettale (CRC) deriva da un processo a più fasi in cui l'epitelio sano si sviluppa lentamente in adenomi o polipi, che progrediscono in carcinomi maligni nel tempo1,2. Nonostante l'elevato tasso di incidenza del CRC, nell'ultimo decennio3è stata osservata una tendenza al ribasso della percentuale di decessi. Infatti, i primi strumenti diagnostici adottati nei programmi di screening hanno portato alla diagnosi precoce e alla rimozione di adenomas pre-neoplastici o polipi4. Tuttavia, a causa dei diversi limiti tecnici, nessuno di questi metodi è ottimale. Infatti, al fine di migliorare la sensibilità e la specificità, molti test del DNA delle feci sono stati proposti da soli o in combinazione con gli attuali test diagnostici di routine5,6.
Tipicamente, mucosa sana getta nel flusso fecale colonciti apoptotici, mentre mucosa malati esfolia colonciti non apoptotici. Frammenti di 200 bp o più di lunghezza caratterizzano il DNA non apoptotico. Questo DNA è chiamato DNA lungo (L-DNA) ed è diventato un biomarcatore utilizzabile per la diagnosi precoce del CRC. Il L-DNA può essere isolato dal campione di feci e quantificato da qPCR utilizzando un kit in vitro diagnostico FL-DNA7,8,9,10,11,12.
Il test consiste in due analisi per il rilevamento di frammenti di FL-DNA che vanno da 138 bp a 339 bp. Ogni analisi consente l'amplificazione del FL-DNA (FAM) e del DNA spike-in (HEX). Per garantire un'amplificazione ottimale di tutti i frammenti, il test è stato suddiviso in due saggi (denominati "A" e "B"). Il saggio A rileva due regioni dell'esone 14 del gene APC (NM_001127511) e un frammento di esone 7 del gene TP53 (NM_001276760). Il saggio B rileva un frammento di esone 14 del gene APC (NM_001127511) e due regioni di esoni 5 e 8 del gene TP53 (NM_001276760). I saggi non distinguono tra le regioni rilevate. Il DNA spike-in corrisponde al DNA del salmone di Oncorhynchus e consente di verificare che la procedura sia stata eseguita correttamente e che controlli la possibile presenza di inibitori, che possono produrre falsi risultati negativi. La concentrazione di FL-DNA viene valutata mediante quantificazione assoluta utilizzando il metodo della curva standard ed è espressa come ng/reazione.
Il metodo FL-DNA è un test del DNA delle feci non invasivo e poco costoso che, combinato con l'esame del sangue occulto fecale a base immunochimica (iFOBT), è attualmente utilizzato nei programmi di screening CRC e consente migliori previsioni di CRC e/o adenoma lesioni ad alto rischio12.
Protocollo
I pazienti sono stati assunti presso l'Istituto Roma Romagnolo per lo Studio e la Cura del Tumori (IRST) di Meldola (FC, Italia) tra il 2013 e il 2015. I pazienti arruolati erano nel protocollo IRSTB002, approvato dal Comitato Etico dell'IRST - IRCCS AVR (25/10/2012, ver. 1). Tutti i metodi sono stati eseguiti in conformità con le linee guida e i regolamenti pertinenti. Il consenso informato scritto è stato ottenuto da tutti i pazienti.
1. Estrazione del DNA dalle feci
- Utilizzare un kit per preparare esempi di feci (vedere Tabella dei materiali). Selezionare e trattare il materiale fecale eseguendo l'estrazione secondo le istruzioni del produttore. Amplificare direttamente il DNA purificato o conservarlo a -20 gradi centigradi per un'analisi successiva.
2. Preparazione di controlli positivi, standard, DNA spike-in e campioni clinici
- Preparazione di norme e campioni
- Per preparare il controllo positivo, gli standard, il DNA spike-in e tutti i campioni clinici, centrifugare una aliquota di controllo positivo, standard e DNA spike-in, quindi rispendere ogni reagente aggiungendo la quantità corretta di acqua fornita (vedi sotto). Quindi, vortice con attenzione il controllo positivo, standard, e picco-in DNA, poi centrifugare per 10 s. Per ottenere una completa sospensione dei reagenti a secco, conservare i reagenti liquidi a temperatura ambiente (RT) per 30 minuti prima dell'uso.
- Il controllo positivo è il DNA umano in formato asciutto. Risospendere ogni aliquota con 750 l' di acqua.
- Il DNA spike-in è il DNA salmone (Oncorhynchus keta), che viene utilizzato come controllo interno esogeno per verificare la possibile presenza di inibitori nei campioni di DNA estratti dalle feci. Risospendere ogni aliquota con 100 l' di acqua.
- Per preparare la curva standard, produrre quattro diluizioni 1:5 a partire dalla soluzione stock. I punti standard devono essere 10 ng/reazione, 2 ng/reazione, 0.4 ng/reaction, e 0.08 ng/reaction.
- Per preparare il controllo positivo, gli standard, il DNA spike-in e tutti i campioni clinici, centrifugare una aliquota di controllo positivo, standard e DNA spike-in, quindi rispendere ogni reagente aggiungendo la quantità corretta di acqua fornita (vedi sotto). Quindi, vortice con attenzione il controllo positivo, standard, e picco-in DNA, poi centrifugare per 10 s. Per ottenere una completa sospensione dei reagenti a secco, conservare i reagenti liquidi a temperatura ambiente (RT) per 30 minuti prima dell'uso.
- Preparazione del DNA 1x spike-in
- Preparare il controllo del DNA spike-in direttamente prima dell'uso.
- Preparare il controllo del DNA di 1x spike-in mescolando 5 -L di picco FL-DNa con 20 -L di acqua sterile. Il numero di campioni di controllo del DNA da 1x spike-in sarà preparato in base al numero di campioni da analizzare, più il controllo positivo.
- Preparazione di campioni
- Mescolare 75 -L dei campioni (campioni clinici o controllo positivo) con 25 : L di 1x di DNA spike-in, producendo un volume totale di 100 .
3. Amplificazione e determinazione del valore FL-DNA utilizzando qPCR Easy PGX
NOTA: miscele di amplificazione complete contenenti primer e sonde specifiche che prendono di mira il DNA umano e il controllo interno sono fornite in un formato liofilizzato in 8 strisce di pozzo per FL-DNA Mix A e FL-DNA Mix B. Standards, controlli positivi e negativi, e i campioni devono essere amplificati con entrambe le miscele di liofilazione. I campioni clinici devono essere amplificati solo in duplicato con entrambe le miscele loofilizzate.
- Consultare la Tabella dei materiali per lo strumento qPCR e il software operativo.
- Aprire il software operativo e impostare la piastra e il profilo termico:
- Impostare la piastra come mostrato nella Tabella 1.
- Impostare il tipo di pozzo per tutte e otto le posizioni nella colonna 1 come Standard.
- Impostare il tipo di pozzo per i pozzi A2 e B2 come NTC.
- Impostare il tipo di pozzo per C2 e D2 (controlli positivi) su Sconosciuto.
- Impostare il tipo di pozzo per tutte le altre posizioni su Sconosciuto.
- Selezionare tutte le 96 posizioni e aggiungere i dyes FAM e HEX. Fare clic su Piastra di sincronizzazione.
- Impostare il profilo termico in base alla tabella 2.
- Impostare la piastra come mostrato nella Tabella 1.
- Centrifugare il numero necessario di strisce per 10 s per portare il contenuto sul fondo del tubo.
- Rimuovere delicatamente le guarnizioni dalle strisce, prestando attenzione a trattenere il contenuto, e aggiungere alle rispettive strisce: controllo negativo: 20 -L di acqua; campione: 20 - L di DNA; curva standard: 20 L di standard 1, 2, 3 o 4; controllo positivo: 20 l di controllo positivo.
- Chiudere con attenzione tutte le strisce utilizzando i tappi ottici piatti a 8 strisce e il vortice per alcuni secondi.
- Centrifugare le strisce per 10 s e caricarle nello strumento. Quindi, avviare la corsa.
- Aprire il software operativo e impostare la piastra e il profilo termico:
4. Analisi dei dati
NOTA: l'analisi dei dati può essere eseguita automaticamente o manualmente a seconda del software (vedere Tabella dei materiali).
- Al termine della corsa, selezionare le colonne A, C, E, G per "FAM: FL-DNA-A " e "HEX: IC"e le colonne B, D, F, H per "FAM: FL-DNA-B" e "HEX: IC".
- Impostare quanto segue per la quantità standard di quantità a partire: 10 ng/reazione per i pozzi A1 e B1, 2 ng/reazione per C1 e D1, 0,4 ng/reazione per E1 e F1 e 0.08 ng/reazione per G1 e H1.
- Impostare i valori di fluorescenza di soglia su 150 per entrambi i canali FAM (FL-DNA A e FL-DNA B) e HEX (IC).
- Nella casella Tabella risultati, fare clic su Opzioni colonna . Proprietà Select All OK per ottenere i risultati in entrambi i canali con i rispettivi valori Cq ( ) e R.
NOTA: questi valori sono forniti dal software dello strumento PCR in tempo reale. L'ultima volta che la fluorescenza viene normalizzata all'ultimo ciclo di amplificazione. - Nella casella Tabella risultati fare clic con il pulsante destro del mouse sulla tabella per aprire il menu di scelta rapida e scegliere Invia a Excel per esportare i dati non elaborati.
- Controllare i valori degli standard per verificare l'idoneità della curva standard.
- Per ogni mix FL-DNA, controllare la colonna R2 ["R2 (-R)" e l'efficienza ["Efficienza (%)" colonna] valori. Se si trovano in un intervallo accettabile, è possibile procedere con l'analisi di conseguenza alle istruzioni del produttore (Tabella 3).
- Se i risultati del canale FAM non sono compresi nell'intervallo previsto, omettere un punto della curva standard e analizzare nuovamente la conduzione.
- Determinare i valori dei controlli negativi e positivi con la formula seguente, considerando i valori "No Cq" come zero:

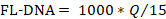
- Confrontare i valori ottenuti con quelli riportati nella tabella 4.
- Se i controlli di reazione sono nell'intervallo dei valori previsti, procedere con l'analisi dei campioni.
NOTA: verificare che i valori Cq ottenuti siano generati da una vera e propria reazione di amplificazione (curva di fluorescenza sigmoidale) e non da un artefatto (curva di fluorescenza lineare). - Per analizzare l'idoneità del campione per ogni mix FL-DNA, confrontare i valori Cq del canale HEX. Se il valore è di 16, procedere con l'analisi dei campioni. Se il valore è <16 o non è presente alcun Cq, è probabile che sia dovuto a un errore di erogazione del raccoglitore FL-DNA. Pertanto, non è possibile analizzare i campioni.
- Calcolare la media dei valori Cq nel canale "HEX" del controllo positivo utilizzando la seguente formula:
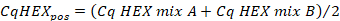
- Calcolare la media dei valori Cq nel canale "HEX" del campione replica utilizzando la seguente formula:
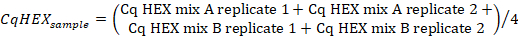
- Calcolare i valori di CqHEX in base alla seguente formula:

- Confrontare i valori di CqHEX dei campioni con quelli riportati nella tabella 5.
- Per ogni mix (Mix A e Mix B), confrontare i valori Cq del canale FAM con quelli riportati nella tabella 6.
- Per determinare il valore FL-DNA di ogni campione adatto, utilizzare la seguente formula, considerando i valori "No Cq" come zero:
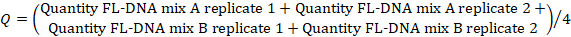
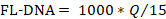
NOTA: il rischio e la prevalenza del cancro colorettale è una funzione delle valutazioni di iFOBT e FL-DNA secondo i risultati nomogrammi Fagan ottenuti da Rengucci et al.12 (tabella 7).
Risultati
Il flusso di lavoro di questo protocollo è illustrato nella Figura 1. Il flusso di lavoro fornisce due passaggi di controllo e azioni diverse in base a questi risultati. In primo luogo, se un campione presenta controlli inadatti, l'amplificazione deve essere ripetuta. In secondo luogo, se l'amplificazione è inibita, il campione deve essere rielaborato dall'inizio o classificato come non prezioso.
La figura 2 mostra le curve di fluorescenza prodotte da campioni positivi e negativi. (A) Mostrato è un esempio di un campione positivo adatto. Il segnale campione sul canale HEX è compreso nell'intervallo accettabile. Il segnale positivo è al di sopra della soglia sul canale FAM. (B) Mostrato è un esempio di un campione positivo/non positivo adatto. Il segnale campione è compreso nell'intervallo accettabile sul canale HEX. Il segnale di controllo negativo è al di sotto della soglia sul canale FAM. (C) Mostrato è un esempio di un campione non adatto. Il segnale del campione non è compreso nell'intervallo accettabile sul canale HEX; pertanto, si può presumere una potenziale inibizione. Questo campione deve essere ripetuto, a partire dall'estrazione.
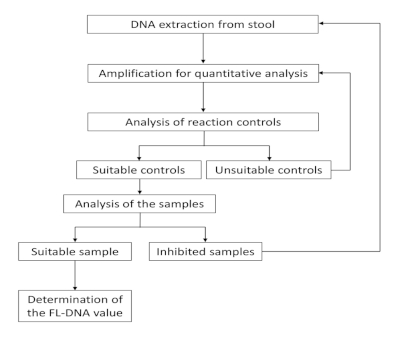
Figura 1: Flusso di lavoro per la quantificazione FL-DNA. Fare clic qui per visualizzare una versione più grande di questa figura.
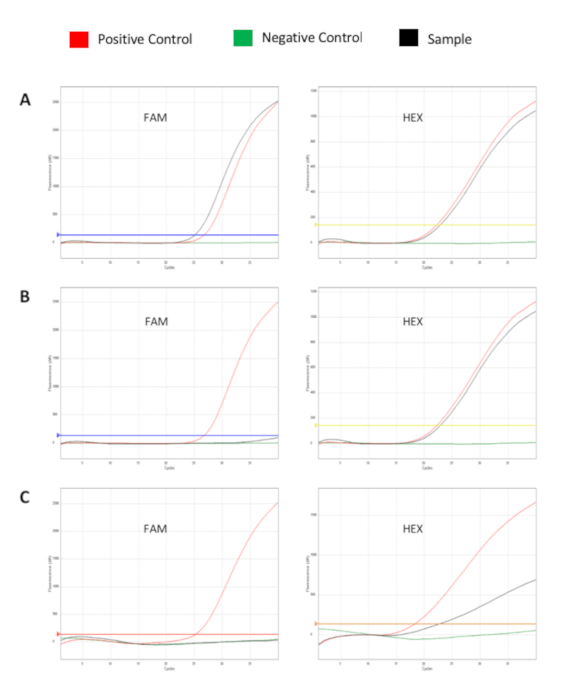
Figura 2: Curve di fluorescenza che mostrano l'amplificazione dei geni bersaglio Mix A (o Mix B) (canale FAM) e controllo interno (canale HEX). (A) Campione positivo FL-DNA. (B) campione negativo FL-DNA. (C) Inibizione dell'amplificazione del campione. Curva rossa: controllo positivo; curva verde: controllo negativo; curva nera: campione clinico. Fare clic qui per visualizzare una versione più grande di questa figura.
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | X | |||
| Un | Standard 1 | Acqua | DNA2 | DNA4 | DNA6 | DNA8 | DNA10 | DNA12 (in questo stato del 200 | DNA14 | DNA16 | DNA18 | DNA20 | 1 | FL-DNA Mix A | |
| B | Standard 1 | Acqua | DNA2 | DNA4 | DNA6 | DNA8 | DNA10 | DNA12 (in questo stato del 200 | DNA14 | DNA16 | DNA18 | DNA20 | 2 | FL-DNA Mix B | |
| C | Standard 2 | Pos | DNA2 | DNA4 | DNA6 | DNA8 | DNA10 | DNA12 (in questo stato del 200 | DNA14 | DNA16 | DNA18 | DNA20 | 3 | FL-DNA Mix A | |
| D | Standard 2 | Pos | DNA2 | DNA4 | DNA6 | DNA8 | DNA10 | DNA12 (in questo stato del 200 | DNA14 | DNA16 | DNA18 | DNA20 | 4 | FL-DNA Mix B | |
| E (in questo modo | Standard 3 | DNA1 | DNA3 | DNA5 | DNA7 | DNA9 | DNA11 | DNA13 | DNA15 | DNA17 | DNA19 | DNA21 | 5 | FL-DNA Mix A | |
| F | Standard 3 | DNA1 | DNA3 | DNA5 | DNA7 | DNA9 | DNA11 | DNA13 | DNA15 | DNA17 | DNA19 | DNA21 | 6 | FL-DNA Mix B | |
| G | Standard 4 | DNA1 | DNA3 | DNA5 | DNA7 | DNA9 | DNA11 | DNA13 | DNA15 | DNA17 | DNA19 | DNA21 | 7 | FL-DNA Mix A | |
| H | Standard 4 | DNA1 | DNA3 | DNA5 | DNA7 | DNA9 | DNA11 | DNA13 | DNA15 | DNA17 | DNA19 | DNA21 | 8 | FL-DNA Mix B |
Tabella 1: piastra di set-up con distribuzione di controllo, curva e campioni. La colonna X indica il numero impresso sulla parte superiore della striscia.
| Passo | Temperatura e tempo |
| Avvio a caldo (1 ciclo) | 95 gradi centigradi per 5 min |
| Amplificazione (40 cicli) | 95 gradi centigradi per 15 s 54 gradi centigradi per 15 s 60 s per 45 s (Raccolta dati) |
Tabella 2: Profilo termico per l'amplificazione del DNA.
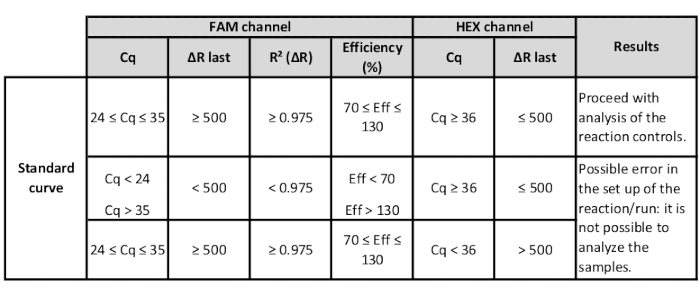
Tabella 3: Gamma di valori dei canali HEX e FAM degli standard per verificare l'idoneità della curva standard.

Tabella 4: Intervallo di valori di canale HEX e FAM di controlli negativi e positivi per verificare l'idoneità della corsa.

Tabella 5: Intervallo di valori di canale HEX e FAM dei campioni per verificare l'idoneità dell'analisi dei campioni.

Tabella 6: Mix A e Mix B Cq valori del canale FAM per verificare l'idoneità dell'analisi FL-DNA.
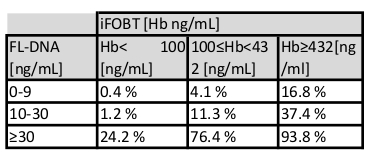
Tabella 7: Valutazione del rischio di cancro in funzione dei valori di iFOBT e FL-DNA. In base alla relazione tra i valori iFOBT e FL-DNA, la tabella stima la probabilità di lesioni neoplastiche colorettali.
Discussione
Studi precedenti hanno dimostrato che l'analisi dell'integrità del DNA delle feci estratte da approcci manuali e semiautomatici può rappresentare uno strumento alternativo per la diagnosi precoce delle lesioni colorettali7,8,9,,10,11,12. Nel corso degli anni sono stati sviluppati test molecolari di screening non invasivi per la rilevazione del cancro colorettale, ma la diffusione diffusa di questi metodi è limitata a causa di approcci che richiedono tempo e di scarsa efficacia in termini di costi rispetto ad altri test di screening.
Questo approccio è relativamente economico e non troppo dispendioso in termini di tempo. Ha anche una maggiore precisione nel rilevamento delle lesioni colorettali a causa di una nuova procedura che richiede pochi passaggi manuali. L'approccio descritto qui è veloce con meno passaggi manuali ed è in grado di essere eseguito facilmente su molti campioni a settimana. L'estrazione del DNA non presenta particolari problemi e può essere eseguita attraverso semplici passaggi manuali o l'uso di uno strumento automatico. In quest'ultimo caso, è necessario determinare lo strumento di estrazione automatica del DNA, consentendo i risultati più riproducibili. La fase più critica dell'estrazione del DNA è la raccolta delle feci e il metodo della sua conservazione prima dell'estrazione del DNA. Si consiglia di mantenere lo sgabello congelato e procedere con l'estrazione il prima possibile.
Un altro problema critico è rappresentato da possibili inibitori delle reazioni di amplificazione. L'estrazione fecale non è in grado di purificare il DNA genomico, poiché le impurità compromettono la corretta reazione di amplificazione. A questo proposito, il protocollo richiede l'uso di DNA spike-in per verificare la presenza/assenza di inibitori di reazione.
Fino a poco tempo fa, l'esame del sangue occulto fecale è l'approccio principale utilizzato per rilevare le lesioni colorettali nei programmi di screening; anche se, presenta alcuni limiti in termini di precisione. Una strategia alternativa per la diagnosi del cancro colorettale si basa sull'analisi del DNA delle cellule esfoliate esfoliate nelle feci. Nell'ultimo anno sono stati valutati diversi approcci, ma nessuno è disponibile per l'uso nei programmi di screening.
Il valore di integrità FL-DNA può essere un'utile alternativa, che in combinazione con il valore di test iFOBT di screening standard, può prevedere la presenza di tumori e/o adenomaade ad alto rischio11,12 da un approccio Fagan Nomogram10 (Tabella 7). Questo approccio stima la probabilità combinata di test della presenza di lesioni neoplastiche, migliorando la precisione diagnostica rispetto all'approccio standard utilizzato da solo.
Queste informazioni possono aiutare i medici a pianificare i test diagnostici oltre a una colonscopia appropriata e a personalizzarne la sorveglianza. Infatti, il kit FL-DNA semplifica i passaggi manuali e garantisce risultati stabili e coerenti. Tuttavia, alcuni problemi rimangono da chiarire per migliorare l'accuratezza diagnostica del metodo. Ad esempio, la frequenza con cui devono essere eseguite le prove e il numero di campioni di feci che devono essere analizzati in momenti specifici per ogni individuo devono essere accuratamente selezionati. Dato che questo test deve essere eseguito contemporaneamente a iFOBT, è necessario un metodo che richiede la raccolta ogni 2 anni, come di contesto in numerosi protocolli di screening, per verificare l'efficacia di questo test in sostituzione o in alternativa agli attuali test di screening.
Divulgazioni
Maura Menghi è dipendente a tempo pieno di Diatech Pharmacogenetics srl.
Riconoscimenti
Gli autori non hanno alcun riconoscimento.
Materiali
| Name | Company | Catalog Number | Comments |
| 1.5 mL and 2 mL polypropylene twist-lock tubes (DNase-, RNase-, DNA-, PCR inhibitor-free) | Consumables required for DNA extraction and Real Time PCR | ||
| Absolute Ethanol (quality of analytical degree) | Reagent required for DNA extraction | ||
| Benchtop centrifuge | Maximum speed of 20000 x g. Instrument required for DNA extraction | ||
| EasyPGX analysis software version 2.0.0 | Diatech Pharmacogenetics | RT800-SW | Analysis software |
| EasyPGX centrifuge/vortex 8-well strips | Diatech Pharmacogenetics | RT803 | Instrument recommended for the Real Time PCR assay |
| EasyPGX qPCR instrument 96 | Diatech Pharmacogenetics | RT800-96 | Instrument recommended for the Real Time PCR assay |
| EasyPGX ready FL-DNA | Diatech Pharmacogenetics | RT029 | Kit required for the Real Time PCR assay |
| Micropipettes (volumes from 1 to 1.000 µL) | Consumables required for DNA extraction and Real Time PCR | ||
| Powder-free disposable gloves | Consumables required for DNA extraction and Real Time PCR | ||
| QIAamp Fast DNA Stool | Qiagen | 51604 | Kit recommended for the DNA extraction and purification from stool |
| Sterile filter tips DNase-, RNase-free (volumes from 1 to 1.000 µL) | Consumables required for DNA extraction and Real Time PCR | ||
| Thermal block e.g. EasyPGX dry block | Diatech Pharmacogenetics | RT801 | Instrument required for DNA extraction |
| Vortex e.g. EasyPGX centrifuge/vortex 1.5 ml | Diatech Pharmacogenetics | RT802 | Instrument required for DNA extraction |
Riferimenti
- Fearon, E. R. Molecular Genetics of Colorectal Cancer. Annual Review of Pathology. 6, 479-507 (2011).
- Sears, C. L., Garrett, W. S. Microbes, Microbiota, and Colon Cancer. Cell Host and Microbe. 15, 317-328 (2014).
- Levin, B., et al. Screening and Surveillance for the Early Detection of Colorectal Cancer and Adenomatous Polyps, 2008: A Joint Guideline From the American Cancer Society, the US Multi-Society Task Force on Colorectal Cancer, and the American College of Radiology. Gastroenterology. 134, 1570-1595 (2008).
- Bosch, L. J., et al. Molecular tests for colorectal cancer screening. Clinical Colorectal Cancer. 10, 8-23 (2011).
- Ahlquist, D. A. Molecular detection of colorectal neoplasia. Gastroenterology. 138, 2127-2139 (2010).
- Calistri, D., et al. Fecal multiple molecular tests to detect colorectal cancer in stool. Clinical Gastroenterology and Hepatology. 1, 377-383 (2003).
- Calistri, D., et al. Detection of colorectal cancer by a quantitative fluorescence determination of DNA amplification in stool. Neoplasia. 6, 536-540 (2004).
- Calistri, D., et al. Quantitative fluorescence determination of long-fragment DNA in stool as a marker for the early detection of colorectal cancer. Cellular Oncology. 31, 11-17 (2009).
- Calistri, D., et al. Fecal DNA for noninvasive diagnosis of colorectal cancer in immunochemical fecal occult blood test-positive individuals. Cancer Epidemiology Biomarkers and Prevention. 19, 2647-2654 (2010).
- De Maio, G., et al. Circulating and stool nucleic acid analysis for colorectal cancer diagnosis. World Journal of Gastroenterology. 20, 957-967 (2014).
- Rengucci, C., et al. Improved stool DNA integrity method for early colorectal cancer diagnosis. Cancer Epidemiology Biomarkers and Prevention. 23, 2553-2560 (2014).
Erratum
Formal Correction: Erratum: Evaluation of Colorectal Cancer Risk and Prevalence by Stool DNA Integrity Detection
Posted by JoVE Editors on 9/28/2020. Citeable Link.
An erratum was issued for: Evaluation of Colorectal Cancer Risk and Prevalence by Stool DNA Integrity Detection. An affiliation was updated.
The first affiliation was updated from:
Istituto Scientifico Romagnolo per lo Studio e la Cura dei Tumori (IRST)
to:
Istituto Scientifico Romagnolo per lo Studio e la Cura dei Tumori (IRST) IRCCS, Meldola, Italy
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon