Method Article
Bewertung des Darmkrebsrisikos und der Prävalenz durch Stuhl-DNA-Integritätsnachweis
In diesem Artikel
Erratum Notice
Zusammenfassung
Das vorgestellte diagnostische FL-DNA-Kit ist eine zeitsparende und benutzerfreundliche Methode, um die zuverlässige Wahrscheinlichkeit des Vorhandenseins von Darmkrebsläsionen zu bestimmen.
Zusammenfassung
Heute kann Stuhl-DNA mit mehreren Methoden isoliert und analysiert werden. Die langen DNA-Fragmente im Stuhl können durch einen qPCR-Assay nachgewiesen werden, der eine zuverlässige Wahrscheinlichkeit für das Vorhandensein von präneoplastischen oder neoplastischen dickdarmen Läsionen bietet. Diese Methode, die fluoreszenzlange DNA (FL-DNA) genannt wird, ist ein schnelles, nicht-invasives Verfahren, das eine Verbesserung gegenüber dem primären Präventionssystem darstellt. Diese Methode basiert auf der Bewertung der Fäkal-DNA-Integrität durch quantitative Verstärkung spezifischer Ziele der genomischen DNA. Insbesondere die Auswertung von DNA-Fragmenten von mehr als 200 bp ermöglicht den Nachweis von Patienten mit kolorektalen Läsionen mit sehr hoher Spezifität. Dieses System und alle derzeit verfügbaren Stuhl-DNA-Tests stellen jedoch einige allgemeine Probleme dar, die angegangen werden müssen (z. B. die Häufigkeit, mit der Tests durchgeführt werden sollten, und eine optimale Anzahl von Stuhlproben, die zu jedem Zeitpunkt für jeden einzelnen Personen gesammelt werden). Der Hauptvorteil von FL-DNA ist jedoch die Möglichkeit, sie in Verbindung mit einem Test zu verwenden, der derzeit im CRC-Screening-Programm verwendet wird, dem immunchemischen fekten okkulten Bluttest (iFOBT). Tatsächlich können beide Tests an derselben Probe durchgeführt werden, wodurch die Kosten gesenkt und eine bessere Vorhersage des eventuellen Vorhandenseins von kolorektalen Läsionen erreicht werden kann.
Einleitung
Darmkrebs (CRC) leitet sich von einem mehrstufigen Prozess ab, bei dem sich gesundes Epithel langsam zu Adenomen oder Polypen entwickelt, die im Laufe der Zeit zu bösartigen Karzinomen übergehen1,2. Trotz der hohen Inzidenzrate des SCR wurde in den letzten zehn Jahren ein rückläufiger Trend beim Anteil der Todesfälle beobachtet3. Tatsächlich haben frühdiagnostische Instrumente, die in Screening-Programmen eingesetzt werden, zur Früherkennung und Entfernung von präneoplastischen Adenomen oder Polypen4geführt. Aufgrund der unterschiedlichen technischen Grenzen ist jedoch keine dieser Methoden optimal. Um die Empfindlichkeit und Spezifität zu verbessern, wurden viele Stuhl-DNA-Tests allein oder in Kombination mit aktuellen Routinediagnostiktestsvorgeschlagen 5,6.
Typischerweise, gesunde Schleimhaut vergießt in den Fäkalstrom apoptotische Kolonozyten, während kranke Schleimhaut Peeling nicht-apoptotische Kolonozyten. Fragmente von 200 bp oder mehr in der Länge charakterisieren nicht-apoptotische DNA. Diese DNA wird lange DNA (L-DNA) genannt und ist zu einem nutzbaren Biomarker für die CRC-Frühdiagnose geworden. Die L-DNA kann aus Stuhlproben isoliert und mit qPCR mit einem in vitro diagnostischen FL-DNA Kit7,8,9,10,11,12quantifiziert werden.
Der Test besteht aus zwei Assays zum Nachweis von FL-DNA-Fragmenten im Bereich von 138 bp bis 339 bp. Jeder Assay ermöglicht die Amplifikation von FL-DNA (FAM) sowie Spike-in-DNA (HEX). Um eine optimale Verstärkung aller Fragmente zu gewährleisten, wurde der Test in zwei Assays (genannt "A" und "B") unterteilt. Der A-Test erkennt zwei Regionen von Exon 14 des APC-Gens (NM_001127511) und ein Fragment von Exon 7 des TP53-Gens (NM_001276760). Der B-Test erkennt ein Fragment von Exon 14 des APC-Gens (NM_001127511) und zwei Regionen der Exons 5 und 8 des TP53-Gens (NM_001276760). Die Assays unterscheiden nicht zwischen den erkannten Regionen. Die Spike-in-DNA entspricht der Oncorhynchus Keta Lachs DNA und ermöglicht die Überprüfung, ob das Verfahren ordnungsgemäß durchgeführt wurde und überprüft das mögliche Vorhandensein von Inhibitoren, die zu falschen negativen Ergebnissen führen können. Die FL-DNA-Konzentration wird durch absolute Quantifizierung nach der Standardkurvenmethode ausgewertet und als ng/Reaktion ausgedrückt.
Die FL-DNA-Methode ist ein nicht-invasiver und kostengünstiger Stuhl-DNA-Test, der in Kombination mit dem immunchemischen fäkalen okkulten Bluttest (iFOBT) derzeit in CRC-Screening-Programmen verwendet wird und bessere Vorhersagen von CRC- und/oder Hochrisiko-Adenomaläsionenermöglicht 12.
Protokoll
Die Patienten wurden zwischen 2013 und 2015 am Istituto Scientifico Romagnolo per lo Studio e la Cura dei Tumori (IRST) in Meldola (FC, Italien) rekrutiert. Die eingeschriebenen Patienten wurden in das Protokoll IRSTB002 aufgenommen, das von der Ethikkommission von IRST - IRCCS AVR genehmigt wurde (25/10/2012, ver. 1). Alle Methoden wurden in Übereinstimmung mit den einschlägigen Richtlinien und Vorschriften durchgeführt. Von allen Patienten wurde eine schriftliche Einwilligung eingeholt.
1. DNA-Extraktion aus dem Stuhl
- Verwenden Sie ein Kit, um Stuhlproben vorzubereiten (siehe Materialtabelle). Wählen und behandeln Sie das Fäkalienmaterial, indem Sie die Extraktion gemäß den Anweisungen des Herstellers durchführen. Verstärken Sie die gereinigte DNA direkt oder speichern Sie sie bei -20 °C für die nachfolgende Analyse.
2. Herstellung von Positivkontrolle, Standards, Spike-in-DNA und klinischen Proben
- Erstellung von Normen und Proben
- Um die positive Kontrolle, Standards, Spike-in-DNA und alle klinischen Proben vorzubereiten, zentrifugieren Sie ein Aliquot aus positiver Kontrolle, Standards und Spike-in-DNA, und setzen Sie dann jedes Reagenz durch Zugabe der richtigen Menge an bereitgestelltem Wasser wieder aus (siehe unten). Dann, vorsichtig Wirbel die positive Kontrolle, Standard, und Spike-in-DNA, dann Zentrifuge für 10 s. Um eine vollständige Resuspension der trockenen Reagenzien zu erreichen, lagern Sie die flüssigen Reagenzien vor Gebrauch 30 min bei Raumtemperatur (RT).
- Die positive Kontrolle ist menschliche DNA in einem trockenen Format. Setzen Sie jedes Aliquot mit 750 l Wasser auf.
- Die Spike-in-DNA ist Lachs (Oncorhynchus keta) DNA, die als exogene interne Kontrolle verwendet wird, um das mögliche Vorhandensein von Inhibitoren in DNA-Proben aus Stuhl extrahiert zu überprüfen. Setzen Sie jedes Aliquot mit 100 l Wasser auf.
- Um die Standardkurve vorzubereiten, produzieren Sie vier 1:5 Verdünnungen ausgehend von der Lagerlösung. Die Standardpunkte müssen 10 ng/Reaktion, 2 ng/Reaktion, 0,4 ng/Reaktion und 0,08 ng/Reaktion sein.
- Um die positive Kontrolle, Standards, Spike-in-DNA und alle klinischen Proben vorzubereiten, zentrifugieren Sie ein Aliquot aus positiver Kontrolle, Standards und Spike-in-DNA, und setzen Sie dann jedes Reagenz durch Zugabe der richtigen Menge an bereitgestelltem Wasser wieder aus (siehe unten). Dann, vorsichtig Wirbel die positive Kontrolle, Standard, und Spike-in-DNA, dann Zentrifuge für 10 s. Um eine vollständige Resuspension der trockenen Reagenzien zu erreichen, lagern Sie die flüssigen Reagenzien vor Gebrauch 30 min bei Raumtemperatur (RT).
- Vorbereitung der 1x Spike-in-DNA
- Bereiten Sie die Spike-in-DNA-Kontrolle direkt vor der Verwendung vor.
- Bereiten Sie die 1x Spike-in-DNA-Kontrolle vor, indem Sie 5 l FL-DNa-Spitze mit 20 l sterilem Wasser mischen. Die Anzahl der 1x Spike-in-DNA-Kontrollproben wird entsprechend der Anzahl der zu analysierenden Proben sowie der positiven Kontrolle erstellt.
- Vorbereitung von Proben
- Mischen Sie 75 l der Proben (klinische Proben oder Positivkontrolle) mit 25 l 1x Spike-in-DNA, was zu einem Gesamtvolumen von 100 l führt.
3. Amplifikation und Bestimmung des FL-DNA-Wertes mit qPCR Easy PGX
HINWEIS: Vollständige Amplifikationsmischungen mit spezifischen Primern und Sonden, die auf die menschliche DNA und die interne Kontrolle abzielen, werden in einem lyophilisierten Format in 8 Well-Streifen für FL-DNA Mix A und FL-DNA Mix B bereitgestellt. Standards, positive und negative Kontrollen sowie Proben müssen mit beiden lyophilisierten Mischungen verstärkt werden. Klinische Proben dürfen nur mit beiden lyophilisierten Mischungen doppelt verstärkt werden.
- Siehe Tabelle der Materialien für qPCR-Instrument und Bediensoftware.
- Öffnen Sie die Bediensoftware und richten Sie die Platte und das Wärmeprofil ein:
- Richten Sie die Platte wie in Tabelle 1dargestellt ein.
- Legen Sie den Brunnentyp für alle acht Positionen in Spalte 1 als Standardfest.
- Stellen Sie den Brunnentyp für die Brunnen A2 und B2 als NTCein.
- Legen Sie den Brunnentyp für C2 und D2 (die positiven Steuerelemente) als Unbekanntfest.
- Legen Sie den Brunnentyp für alle anderen Positionen als Unbekannt fest.
- Wählen Sie alle 96 Positionen aus, und fügen Sie die Dyes FAM und HEXhinzu. Klicken Sie auf Sync Plate.
- Stellen Sie das Wärmeprofil gemäß Tabelle 2ein.
- Richten Sie die Platte wie in Tabelle 1dargestellt ein.
- Zentrifugieren Sie die benötigte Anzahl von Streifen für 10 s, um den Inhalt auf den Boden des Rohres zu bringen.
- Entfernen Sie vorsichtig die Dichtungen von den Streifen, während Sie darauf achten, den Inhalt zu behalten, und fügen Sie zu den jeweiligen Streifen: Negativkontrolle: 20 L Wasser; Probe: 20 L DNA; Standardkurve: 20 l der Norm 1, 2, 3 oder 4; Positivkontrolle: 20 L positiv kontrollierbar.
- Schließen Sie alle Streifen mit den 8 Streifen flachen optischen Kappen und Wirbel für einige Sekunden sorgfältig.
- Zentrifugieren Sie die Streifen für 10 s und laden Sie sie in das Instrument. Starten Sie dann den Lauf.
- Öffnen Sie die Bediensoftware und richten Sie die Platte und das Wärmeprofil ein:
4. Datenanalyse
HINWEIS: Die Datenanalyse kann je nach Software automatisch oder manuell durchgeführt werden (siehe Materialtabelle).
- Wählen Sie am Ende des Laufs die Spalten A, C, E, G für "FAM: FL-DNA-A" und "HEX: IC"und die Spalten B, D, F, H für "FAM: FL-DNA-B" und "HEX: IC"aus.
- Stellen Sie für die Standardmengen Ausgangsmengefolgendes auf: 10 ng/Reaktion für A1- und B1-Bohrungen, 2 ng/Reaktion für C1 und D1, 0,4 ng/Reaktion für E1 und F1 und 0,08 ng/Reaktion für G1 und H1.
- Legen Sie Schwellenwertfluoreszenzwerte für FAM-Kanäle (FL-DNA A und FL-DNA B) und HEX (IC) auf 150 fest.
- KlickenSie im Feld Ergebnistabelleauf Spaltenoptionen | Alle auswählen | Ok, um die Ergebnisse in beiden Kanälen mit ihren jeweiligen Cq-Werten (-R) und -R-Lastwerten zu erhalten.
HINWEIS: Diese Werte werden von der Real Time PCR-Instrumentensoftware bereitgestellt. Die letzte R entspricht dem fluoreszenzwert, der auf den letzten Amplifikationszyklus normalisiert wurde. - Klicken Sie im Feld Ergebnistabelle mit der rechten Maustaste auf die Tabelle, um das Kontextmenü zu öffnen, und klicken Sie auf An Excel senden, um die Rohdaten zu exportieren.
- Überprüfen Sie die Werte der Standards, um die Eignung der Standardkurve zu überprüfen.
- Überprüfen Sie für jeden FL-DNA-Mix dieR2-Spalte ["R2 (R)" und die Effizienz ["Effizienz (%)" Spaltenwerte. Wenn sie sich in einem akzeptablen Bereich befinden, ist es möglich, die Analyse entsprechend den Anweisungen des Herstellers fortzusetzen (Tabelle 3).
- Wenn die Ergebnisse des FAM-Kanals nicht im erwarteten Bereich liegen, lassen Sie einen Punkt der Standardkurve weg, und analysieren Sie den Lauf erneut.
- Bestimmen Sie die Werte der negativen und positiven Steuerelemente mit der folgenden Formel, wobei die "No Cq"-Werte als Null berücksichtigt werden:

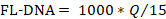
- Vergleichen Sie die erhaltenen Werte mit den in Tabelle 4genannten Werten.
- Wenn sich die Reaktionssteuerungen im Bereich der erwarteten Werte befinden, fahren Sie mit der Analyse der Proben fort.
ANMERKUNG: Stellen Sie sicher, dass die erhaltenen Cq-Werte aus einer realen Amplifikationsreaktion (sigmoidale Fluoreszenzkurve) und nicht aus einem Artefakt (lineare Fluoreszenzkurve) generiert werden. - Um die Eignung der Probe für jeden FL-DNA-Mix zu analysieren, vergleichen Sie die Cq-Werte des HEX-Kanals. Wenn der Wert 16 € beträgt, fahren Sie mit der Analyse der Proben fort. Wenn der Wert <16 ist oder kein Cq vorhanden ist, ist dies wahrscheinlich auf einen Dosierfehler des FL-DNA Spike zurückzuführen. Daher ist es nicht möglich, die Proben zu analysieren.
- Berechnen Sie den Mittelwert der Cq-Werte im "HEX"-Kanal der Positivsteuerung mit der folgenden Formel:
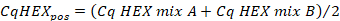
- Berechnen Sie den Mittelwert der Cq-Werte im "HEX"-Kanal der Stichprobenreplikationen mit der folgenden Formel:
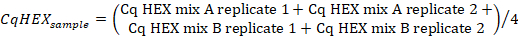
- Berechnen Sie die CqHEX-Werte nach der folgenden Formel:

- Vergleichen Sie die CqHEX-Werte der Stichproben mit den in Tabelle 5.
- Vergleichen Sie für jede Mischung (Mix A und Mix B) die Cq-Werte des FAM-Kanals mit den in Tabelle 6 gemeldeten Werten.
- Um den FL-DNA-Wert jeder geeigneten Probe zu bestimmen, verwenden Sie die folgende Formel, wobei die "No Cq"-Werte als Null betrachtet werden:
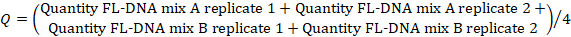
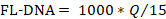
HINWEIS: Das Darmkrebsrisiko und die Prävalenz sind eine Funktion von iFOBT- und FL-DNA-Auswertungen gemäß den Von Rengucci et al.12 (Tabelle 7) erhaltenen Fagan-Nomogramm-Ergebnissen.
Ergebnisse
Der Workflow dieses Protokolls ist in Abbildung 1dargestellt. Der Workflow bietet zwei Steuerungsschritte und verschiedene Aktionen entsprechend diesen Schrittergebnissen. Erstens: Wenn eine Probe ungeeignete Kontrollen enthält, muss die Verstärkung wiederholt werden. Zweitens: Wenn die Verstärkung gehemmt wird, muss die Probe von Anfang an aufbereitet oder als nicht wertvoll eingestuft werden.
Abbildung 2 zeigt die Fluoreszenzkurven, die durch positive und negative Proben erzeugt werden. (A) Gezeigt ist ein Beispiel für eine geeignete positive Probe. Das Probensignal auf dem HEX-Kanal liegt im akzeptablen Bereich. Das positive Signal liegt über dem Schwellenwert auf dem FAM-Kanal. (B) Gezeigt ist ein Beispiel für eine geeignete negative/nicht positive Probe. Das Sample-Signal liegt innerhalb des akzeptablen Bereichs auf dem HEX-Kanal. Das negative Steuersignal liegt unterhalb des Schwellenwerts auf dem FAM-Kanal. (C) Gezeigt ist ein Beispiel für eine nicht geeignete Probe. Das Sample-Signal liegt nicht innerhalb des akzeptablen Bereichs auf dem HEX-Kanal; somit kann von einer möglichen Hemmung ausgegangen werden. Diese Probe muss wiederholt werden, beginnend mit der Extraktion.
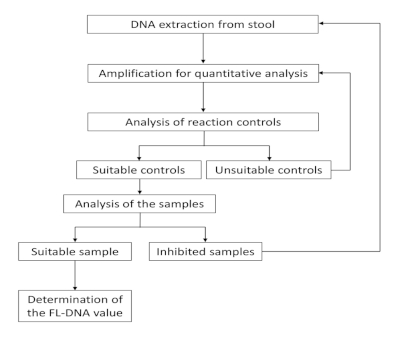
Abbildung 1: Workflow für FL-DNA-Quantifizierung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
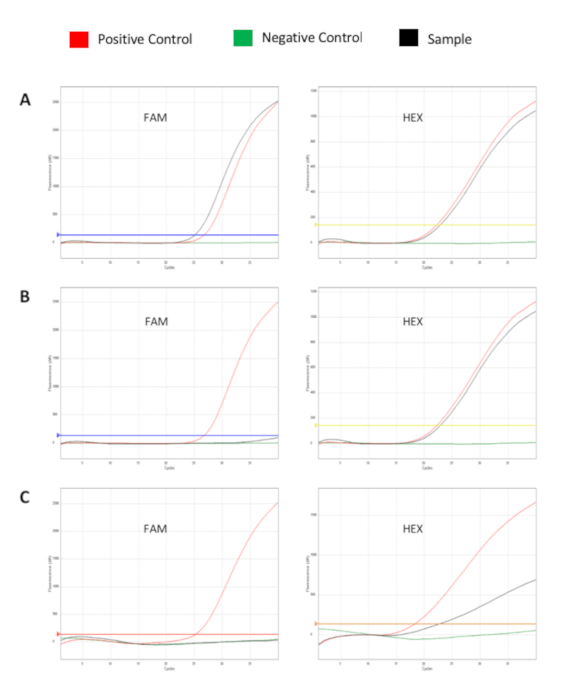
Abbildung 2: Fluoreszenzkurven, die die Verstärkung von Mix A (oder Mix B) Zielgenen (Kanal FAM) und interner Kontrolle (Kanal HEX) anzeigen. (A) FL-DNA-positive Probe. (B) FL-DNA-Negativprobe. (C) Hemmung der Probenverstärkung. Rote Kurve: positive Kontrolle; grüne Kurve: Negativkontrolle; schwarze Kurve: klinische Probe. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | X | |||
| Eine | Standard 1 | Wasser | DNA2 | DNA4 | DNA6 | DNA8 | DNA10 | DNA12 | DNA14 | DNA16 | DNA18 | DNA20 | 1 | FL-DNA Mix A | |
| B | Standard 1 | Wasser | DNA2 | DNA4 | DNA6 | DNA8 | DNA10 | DNA12 | DNA14 | DNA16 | DNA18 | DNA20 | 2 | FL-DNA Mix B | |
| C | Standard 2 | Pos | DNA2 | DNA4 | DNA6 | DNA8 | DNA10 | DNA12 | DNA14 | DNA16 | DNA18 | DNA20 | 3 | FL-DNA Mix A | |
| D | Standard 2 | Pos | DNA2 | DNA4 | DNA6 | DNA8 | DNA10 | DNA12 | DNA14 | DNA16 | DNA18 | DNA20 | 4 | FL-DNA Mix B | |
| E | Standard 3 | DNA1 | DNA3 | DNA5 | DNA7 | DNA9 | DNA11 | DNA13 | DNA15 | DNA17 | DNA19 | DNA21 | 5 | FL-DNA Mix A | |
| F | Standard 3 | DNA1 | DNA3 | DNA5 | DNA7 | DNA9 | DNA11 | DNA13 | DNA15 | DNA17 | DNA19 | DNA21 | 6 | FL-DNA Mix B | |
| G | Standard 4 | DNA1 | DNA3 | DNA5 | DNA7 | DNA9 | DNA11 | DNA13 | DNA15 | DNA17 | DNA19 | DNA21 | 7 | FL-DNA Mix A | |
| H | Standard 4 | DNA1 | DNA3 | DNA5 | DNA7 | DNA9 | DNA11 | DNA13 | DNA15 | DNA17 | DNA19 | DNA21 | 8 | FL-DNA Mix B |
Tabelle 1: Rüstplatte mit Verteilung von Steuerung, Kurve und Proben. Spalte X gibt die Aufdrucknummer oben auf dem Streifen an.
| Schritt | Temperatur und Zeit |
| Heißstart (1 Zyklus) | 95 °C für 5 min |
| Verstärkung (40 Zyklen) | 95 °C für 15 s 54 °C für 15 s 60 °C für 45 s (Datenerfassung) |
Tabelle 2: Thermisches Profil zur DNA-Verstärkung.
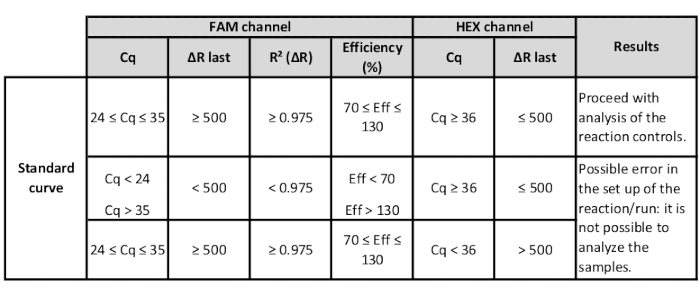
Tabelle 3: Bereich der HEX- und FAM-Kanalwerte der Standards zur Überprüfung der Eignung der Standardkurve.

Tabelle 4: Bereich der HEX- und FAM-Kanalwerte von negativen und positiven Steuerelementen, um die Eignung des Laufs zu überprüfen.

Tabelle 5: Bereich der HEX- und FAM-Kanalwerte von Proben zur Überprüfung der Eignung der Probenanalyse.

Tabelle 6: Mischen Sie A- und Mix B-Cq-Werte des FAM-Kanals, um die Eignung der FL-DNA-Analyse zu überprüfen.
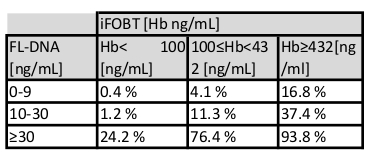
Tabelle 7: Krebsrisikobewertung in Abhängigkeit von iFOBT- und FL-DNA-Werten. Entsprechend der Beziehung zwischen iFOBT- und FL-DNA-Werten schätzt die Tabelle die Wahrscheinlichkeit von kolorektalen neoplastischen Läsionen.
Diskussion
Frühere Studien haben gezeigt, dass die DNA-Integritätsanalyse von Hockern, die durch manuelle und halbautomatische Ansätze extrahiert wurden, ein alternatives Werkzeug zur Früherkennung von kolorektalen Läsionen darstellen kann7,8,9,,10,11,12. Molekulare, nichtinvasive Screening-Tests wurden im Laufe der Jahre für den Nachweis von Dickdarmkrebs entwickelt, aber die weit verbreitete Verbreitung dieser Methoden ist aufgrund zeitaufwändiger Ansätze und schlechter Wirtschaftlichkeit im Vergleich zu anderen Screening-Tests begrenzt.
Dieser Ansatz ist relativ billig und nicht zu zeitaufwändig. Es hat auch erhöhte Genauigkeit bei der Erkennung von kolorektalen Läsionen aufgrund eines neuen Verfahrens, das nur wenige manuelle Schritte erfordert. Der hier beschriebene Ansatz ist schnell mit weniger manuellen Schritten, und es ist in der Lage, leicht auf viele Proben pro Woche durchgeführt werden. Die DNA-Extraktion stellt keine besonderen Probleme dar und kann durch einfache manuelle Schritte oder die Verwendung eines automatischen Instruments durchgeführt werden. Im letzteren Fall ist es notwendig, das automatische DNA-Extraktionsinstrument zu bestimmen, so dass die reproduzierbarsten Ergebnisse möglich sind. Der wichtigste Schritt der DNA-Extraktion ist die Sammlung von Stuhl und Methode seiner Lagerung vor der DNA-Extraktion. Es ist ratsam, den Stuhl gefroren zu halten und so schnell wie möglich mit der Extraktion fortzufahren.
Ein weiteres kritisches Problem sind mögliche Inhibitoren der Amplifikationsreaktionen. Die Fäkalienextraktion ist nicht in der Lage, die genomische DNA zu reinigen, da Verunreinigungen die richtige Amplifikationsreaktion beeinträchtigen. In diesem Zusammenhang erfordert das Protokoll die Verwendung von Spike-in-DNA zur Überprüfung des Vorhandenseins/Fehlens von Reaktionshemmern.
Bis vor kurzem ist der okkulte Fäkalien-Bluttest der Hauptansatz, der verwendet wird, um kolorektale Läsionen in Screening-Programmen zu erkennen; obwohl, es stellt einige Grenzen in Bezug auf die Genauigkeit. Eine alternative Strategie zur Diagnose von Dickdarmkrebs basiert auf der Analyse von DNA aus Peelingzellen, die im Stuhl ausgeschieden werden. Im vergangenen Jahr wurden mehrere Ansätze evaluiert, aber keine sonnbar für Screening-Programme zur Verfügung.
Der FL-DNA-Integritätswert kann eine nützliche Alternative sein, die in Kombination mit dem Standard-Screening-iFOBT-Testwert das Vorhandensein von Tumoren und/oder Hochrisiko-Adenomen11,12 durch einen Fagan Nomogram-Ansatz10 vorhersagen kann (Tabelle 7). Dieser Ansatz schätzt die kombinierte Testwahrscheinlichkeit der neoplastischen Läsionspräsenz und verbessert die diagnostische Genauigkeit im Vergleich zum Standardansatz, der allein verwendet wird.
Diese Informationen können Ärzten bei der Planung diagnostischer Tests zusätzlich zu einer geeigneten Koloskopie und Personalisierung ihrer Überwachung helfen. Tatsächlich vereinfacht das FL-DNA-Kit die manuellen Schritte und sorgt für stabile und konsistente Ergebnisse. Allerdings müssen noch einige Fragen geklärt werden, um die diagnostische Genauigkeit der Methode zu verbessern. Beispielsweise sollten die Häufigkeit, mit der die Tests durchgeführt werden sollen, und die Anzahl der Stuhlproben, die zu bestimmten Zeitpunkten für jede Person analysiert werden müssen, sorgfältig ausgewählt werden. Da dieser Test gleichzeitig mit iFOBT durchgeführt werden muss, ist eine Methode erforderlich, die alle 2 Jahre aufgenommen werden muss, wie es in zahlreichen Screening-Protokollen üblich ist, um die Wirksamkeit dieses Tests als Ersatz oder Alternative zu den aktuellen Screening-Tests zu überprüfen.
Offenlegungen
Maura Menghi ist hauptamtliche Mitarbeiterin von Diatech Pharmacogenetics srl.
Danksagungen
Die Autoren haben keine Anerkennung.
Materialien
| Name | Company | Catalog Number | Comments |
| 1.5 mL and 2 mL polypropylene twist-lock tubes (DNase-, RNase-, DNA-, PCR inhibitor-free) | Consumables required for DNA extraction and Real Time PCR | ||
| Absolute Ethanol (quality of analytical degree) | Reagent required for DNA extraction | ||
| Benchtop centrifuge | Maximum speed of 20000 x g. Instrument required for DNA extraction | ||
| EasyPGX analysis software version 2.0.0 | Diatech Pharmacogenetics | RT800-SW | Analysis software |
| EasyPGX centrifuge/vortex 8-well strips | Diatech Pharmacogenetics | RT803 | Instrument recommended for the Real Time PCR assay |
| EasyPGX qPCR instrument 96 | Diatech Pharmacogenetics | RT800-96 | Instrument recommended for the Real Time PCR assay |
| EasyPGX ready FL-DNA | Diatech Pharmacogenetics | RT029 | Kit required for the Real Time PCR assay |
| Micropipettes (volumes from 1 to 1.000 µL) | Consumables required for DNA extraction and Real Time PCR | ||
| Powder-free disposable gloves | Consumables required for DNA extraction and Real Time PCR | ||
| QIAamp Fast DNA Stool | Qiagen | 51604 | Kit recommended for the DNA extraction and purification from stool |
| Sterile filter tips DNase-, RNase-free (volumes from 1 to 1.000 µL) | Consumables required for DNA extraction and Real Time PCR | ||
| Thermal block e.g. EasyPGX dry block | Diatech Pharmacogenetics | RT801 | Instrument required for DNA extraction |
| Vortex e.g. EasyPGX centrifuge/vortex 1.5 ml | Diatech Pharmacogenetics | RT802 | Instrument required for DNA extraction |
Referenzen
- Fearon, E. R. Molecular Genetics of Colorectal Cancer. Annual Review of Pathology. 6, 479-507 (2011).
- Sears, C. L., Garrett, W. S. Microbes, Microbiota, and Colon Cancer. Cell Host and Microbe. 15, 317-328 (2014).
- Levin, B., et al. Screening and Surveillance for the Early Detection of Colorectal Cancer and Adenomatous Polyps, 2008: A Joint Guideline From the American Cancer Society, the US Multi-Society Task Force on Colorectal Cancer, and the American College of Radiology. Gastroenterology. 134, 1570-1595 (2008).
- Bosch, L. J., et al. Molecular tests for colorectal cancer screening. Clinical Colorectal Cancer. 10, 8-23 (2011).
- Ahlquist, D. A. Molecular detection of colorectal neoplasia. Gastroenterology. 138, 2127-2139 (2010).
- Calistri, D., et al. Fecal multiple molecular tests to detect colorectal cancer in stool. Clinical Gastroenterology and Hepatology. 1, 377-383 (2003).
- Calistri, D., et al. Detection of colorectal cancer by a quantitative fluorescence determination of DNA amplification in stool. Neoplasia. 6, 536-540 (2004).
- Calistri, D., et al. Quantitative fluorescence determination of long-fragment DNA in stool as a marker for the early detection of colorectal cancer. Cellular Oncology. 31, 11-17 (2009).
- Calistri, D., et al. Fecal DNA for noninvasive diagnosis of colorectal cancer in immunochemical fecal occult blood test-positive individuals. Cancer Epidemiology Biomarkers and Prevention. 19, 2647-2654 (2010).
- De Maio, G., et al. Circulating and stool nucleic acid analysis for colorectal cancer diagnosis. World Journal of Gastroenterology. 20, 957-967 (2014).
- Rengucci, C., et al. Improved stool DNA integrity method for early colorectal cancer diagnosis. Cancer Epidemiology Biomarkers and Prevention. 23, 2553-2560 (2014).
Erratum
Formal Correction: Erratum: Evaluation of Colorectal Cancer Risk and Prevalence by Stool DNA Integrity Detection
Posted by JoVE Editors on 9/28/2020. Citeable Link.
An erratum was issued for: Evaluation of Colorectal Cancer Risk and Prevalence by Stool DNA Integrity Detection. An affiliation was updated.
The first affiliation was updated from:
Istituto Scientifico Romagnolo per lo Studio e la Cura dei Tumori (IRST)
to:
Istituto Scientifico Romagnolo per lo Studio e la Cura dei Tumori (IRST) IRCCS, Meldola, Italy
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten