Method Article
Évaluation du risque et de la prévalence du cancer colorectal par détection de l’intégrité de l’ADN des selles
Dans cet article
Erratum Notice
Résumé
Le kit de DIAGNOSTIC PRÉSENTÉ FL-ADN est une méthode de gain de temps et conviviale pour déterminer la probabilité fiable de la présence de lésions de cancer colorectal.
Résumé
De nos jours, l’ADN des selles peut être isolé et analysé par plusieurs méthodes. Les longs fragments d’ADN dans les selles peuvent être détectés par un essai qPCR, qui fournit une probabilité fiable de la présence de lésions côlorectiques pré-néoplastiques ou néoplastiques. Cette méthode, appelée fluorescence long DNA (FL-ADN), est une procédure rapide et non invasive qui est une amélioration par rapport au système de prévention primaire. Cette méthode est basée sur l’évaluation de l’intégrité de l’ADN fécal par amplification quantitative de cibles spécifiques de l’ADN génomique. En particulier, l’évaluation des fragments d’ADN de plus de 200 bp permet de détecter les patients atteints de lésions côlorectaux avec une spécificité très élevée. Cependant, ce système et tous les tests d’ADN des selles actuellement disponibles présentent des problèmes généraux qui doivent être abordés (p. ex., la fréquence à laquelle les tests devraient être effectués et le nombre optimal d’échantillons de selles prélevés à chaque point de vue pour chaque individu). Cependant, le principal avantage de l’ADN FL est la possibilité de l’utiliser en association avec un test actuellement utilisé dans le programme de dépistage du CRC, connu sous le nom de test sanguin occulte fécical à base d’immunochimie (iFOBT). En effet, les deux tests peuvent être effectués sur le même échantillon, réduisant les coûts et réalisant une meilleure prévision de la présence éventuelle de lésions côlorectaux.
Introduction
Le cancer colorectal (CRC) provient d’un processus en plusieurs étapes dans lequel l’épithélium sain se développe lentement en adéomes ou polypes, qui progressent dans les carcinomes malins au fil du temps1,2. Malgré le taux d’incidence élevé du CRC, on a observé une tendance à la baisse du pourcentage de décès au cours de la dernière décennie3. En effet, les premiers outils de diagnostic adoptés dans les programmes de dépistage ont conduit à la détection et à l’élimination précoces des adéomes pré-néoplastiques ou des polypes4. Cependant, en raison des différentes limites techniques, aucune de ces méthodes n’est optimale. En effet, afin d’améliorer la sensibilité et la spécificité, de nombreux tests d’ADN des selles ont été proposés seuls ou en combinaison avec les tests diagnostiques de routineactuels 5,6.
Typiquement, la muqueuse saine se jette dans les colonocytes apoptotiques de cours d’eau fécal, tandis que la muqueuse malade exfolie les colonocytes non apoptotiques. Des fragments de 200 bp ou plus de longueur caractérisent l’ADN non apoptotique. Cet ADN est appelé l’ADN long (L-ADN) et est devenu un biomarqueur utilisant pour le diagnostic tôt de CRC. L’ADN L peut être isolé du spécimen de selles et quantifié par qPCR à l’aide d’un diagnostic in vitro FL-ADN kit7,8,9,10,11,12.
Le test se compose de deux essais pour la détection de fragments d’ADN FL allant de 138 bp à 339 bp. Chaque essai permet l’amplification de FL-ADN (FAM) ainsi que l’ADN de pointe (HEX). Pour assurer une amplification optimale de tous les fragments, le test a été divisé en deux essais (nommés « A » et « B »). L’essai A détecte deux régions d’exon 14 du gène APC (NM_001127511) et un fragment d’exon 7 du gène TP53 (NM_001276760). L’essai B détecte un fragment d’exon 14 du gène APC (NM_001127511) et deux régions d’exons 5 et 8 du gène TP53 (NM_001276760). Les essais ne font pas de distinction entre les régions détectées. L’ADN de pointe correspond à l’ADN du saumon Oncorhynchus keta et permet de vérifier que la procédure a été effectuée correctement et vérifie la présence possible d’inhibiteurs, ce qui peut donner de faux résultats négatifs. La concentration FL-ADN est évaluée par quantification absolue en utilisant la méthode de courbe standard et est exprimée comme ng/réaction.
La méthode FL-ADN est un test d’ADN de selles non invasif et peu coûteux qui, combiné avec le test sanguin occulte féctique immunochimique (iFOBT), est actuellement utilisé dans les programmes de dépistage du CRC et permet de meilleures prédictions des lésions d’adénonomique de CRC et/ou à haut risque12.
Protocole
Des patients ont été recrutés à l’Istituto Scientifico Romagnolo per lo Studio e la Cura dei Tumori (IRST) de Meldola (FC, Italie) entre 2013 et 2015. Les patients inscrits étaient dans le protocole IRSTB002, approuvé par le Comité d’éthique de l’IRST - IRCCS AVR (25/10/2012, ver. 1). Toutes les méthodes ont été exécutées conformément aux lignes directrices et règlements pertinents. Le consentement éclairé écrit a été obtenu de tous les patients.
1. Extraction d’ADN à partir de selles
- Utilisez un kit pour préparer des échantillons de selles (voir Tableau des matériaux). Sélectionnez et traitez le matériel fécal en effectuant l’extraction selon les instructions du fabricant. Amplifier l’ADN purifié directement ou stocker à -20 oC pour une analyse ultérieure.
2. Préparation d’un contrôle positif, de normes, d’un pic d’ADN et d’échantillons cliniques
- Préparation des normes et des échantillons
- Pour préparer le contrôle positif, les normes, l’ADN à pointe et tous les échantillons cliniques, la centrifugeuse un aliquot de contrôle positif, les normes et l’ADN de pointe, puis résout chaque réactif en ajoutant la bonne quantité d’eau fournie (voir ci-dessous). Puis, soigneusement vortex le contrôle positif, la norme, et l’ADN de pointe-dans, puis centrifugeuse pour 10 s. Pour obtenir une résuspension complète des réactifs secs, entreposez les réactifs liquides à température ambiante (RT) pendant 30 minutes avant utilisation.
- Le contrôle positif est l’ADN humain dans un format sec. Resuspendez chaque aliquot avec 750 ll d’eau.
- L’ADN de pointe est le saumon (Oncorhynchus keta) ADN, qui est utilisé comme un contrôle interne exogène pour vérifier la présence possible d’inhibiteurs dans des échantillons d’ADN extraits de selles. Réutilisez chaque aliquot à 100 ll d’eau.
- Pour préparer la courbe standard, produisez quatre dilutions de 1:5 à partir de la solution de stock. Les points standard doivent être 10 ng/réaction, 2 ng/réaction, 0,4 ng/réaction, et 0,08 ng/réaction.
- Pour préparer le contrôle positif, les normes, l’ADN à pointe et tous les échantillons cliniques, la centrifugeuse un aliquot de contrôle positif, les normes et l’ADN de pointe, puis résout chaque réactif en ajoutant la bonne quantité d’eau fournie (voir ci-dessous). Puis, soigneusement vortex le contrôle positif, la norme, et l’ADN de pointe-dans, puis centrifugeuse pour 10 s. Pour obtenir une résuspension complète des réactifs secs, entreposez les réactifs liquides à température ambiante (RT) pendant 30 minutes avant utilisation.
- Préparation de l’ADN 1x spike-in
- Préparer le contrôle de l’ADN avant l’utilisation.
- Préparer le contrôle de l’ADN 1x en mélangeant 5 L de pointe FL-DNa avec 20 L d’eau stérile. Le nombre d’échantillons de contrôle de l’ADN à pointe de 1x sera préparé en fonction du nombre d’échantillons à analyser, ainsi que du contrôle positif.
- Préparation d’échantillons
- Mélanger 75 l’un des échantillons (échantillons cliniques ou contrôle positif) avec 25 lL d’ADN de pointe de 1x, ce qui donne un volume total de 100 ll.
3. Amplification et détermination de la valeur FL-ADN à l’aide de QPCR Easy PGX
REMARQUE : Des mélanges complets d’amplification contenant des amorces et des sondes spécifiques ciblant l’ADN humain et le contrôle interne sont fournis dans un format lyophilisé en 8 bandes de puits pour le mélange DE FL-ADN A et le mix B de FL-ADN, les contrôles positifs et négatifs, et les échantillons doivent être amplifiés avec les deux mélanges lyophilisés. Les échantillons cliniques ne doivent être amplifiés qu’en double avec les deux mélanges lyophilisés.
- Consultez le tableau des matériaux pour l’instrument qPCR et les logiciels d’exploitation.
- Ouvrez le logiciel d’exploitation et configurez la plaque et le profil thermique :
- Configurez la plaque comme indiqué dans le tableau 1.
- Définissez le type de puits pour les huit positions dans la colonne 1 comme Standard.
- Définissez le type de puits pour les puits A2 et B2 en tant que CNT.
- Définir le type de puits pour C2 et D2 (les contrôles positifs) comme inconnu.
- Définissez le type de puits pour toutes les autres positions comme inconnu.
- Sélectionnez les 96 positions et ajoutez les Dyes FAM et HEX. Cliquez sur Sync Plate.
- Réglez le profil thermique selon le tableau 2.
- Configurez la plaque comme indiqué dans le tableau 1.
- Centrifugez le nombre nécessaire de bandes pour 10 s pour apporter le contenu au fond du tube.
- Retirez délicatement les joints des bandes, tout en prêtant attention à conserver le contenu, et ajoutez aux bandes respectives : contrôle négatif : 20 l d’eau ; échantillon : 20 l d’ADN; courbe standard : 20 l de la norme 1, 2, 3 ou 4; contrôle positif : 20 L de contrôle positif.
- Fermez soigneusement toutes les bandes à l’aide des 8 bandes de bouchons optiques plats et de vortex pendant quelques secondes.
- Centrifugez les bandes pour 10 s et chargez-les dans l’instrument. Ensuite, commencez la course.
- Ouvrez le logiciel d’exploitation et configurez la plaque et le profil thermique :
4. Analyse des données
REMARQUE : L’analyse des données peut être effectuée automatiquement ou manuellement en fonction du logiciel (voir tableau des matériaux).
- A la fin de la course, sélectionnez les colonnes A, C, E, G pour "FAM: FL-DNA-A " et "HEX: IC « ,et les colonnes B, D, F, H pour "FAM: FL-DNA-B " et "HEX: IC ".
- Définir ce qui suit pour le montant de départ des quantités standard: 10 ng/réaction pour les puits A1 et B1, 2 ng/réaction pour C1 et D1, 0,4 ng/réaction pour E1 et F1, et 0,08 ng/réaction pour G1 et H1.
- Définir les valeurs de fluorescence seuil à 150 pour les canaux FAM (FL-DNA A et FL-DNA B) et HEX (IC).
- Dans la case Tableau des résultats, cliquez sur Options de colonnes Sélectionnez tous les Ok pour obtenir les résultats dans les deux canaux avec leurs valeurs respectives de Cq et de R.
REMARQUE : Ces valeurs sont fournies par le logiciel d’instrument PCR en temps réel. Le dernier de la période correspond à la valeur de fluorescence normalisée au dernier cycle d’amplification. - Dans la case Tableau de résultat, cliquez à droite sur la table pour ouvrir le menu contextuelle et cliquez sur Envoyer à Excel pour exporter les données brutes.
- Vérifiez les valeurs des normes pour vérifier la pertinence de la courbe standard.
- Pour chaque mix FL-ADN, vérifiez la colonne R2 [R2[R) » et l’efficacité ["Efficacité (%) » colonne] valeurs. S’ils sont dans une fourchette acceptable, il est possible de procéder à l’analyse en conséquence des instructions du fabricant(tableau 3).
- Si les résultats du canal FAM ne sont pas dans la plage prévue, omettre un point de la courbe standard et réanalysez la course.
- Déterminer les valeurs des contrôles négatifs et positifs avec la formule suivante, considérant les valeurs « No Cq » comme étant nulles :

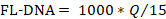
- Comparez les valeurs obtenues avec celles indiquées dans le tableau 4.
- Si les contrôles de réaction sont dans la fourchette des valeurs attendues, procédez à l’analyse des échantillons.
REMARQUE : Vérifiez que les valeurs Cq obtenues sont générées à partir d’une véritable réaction d’amplification (courbe de fluorescence sinoïdale) et non d’un artefact (courbe de fluorescence linéaire). - Pour analyser la pertinence de l’échantillon pour chaque mix FL-ADN, comparez les valeurs Cq du canal HEX. Si la valeur est de 16 euros, procéder à l’analyse des échantillons. Si la valeur est de 'lt;16 ou il n’y a pas de Cq, il est probablement dû à une erreur de distribution de la pointe FL-ADN. Par conséquent, il n’est pas possible d’analyser les échantillons.
- Calculez la moyenne des valeurs Cq dans le canal « HEX » du contrôle positif à l’aide de la formule suivante :
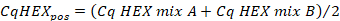
- Calculer la moyenne des valeurs Cq dans le canal « HEX » de l’échantillon se réplique à l’aide de la formule suivante:
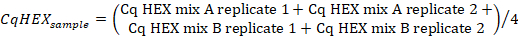
- Calculez les valeurs de CqHEX selon la formule suivante :

- Comparez les valeurs de l’échantillon à l’CQHEX avec celles indiquées dans le tableau 5.
- Pour chaque mix (Mix A et Mix B), comparez les valeurs Cq du canal FAM avec celles indiquées dans le tableau 6.
- Pour déterminer la valeur FL-ADN de chaque échantillon approprié, utilisez la formule suivante, considérant les valeurs « No Cq » comme étant nulles :
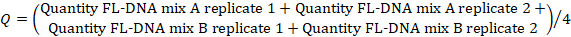
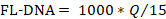
REMARQUE : Le risque et la prévalence du cancer colorectal sont fonction des évaluations iFOBT et FL-ADN selon les résultats du nomogramme Fagan obtenus par Rengucci et coll.12 (tableau 7).
Résultats
Le flux de travail de ce protocole est indiqué dans la figure 1. Le flux de travail fournit deux étapes de contrôle et des actions différentes selon ces résultats d’étape. Tout d’abord, si un échantillon présente des contrôles inappropriés, l’amplification doit être répétée. Deuxièmement, si l’amplification est inhibée, l’échantillon doit être retraité dès le début ou classé comme n’étant pas utile.
La figure 2 montre les courbes de fluorescence produites par des échantillons positifs et négatifs. (A) Montré est un exemple d’un échantillon positif approprié. Le signal de l’échantillon sur le canal HEX se situe dans la plage acceptable. Le signal positif est au-dessus du seuil sur le canal FAM. (B) Montré est un exemple d’un échantillon négatif/non positif approprié. Le signal de l’échantillon se situe dans la plage acceptable du canal HEX. Le signal de contrôle négatif est inférieur au seuil du canal FAM. (C) Montré est un exemple d’un échantillon non approprié. Le signal de l’échantillon n’est pas dans la plage acceptable sur le canal HEX; ainsi, une inhibition potentielle peut être supposée. Cet échantillon doit être répété, à partir de l’extraction.
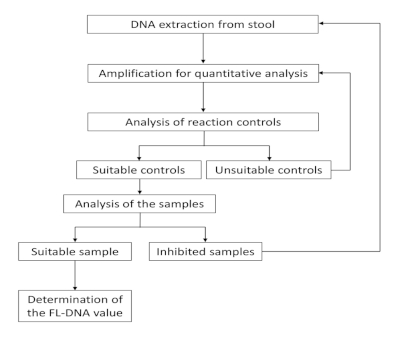
Figure 1 : Flux de travail pour la quantification DE l’ADN FL. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
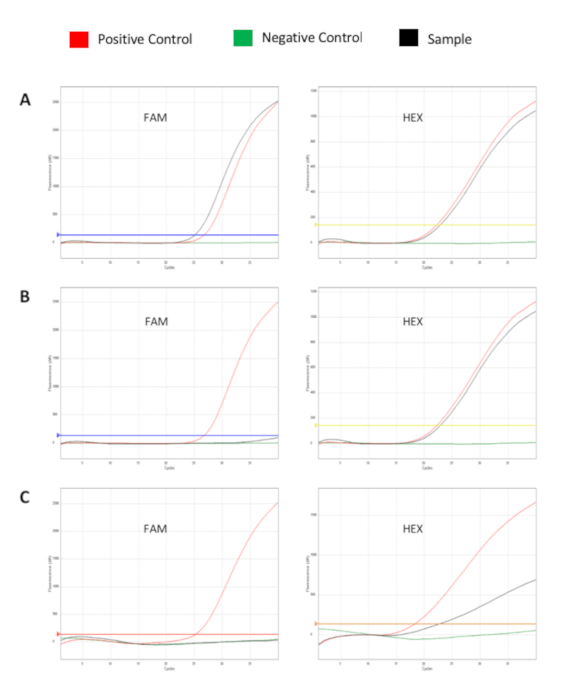
Figure 2 : Courbes de fluorescence montrant l’amplification des gènes cibles du mélange A (ou Mix B) (canal FAM) et du contrôle interne (canal HEX). (A) ÉCHANTILLON positif FL-ADN. (B) ÉCHANTILLON négatif FL-ADN. (C) Inhibition de l’amplification de l’échantillon. Courbe rouge : contrôle positif; courbe verte : contrôle négatif; courbe noire : échantillon clinique. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | X | |||
| Un | Norme 1 | Eau | DNA2 | DNA4 | DNA6 | DNA8 | DNA10 | DNA12 | DNA14 | DNA16 | DNA18 | DNA20 | 1 | MÉLANGE FL-ADN A | |
| B | Norme 1 | Eau | DNA2 | DNA4 | DNA6 | DNA8 | DNA10 | DNA12 | DNA14 | DNA16 | DNA18 | DNA20 | 2 | FL-ADN Mix B | |
| C | Norme 2 | Pos | DNA2 | DNA4 | DNA6 | DNA8 | DNA10 | DNA12 | DNA14 | DNA16 | DNA18 | DNA20 | 3 | MÉLANGE FL-ADN A | |
| D | Norme 2 | Pos | DNA2 | DNA4 | DNA6 | DNA8 | DNA10 | DNA12 | DNA14 | DNA16 | DNA18 | DNA20 | 4 | FL-ADN Mix B | |
| E | Norme 3 | DNA1 | DNA3 | DNA5 | DNA7 | DNA9 | DNA11 | DNA13 | DNA15 | DNA17 | DNA19 | DNA21 | 5 | MÉLANGE FL-ADN A | |
| F | Norme 3 | DNA1 | DNA3 | DNA5 | DNA7 | DNA9 | DNA11 | DNA13 | DNA15 | DNA17 | DNA19 | DNA21 | 6 | FL-ADN Mix B | |
| G | Norme 4 | DNA1 | DNA3 | DNA5 | DNA7 | DNA9 | DNA11 | DNA13 | DNA15 | DNA17 | DNA19 | DNA21 | 7 | MÉLANGE FL-ADN A | |
| H (en) | Norme 4 | DNA1 | DNA3 | DNA5 | DNA7 | DNA9 | DNA11 | DNA13 | DNA15 | DNA17 | DNA19 | DNA21 | 8 | FL-ADN Mix B |
Tableau 1 : Plaque d’ensemble avec distribution de contrôle, de courbe et d’échantillons. La colonne X indique le nombre imprimé sur le dessus de la bande.
| Étape | Température et temps |
| Hot Start (1 Cycle) | 95 oC pour 5 min |
| Amplification (40 cycles) | 95 oC pour 15 s 54 oC pour 15 s 60 oC pour 45 s (Data Collection) |
Tableau 2 : Profil thermique pour l’amplification de l’ADN.
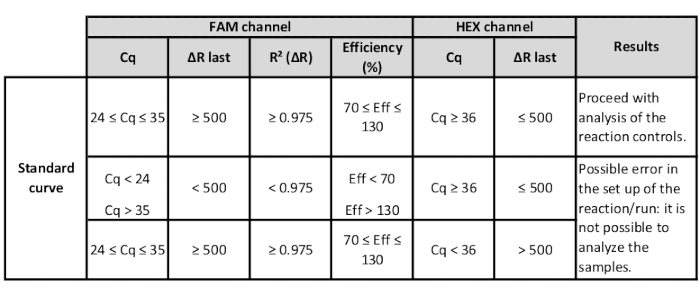
Tableau 3 : Gamme de valeurs des canaux HEX et FAM des normes pour vérifier l’adéquation de la courbe standard.

Tableau 4 : Gamme de valeurs de canaux HEX et FAM de contrôles négatifs et positifs pour vérifier l’adéquation de l’exécution.

Tableau 5 : Gamme de valeurs d’échantillons de canaux HEX et FAM pour vérifier la pertinence de l’analyse de l’échantillon.

Tableau 6 : Mélanger les valeurs A et Mix B Cq du canal FAM pour vérifier l’adéquation de l’analyse FL-ADN.
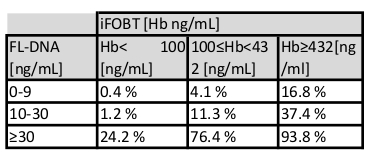
Tableau 7 : Évaluation du risque de cancer en fonction des valeurs iFOBT et FL-ADN. Selon la relation entre les valeurs d’iFOBT et d’ADN de FL, le tableau estime la probabilité des lésions néoplastiques côlorectales.
Discussion
Des études antérieures ont démontré que l’analyse de l’intégrité de l’ADN des selles extraites par des approches manuelles et semi-automatiques peut représenter un outil alternatif pour la détection précoce des lésions colorectales7,8,9,10,11,12. Des tests de dépistage moléculaires et non invasifs ont été mis au point au fil des ans pour la détection du cancer colorectal, mais la diffusion généralisée de ces méthodes est limitée en raison d’approches chronophages et d’une faible rentabilité par rapport à d’autres tests de dépistage.
Cette approche est relativement bon marché et ne prend pas trop de temps. Il a également une précision accrue dans la détection des lésions côlorectiques en raison d’une nouvelle procédure nécessitant peu d’étapes manuelles. L’approche décrite ici est rapide avec moins d’étapes manuelles, et il est en mesure d’être facilement effectué sur de nombreux échantillons par semaine. L’extraction de l’ADN ne présente pas de problèmes particuliers et peut être effectuée par des étapes manuelles faciles ou l’utilisation d’un instrument automatique. Dans ce dernier cas, il est nécessaire de déterminer l’instrument automatique d’extraction d’ADN, permettant les résultats les plus reproductibles. L’étape la plus critique de l’extraction de l’ADN est la collecte des selles et la méthode de son stockage avant l’extraction de l’ADN. Il est conseillé de garder le tabouret congelé et de procéder à l’extraction dès que possible.
Un autre problème critique est représenté par des inhibiteurs possibles des réactions d’amplification. L’extraction fécale n’est pas en mesure de purifier l’ADN génomique, car les impuretés compromettent la réaction d’amplification correcte. À cet égard, le protocole exige l’utilisation de l’ADN de pointe-in pour vérifier la présence /absence des inhibiteurs de réaction.
Jusqu’à récemment, le test sanguin occulte fécal est la principale approche utilisée pour détecter les lésions colorectales dans les programmes de dépistage; bien que, il présente certaines limites en termes de précision. Une stratégie alternative pour le diagnostic du cancer colorectal est basée sur l’analyse de l’ADN des cellules exfoliées excrétées dans les selles. Plusieurs approches ont été évaluées au cours de la dernière année, mais aucune n’est disponible pour les programmes de dépistage.
La valeur d’intégrité FL-ADN peut être une alternative utile, qui, en combinaison avec la valeur standard de test iFOBT de dépistage, peut prédire la présence de tumeurs et/ou d’adénomes à haut risque11,12 par une approche De Nomogram Fagan10 (tableau 7). Cette approche estime la probabilité combinée d’essai de la présence de lésion néoplastique, améliorant l’exactitude diagnostique comparée à l’approche standard utilisée seule.
Cette information peut aider les cliniciens à planifier des tests diagnostiques en plus d’une coloscopie appropriée et à personnaliser sa surveillance. En effet, le kit FL-ADN simplifie les étapes manuelles et assure des résultats stables et cohérents. Cependant, certaines questions restent à clarifier pour améliorer l’exactitude diagnostique de la méthode. Par exemple, la fréquence à laquelle les tests doivent être effectués et le nombre d’échantillons de selles qui doivent être analysés à des délais spécifiques pour chaque individu doivent être soigneusement sélectionnés. Étant donné que ce test doit être effectué en même temps que l’iFOBT, une méthode qui nécessite une collecte tous les 2 ans, comme c’est la norme dans de nombreux protocoles de dépistage, est nécessaire pour vérifier l’efficacité de ce test en remplacement ou en alternative aux tests de dépistage actuels.
Déclarations de divulgation
Maura Menghi est employée à plein temps de Diatech Pharmacogenetics srl.
Remerciements
Les auteurs n’ont aucune reconnaissance.
matériels
| Name | Company | Catalog Number | Comments |
| 1.5 mL and 2 mL polypropylene twist-lock tubes (DNase-, RNase-, DNA-, PCR inhibitor-free) | Consumables required for DNA extraction and Real Time PCR | ||
| Absolute Ethanol (quality of analytical degree) | Reagent required for DNA extraction | ||
| Benchtop centrifuge | Maximum speed of 20000 x g. Instrument required for DNA extraction | ||
| EasyPGX analysis software version 2.0.0 | Diatech Pharmacogenetics | RT800-SW | Analysis software |
| EasyPGX centrifuge/vortex 8-well strips | Diatech Pharmacogenetics | RT803 | Instrument recommended for the Real Time PCR assay |
| EasyPGX qPCR instrument 96 | Diatech Pharmacogenetics | RT800-96 | Instrument recommended for the Real Time PCR assay |
| EasyPGX ready FL-DNA | Diatech Pharmacogenetics | RT029 | Kit required for the Real Time PCR assay |
| Micropipettes (volumes from 1 to 1.000 µL) | Consumables required for DNA extraction and Real Time PCR | ||
| Powder-free disposable gloves | Consumables required for DNA extraction and Real Time PCR | ||
| QIAamp Fast DNA Stool | Qiagen | 51604 | Kit recommended for the DNA extraction and purification from stool |
| Sterile filter tips DNase-, RNase-free (volumes from 1 to 1.000 µL) | Consumables required for DNA extraction and Real Time PCR | ||
| Thermal block e.g. EasyPGX dry block | Diatech Pharmacogenetics | RT801 | Instrument required for DNA extraction |
| Vortex e.g. EasyPGX centrifuge/vortex 1.5 ml | Diatech Pharmacogenetics | RT802 | Instrument required for DNA extraction |
Références
- Fearon, E. R. Molecular Genetics of Colorectal Cancer. Annual Review of Pathology. 6, 479-507 (2011).
- Sears, C. L., Garrett, W. S. Microbes, Microbiota, and Colon Cancer. Cell Host and Microbe. 15, 317-328 (2014).
- Levin, B., et al. Screening and Surveillance for the Early Detection of Colorectal Cancer and Adenomatous Polyps, 2008: A Joint Guideline From the American Cancer Society, the US Multi-Society Task Force on Colorectal Cancer, and the American College of Radiology. Gastroenterology. 134, 1570-1595 (2008).
- Bosch, L. J., et al. Molecular tests for colorectal cancer screening. Clinical Colorectal Cancer. 10, 8-23 (2011).
- Ahlquist, D. A. Molecular detection of colorectal neoplasia. Gastroenterology. 138, 2127-2139 (2010).
- Calistri, D., et al. Fecal multiple molecular tests to detect colorectal cancer in stool. Clinical Gastroenterology and Hepatology. 1, 377-383 (2003).
- Calistri, D., et al. Detection of colorectal cancer by a quantitative fluorescence determination of DNA amplification in stool. Neoplasia. 6, 536-540 (2004).
- Calistri, D., et al. Quantitative fluorescence determination of long-fragment DNA in stool as a marker for the early detection of colorectal cancer. Cellular Oncology. 31, 11-17 (2009).
- Calistri, D., et al. Fecal DNA for noninvasive diagnosis of colorectal cancer in immunochemical fecal occult blood test-positive individuals. Cancer Epidemiology Biomarkers and Prevention. 19, 2647-2654 (2010).
- De Maio, G., et al. Circulating and stool nucleic acid analysis for colorectal cancer diagnosis. World Journal of Gastroenterology. 20, 957-967 (2014).
- Rengucci, C., et al. Improved stool DNA integrity method for early colorectal cancer diagnosis. Cancer Epidemiology Biomarkers and Prevention. 23, 2553-2560 (2014).
Erratum
Formal Correction: Erratum: Evaluation of Colorectal Cancer Risk and Prevalence by Stool DNA Integrity Detection
Posted by JoVE Editors on 9/28/2020. Citeable Link.
An erratum was issued for: Evaluation of Colorectal Cancer Risk and Prevalence by Stool DNA Integrity Detection. An affiliation was updated.
The first affiliation was updated from:
Istituto Scientifico Romagnolo per lo Studio e la Cura dei Tumori (IRST)
to:
Istituto Scientifico Romagnolo per lo Studio e la Cura dei Tumori (IRST) IRCCS, Meldola, Italy
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon