Method Article
Analisi della camera cardiaca sviluppo durante l'embriogenesi del Mouse utilizzando l'intero Monte epifluorescenza
In questo articolo
Riepilogo
Vi presentiamo i protocolli per esaminare lo sviluppo del cuore del mouse usando la microscopia intero Monte epifluorescente su embrioni di topo sezionati da ventricolare specifico MLC-2v-tdTomato reporter topi knock-in. Questo metodo permette di visualizzare direttamente ogni fase della formazione ventricolare durante lo sviluppo del cuore del mouse senza laboriosi metodi istochimici.
Abstract
L'obiettivo del presente protocollo è di descrivere un metodo per la dissezione degli embrioni del mouse e visualizzazione delle camere ventricolari embrionali del mouse durante lo sviluppo del cuore usando topi knock-in reporter fluorescenti specifici ventricolare (MLC-2v-tdTomato topi). Sviluppo del cuore implica una formazione cuore lineare del tubo, il tubo di cuore looping e quattro camera settazione. Questi complessi processi sono altamente conservati in tutti i vertebrati. Il cuore di embrionali del topo è stato ampiamente usato per studi evolutivi di cuore. Tuttavia, a causa della loro dimensioni estremamente ridotte, cuori embrionali del topo di dissezione è tecnicamente impegnativo. Inoltre, visualizzazione di formazione camera cardiaca spesso ha bisogno di ibridazione in situ, beta-galattosidasi che macchia usando topi reporter LacZ, o immunostaining del cuore embrionale sezionato. Qui, descriviamo come sezionare cuori embrionali del mouse e visualizzare direttamente la formazione di camera ventricolare di MLC-2v-tdTomato topi usando la microscopia intero Monte epifluorescente. Con questo metodo, è possibile esaminare direttamente la formazione del tubo di cuore e loop e quattro formazione di camera senza ulteriore manipolazione sperimentale degli embrioni del mouse. Anche se la linea di topo knock-in reporter MLC-2v-tdTomato è utilizzata in questo protocollo come esempio, questo protocollo può essere applicato ad altre linee di topi transgenici reporter fluorescenti specifici del cuore.
Introduzione
Formazione di camera durante lo sviluppo del cuore è un processo complesso, passando attraverso diversi stadi embrionali morfologicamente distinti1,2. La forma a mezzaluna cardiaco della popolazione delle cellule del progenitore forma un tubo lineare cuore e poi subisce allungamento e looping per formare la forma a spirale del cuore in via di sviluppo. Dopo il suo processo di settazione, cuore in via di sviluppo si trasforma in cuore quattro-a temperatura ambiente. Interruzione di uno qualsiasi di questi processi si traduce in difetti inerenti allo sviluppo del cuore. Pertanto, è importante capire i meccanismi molecolari sottostanti formazione camera durante lo sviluppo del cuore. Nonostante i numerosi studi precedenti su sviluppo del cuore, la nostra comprensione di questo complesso processo resta limitata.
L'ibridazione in situ, immunoistochimica e beta-galattosidasi che macchia usando topi reporter LacZ sono stati ampiamente usati per studiare la formazione di camera durante lo sviluppo del cuore del mouse dall'etichettatura cardiaci specifici o camera di specifici geni strutturali o proteine (ad es., Nppa, colpo di stato-TFII, Irx4, MLC-2a e MLC-2v)3,4,5,6,7,8,9,10. Tuttavia, questi esperimenti con embrioni di topo richiedono molto tempo e competenze, perché diverse fasi sperimentali devono essere eseguite in sequenza11. Qui, descriviamo un metodo di microscopia semplice intero Monte epifluorescente per visualizzare i ventricoli sviluppo utilizzando embrioni dissecati dal MLC-2v-tdTomato reporter topi knock-in12. Il vantaggio di questo metodo rispetto ai metodi precedenti è di evitare la complessa procedura sperimentale che spesso può creare variazioni sperimentali. Lo scopo principale di questo protocollo è descrivere come dissezionare embrioni di topo e cuori in via di sviluppo e di esaminare ogni fase dello sviluppo di alloggiamento cardiaco del mouse senza esperimenti istochimici noiosi. Questo metodo può essere applicato facilmente per valutare lo sviluppo di cuore utilizzando varie altre linee di topi transgenici etichettatura marcatori cardiaci precoci (ad es., Mesp1Cre: Rosa26EYFP13, Isl1Cre: Rosa26EYFP13, Hcn4H2BGFP14, Hcn4Cre: Rosa mT /mG14, Nkx2-5Cre: Rosa mT/mG14, Hcn4-eGFP15, Isl1Cre: Rosa mT/mG14, Nkx2.5Cre: Rosa26tdTomato15e topi16 TgMef2c-AHF-GFP).
Protocollo
Tutte le procedure degli animali sono state effettuate con l'approvazione del Comitato di uso e Vanderbilt University Medical Center istituzionale Animal Care.
1. mouse raccolta degli embrioni e dissezione
- Compagno di 8-10 settimane vecchio MLC-2v-tdTomato+ /- topi femmina con 8-10 settimane vecchio MLC-2v-tdTomato+ /- topo maschio per ottenere MLC-2v-tdTomato+ +, MLC-2v-tdTomato+ /- e MLC-2v-tdTomato- / - embrioni.
- Controllare le dighe per spine vaginale ogni mattina. Mezzogiorno del giorno di rilevamento plug vaginale è considerato come 0.5.
Nota: Esame vaginale per la rilevazione di un plug vaginale deve essere eseguita al mattino (entro 8-24 h dopo attività sessuale), poiché un plug vaginale può essere perso durante tutta la giornata. - Eutanasia le dighe incinte al coitum dell'alberino di giorni diversi (ad es., E8.5, e 10.5 e 12.5) usando l'inalazione di CO2 seguita da dislocazione cervicale.
- Posare i topi supino e spruzzare etanolo al 70% sull'addome di topi per evitare la contaminazione di capelli del mouse durante la dissezione.
- Aprire la cavità addominale da incisione della pelle e della parete addominale usando forbici chirurgiche e una pinza.
- Individuare bilaterali corni uterini nella parte dorsale della cavità addominale.
- Separare l'utero intero tagliando con attenzione sopra ovidotti su entrambi i lati utilizzando forbici chirurgiche e una pinza.
- Posizionare l'intero utero dissecato in un 10cm Petri con PBS ghiacciata e separare con cura ogni sacco amniotico lungo il corno uterino utilizzando forbici chirurgiche e una pinza.
- Trasferire ogni embrione in singoli pozzetti della piastra ben 6 riempito con gelida PBS utilizzando una pipetta di trasferimento.
- Sotto un microscopio per dissezione, aprire un sacco amniotico ed esporre ogni embrione, tagliando il cordone ombelicale utilizzando forbici chirurgiche e una pinza in un pozzo individuo di una piastra ben 6 con PBS ghiacciata.
- Ritagliare i tessuti extra-embrionali per quanto possibili senza danneggiare l'embrione utilizzando forbici chirurgiche e una pinza.
Nota: Intero Monte epifluorescente imaging di embrione di topo intero viene solitamente eseguita prima dissezione cuore in via di sviluppo, come descritto di seguito. - Tagliare la testa dell'embrione utilizzando forbici chirurgiche e una pinza e trasferirlo in una provetta da 1,5 mL con 100 µ l di tampone A (25 mM EDTA NaOH e 0,2 mM) per la genotipizzazione di correlare con i risultati di formazione immagine epifluorescente.
- Aprire lo scrigno dell'embrione utilizzando una pinzetta, togliere il cuore lontano da polmoni e sistema vascolare utilizzando forbici chirurgiche e forcipe e trasferire il cuore embrionale dissecato in un pozzetto di una piastra ben 12 con PBS utilizzando una pipetta di trasferimento. Tutte le procedure di dissezione sono compiuti sotto un microscopio per dissezione con un fibra ottica microscopio Illuminatore.
Nota: Era tecnicamente difficile sezionare il cuori embrionali del mouse al E8.0 o E8.5, a causa delle loro dimensioni estremamente ridotte e la fragile struttura. Cuori di embrionali precoce (cioè E8.0 ed E8.5) possono essere esaminati all'interno dell'embrione intero supporto senza dissezione.
2. intero-monta epifluorescenza imaging
- Posizionare la piastra ben 12 con cuori embrionali del mouse sotto un microscopio per dissezione di epifluorescente.
- Sotto un epifluorescente microscopio utilizzando una pinzetta per dissezione, posizionare il cuore embrionale in modo tale che i ventricoli in via di sviluppo si trovano vicino l'esaminatore.
- Regolare la messa a fuoco dell'immagine usando un obiettivo x 0.63 (intervallo di zoom tra x 3.15 e 18,9 x) in modalità campo luminoso.
- Le esposizioni del campo luminoso e catturare immagini multiple. Le immagini sono state ottenute solitamente una seconda esposizione. Tuttavia, tempi di esposizione possono variare a seconda della illuminazione e telecamera specificationss e devono essere ottimizzate per ogni installazione.
- Disattivare un fibra ottica microscopio Illuminatore e impostare il filtro per fluorescenza rossa (Ex545 nm/Em 605 nm) di visualizzare tdTomato espressione.
- Regolare nuovamente la messa a fuoco dell'immagine, se necessario.
- Regolare luminosità e contrasto, prendere esposizioni fluorescente rosse e catturare immagini multiple.
Nota: La seguente impostazione di immagine è stata usata solitamente: esposizione di s 1 volta, 2 x guadagno, 1,0 saturazione e correzione 1,0 gamma. L'impostazione ottimale deve essere ottimizzato per ogni esperimento. Una volta determinata l'impostazione ottimale, la stessa impostazione deve essere utilizzata per un esperimento intero.
3. genotipizzazione
- Bollire i campioni dal passaggio 1.12 per 1 h a 100 ° C.
- Centrifugare per 2 min a 11.360 x g, trasferire 20 µ l del surnatante in una nuova provetta da 1,5 mL con 20 µ l di tampone B (40 mM Tris-HCl, pH 5.5) e mescolare.
- Prendere 4,5 µ l del surnatante misto da passo 3.2 come un modello di DNA, combinarlo con 0,5 µ l di ciascuno dei specifici in avanti ed iniettori d'inversione (10 µM), 10 µ l di tampone della polimerasi e reazione premiscelato (2x) (Vedi Tabella materiali) e quindi aggiungere l'acqua per un totale v olazione di 20 µ l. sequenze dell'iniettore sono come di seguito.
F1: 5'-TACCCACGGAGAAGAGAAGGACT-3'
R1: 5'-TGGACTTCTTGGAACTGACTCTGT-3'
F2: 5'-ACGGCACGCTGATCTACAAGGT-3'
R2: 5'-TTTGCGCACAGCCCTGGGAT-3' - Eseguire una reazione a catena della polimerasi (PCR) con il seguente programma PCR (tabella 1 e tabella 2).
- Eseguire PCR campioni e scaletta del DNA su un gel di agarosio 1% a 140 V in 1 buffer x TAE (Tris-acetato-EDTA) (40 mM Tris-acetato e 1 mM EDTA) per 25 min. usare una scala di DNA di bp 100 per stimare le dimensioni delle bande PCR.
- Collocare il gel del DNA su un transilluminatore UV per individuare le bande di DNA e accendere il UV luce.
Risultati
Durante lo sviluppo del cuore, MLC-2v è considerato essere l'indicatore più in anticipo per camera ventricolare specifica:17. Come descritto in Figura 1, abbiamo dissecato fuori interi embrioni del mouse e hearts embrionali di topi knock-in reporter di MLC-2v-tdTomato ed esaminato MLC-2v-tdTomato reporter espressione durante lo sviluppo del cuore. In MLC-2v-tdTomato reporter topi knock-in, espressione costitutiva tdTomato nel cuore in via di sviluppo è visualizzato tramite epifluorescenza intero supporto immagini più presto a E8.012 (Figura 2). Relativamente debole espressione di tdTomato nel tubo lineare cuore alle E8.0 diventa più forte al E8.5. A e 10.5, MLC-2v-tdTomato reporter espressione è stata dimostrata nella parte ventricolare del cuore loop dissecato da un embrione di topo intero, mentre non è stato mostrato nel tratto di afflusso, il tratto di efflusso o atri futuri. A 12.5-E13.5, intero Monte epifluorescente imaging del cuore dissecato di embrione di topo knock-in reporter MLC-2v-tdTomato ha mostrato che il reporter di tdTomato è espressa esclusivamente nei ventricoli del cuore quattro-a temperatura ambiente. Il simile modello ventricolare espressione specifica di MLC-2v-tdTomato reporter è stato mostrato nell'embrione del mouse dissecata presso E16.5. Utilizzando questo metodo, possiamo facilmente rintracciare formazione camera ventricolare durante lo sviluppo del cuore del mouse.
Dopo formazione immagine intera Monte epifluorescente di embrioni di topo o dissecati cuori in via di sviluppo, abbiamo confermato in modo retrospettivo il genotipo dell'embrione usando la testa degli embrioni del mouse. Usando due insiemi degli iniettori come illustrato in Figura 3A, abbiamo effettuato la genotipizzazione PCR. Gli embrioni che trasportano l'allele wild-type ha mostrato un prodotto PCR bp 383 utilizzando il set di primer F1 e R1. Gli embrioni che trasportano il tdTomato knock-in allele ha mostrato il prodotto PCR bp 497 utilizzando il primer F2 e R2 impostare (Figura 3B). Eterozigoti embrioni sono stati definiti dimostrando entrambi il 383 bp e le 487 bande di bp, mentre il tipo selvaggio o genotipo omozigotico è stato determinato dimostrando un singolo 383 bp o 497 bp band, rispettivamente.
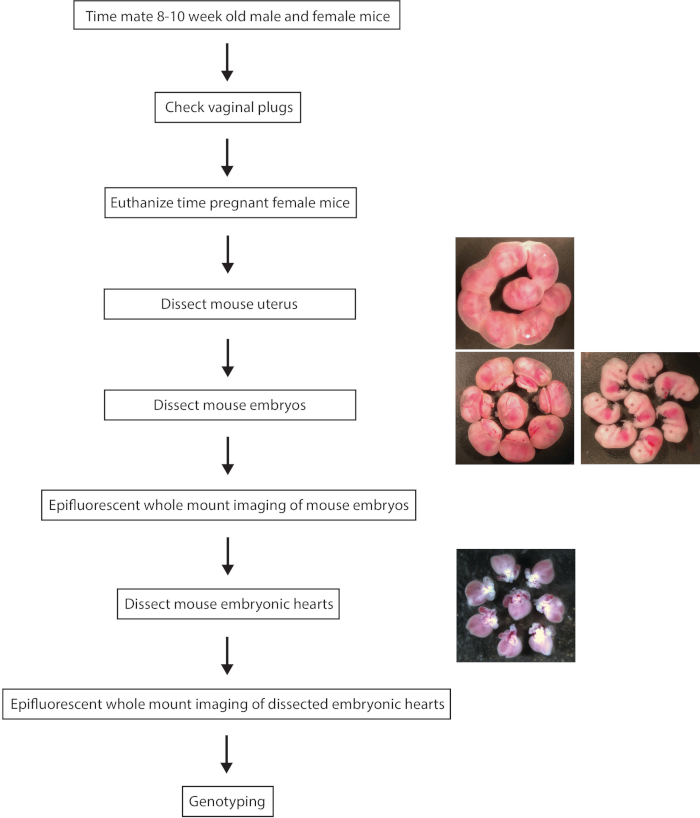
Figura 1. Contorno di graduale procedura sperimentale per l'intero Monte epifluorescente imaging dei cuori embrionali del mouse MLC-2v-tdTomato reporter. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2. Rappresentante epifluorescente immagini di interi embrioni e sviluppo cuori sezionati da topi knock-in reporter di MLC-2v-tdTomato. La formazione immagine epifluorescente di intero supporto embrioni e cuori sezionati a diversi stadi embrionali dimostra espressione specifica di tdTomato nei ventricoli di sviluppare i cuori. (A) intero embrione allo E8.0, (B) intero embrione allo E8.5, (C) intero embrione allo e 9.0, (D) intero embrione allo e 10.5, (E) intero embrione allo E13.5, embrione intero (F) a E16.5, (G) cuore embrionale alle e 9.0, (H ) Cuore embrionale e 10.5, (io) embrionali cuore a E13.5 e il cuore embrionale (J) al E16.5. Un, atrio; V, ventricolo; IFT, tratto di afflusso; OFT, tratto di efflusso. Scala bar (A\u2012F) = 1 mm; Scala bar (G-J) = 500 µm. Questa figura è stata modificata da riferimento #12 con permesso. Clicca qui per visualizzare una versione più grande di questa figura.
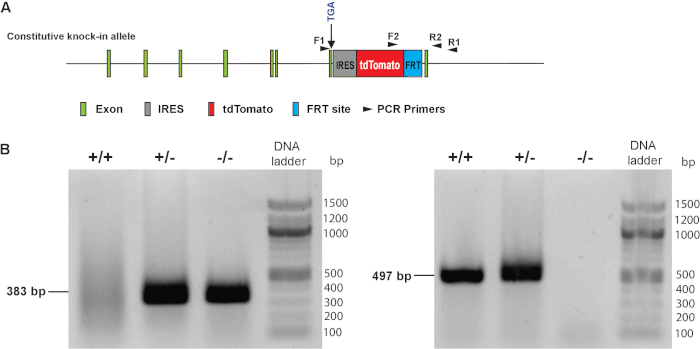
Figura 3. Genotipizzazione di MLC-2v-tdTomato reporter knock-in embrioni. (A) illustrazione di disegno dell'iniettore di genotipizzazione. (B) rappresentante genotipizzazione risultati utilizzando primer F1 e R1 (a sinistra) e F2 e R2 primer (a destra). + +: omozigotica, + /-: eterozigoti,- / -: wild-type. Esone: un segmento di un gene che contiene informazioni necessarie per la sintesi proteica; IRES: Sito Internal ribosome entry; FRT: flippase ricombinasiricombinazione bersaglio. Clicca qui per visualizzare una versione più grande di questa figura.
| Passo | Temp ° C | Tempo | Nota |
| 1 | 94 | 3 min | |
| 2 | 94 | 30 s | |
| 3 | 60 | 35 s | |
| 4 | 72 | 35 s | |
| 5 | Ripetere i passaggi 2-4 per 38 cicli | ||
| 6 | 72 | 5 min | |
| 7 | 10 | Tenere premuto |
Tabella 1: Programma PCR utilizzando primers F1 e R1
| Passo | Temp ° C | Tempo | Nota |
| 1 | 94 | 3 min | |
| 2 | 94 | 30 s | |
| 3 | 61,7 | 35 s | |
| 4 | 72 | 35 s | |
| 5 | Ripetere i passaggi 2-4 per 38 cicli | ||
| 6 | 72 | 5 min | |
| 7 | 10 | Tenere premuto |
Tabella 2: Programma PCR utilizzando primers F2 e R2
Discussione
Il metodo descritto qui è relativamente semplice per esaminare lo sviluppo camera ventricolare, senza laboriosi esperimenti per etichettare ventricolare o cardiaci specifici geni strutturali o proteine. Quindi, questo metodo riduce al minimo tecnico variabilities che spesso sono stati trovati nelle sezioni immunostained cuore.
Ci sono due passaggi critici per l'esecuzione con successo questo metodo tra cui stima precisa dell'età embrionali di topi e dissezione del cuore embrionale. Stimiamo praticamente l'età embrionale dei topi identificando vaginale spine nei topi femmina. Tuttavia, la presenza di un plug vaginale non indica necessariamente gravidanza. Indica solo che il rapporto sessuale si è verificato entro un periodo di circa 8-24 h. Anche se si trovano spine vaginali, è spesso difficile determinare se topi sono infatti incinto o non fino a 10-11 giorni post coitum dell'esaminando l'addome dei topi femminili. Allevamento più coppie di topi maschi e femmine è una strategia di allevamento sicuro per ottenere l'età prevista del mouse embrioni. Isolare lo sviluppo di cuori del mouse senza danni significativi alle loro strutture è fondamentale per esaminare accuratamente camera cardiaca sviluppo durante l'embriogenesi del mouse come illustrato nella Figura 1. La dissezione attenta sotto un microscopio per dissezione e la comprensione dello sviluppo anatomia cardiaca ogni fase embrionale prima della dissezione sono necessari per svolgere con successo questo protocollo.
Poiché in primo luogo è stata osservata, alle E8.0, MLC-2v-tdTomato reporter espressione, non è possibile esaminare i precedenti cuore embrionale tubo o cardiaco mezzaluna fase12. Le varie linee del mouse reporter etichettatura precedenti indicatori cardiaci (ad es. Mesp1Cre: Rosa26EYFP13, Isl1Cre: Rosa26EYFP13, Hcn4H2BGFP14, Hcn4Cre: Rosa mT/mG14, Nkx2-5Cre: Rosa mT/mG14, Hcn4-eGFP15 Isl1Cre: Rosa mT/mG14e Nkx2.5Cre: Rosa26tdTomato15, topi16 TgMef2c-AHF-GFP) può essere utilizzato per esaminare le fasi precedenti dello sviluppo camera utilizzando il metodo descritto in questo articolo.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è stato supportato da NIH R03 HL140264 (Y.-J. N) e programma di Gilead Sciences Research Scholar (Y.-J. N).
Materiali
| Name | Company | Catalog Number | Comments |
| dissecting microscope | Leica | MZ125 | |
| DNA ladder (100 bp) | Promega | G2101 | |
| epifluorescence dissecting microscope | Leica | M165 FC | |
| GoTaq Green master Mix | Promega | M712 | |
| PCR machine (master cycler) | Eppendorf | 6336000023 |
Riferimenti
- Evans, S. M., Yelon, D., Conlon, F. L., Kirby, M. L. Myocardial lineage development. Circulation Research. 107 (12), 1428-1444 (2010).
- Paige, S. L., Plonowska, K., Xu, A., Wu, S. M. Molecular regulation of cardiomyocyte differentiation. Circulation Research. 116 (2), 341-353 (2015).
- Lee, K. J., et al. Myosin light chain-2 luciferase transgenic mice reveal distinct regulatory programs for cardiac and skeletal muscle-specific expression of a single contractile protein gene. Journal of Biological Chemistry. 267 (22), 15875-15885 (1992).
- Chen, J., et al. Selective requirement of myosin light chain 2v in embryonic heart function. Journal of Biological Chemistry. 273 (2), 1252-1256 (1998).
- Ross, R. S., Navankasattusas, S., Harvey, R. P., Chien, K. R. An HF-1a/HF-1b/MEF-2 combinatorial element confers cardiac ventricular specificity and established an anterior-posterior gradient of expression. Development. 122 (6), 1799-1809 (1996).
- Christoffels, V. M., et al. Chamber formation and morphogenesis in the developing mammalian heart. Dev Biol. 223 (2), 266-278 (2000).
- Wu, S. P., et al. Atrial identity is determined by a COUP-TFII regulatory network. Developmental Cell. 25 (4), 417-426 (2013).
- Nelson, D. O., Jin, D. X., Downs, K. M., Kamp, T. J., Lyons, G. E. Irx4 identifies a chamber-specific cell population that contributes to ventricular myocardium development. Developmental Dynamics. 243 (3), 381-392 (2014).
- Bao, Z. Z., Bruneau, B. G., Seidman, J. G., Seidman, C. E., Cepko, C. L. Regulation of chamber-specific gene expression in the developing heart by Irx4. Science. 283 (5405), 1161-1164 (1999).
- Kubalak, S. W., Miller-Hance, W. C., O'Brien, T. X., Dyson, E., Chien, K. R. Chamber specification of atrial myosin light chain-2 expression precedes septation during murine cardiogenesis. Journal of Biological Chemistry. 269 (24), 16961-16970 (1994).
- Bardot, P., et al. The TAF10-containing TFIID and SAGA transcriptional complexes are dispensable for early somitogenesis in the mouse embryo. Development. 144 (20), 3808-3818 (2017).
- Zhang, Z., Nam, Y. J. Generation of MLC-2v-tdTomato knock-in reporter mouse line. Genesis. , (2018).
- Ieda, M., et al. Direct reprogramming of fibroblasts into functional cardiomyocytes by defined factors. Cell. 142 (3), 375-386 (2010).
- Liang, X., et al. HCN4 dynamically marks the first heart field and conduction system precursors. Circulation Research. 113 (4), 399-407 (2013).
- Nam, Y. J., et al. Induction of diverse cardiac cell types by reprogramming fibroblasts with cardiac transcription factors. Development. 141 (22), 4267-4278 (2014).
- Spater, D., et al. A HCN4+ cardiomyogenic progenitor derived from the first heart field and human pluripotent stem cells. Nature Cell Biology. 15 (9), 1098-1106 (2013).
- O'Brien, T. X., Lee, K. J., Chien, K. R. Positional specification of ventricular myosin light chain 2 expression in the primitive murine heart tube. Proceedings of the National Academy of Sciences of the United States of America. 90 (11), 5157-5161 (1993).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon