Method Article
PCR digitale per quantificare circolanti microRNA nell'infarto miocardico acuto e la malattia cardiovascolare
In questo articolo
Riepilogo
MicroRNA circolanti hanno indicato la promessa come biomarcatori per malattie cardiovascolari e infarti miocardici acuti. In questo studio, descriviamo un protocollo per l'estrazione di miRNA, trascrizione d'inversione e digitali PCR per la quantificazione assoluta dei miRNA nel siero dei pazienti con la malattia cardiovascolare.
Abstract
Circolanti nel siero i microRNA (Mirna) hanno indicato la promessa come biomarcatori per la malattia cardiovascolare ed infarto miocardico acuto (AMI), rilasciato dalle cellule cardiovascolari nella circolazione. Mirna circolanti sono altamente stabili e possono essere quantificati. L'espressione quantitativa di miRNA specifici possa essere collegato per la patologia e alcuni miRNA Visualizza alta tessuto e specificità di malattia. Trovare nuovi biomarcatori per le malattie cardiovascolari è di importanza per la ricerca medica. Abbastanza recentemente, reazione a catena della polimerasi digitale (dPCR) è stata inventata. dPCR, combinato con sonde fluorescenti idrolisi, consente specifica diretta quantificazione assoluta. dPCR esibisce qualità tecniche superiori, tra cui una bassa variabilità, alta linearità e alta sensibilità rispetto alla reazione a catena della polimerasi quantitativa (qPCR). Così, dPCR è un metodo più accurato e riproducibile per quantificare direttamente Mirna, specialmente per l'uso in ampi studi clinici cardiovascolari del multi-center. In questa pubblicazione, descriviamo come eseguire efficacemente PCR digitale al fine di valutare il numero di copia assoluta nei campioni di siero.
Introduzione
Mirna circolanti sono stati identificati come promettenti marcatori per un certo numero di malattie, compreso la malattia cardiovascolare1. I miRNA sono piccoli, non codificanti molecole di RNA singolo-incagliato (lunghe circa 22 nucleotidi) sono coinvolti nella regolazione post-trascrizionale attraverso l'alterazione della traduzione di RNA messaggero e d'influenza gene expression2 e vengono rilasciati nella circolazione negli Stati sia fisiologiche che patologiche. L'espressione quantitativa di miRNA specifici possa essere collegato alla patologia e alcuni miRNA Mostra alto tessuto e malattia specificità1. Nelle malattie cardiovascolari, Mirna sono diventati i candidati attraenti come nuovi biomarcatori perché sono notevolmente stabili nel siero e può facilmente essere quantificati con l'aiuto di PCR metodologia3. Il valore potenziale di Mirna come biomarcatori per infarto del miocardio è stato valutato in piccoli studi, ma una convalida in larghe coorti è carente2. Ad esempio, miR-499 è trovato altamente espressi nel muscolo del miocardio, e ha dimostrato di essere significativamente aumentata in un AMI4,5,6. Inoltre, esso regola programmato la morte delle cellule (apoptosi) e la differenziazione dei cardiomiociti e così è compreso in parecchi meccanismi segue un AMI7. A parte alcuni piccoli studi che riferiscono una superiorità e il valore incrementale di Mirna per la diagnosi di AMI, la superiorità o uguaglianza di alto-sensibilità troponine cardiache non è ancora stato dimostrato in studi su larga scala2,5 ,6,8. Ulteriori studi prospettici in larghe coorti sono, pertanto, necessari per valutare il potenziale valore diagnostico di Mirna. Inoltre, metodi di quantificazione di miRNA devono essere ottimizzate e9di protocolli standardizzati utilizzando comparabili. Analisi standardizzate possono ridurre risultati incoerenti e possono aiutare Mirna a diventare potenziali biomarcatori per l'applicazione clinica di routine, come biomarcatori devono essere quantificati in modo riproducibile per garantire la loro applicabilità clinica.
Recentemente, dPCR è stato introdotto come un'analisi di punto finale. Esso suddivide il campione in circa 20.000 reazioni individuali10. Il sistema di dPCR utilizza quindi una matematico Poisson analisi statistica dei segnali fluorescenti (reazioni positive e negative), che consente una quantificazione assoluta senza una curva standard10. Quando si combinano dPCR con sonde fluorescenti idrolisi, la quantificazione assoluta diretta altamente specifica dei miRNA è reso possibile. Reazione a catena della polimerasi digitale ha dimostrato che mostra qualità tecniche superiori (tra cui una variabilità in diminuzione, una maggiore riproducibilità quotidiana, un alto grado di linearità e un'alta sensibilità) per quantificare i livelli di miRNA nella circolazione rispetto al quantitativo in tempo reale PCR10,11. Queste qualità tecniche superiori potrebbe contribuire ad per attenuare le attuali limitazioni sull'utilizzo di Mirna circolanti come biomarcatori e può condurre all'istituzione di Mirna come biomarcatori in ampi studi clinici cardiovascolari del multi-center e come metodo diagnostico in generale. In uno studio precedente, abbiamo recentemente applicato dPCR per la quantificazione assoluta di Mirna in pazienti con IMA in circolazione e sono stati in grado di dimostrare il potenziale diagnostico superiore rispetto alla quantificazione dei miRNA qPCR12.
In questa pubblicazione, vogliamo dimostrare che l'utilizzo di dPCR è un metodo accurato e riproducibile per quantificare direttamente la circolazione cardiovascolari Mirna. La quantificazione assoluta di miRNA livelli nel siero, usando la PCR digitale, Mostra il potenziale per l'uso in ampi studi clinici cardiovascolari del multi-center. In questa pubblicazione, descriviamo in dettaglio come eseguire efficacemente PCR digitale e come rilevare il numero di copie di miRNA assoluta nel siero.
Protocollo
1. estrazione di miRNA da Plasma/siero
Nota: Al fine di quantificare i miRNA in modo appropriato, l'isolamento di microRNA corretta dal plasma/siero è un passo cruciale. Una cosa fondamentale da tenere a mente, soprattutto perché esistono diversi protocolli, è di aderire al flusso di lavoro stesso durante l'elaborazione dei campioni. In questo protocollo, miRNA è estratta da 50 µ l di siero. Non utilizzare più di 200 µ l come questo limite il processo di estrazione corretta.
- Preparare il siero o scongelare i campioni congelati.
- Aggiungere 250 µ l (5 volumi) di reagente di lisi commerciale 50 µ l di siero. Sostenere la lisi nel Vortex. Posizionare il tubo con il lisato sul banco a temperatura ambiente per 5 min.
- Spike il lisato con 3,5 µ l di controllo di spike (1,6 x 107 copie / µ l) e mescolare nel Vortex.
- Aggiungere 50 µ l di cloroformio (cioè, un importo pari per quanto riguarda il campione di siero di partenza) per la separazione di fase. Agitare energicamente il tubo per 15 s. posto il tubo sul banco a temperatura ambiente per 2-3 min.
- Per la separazione di fase, centrifugare la provetta a 12.000 x g a 4 ° C per 15 min.
Nota: I campioni sono ora separati in una fase acquosa superiore contenente il RNA, un bianco interfase e una fase organica inferiore, rosa. - Trasferire la fase acquosa superiore (100 µ l) contenente il RNA in un nuovo tubo. Non trasferire qualsiasi materiale bianco interfase come questo falsifica il conteggio corretto di RNA.
- Aggiungere 150 µ l (cioè, 1,5 volte il volume del campione trasferito) di etanolo al 100%. Mescolare i materiali li pipettando su e giù. Non centrifugare li e rapidamente passare al passaggio successivo.
- Pipettare 250 µ l del campione su una colonna commerciale spin in un tubo di raccolta da 2 mL. Chiudere il coperchio. Centrifugare la colonna > 8.000 x g e a temperatura ambiente per 15 s. scartare il flusso continuo.
- Aggiungere 700 µ l di numero di buffer di lavaggio commerciale 1 nella colonna di selezione. Chiudere il coperchio e centrifugare la colonna > 8.000 x g e a temperatura ambiente per 15 s. scartare il tampone di lavaggio di prova generale.
- Aggiungere 500 µ l di numero di buffer di lavaggio commerciale 2 nella colonna di selezione. Chiudere il coperchio e centrifugare colonna in > 8, 000 x g a temperatura ambiente per 15 s. scartare il tampone di lavaggio di prova generale.
- Pipettare 500 µ l di etanolo di 80% per la colonna di spin. Chiudere il coperchio, centrifugare la colonna a > 8, 000 x g e a temperatura ambiente per 2 min. gettare la provetta di raccolta con il flusso continuo.
- Trasferire la colonna di rotazione ad un nuovo tubo di raccolta da 2 mL. Centrifugare la colonna a tutta velocità con un coperchio aperto ad asciugare la membrana. Gettare la provetta di raccolta con il flusso continuo.
- Trasferire la colonna di spin in un nuovo tubo di raccolta di 1,5 mL. Eluire il RNA applicando 30 µ l di acqua RNAsi-libera direttamente al centro della membrana. Chiudere il coperchio. Centrifugare la colonna a tutta velocità per 1 min.
- Conservare il RNA-70 ° C.
Nota: Il deposito di RNA purificato in acqua RNAsi-libera tra-70 ° C a-20 ° C non assicura nessuna degradazione del RNA per 1 anno. Seguendo il protocollo del produttore, l'importo minimo di RNAsi-libera di acqua per diluire il RNA è 10 µ l. utilizzando meno di 10 µ l insufficientemente può idratare la membrana e riducono il rendimento di RNA totale.
2. trascrizione inversa
Nota: Il DNA complementare (cDNA) è stato sintetizzato utilizzando il seguente protocollo di trascrizione inversa (RT) di 15 µ l (volume di reazione totale).
- Scongelare il kit RT sul ghiaccio. Mantenere gli enzimi nel congelatore finchè possibile impedire loro di degradante e loro si sono fermati prima dell'uso. Scongelare i primer RT sul ghiaccio e li spin giù prima dell'uso.
- Preparare il mix master come descritto nella tabella 1.
- Unire 10 µ l di mix master e 5 µ l di RNA estratto per pozzetto di una piastra a 96 pozzetti.
- Centrifugare la piastra ben a 2.000 x g a 4 ° C per 2 min.
- Utilizzare il protocollo di ciclo termico descritto in tabella 2 per la trascrizione d'inversione.
Nota: Come descritto di seguito, cDNA può direttamente essere trattato nella macchina digitale PCR (Vedi Tabella materiali); in alternativa, può essere conservato a-20 ° C.
3. gocciolina generazione e digitali PCR
Nota: Digital PCR è stata effettuata utilizzando il seguente protocollo di 40 µ l.
- Scongelare il mix commerciale dPCR, il cDNA preparato e gli iniettori PCR sul ghiaccio. Centrifugare a loro poco prima dell'uso.
- Preparare il mix master, come descritto in tabella 3.
- Aggiungere 1,33 μL/pozzetto del prodotto RT ed e centrifugare.
- Pipettare 20 μL del campione in ciascun pozzetto della cassetta 8 pozzetti (media riga).
Nota: Se la cassetta 8 pozzetti non è completamente riempita, riempire i pozzetti rimanenti con controlli non modello (NTCs) che omettono di cDNA (cioè, il prodotto di RT producerat con acqua invece di RNA estratto) o buffer commerciale dPCR. La NTC serve come un controllo di contaminazione. Acqua non deve essere utilizzato nei restanti pozzetti, perché che potrebbe compromettere la qualità della formazione delle gocce. - Pipettare 70 μL dell'olio di generazione di goccia per le sonde in pozzi di petrolio (riga inferiore) della cassetta 8 pozzetti.
- Posizionare una guarnizione sopra la cassetta 8 pozzetti e inserire la cassetta nel generatore di goccia. Chiudere il generatore di goccia. Attendere che le spie 3 sono verde fisso.
Nota: Le goccioline vengono ora generate. - Lentamente e con cautela trasferire 40 μL del campione formato della gocciolina (riga superiore) in pozzetti separati di una piastra a 96 pozzetti di pipettaggio.
- Dopo aver completato la formazione della gocciolina sui campioni, sigillare la piastra PCR con un foglio di dPCR PERFORABILE a 180 ° C per 4 s.
- Posizionare la piastra PCR a lamina-sigillato nel ciclatore e azionarla come descritto nella tabella 4, ad un tasso di rampa di 2,5 ° C/s.
4. gocciolina lettura e l'analisi
- Trasferire la piastra dal termociclatore alla base del proprietario del piatto. Serrare la piastra PCR posizionando ilvertice il portatarga sulla piastra di PCR.
- Avviare il software commerciale dPCR.
- Immettere il nome di esempio, il nome dell'esperimento e il bersaglio nel setup. Selezionare la quantificazione assoluta.
- Avviare la gocciolina lettura facendo clic su Esegui. Selezionare FAM/HEX o FAM/VIC come chimica di rilevamento quando si lavora con sonde di idrolisi.
Nota: Il software commerciale dPCR quindi auto-analizzerà i dati. - Controllare i numeri di goccia nella tabella dei risultati per garantire una generazione corretta della gocciolina.
- Selezionare tutti i pozzetti con la stessa miRNA quantificato e fare clic su analizza. Utilizzare il blot 2D per contrassegnare le goccioline positive e correggere manualmente i dati auto-analizzati (Figura 1).
5. correggere i calcoli sul campione
- Normalizzare l'esempio moltiplicando il campione per il fattore di normalizzazione (NF).
NF = mediana [c. elegans miR-39 misure]tutti i campioni/ [c. elegans miR-39]dato campione - Calcolare la concentrazione nel siero di miRNA finale, regolazione per fattori di diluizione (DF).
[miRNA] finale = [miRNA]valore raw x DFRT x DFdPCR x DFEx
Dove:
[miRNA] valore non elaborato = il miRNA quantificato dal software commerciale dPCR;
DFRT = il fattore di diluizione del modello nella trascrizione d'inversione;
DFdPCR = il fattore di diluizione del modello in dPCR;
DFEx = il fattore di diluizione nell'estrazione.
6. serie di diluizioni di Oligonucleotide sintetico
- Scongelare i liofilizzati oligonucleotide sintetico sul ghiaccio. Brevemente e centrifugare.
- Diluire il liofilizzato oligonucleotide sintetico in acqua priva di nucleasi a una concentrazione finale di 10 pmol / µ l. brevemente centrifuga esso.
- Diluire in acqua priva di nucleasi come descritto nella tabella 5. Centrifugare la diluizione brevemente tra ogni fase di diluizione a 8.000 x g per 10 s.
- Proseguire con la trascrizione d'inversione come descritto nel passaggio 2 e analizzare i campioni con PCR digitale come descritto nei passaggi 3 e 4.
Risultati
PCR digitale combinato con sonde fluorescenti idrolisi consente ai ricercatori di quantificare direttamente gli importi assoluti di miRNA specifici in copie / µ l. Come l'esempio in dPCR è partizionata in circa 20.000 singole reazioni di PCR, dPCR non richiede tecniche replica10. Il sistema di dPCR utilizza un'analisi statistica di Poisson matematica dei segnali fluorescenti (differiscono tra reazioni positive e negative) che consente una quantificazione assoluta senza la necessità di uno standard curva10. Al fine di calcolare correttamente le concentrazioni, è necessario un conteggio di goccia accettabile (> 15.000). Nessun controllo di modello assicurare l'esclusione di una notevole amplificazione aspecifico. Tutti i campioni inclusi nell'analisi vengono elaborati utilizzando il volume stesso, il metodo e il protocollo di PCR per rafforzare la validità dei risultati ottenuti. Le concentrazioni ottenute in dPCR sono normalizzate utilizzando una procedura di normalizzazione mediano attraverso tutti i campioni analizzati con il sintetico a spillo-in c. elegans miR-3913. Normalizzando i dati per la quantità misurata di spillo-in c. elegans miR-39 corregge per la variazione del campione a campione dell'efficienza di estrazione di RNA e serve come un controllo di reazione di PCR, aggiungendo ulteriore per la validità dei risultati. Non c'è nessun stabilito gold standard per la normalizzazione del siero Mirna; Tuttavia, controlli esogeni quali lo spillo-in c. elegans miR-39 sono una soluzione elegante per le procedure di normalizzazione, come Mirna endogeni sono spesso alterati in vari Stati di malattia9.
I campioni di siero analizzati sono stati acquisiti prima di un intervento coronarico percutaneo (PCI), 8 h dopo un PCI e 16 h dopo un PCI, dai pazienti con un infarto miocardico acuto ST-segmento elevazione (STEMI). STEMI pazienti esibiscono ischemia severa e così qualificano per la valutazione di nuovi biomarcatori di ischemia. Per valutare la cinetica di rilascio miR-499 e l'uso potenziale di miR-499 come biomarcatore per ischemia nella malattia cardiovascolare, miR-499 è stato analizzato con dPCR in tutti i pazienti per i tre tempi diversi punti14.
Figura 1 viene illustrata la selezione di goccioline positive dando la concentrazione di miRNA finale calcolata dal software. La frazione di positivi in un campione determina la concentrazione di copie / µ l come calcolato dal software commerciale dPCR. I risultati possono anche essere visualizzati in un grafico 2D e gli aspetti positivi possono essere contrassegnati manualmente con i cerchi. Le concentrazioni sono considerate solo se sono di sopra l'amplificazione noa specifico della NTCs.
La figura 2 Mostra la linearità e la bontà di adattamento di una serie di diluizioni di miRNA sintetico del oligonucleotide ha-miR-499-5 p in duplicati. Una serie di diluizioni di 8 fasi è stato effettuato da 2.500 copie / µ l a 0 copie / µ l. Il calcolato previsto copie / µ l come descritto nella tabella 5 si supponga 100% RT e l'efficienza digitale di PCR. In questa configurazione, PCR digitale dimostra un elevato grado di linearità (r2 = 0,99). Il limite di rilevabilità (LoD = 0.12) e il limite di quantificazione (LoQ = 0.23) inoltre sono presentati in Figura 2, Mostra che anche un'espressione di miRNA basso può essere rilevata con successo. Così, dPCR può essere utilizzato per quantificare anche bassi livelli del siero di Mirna in circolazione. Il limite di rilevabilità (LoD) e limite di quantificazione (LoQ) sono calcolati seguendo l'approccio di Forootan et al. 15 e sono definiti come LoD = LoB + 1.645 x σbassoconcentrazionecampione, dove è calcolato il limite di spazio vuoto (LoB) come LoB = mediavuoto + 1.645 x σvuoto. LoQ è poi stimato replicare in esecuzione standard curve15. Per garantire la quantificazione riproducibile dei miRNA, miRNA soltanto le concentrazioni che si trovano di sopra sia il LoD e LoQ sono considerate come un valido valore esatto.
La figura 3 Mostra i risultati rappresentativi dei livelli di miR-499 di pazienti con un infarto miocardico di altezza di ST (STEMI), n = 16 in ogni momento. Campioni sono stati prelevati prima di un intervento percutaneo (PCI) (t = 0), 8 h dopo un PCI (t = 8) e 16 h dopo un PCI (t = 16), e i livelli di miR-499 erano rispetto ai livelli di miR-499 pazienti con coronaropatia stabile (CAD, n = 20). Le principali caratteristiche del paziente possono essere trovate nella tabella 6. Dopo il rilascio iniziale di miR-499 nel siero nelle prime 8 ore dopo un infarto miocardico, livelli di miR-499 diminuiscono nuovamente dopo le 16h. Una tendenza in aumento può essere già visto all'inizio del STEMI (t = 0), e un aumento significativo dei livelli di miR-499 rispetto ai pazienti con CAD può essere visto 8 h dopo la STEMI (t = 8, p < 0.01) quando si confrontano i livelli di miR-499 livelli di miR-499 di pazienti stabili di CAD. Il funzionamento del ricevitore curve Visualizza (ROC) la possibile utilità di miR-499 come un nuovo biomarcatore per differenziare fra i pazienti con CAD e pazienti con un STEMI [l'area sotto la curva (AUC) = 0,62 per campioni prelevati prima di un intervento percutaneo (PCI), l'AUC = 0,75 per campioni prelevati 8 h dopo un PCI e AUC = 0,78 per campioni prelevati 16 h dopo un PCI rispetto ai livelli del siero di miR-499 in pazienti con CAD].

Figura 1: selezione di goccioline positive in una serie di diluizioni eseguite in duplicati. (A) la soglia è impostata manualmente sopra le goccioline negative. (B) i risultati sono visualizzati in una macchia 2D, i lati positivi, selezionati da un cerchietto. Clicca qui per visualizzare una versione più grande di questa figura.
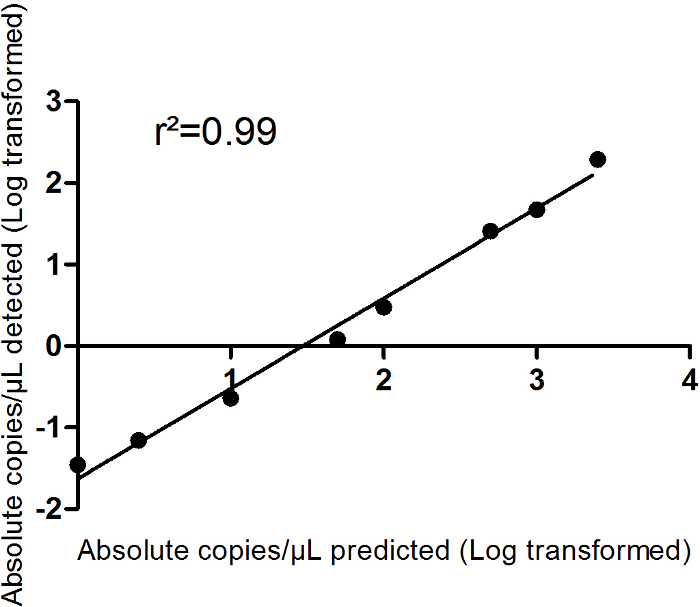
Figura 2: serie di diluizioni di oligonucleotide sintetico di miR-499. (A) le misurazioni sono state eseguite in duplicato. I dati sono presentati registro trasformato come assolute copie per µ l. regressioni lineari e bontà di adattamento (r2-valore) sono mostrati. I dati sono presentati come media ± SEM. (B), questo pannello mostra il limite di rilevabilità (LoD) e limite di quantificazione (LoQ). Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Quantificazione di circolanti nel siero miR-499-5p in pazienti con infarto miocardico di altezza di ST (n = 16) prima di intervento coronarico percutaneo (PCI), 8 h dopo PCI e 16 h dopo PCI, rispetto ai pazienti con coronaropatia stabile malattia (n = 20). (A) la circolazione del siero miR-499-5p è rappresentato come media con l'errore standard della media con il corrispondente p-valore calcolato da One-way ANOVA seguita da un test post hoc di Bonferroni (p < 0,05 è stato considerato come statisticamente significativa). (B) un ricevitore opera analisi della curva caratteristica (ROC) viene eseguito sui dati dal pannello A. L'analisi ROC dimostra la sensibilità e la specificità del biomarcatore. Inoltre, l'area corrispondente sotto i valori della curva (AUC) sono mostrati. Clicca qui per visualizzare una versione più grande di questa figura.
| quantità di campione | ||||
| n = 1 | / bene [µ l] | |||
| acqua priva di nucleasi | 4,16 | |||
| 10x buffer della trascrizione inversa | 1.5 | |||
| dNTP 100nm | 0.15 | |||
| Inibitore di RNAsi | 0,19 | |||
| Primer specifico RT | 3 | |||
| Commerciale Trascrittasi inversa enzima 50 U/ul | 1 | |||
| Master-Mix totale | 10 | |||
Tabella 1: Trascrizione inversa reagenti Mix.
| Temperatura | Tempo |
| 16 ° C | 30 minuti |
| 42 ° C | 30 minuti |
| 85 ° C | 5 minuti |
| 4 ° C | ∞ |
Tabella 2: Condizioni termiche in bicicletta per trascrizione inversa.
| quantità di campione | ||||
| n = 1 | / bene [µ l] | |||
| acqua priva di nucleasi | 7.67 | |||
| commerciale dPCR mix | 10 | |||
| 20 x idrolisi specifici primer/sonda | 1 | |||
| Master-Mix totale | 18.67 | |||
Tabella 3: PCR digitale agitare.
| Temperatura | Tempo |
| 95 ° C | 10 minuti |
| 94 ° C | 30 secondi (x40) |
| 60 ° C | 1 min |
| 98 ° C | 10 minuti |
| 4 ° C | ∞ |
Tabella 4: Condizioni di ciclismo termica per PCR digitale.
| Tubo | Trasferito il oligonucleotide sintetico miR-499 (µ l) | Diluente (µ l) | miRNA (copie / µ l) | Previsto copie / µ l in RT | Previsto copie / µ l in dPCR |
| Originale | 6.022 x 1012 | ||||
| 1 | 10 | 990 | 6.022 x 1010 | ||
| 2 | 10 | 990 | 6.022 x 108 | ||
| 3 | 10 | 990 | 6.022 x 106 | ||
| 4 | 18,7 | 981.3 | 1.128 x 105 | 37600 | 2500 |
| 5 | 40 | 60 | 4.511 x 104 | 15040 | 1000 |
| 6 | 50 | 50 | 2.256 x 104 | 7520 | 500 |
| 7 | 40 | 160 | 4.511 x 103 | 1504 | 100 |
| 8 | 50 | 50 | 2.256 x 103 | 752 | 50 |
| 9 | 40 | 160 | 4.511 x 102 | 150.4 | 10 |
| 10 | 50 | 150 | 1.128 x 102 | 37,6 | 2.5 |
| 11 | 0 | 50 | 0 | 0 | 0 |
Tabella 5: Serie di diluizione sintetica per miR-499.
| Caratteristica | Tutti i | CAD pazienti stabili (n = 20) | STEMI di pazienti (n = 24) | valore di p |
| Età | 64,7 ± 11,9 | 66,7 ± 13,1 | 62,2 ± 9,9 | 0.2810 |
| Maschi | 77,8% | 65% | 93,8% | 0.0392 |
| DM | 33,3% | 40% | 25% | 0.3428 |
| HTN | 61,1% | 60% | 62,5% | 0.8785 |
| Dislipidemia | 38,9% | 65% | 6,3% | 0,0003 |
| Fumatore | 47,2% | 55% | 37,5% | 0.3796 |
| Storia di famiglia | 25% | 35% | 12,5% | 0.1213 |
| Sovrappeso | 58,3% | 55% | 62,5% | 0.6501 |
| Creatinina sierica (mg/dL) | 0,94 (0.83 - 1) | 0,98 (0,82 - 1.01) | 0.93 (0.83 - 1) | 0,7255 |
| Picco CK (U / I) | 161 (102-611) | 126 (81-161) | 492 (228-3208) | 0,0004 |
| cTnT Peak (ng/L) | 322,5 (23,8-4533) | 18 (7-25.3) | 2492 (240-5586) | 0,0002 |
| LVEF % | 45% (40% - 50%) | 45% (32,5% - 55%) | 45% (45% - 50%) | 0.9596 |
| I valori sono presentati come media ± deviazione standard; valore mediano (intervallo di 25 al 75 ° percentile) o % | ||||
| DM: diabete mellito | ||||
| HTN: ipertensione | ||||
| CK: creatinchinasi | ||||
| cTnT: troponina cardiaca T | ||||
| LVEF: Frazione di eiezione ventricolare sinistra | ||||
Tabella 6: Caratteristiche principali del paziente.
Discussione
Digitale PCR è un metodo relativamente nuovo end-point di PCR che permette la quantificazione assoluta diretta degli acidi nucleici all'interno di un campione. Il metodo possiede particolari vantaggi, tra cui una variabilità in diminuzione, una maggiore riproducibilità quotidiana e una sensibilità superiore11,12. Inoltre, dovuto il partizionamento del campione in analisi di endpoint e circa 20.000 singole reazioni, dPCR è più robusto per sostanze interferenti nella PCR rispetto al quantitativo di RT-PCR16. Queste qualità in dPCR lo rendono un'alternativa attraente per RT-PCR quantitativa come strumento diagnostico per la quantificazione. Come miRNA di circolazione sono spesso presenti alle concentrazioni basse nel siero, finora, gli scienziati si sono sfidati per quantificare in modo appropriato i miRNA da PCR9. D'altra parte, dPCR è in grado di quantificare in modo appropriato anche un'espressione di miRNA bassa nel siero, attenuare i problemi osservati nel conteggio basso miRNA quantificazione17. La capacità di dPCR di dare direttamente i conteggi / µ l, anche in un'espressione di miRNA molto basso, così rende un attraente strumento diagnostico per la comunità di ricerca cardiovascolare negli studi di biomarcatore di miRNA. Come la concentrazione data in conteggi / µ l può essere moltiplicata per il fattore di diluizione utilizzato nell'estrazione, reazione di RT e dPCR, è possibile ottenere il numero esatto di copie in 1 µ l di siero. Il flusso di lavoro qui presentato può essere eseguita in fino a 96 campioni su un piatto, quindi fornire uno strumento per grandi studi cardiovascolari. Anche se dPCR presenta parecchi vantaggi sopra RT-PCR quantitativa, non è ancora ordinariamente applicato per la quantificazione dei miRNA nelle prove cardiovascolari. Le procedure di normalizzazione di dati ulteriori, standardizzati sono carenti.
In questo approccio, dimostriamo che dPCR, combinato con sonde fluorescenti idrolisi, consente la quantificazione assoluta diretta specifica del circolante Mirna collegati alla malattia cardiovascolare. Con questa relazione, abbiamo mirato a dimostrare un protocollo ottimizzato per rilevazione di miRNA via dPCR sia confermare i vantaggi di dPCR rispetto RT-PCR quantitativa.
Abbiamo confermato i reperti di dPCR buona linearità e i limiti bassi della rilevazione e quantificazione di dPCR per quantificare i miRNA. Da chiodare il campione con un oligonucleotide sintetico, la normalizzazione del campione è possibile, regolando per efficienza di estrazione e campione a campione variazione.
In conclusione, PCR digitale, come esibisce superiorità sia di competenza tecnica e di potenziale diagnostico, è il miglior metodo corrente per la quantificazione di miRNA e possono essere ulteriormente utilizzati per studi di biomarcatore grande multi-centro cardiovascolare miRNA.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Gli autori non hanno nessun ringraziamenti.
Materiali
| Name | Company | Catalog Number | Comments |
| RNA-Extraction | |||
| miRNeasy Serum/Plasma Kit (50) | Qiagen-Sample & Assay Technologies, Hilden, Deutschland | 217184 | Kit for microRNA extraction. Kit contains commercial buffer RWT (called number one in the manuscript) and RPE (called number two in manuscript). |
| miRNeasy Serum/Plasma Spike-in-Control; Syn-cel-miR-39 miRNA; 10pmol | Qiagen-Sample & Assay Technologies, Hilden, Deutschland | 219610 | Spike-in for normalisation , Sequence: 5'-UCACCGGGUGUAAAUCAGCUUG-3' |
| Reverse Transcription | |||
| TaqMan MicroRNA Reverse Transcription Kit (1000 Reactions) | Applied Biosystems, Inc., Foster City, CA, USA | 4366597 | Kit for microRNA reverse transcription |
| TaqMan MicroRNA Assays M | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assays used in reverse transcription |
| hsa-miR-499 (750 RT/750 PCR rxns) | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assay Number 001352 |
| cel-miR-39 (750 RT/750 PCR rxns) | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assay Number 000200 |
| PCR Plate, 96-well, segmented, semi-skirted | Thermo Fisher Scientific, Waltham, MA, USA | AB0900 | 96 well plate for reverse transcription |
| Microseal ‘B’ seal Seals | Bio-Rad Laboratories, Inc., Hercules, CA, USA | MSB1001 | Foil to ensure proper storage |
| C1000 Touch Thermal Cycler | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1851196 | Cycler used for reverse transcription |
| Droplet Digital PCR | |||
| 100 nmole RNA oligo hsa-miR-499-5p | Integrated DNA Technologies | Custom | Sequence: 5'-phos-UUAAGACUUGCAGUGAUGUUU-3' |
| ddPCR Supermix for Probes (No dUTP) | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863024 | Supermix used in droplet generation |
| TaqMan MicroRNA Assays | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assays used in digital PCR (fluorescent hydrolysis probe) |
| hsa-miR-499 (750 RT/750 PCR rxns) | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assay Number 001352, commercial primers |
| cel-miR-39 (750 RT/750 PCR rxns) | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assay Number 000200, commercial primers |
| DG8 Cartridges and Gaskets | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1864007 | Cartridge takes up to 8 samples for droplet generation |
| DG8 Cartridge Holder | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863051 | Holds cartridges in droplet generation |
| Droplet Generation Oil for Probes | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863005 | Oil used in droplet generation |
| ddPCR 96-Well Plates | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 12001925 | 96 well plate for ddPCR |
| PCR Plate Heat Seal, foil, pierceable | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1814040 | Pierceable foil, compatible with droplet reader |
| ddPCR Droplet Reader Oil | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863004 | Oil used in droplet reading |
| QX100 or QX200 Droplet Generator | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863002 | Droplet Generator, generates the droplets from sample/oil emulsion |
| PX1 PCR Plate Sealer | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1814000 | Seals the plate before PCR |
| C1000 Touch Thermal Cycler | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1851196 | Cycler used for ddPCR |
| QX100 or QX200 Droplet Reader | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863003 | Reads PCR-positive and PCR-negative droplets with an optical detector |
| ddPCR Buffer Control for Probes | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863052 | Blank control and to fill up the remaining wells of 8-well cassette |
| Software | |||
| QuantaSof Software | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1864011 | Program for droplet reading |
| Prism Windows 5 | GraphPad Software Inc., La Jolla, CA, USA | Program for statistical analysis |
Riferimenti
- Schulte, C., Zeller, T. microRNA-based diagnostics and therapy in cardiovascular disease - summing up the facts. Cardiovascular Diagnosis and Therapy. 5, 17-36 (2015).
- Sun, T., et al. The role of microRNAs in myocardial infarction: from molecular mechanism to clinical application. International Journal of Molecular Sciences. 18, (2017).
- Dimmeler, S., Zeiher, A. M. Circulating microRNAs: novel biomarkers for cardiovascular diseases. European Heart Journal. 31, 2705-2707 (2010).
- Creemers, E. E., Tijsen, A. J., Pinto, Y. M. Circulating microRNAs: novel biomarkers and extracellular communicators in cardiovascular disease. Circulation Research. 110, 483-495 (2012).
- Oerlemans, M. I., et al. Early assessment of acute coronary syndromes in the emergency department: the potential diagnostic value of circulating microRNAs. EMBO Molecular Medicine. 4, 1176-1185 (2012).
- Olivieri, F., et al. Diagnostic potential of circulating miR-499-5p in elderly patients with acute non ST-elevation myocardial infarction. Internation Journal of Cardiology. 167, 531-536 (2013).
- Navickas, R., et al. Identifying circulating microRNAs as biomarkers of cardiovascular disease: a systematic review. Cardiovascular Research. 111, 322-337 (2016).
- Devaux, Y., et al. Diagnostic and prognostic value of circulating microRNAs in patients with acute chest pain. Journal of Internal Medicine. 277, 260-271 (2015).
- Schwarzenbach, H., da Silva, A. M., Calin, G., Pantel, K. Data normalization strategies for microRNA quantification. Clinical Chemistry. 61, 1333-1342 (2015).
- Hindson, B. J., et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number. Analytical Chemistry. 83, 8604-8610 (2011).
- Hindson, C. M., et al. Absolute quantification by droplet digital PCR versus analog real-time PCR. Nature Methods. 10, 1003-1005 (2013).
- Robinson, S., et al. Droplet digital PCR as a novel detection method for quantifying microRNAs in acute myocardial infarction. Internation Journal of Cardiology. 257, 247-254 (2018).
- Mitchell, P. S., et al. Circulating microRNAs as stable blood-based markers for cancer detection. Proceedings of the National Academy of Sciences of the United States of America. , 10513-10518 (2008).
- D'Alessandra, Y., et al. Circulating microRNAs are new and sensitive biomarkers of myocardial infarction. European Heart Journal. 31, 2765-2773 (2010).
- Forootan, A., et al. Methods to determine limit of detection and limit of quantification in quantitative real-time PCR (qPCR). Biomolecular Detection and Quantification. 12, 1-6 (2017).
- Dingle, T. C., Sedlak, R. H., Cook, L., Jerome, K. R. Tolerance of droplet-digital PCR vs real-time quantitative PCR to inhibitory substances. Clinical Chemistry. 59, 1670-1672 (2013).
- Taylor, S. C., Laperriere, G., Germain, H. Droplet digital PCR versus qPCR for gene expression analysis with low abundant targets: from variable nonsense to publication quality data. Scientific Reports. 7, 2409(2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon