Method Article
Digitale PCR zur Quantifizierung der zirkulierenden MicroRNAs in akuten Myokardinfarkt und kardiovaskulären Erkrankungen
In diesem Artikel
Zusammenfassung
Zirkulierenden MicroRNAs haben Versprechen als Biomarker für kardiovaskuläre Erkrankungen und akute Myokardinfarkte gezeigt. In dieser Studie beschreiben wir ein Protokoll für die MiRNA-Extraktion, reverse Transkription und digitale PCR für die absolute Quantifizierung von MiRNAs im Serum von Patienten mit kardiovaskulären Erkrankungen.
Zusammenfassung
Zirkulierende Serum Mikro-RNAs (MiRNAs) haben gezeigt, dass Versprechen als Biomarker für die Herz-Kreislauf-Krankheiten und akutem Myokardinfarkt (AMI), wird von den Herz-Kreislauf-Zellen in den Blutkreislauf freigesetzt. Zirkulierenden MiRNAs sind sehr stabil und quantifiziert werden kann. Quantitative Ausdruck der spezifischen MiRNAs kann mit der Pathologie und einige MiRNAs zeigen hohe Gewebe und Krankheit Spezifität verknüpft werden. Neue Biomarker für Herz-Kreislauf-Krankheiten zu finden ist von Bedeutung für die medizinische Forschung. Vor kurzem wurde digital Polymerase-Kettenreaktion (dPCR) erfunden. dPCR, kombiniert mit fluoreszierenden Hydrolyse Sonden ermöglicht eine spezifische direkte absolute Quantifizierung. dPCR stellt überlegene technische Eigenschaften, einschließlich eine geringe Variabilität, hohe Linearität und hohe Empfindlichkeit im Vergleich zu den quantitativen Polymerase-Kettenreaktion (qPCR). Somit ist dPCR eine genaue und reproduzierbare Methode zur Quantifizierung der MiRNAs, speziell für den Einsatz in großen multizentrischen klinischen Herz-Kreislauf-direkt. In dieser Publikation beschreiben wir, wie effektiv digitale PCR durchzuführen, um die absolute Kopienzahl in Serumproben zu beurteilen.
Einleitung
Zirkulierenden MiRNAs wurden als vielversprechender Marker für eine Reihe von Krankheiten, einschließlich Herz-Kreislauf-Krankheit1identifiziert. Die MiRNAs sind klein, nicht-kodierenden einsträngige RNA-Moleküle (ca. 22 Nukleotide lang) engagieren sich in der Posttranskriptionale Verordnung über die Änderung der Boten-RNA Übersetzung und beeinflussende Gene Expression2, und in den Blutkreislauf in physiologischen und pathologischen Zuständen freigegeben werden. Quantitative Ausdruck der spezifischen MiRNAs der Pathologie zugeordnet werden kann, und einige MiRNAs zeigen hohe Gewebe und Krankheit Spezifität1. Im Herz-Kreislauf-Krankheiten sind MiRNAs attraktive Kandidaten geworden, da neue Biomarker weil sie bemerkenswert stabil im Serum sind und leicht können mit Hilfe der PCR Methode3quantifiziert werden. Der potenzielle Wert der MiRNAs als Biomarker für Myokardinfarkt wurde in kleinen Studien ausgewertet, aber eine Validierung in großen Kohorten fehlt2. Zum Beispiel MiR-499 findet man hoch in den Herzmuskel zum Ausdruck gebracht, und es hat gezeigt, dass ein AMI4,5,6deutlich erhöht werden. Darüber hinaus regelt es programmierte Zelltod (Apoptose) und die Differenzierung von Kardiomyozyten und engagiert sich daher in mehrere Mechanismen, die nach einem AMI-7. Abgesehen von einigen kleinen Studien Berichterstattung eine Überlegenheit und inkrementellen Wert von MiRNAs für die Diagnose von AMI Überlegenheit oder Gleichheit zu hoher Empfindlichkeit kardiale Troponins nicht noch in groß angelegten Studien2,5 nachweislich ,6,8. Weitere prospektiven Studien in großen Kohorten sind daher erforderlich, um die mögliche diagnostische Wertigkeit von MiRNAs zu beurteilen. Darüber hinaus Methoden zur Quantifizierung der MiRNA optimiert werden müssen und standardisierte Verwendung vergleichbarer Protokolle9. Standardisierte Tests möglicherweise inkonsistente Ergebnisse reduzieren und können helfen MiRNAs, potenzielle Biomarker für die routinemäßige klinische Anwendung werden als Biomarker müssen reproduzierbar Sicherstellung ihrer klinischen Anwendbarkeit quantifiziert werden.
Vor kurzem, hat dPCR als eine Endpunktanalyse eingeführt. Es Partitionen die Probe in etwa 20.000 individuelle Reaktionen10. Das dPCR System nutzt dann eine mathematische Poisson statistische fluoreszierende Signale (positive und negative Reaktionen), wodurch eine absolute Quantifizierung ohne eine Standardkurve10. Bei der Kombination von dPCR mit fluoreszierenden Hydrolyse Sonden wird die hochspezifische direkte absolute Quantifizierung von MiRNAs ermöglicht. Digitalen Polymerase-Kettenreaktion hat gezeigt, dass überlegene technische Eigenschaften (einschließlich eine verminderte Variabilität, einer erhöhten täglichen Reproduzierbarkeit, ein hohes Maß an Linearität und eine hohe Empfindlichkeit) zur Quantifizierung der MiRNA-Ebenen in weisen die Zirkulation im Vergleich zu quantitative Echtzeit-PCR-10,11. Diese überlegene technische Qualitäten könnte dazu beitragen, um aktuelle Beschränkungen bei der Verwendung von zirkulierenden MiRNAs als Biomarker zu mildern und führen zur Gründung der MiRNAs als Biomarker in großen multizentrischen klinischen Herz-Kreislauf- sowie eine diagnostische Methode in allgemeine. In einer früheren Studie haben wir vor kurzem angewendet dPCR für die absolute Quantifizierung der zirkulierenden MiRNAs bei Patienten mit einem AMI und konnten überlegene diagnostische Potenzial im Vergleich zu der Quantifizierung von MiRNAs qPCR12zeigen.
In dieser Publikation wollen wir zeigen, dass mit dPCR ist eine genaue und reproduzierbare Methode zur Quantifizierung der zirkulierenden Herz-Kreislauf-MiRNAs direkt. Die absolute Quantifizierung der MiRNA-Niveaus im Serum, mit digitalen PCR zeigt Potenzial für den Einsatz in großen multizentrischen Herz-Kreislauf-klinische Studien. In dieser Publikation beschreiben wir ausführlich, wie Sie digitale PCR effizient zu erfüllen und um die absolute MiRNA Kopienzahl im Serum zu erkennen.
Protokoll
1. Gewinnung von MiRNA aus Plasma/Serum
Hinweis: Um MiRNAs angemessen zu quantifizieren, die richtige MicroRNA-Isolierung aus dem Plasma/Serum ist ein entscheidender Schritt. Eine wichtige Sache zu beachten, vor allem, weil unterschiedliche Protokolle vorhanden sind, ist zur Einhaltung des gleichen Workflows bei der Verarbeitung der Proben. In diesem Protokoll wird MiRNA aus 50 µL Serum gewonnen. Verwenden Sie nicht mehr als 200 µL als dieser Grenzwert die korrekte Extraktion.
- Bereiten Sie Serum oder tauen Sie gefrorene Proben auf.
- 50 µL Serum 250 µL (5 Bände) kommerzielle Lyse-Reagenz hinzufügen. Unterstützen Sie die Lyse durch aufschütteln. Legen Sie den Schlauch mit der lysate auf der Bank bei Raumtemperatur für 5 min.
- Spike lysate mit 3,5 µL Spike-Steuerelement (1.6 x 107 Kopien/µL) und mischen sie durch aufschütteln.
- Fügen Sie 50 µL Chloroform (d.h. den gleichen Betrag, der Ausgangspunkt Serumprobe) für die Phasentrennung. Schütteln Sie das Rohr für 15 S. Ort das Rohr auf der Bank bei Raumtemperatur für 2 – 3 min. kräftig.
- Zentrifugieren Sie für die Phasentrennung das Rohr auf 12.000 x g bei 4 ° C für 15 Minuten.
Hinweis: Die Proben werden nun in eine obere, wässrige Phase mit der RNA, eine weiße Interphase und eine niedrigere, Rosa organische Phase getrennt. - Übertragung der oberen wässrigen Phase (100 µL), enthält die RNA in einen neuen Schlauch. Übertragen Sie weiße Interphase Material nicht, da dies die Anzahl der richtigen RNA verfälscht.
- Fügen Sie 150 µL (d.h.1,5 Mal die Menge der übertragenen Probe) von 100 % Ethanol. Mischen Sie die Materialien durch pipettieren sie rauf und runter. Zentrifugieren sie nicht und weitermachen Sie schnell mit dem nächsten Schritt.
- Pipette 250 µL der Probe auf eine kommerzielle Spin-Spalte in einer 2 mL-Kollektion-Röhre. Schließen Sie den Deckel. Zentrifugieren die Spalte > 8.000 x g bei Raumtemperatur 15 S. verwerfen der durchströmten.
- Die Spin-Spalte 700 µL kommerzielle waschen-Puffer-Nummer 1 hinzufügen. Schließen Sie den Deckel und Zentrifugieren die Spalte > 8.000 x g bei Raumtemperatur 15 S. verwerfen der Durchlauf-waschen-Puffer.
- Die Spin-Spalte 500 µL kommerzielle waschen-Puffer-Nummer 2 hinzufügen. Schließen Sie den Deckel und Zentrifugieren die Spalte > 8, 000 x g bei Raumtemperatur 15 S. verwerfen der Durchlauf-waschen-Puffer.
- Pipette 500 µL Ethanol 80 % auf die Spin-Spalte. Schließen Sie den Deckel, Zentrifugieren die Spalte > 8, 000 X g bei Raumtemperatur für 2 min. verwerfen das Sammelrohr mit dem Durchfluss.
- Übertragen Sie die Spin-Spalte auf eine neue 2 mL Sammelrohr. Zentrifugieren Sie die Spalte auf Hochtouren mit geöffneten Deckel um die Membran zu trocknen. Das sammelröhrchen mit Flow-through zu verwerfen.
- Übertragen Sie die Spin-Spalte in eine neue 1,5 mL sammelröhrchen. Eluieren Sie RNA indem 30 µL RNase-freies Wasser direkt auf der Mitte der Membran. Schließen Sie den Deckel. Zentrifugieren Sie die Spalte auf Hochtouren für 1 min.
- Speichern der RNS bei-70 ° C.
Hinweis: Die Lagerung von gereinigten RNA in RNase-freies Wasser zwischen-70 ° C bis-20 ° C garantiert kein Abbau der RNA für 1 Jahr. Nach Protokoll des Herstellers ist der Mindestbetrag der RNase-freies Wasser zum Verdünnen der RNS 10 µL. mit weniger als 10 µL können nur unzureichend Hydrat die Membran und reduzieren die Gesamtausbeute RNA.
(2) reverse Transkription
Hinweis: Ergänzende DNA (cDNA) wurde unter Verwendung des folgenden 15 µL reverse Transkription (RT) Protokolls (gesamte Reaktionsvolumen) synthetisiert.
- Tauen Sie das RT-Kit auf dem Eis. Halten Sie die Enzyme in den Gefrierschrank so lange wie möglich zu verhindern, dass sie beeinträchtigt und drehen sie Sie vor dem Gebrauch. Tauen Sie RT Primer auf Eis auf und drehen sie Sie vor dem Gebrauch.
- Bereiten Sie den master-Mix, wie in Tabelle 1beschrieben.
- Kombinieren Sie 10 µL des master-Mix und 5 µL der extrahierten RNA pro Bohrloch in einer 96-Well-Platte.
- Zentrifugieren Sie die well-Platte bei 2.000 x g bei 4 ° C für 2 min.
- Verwenden Sie das thermischen Zyklus-Protokoll für die reverse Transkription in Tabelle 2 beschrieben.
Hinweis: Wie unten beschrieben, cDNA kann direkt verarbeitet werden in der digitalen PCR-Maschine (siehe Tabelle der Materialien); Alternativ kann es bei-20 ° c gelagert werden
(3) Tröpfchen Generation und digitale PCR
Hinweis: Digitale PCR wurde durchgeführt unter Verwendung des folgenden 40 µL-Protokolls.
- Tauen Sie den kommerziellen dPCR Mix, die vorbereiteten cDNA und die PCR-Primer auf Eis. Zentrifugieren sie kurz vor der Verwendung.
- Die master-Mix vorzubereiten, wie in beschrieben Tabelle 3.
- 1,33 μl/Well des Messguts RT hinzufügen und kurz Zentrifugieren.
- Pipette 20 μl der Probe in jede Vertiefung der Kassette 8-Brunnen (mittlere Reihe).
Hinweis: Wenn die 8-Brunnen-Kassette nicht vollständig gefüllt ist, füllen Sie die restlichen Brunnen mit nicht-Template-Steuerelemente (NTCs), die cDNA (d. h., das RT-Produkt mit Wasser anstelle von extrahierten RNA hergestellt) weglassen oder dPCR kommerziellen Puffer. Der NTC dient als eine Kontaminationskontrolle. Wasser darf nicht in den verbleibenden Brunnen verwendet werden, weil die die Qualität der Tropfenbildung beeinträchtigen wird. - Pipette 70 μL des tröpfchens Generation Öl für die Sonden in den Ölquellen (untere Zeile) der 8-Brunnen-Kassette.
- Legen Sie eine Dichtung auf die 8-Brunnen-Kassette und legen Sie die Kassette in die tropfengenerators. Schließen Sie die tropfengenerators. Warten Sie, bis die 3 leuchtet durchgehend grün sind.
Hinweis: Tröpfchen sind nun generiert. - Vorsichtig und langsam transfer 40 μL der Tröpfchen gebildet Probe (obere Zeile) in separaten Vertiefungen einer 96-Well-Platte pipettieren.
- Nach Abschluss der Tropfenbildung an den Proben, versiegeln die PCR-Platte mit einer durchstechbare dPCR Folie bei 180 ° C für 4 s.
- Legen Sie die Folie versiegelt PCR-Platte in der Cycler und Zyklus, wie beschrieben in Tabelle 4, mit einer Rampe-Rate von 2,5 ° C/s.
(4) Tropfen lesen und analysieren
- Übertragen Sie die Platte aus der Thermocycler auf der Basis der Kennzeichenhalter. Ziehen Sie die PCR-Platte, indem man oben auf den Kennzeichenhalter auf die PCR-Platte.
- Starten Sie die kommerzielle dPCR Software.
- Geben Sie die probennamen, den Namen des Experiments und das Ziel in der Einrichtung. Wählen Sie die absolute Quantifizierung.
- Starten Sie das Droplet lesen durch Klicken auf Ausführen. Wählen Sie FAM/HEX oder FAM/VIC als Erkennung Chemie, bei der Arbeit mit Hydrolyse-Sonden.
Hinweis: Die kommerzielle dPCR Software wird dann automatisch-die Daten analysieren. - Überprüfen Sie die Tröpfchen-Nummern in der Ergebnistabelle um eine richtige Tröpfchen-Generation zu gewährleisten.
- Wählen Sie alle Brunnen mit der gleichen quantifizierte MiRNA und klicken Sie auf analysieren. Verwenden Sie den 2D Blot positivere Tröpfchen markieren und manuell korrigieren Sie die Auto-analysiert Daten (Abbildung 1).
5. korrekte Berechnungen auf die Probe
- Normalisieren Sie die Probe die Probe mit der Normalisierungsfaktor (NF) multipliziert.
NF = Medianalle Proben[C. Elegans MiR-39 Messungen] / [C. Elegans MiR-39]gegebene Probe - Berechnen Sie die endgültige MiRNA-Serum-Konzentration, bereinigt um Verdünnungsfaktoren (DF).
[MiRNA] Final = [MiRNA]Rohwert x DFRT x DFdPCR x DFEx
Wo:
[MiRNA] Rohwert = die MiRNA quantifiziert durch kommerzielle dPCR Software;
DFRT = Verdünnungsfaktor der Vorlage in reverse Transkription;
DFdPCR = Verdünnungsfaktor der Vorlage im dPCR;
DF-Ex = Verdünnungsfaktor bei der Extraktion.
(6) synthetisches Oligonukleotid Verdünnungsreihe
- Lyophilisierter synthetisches Oligonukleotid auf Eis Auftauen. Zentrifugieren Sie es kurz.
- Verdünnen Sie die lyophilisierten synthetisches Oligonukleotid in Nuklease-freies Wasser, eine Endkonzentration von 10 Pmol / µL. kurz Zentrifugieren.
- Verdünnen Sie es in Nuklease-freies Wasser, wie in Tabelle 5beschrieben. Zentrifugieren Sie die Verdünnung kurz zwischen jedem Verdünnungsschritt bei 8.000 x g für 10 s.
- Fahren Sie mit der reversen Transkription wie in Schritt 2 beschrieben und analysieren Sie die Proben mit digitalen PCR zu, wie in Schritt 3 und Schritt 4 beschrieben.
Ergebnisse
Digitale PCR kombiniert mit fluoreszierenden Hydrolyse Sonden ermöglicht Forschern direkt die absoluten Beträge der spezifischen MiRNAs in Kopien/µL zu quantifizieren. Wie das Beispiel in dPCR in etwa 20.000 einzelnen PCR-Reaktionen, partitioniert ist dPCR erfordert keine technischen repliziert10. Das dPCR System nutzt eine mathematische Poisson statistische Analyse der fluoreszierenden Signale (die sich zwischen positiven und negativen Reaktionen) eine absolute Quantifizierung ermöglichen, ohne die Notwendigkeit für einen Standard Kurve10. Um die Konzentrationen korrekt zu berechnen, benötigt man eine akzeptable Tröpfchen Graf (> 15.000). Keine Vorlage Kontrollen sichern den Ausschluss von erheblichen unspezifische Verstärkung. Alle Proben, die in die Analyse einbezogen werden mit der gleichen Lautstärke, Methode und PCR-Protokoll, um die Gültigkeit der erzielten Ergebnisse zu stärken verarbeitet. Die ermittelten Konzentrationen im dPCR werden normalisiert, mit einem Median Normalisierung Verfahren über alle Proben analysiert mit dem synthetischen versetzt-in C. Elegans MiR-3913. Normalisierung der Daten auf die gemessene Höhe der Pfennigabsatz in C. Elegans MiR-39 korrigiert für die Probe zu Probe Variation der RNA-Extraktion-Effizienz und dient als eine PCR Reaktionskontrolle, die Gültigkeit der Ergebnisse weitere hinzufügen. Es gibt keine etablierte Goldstandard für die Normalisierung der Serum-MiRNAs; jedoch exogene Kontrollen wie die versetzt-in C. Elegans MiR-39 sind eine elegante Lösung für Normalisierung Verfahren wie endogene MiRNAs oft in verschiedenen Krankheit Staaten9verändert werden.
Die analysierten Serumproben wurden vor eine perkutane Koronarintervention (PCI), 8 h nach einer PCI und 16 h nach einer PCI von Patienten mit einem akuten ST-Segment Elevation Myokardinfarkt (STEMI) erworben. STEMI-Patienten weisen schwere Ischämie und somit qualifizieren sich für die Bewertung neuer Biomarker der Ischämie. Um die Kinetik der MiR-499-Version und die mögliche Verwendung von MiR-499 als Biomarker für Ischämie bei Herz-Kreislauf-Krankheit zu bewerten, wurde MiR-499 mit dPCR über alle Patienten für die drei verschiedenen Zeit Punkte14analysiert.
Abbildung 1 zeigt die Auswahl der positiven Tröpfchen geben die endgültige MiRNA-Konzentration von der Software berechnet. Der Anteil von positiven Ergebnissen in einer Probe bestimmt die Konzentration von Kopien/µL von der kommerziellen dPCR Software berechnet. Die Ergebnisse können auch in einem 2D Grundstück visualisiert werden und die positiven Aspekte können manuell mit Kreisen markiert werden. Konzentrationen werden nur berücksichtigt, wenn sie über die unspezifische Verstärkung der NTCs sind.
Abbildung 2 zeigt die Linearität und die Güte der Anpassung von einer synthetischen MiRNA Oligonukleotid hat-MiR-499-5 p-Verdünnungsreihe in Duplikate. Ein 8-stufiges Verdünnungsreihen wurde von 2.500 Kopien/µL nach 0 Kopien/µL durchgeführt. Die berechnete erwartete Kopien/µL übernehmen wie in Tabelle 5 beschrieben 100 % RT und digitale PCR-Effizienz. In diesem Setup digitale PCR zeigt ein hohes Maß an Linearität (R2 = 0,99). Der Nachweisgrenze (LoD = 0,12) und die Grenze der Quantifizierung (LoQ = 0,23) werden auch dargestellt in Abbildung 2zeigt, dass sogar ein geringe MiRNA Ausdruck erfolgreich erkannt werden kann. So kann dPCR verwendet werden, auch niedrige Serumspiegel von zirkulierenden MiRNAs zu quantifizieren. Der Nachweisgrenze (LoD) und die Grenze der Quantifizierung (LoQ) werden berechnet nach dem Ansatz von Forootan Et al. 15 und sind definiert als LoD = LoB + 1.645 X σniedrigKonzentrationProbe, wo die Grenze des Rohlings (LoB) sich als LoB errechnet = mittlereleer + 1.645 X σleer. Das LoQ ist dann geschätzte laufende replizieren standard15Kurven. Um die reproduzierbare Quantifizierung von MiRNAs zu gewährleisten, werden nur MiRNA-Konzentrationen, die über die Detailgenauigkeit und die LoQ liegen als einen gültigen exakten Wert betrachtet.
Abbildung 3 zeigt repräsentative Ergebnisse von MiR-499 Ebenen von Patienten mit einem ST-Elevation Myokardinfarkt (STEMI), n = 16 zu jedem Zeitpunkt. Wurden Proben genommen, bevor eine perkutane Intervention (PCI) (t = 0), 8 h nach einer PCI (t = 8), und 16 h nach einer PCI (t = 16), und die MiR-499-Ebenen waren im Vergleich zu MiR-499 Ebenen von Patienten mit stabiler koronarer Herzkrankheit (KHK, n = 20). Die wichtigsten Patientenmerkmalen finden Sie in Tabelle 6. Nach der ersten Veröffentlichung von MiR-499 in das Serum in den ersten 8 Stunden nach einem Herzinfarkt senken MiR-499 nach 16 h wieder. Ein Trend in der Erhöhung kann schon zu Jahresbeginn die STEMI (t = 0), und ein deutliches Plus von MiR-499-Ebenen im Vergleich zu Patienten mit CAD kann gesehen 8 h nach dem STEMI (t = 8, p < 0,01) beim Vergleich von MiR-499-Ebenen, MiR-499 Ebenen stabil CAD-Patienten. Der Empfänger Betrieb Kurven (ROC) zeigen das mögliche MiR-499-Dienstprogramm als neuartige Biomarker Unterscheidung zwischen Patienten mit CAD und Patienten mit einem STEMI [die Fläche unter der Kurve (AUC) = 0,62 für Proben, die zuvor eine perkutane Intervention (PCI), AUC = 0,75 für Proben, die 8 h nach einer PCI und AUC = 0,78 für Proben 16 h nach einer PCI gegenüber Serumspiegel von MiR-499 bei Patienten mit CAD].

Abbildung 1: Auswahl der positiven Tröpfchen in einer Verdünnungsreihe durchgeführt in Duplikate. (A) die Schwelle ist über die negativen Tröpfchen manuell einstellen. (B) die Ergebnisse werden in ein 2D Schandfleck, das positive ausgewählten Kreisen visualisiert. Bitte klicken Sie hier für eine größere Version dieser Figur.
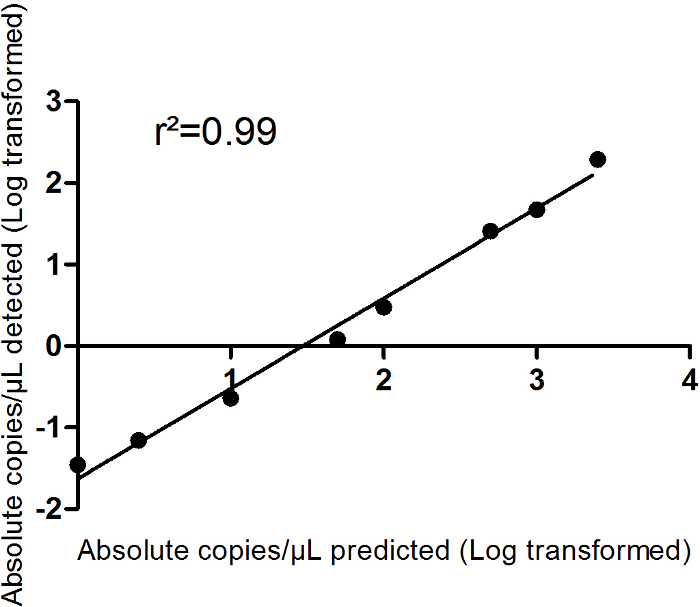
Abbildung 2: synthetische MiR-499 Oligonukleotid Verdünnungsreihe. (A) die Messungen wurden in zweifacher Ausfertigung. Die Daten werden Log umgewandelt als absolute Kopien pro µL. lineare Regressionen und Güte der Anpassung dargestellt (R2-Wert) werden angezeigt. Die Daten sind als Mittelwert ± SEM (B) dieses Panel der Nachweisgrenze (LoD) und die Grenze der Quantifizierung (LoQ zeigt) dargestellt. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3: Quantifizierung der zirkulierenden Serum MiR-499-5 p bei Patienten mit Myokardinfarkt ST-Elevation (n = 16) vor 8 h nach PCI perkutane Koronarintervention (PCI) und 16 h nach PCI, im Vergleich zu Patienten mit stabiler koronarer Krankheit (n = 20). (A) die zirkulierende Serum MiR-499-5 p wird dargestellt als Mittelwert mit den Standardfehler des Mittelwerts mit der entsprechenden p-Wert berechnet, indem einfache ANOVA gefolgt von einer Bonferroni Post Hoc Test (p < 0,05 galt als "statistisch signifikant). (B) ein Empfänger Betrieb Kennlinie (ROC) Analyse wird auf die Daten aus Schalttafel Adurchgeführt. Die ROC-Analyse zeigt die Sensitivität und Spezifität der Biomarker. Darüber hinaus werden der entsprechende Bereich unter der Kurve (AUC) Werte angezeigt. Bitte klicken Sie hier für eine größere Version dieser Figur.
| Probenmenge | ||||
| n = 1 | / sowie [µl] | |||
| Nuklease-freies Wasser | 4.16 | |||
| 10 x Reverse Transkription Puffer | 1.5 | |||
| 100nM dNTP | 0,15 | |||
| RNAse-inhibitor | 0,19 | |||
| spezifischen RT Primer | 3 | |||
| Kommerzielle Reverse Transkriptase Enzym 50 U/ul | 1 | |||
| Master-Mix Gesamt | 10 | |||
Tabelle 1: Reverse Transkription Reagenzien Mix.
| Temperatur | Zeit |
| 16 ° C | 30 Minuten |
| 42 ° C | 30 Minuten |
| 85 ° C | 5 Minuten |
| 4 ° C | ∞ |
Tabelle 2: Radsport Thermik für Reverse Transkription.
| Probenmenge | ||||
| n = 1 | / sowie [µl] | |||
| Nuklease-freies Wasser | 7,67 | |||
| kommerzielle dPCR Mischung | 10 | |||
| 20 x spezifische Hydrolyse Primer/Sonden | 1 | |||
| Master-Mix Gesamt | 18,67 | |||
Tabelle 3: Digitale PCR Reagenz Mix.
| Temperatur | Zeit |
| 95 ° C | 10 min. |
| 94 ° C | 30 Sek. (x40) |
| 60 ° C | 1 min |
| 98 ° C | 10 min. |
| 4 ° C | ∞ |
Tabelle 4: Radfahren Thermik für digitale PCR.
| Rohr | Synthetisches Oligonukleotid MiR-499 (µL) übertragen | Verdünnungsmittel (µL) | MiRNA (Kopien/µL) | Erwarteten Kopien/µL in RT | Erwarteten Kopien/µL in dPCR |
| Original | 6,022 x 1012 | ||||
| 1 | 10 | 990 | 6,022 x 1010 | ||
| 2 | 10 | 990 | 6,022 x 108 | ||
| 3 | 10 | 990 | 6,022 x 106 | ||
| 4 | 18.7 | 981,3 | 1,128 x 105 | 37600 | 2500 |
| 5 | 40 | 60 | 4.511 x 104 | 15040 | 1000 |
| 6 | 50 | 50 | 2.256 x 104 | 7520 | 500 |
| 7 | 40 | 160 | 4.511 x 103 | 1504 | 100 |
| 8 | 50 | 50 | 2.256 x 103 | 752 | 50 |
| 9 | 40 | 160 | 4.511 x 102 | 150,4 | 10 |
| 10 | 50 | 150 | 1,128 x 102 | 37,6 | 2.5 |
| 11 | 0 | 50 | 0 | 0 | 0 |
Tabelle 5: Synthetische Verdünnungsreihen für MiR-499.
| Charakteristisch | Alle | Stabile CAD Patienten (n = 20) | STEMI-Patienten (n = 24) | p-Wert |
| Alter | 64,7 ± 11,9 | 66,7 ± 13.1 | 62,2 ± 9,9 | 0.2810 |
| Männer | 77,8 % | 65 % | 93,8 % | 0.0392 |
| DM | 33,3 % | 40 % | 25 % | 0.3428 |
| HTN | 61,1 % | 60 % | 62,5 % | 0.8785 |
| Dyslipidämie | 38,9 % | 65 % | 6,3 % | 0,0003 |
| Raucher | 47,2 % | 55 % | 37,5 % | 0.3796 |
| Familiengeschichte | 25 % | 35 % | 12,5 % | 0.1213 |
| Übergewicht | 58,3 % | 55 % | 62,5 % | 0.6501 |
| Serum-Kreatinin (mg/dL) | 0,94 (0,83 - 1) | 0,98 (0,82 - 1,01) | 0.93 (0,83 - 1) | 0.7255 |
| CK-Peak (U / ich) | 161 (102-611) | 126 (81-161) | 492 (228-3208) | 0,0004 |
| cTnT Peak (ng/L) | 322,5 (23,8-4533) | 18 (7-25,3) | 2492 (240-5586) | 0,0002 |
| LVEF % | 45 % (40-50 %) | 45 % (32,5 % - 55 %) | 45 % (45-50 %) | 0.9596 |
| Werte werden als ± SD bedeuten präsentiert; Median Wertebereich (25.-75. Perzentile) oder % | ||||
| DM: Diabetes mellitus | ||||
| HTN: Bluthochdruck | ||||
| CK: Kreatinkinase | ||||
| cTnT: kardiale Troponin T | ||||
| LVEF: Linke linksventrikulären Ejektionsfraktion | ||||
Tabelle 6: Main Patientenmerkmalen.
Diskussion
Digitale PCR ist ein relativ neuer Endpunkt-Methode von PCR, die die direkte absolute Quantifizierung von Nukleinsäuren innerhalb einer Probe ermöglicht. Die Methode besitzt bestimmte Vorteile, wie eine verminderte Variabilität, einer erhöhten täglichen Reproduzierbarkeit und eine höhere Sensitivität11,12. Darüber hinaus durch die Partitionierung der Probe in etwa 20.000 einzelne Reaktionen und Endpunkt Analysen dPCR ist robuster, störender Substanzen in der PCR im Vergleich zu quantitativen RT-PCR-16. Diese Qualitäten in dPCR machen es eine attraktive Alternative zur quantitativen RT-PCR als diagnostisches Instrument zur Quantifizierung. Als zirkulierende MiRNAs sind häufig bei niedrigen Serumkonzentrationen, so weit, Wissenschaftler wurden gestellt entsprechend MiRNAs Quantifizierung von PCR-9. Auf der anderen Seite kann dPCR entsprechend auch einen niedrigen MiRNA Ausdruck im Serum, Minderung der Probleme, die in geringer Anzahl MiRNA Quantifizierung17beobachtet quantifizieren. Die Fähigkeit der dPCR direkt den Grafen/µL, sogar in einem sehr niedrigen MiRNA Ausdruck geben macht es somit eine attraktive Diagnose-Tool für die Herz-Kreislauf-Forschungsgemeinschaft in MiRNA Biomarker Studien. Da die Konzentration in Grafen/µL gegeben mit der Verdünnungsfaktor verwendet in Extraktion, RT-Reaktion und dPCR multipliziert werden kann, ist es möglich, die genaue Anzahl der Exemplare in 1 µL Serum zu erreichen. Die hier vorgestellten Workflow kann in bis zu 96 Proben auf einem Teller, daher bietet eine Tool für große Herz-Kreislauf-Studien durchgeführt werden. Obwohl dPCR mehrere Vorteile gegenüber quantitativen RT-PCR aufweist, ist es noch nicht routinemäßig für die Quantifizierung von MiRNAs in Herz-Kreislauf-Studien angewendet. Weitere, standardisierte Daten Normalisierung Verfahren fehlen.
Bei diesem Ansatz zeigen wir, dass dPCR, kombiniert mit fluoreszierenden Hydrolyse Sonden, spezifischen direkten absolute Quantifizierung der zirkulierenden MiRNAs Zusammenhang mit kardiovaskulären Erkrankungen ermöglicht. Mit diesem Bericht wollten wir sowohl ein optimiertes Protokoll für MiRNA-Erkennung über dPCR zeigen und bestätigen die Vorteile der dPCR über quantitative RT-PCR.
Wir bestätigen die gute Linearität dPCR Exponate und den niedrigen Limits der Erkennung und Quantifizierung von dPCR zur Quantifizierung der MiRNAs. Durch die Probe mit ein synthetisches Oligonukleotid Spick, ist die Normalisierung der Probe möglich, bereinigt um absaugeffizienz und Probe zu Probe Variation.
Zusammenfassend, digitale PCR als es Überlegenheit in technische Kenntnisse und diagnostischen Möglichkeiten, Ausstellungen ist die beste aktuelle Methode zur Quantifizierung der MiRNA und weiter verwendet werden, für große multizentrische Herz-Kreislauf-MiRNA Biomarker Studien.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Die Autoren haben keine Bestätigungen.
Materialien
| Name | Company | Catalog Number | Comments |
| RNA-Extraction | |||
| miRNeasy Serum/Plasma Kit (50) | Qiagen-Sample & Assay Technologies, Hilden, Deutschland | 217184 | Kit for microRNA extraction. Kit contains commercial buffer RWT (called number one in the manuscript) and RPE (called number two in manuscript). |
| miRNeasy Serum/Plasma Spike-in-Control; Syn-cel-miR-39 miRNA; 10pmol | Qiagen-Sample & Assay Technologies, Hilden, Deutschland | 219610 | Spike-in for normalisation , Sequence: 5'-UCACCGGGUGUAAAUCAGCUUG-3' |
| Reverse Transcription | |||
| TaqMan MicroRNA Reverse Transcription Kit (1000 Reactions) | Applied Biosystems, Inc., Foster City, CA, USA | 4366597 | Kit for microRNA reverse transcription |
| TaqMan MicroRNA Assays M | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assays used in reverse transcription |
| hsa-miR-499 (750 RT/750 PCR rxns) | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assay Number 001352 |
| cel-miR-39 (750 RT/750 PCR rxns) | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assay Number 000200 |
| PCR Plate, 96-well, segmented, semi-skirted | Thermo Fisher Scientific, Waltham, MA, USA | AB0900 | 96 well plate for reverse transcription |
| Microseal ‘B’ seal Seals | Bio-Rad Laboratories, Inc., Hercules, CA, USA | MSB1001 | Foil to ensure proper storage |
| C1000 Touch Thermal Cycler | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1851196 | Cycler used for reverse transcription |
| Droplet Digital PCR | |||
| 100 nmole RNA oligo hsa-miR-499-5p | Integrated DNA Technologies | Custom | Sequence: 5'-phos-UUAAGACUUGCAGUGAUGUUU-3' |
| ddPCR Supermix for Probes (No dUTP) | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863024 | Supermix used in droplet generation |
| TaqMan MicroRNA Assays | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assays used in digital PCR (fluorescent hydrolysis probe) |
| hsa-miR-499 (750 RT/750 PCR rxns) | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assay Number 001352, commercial primers |
| cel-miR-39 (750 RT/750 PCR rxns) | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assay Number 000200, commercial primers |
| DG8 Cartridges and Gaskets | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1864007 | Cartridge takes up to 8 samples for droplet generation |
| DG8 Cartridge Holder | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863051 | Holds cartridges in droplet generation |
| Droplet Generation Oil for Probes | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863005 | Oil used in droplet generation |
| ddPCR 96-Well Plates | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 12001925 | 96 well plate for ddPCR |
| PCR Plate Heat Seal, foil, pierceable | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1814040 | Pierceable foil, compatible with droplet reader |
| ddPCR Droplet Reader Oil | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863004 | Oil used in droplet reading |
| QX100 or QX200 Droplet Generator | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863002 | Droplet Generator, generates the droplets from sample/oil emulsion |
| PX1 PCR Plate Sealer | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1814000 | Seals the plate before PCR |
| C1000 Touch Thermal Cycler | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1851196 | Cycler used for ddPCR |
| QX100 or QX200 Droplet Reader | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863003 | Reads PCR-positive and PCR-negative droplets with an optical detector |
| ddPCR Buffer Control for Probes | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863052 | Blank control and to fill up the remaining wells of 8-well cassette |
| Software | |||
| QuantaSof Software | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1864011 | Program for droplet reading |
| Prism Windows 5 | GraphPad Software Inc., La Jolla, CA, USA | Program for statistical analysis |
Referenzen
- Schulte, C., Zeller, T. microRNA-based diagnostics and therapy in cardiovascular disease - summing up the facts. Cardiovascular Diagnosis and Therapy. 5, 17-36 (2015).
- Sun, T., et al. The role of microRNAs in myocardial infarction: from molecular mechanism to clinical application. International Journal of Molecular Sciences. 18, (2017).
- Dimmeler, S., Zeiher, A. M. Circulating microRNAs: novel biomarkers for cardiovascular diseases. European Heart Journal. 31, 2705-2707 (2010).
- Creemers, E. E., Tijsen, A. J., Pinto, Y. M. Circulating microRNAs: novel biomarkers and extracellular communicators in cardiovascular disease. Circulation Research. 110, 483-495 (2012).
- Oerlemans, M. I., et al. Early assessment of acute coronary syndromes in the emergency department: the potential diagnostic value of circulating microRNAs. EMBO Molecular Medicine. 4, 1176-1185 (2012).
- Olivieri, F., et al. Diagnostic potential of circulating miR-499-5p in elderly patients with acute non ST-elevation myocardial infarction. Internation Journal of Cardiology. 167, 531-536 (2013).
- Navickas, R., et al. Identifying circulating microRNAs as biomarkers of cardiovascular disease: a systematic review. Cardiovascular Research. 111, 322-337 (2016).
- Devaux, Y., et al. Diagnostic and prognostic value of circulating microRNAs in patients with acute chest pain. Journal of Internal Medicine. 277, 260-271 (2015).
- Schwarzenbach, H., da Silva, A. M., Calin, G., Pantel, K. Data normalization strategies for microRNA quantification. Clinical Chemistry. 61, 1333-1342 (2015).
- Hindson, B. J., et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number. Analytical Chemistry. 83, 8604-8610 (2011).
- Hindson, C. M., et al. Absolute quantification by droplet digital PCR versus analog real-time PCR. Nature Methods. 10, 1003-1005 (2013).
- Robinson, S., et al. Droplet digital PCR as a novel detection method for quantifying microRNAs in acute myocardial infarction. Internation Journal of Cardiology. 257, 247-254 (2018).
- Mitchell, P. S., et al. Circulating microRNAs as stable blood-based markers for cancer detection. Proceedings of the National Academy of Sciences of the United States of America. , 10513-10518 (2008).
- D'Alessandra, Y., et al. Circulating microRNAs are new and sensitive biomarkers of myocardial infarction. European Heart Journal. 31, 2765-2773 (2010).
- Forootan, A., et al. Methods to determine limit of detection and limit of quantification in quantitative real-time PCR (qPCR). Biomolecular Detection and Quantification. 12, 1-6 (2017).
- Dingle, T. C., Sedlak, R. H., Cook, L., Jerome, K. R. Tolerance of droplet-digital PCR vs real-time quantitative PCR to inhibitory substances. Clinical Chemistry. 59, 1670-1672 (2013).
- Taylor, S. C., Laperriere, G., Germain, H. Droplet digital PCR versus qPCR for gene expression analysis with low abundant targets: from variable nonsense to publication quality data. Scientific Reports. 7, 2409(2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten