Method Article
PCR numérique pour quantifier les microARN dans l’infarctus aigu du myocarde et les maladies cardiovasculaires en circulation
Dans cet article
Résumé
Les microARN circulants ont montré prometteur comme biomarqueurs des maladies cardiovasculaires et d’infarctus du myocarde aigus. Dans cette étude, les auteurs décrivent un protocole pour l’extraction de miRNA, transcription inverse et ACP numérique pour la quantification absolue des miARN dans le sérum des patients atteints de maladies cardiovasculaires.
Résumé
Circulants sérum microARN (miARN) ont montré prometteur comme biomarqueurs pour les maladies cardiovasculaires et infarctus aigu du myocarde (IAM), étant libérée des cellules cardiovasculaires dans la circulation. MiARN circulants est très stables et peut être quantifiées. L’expression quantitative des miARN spécifiques peut être liée à la pathologie et certains miARN Voir la haute tissu et la spécificité de la maladie. Trouver de nouveaux biomarqueurs des maladies cardiovasculaires est d’importance pour la recherche médicale. Tout récemment, réaction en chaîne de polymérase numérique (dPCR) a été inventée. dPCR, combiné avec les sondes fluorescentes hydrolyse, permet une quantification absolue directe spécifique. dPCR présente des qualités techniques supérieures, y compris une faible variabilité, une linéarité élevée et une sensibilité élevée par rapport à la réaction en chaîne par polymérase quantitative (qPCR). DPCR est donc une méthode plus précise et reproductible pour quantifier directement les miARN, particulièrement pour l’utilisation lors d’essais cliniques cardiovasculaires de grandes multi-center. Dans cette publication, nous décrivons comment effectuer efficacement numérique PCR afin d’évaluer le nombre de copies absolue dans le sérum.
Introduction
MiARN circulants ont été identifiés comme marqueurs prometteurs pour un certain nombre de maladies, y compris les maladies cardiovasculaires,1. Les miARN est petites, de molécules d’ARN simple brin non codant (environ 22 nucléotides de long) sont impliqués dans la post-transcriptionnelle via la modification de la traduction de l’ARN messager et l’expression de gène influençant2, et sont libérés dans la circulation dans les États physiologiques et pathologiques. L’expression quantitative des miARN spécifique peut être liée à la pathologie, et certains miARN montre tissu haut et spécificité de la maladie1. Dans les maladies cardiovasculaires, miARN est devenus candidats attrayants comme nouveaux biomarqueurs parce qu’ils sont remarquablement stables dans le sérum et peuvent facilement être quantifiées à l’aide de la PCR méthodologie3. La valeur potentielle des miARN comme biomarqueurs pour infarctus du myocarde a été évaluée dans des études de petites envergure, mais une validation dans des cohortes manque2. Par exemple, miR-499 se trouve fortement exprimée dans le muscle myocardique, et il s’est avéré être une augmentation significative chez un AMI4,5,6. En outre, il réglemente programmé la mort cellulaire (apoptose) et la différenciation des cardiomyocytes et participe ainsi à plusieurs mécanismes suivant un AMI7. Mis à part quelques petites études rapports une supériorité et une valeur incrémentielle des miARN pour le diagnostic de l’AMI, la supériorité ou l’égalité à haute sensibilité troponines cardiaques n'a pas encore été prouvée dans des études à grande échelle2,5 ,6,8. Plusieurs études prospectives dans des cohortes sont, par conséquent, nécessaires pour évaluer la valeur diagnostique potentielle des miARN. En outre, méthodes de quantification de miRNA doivent être optimisées et9des protocoles normalisés utilisant comparables. Essais normalisés peuvent réduire des résultats incohérents et peuvent aider les miARN pour devenir des biomarqueurs potentiels pour l’application clinique de routine, comme biomarqueurs doivent être quantifiés de manière reproductible pour assurer leur application clinique.
Récemment, dPCR a été présenté comme une analyse de point final. Il répartit l’échantillon dans environ 20 000 réactions individuelles10. Le système dPCR utilise ensuite une Poisson statistique analyse mathématique des signaux fluorescents (réactions positives et négatives), ce qui permet une quantification absolue sans une courbe standard10. Pour combiner dPCR avec sondes fluorescentes hydrolyse, la quantification absolue directe hautement spécifique des miARN est rendue possible. Réaction en chaîne de polymérase numérique a démontré que présentent des qualités techniques supérieures (y compris une variabilité réduite, une reproductibilité quotidienne accrue, un haut degré de linéarité et une sensibilité élevée) pour quantifier les niveaux de miRNA dans le circulation par rapport à quantitative en temps réel PCR10,11. Ces qualités techniques supérieures pourraient aider à atténuer les limites en vigueur sur l’utilisation des miARN circulant comme biomarqueurs et pourraient mener à l’établissement des miARN comme biomarqueurs dans grands multi-centres les essais cliniques cardiovasculaires et comme une méthode de diagnostic en en général. Dans une étude précédente, nous avons récemment appliqué dPCR pour la quantification absolue des miARN chez les patients avec une AMI en circulation et ont pu démontrer le potentiel de diagnostique supérieure par rapport à la quantification des miARN de qPCR12.
Dans cette publication, nous voulons démontrer que l’utilisation dPCR est une méthode précise et reproductible pour quantifier directement miARN cardiovasculaire en circulation. La quantification absolue de miRNA niveaux dans le sérum par PCR digital, montre potentielle pour l’utilisation lors d’essais cliniques cardiovasculaires de grandes multi-center. Dans cette publication, nous décrivons en détail comment effectuer efficacement PCR numérique et comment détecter le nombre de copies de miRNA absolue dans le sérum.
Protocole
1. extraction de miRNA du Plasma/sérum
Remarque : Afin de quantifier adéquatement les miARN, l’isolement de microARN correcte du plasma/sérum est une étape cruciale. Un essentiel de garder à l’esprit, surtout parce qu’il existe des protocoles différents, est d’adhérer au même flux de travail lors du traitement des échantillons. Dans ce protocole, miRNA est extrait de 50 µL de sérum. Ne pas utiliser plus de 200 µL comme cette limite le processus d’extraction correcte.
- Préparer le sérum ou décongeler les échantillons congelés.
- Ajouter 250 µL (5 volumes) de réactif de lyse commerciale à 50 µL de sérum. Soutenir la lyse au vortex. Placer le tube avec le lysat sur le banc à température ambiante pendant 5 min.
- Doper le lysat 3,5 µl de contrôle spike (1.6 x 107 copies/µL) et les mélanger au vortex.
- Ajouter 50 µL de chloroforme (c'est-à-dire, une quantité égale à l’échantillon de sérum départ) pour la séparation de phase. Agiter vigoureusement le tube pour 15 s. Place le tube sur le banc à température ambiante pendant 2-3 min.
- Pour la séparation de phase, centrifuger le tube à 12 000 x g à 4 ° C pendant 15 minutes.
Remarque : Les échantillons sont maintenant séparés en phase aqueuse supérieure contenant l’ARN, une interphase blanc et une phase organique inférieure, rose. - Transférer la phase aqueuse supérieure (100 µL) contenant l’ARN dans un nouveau tube. Ne transférez pas n’importe quel matériel interphase blanc comme cela falsifie le nombre correct de RNA.
- Ajouter 150 µL (c'est-à-dire1,5 fois le volume de l’échantillon transféré) d’éthanol à 100 %. Les matériaux de la composition de leur pipetage de haut en bas. Ne pas centrifuger à eux et rapidement passer à l’étape suivante.
- Distribuer 250 µL de l’échantillon sur une colonne commerciale dans un tube de prélèvement de 2 mL. Fermer le couvercle. Centrifuger la colonne > 8 000 x g à température ambiante pendant 15 s. jeter le cheminement.
- Ajouter 700 µL de numéro de tampon de lavage commercial 1 dans la colonne. Fermer le couvercle et centrifuger la colonne > 8 000 x g à température ambiante pendant 15 s. jeter le tampon de lavage des trains directs.
- Ajouter 500 µL de numéro de tampon de lavage commercial 2 à la colonne. Fermer le couvercle et centrifuger la colonne > 8, 000 x g à température ambiante pendant 15 s. jeter le tampon de lavage des trains directs.
- Pipeter 500 µL d’éthanol à 80 % pour la colonne. Fermez le couvercle, centrifuger la colonne > 8, 000 x g à température ambiante pendant 2 min. jetez le tube de prélèvement avec le cheminement.
- Transférer la colonne vers un nouveau tube de prélèvement de 2 mL. Centrifuger la colonne à pleine vitesse avec un couvercle ouvert pour sécher la membrane. Jeter le tube de prélèvement avec le cheminement.
- Transférer la colonne de spin dans un nouveau tube de prélèvement de 1,5 mL. Éluer l’ARN en appliquer 30 µL d’eau exempte de RNase directement au centre de la membrane. Fermer le couvercle. Centrifuger la colonne à pleine vitesse pendant 1 min.
- Magasin l’ARN à-70 ° C.
Remarque : Le stockage de l’ARN purifié dans l’eau sans RNase entre-70 ° C à-20 ° C ne garantit aucune dégradation de l’ARN pendant 1 an. Suite de protocole du fabricant, la quantité minimale d’eau exempte de RNase pour diluer l’ARN est de 10 µL. à l’aide de moins de 10 µL peut pas suffisamment hydrater la membrane et réduire le rendement total de RNA.
2. Transcription inverse
Remarque : L’ADN complémentaire (ADNc) a été synthétisé en utilisant le protocole suivant de la transcription inverse (RT) 15 µL (volume de réaction totale).
- Décongeler le kit RT sur la glace. Garder les enzymes dans le congélateur le plus longtemps possible d’empêcher leur dégradants et leur spin down avant utilisation. Décongeler les amorces RT sur la glace et leur spin down avant utilisation.
- Préparer le mélange maître tel que décrit dans le tableau 1.
- Mélanger 10 µL du mélange maître et 5 µL de l’ARN extrait par puits dans une plaque 96 puits.
- Centrifuger la plaque bien à 2 000 x g à 4 ° C pendant 2 min.
- Utiliser le protocole de cycle thermique décrit dans le tableau 2 pour la transcriptase inverse.
Remarque : Comme décrit ci-dessous, cDNA peut-être directement être traité dans la machine de PCR numérique (voir Table des matières) ; Alternativement, il peut être conservé à-20 ° C.
3. droplet Generation et PCR numérique
Remarque : Digital PCR a été réalisée en utilisant le protocole suivant de 40 µL.
- Décongeler le mélange commercial dPCR, l’ADNc préparé et les amorces PCR sur la glace. Centrifuger à eux peu avant son utilisation.
- Préparer le mélange maître comme décrit dans tableau 3.
- Ajouter 1,33 μL/puits du produit RT et il centrifuger brièvement.
- Pipeter 20 μl de l’échantillon dans chaque puits de la cassette de 8 puits (rangée du milieu).
Remarque : Si la cassette de 8 puits n’est pas complètement remplie, remplir les puits restants avec contrôles sans modèle (NTCs) omettent d’ADNc (c'est-à-direle produit de RT fabriqué avec de l’eau au lieu de RNA extraction) ou dPCR tampon commercial. Le CNT est un contrôle de la contamination. Eau ne doit pas être utilisée dans les puits restants, car qui nuira à la qualité de la formation de gouttelettes. - Pipetter 70 μL de l’huile goutte de génération des sondes dans les puits de pétrole (rangée du bas) de la cassette de 8 puits.
- Placer un joint d’étanchéité sur le dessus de la cassette de 8 puits et placez la cassette dans le générateur de gouttelettes. Fermer le générateur de gouttelettes. Attendez que les 3 voyants sont vert fixe.
Remarque : Gouttelettes sont maintenant générés. - Soigneusement et lentement transfert 40 μl de l’échantillon formé de gouttelettes (rangée du haut) dans les puits séparés d’une plaque de 96 puits en pipettant également.
- À l’issue de la formation de gouttelettes sur les échantillons, sceller la plaque PCR avec une feuille de dPCR perçable à 180 ° C pendant 4 s.
- Placer la PCR-plaque aluminium scellées dans le cycleur et il cycle tel que décrit dans le tableau 4, à une vitesse de montée de 2.5 ° C/s.
4. gouttelette lecture et analyse
- Transférez la plaque le thermocycleur jusqu'à la base du support de la plaque. Serrer la plaque PCR en plaçant le haut du support de plaque sur la plaque de la PCR.
- Démarrez le logiciel commercial dPCR.
- Entrez le nom de l’échantillon, le nom de l’expérience et la cible dans la configuration. Sélectionnez la quantification absolue.
- Commencer la gouttelette lecture en cliquant sur exécuter. Sélectionnez FAM/HEX ou FAM/VIC comme chimie détection lorsque vous travaillez avec des sondes d’hydrolyse.
Remarque : Le logiciel commercial dPCR va alors auto-analyser les données. - Vérifier les numéros de gouttelettes dans le tableau de résultats afin d’assurer une génération de gouttelettes correct.
- Sélectionnez tous les puits avec le même miRNA quantifié et cliquez sur ANALYZE. La tache 2D permet de marquer les gouttelettes positives et corriger manuellement les données d’analyse automatique (Figure 1).
5. corriger les calculs sur l’échantillon
- Normaliser l’échantillon en multipliant l’échantillon par le facteur de normalisation (NF).
NF = médiane [c. elegans miR-39 mesures]tous les échantillons/ [c. elegans miR-39]échantillon donné - Calculer la concentration sérique de miRNA final, ajustement pour les facteurs de dilution (DF).
[miRNA] finale = [miRNA]valeur brute x DF,RT , x DFdPCR x DFEx
Où :
[miRNA] valeur brute = la miRNA quantifié par dPCR commercial software ;
DFRT = le facteur de dilution du modèle dans la transcription inverse ;
DFdPCR = le facteur de dilution du modèle dans dPCR ;
DFEx = le facteur de dilution dans l’extraction.
6. synthèse d’oligonucléotide Dilution série
- Dégelez lyophilisée oligonucléotide synthétique sur la glace. Brièvement, centrifuger il.
- Diluer l’oligonucléotide synthétique lyophilisé dans une eau exempte de nucléase à une concentration finale de 10 pmol / µL. brièvement centrifuger il.
- Le diluer dans de l’eau exempte de nucléase tel que décrit dans le tableau 5. Centrifuger la dilution brièvement entre chaque étape de dilution à 8 000 x g pendant 10 s.
- Poursuivez la transcriptase inverse, tel que décrit à l’étape 2 et analyser les échantillons avec PCR numérique tel que décrit dans les étapes 3 et 4.
Résultats
PCR numérique combiné avec les sondes fluorescentes hydrolyse permet aux chercheurs de quantifier directement les quantités absolues des miARN spécifique en copies/µL. Comme l’échantillon en dPCR est partitionnée en environ 20 000 réactions individuelles de PCR, dPCR ne nécessite pas de technique réplique10. Le système dPCR utilise une analyse statistique mathématique de Poisson des signaux fluorescents (différant entre les réactions positives et négatives) qui permet une quantification absolue sans la nécessité d’une norme courbe10. Afin de calculer correctement les concentrations, il faut un nombre acceptable de gouttelettes (> 15 000). Aucun contrôle de modèle n’assurer l’exclusion d’amplification non-spécifique considérable. Tous les échantillons inclus dans l’analyse sont traitées en utilisant le même volume, méthode et protocole ACP à renforcer la validité des résultats obtenus. Les concentrations obtenues dans dPCR sont normalisées en utilisant une procédure de normalisation médian dans l’ensemble de tous les échantillons analysés avec le synthétique enrichi en c. elegans miR-3913. Normaliser les données pour la quantité mesurée de dopés-in c. elegans miR-39 corrige pour l’échantillon à la variation du rendement d’extraction de RNA et agit comme un contrôle de réaction PCR, ajoutant encore à la validité des résultats. Il n’y a aucune norme établie d’or pour normaliser les miARN de sérum ; Toutefois, des contrôles exogènes, par exemple les dopés c. elegans miR-39 sont une solution élégante pour les procédures de normalisation, comme endogène miARN est souvent altérées dans diverses maladies États9.
Les échantillons de sérum analysées ont été acquis avant une intervention coronarienne percutanée (ICP), 8 h après une carte PCI et à 16 h suivant une ICP, provenant de patients avec un segment ST infarctus du myocarde aigu (STEMI). STEMI patients présentent une ischémie sévère et ainsi se qualifier pour l’évaluation de nouveaux biomarqueurs de l’ischémie. Pour évaluer la cinétique de libération miR-499 et l’utilisation potentielle de miR-499 comme biomarqueur pour ischémie dans les maladies cardiovasculaires, miR-499 a été analysée avec dPCR dans l’ensemble de tous les patients pour les trois temps différents points14.
La figure 1 illustre la sélection de gouttelettes positifs donnant la concentration finale de miRNA calculée par le logiciel. La fraction de positifs dans un échantillon détermine la concentration des copies/µL, telle que calculée par le logiciel commercial dPCR. Les résultats peuvent également être visualisées dans un graphique 2D, et les points positifs peuvent être marqués manuellement avec des cercles. Les concentrations sont considérées uniquement si elles sont au-dessus de l’amplification non spécifique des NTCs.
La figure 2 montre la linéarité et la qualité de l’ajustement d’une série de dilutions d’a-miR-499-5 p oligonucléotide synthétique miRNA des doublons. Une série de dilution de 8 étapes a été réalisée de 2 500 exemplaires/µL à 0 copies/µL. Les copies/µL attendue calculée comme indiqué dans le tableau 5 assumer 100 % RT et efficacité PCR numérique. Dans cette configuration, PCR numérique montre un haut degré de linéarité (r2 = 0,99). La limite de détection (LoD = 0,12) et la limite de quantification (LQ = 0,23) sont également présentés dans la Figure 2, montrant que même une expression faible de miRNA peut être détectée avec succès. Ainsi, dPCR peut être utilisé pour quantifier la même faible taux sériques des miARN en circulation. La limite de détection (LD) et la limite de quantification (LQ) sont calculés conformément à l’approche de Fouquet et al. 15 et sont définis comme LoD = LoB + 1,645 x σbasconcentrationexemple, où la limite du blanc (LoB) est calculée comme LoB = moyenneblanc + 1,645 x σvide. La LDD est alors estimée répliquer en cours d’exécution standard courbes15. Afin d’assurer la quantification reproductible des miARN, seulement les concentrations de miRNA qui se trouvent au-dessus de fois la limite de détection et la limite de dosage sont considérées comme une valeur exacte valide.
La figure 3 illustre les résultats représentatifs de miR-499 niveaux des patients avec un ST-du myocarde (IDM), n = 16 à chaque instant. Des échantillons ont été prélevés avant une intervention percutanée (ICP) (t = 0), 8 h après un PCI (t = 8) et 16 h après un PCI (t = 16), et les niveaux de miR-499 ont par rapport aux niveaux de miR-499 des patients avec maladie coronarienne stable (CAD, n = 20). On trouvera au tableau 6, les principales caractéristiques du patient. Après la sortie initiale de miR-499 dans le sérum au cours des 8 premières heures, suite à un infarctus du myocarde, miR-499 niveaux diminuent à nouveau à partir de 16 h. Une tendance à la hausse peut déjà être vu au début de l’infarctus (t = 0), et une augmentation significative des concentrations de miR-499 comparée aux patients avec CAD peut être vu à 8 h après l’infarctus (t = 8, p < 0,01) lors de la comparaison des niveaux de miR-499 à miR-499 niveaux des patients stables de CAD. Le fonctionnement du récepteur courbes Voir la (ROC) l’utilité possible de miR-499 comme nouveaux biomarqueurs pour différencier les patients avec CAD et patients présentant un SCA ST + [l’aire sous la courbe (AUC) = 0,62 sur des échantillons prélevés avant une intervention percutanée (ICP), AUC = 0,75 pour les échantillons prélevés 8 h après une PCI et AUC = 0,78 pour échantillons prélevés 16 h suivant une ICP par rapport aux niveaux de sérum de miR-499 chez les patients avec CAD].

Figure 1 : sélection des gouttelettes positives dans une série de dilution effectuée en doublons. (A) le seuil est défini manuellement au-dessus les gouttelettes négatifs. (B) les résultats sont visualisés dans une tache 2D, les points positifs sélectionnés en encerclant. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
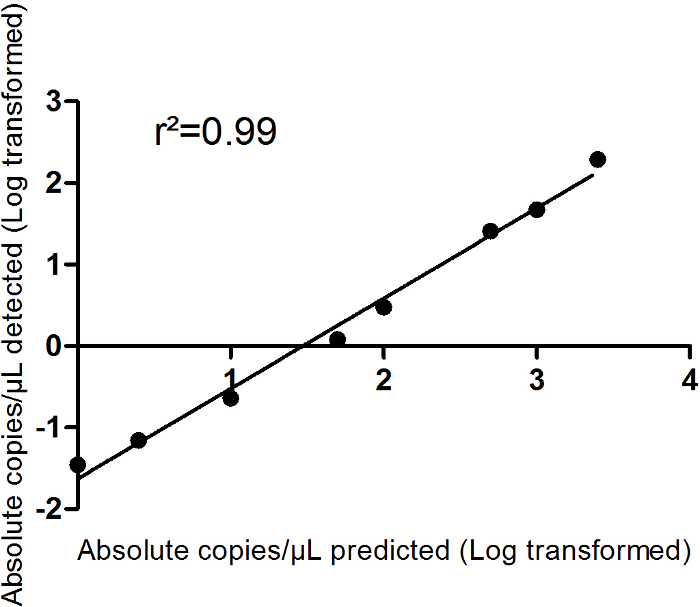
Figure 2 : série de dilutions d’oligonucléotide synthétique de miR-499. (A) les mesures ont été effectuées en double. Les données sont présentées journal transformé sous forme de copies absolues par µL. régressions linéaires et d’ajustement (r2-valeur) sont indiquées. Les données sont présentées comme moyenne ± SEM. (B), ce panneau montre la limite de détection (LD) et la limite de quantification (LQ). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 3 : Quantification des p miR-499-5 sérique chez les patients atteints d’infarctus du myocarde-ST en circulation (n = 16) avant l’intervention coronarienne percutanée (ICP), 8 h après PCI et 16 h après PCI, comparativement aux patients avec coronarienne stable maladie (n = 20). (A) la circulation sérum miR-499-5p est représenté sous forme de moyenne avec l’erreur-type de la moyenne avec le correspondant p-valeur calculée par ANOVA à suivie d’un test post hoc de Bonferroni (p < 0,05 était considérée comme statistiquement significative). Récepteur (B) une analyse de la courbe caractéristique (ROC) est effectué sur les données du groupe A. L’analyse ROC montre la sensibilité et la spécificité du biomarqueur. En outre, la zone correspondante sous les valeurs de la courbe (ASC) sont indiquées. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
| quantité d’échantillon | ||||
| n = 1 | / bien [μl] | |||
| eau exempte de nucléase | 4.16 | |||
| 10 x tampon Reverse Transcription | 1.5 | |||
| 100nM dNTP | 0,15 | |||
| Inhibiteur de RNAse | 0,19 | |||
| Amorces spécifiques de RT | 3 | |||
| Commerciale Reverse Transcriptase Enzyme 50 U/ul | 1 | |||
| Total Master-Mix | 10 | |||
Tableau 1 : Mélange de réactifs transcriptase inverse.
| Température | Temps |
| 16 ° C | 30 mn |
| 42 ° C | 30 mn |
| 85 ° C | 5 min. |
| 4 ° C | ∞ |
Tableau 2 : Cyclisme Conditions thermiques pour la Transcription inverse.
| quantité d’échantillon | ||||
| n = 1 | / bien [μl] | |||
| eau exempte de nucléase | 7,67 | |||
| mélange commercial dPCR | 10 | |||
| 20 x hydrolyse spécifique apprêt/sonde | 1 | |||
| Total Master-Mix | 18.67 | |||
Tableau 3 : PCR numérique réactif Mix.
| Température | Temps |
| 95 ° C | 10 mins. |
| 94 ° C | 30 secondes (x40) |
| 60 ° C | 1 min |
| 98 ° C | 10 mins. |
| 4 ° C | ∞ |
Tableau 4 : Cyclisme des Conditions thermiques pour l’ACP numérique.
| Tube | Transférés d’oligonucléotide synthétique miR-499 (µL) | Diluant (µL) | miRNA (copies/µL) | Attendus des Copies/µL en RT | Copies/µL attendue dans dPCR |
| Langue source | 6.022 x 1012 | ||||
| 1 | 10 | 990 | 6.022 x 1010 | ||
| 2 | 10 | 990 | 6.022 x 108 | ||
| 3 | 10 | 990 | 6.022 x 106 | ||
| 4 | 18,7 | 981.3 | 1,128 x 105 | 37600 | 2500 |
| 5 | 40 | 60 | 4.511 x 104 | 15040 | 1000 |
| 6 | 50 | 50 | 2,256 x 104 | 7520 | 500 |
| 7 | 40 | 160 | 4.511 x 103 | 1504 | 100 |
| 8 | 50 | 50 | 2,256 x 103 | 752 | 50 |
| 9 | 40 | 160 | 4.511 x 102 | 150,4 | 10 |
| 10 | 50 | 150 | 1,128 x 102 | 37,6 | 2.5 |
| 11 | 0 | 50 | 0 | 0 | 0 |
Tableau 5 : Les séries de dilutions synthétique pour miR-499.
| Caractéristique | Tous les | Stable CAD Patients (n = 20) | STEMI Patients (n = 24) | p-valeur |
| Age | 64,7 ± 11,9 | 66,7 ± 13,1 | 62,2 ± 9,9 | 0.2810 |
| Mâles | 77,8 % | 65 % | 93,8 % | 0.0392 |
| DM | 33,3 % | 40 % | 25 % | 0.3428 |
| HTN | 61,1 % | 60 % | 62,5 % | 0.8785 |
| Dyslipidémie | 38,9 % | 65 % | 6.3 % | 0,0003 |
| Fumeur | 47,2 % | 55 % | 37,5 % | 0.3796 |
| Antécédents familiaux | 25 % | 35 % | 12,5 % | 0.1213 |
| Surcharge pondérale | 58,3 % | 55 % | 62,5 % | 0.6501 |
| Créatinine sérique (mg/dL) | 0,94 (0,83 - 1) | 0,98 (0,82 - 1.01) | 0,93 (0,83 - 1) | 0.7255 |
| CK Peak (U / I) | 161 (102-611) | 126 (81-161) | 492 (228-3208) | 0,0004 |
| cTnT Peak (ng/L) | 322,5 (23,8-4533) | 18 (7-25,3) | 2492 (240-5586) | 0,0002 |
| FEVG % | 45 % (40 % - 50 %) | 45 % (32,5 % - 55 %) | 45 % (45-50 %) | 0.9596 |
| Les valeurs sont présentées comme moyenne ± écart-type ; valeur médiane (25e au 75e centile) ou % | ||||
| DM : diabète sucré | ||||
| HTN : hypertension artérielle | ||||
| CK : créatine kinase | ||||
| cTnT : troponine cardiaque T | ||||
| FEVG : Fraction d’éjection ventriculaire gauche | ||||
Tableau 6 : Principales caractéristiques de patient.
Discussion
PCR numérique est une méthode relativement nouvelle butée de PCR qui permet la quantification absolue directe d’acides nucléiques dans un échantillon. La méthode possède des avantages particuliers, notamment une diminution de la variabilité, une reproductibilité quotidienne accrue et une sensibilité supérieure11,12. En outre, en raison de la répartition de l’échantillon dans environ 20 000 réactions simples et des analyses de point de terminaison, dPCR est plus robuste aux substances interférentes en PCR par rapport à de RT-PCR quantitative16. Ces qualités dans dPCR en font une alternative intéressante à la RT-PCR quantitative comme outil de diagnostic pour la quantification. Comme circulation miARN sont souvent présents en faibles concentrations sériques, jusqu’ici, scientifiques ont été contestées de quantifier adéquatement miARN par PCR9. En revanche, dPCR peut quantifier adéquatement même une expression faible de miRNA dans le sérum, atténuer les problèmes observés dans la faible numération de quantification miRNA17. La capacité de dPCR à donner directement les chefs d’accusation/µL, même dans une expression très faible miRNA, rend donc un outil de diagnostic intéressant pour la communauté de recherche cardiovasculaire dans les études de biomarqueurs de miRNA. Comme la concentration indiquée en chiffres/µL peut être multipliée par le facteur de dilution utilisé dans l’extraction, RT-réaction et dPCR, il est possible d’atteindre le nombre exact de copies en 1 µL de sérum. Le flux de travail présentée ici peut être effectuée jusqu'à 96 échantillons sur une plaque, offrant ainsi un outil pour les grandes études cardiovasculaires. Bien que dPCR présente plusieurs avantages par rapport aux RT-PCR quantitative, il n’est pas encore systématiquement appliqué pour la quantification des miARN dans les essais cardiovasculaires. Procédures de normalisation des données supplémentaires, normalisé ne manquent pas.
Dans cette approche, nous démontrons que dPCR, combiné avec les sondes fluorescentes hydrolyse, permet la quantification absolue directe spécifique consistant à diffuser les miARN liés aux maladies cardiovasculaires. Avec ce rapport, nous avons cherché à démontrer un protocole optimisé pour la détection de miRNA via dPCR et confirmer les avantages de dPCR par RT-PCR quantitative.
Nous avons confirmé les bonne linéarité dPCR expositions et les limites basses de la détection et la quantification des dPCR pour quantifier les miARN. De l’échantillon avec un oligonucléotide synthétique de fortification, la normalisation de l’échantillon est possible, ajustement pour l’efficacité de l’extraction et de la variation de l’échantillon à.
En conclusion, PCR numérique, car il montre la supériorité dans les compétences techniques et le potentiel diagnostique, est la meilleure méthode actuelle pour la quantification de miRNA et plus peut-être être utilisé pour les études de biomarqueurs grand multicentrique cardiovasculaire miRNA.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs n’ont aucuns accusés de réception.
matériels
| Name | Company | Catalog Number | Comments |
| RNA-Extraction | |||
| miRNeasy Serum/Plasma Kit (50) | Qiagen-Sample & Assay Technologies, Hilden, Deutschland | 217184 | Kit for microRNA extraction. Kit contains commercial buffer RWT (called number one in the manuscript) and RPE (called number two in manuscript). |
| miRNeasy Serum/Plasma Spike-in-Control; Syn-cel-miR-39 miRNA; 10pmol | Qiagen-Sample & Assay Technologies, Hilden, Deutschland | 219610 | Spike-in for normalisation , Sequence: 5'-UCACCGGGUGUAAAUCAGCUUG-3' |
| Reverse Transcription | |||
| TaqMan MicroRNA Reverse Transcription Kit (1000 Reactions) | Applied Biosystems, Inc., Foster City, CA, USA | 4366597 | Kit for microRNA reverse transcription |
| TaqMan MicroRNA Assays M | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assays used in reverse transcription |
| hsa-miR-499 (750 RT/750 PCR rxns) | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assay Number 001352 |
| cel-miR-39 (750 RT/750 PCR rxns) | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assay Number 000200 |
| PCR Plate, 96-well, segmented, semi-skirted | Thermo Fisher Scientific, Waltham, MA, USA | AB0900 | 96 well plate for reverse transcription |
| Microseal ‘B’ seal Seals | Bio-Rad Laboratories, Inc., Hercules, CA, USA | MSB1001 | Foil to ensure proper storage |
| C1000 Touch Thermal Cycler | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1851196 | Cycler used for reverse transcription |
| Droplet Digital PCR | |||
| 100 nmole RNA oligo hsa-miR-499-5p | Integrated DNA Technologies | Custom | Sequence: 5'-phos-UUAAGACUUGCAGUGAUGUUU-3' |
| ddPCR Supermix for Probes (No dUTP) | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863024 | Supermix used in droplet generation |
| TaqMan MicroRNA Assays | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assays used in digital PCR (fluorescent hydrolysis probe) |
| hsa-miR-499 (750 RT/750 PCR rxns) | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assay Number 001352, commercial primers |
| cel-miR-39 (750 RT/750 PCR rxns) | Applied Biosystems, Inc., Foster City, CA, USA | 4440887 | Assay Number 000200, commercial primers |
| DG8 Cartridges and Gaskets | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1864007 | Cartridge takes up to 8 samples for droplet generation |
| DG8 Cartridge Holder | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863051 | Holds cartridges in droplet generation |
| Droplet Generation Oil for Probes | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863005 | Oil used in droplet generation |
| ddPCR 96-Well Plates | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 12001925 | 96 well plate for ddPCR |
| PCR Plate Heat Seal, foil, pierceable | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1814040 | Pierceable foil, compatible with droplet reader |
| ddPCR Droplet Reader Oil | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863004 | Oil used in droplet reading |
| QX100 or QX200 Droplet Generator | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863002 | Droplet Generator, generates the droplets from sample/oil emulsion |
| PX1 PCR Plate Sealer | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1814000 | Seals the plate before PCR |
| C1000 Touch Thermal Cycler | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1851196 | Cycler used for ddPCR |
| QX100 or QX200 Droplet Reader | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863003 | Reads PCR-positive and PCR-negative droplets with an optical detector |
| ddPCR Buffer Control for Probes | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1863052 | Blank control and to fill up the remaining wells of 8-well cassette |
| Software | |||
| QuantaSof Software | Bio-Rad Laboratories, Inc., Hercules, CA, USA | 1864011 | Program for droplet reading |
| Prism Windows 5 | GraphPad Software Inc., La Jolla, CA, USA | Program for statistical analysis |
Références
- Schulte, C., Zeller, T. microRNA-based diagnostics and therapy in cardiovascular disease - summing up the facts. Cardiovascular Diagnosis and Therapy. 5, 17-36 (2015).
- Sun, T., et al. The role of microRNAs in myocardial infarction: from molecular mechanism to clinical application. International Journal of Molecular Sciences. 18, (2017).
- Dimmeler, S., Zeiher, A. M. Circulating microRNAs: novel biomarkers for cardiovascular diseases. European Heart Journal. 31, 2705-2707 (2010).
- Creemers, E. E., Tijsen, A. J., Pinto, Y. M. Circulating microRNAs: novel biomarkers and extracellular communicators in cardiovascular disease. Circulation Research. 110, 483-495 (2012).
- Oerlemans, M. I., et al. Early assessment of acute coronary syndromes in the emergency department: the potential diagnostic value of circulating microRNAs. EMBO Molecular Medicine. 4, 1176-1185 (2012).
- Olivieri, F., et al. Diagnostic potential of circulating miR-499-5p in elderly patients with acute non ST-elevation myocardial infarction. Internation Journal of Cardiology. 167, 531-536 (2013).
- Navickas, R., et al. Identifying circulating microRNAs as biomarkers of cardiovascular disease: a systematic review. Cardiovascular Research. 111, 322-337 (2016).
- Devaux, Y., et al. Diagnostic and prognostic value of circulating microRNAs in patients with acute chest pain. Journal of Internal Medicine. 277, 260-271 (2015).
- Schwarzenbach, H., da Silva, A. M., Calin, G., Pantel, K. Data normalization strategies for microRNA quantification. Clinical Chemistry. 61, 1333-1342 (2015).
- Hindson, B. J., et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number. Analytical Chemistry. 83, 8604-8610 (2011).
- Hindson, C. M., et al. Absolute quantification by droplet digital PCR versus analog real-time PCR. Nature Methods. 10, 1003-1005 (2013).
- Robinson, S., et al. Droplet digital PCR as a novel detection method for quantifying microRNAs in acute myocardial infarction. Internation Journal of Cardiology. 257, 247-254 (2018).
- Mitchell, P. S., et al. Circulating microRNAs as stable blood-based markers for cancer detection. Proceedings of the National Academy of Sciences of the United States of America. , 10513-10518 (2008).
- D'Alessandra, Y., et al. Circulating microRNAs are new and sensitive biomarkers of myocardial infarction. European Heart Journal. 31, 2765-2773 (2010).
- Forootan, A., et al. Methods to determine limit of detection and limit of quantification in quantitative real-time PCR (qPCR). Biomolecular Detection and Quantification. 12, 1-6 (2017).
- Dingle, T. C., Sedlak, R. H., Cook, L., Jerome, K. R. Tolerance of droplet-digital PCR vs real-time quantitative PCR to inhibitory substances. Clinical Chemistry. 59, 1670-1672 (2013).
- Taylor, S. C., Laperriere, G., Germain, H. Droplet digital PCR versus qPCR for gene expression analysis with low abundant targets: from variable nonsense to publication quality data. Scientific Reports. 7, 2409(2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon