Method Article
Istologico quantificazione per determinare il fardello fungine del polmone nell'aspergillosi sperimentale
In questo articolo
Riepilogo
Qui descriviamo un protocollo per determinare l'onere fungine polmonari nei topi con aspergillosi invasiva mediante la quantificazione di argento methanamine modificate di Gomori colorazione nelle sezioni istologiche. Utilizzo di questo metodo ha provocato risultati comparabili con meno animali rispetto alla valutazione dell'onere fungine mediante PCR quantitativa di DNA fungoso del polmone.
Abstract
La quantificazione degli oneri fungine del polmone è fondamentale per la determinazione dei livelli relativi di protezione immunitaria e virulenza fungina in modelli murini di micosi polmonare. Anche se più metodi sono utilizzati per valutare l'onere fungine, reazione a catena della polimerasi quantitativa (qPCR) di DNA fungoso è emerso come una tecnica con diversi vantaggi rispetto ai metodi basati su cultura precedenti. Attualmente, una valutazione completa della patologia del polmone, reclutamento leucocitario, onere fungine ed espressione genica nei topi con aspergillosi invasiva (IA) richiede l'uso di un numero significativo di sperimentale e animali di controllo. Qui la quantificazione del polmone istologico macchiatura per determinare l'onere fungine utilizzando un ridotto numero di animali è stata esaminata in dettaglio. Sezioni del polmone sono stati macchiati per identificare le strutture fungine con argento di Gomori modificata methanamine (GMS) colorazione. Immagini sono state prese dalle sezioni GMS-macchiato da 4 campi discreti di ciascun polmone di paraffina-incastonato formalina-fisso. Il GMS macchiato aree all'interno di ogni immagine sono stato quantificato utilizzando un programma di analisi di immagine, e da questa quantificazione, la percentuale media di zona macchiata è stata determinata per ciascun campione. Utilizzando questa strategia, topi eosinofilo-carenti hanno esibiti diminuito onere fungine e malattia con la terapia del caspofungin, mentre topi wild-type con IA non sono migliorato con caspofungin. Allo stesso modo, onere fungine in topi privi di cellule T γδ inoltre sono stati migliorati da caspofungin, come misurato da qPCR e quantificazione di GMS. Quantificazione di GMS pertanto è stato introdotto come un metodo per la determinazione degli oneri fungine relativo polmone che possono in ultima analisi, ridurre la quantità di animali sperimentali necessari per studi completi dell'aspergillosi dilagante.
Introduzione
IA è un'infezione opportunistica che possono svilupparsi in individui suscettibili con immunodeficienze congenite o acquisite a causa di terapia soppressiva immune o infezione cronica1,2. Spesso l'infezione primaria si verifica nei polmoni, anche se in alcuni diffusione di istanze di Aspergillus fumigatus a fegato, reni, cuore e cervello può verificarsi, con conseguente invasione di vasto tessuto di ife accompagnata dalla malattia severa e alta tassi di mortalità1,2. Inoltre, l'efficacia di farmacoterapie esistente è limitata e può essere ulteriormente indebolita dall'emergere di ceppi resistenti antimicotico in ambiente3. È quindi importante capire i meccanismi della patologia fungina di virulenza e host che promuovono lo sviluppo o esacerbazione della malattia fungosa dilagante.
Modelli murini rimangono importanti per studi meccanicistici IA, in quanto consentono ai ricercatori di valutare il ruolo dei geni di virulenza fungina e ospitare gli effettori immunitari per l'istituzione e lo sviluppo di a. fumigatus in vivo4,5. Di conseguenza, più strategie sono state escogitate per effettivamente quantificare o confrontare fungine onere nei gruppi di animali da esperimento6,7. Queste strategie coinvolgono basati su cultura, biochimico, immunologico o metodi qPCR, ognuno con vantaggi e svantaggi. Inoltre, ciascuno di questi metodi implicano la dedizione di un sottoinsieme degli animali oltre a quelli sacrificati per le valutazioni della funzione effector immuni, analisi di espressione genica e l'istopatologia comparativa7. Così, studi completi IA richiedono spesso un numero significativo di animali di ricerca ad un costo significativo. Strategie efficaci che riducono il tempo sperimentale, i costi degli animali e considerazioni etiche utilizzando tessuti animali per analisi multiple sono quindi estremamente prezioso7.
In questo rapporto, è stato introdotto un metodo che descrive la quantificazione di GMS colorazione nelle sezioni istologiche per il confronto dell'onere relativo fungina tra gruppi sperimentali dei topi con IA. Ogni passaggio da coltura fungosa di infezione, tessuto raccolta e lavorazione e acquisizione di immagini e analisi dei dati, è descritto in dettaglio. Fungini oneri ottenuti mediante la quantificazione di GMS sono stati confrontati con qPCR in neutropenic modelli di IA e nei topi wild-type o eosinofilo-carenti trattati con caspofungin con IA8. I risultati mostrano somiglianza con GMS quantificazione e qPCR di DNA fungoso. Questo suggerisce che la quantificazione di GMS può essere utile ai ricercatori impegnati in analisi istologiche come metodo alternativo o supplementare di confronto dell'onere relativo fungine in topi con IA e, infine, può ridurre i costi e l'uso di animali di ricerca in complessi studi meccanicistici.
Protocollo
Tutte le procedure di animali sono state approvate dalla cura degli animali e uso Comitato di Indiana State University, il campus di host dell'Indiana University School of Medicine — Terre Haute.
1. preparazione di a. fumigatus conidi per infezione
- Mentre si lavora in una cappa ben ventilata, aggiungere 125 µ l di soluzione madre di a. fumigatus 293 900 µ l cella cultura acqua di qualità (CCGW). Quindi trasferire la soluzione per piastra di agar (MEA) estratto tramite pipetta di malto e distribuire uniformemente con un'ansa da inoculo.
- Conservare la piastra fungina al buio a 24 ° C, per almeno 14 giorni e non più di 28 giorni.
- Conidi raccolto usando i branelli di vetro come precedentemente descritto9.
- Versare 1,5 g di 0,5 mm perle di vetro sulla piastra e inclinare delicatamente avanti e indietro fino a quando le perline sono rivestite per estrarre conidi. Raccogliere la miscela di tallone/conidi in una provetta conica da 15 mL. Sospendere in tampone fosfato salino (DPBS) di 5ml Dulbecco e vortex.
- Contare i conidi in una diluzione di 50x del surnatante in DPBS utilizzando un emocitometro. Contare il numero di conidi nella zona indicata in Figura 1 e utilizzare 1 equazione per calcolare la concentrazione.
Fattore di diluizione × #Conidia × 104= conidi/mL (equazione 1)
Nota: Ulteriori aree di angolo quadrato 4x4 possono essere contati, con la media di queste aree usate come #Conidia in equazione 1.
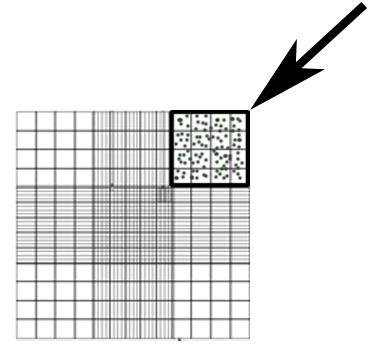
Figura 1: Emocitometro rappresentanza area utilizzata per conidi contando (scatola, freccia).
- Diluire la sospensione di conidi con soluzione salina sterile (DPBS) per ottenere la concentrazione desiderata, 1.0 x 108 conidi/mL per l'infezione con 5 x 106 conidi/50 µ l. preparare 50 µ l di soluzione di conidi a ' n + 2' topi infetti al conto per il pipettaggio errore.
Nota: Standard fungo raccolto e preparazione/filtrazione possono essere usati in alternativa10. Il metodo del branello di vetro è meglio utilizzato per garantire che i campioni di conidi con minimi piccolo/solubile antigeni hyphal sono ottenuti per aspirazione.
2. l'immunosoppressione e infezione del modello murino
- Utilizzare 10-week-old 7 topi.
Nota: BALB/c, C57BL/6 (B6), eosinofilo-carenti (ΔdblGATA, sfondo BALB/c) e delle cellule T γδ-carenti (TCRδ-/-, sfondo B6) topi sono stati usati per questo studio. Per ogni esperimento, età e genere-abbinato topi (sia maschi che femmine) sono stati usati per ogni gruppo. - Impoveriscono neutrofili via (IP) iniezione intraperitoneale dell'anticorpo di anti-mouse-Ly - 6G di 0,1 mL (0,5 mg) in soluzione fisiologica sterile a 45° nel quadrante destro inferiore laterale (per un programma sperimentale, Vedi Figura 2).
- 24 ore più tardi, infettare topi dall'aspirazione con 5,0 x 106a. fumigatus conidi sospesi in 50 soluzione salina sterile µ l (vedere i passaggi 2.4-2.10) come descritto in precedenza11. Dopo l'infezione, iniettare un sottoinsieme degli animali con agente antimicotico come diacetato di caspofungin 5 mg/kg in DPBS utilizzato in questo studio (tutti i giorni, Vedi Figura 2).
- Anestetizzare topi con isoflurano 99,9% utilizzando una macchina di anestesia veterinaria.
- Posizionare un tovagliolo di carta sul pavimento della camera ad induzione per catturare le feci e nelle urine.
- Posizionare il mouse nella camera di induzione, avvitare il coperchio e impostare il vaporizzatore per isoflurano 5% per 2 min e 30 s.
Nota: Unguento veterinario non viene utilizzato perché il tempo totale in anestesia è meno di 5 min. - Ridurre il vaporizzatore a metà l'impostazione massima per 30 s.
- Confermare che il mouse è adeguatamente anestetizzato osservando rallentato la respirazione, mancanza di movimento e la mancanza di risposta allo stimolo di punta-pizzico.
- Rimuovere il mouse dall'aula di induzione e posto su un asse inclinato. Posizionare il mouse con le spalle contro il bordo. Assicurarsi che i suoi incisivi superiori sono delicatamente trattenuti con un elastico e gli incisivi inferiori sono delicatamente trattenuti con un filo metallico.
- Tenere attentamente la lingua alla massima estensione con piccole pinze e dispensare 50 µ l della sospensione di conidi/PBS alla base della linguetta.
- Tieni a freno la lingua alla massima estensione; Ascolta per respirazione rapida e il distinto rumore di aspirazione. Tenere premuto per 20 s o fino a quando la sospensione è completamente aspirato.
- Rimuovere il mouse dal bordo inclinato e posizionare con delicatezza in una gabbia per l'osservazione.
- A seguito di aspirazione, il mouse dovrebbe riprendere coscienza entro 1 min Monitor mouse finché non pienamente cosciente e in grado di camminare intorno alla gabbia. Gli animali dovrebbero essere ospitati a 21 ° C e controllati due volte al giorno fino a che i polmoni sono raccolte.
- Ripetere l'esaurimento dei neutrofili (punto 2.2) 24h post-infettiva (Figura 2).

Figura 2: pianificazione sperimentale. Questa figura è stata modificata da una precedente pubblicazione8.
3. raccolta e conservazione dei polmoni di Mouse per analisi istologica
- Profondamente anestetizzare l'animale con una dose fatale di 0,1 ml di 390 mg/ml sodio pentobarbital soluzione IP.
- Confermare l'eutanasia osservando la mancanza di respirazione spontanea e l'assenza di risposta alla stimolazione degli arti con le pinzette. Una volta che l'eutanasia, spruzzare la carcassa con etanolo al 70% per sterilizzare.
- Posto la carcassa su un bordo della gomma piuma è ricoperta da un tampone assorbente.
Nota: La carcassa deve essere inserita sul dorso con perni fissaggio tutte e quattro le membra per il bordo della gomma piuma. - Aprire la cavità di cassa superiore utilizzando le forbici con cautela. Tagliare via la gabbia toracica al fine di esporre i polmoni e il cuore. Tagliare la vena cava inferiore per consentire per aspersione. Evitare i vasi sanguigni di taglio e perforazione di organi.
- Irrorare lentamente con 5 mL di DPBS freddo nel ventricolo destro del cuore ad un angolo di 45° con un ago da 25-G. Ripetere la perfusione con 5 mL di formalina al 10%.
- Attentamente esporre la trachea e staccare i cuscinetti di grasso circostanti. Prestare attenzione ed evitare di perforare la trachea. Assicurarsi che il tessuto connettivo in eccesso viene rimosso al fine di visualizzare completamente la trachea prima di procedere. Una volta che la trachea è esposto, collocare la pinza sotto la trachea per elevarlo.
- Colpire un piccolo foro nella trachea superiore utilizzando un ago 25-G.
Nota: Il foro deve essere posizionato in modo che la cannula intera si inserisce nella trachea. Usare cautela per evitare la rottura della trachea. - Inserire una cannula attraverso il foro e verso i polmoni. Fissare la cannula con un filo chirurgico legato senza bloccare intorno alla trachea.
Nota: Se si rompe la trachea, la cannula può essere inserita passato la trachea per quanto possibile nelle vie respiratorie. - Usando una siringa, gonfiare i polmoni con 1 mL di tampone di formalina 10% attraverso la cannula. Assicurarsi che l'inflazione dei polmoni è visibile. In caso contrario, regolare il catetere e ripetere. Una volta che i polmoni sono gonfiati, rapidamente rimuovere la cannula con cautela e stringere il filo in modo sicuro per sigillare la trachea.
Nota: Se la trachea è rotto e non possono essere gonfiati i polmoni, i polmoni possono ancora essere raccolti ma questo potrebbe impedire il corretto fissaggio. - Rimuovere delicatamente la trachea e i polmoni dal petto staccando accuratamente il tessuto connettivo della trachea con il forcipe tirando delicatamente sul thread. Posizionare i polmoni e la trachea in una provetta conica da 50 mL con 15 mL di buffer di formalina 10%. Inserire un'estremità del filo esterno del tubo e il tappo di tenuta. Capovolgere la provetta per immergere completamente il campione nel fissativo.
- Conservare i campioni a temperatura ambiente per almeno 24 ore o fino a ulteriore elaborazione.
Attenzione: La formalina è pericolosa. Indossare adeguati dispositivi di protezione personali compresi occhiali e mascherina facciale. Lavorare sotto una cappa chimica durante l'elaborazione di campioni.
4. raccolta e conservazione dei polmoni di Mouse per DNA fungine onere
- Seguire i passaggi 3.1-3.5 come descritto in precedenza su un mouse separato.
- Irrorare lentamente con 10 mL di DPBS freddo nel ventricolo destro del cuore ad un angolo di 45° con un ago da 25-G.
- Asportare i polmoni con le forbici e posto in provette da 1,5 mL con etichetta.
- Memorizzare i polmoni a-80 ° C fino a ulteriore elaborazione.
- Utilizzare le tecniche standard come descritto (incorporamento e sezionamento) e secondo i protocolli del produttore (Vedi Tabella materiali).
5. microscopia e Imaging
- Aprire il programma imaging di quantificazione (Vedi Tabella materiali).
- Utilizzando un microscopio ottico, ottenere almeno 4 campi distinti di GMS (obiettivo 10x) vetrini colorati. Garantire che le immagini sono rappresentative delle vie aeree all'interno del polmone con simile densità del tessuto infettate.
Nota: Se la densità e la distribuzione dei fuochi ifali appaiono marcatamente differenti fra gruppi sperimentali e di controllo, campi aggiuntivi possono essere adottate, o l'area di GMS della sezione intero polmone può essere imaged (obiettivo 4x). Ottenere immagini delle stesse zone del polmone (identificato dalla morfologia del tessuto equivalente) su una sezione di polmone seriale colorati con ematossilina ed eosina, se lo si desidera. - Aggiungere barre di scala per le immagini, ove necessario e salvare le immagini da quantificare. Selezionate, modifica > Aggiungi/modifica taratura marchi > Menu: Scegli la dimensione barra di spessore, colore e scala di riga > Clicca sull'immagine per la quale la barra della scala deve essere messo. Chiudere il Menu e scegliete, modifica > Merge > Merge calibrazione marchi > scegliere Salva con nome > nome del file e fare clic su Salva.
6. utilizzando un programma di elaborazione immagini per calcolare l'onere fungina (Figura 3)
- Aprire un programma di elaborazione di immagini. Selezionare File > cartella con campioni di quantificare > immagine (Figura 3A). Selezionare immagine > tipo > convertire "RGB stack" (Figura 3B).
- Utilizzare il tasto freccia destra per selezionare la seconda delle tre immagini data (Figura 3B). Premere "CTRL + MAIUSC + T" per far apparire il flusso di "Soglia" dal menu impostazioni (Figura 3). Individuare l'immagine TIFF o jpg originale come riferimento e aprire con una programma (Figura 3) la visualizzazione di immagini.
- Tirare il cursore in alto a sinistra e regolare il dispositivo di scorrimento inferiore fino a quando l'area selezionata (in rosso) è rappresentativo dell'immagine di microscopia (in genere che vanno da 130-160 come indicato a destra del dispositivo di scorrimento (Figura 3).
- Una volta selezionato, premere il tasto "Set" > "OK" (Figura 3D). Selezionare "Analizza" > "Impostare misure" > Verifica "Area, frazione di zona, limite di soglia, etichetta visualizzata" e lasciare le impostazioni di fondo (il menu a discesa e decimali) come predefinito > "OK" (Figura 3D).
- Aree significative di spazi vuoti o una colorazione di fondo possono essere esclusi dalla selezione del tessuto appropriato con uno strumento di area o poligono.
- Infine seleziona, "Analizza" > "Misura" (dovrebbe apparire una finestra con i dati, o verrà visualizzata una scheda della barra delle applicazioni di windows con l'etichetta, "Risultati") (Figura 3E). Trasferire i dati ad un programma di analisi di dati per grafici e analisi statistica.
- Inserire la media delle percentuali di quattro area per ogni campione del polmone. 5-8 campioni sono generalmente sufficienti per rilevare differenze significative tra i gruppi. Se si confrontano due gruppi sperimentali, eseguire un spaiata dello studente t-test per determinare il significato.

Figura 3: quantificazione di campo rappresentante screenshots per ogni passaggio di GMS istologico. (A) selezionare File > cartella con campioni di quantificare > Seleziona immagine. (B) selezionare immagine > tipo > convertire "RGB stack". Utilizzare il tasto freccia destra per selezionare la seconda delle tre immagini specificate. (C) Hit "CTRL + MAIUSC + T" per far apparire il flusso di "Soglia" dal menu impostazioni. Individuare l'immagine TIFF o jpg originale come riferimento e aprire con il programma Visualizzatore di foto. Tirare il cursore in alto tutta la strada a sinistra e regolare il dispositivo di scorrimento inferiore fino a quando l'area selezionata (in rosso) è rappresentativo dell'immagine di microscopia (in genere che vanno da 130-160 come indicato a destra del cursore. (D) una volta selezionato premere il tasto "Set" > "OK". Selezionare "Analizza" > "Impostare misure" > Verifica "Area, frazione di zona, limite di soglia, etichetta visualizzata" e lasciare le impostazioni di fondo (il menu a discesa e decimali) come predefinito > "OK". (E), infine seleziona "analizza" > "Misura" (dovrebbe apparire una finestra con i dati, o verrà visualizzata una scheda della barra delle applicazioni di windows con l'etichetta "Risultati"). Clicca qui per visualizzare una versione più grande di questa figura.
7. DNA fungine onere
- Rimuovere i polmoni da-80 ° C e lyophilize per almeno 4 ore (durante la notte è ottimale) o fino a quando non completamente asciugato da un sistema a secco di congelare.
- Mentre i polmoni stanno asciugando, preparare il tampone di estrazione del DNA (0,1 M NaCl, 10 mM EDTA pH 8.0, 10 mM Tris-HCl pH 8.0 e 0,5% SDS).
- Posizionare il tampone di estrazione del DNA preparato in bagnomaria a 60 ° C per pre-riscaldare. Inoltre, posto il fenolo: cloroformio: alcool isoamilico (25:24:1) in una provetta conica da 50 mL e consentire per la separazione.
- Collocare i polmoni liofilizzati in una provetta 2 mL tappo a vite con 0,2 mL di perline di vetro. Utilizzando un battitore di perlina, macinare il tessuto polmonare asciutto per 15-30 s fino a formare una polvere fine.
- Aggiungere 0,8 mL di tampone di estrazione del DNA caldo per ogni provetta e agitare bene. Incubare ogni tubo per 15 min in un bagno di perlina di 65 ° C e poi vortice prima incubazione per altri 15 minuti. Dopo incubazione, vortice e poi Centrifugare le provette a 25.000 x g per 10 min.
- Etichetta 3 set di provette da 1,5 mL per ciascuno dei campioni e trasferire lo strato superiore del surnatante in un unico set di tubi. Usare cautela durante il pipettaggio fuori lo strato acquoso superiore per non disturbare lo strato intermedio.
- Aggiungere volume uguale del fenolo: cloroformio: alcool isoamilico e mescolare bene. Poi Centrifugare a 25.000 x g per 15 min prima di dispensazione fuori lo strato superiore nella seconda serie di tubi.
- Aggiungere un volume equivalente di cloroformio: isoamyl cloroformio, mescolare bene e centrifugare a 25.000 x g per 10 min attentamente pipetta fuori lo strato superiore nel terzo set di tubi.
- Aggiungere 500 µ l di isopropanolo e 50 µ l NaoAc (pH 5.2), mescolare bene e lasciare i campioni per incubare a temperatura ambiente per 10 min. Poi Centrifugare a 25.000 x g per 30 min e decantare il supernatante.
- Lavare il pellet di DNA con 750 µ l di etanolo 70% freddo di ghiaccio e centrifugare per 5 min a 25.000 x g. decantare l'etanolo e lasciare che asciugare il pellet all'aria nei tubi in un cappe per 40 min.
- Risospendere il pellet asciutto in 300 µ l di buffer di TE.
Nota: Il DNA può essere conservato a-20 ° C fino a quando non quantificato. - Quantificazione del DNA utilizzando uno spettrofotometro e preparare 100 µ l di 0,2 µ g / µ l di ciascun campione di DNA per l'analisi qPCR.
- Eseguire un qPCR standard in triplice copia come precedentemente descritto con 1 µ g di campione di DNA, primer di rDNA 18S fungine, e sonda imposta con un quencher modificate sonda (5'- / 56-FAM/AGC CAG CGG/ZEN/GCA CCC AAT G/3IABkFQ-3')12,13.
- Preparare una curva standard da DNA genomic a. fumigatus e calcolare la concentrazione di DNA fungoso in pg in ogni campione utilizzando la media reporter tinta i valori Ct.
- Tracciare il pg / µ g di DNA fungoso con errore standard della media. Esecuzione di uno studente t-analisi per determinare il significato del test.
Nota: come un meno tossico alternativa per l'estrazione di DNA fenolo-cloroformio utilizzato nel presente documento, colonna basato estrazione del DNA kit possono essere utilizzati.
Risultati
Figura 4 include un grafico di sopravvivenza di tipo selvaggio o topi eosinofilo-carenti con IA trattati con caspofungin. I risultati mostrano che topi eosinofilo-carenti hanno esibito la sopravvivenza aumentata rispetto ai topi wild-type (50% di mortalità rispetto al 100% di mortalità, rispettivamente). Figura 5 spettacoli rappresentante GMS colorazione da topi wild-type ed eosinofilo-carenti neutropenic con IA trattati o non trattati con caspofungin. Trattamento caspofungin provocato la relativa distanza fungina in eosinofilo-carenti, ma topi non wild-type (pannelli di destra), mentre entrambi i gruppi che non hanno ricevuto caspofungin erano simili (pannelli a sinistra). Figura 6 Mostra il confronto tra onere fungine in caspofungin-trattati e non trattati topi wild-type o eosinofilo-carenti di controllo, utilizzando entrambi qPCR di DNA fungoso (Figura 6A) e rappresentante GMS quantificazione di 4 (obiettivo 10x) campi ( Figura 6B). I risultati generati da entrambe le tecniche mostrano che caspofungin risultati di trattamento dell'onere fungine più significativa diminuiscono tra topi wild-type ed eosinofilo-carenti (Figura 6A). Tuttavia, in topi non trattati, solo quantificazione di GMS ha provocato una diminuzione significativa in topi che mancano di eosinofili (Figura 6B). Quando sono stati calcolati l'area media di sezioni di GMS-macchiate del intero-polmone (obiettivo 4x), le differenze erano simili a quelli ottenuti con 4 campi di rappresentante 10 X, anche se con meno significato statistico (Figura 6). La figura 7 Mostra sopravvivenza, immagini di macchiatura rappresentante GMS (4 10 X campi) e quantificazione degli oneri fungini con entrambi i metodi in γδ topi T cellula-carenti (TCRδKO) che sono stati trattati con caspofungin rispetto al controllo, non trattata di topi. Caspofungin trattamento ha migliorato la sopravvivenza (figura 7A) e fungine onere (figura 7B-D) in topi TCRδKO. Simile ai risultati di Figura 6, i risultati dell'onere fungine come misurato da qPCR (figura 7B) e rappresentante quantificazione GMS (Figura 7) erano comparabili.
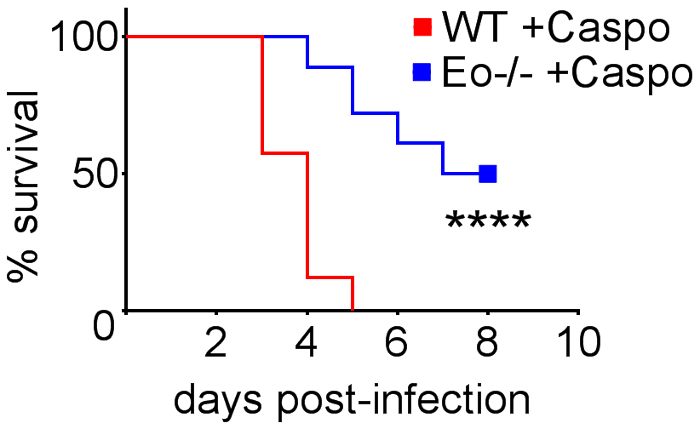
Figura 4: ha aumentato la sopravvivenza in topi eosinofilo-carenti (ΔdblGATA) con IA dopo il trattamento con caspofungin. 15-30 topi/gruppo. Riepilogo di esperimenti di 3-6. p < 0,0001. Questa figura è stata modificata da una precedente pubblicazione8. Clicca qui per visualizzare una versione più grande di questa figura.
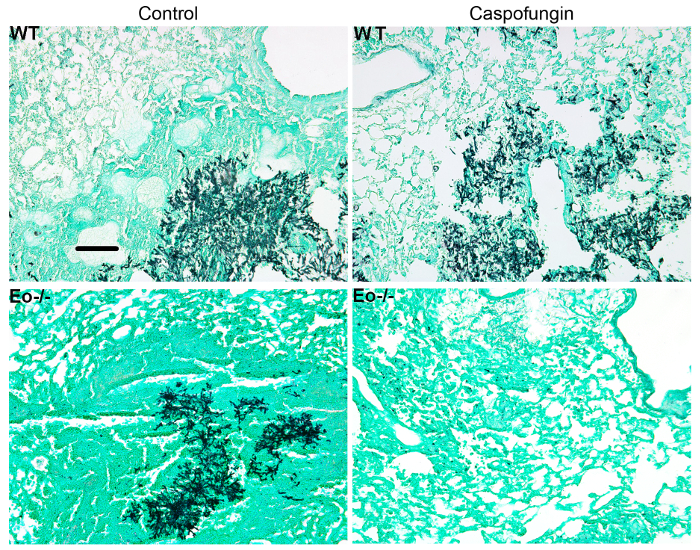
Figura 5: immagini rappresentative delle sezioni di GMS-macchiato del polmone da wild-type, eosinofilo carente e trattati con caspofungin o controllo non trattato topi con Ia Rappresentante di topi/gruppo di 3-4. Barra della scala è equivalente a 100 µm. Questa figura è stata modificata da una precedente pubblicazione8. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6: fungine onere nel selvaggio-tipo, topi eosinofilo-carenti con IA trattati o controllo trattati con caspofungin. (A) qPCR del DNA fungoso. 15-30 topi/gruppo, sintesi di 3-6 esperimenti. (B), GMS quantificazione delle sezioni istologiche, media % di GMS colorazione da 4 campi (obiettivo 10x). (C), GMS quantificazione, media % della sezione del polmone intero (obiettivo 4x). B e C sono una sintesi di 5 topi/gruppo. p < 0.05. p < 0,001. p < 0,0001. Questa figura è stata generata utilizzando dati che sono stati segnalati in una precedente pubblicazione8. Clicca qui per visualizzare una versione più grande di questa figura.
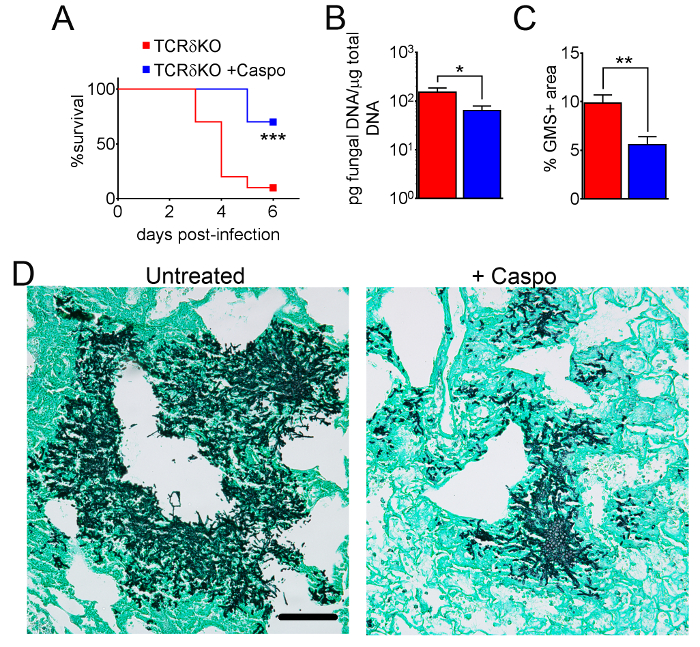
Figura 7: fatto diminuire la severità di IA in topi γδ cellule T-carenti dopo trattamento caspofungin. Sopravvivenza (A). (B) qPCR del DNA fungoso. (C), GMS quantificazione dell'istologia. (D) immagini rappresentative delle sezioni GMS-macchiate dalle cellule T γδ con IA, trattati o non trattati con caspofungin. A e B sono una sintesi di due esperimenti con topi/gruppo di 7-10. C e D sono una sintesi dei topi/gruppo 4. p < 0.05. p < 0.01. p < 0,001. Questa figura è stata modificata da una precedente pubblicazione8. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Lo scopo di questo articolo era di introdurre un metodo per la determinazione degli oneri fungine del polmone in topi con IA utilizzando le sezioni istologiche GMS-macchiato del polmone per analisi dell'immagine e la quantificazione. In questo studio, il trattamento con il farmaco antifungino β-glucano-sintesi-targeting caspofungin14 non ha migliorato la sopravvivenza o fungine onere in topi wild-type neutropenic con IA8. Tuttavia, in assenza di eosinofilo o T γδ cellule, sopravvivenza e fungine onere migliorata. I risultati del nostro studio ha anche dimostrano che risultati comparabili sono ottenibili con il fardello fungo GMS rispetto ai diffusi fungine DNA qPCR metodo6.
Ci sono diversi vantaggi ad utilizzare quantificazione fungine onere GMS. In primo luogo, il processo può utilizzare esistenti campioni istologici, potenzialmente riducendo così il numero degli esperimenti necessari per determinare le differenze significative. In secondo luogo, in questo studio, meno animali sono stati richiesti per la quantificazione di onere fungine GMS ottenere differenze significative rispetto ai qPCR di DNA fungoso (Figura 6, Figura 7). In terzo luogo, confronto di onere fungine in diversi isolati di qPCR può essere influenzato da differenze di isolare-dipendente ribosomal DNA copia numero15. Al contrario, quantificazione di GMS non risente numero di copia, come onere fungine è determinato dai relativi livelli di crescita fungina del polmone. Pertanto, l'uso di quantificazione di GMS per fungine onere riduce l'utilizzo di animali vertebrati e non richiede pre-determinazione del numero di copie di rDNA. Infine, oltre a modificare la morfologia fungina, caspofungin terapia aumenta la frammentazione fungine e quindi può aumentare artificialmente fungine onere quando misurato tramite isolamento delle unità formanti colonie dal polmone omogeneati12,16 ,17. Quantificazione di GMS di onere fungine evitando parecchie limitazioni inerenti con altri metodi comunemente utilizzati.
Tuttavia, i limiti di quantificazione GMS e/o di questo studio sono importanti da notare. In primo luogo, gli autori presuppone una distribuzione paragonabile di crescita ifale durante i polmoni di ogni gruppo sperimentale e pertanto utilizzata quantificazione da 4 rappresentante 10 x campi oggettivi come una misura rappresentativa per l'onere fungina nel polmone intero (Figura 6B). È possibile che in alcuni casi la distribuzione relativa, dimensioni e densità dei fuochi ifali sufficientemente sarebbe diverso affinché l'onere fungina appaiano diverso con questo metodo e l'equivalente dal metodo qPCR. Tuttavia, i nostri risultati ulteriori con quantificazione di sezione intero polmone utilizzando i 4 campi obiettivi X ha mostrato simile, anche se meno differenze statisticamente significative, tra gruppi (Figura 6). L'errore standard di questa quantificazione è stato aumentato con questa strategia, probabilmente a causa della risoluzione in diminuzione hyphal con 4x obiettivo e sfondo aumentato nei campi non ottimali. Di conseguenza, una quantificazione rappresentativa di meno campi a maggiore ingrandimento è preferita. In secondo luogo, solo una singola sezione centrale è stata utilizzata per ciascun campione. È possibile, basato su isolati fungosi o utilizzati ceppi di topi, che alcuni studi possono provocare una distribuzione irregolare di crescita ifale. In questi casi, devono essere quantificate sezioni aggiuntive in ogni blocco di paraffina per ottenere un onere più rappresentativo. In terzo luogo, negli esperimenti che inducono la notevole produzione di mucine (cioè, quantificare crescita fungina delle vie aeree in broncopolmonare allergica (ABPA) l'aspergillosi18 o fibrosi cistica (CF)18), reattività GMS con ricco di polisaccaridi mucine19 potrebbe produrre aspecifici GMS + risultati e inclinare così alcuni campioni in favore di maggiore carico fungina. Poiché solo il modello neutropenic di IA è stato usato in questo studio, è possibile che l'utilizzo di altri modelli immuni ai competenti o soppressive potrebbe risultati in risultati meno comparabili. Nonostante queste riserve, quantificazione di GMS fornisce una tecnica paragonabile per determinare l'onere fungine e relativo uso continuato negli studi supplementari può convalidare ulteriormente l'utilità di questo metodo come coerente, affidabile e conveniente.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo studio è stato sostenuto in parte da un Indiana University School di medicina Research Enhancement Grant e da NIH-NIAID 1R03AI122127-01. N.A. è stato in parte sostenuto durante questo periodo di una carriera in immunologia Fellowship dal Associazione americana di immunologi.
Materiali
| Name | Company | Catalog Number | Comments |
| Aspergillus fumigatus 293 Stock Solution | Fungal Genetics Stock Center | FGSC #A1100 | |
| HyPure Cell Culture Grade Water | Thermo Fisher Scientific | SH30529.03 | |
| Malt Extract | MP Biomedicals | 2155315 | White Powder |
| BD BBL Acidicase Dehydrated Culture Media: Peptone | Fisher Scientific | L11843 | |
| Dextrose (D-Glucose) Anhydrous (Granular Powder/Certified ACS) | Fisher Scientific | D16-3 | |
| Fisher BioReagents Agar, Powder / Flakes, Fisher BioReagents | Fisher Scientific | BP1423-500 | |
| Auto Dry Cabinet | Shanghai Hasuc Instrument Manufacture Co.,LTD | HSFC160FD | |
| 1300 Series Class II, Type A2 Biological Safety Cabinet | Thermo Fisher Scientific | 1375 | |
| 0.5 mm Glass Beads | BioSpec Products | 11079105 | |
| 15 ml Conical Tubes | Thermo Fisher Scientific | 339650 | |
| Hemacytometer | Fisher Scientific | # 0267110 | |
| Leica Model DME Microscope | Leica | 13595XXX | |
| Dulbecco's Phosphate Buffered Saline | Sigma-Aldrich | D8662-500 ml | |
| 1.5 ml tubes | Fisher Scientific | # 05-408-129 | |
| BD Precisionglide syringe needles, gauge 27, L 1/2 in. | Sigma-Aldrich | Z192384 | |
| Anti-mouse-Ly-6G antibody | BioXCell | BP0075-1 | Clone 1A8 |
| Caspofungin diacetate | Sigma-Aldrich | SML0425 | |
| Isoflurane | Henry Schein Animal Health | 1169567762 | |
| Non-Rebreathing Table Top Veterinary Anesthesia Machine | Supera Anesthesia Innovations | M3000 | |
| Pureline Oxygen Concentrator | Supera Anesthesia Innovations | OC8000 | |
| Slant Board Restraint | Indiana State University Facilities Management | Custom made | |
| Gilson PIPETMAN Classic 200 ml Pipets | Fisher Scientific | F123601G | |
| Pentobarbital Sodium (Fatal-Plus) | Vortech Pharmaceuticals | 0298-9373-68 | |
| General-Purpose Broad-Tipped Forceps | Fisher Scientific | 10-300 | |
| Fisherbrand Standard Dissecting Scissors | Fisher Scientific | 08-951-20 | |
| Fisherbrand General-Purpose Curved Forceps | Fisher Scientific | 10-275 | |
| Ethyl Alcohol-200 Proof | PHARMCO-AAPER | 111000200 | |
| Fisherbrand Absorbent Underpads | Fisher Scientific | 14-206-63 | |
| BD Precisionglide syringe needles, gauge 25, L 1 in. | Fisher Scientific | Z192406 | |
| All-Plastic Norm-Ject Syringes | Fisher Scientific | 14-817-30 | |
| IV CATH ANGIOCATH 22GX1GIN 50B | Fisher Scientific | NC9742754 | |
| Formalin Solution, neutral buffered, 10% | Sigma-Aldrich | HT501128-4L | |
| 50 ml Conical Tubes | Thermo Fisher Scientific | 339652 | |
| Thermo Scientific Shandon Embedding Cassettes II in Tube Packs | Fisher Scientific | B1000729 | |
| Fisherfinest Histoplast Paraffin Wax | Fisher Scientific | 22-900-700 | |
| Disposable Base Molds | Fisher Scientific | 22-363-554 | |
| Reichert Jung Histocut 820 Microtome | Labequip.com | 31930 | |
| Water Bath | Precision Scientific | 66630-23 | |
| CO2 Incubator | Fisher Scientific | 116875H | |
| Silver Stain (Modified GMS) Kit | Sigma-Aldrich | HT100A-1KT | |
| Fast Green FCF | Fisher Scientific | AC410530250 | |
| Frosted Microscope Slides | Fisher Scientific | 12-550-343 | |
| Fisherfinest Superslip Cover Glass | Fisher Scientific | 12-545-89 | |
| Cytoseal XYL | Fisher Scientific | 22-050-262 | |
| Olympus Provis AX70 Microscope | Olympus | OLYMPUS-AX70 | |
| U-PHOTO Universal Photo System | Olympus | OLYMPUS-U-PHOTO | |
| U-MCB-2 MULTI CONTROL BOX | Olympus | OLYMPUS-U-MCB-2 | |
| U-PS POWER SUPPLY UNIT | Olympus | OLYMPUS-U-PS | |
| FreeZone 1 Liter Benchtop Freeze Dry System | LABCONCO | 7740020 | |
| Maxima C Plus Vacuum Pump | Fisher Scientific | 01-257-80 | Displacement- 6.1 cfm |
| 1-Butanol | Fisher Scientific | A383-4 | |
| AMRESCO PHENOL-CHLORFORM-OSOAMYL 100ML DFS | Fisher Scientific | NC9573988 | |
| Free-Standing Microcentrifuge Tubes with Screw Caps | Fisher Scientific | # 02-682-557 | |
| Mini-Beadbeater-24 | BioSpec Products | 112011 | |
| BioSpec ProductsSupplier Diversity Partner 2.3 MM ZIRCONIA BEADS | Fisher Scientific | NC0451999 | |
| Tris Base (White Crystals or Crystalline Powder/Molecular Biology) | Fisher Scientific | BP152-1 | |
| Hydrochloric Acid, Certified ACS Plus | Fisher Scientific | A144S | |
| Ethylenediaminetetraacetic Acid, Disodium Salt Dihydrate | Fisher Scientific | S311 | |
| Sodium Dodecyl Sulfate (SDS), White Powder, Electrophoresis | Fisher Scientific | BP166 | |
| Chloroform/isoamyl alcohol 24:1 | Fisher Scientific | AC327155000 | |
| Biotek Epoch Microplate Spectrophotometer | Fisher Scientific | 11-120-570 | |
| Thermo Scientific™ ABsolute Blue qPCR Mixes | Fisher Scientific | AB4137A | |
| Hybridization Probe, 5′-FAM-AGCCAGCGGCCCGCAAATG-TAMRA-3′ | Integrated DNA Technologies | N/A | |
| Sense Amplification Primer, 5′-GGCCCTTAAATAGCCCGGT-3′ | Integrated DNA Technologies | N/A | |
| Antisense Amplification Primer, 5′-TGAGCCGATAGTCCCCCTAA-3′ | Integrated DNA Technologies | N/A | |
| Applied Biosystems™ MicroAmp™ Optical 8-Tube Strip, 0.2mL | Fisher Scientific | 43-165-67 | |
| Thermo Scientific Domed and Flat PCR Cap Strips | Fisher Scientific | AB-0386 | |
| Mx3005P QPCR System, 110 Volt | Agilent | 401443 | Stratagene is now owned by Agilent |
| BALB/c mice | The Jackson Laboratory | 000651 | |
| C57BL/6 (B6) mice | The Jackson Laboratory | 000664 | |
| Eosinophil-deficient (ΔdblGATA, BALB/c background) mice | The Jackson Laboratory | 005653 | |
| γδ T cell-deficient (TCRδ-/-, B6 background) mice | The Jackson Laboratory | 002120 | |
| ImageJ Software | National Institutes of Health | N/A | https://imagej.nih.gov/ij/ |
| Spot Advanced Software | Spot Imaging | SPOT53A | http://www.spotimaging.com/software/spot-advanced/ |
| GraphPad Prism 6 | GraphPad Software | N/A | https://www.graphpad.com/scientific-software/prism/ |
| Fisherbrand Absorbent Underpads | Fisher Scientific | 14-206-62 | |
| Step 4.5 | http://www.bdbiosciences.com/sg/resources/protocols/paraffin_sections.jsp ; https://www-jove-com.remotexs.ntu.edu.sg/science-education/5039 ; http://www.sigmaaldrich.com/content/dam/sigma-aldrich/docs/Sigma/General_Information/1/ht100.pdf |
Riferimenti
- Hohl, T. M., Feldmesser, M. Aspergillus fumigatus: principles of pathogenesis and host defense. Eukaryot Cell. 6 (11), 1953-1963 (2007).
- Kwon-Chung, K. J., Sugui, J. A. Aspergillus fumigatus--what makes the species a ubiquitous human fungal pathogen?. PLoS Pathog. 9 (12), e1003743 (2013).
- Ostrosky-Zeichner, L., Casadevall, A., Galgiani, J. N., Odds, F. C., Rex, J. H. An insight into the antifungal pipeline: selected new molecules and beyond. Nat Rev Drug Discov. 9 (9), 719-727 (2010).
- Desoubeaux, G., Cray, C. Rodent Models of Invasive Aspergillosis due to Aspergillus fumigatus: Still a Long Path toward Standardization. Front Microbiol. 8, 841 (2017).
- Hohl, T. M. Overview of vertebrate animal models of fungal infection. J Immunol Methods. 410, 100-112 (2014).
- Sheppard, D. C., et al. Comparison of three methodologies for the determination of pulmonary fungal burden in experimental murine aspergillosis. Clin Microbiol Infect. 12 (4), 376-380 (2006).
- Patterson, T. F. The future of animal models of invasive aspergillosis. Med Mycol. 43 Suppl 1, S115-S119 (2005).
- Amarsaikhan, N., et al. Caspofungin Increases Fungal Chitin and Eosinophil and gammadelta T Cell-Dependent Pathology in Invasive Aspergillosis. J Immunol. 199 (2), 624-632 (2017).
- Templeton, S. P., Buskirk, A. D., Law, B., Green, B. J., Beezhold, D. H. Role of germination in murine airway CD8+ T-cell responses to Aspergillus conidia. PLoS One. 6 (4), e18777 (2011).
- Brunel, S. F., et al. Live Imaging of Antifungal Activity by Human Primary Neutrophils and Monocytes in Response to A. fumigatus. J Vis Exp. (122), (2017).
- Rao, G. V., et al. Efficacy of a technique for exposing the mouse lung to particles aspirated from the pharynx. J Toxicol Environ Health A. 66 (15), 1441-1452 (2003).
- Bowman, J. C., et al. Quantitative PCR assay to measure Aspergillus fumigatus burden in a murine model of disseminated aspergillosis: demonstration of efficacy of caspofungin acetate. Antimicrob Agents Chemother. 45 (12), 3474-3481 (2001).
- Li, H., et al. The small GTPase RacA mediates intracellular reactive oxygen species production, polarized growth, and virulence in the human fungal pathogen Aspergillus fumigatus. Eukaryot Cell. 10 (2), 174-186 (2011).
- Sucher, A. J., Chahine, E. B., Balcer, H. E. Echinocandins: the newest class of antifungals. Ann Pharmacother. 43 (10), 1647-1657 (2009).
- Herrera, M. L., Vallor, A. C., Gelfond, J. A., Patterson, T. F., Wickes, B. L. Strain-dependent variation in 18S ribosomal DNA Copy numbers in Aspergillus fumigatus. J Clin Microbiol. 47 (5), 1325-1332 (2009).
- Kirkpatrick, W. R., Perea, S., Coco, B. J., Patterson, T. F. Efficacy of caspofungin alone and in combination with voriconazole in a Guinea pig model of invasive aspergillosis. Antimicrob Agents Chemother. 46 (8), 2564-2568 (2002).
- Petraitiene, R., et al. Antifungal efficacy of caspofungin (MK-0991) in experimental pulmonary aspergillosis in persistently neutropenic rabbits: pharmacokinetics, drug disposition, and relationship to galactomannan antigenemia. Antimicrob Agents Chemother. 46 (1), 12-23 (2002).
- Cowley, A. C., Thornton, D. J., Denning, D. W., Horsley, A. Aspergillosis and the role of mucins in cystic fibrosis. Pediatr Pulmonol. 52 (4), 548-555 (2017).
- Taylor, M. J., et al. Detection of fungal organisms in eosinophilic mucin using a fluorescein-labeled chitin-specific binding protein. Otolaryngol Head Neck Surg. 127 (5), 377-383 (2002).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon