Method Article
Alta risoluzione Volume Imaging di neuroni nell'esclusione di uso di fluorescenza metodo e dedicato dispositivi microfluidici
In questo articolo
Riepilogo
Il volume è un parametro importante per quanto riguarda le caratteristiche fisiologiche e patologiche delle cellule. Descriviamo un metodo di esclusione fluorescente che permette di pieno-campo misura di volume di un neurone in vitro con sub-micrometrica risoluzione assiale richiesto per l'analisi dei neuriti e strutture dinamiche implicite nella crescita neuronale.
Abstract
Il volume è un parametro importante per quanto riguarda le caratteristiche fisiologiche e patologiche dei neuroni alle diverse scale temporali. I neuroni sono cellule piuttosto uniche per quanto riguarda la loro estese morfologie ramificate e conseguenza genera diverse sfide metodologiche per la misurazione del volume. Nel caso particolare di in vitro la crescita neuronale, la metodologia scelta dovrebbe includere sub-micrometrica risoluzione assiale combinato con osservazione di pieno campo su scale temporali da minuti a ore o giorni. A differenza di altri metodi come la cellula forma ricostruzione usando la formazione immagine confocal, misure basate su elettricamente o microscopia a forza atomica, il metodo di esclusione di fluorescenza (FXm) recentemente sviluppato ha il potenziale per soddisfare queste sfide. Tuttavia, pur essendo semplice nel suo principio, l'implementazione di un FXm ad alta risoluzione per i neuroni richiede regolazioni multiple e una metodologia dedicata. Presentiamo qui un metodo basato sulla combinazione di esclusione di fluorescenza, dispositivi microfluidici multi-compartimenti bassa rugosità e infine micropatterning per ottenere misurazioni in vitro del volume locale di un neurone. L'alta risoluzione fornita dal dispositivo ci ha permesso di misurare il volume locale di processi neuronali (neuriti) e il volume di alcune strutture specifici coinvolti nella crescita neuronale, come coni di crescita (GCs).
Introduzione
La conoscenza precisa del volume cellulare ha attirato l'attenzione aumentata negli ultimi anni, guidato dall'emissione di omeostasi di dimensione delle cellule in microrganismi unicellulari 1 e, più in generale, in cellule mitotiche 2. Tuttavia, la questione del volume delle cellule è pertinente anche per cellule post-mitotiche, per il quale i neuroni costituiscono un esempio paradigmatico.
Volume è infatti una firma importante degli eventi fisiologici e patologici alle diverse scale e intervalli di tempo in vita neuronale, da transitoria deformazione assonale associata alla attività elettrica (scala di millisecondo) 3 per l'irreversibile un neurone gonfiore che si verificano durante la fase asintomatica di neurodegenerative malattie (negli anni in esseri umani) 4. Tuttavia, la più grande variazione di volume si verifica in una scala di tempo intermedio di giorni o settimane (a seconda del microrganismo considerato) durante lo sviluppo neuronale. L'estesa e complessa morfologia dei neuroni solleva problemi multipli, tra cui la regolazione della dimensione della cella. Diametro e lunghezza assonale sono infatti strettamente regolato in vivo, con valori specifici per ogni tipo di un neurone 5,6.
Queste questioni, complessi da affrontare in vivo, anche possono essere affrontate in modo semplificato in vitro. A tale scopo, un metodo dedicato alla misurazione del volume veloce basta seguire il tasso di crescita (cioè in una scala di tempo di minuti) e compatibile con l'osservazione nel corso di ore o giorni è richiesto. Diversi metodi sono stati sviluppati nel corso degli anni per fornire un accesso diretto o indiretto a volume cellulare in vitro. Ricostruzione delle cellule da imaging confocale è uno di loro, ma questo metodo implica d'etichettatura e ripetute esposizioni alla luce pur mostrando una limitata risoluzione assiale di circa 500 nm 7. Si noti che questi due ultimi inconvenienti sono parzialmente superati mediante un metodo più recente e sofisticato denominato reticolo microscopia luce-foglio 8. Microscopia a forza atomica è stato usato 9 ma questo metodo di scansione è di essenza lento e noioso. Inoltre, il contatto fisico che richiede con la cella potrebbe interferire con la misurazione considerando l'estrema morbidezza di neuroni 10. Metodo indiretto tramite impedenza o risonanza sono state impiegate per cellulari differenti tipi di 11, ma sono insufficienti per esteso adesive cellule come i neuroni.
Uno dei metodi più promettenti si basa sulla misura del volume escluso delle cellule in una camera stretta, piena di una tintura fluorescente. Il metodo di esclusione di fluorescenza (FXm) è semplice nel suo principio in quanto non richiede alcuna etichettatura ed è adatto per l'imaging ottico veloce, a lungo termine delle popolazioni di cellule con una risoluzione assiale potenzialmente Sub-ottica. Più precisamente, la risoluzione in z dipende l'intensità della fluorescenza massima nella camera di coltura (cioè nella regione privo di cellule) divisa per la gamma dinamica della fotocamera, anche se diverse fonti di rumore limitano questa risoluzione definitiva. Questo metodo è stato molto potente per seguire il volume di migrazione delle cellule aderenti 12 o per studiare la variazione di volume durante la mitosi delle cellule di mammiferi, come accuratamente descritto in 13. Tuttavia, i neuroni costituiscano una sfida metodologica per FXm considerando la loro ampia ramificazione nei processi sub-micrometrici.
Presentiamo qui un metodo che conduce alla realizzazione di strutture liscia FXm alloggiamenti per accedere con alta precisione il volume e l'altezza di un neurone filiali e strutture dinamiche coinvolte nella crescita neuronale come coni di crescita.
Chambers dovrebbe avere altezze simili rispetto all'oggetto da misurare al fine di ottimizzare la risoluzione assiale. Pertanto, abbiamo progettato diversi dispositivi FXm caratterizzati da sezioni di misura centrale di tre diverse altezze. Il più sottile (3 µm in altezza) è dedicato alla misurazione del neurite: questa bassa altezza esclude soma, che rimangono nella camera intermedia alta, vicino 15 µm. Camere centrali più spesse (10 e 12 µm) sono sufficientemente elevate per seguire la crescita di cellule intere. Il dispositivo include anche due serbatoi situati su entrambi i lati della camera centrale. Quattro fori di iniezione (IH) così vengono implementate e sono designati come segue: l'ingresso e l'uscita servono ad introdurre le sospensioni cellulari nel chip, mentre gli altri due alimentare i serbatoi.
Abbiamo prima calibrazione fabbricato coprioggetti per le misurazioni di altezza con photoresist strutture geometriche conosciute. Abbiamo quindi abbiamo ripreso neuroni crescenti gratis, ma i neuroni anche morfologicamente vincolati in microfantasie di adesione.
Protocollo
Lo studio è stato effettuato conformemente agli orientamenti della Comunità europea sull'uso degli animali da laboratorio e manutenzione: 86/609/CEE. Lo scopo di ricerca e il protocollo sono descritti nel progetto etico allegato di ERCadg violoncello, che è stato approvato ed è regolarmente esaminato dalla ERCEA. Institut Curie animal facility ha ricevuto licenza #C75-05-18, 24/04/2012, riportando al Comité d'Ethique en matière d' ' expérimentation animale Paris Centre et Sud (numero di registrazione nazionale: #59).
1. fabbricazione della muffa
Nota: Lo stampo comprende camere centrali e intermedi collegate ad un ingresso e un'uscita, plus due serbatoi (e insenature) situato su entrambi i lati della camera centrale.
- Utilizzare una pinzetta piatta per manipolare i wafer di silicio del diametro di 51 mm e trasferirli da un luogo a altro.
- Muffa del silicone camera centrale
- Riempire una pipetta di plastica di 2 mL con photoresist positivo. Posizionare la pipetta al centro di un wafer di silicio del diametro di 51 mm e premere sul suo serbatoio fino a coprire circa il 75% della superficie del wafer con il photoresist. Spin-cappotto a 3000 rpm per 30 s.
- Trasferire la cialda da spin-spalmatrice a una piastra riscaldante, con una temperatura superficiale di 100 ° C, per 50 s.
- Trasferire la cialda dalla piastra riscaldante al titolare di substrato (chuck) di aligner maschera. Esporre attraverso la "maschera DRIE" (Vedi file supplementari "masks_neuron_volume_chips.tiff" e "masks_neuron_volume_chips.dxf").
- Rimuovere la cialda dal mandrino quindi fare immersioni in un bicchiere da 100 mL cristallizzare il piatto contenente lo sviluppatore (diluizione 1:4 in acqua deionizzata). Rilasciare la cialda e mantenerla per 1 min, rimestando continuamente e delicatamente il piatto di cristallizzazione.
Nota: Per salvare i reagenti, riempire il piatto di cristallizzazione a 1 cm di altezza al massimo. - Riprendere la cialda da parte dello sviluppatore e immergerlo in un piatto di cristallizzazione di 100 mL con acqua deionizzata per circa 10 s.Then posto la cialda su una carta assorbente e asciugare con azoto pressurizzato usando una pistola di soffiaggio di aria.
- Posizionare la cialda per 50 s su una piastra calda con una temperatura superficiale di 115 ° C.
- Eseguire profondo-Reactive Ion Etching (DRIE) con i seguenti parametri (ulteriori informazioni sulla tecnica DRIE possono essere trovati in riferimenti 14 e 15); Passo di passivazione: 50 sccm di C4F8, ha supporto di flusso 10 sccm, CP 10 W, Inductively Coupled Plasma (ICP) 1500 W, totale pressione 24 mbar, temperatura 18 ° C; Passo di incisione: 100 sccm di SF6, ha supporto di flusso 10 sccm, CP 11 W, ICP 1500 W, totale pressione 24 mbar, temperatura 18 ° C; Tempo di passivazione: 4 s; Acquaforte di tempo: 7 s; Durata del processo totale: in genere 5 min per circa 10 µm inciso profondità.
- Sciogliere il photoresist da immersione il substrato in un piatto di cristallizzazione riempito con acetone.
- Immergere la cialda in una soluzione piranha (2/3 di H2O2 (30%) e 1/3 di H2SO4 (95 %)) per 5 min.
Attenzione: Aggiungere sempre H2O2 prima e quindi H2così4 e sciacquare almeno 3 volte acqua deionizzata dopo la pulizia.
- Fabbricare stampi corrispondenti agli alloggiamenti intermedi attenendosi alla procedura 1.2.1 a 1.2.11 seguendo i parametri indicati nelle tabelle 1-3.
Nota: Ogni tabella corrisponde a un determinato dispositivo caratterizzato da una specifica altezza della camera centrale di osservazione.
Nota: Eseguire l'intero processo utilizzando crescente i photoresists altezze (maschere 1 a 3 per ogni dispositivo, vedere file supplementari "masks_neuron_volume_chips.tiff" e "masks_neuron_volume_chips.dxf").- Posizionare il wafer di silicio inciso sul titolare del substrato di spin coater.
- Verifica che la spin coater funzioni correttamente verificando che l'aspirazione del substrato è efficace (il substrato dovrebbe spin e rimanere in posizione durante la velocità di rotazione nominale).
- La viscosità del SU-8 aumenta con l'altezza della gamma spessore mirati. Sempre utilizzare bottiglie di 20-30 mL per memorizzare i photoresists SU-8 e versatelo sul wafer prima di rivestimento per rotazione (SU-8 potrebbe essere troppo viscoso ad essere manipolato con una pipetta di plastica).
- Versare la resina epossidica-tipo negativo photoresist SU-8 al centro del substrato fino a coprire circa il 75% dei wafer di silicio, poi spin-cappotto utilizzando i parametri indicati nella riga "Spincoating" delle tabelle.
- Posto il wafer rivestito su una piastra per la durata e la temperatura indicata nella "Soft cuoce" riga.
- Montare la cialda acidata e l'adeguata "maschera di SU-8" aligner maschera.
- Allineare la cialda acidata con la maschera utilizzando le croci di allineamento dedicato (dimensione tipica di queste croci: 50 µm × 150 µm) progettato su ogni maschera.
Nota: Due croci su ogni lato del dispositivo sono sufficiente (uno in basso a sinistra) e uno in alto a destra. - Expose utilizzando la-linea di aligner maschera (lunghezza d'onda 365 nm) con la dose di UV appropriata, come indicato nella riga "Energia di esposizione".
Nota: Il tempo di esposizione è calcolato dividendo la potenza effettiva della lampada UV la specifica di energia E di esposizione per ogni photoresist, modulata dall'assorbimento della maschera: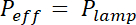 x (1 - assorbimentomaschera). L'assorbimento è di circa il 20% per le maschere di flessibile e trascurabile per maschera di cromo duro.
x (1 - assorbimentomaschera). L'assorbimento è di circa il 20% per le maschere di flessibile e trascurabile per maschera di cromo duro. - Posizionare la cialda rivestita su una piastra per la durata e la temperatura indicata nella riga "Post cuoce".
- Preparare due 100 mL vetro-cristallizzazione piatti, uno contenente lo sviluppatore, l'altro vuoto. Immersione della cialda in developer per la durata indicata nella riga "Sviluppo". Agitare delicatamente il piatto cristallizzazione lungo tutto lo sviluppo
- Cospargere la cialda con isopropanolo sopra il piatto di cristallizzazione vuoto per circa 5 s. Infine, inserire la cialda su una carta assorbente e asciugare con azoto pressurizzato usando una pistola di soffiaggio di aria.
- Posto il wafer rivestito su una piastra per la durata e la temperatura indicato in "Hard bake" riga (opzionale).
Nota: Questo passaggio è utile per evitare crepe nel photoresist e offrono una superficie piana omogenea per i passaggi successivi. - Ripetere i passaggi 1.3.1 - 1.3.10 per completare i processi elencati nella tabella 1-3.
- Dopo l'ultimo passo della fotolitografia, silanizzare il master finale di stampo che consiste di 3 strati di fotoresist negativi inviando due 100 µ l gocce di Trichloro(1H,1H,2H,2H-perfluoro-octyl) Silano su ciascun lato del master in una capsula di Petri 100mm. Il disco di Petri con una pellicola di plastica paraffina ed incubare 20 min a temperatura ambiente (TA).
Nota: Lo stampo master è pronto e può essere utilizzato più volte.
- Posizionare il wafer di silicio inciso sul titolare del substrato di spin coater.
2. fabbricazione di chip di PDMS
- Versare 90 g di polimero organico a base di silicio (polidimetilsilossano: PDMS) in un bicchiere di plastica da 100 mL. Aggiungere 10 g di suo agente indurente (01:10 in peso). Mescolare il composto utilizzando una pipetta di plastica per 2-3 min.
Nota: Mescolare 90 g di PDMS con 10 g di agente indurente per la realizzazione di 6 gettoni. - Mettere il composto all'interno di un essiccatore sotto vuoto e la pompa per circa 30 min per rimuovere le bolle d'aria intrappolate nel PDMS.
- Posizionare lo stampo master in una capsula di Petri P100 e versare 15 mL della miscela sullo stampo usando una siringa.
Nota: 15ml conduce ad un'altezza di chip totale di 1,5 mm. - Posizionare la struttura di PDMS-muffa all'interno di un essiccatore sotto vuoto e la pompa fino a quando bolle d'aria più scoppio alla superficie del PDMS.
Nota: Questo passaggio richiede circa 30 min. - Spingere la cialda nella parte inferiore del piatto Petri utilizzando una punta di cono per evitare volume PDMS morto sotto il wafer. Posizionare il dispositivo di PDMS-stampo in forno a 70 ° C per almeno 2 h.
- Sformatura del blocco PDMS sotto una cappa chimica usando una spatola piatta in acciaio inox e in isopropanolo versando sul chip. Posizionarlo sulla panchina della cappa in acciaio inox.
- Tagliare intorno il wafer di silicio e sformatura la replica PDMS utilizzando un bisturi.
- Tagliare intorno (lasciare un 2 mm margine) il chip usando un bisturi o una lama di rasoio.
- Punch insenature premendo saldamente il puncher del diametro di 1,5 mm e azionare per tagliare e scolpire il foro di ingresso. Fare lo stesso alle quattro zone dedicate del chip dove sarà iniettato liquido.
- Pulire il chip di attaccare e peeling nastro adesivo sul lato microstrutturato. Cospargere di isopropanolo su entrambi i lati. Quindi asciugare il chip con azoto pressurizzato usando una pistola di soffiaggio di aria.
3. fabbricazione di fantasia coprioggetti (24 × 24 mm2)
Nota: Manipolare le lamelle con Pinzette curve.
- Modelli di poli-ornitina (PLO)
- Applicare una O2 pulizia al plasma su vetrini coprioggetti. Parametri del plasma: pressione di pompaggio: 0,25 mbar; O2 alimentazione Durata: 3 min; flusso del gas: 10 sccm; deviazione massima: ± 5 sccm; durata del plasma: 3 min; pressione impostata: 0.36 mbar; deviazione massima: ± 0,10 mbar; regolare la potenza: 50 W; deviazione massima: 5%; durata di ventilazione: 45 s.
- Mix 484 µ l di acido acetico e 56 µ l di 3-methacryloxypropyl-trimetossisilano, completare con etanolo assoluto per ottenere un volume totale di 15 mL.
- Utilizzando un cono di punta di 1 mL, mettere una goccia di 500 µ l di questa soluzione su ogni vetrino coprioggetti, attendere 2-3 min a secco utilizzando un camera bianca microfibra tergicristallo.
Nota: Lamelle di vetro silanizzata possono essere conservate fino a 1 mese a temperatura ambiente all'interno di scatole di plastica sigillato con una pellicola di plastica paraffina. - Posizionare ogni vetrino coprioggetti su una macchina a rotazione che si trova in un ambiente della stanza pulita. Mettere una goccia di un photoresist positivo che coprono circa il 75% del vetrino coprioggetti (circa 500 µ l) e spin-cappotto a 4000 rpm per 30 s a raggiungere uno spessore finale di 0,45 µm.
- Posizionare i vetrini coprioggetto per 1 min su una piastra calda con una temperatura superficiale di 115 ° C.
- Utilizzando un aligner maschera, esporre ogni vetrino coprioggetti alla lunghezza d'onda di 435 nm (linea G) attraverso la maschera dedicata secondo i parametri fabricant (dose UV circa 50-60 mJ.cm–1)
- Preparare 2 piatti cristallizzazione di vetro, uno contenente lo sviluppatore (Nessuna diluizione), l'altra contenente acqua deionizzata.
- Immersioni uno ogni vetrino coprioggetti in developer per 1 min mescolando continuamente e delicatamente il piatto di cristallizzazione. Immergere la cialda in acqua deionizzata per circa 5 s. posto il wafer su una carta assorbente, quindi asciugare con azoto pressurizzato usando una pistola di soffiaggio aria.
- Applicare un plasma di2 attivazione O con gli stessi parametri come in 3.1.1.
- Sotto il cofano, deporre quattro 170 µ l gocce di una soluzione di 100 µ g/mL PLO per P100 di Petri. Mettere la faccia fantasia di lamelle su ognuna di queste gocce. Sigillare la capsula di Petri con una pellicola di plastica paraffina per evitare la disidratazione. Incubare per una notte a TA.
Nota: La soluzione di PLO deve rimanere agganciata a lamelle per capillarità. - Preparare quattro destinatari (in genere di P60 Petri), tre di loro riempire con PBS e la quarta con acqua deionizzata. Preparare due piastre di vetro cristallizzazione di etanolo puro.
- Stipulare ogni vetrino coprioggetti dai piatti Petri, immergerlo nel primo bagno PBS per 10-15 s, evacuare il liquido su un tessuto assorbente mettendo il coprioggetto verticalmente sul lato, inserirlo con per esempio il modellato verso l'alto all'interno del bagno di etanolo.
Nota: Una volta completata la dissoluzione del photoresist, diventa difficile individuare il lato disegnato del coprivetrino. Pertanto, è importante in questa fase per rintracciare la sua posizione. - Posizionare l'etanolo cristallizzare il piatto all'interno di un sonicatore bagno ad ultrasuoni (120 W / 35 kHz) e lasciare che il photoresist essere dissolto per 3 min.
Nota: Modificare il bagno di etanolo ogni 4 vetrini coprioggetti per limitare la diluizione di PBS che potrebbe compromettere la dissoluzione del photoresist. - Estrarre il vetrino coprioggetto dal bagno di etanolo, poi tuffarsi esso parecchie volte il secondo bagno di PBS. Controllare l'aspetto di superficie ripetutamente finché la superficie grassa-come che risultati da pellicola liquida restante di etanolo scompare.
- Immergere per 5-10 s il coprioggetto in terzo bagno di PBS. Quindi immediatamente trasferire a bagno d'acqua deionizzata. Posizionare il vetrino coprioggetto su una carta assorbente e asciugare con azoto pressurizzato usando una pistola di soffiaggio di aria.
Nota: L'ultimo risciacquo in acqua deionizzata viene utilizzato per evitare la formazione di cristalli di PBS durante la fase di asciugatura.
- Photoresist strutture per la calibrazione di altezza
- Effettuare solo operazioni 3.1.4. a 3.1.8. utilizzando la maschera dedicata (maschera "Photoresist stripes", vedi file supplementari "Mask_Photoresist-stripes.dxf").
4. chip di montaggio e finali di esecuzione
- Chip di montaggio su piatti di vetro inferiore
- Mettere sia il chip PDMS e il piatto di vetro su cui verrà essere incollato nella camera del plasma per l'attivazione superficiale. Parametri: pompaggio giù pressione: 0,25 mbar; O2 alimentazione Durata: 3 min; flusso del gas: 10 sccm; deviazione massima: ± 5 sccm; durata del plasma: 30 s; pressione impostata: 0.40 mbar; deviazione massima: ± 0,10 mbar; regolare la potenza: 50 W; deviazione massima: 5%; durata di ventilazione: 45 s.
- Mettere delicatamente il chip PDMS attivato a contatto con il vetrino coprioggetti e applicare delicatamente la pressione sui bordi del chip per legare il chip per il vetrino coprioggetti. Per aumentare la forza di legame, posizionare il dispositivo in forno a 70 ° C per 5/10 minuti.
Nota: Non premere sulle parti contenenti pilastri, potrebbe comprimere troppo sotto pressione. - Sotto il cofano (cioè a RT) ed entro 30 min dopo l'incollaggio, posizionare un cono di punta 10-µ l riempito con una soluzione di 100 µ g/mL PLO presso IHs, quindi iniettare il liquido. Regolare il volume al fine di formare una goccia nella parte superiore di ogni IHs. Quindi, utilizzando un cono di punta di 1 mL, aggiungere PBS nel piatto Petri tutto intorno il chip.
- Lasciate che il chip a RT con un minimo di tempo di incubazione di 2 h. Per incubazione overnight, sigillo di Petri utilizzando una pellicola di plastica paraffina per evitare la disidratazione.
Nota: Il liquido non dovesse fuoriuscire fuori il chip altrimenti che il chip deve essere scartato. - Premere leggermente un cono di punta 10-µ l in ogni IH e aspirare l'eccesso di liquido. Quindi aderire completamente al cono all'interno dell'outlet e redigere il liquido rimanente.
- Sostituire PLO laminin seguendo le istruzioni riportate nei passaggi 4.1.5 (svuotamento) e 4.1.3 (riempimento). Incubare per 1h a RT.
- Sostituire laminin dal terreno di coltura seguente le istruzioni riportate nei passaggi 4.1.5 (svuotamento) e 4.1.3 (riempimento). Composizione del terreno di coltura: MEM 81,8%; Piruvato di sodio 100 mM 1%; Glutamax 200 mM 1%; Cavallo del siero 5%; B27 supplemento 2%, N2 supplemento 1%, gentamicina 0,2%; filtrare la soluzione utilizzando un filtro di 220 nm. Utilizzando un cono di punta di 1 mL, sostituire anche il PBS che circondano il chip da questo mezzo.
- Mettere il chip in incubatrice regolamentato a 37 ° C e 5% di CO2 per almeno 5 ore (o durante la notte) prima del neurone di semina.
- Chip di montaggio utilizzando fantasia vetrini coprioggetti
Nota: Se i coprioggetti modellati includono photoresist positivo fanno riferimento a oggetti, eseguire passaggi 4.2.1 a 4.2.9. In caso contrario, eseguire solo passaggio 4.2.3, bastone il dispositivo PDMS il coprivetrino PLO modellato come indicato al punto 4.1.2, messo terreno di coltura all'interno e intorno il chip quindi andare al passaggio 4.2.10.- Depositare una goccia d'acqua su un vetrino per microscopio rettangolare di spessore e attaccare il coprioggetto sul vetrino per capillarità (non-fantasia lato rivolto verso il vetrino). Sotto un microscopio, segnare la posizione delle strisce photoresist nella parte posteriore il vetrino utilizzando un pennarello.
- Mettete il coprioggetto di vetro modellato sul supporto maschera di aligner maschera. Si affidano il segno fatto con il pennarello per centrare gli oggetti di riferimento del photoresist.
- Eseguire il passaggio del plasma come descritto in 4.1.1 sul chip PDMS.
- Mettete il chip PDMS sul supporto mobile del substrato (chuck) di aligner maschera.
Nota: Per aumentare il contrasto ottico, posizionare un wafer di silicio sotto il chip PDMS. Il wafer di silicio deve rimanere saldamente agganciato sul mandrino durante il processo di allineamento (uso un nastro trasparente per attaccarlo al mandrino). - Sollevare il mandrino al limite del contatto meccanico per allineare il chip con la matrice di photoresist strisce situate il vetrino coprioggetti.
- Ottenere un contatto meccanico tra il chip e il vetrino coprioggetti finendo il mandrino di sollevamento fino a quando la superficie del PDMS pilastri tocco il vetrino coprioggetti.
- Abbassare il mandrino. Rimuovere il vetrino coprioggetto ora legato al chip da parte del titolare di maschera. Quindi posizionare il dispositivo in una capsula Petri 35 mm e trasferire tutto in forno (temperatura: 70 ° C) per 5-10 min aumentare la forza di legame.
- Eseguire come descritto al punto 4.1.3.
Nota: se utilizzando PLO modellata vetrini coprioggetti, andare direttamente dal passaggio 4.2.7 a passo 4.2.9. - Sostituire PLO dal seguente media placcatura che le procedure descritte nei passaggi 4.1.5 (svuotamento) e 4.1.3 (riempimento). Utilizzando un cono di punta di 1 mL, sostituire anche il PBS che circondano il chip da questo mezzo.
- Mettere il chip in incubatrice regolamentato a 37 ° C e 5% CO2 fino al neurone semina, con un minimo di tempo di incubazione di 5 h.
5. cultura di neurone
- Preparare 100 mL di terreno di dissezione (medio HH) mescolando 10 mL di HBSS 10 x, 2 mL di Hepes 1 M e 88 mL di acqua sterile in un recipiente di plastica di 200 mL.
Nota: Il medium HH può essere preparato il giorno prima la cultura. - Sezionare ippocampo da embrione di topi E18 estratte da una madre eutanasizzata di dislocazione cervicale (topi C57BL/6J da Charles Rivers). Passaggi della dissezione sono ad esempio dettagliato in 16.
- Posto ippocampo in un tubo di plastica contenente tripsina (0,3 mL di tripsina 2.5%, w/o EDTA in 2,7 mL di medium HH) per 10 min a 37 ° C al fine di avviare dissociazione chimica.
- Eliminare quasi tutto il liquido e sostituirlo con 5-10 mL di HH utilizzando pipette di plastica usa e gettare. Fare 3 volte. Per il riempimento di ultimo, usare 1 mL di placcatura media invece di HH.
- Meccanicamente dissociare tessuti utilizzando un punta-cono di 1 mL di aspiranti ed espellere il volume completo più volte, evitando che fa le bolle e utilizzando non più di 15-20 passaggi.
- In un destinatario separato di 500 µ l, preparare una soluzione usando 5 µ l di sospensione cellulare diluito in 45 µ l di PBS. Prendere 1 µ l di questa soluzione utilizzando una pipetta µ l 10 e inserire la sospensione diluita in un contatore di cellule di Malassez. Utilizzare le indicazioni fornite in 17 per stimare il numero di cellule.
Nota: Un singolo ippocampo fornisce solitamente circa 0,5 milioni di neuroni. - Centrifuga a 100 x g per 6 min a RT.
- Scartare il surnatante e sostituirlo con il volume di placcatura medio richiesto per raggiungere una concentrazione di 10 milioni di cellule/mL. Risospendere le cellule da successivamente aspire ed espellere la sospensione cellulare con un cono di punta di 1 mL.
- Redigere il mezzo di placcatura presente nel chip (fare riferimento al punto 4.1.5). Raccogliere 2-3 µ l della soluzione appena sedimento usando una pipetta 10-µ l e iniettarla all'ingresso (fare riferimento alla procedura di iniezione descritta al punto 4.1.3). Immediatamente ripetere la stessa operazione in uscita.
- Iniettare circa lo stesso volume di mezzo di placcatura in ciascun serbatoio (fare riferimento al punto 4.1.3).
Nota: Osservare rapidamente il chip al microscopio per verificare la densità delle cellule. L'ordine di grandezza della densità cellulare ottimale corrisponde a circa 5-10 celle all'interno della superficie quadrata delimitata da 4 pilastri (circa 0,3 mm2, cioè circa 15-20 cellule per mm2). - Eventualmente ripetere il punto 5.10 utilizzando invece di 0,5-1 µ l di sospensione cellulare per raggiungere la densità delle cellule mirati.
- Inserire il chip seminato in un'incubatrice regolamentata a 37 ° C 5% CO2.
6. osservazione di esclusione fluorescenza
- Sostituzione del mezzo di coltura mediante il mezzo di imaging.
- Preparare il supporto di imaging come 4.1.7, ma utilizzare invece MEM privo di rosso fenolo e aggiungere destrano fluorescente. A tale scopo, diluire destrano (peso molecolare 10.000 g/mol, stock soluzione concentrata a 10 mg/mL in PBS) al fine di ottenere una concentrazione finale di 0.5-1 mg/mL nel mezzo di imaging.
Nota: Uso destrano coniugati con i massimi di assorbimento/emissione 496/524 o 650/668. Preferisco la prima immagine di 0,45 µm alta photoresist positivo strutture (per sbarazzarsi di loro fluorescenza di auto nella banda rossa) e la seconda ai neuroni di immagine (meno tossici). - Svuotare tutti gli ingressi utilizzando una pipetta 10-µ l e ri-riempirli completamente con il supporto di formazione immagine (fare riferimento ai punti 4.1.3 e 4.1.5 per la precisa metodologia di sostituzione media).
- Preparare il supporto di imaging come 4.1.7, ma utilizzare invece MEM privo di rosso fenolo e aggiungere destrano fluorescente. A tale scopo, diluire destrano (peso molecolare 10.000 g/mol, stock soluzione concentrata a 10 mg/mL in PBS) al fine di ottenere una concentrazione finale di 0.5-1 mg/mL nel mezzo di imaging.
- Di imaging
- Inserire il chip sotto un microscopio a epifluorescenza dotato di una camera climatica, regolamentata a 37 ° C e 5% di CO2. Usare un 40 X, apertura numerica (NA) 0,8 secco obiettivo, 30% di piena potenza (piena potenza: 3 W) e 30 ms di tempo di esposizione. Acquisizione di immagini di celle al focus (da singolo a più immagini successive in caso di esperimenti di time-lapse).
- Analisi dell'immagine
- Normalizzare le immagini utilizzando la routine di riduzione di sfondo implementata in software dedicato per ottenere uno sfondo omogeneo. Vedi 13 per i dettagli dei passaggi inclusi in questo software di elaborazione delle immagini. L'immagine di uscita ha una. Formato di MAT.
- Convertire. File MAT in. TIFF 8 bit immagini utilizzando la routine messi in materiale supplementare (conversion_mattotiff.m, che chiama per importfilevol.m).
- Eseguire importazione > sequenza di immagini su ImageJ per costruire un video dalla. Immagini TIFF.
- Calcolare l'intensità media P di un'area quadrata localizzata al centro di un pilastro (oggetto reference) e l'intensità media B di un'area quadrata dell'alloggiamento privo di cellule (sfondo, cioè altezza zero). Vedi 18 per un esempio di metodologia dettagliata dell'elaborazione delle immagini.
Nota: La dimensione laterale delle zone quadrate utilizzati come riferimenti di intensità devono essere circa la metà del diametro del pilastro per ottenere un numero sufficiente di pixel evitando inquinamento luminoso dai bordi di pilastro. - Utilizzare i valori di P e B per stabilire la legge di conversione lineare da intensità sono di altezza h:

con hc altezza nota dell'alloggiamento, e
e
Nota: PDMS non visualizza nessun autofluorescence rilevabile. - Selezionare un'area attorno alla zona di interesse, integrare intensità usando ImageJ (Vedi 19 per maggiori dettagli) e applicare la legge di conversione ottenuta in 6.3.5 per misurare il volume di un compartimento cellulare.
Nota: La zona di interesse potrebbe essere selezionata basato sulla fluorescenza degli elementi sub-cellulari come l'actina, per esempio, per la selezione di crescita coni in neuroni di GFP-LifeAct, selezionare la zona di interesse per il canale di emissione di GFP, Risparmia il contorno di questo zona tramite un strumento di gestione di ROI, quindi misura il volume di cella racchiusi all'interno della stessa zona del canale di emissione di destrano (in rosso).
Risultati
Il risultato del processo di fabbricazione descritto nelle sezioni 1 e 2 è illustrato dalle immagini di Figura 1A-1B e la curva di Figura 1. La tabella di Figura 1 Visualizza i valori di rugosità di due diverse aree rappresentative del chip PDMS, cioè nella parte centrale e la camera intermedia successiva alta 20 µm. Una diminuzione della rugosità di un fattore di circa 7 è stata ottenuta utilizzando wafer Si acidato anziché SU-8 photoresist. Quindi, FXm in primo luogo è stato applicato su una striscia di photoresist della geometria noto (Figura 2A) all'interno di una camera alta di 10 µm. Dopo l'elaborazione delle immagini e intensità alla conversione di altezza (Vedi il grafico della Figura 2B), profili FXm eseguiti su sezioni trasversali lungo questa striscia (Figura 2) forniscono i profili di altezza desiderata (Figura 2D). Figura 2D Mostra il confronto tra profili ottenuti utilizzando profilometria meccanica e metodi FXm. Questi profili, incluso il valore dei margini e plateau, sono molto simili, il metodo di convalida. Si noti che la dispersione dei dati FXm non è rappresentativa della risoluzione finale del metodo, come ulteriormente valutato nella Figura 3 e Figura 4, ma i risultati di bassa intensità impiegate per evitare un possibile effetto di molto debole auto-fluorescenza di photoresist nel canale di GFP.
Quindi, abbiamo osservato i neurites a 3 µm e 10 µm alta chambers (Figura 3). La deviazione standard del rumore di fondo è circa 18 nm dopo intensità alla correzione di conversione e sfondo di altezza. Questo valore è leggermente superiore il fisico rugosità delle superfici PDMS castate su superfici di silicio (12 nm, vedere la tabella di Figura 1) ma molto più basso di quanto la rugosità misurata su PDMS ottenuti da stampi SU-8. Questi risultati evidenziano il valore aggiunto di perforazione di pozzi in wafer di silicio, anziché aprire fori in SU-8 photoresist eseguire il cast di pilastri. Un basso valore permette un alto segnale rumore e immagini molto chiare nel volume come quello mostrato in Figura 3A. Come esempio dei dati che possono essere recuperati da tali immagini, abbiamo calcolato il volume di 1,6 µm (cioè 10 pixel) del neurite ampia fetta (Vedi il grafico della Figura 3B). Utilizzo in prima approssimazione una misura lineare di questi dati fornisce un valore di altezza media del neurite di circa 400 nm, da confrontare con per esempio il diametro assonale 500 nm trovato in 10 giorni vecchi cuccioli all'interno del corpo calloso 5. Abbiamo anche combinato FXm con microfantasie di adesione consistente in serie intestati 2 µm e 6 µm strisce di larghezza di 30 µm di lunghezza. Il nostro scopo era di studiare l'influenza della larghezza del neurite sulla sua forma 3D. Figura 3 Mostra una rappresentazione 3D in falsi colori di un'immagine di intero neurone ottenuta in una camera alta di 10 µm. Neurites stanno diffondendo su 2 µm e strisce di larghezza 6 µm, mentre il soma è situato all'estremità della striscia più grande. Profili di altezza sono stati disegnati in tre diverse sezioni trasversali. In coerenza con il grafico visualizzato in Figura 3A, la superficie integrata per l'aumento di sezioni trasversali con la larghezza del neurite (Figura 3D).
Ci siamo concentrati anche su strutture 3D di crescita cono (GC). Figura 4 A-B consente di visualizzare due diversi profili GC ottenuti in una camera alta di 3 µm, che mettono in evidenza loro sub-struttura ramificata. Inoltre, abbiamo effettuato esperimenti di time-lapse per seguire la dinamica del volume di GCs in una camera alta di 12 µm. Figura 4 Mostra un ciclo di compattazione e riattivazione di un GC specificato all'interno di una scala di tempo di poche decine di minuti. Grazie all'utilizzo di topi GFP-lifeact, coni di crescita sono stati localizzati nella lunghezza d'onda di emissione di GFP (510 nm) da loro concentrazione alta actina. La superficie identificata nella lunghezza d'onda è stata utilizzata per integrare sopra la lunghezza d'onda emissione dextrano a 647 nm per calcolare il volume di GC. Figura 4 Mostra infine la distribuzione del volume di GC ad intervalli di tempo differenti e posizione su tre diversi neuroni, centrati su un valore di circa 6 µm3.

Figura 1: chambers FXm PDMS. (A) schemi delle quattro fasi principali differenti di microfabbricazione che conduce allo stampo finale. La posizione dell'entrata, uscita e serbatoi sono indicati. Scala bar: 1 mm. (B) immagine della camera PDMS FXm ottenuta con un profilometro ottico. Questa immagine mostra la camera centrale contenente 3 file di 10 µm alti pilastri e gli alloggiamenti intermedi di µm 20, 50 e 90 di altezza. Barra della scala: 500 µm. (C) vista di sezione trasversale del chip lungo le due linee tratteggiate disegnate in (B). Giallo/oro: sezione trasversale lungo pilastri, blu: sezione trasversale tra i pilastri. (D) significa valori della rugosità PDMS misurati su 50 × 50 µm2 aree modellate su silicio e su 20 µm alta SU-8 camera intermedia (vedi frecce per la posizione di queste zone). I valori medi sono stati ottenuti dalle misurazioni di tre diverse aree. Clicca qui per visualizzare una versione più grande di questa figura.
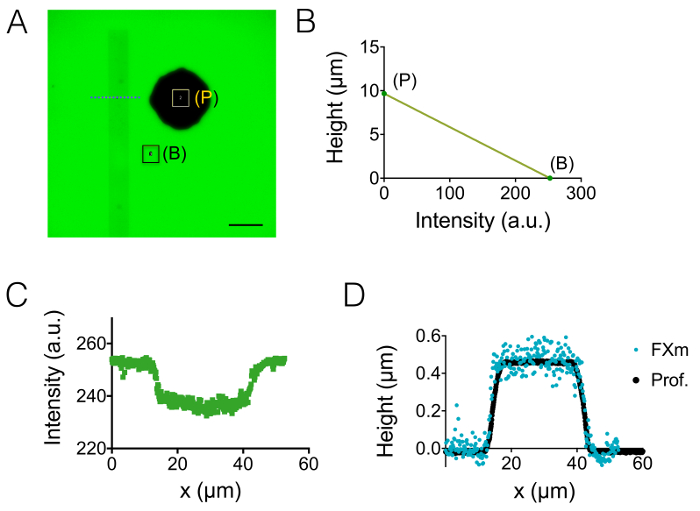
Figura 2: calibrazione del metodo FXm utilizzando una striscia di photoresist come oggetto di interesse. (A) immagine di fluorescenza GFP presi in una camera alta di 10 µm riempito con 10.000 destrano MW assorbente a 488 nm a 1 mg/mL. (B: sfondo, p: pilastro). Osservazione con un obiettivo di NA X 0,8 40 asciutto. Barra della scala: 50 µm. (B) diritto di taratura lineare ottenuta dall'intensità media dei due rettangoli colorati mostrato nel profilo di intensità a. (C) fluorescenza ottenuto a livello della linea tratteggiata blu visualizzato in (A), attraversando il photoresist striscia (0,45 µm alta photoresist positivo). (D) confronto tra i profili ottenuti da profilometro meccanico (punti neri) e FXm dopo intensità alla conversione di altezza dei dati di (B) (punti blu). Clicca qui per visualizzare una versione più grande di questa figura.
Figura 3: imaging con volumi del Neurite. (A) dei neuriti che si estende nella camera alta 3 µm centrale da soma situato nella camera intermedia prossimi 15 µm. Formazione immagine effettuata utilizzando 10.000 destrano MW assorbente a 488 nm e 40 X, NA 0.8 a secco obiettivo. L'inserto ottenuti dopo l'utilizzo della routine sfondo riduzione evidenzia i due neurites e scelto di tracciare il grafico sulla destra. Scala bar: 30 µm. (B) del Neurite fetta volume in funzione della larghezza del neurite ottenuti dai 22 profili (media su 10 pixel, cioè su un 1,6 µm "neurite fetta") mostrati in (A). La linea continua rappresenta una misura lineare di pendenza 0,4 µm passando attraverso l'origine. (C) falso colore immagine di un neurone modellato su una striscia adesiva in successivi 2 µm e il µm 6 ceppi ampia (rappresentati in bianco). Le misure sono state effettuate in una camera alta di 10 µm piena di 10.000 destrano MW assorbente a 647 nm e utilizzando un NA 40 x 0,8 a secco obiettivo. (D) profili di altezza corrispondente per le colorate righe tratteggiate in (C), mantenendo lo stesso codice di colore. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: formazione immagine statica e dinamica crescita cono. (A-B) Profili di altezza cono crescita ottenuti in una camera alta di 3 µm dopo intensità alla conversione di altezza lungo le linee gialle visualizzati in immagini associate. Osservazione eseguita utilizzando un riempimento con 10.000 destrano MW assorbente a 488 nm e 40 X, NA 0.8 a secco obiettivo. (C) formazione immagine intero neurone in una camera alta 12 µm riempito con 10.000 destrano MW assorbente a 647 nm. Sono state fatte osservazioni in due canali fluorescente: GFP per la localizzazione di cono di crescita (linee gialle tratteggiate) e CY5 per calcolare il volume di GC dall'esclusione di fluorescenza. La superficie inclusa da linee gialle tratteggiate è stata utilizzata per calcolare il volume di GC. Il grafico mostra la variazione del volume di GC nel tempo e morfologie associate nei canali sia GFP e CY5 in due diversi momenti rappresentativi. Tutti i dati sono stati acquisiti utilizzando un NA 40 x 0,8 a secco obiettivo barre della scala ogni 3 min: 10 µm. Clicca qui per visualizzare una versione più grande di questa figura.
| Passo | Maschera 1:8 µm strato | Maschera 02:30 µm strato | Maschera 03:40 µm strato |
| Tipo SU-8 | 2007 | 2025 | 2050 |
| Spincoating | 30 s @ 2000 giri/min | 30 s @ 3050 rpm | 30 s @ 3250 giri/min |
| Cuocere morbido | 3 min a 95 ° C | 2 min @ 65 ° C + 6 min a 95 ° C | 3 min @ 65 ° C + 7 min a 95 ° C |
| Energia di esposizione | 110 mJ/cm2 | 155 mJ/cm2 | 170 mJ/cm2 |
| Cuocere post-esposizione | 4 min a 95 ° C | 1 min a 65 ° C + 6 min a 95 ° C | 2 min @ 65 ° C + 7 min a 95 ° C |
| Sviluppo | 2 min 30 s | 5 min | 6 min |
| Difficile cuocere (opzionale) | 3-5 min a 200 ° C | 3-5 min a 200 ° C | 3-5 min a 200 ° C |
Tabella 1: fotolitografia passaggi eseguiti per costruire un dispositivo contenente una camera centrale di 12 μm in altezza. Altezze delle sezioni intermedie: µm 20, 50 e 90.
| Passo | Maschera 01:10 µm strato | Maschera 02:30 µm strato | Maschera 03:40 µm strato |
| Tipo SU-8 | 2007 | 2025 | 2050 |
| Spincoating | 30 s @ 1500 giri/min | 30 s @ 3050 rpm | 30 s @ 3250 giri/min |
| Cuocere morbido | 3 min a 95 ° C | 2 min @ 65 ° C + 6 min a 95 ° C | 3 min @ 65 ° C + 7 min a 95 ° C |
| Energia di esposizione | 125 mJ/cm2 | 155 mJ/cm2 | 170 mJ/cm2 |
| Cuocere post-esposizione | 4 min a 95 ° C | 1 min a 65 ° C + 6 min a 95 ° C | 2 min @ 65 ° C + 7 min a 95 ° C |
| Sviluppo | 2 min 30 s | 5 min | 6 min |
| Difficile cuocere (opzionale) | 3-5 min a 200 ° C | 3-5 min a 200 ° C | 3-5 min a 200 ° C |
Tabella 2: fotolitografia passaggi eseguiti per costruire un dispositivo contenente una camera centrale di 10 μm in altezza. Altezze delle sezioni intermedie: µm 20, 50 e 90.
| Passo | Maschera 01:12 µm strato | Maschera 02:32 µm strato | Maschera 03:40 µm strato |
| Tipo SU-8 | 2015 | 2025 | 2050 |
| Spincoating | 30 s @ 3250 giri/min | 30 s @ 2500 giri/min | 30 s @ 3250 giri/min |
| Cuocere morbido | 3 min a 95 ° C | 2 min @ 65 ° C + 5 min a 95 ° C | 3 min @ 65 ° C + 7 min a 95 ° C |
| Tempo di esposizione | 140 mJ/cm2 | 157 di mJ/cm2 | 170 mJ/cm2 |
| Cuocere post-esposizione | 4 min a 95 ° C | 1 min a 65 ° C + 5 min a 95 ° C | 2 min @ 65 ° C + 7 min a 95 ° C |
| Sviluppo | 3 min | 5 min | 6 min |
| Difficile cuocere (opzionale) | 3-5 min a 200 ° C | 3-5 min a 200 ° C | 3-5 min a 200 ° C |
Tabella 3: fotolitografia passaggi eseguiti per costruire un dispositivo contenente una camera centrale di 3 μm in altezza. Altezze delle sezioni intermedie: 18, 50 e 90 µm.
Dati supplementari 1: masks_neuron_volume_chips.tiff. Rappresentazione schematica delle maschere usate per fabbricare il dispositivo PDMS (DRIE maschera e maschere 1-3). Per favore clicca qui per scaricare questo file.
Dati supplementari 2: file "masks_neuron_volume_chips.dxf". File elettronici che permette di fabbricare la maschera di DRIE e maschere 1-3. Per favore clicca qui per scaricare questo file.
Dati supplementari 3: "Mask_Photoresist-stripes.dxf". File elettronici che permette di fabbricare la maschera utilizzata per la fotolitografia di strisce di photoresist. Per favore clicca qui per scaricare questo file.
Dati supplementari 4: file conversion_mattotiff.m Clicca qui per scaricare questo file.
Dati supplementari 5: file importfilevol.m Clicca qui per scaricare questo file.
Discussione
Imaging con volumi di neuroni costituisce una sfida per la tecnica di FXm dovuto le estensioni lunghe e sottili di queste cellule. Questo protocollo descrive varianti dello stesso tipo di dispositivo microfluidico dedicata all'imaging del neurone.
Al lato degli aspetti della progettazione microfluidica, la scelta dell'obiettivo è fondamentale per l'imaging di fluorescenza esclusione e implica un compromesso tra la risoluzione laterale e la chiarezza di immagine. È stato dimostrato in 13 che un NA alta che porta ad una profondità di messa a fuoco più piccolo dell'altezza della camera non era dannoso per la precisione di misurazione di volume se imaging è stata eseguita a fuoco e se viene lasciato un margine sufficiente tra il contorno dell'oggetto di i nterest e i limiti della superficie di integrazione. Tuttavia, l'uso di una camera molto più alta rispetto alla profondità di messa a fuoco altera la chiarezza di immagine a causa della diffusione del fotone, che smussa i bordi degli oggetti di interesse. La realizzazione di una camera alta di 3 µm ridotto questo offuscamento laterale e fornito immagini eccezionalmente ben definita esclusione fluorescente anche utilizzando alta NA (0,8) 40 X obiettivi per visualizzare un neurone rami con elevata risoluzione laterale.
Chip di montaggio è un passo fondamentale, in particolare nel caso di 3 µm alta chambers, ma la manipolazione attenta come descritto al punto 4.1.2 evita il crollo del tetto. L'alta superficie in rapporto al volume associato a questi alloggiamenti sottili anche sollevato la questione della stabilità della concentrazione di destrano nel corso del tempo. Abbiamo controllato che l'assorbimento superficiale del destrano dopo una notte di incubazione era trascurabile: dopo la sostituzione del dextrano di PBS, la differenza di intensità tra il pilastro e lo sfondo era circa 1 / 1000 del contrasto tra intensità iniziale Queste due regioni in presenza di destrano. Si noti che i neuroni possono aderire il coprioggetto di fondo sia sul tetto PDMS. Questo effetto scomparirà quando usando fantasia coprioggetti (cioè quando noi non Incubare molecole adesive all'interno della camera PDMS), come il rivestimento è pertanto strettamente localizzato sul fondo della camera.
Oltre alla loro morfologia impegnativo, i neuroni sono piuttosto adatti a FXm dovuto al fatto che una delle principali limitazioni del metodo, cioè endocitosi di destrano, è molto limitata in queste cellule. Scegliamo un 10 kDa formulazione per sopprimere nel lungo intervallo (ore) eventuali fenomeni di endocitosi visibile.
In conclusione, la semplicità concettuale del FXm è bilanciata da un insieme di problemi sperimentali che sono stati risolti dal presente protocollo, ad esempio nanometriche PDMS rugosità e altezza della camera micrometrica o correzione del fondo per correggere le irregolarità del il soffitto di PDMS tra pilastri. Tuttavia, l'uso di una camera stretta microfluidici per confinare il mezzo fluorescente produce pochi vincoli specifici come la necessità di pilastri di sostegno, che abbassa la superficie efficace disponibile per adesione delle cellule, o la necessità di escludere soma dalla centrale Camera per osservare un neurone estensioni con la massima chiarezza, che limita le regioni della cellula accessibile all'osservazione ad alta risoluzione. Una possibile evoluzione di questo metodo sarebbe quello di sbarazzarsi di questo contenimento fisico, per essere sostituita da un ottico. Il nuovo sviluppo della microscopia foglio leggero potrebbe essere abbinato vantaggiosamente FXm in futuro.
Divulgazioni
Gli autori non dichiarano alcun conflitto di interessi.
Riconoscimenti
Gli autori vogliono riconoscere ChiLab, materiali e microsistemi laboratorio - Politecnico di Torino - DISAT, nella persona del Prof C F Pirri, Dr. M Cocuzza e Dr. S L Marasso, per il loro prezioso aiuto nel processo di sviluppo e fabbricazione di dispositivi. Ringraziamo Victor Racine da Quantacell di discussione e supporto nella elaborazione delle immagini. Siamo grati a Isabelle Grandjean e Manon Chartier da Animal Facility del Institut Curie per il loro sostegno per i topi e Pablo Vargas e Ana-Maria Lennon (Institut Curie) per averci fornito i topi GFP LifeAct. Siamo grati a Olivier Thouvenin dal Institut Langevin e Clotilde Cadart, Larisa Venkova e Matthieu Piel dall'Institut Curie - UMR 144, per il loro aiuto nella comprensione dell'esclusione fluorescenza metodo. Infine, ringraziamo la piattaforma tecnologica del Institut Pierre-Gilles de Gennes (CNRS UMS 3750) per il loro sostegno nella microfabbricazione. Questo lavoro è stato in parte sostenuto dall'europeo ricerca Consiglio Advanced Grant No. 321107 "Violoncello", Université PSL (progetto SwithNeuroTrails), ANR Investissement Avenir e la IPGG Labex ed Equipex.
Materiali
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| Plasmalab System 100 | Oxford Instruments | To perform DRIE | |
| MJB4 mask aligner | SUSS MicroTec | SU-8 photolithography | |
| NXQ 4006 Mask aligner | Neutronix Quintel | Photolithography associated to DRIE | |
| Plasma cleaner | Diener Electronic | Pico PCCE | |
| Cell culture hood | ADS Laminaire | Optimale 12 | |
| Centrifuge | Thermo Fisher Scientific | Heraeus Multifuge X1R | |
| Incubator 37 °C 5% CO2 | Panasonic | MCO-170AICUVH-PE IncuSafe CO2 Incubator | |
| Epifluorescence microscope | Leica | DMi8 | |
| PDMS Oven between 65 and 80 °C | Memmert | ||
| Mechanical profilometer | Veeco | Dektak 6M Stylus Profiler | |
| Optical profilometer | Veeco | Wyko NT9100 | |
| Vacuum desiccator | Verrerie Villeurbanaise (Kartel Labware) | 230KAR | Diameter 200 mm |
| Ultrasonic bath sonicator | Labo Moderne | SHE1000 | Volume of the bath: 0.8 liter |
| Hotplate | Stuart SD162 | SD162 | |
| Masks | |||
| DRIE | Supplementary data | ||
| Su8 Mask 1 | Supplementary data | ||
| Su8 Mask 2 | Supplementary data | ||
| Su8 Mask 3 | Supplementary data | ||
| S1805 calibration stripes | Supplementary data | ||
| Small laboratory equipment | |||
| 1.5 mm hole puncher | Sigma-Aldrich | 29002519 (US reference) | |
| Scalpel or razor blade | |||
| 9" Stainless Steel Flat Spatula with Spoon | VWR International | 82027-532 | To demold PDMS |
| Top Lip Wafer Handling | VWR International | 63042-096 | |
| Curved tweezer | FST | Dumont #7 Forceps - Standard / Dumoxel | To manipulate glass coverslips |
| Substrates | |||
| Silicon wafer | Prolog Semicor Ltd | ||
| 24x24 mm glass coverslips | VWR | 631-0127 | |
| Photoresists and developpers | |||
| AZ 1518 positive photoresist | Microchemicals GmbH | Before DRIE process, thicknes 1.8 µm | |
| AZ 351B developer | Microchemicals GmbH | To develop positive AZ 1518 photoresist | |
| SU-8 2007 | MicroChem | ||
| SU-8 2025 | MicroChem | ||
| SU-8 2050 | MicroChem | ||
| PGMA developer | Technic | To develop SU-8 negative photoresist | |
| Microposit S1805 resist | Chimie Tech Services | Positive photoresist used to obtain 0.5µm high structures | |
| MF 26A developer | Chimie Tech Services | To develop positive S1805 photoresist | |
| Laboratory consumables | |||
| Disposable plastic pipette 3 mL | LifeTechnologies - ThermoFisher | ||
| P100 Petri dishes | TPP | 93100 | |
| 20 mL syringe | Terumo | SS+20ES1 | |
| Transparent scotch tape | |||
| Square wipes | VWR | 115-2148 | |
| Parafilm | DUTSCHER | 90260 | Plastic paraffin film |
| Chemicals | |||
| (3-Methacryloxypropyl)trichlorosilane | abcr | AB 109004 | |
| PDMS and curing agent Sylgard 184 | Sigma-Aldrich | 761036 | |
| isopropanol | W292907 | ||
| poly-ornithine | Sigma-Aldrich | P4957 - 50 mL | |
| Ethanol absolute | Sigma-aldrich | 02865 | 99.8% |
| 3-methacryloxypropyl-trimethoxysilane | Sigma-aldrich | M6514-25ML | (C4H5O2)-(CH2)3- Si(OCH3)3 |
| acetic acid | Sigma-aldrich | 71251-5ML-F | |
| Dextran 10kW conjugated with Alexa488 | LifeTechnologies - ThermoFisher | D22910 | Absoprtion at 488 nm |
| Dextran 10kW conjugated with Alexa647 | LifeTechnologies - ThermoFisher | D22914 | Absoprtion at 647 nm |
| Culture medium | |||
| MEM | LifeTechnologies - ThermoFisher | 21090-022 | |
| Horse Serum | LifeTechnologies - ThermoFisher | 26050088 | |
| B27 | LifeTechnologies - ThermoFisher | 12587-010 | |
| Glutamax 200 mM | LifeTechnologies - ThermoFisher | 35050-061 | |
| Sodium Pyruvate GIBCO 100 mM | LifeTechnologies - ThermoFisher | 11360-070 | |
| Gentamicin | LifeTechnologies - ThermoFisher | 15710-049 | |
| PBS | Sigma-Aldrich | D8537-500ML | |
| HBSS 10x | LifeTechnologies - ThermoFisher | 14180-046 | |
| Hepes 1M | LifeTechnologies - ThermoFisher | 15630-056 | |
| trypsin-EDTA | Sigma-Aldrich | 59418C-100ML | |
| Neurobasal | LifeTechnologies - ThermoFisher | 21103-049 | |
| Neurobasal without phenol red | LifeTechnologies - ThermoFisher | 12348-017 | |
| Softwares | |||
| Routine in Matlab for background normalization | Quantacell | Contact Victor Racine: victor.racine@quantacell.com | |
| ImageJ | To select specific ROI for image analysis | ||
| Routine | ImageJ | Supplementary data | |
| Routine | Matlab | Supplementary data |
Riferimenti
- Jun, S., Taheri-Araghi, S. Cell-size maintenance: universal strategy revealed. Trends Microbiol. 23 (1), 4-6 (2015).
- Zlotek-Zlotkiewicz, E., Monnier, S., Cappello, G., Le Berre, M., Piel, M. Optical volume and mass measurements show that mammalian cells swell during mitosis. J. Cell Biol. 211 (4), 765-774 (2015).
- Kim, G. H., Kosterin, P., Obaid, A. L., Salzberg, B. M. A mechanical spike accompanies the action potential in Mammalian nerve terminals. Biophys J. 92 (9), 3122-3129 (2007).
- Iacono, D., et al. Neuronal Hypertrophy in Asymptomatic Alzheimer Disease. J Neuropathol Exp Neurol. 67 (6), 578-589 (2008).
- Cheli, V. T., et al. Conditional Deletion of the L-Type Calcium Channel Cav1. 2 in Oligodendrocyte Progenitor Cells Affects Postnatal Myelination in Mice. J Neurosci. 36 (42), 10853-10869 (2016).
- Kneynsberg, A., Collier, T. J., Manfredsson, F. P., Kanaan, N. M. Quantitative and semi-quantitative measurements of axonal degeneration in tissue and primary neuron cultures. J Neurosci Methods. 266, 32-41 (2016).
- Fouquet, C., et al. Improving axial resolution in confocal microscopy with new high refractive index mounting media. PloS one. 10 (3), (2015).
- Aguet, F., et al. Membrane dynamics of dividing cells imaged by lattice light-sheet microscopy. Mol Biol Cell. 27 (22), 3418-3435 (2016).
- Roland, A. B., et al. Cannabinoid-induced actomyosin contractility shapes neuronal morphology and growth. eLife. 3, (2014).
- Lu, Y. B., et al. Viscoelastic properties of individual glial cells and neurons in the CNS. Proc Natl Acad Sci USA. 103 (47), 17759-17764 (2006).
- Chen, J., Xue, C., Zhao, Y., Chen, D., Wu, M. H., Wang, J. Microfluidic impedance flow cytometry enabling high-throughput single-cell electrical property characterization. Int J Mol Sci. 16 (5), 9804-9830 (2015).
- Bottier, C., et al. Dynamic measurement of the height and volume of migrating cells by a novel fluorescence microscopy technique. Lab Chip. 11, 3855-3863 (2011).
- Cadart, C., et al. Fluorescence eXclusion Measurement of volume in live cells. Methods Cell Biol. 139, 103-120 (2017).
- Marasso, S. L., et al. A novel graphene based nanocomposite for application in 3D flexible micro-supercapacitors. Materials Research Express. 3 (6), 065001 (2016).
- Welch, C. C., Goodyear, A. L., Wahlbrink, T., Lemme, M. C., Mollenhauer, T. Silicon etch process options for micro-and nanotechnology using inductively coupled plasmas. Microelectronic Engineering. 83 (4), 1170-1173 (2006).
- Fath, T., Ke, Y. D., Gunning, P., Götz, J., Ittner, L. M. Primary support cultures of hippocampal and substantia nigra neurons. Nat. Protoc. 4 (1), 78-85 (2008).
- Getting intensity values from single ROI. Image Intensity Processing Available from: https://imagej.net/Image_Intensity_Processing (2017)
- 19. Tools. ImageJ User Guide Available from: https://imagej.nih.gov/ij/docs/guide/146-19.html (2017)
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon