Method Article
Haute résolution Volume Imaging de neurones par l’eXclusion de l’utilisation de la Fluorescence méthode et dédié des dispositifs microfluidiques
Dans cet article
Résumé
Le volume est un paramètre important concernant les caractéristiques physiologiques et pathologiques des cellules. Nous décrivons une méthode d’exclusion fluorescent permettant la mesure de plein champ en vitro neuronale volume avec Sub micrométrique résolution axiale requise pour l’analyse des neurites et des structures dynamiques, impliqués dans la croissance neuronale.
Résumé
Le volume est un paramètre important concernant les caractéristiques physiologiques et pathologiques des neurones à des échelles de temps différentes. Les neurones sont des cellules tout à fait uniques au sujet de leurs morphologies ramifiés étendus et par conséquent relever plusieurs défis méthodologiques pour la mesure de volume. Dans le cas particulier de in vitro la croissance neuronale, la méthodologie choisie devrait inclure Sub micrométrique résolution axiale combinée avec plein champ observation sur des échelles de temps de minutes d’heures ou de jours. Contrairement à d’autres méthodes comme la reconstruction de forme de cellule à l’aide de l’imagerie confocale, mesures électriquement ou microscopie à Force atomique, la méthode d’eXclusion à la Fluorescence récemment développée (FXm) a le potentiel de répondre à ces défis. Cependant, bien qu’elle soit simple dans son principe, mise en œuvre d’un FXm haute résolution pour les neurones nécessite plusieurs ajustements et une méthodologie dédiée. Nous présentons ici une méthode basée sur la combinaison de l’exclusion de la fluorescence, Dispositifs microfluidiques multi-compartiments bas-rugosité et enfin microstructuration à obtenir in vitro des mesures de volume neuronal local. La haute résolution fournie par l’appareil nous a permis de mesurer le volume local des processus neuronaux (neurites) et le volume de certaines structures spécifiques impliqués dans la croissance neuronale, tels que les cônes de croissance (GCs).
Introduction
La connaissance précise du volume cellulaire a attiré une attention accrue dans les dernières années, conduit par la question de l’homéostasie cellulaire taille archaios 1 et plus généralement dans les cellules mitotiques 2. Cependant, la question du volume des cellules est pertinente aussi pour les cellules post-mitotiques, pour lequel les neurones constituent un exemple paradigmatique.
Volume est en effet une signature importante des événements physiologiques et pathologiques à différentes échelles et points dans le temps vie neuronale, des déformation axonale transitoire, liée à l’activité électrique (échelle de la milliseconde) 3 pour l’irréversible neuronale gonflement survenant durant la phase asymptomatique de neurodegenerative maladies (au cours des années chez l’homme) 4. Toutefois, le changement de volume plus important se produit sur une échelle de temps intermédiaires de jours ou de semaines (en fonction de l’organisme considéré) au cours de la croissance neuronale. La morphologie étendue et complexe des neurones soulève des questions multiples, parmi lesquels la réglementation de la taille des cellules. Diamètre et longueur axonal sont en effet étroitement réglementé en vivo, avec des valeurs spécifiques à chaque type neuronal 5,6.
Ces questions, complexes à aborder en vivo, peuvent également être adressées d’une manière simplifiée in vitro. Dans ce but, une méthode dédiée à la mesure de volume rapide assez pour suivre la dynamique de croissance (c'est-à-dire sur une échelle de temps de minutes) et compatible avec l’observation durant des heures ou des jours est requise. Plusieurs méthodes ont été développées au fil des ans pour donner un accès direct ou indirect à volume cellulaire in vitro. Reconstruction de la cellule d’imagerie confocale est l’un d'entre eux, mais cette méthode implique une exposition étiquetage et répétée à la lumière tout en montrant une résolution limitée axiale d’environ 500 nm 7. Notez que ces deux derniers inconvénients sont partiellement surmontés par une méthode plus sophistiquée et plus récente, nommée treillis microscopie de lumière-feuille 8. Microscopie à Force atomique a été utilisé 9 , mais cette méthode de balayage est par essence lent et fastidieux. En outre, le contact physique, qu'elle exige avec la cellule risquent d’interférer avec la mesure compte tenu de l’extrême douceur de neurones 10. Méthode indirecte à l’aide de la résonance ou impédance ont été employés pour différentes cellules types 11, mais insuffisante pour nous adressons des cellules adhésives, comme les neurones.
Une des méthodes plus prometteuses est basée sur la mesure du volume exclu des cellules dans une chambre étroite remplie avec un colorant fluorescent. La méthode par Fluorescence de l’eXclusion (FXm) est simple dans son principe, car il ne nécessite aucun étiquetage et convient pour l’imagerie optique rapide, à long terme des populations de cellules avec une résolution axiale potentiellement l’optique. Plus précisément, la résolution en z dépend de l’intensité de fluorescence maximum dans la chambre de culture (c'est-à-dire dans une région dépourvue de cellules) divisée par la dynamique de la caméra, bien que plusieurs sources de bruit limitent cette résolution ultime. Cette méthode a été très puissante pour suivre le volume de la migration de cellules adhérentes 12 ou pour étudier le changement de volume au cours de la mitose de cellules de mammifères, minutieusement décrit dans 13. Toutefois, les neurones constituent un défi méthodologique pour FXm compte tenu de leur grande ramification dans les processus de Sub micrométriques.
Nous présentons ici une méthode conduisant à la fabrication des lisses FXm chambres d’accéder avec grande précision le volume et la hauteur des branches neuronales et dynamique des structures impliquées dans la croissance neuronale comme les cônes de croissance.
Chambers doit avoir des hauteurs similaires que l’objet à mesurer afin d’optimiser la résolution axiale. Par conséquent, nous avons conçu différents dispositifs FXm caractérisés par des chambres de mesure centrale des trois hauteurs différentes. La plus mince (3 µm de hauteur) est dédié à la mesure des neurites : cette faible hauteur exclut soma, qui restent dans la chambre intermédiaire élevé près de 15 µm. Chambres centrales plus épais (10 à 12 µm) sont suffisamment élevées pour suivre la croissance de cellules entières. Le dispositif comprend également deux réservoirs situés de chaque côté de la chambre centrale. Quatre trous d’injection (IH) sont ainsi mis en œuvre et sont désignées comme suit : l’entrée et la sortie servent à introduire les suspensions cellulaires dans la puce, alors que les deux autres alimentent les réservoirs.
Nous avons la première lamelles fabriqué d’étalonnage pour les mesures de hauteur à l’aide de structures de résine photosensible de géométrie connue. Nous avons ensuite imagés gratuits neurones croissantes, mais aussi morphologiquement contraints neurones dans micromotifs de l’adhérence.
Protocole
L’étude a été réalisée conformément aux directives de la Communauté européenne sur le soin et l’utilisation des animaux de laboratoire : 86/609/CEE. Le but de la recherche et du protocole sont décrites dans le projet éthique annexe de ERCadg violoncelle, qui a été approuvé et est régulièrement examinée par le ERCEA. Institut Curie animalerie a reçu la licence #C75-05-18, 24/04/2012, rapports au Comité d’Ethique en matière de "expérimentation animale Paris Centre et Sud (numéro d’enregistrement National : #59).
1. fabrication du moule
Remarque : Le moule comprend des chambres centrales et intermédiaires reliés à un bras et une prise de courant, plus deux réservoirs (et anses) situés des deux côtés de la chambre centrale.
- Utiliser des pinces plates pour manipuler des plaquettes de silicium diamètre 51 mm et de les transférer d’un endroit à l’autre.
- Moule silicone chambre centrale
- Remplir une pipette en plastique de 2 mL de résine photosensible positive. Positionner la pipette au centre d’une tranche de silicium de 51 mm de diamètre et appuyer sur son réservoir jusqu'à couvrant environ 75 % de la surface de gaufrette avec la résine photosensible. Spin-manteau à 3000 tr/min pendant 30 s.
- Transférer la plaquette de l’essorage-coater à une plaque de cuisson, avec une température de 100 ° C, pendant 50 s.
- Transférer la plaquette de la plaque chauffante dans le porte-substrat (chuck) de l’aligneur de masque. Exposer à travers le « masque DRIE » (voir fichiers supplémentaire « masks_neuron_volume_chips.tiff » et « masks_neuron_volume_chips.dxf »).
- Retirer le mandrin de la plaquette, puis il plonge dans un verre de 100 mL, cristallisant le plat contenant le développeur (dilution 1:4 dans l’eau désionisée). Libérer la plaquette et le maintenir pendant 1 min tout doucement et continuellement en remuant le plat de cristallisation.
Remarque : Pour enregistrer les réactifs, remplissez le plat cristallisant à 1 cm de hauteur au maximum. - Reprendre la plaquette du développeur et il submerge dans un plat de cristallisation de 100 mL rempli d’eau déionisée pour environ 10 s.Then place la plaquette sur un papier absorbant et sécher avec de l’azote sous pression à l’aide d’un pistolet de soufflage.
- Placer la plaquette pour 50 s sur une plaque chauffante avec une température de 115 ° C.
- Effectuer la gravure ionique réactive profonde (DRIE) avec les paramètres suivants (plus d’informations sur la technique DRIE peuvent être trouvés dans références 14 et 15) ; Étape de passivation : 50 sccm de C4F8, il sauvegarde flux sccm 10, CP 10 W, inductivement couplé Plasma (ICP) 1500 W, total pression 24 mbar, température de 18 ° C ; Étape de gravure : 100 sccm de SF6, il sauvegarde flux sccm 10, CP 11 W, 1500 W, total de la PIC pression 24 mbar, température de 18 ° C ; Temps de passivation : 4 s ; Temps de gravure : 7 s ; Processus de durée totale : en général 5 min pour environ 10 µm gravé de profondeur.
- Dissoudre la résine photosensible en plongeant le substrat dans un plat de cristallisation rempli avec de l’acétone.
- Plonger les tranches dans une solution de piranha (2/3 H2O2 (30 %) et 1/3 H2SO4 (95 %)) pendant 5 min.
ATTENTION : Toujours ajouter H2O2 tout d’abord, et ensuite H2donc4 et rinçage au moins 3 fois dans l’eau désionisée après le nettoyage.
- Fabriquer des moules correspondant aux chambres intermédiaires en procédant comme 1.2.1 à 1.2.11 suivant les paramètres répertoriés dans les tableaux 1 à 3.
Remarque : Chaque table correspond à un périphérique donné, caractérisé par une hauteur spécifique de la chambre centrale d’observation.
Remarque : Effectuer l’ensemble du processus utilisant croissant hauteurs de résines photosensibles (masques 1 à 3 pour chaque périphérique, voir fichiers supplémentaire « masks_neuron_volume_chips.tiff » et « masks_neuron_volume_chips.dxf »).- Placer la tranche de silicium gravé sur le porte-substrat-spin coater.
- Vérifiez que le spin-coater fonctionne correctement en vérifiant que l’aspiration du substrat est efficace (le substrat doit tourner et rester en place pendant la vitesse de rotation nominale).
- La viscosité du SU-8 augmente avec la hauteur de la gamme d’épaisseur ciblées. Toujours utiliser des bouteilles de 20 à 30 mL pour stocker les photorésines SU-8 et versez-la sur la plaquette avant enduction centrifuge (SU-8 pourrait être trop visqueux pour être manipulé avec une pipette en plastique).
- Verser le négatif de type époxy résine photosensible SU-8 au centre du substrat jusqu'à couvrant environ 75 % de la tranche de silicium, puis spin-manteau en utilisant les paramètres indiqués dans la ligne « Spincoating » des tableaux.
- Place la plaquette couchée sur une table de travail pour la durée et la température indiquée en ligne « douce cuisson au four ».
- Monter la plaquette gravée et adéquate « SU-8 masque » sur l’aligneur de masque.
- Aligner la plaquette décapée avec le masque à l’aide de la Croix alignement dédié (taille typique de ces croisements : 50 µm × 150 µm) conçu sur chaque masque.
Remarque : Deux croix de chaque côté de l’appareil est suffisante (celui en bas à gauche) et l’autre en haut à droite. - Exposer en utilisant la je-ligne de l’aligneur de masque (longueur d’onde 365 nm) avec la dose d’UV appropriée, comme indiqué dans la rangée « Exposition à l’énergie ».
Remarque : La durée d’exposition est calculé en divisant le spécifique de l’énergie E exposition pour chaque photorésine par la puissance de la lampe UV, modulée par l’absorption du masque :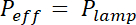 x (1 - Absorptionmasque). Absorption est d’environ 20 % pour des masques souples et négligeable pour masque dur chrome.
x (1 - Absorptionmasque). Absorption est d’environ 20 % pour des masques souples et négligeable pour masque dur chrome. - Placer la plaquette couchée sur une plaque chauffante pour la durée et la température indiquée dans la ligne « Post cuisson au four ».
- Préparer deux 100 mL verre ne cristallise pas plats, l’un contenant le développeur, l’autre vide. Plonger la plaquette dans le développeur pour la durée indiquée dans la rangée « Développement ». Agiter doucement le plat cristallisant tout au long du développement
- Saupoudrer les tranches avec de l’isopropanol dessus plat vide cristallisant pour environ 5 s. Enfin, placer les tranches sur un papier absorbant et sécher avec de l’azote sous pression à l’aide d’un pistolet de soufflage.
- Place la plaquette couchée sur une table de travail pour la durée et la température indiquée en ligne « dur cuisson au four » (en option).
Remarque : Cette étape est utile pour éviter les fissures dans la résine photosensible et fournir une surface plane homogène pour les prochaines étapes. - Répétez les étapes 1.3.1 - 1.3.10 pour terminer les processus répertoriés dans le tableau 1-3.
- Après la dernière étape de la photolithographie, Silyler le capitaine final moule composé de 3 couches de photorésine négatif en dépêchant deux 100 gouttes µL du silane Trichloro(1H,1H,2H,2H-perfluoro-octyl) de chaque côté du maître dans une boîte de Pétri 100 mm. Sceller le disque de Petri avec un film plastique paraffine et incuber 20 min à température ambiante (RT).
Remarque : Le moule principal est prêt et peut être utilisé plusieurs fois.
- Placer la tranche de silicium gravé sur le porte-substrat-spin coater.
2. fabrication de copeaux PDMS
- Versez 90 g de polymère organique à base de silicium (polydiméthylsiloxane : PDMS) dans une tasse en plastique de 100 mL. Ajouter 10 g de son de salaison (01:10 en poids). Incorporer le mélange à l’aide d’une pipette en plastique pendant 2-3 min.
NOTE : Mélanger 90 g de PDMS avec 10 g de polymérisation agent pour la fabrication de 6 jetons. - Placer le mélange dans un dessiccateur à vide et la pompe pendant environ 30 min pour enlever les bulles d’air piégées dans le PDMS.
- Placer le moule principal dans une boîte de Pétri P100 et verser 15 mL du mélange sur le moule à l’aide d’une seringue.
NOTE : 15 mL mène à une hauteur totale de puce de 1,5 mm. - Placer la structure PDMS le moule dans un dessiccateur à vide et la pompe jusqu'à ce qu’il n’y a aucune bulle d’air plus éclatant à la surface de la PDMS.
Remarque : Cette étape prend environ 30 min. - Poussez la gaufrette au bas de la boîte de Pétri en utilisant un embout conique pour éviter le volume mort de PDMS au-dessous de la plaquette. Placez l’appareil de PDMS le moule dans le four réglé à 70 ° C pendant au moins 2 h.
- Demold le bloc PDMS sous une hotte chimique à l’aide d’une spatule plate en acier inoxydable et de l’isopropanol battante sur la puce. Placez-le sur le banc en acier inoxydable de la hotte.
- Coupez autour de la plaquette de silicium et demold la réplique PDMS à l’aide d’un scalpel.
- Découpé (congé a 2 mm de marge) la puce à l’aide d’un bistouri ou une lame de rasoir.
- Punch des entrées en appuyant fermement sur le perforateur de diamètre 1,5 mm et actionner pour couper et tailler le trou de l’entrée. Faire la même chose dans les quatre zones dédiées de la puce où liquide sera injecté.
- Nettoyer la puce par collage et épluchage de ruban adhésif sur le côté MICROSTRUCTURÉ. Isopropanol saupoudrer sur les deux côtés. Puis séchez la puce avec de l’azote sous pression à l’aide d’un pistolet de soufflage.
3. fabrication des lamelles (24 × 24 mm2)
NOTE : Manipuler les lamelles couvre-objet avec pincette courbée.
- Modèles poly-ornithine (OLP)
- Appliquer un O2 nettoyage plasma sur couvre-objet en verre. Paramètres du plasma : baisser la pression de pompage : 0,25 mbar ; O2 approvisionnement Durée : 3 min ; débit de gaz : 10 sccm ; déviation maximale : ± 5 sccm ; durée de plasma : 3 min ; Réglez la pression : 0,36 mbar ; déviation maximale : ±0, 10 mbar ; régler la puissance : 50 W ; déviation maximale : 5 % ; durée de la ventilation : 45 s.
- Mix 484 µL d’acide acétique et 56 µL de 3-methacryloxypropyl-trimethoxysilane, complet avec éthanol absolu pour obtenir un volume total de 15 mL.
- À l’aide d’une embase de 1 mL, mettez une goutte de 500 µL de cette solution sur chaque lamelle, attendre 2-3 min. séchez avec un chiffon microfibre de cleanroom.
NOTE : Lamelles de verre silanisée peuvent être conservés jusqu'à 1 mois à température ambiante dans des boîtes en plastique scellé avec un film plastique paraffine. - Placez chaque lamelle sur une coucheuse spin situé dans un environnement de salle blanche. Mettez une goutte d’un photosensible positive couvrant environ 75 % de la lamelle couvre-objet (environ 500 µL) et spin-manteau à 4000 tr/min pendant 30 s pour atteindre une épaisseur finale de 0,45 µm.
- Placer les lamelles pendant 1 min sur une plaque chauffante avec une température de 115 ° C.
- En utilisant un aligneur de masque, exposer chaque lamelle sur une longueur d’onde de 435 nm (ligne G) à travers le masque dédié selon les paramètres LABELTEX (dose UV environ 50-60 mJ.cm–1)
- Préparer 2 plats cristallisant en verre, un contenant le développeur (sans dilution), l’autre contenant l’eau désionisée.
- Plonger un par un chaque lamelle dans le développeur pendant 1 min tout doucement et continuellement en remuant le plat de cristallisation. Immerger la plaquette dans l’eau désionisée pendant environ 5 s. Place la plaquette sur un papier absorbant, puis sécher avec de l’azote sous pression à l’aide d’un pistolet de soufflage.
- Appliquer un plasma de2 activation O avec les mêmes paramètres qu’en 3.1.1.
- Sous le capot, déposer quatre 170 µL gouttes d’une solution à 100 µg/mL PLO par P100 Pétri. Mettre le visage modelé les lamelles sur chacune de ces gouttes. Sceller la boîte de Pétri avec une pellicule de paraffine en plastique pour éviter le séchage. Incuber une nuit à température ambiante.
Remarque : La solution de PLO doit rester attachée aux lamelles par capillarité. - Préparer quatre récipiendaires (typiquement P60 Pétri), trois d'entre eux remplir avec PBS et le quatrième à l’eau désionisée. Préparer deux plats en verre cristallisation d’éthanol pur.
- Sortez chaque lamelle de la boîtes de Pétri, il submerge dans le premier bain de PBS pour 10-15 s, évacuer le liquide sur un tissu absorbant en mettant la lamelle verticale sur le côté, insérez-le avec par exemple la face à motifs à l’intérieur de la baignoire de l’éthanol.
Remarque : Une fois la dissolution de la résine photosensible est terminée, il devient difficile de localiser le côté motif de la lamelle. Par conséquent, il est important à ce stade de retrouver son emplacement. - Placer l’éthanol cristallisant plat dans un sonicateur bain à ultrasons (120 W / 35 kHz) et laissez la résine photosensible dissous pendant 3 min.
Remarque : Changer le bain d’éthanol chaque 4 lamelles pour limiter la dilution par le PBS susceptible de nuire à la dissolution de la résine photosensible. - Sortent de la lamelle couvre-objet de la baignoire de l’éthanol, puis il plonger plusieurs fois dans le second bain de PBS. Vérifier l’aspect de surface à plusieurs reprises jusqu'à la surface graisseuse ressemblant qui disparaît des résultats du film liquid restant d’éthanol.
- Plonger pendant 5-10 s la lamelle dans la troisième bain de PBS. Puis immédiatement les transférer dans le bain d’eau désionisée. Placez le couvre-objet sur un papier absorbant et sécher avec de l’azote sous pression à l’aide d’un pistolet de soufflage.
Remarque : Le dernier rinçage à l’eau déminéralisée est utilisé pour éviter la formation de cristaux de PBS pendant l’étape de séchage.
- Structures de résine photosensible pour le calibrage de la hauteur
- Procédez uniquement comme 3.1.4. à 3.1.8. en utilisant le masque dédié (masque « Photoresist stripes », voir la fiche complémentaire « Mask_Photoresist-stripes.dxf »).
4. montage et finals d’exécution de chip
- Puce d’assemblage sur la vaisselle en verre bas
- Placer la puce PDMS et le plat en verre sur lequel il va être collé dans la chambre de plasma pour l’activation de la surface. Paramètres : arrêtant de pression : 0,25 mbar ; O2 approvisionnement Durée : 3 min ; débit de gaz : 10 sccm ; déviation maximale : ± 5 sccm ; durée de plasma : 30 s ; Réglez la pression : 0,40 mbar ; déviation maximale : ±0, 10 mbar ; régler la puissance : 50 W ; déviation maximale : 5 % ; durée de la ventilation : 45 s.
- Mettez doucement la puce PDMS activée au contact de la lamelle de verre et appliquer délicatement une pression sur les bords de la puce pour coller la puce de la lamelle. Pour augmenter la force de liaison, placez l’appareil dans le four à 70 ° C pendant 5 à 10 min.
Remarque : N’appuyez pas sur les pièces contenant des piliers, ils pourraient s’effondrer sous trop de pression. - Sous le capot (c'est-à-dire à la droite) et 30 min après collage, placez un cône de pointe 10 µL rempli d’une solution de 100 µg/mL PLO à l’IHs, puis injecter le liquide. Ajuster le volume pour former une goutte en haut de chaque IHs. Puis, à l’aide d’une embase de 1 mL, ajoutez des PBS dans la boîte de Pétri tout autour de la puce.
- Laissez la puce au RT avec un minimum de temps de 2 h d’incubation. Pour une nuit d’incubation, sceller la boîte de Pétri à l’aide d’un film de paraffine en plastique pour éviter le séchage.
Remarque : Le liquide ne doit pas fuir à l’extérieur de la puce sinon que la puce doit être jetée. - Appuyez légèrement sur un porte-cône 10 µL dans chaque IH et aspirer l’excès de liquide. Puis coller complètement le porte-cône à l’intérieur de la prise et dresser le liquide restant.
- Remplacer le PLO par laminine suit les instructions données aux étapes 4.1.5 (vidange) et 4.1.3 (remplissage). Incuber à RT pendant 1 h.
- Remplacer la laminine par le milieu de culture qui suit les instructions données aux étapes 4.1.5 (vidange) et 4.1.3 (remplissage). Composition du milieu de culture : MEM 81,8 % ; Pyruvate de sodium 100 mM 1 % ; Glutamax 200 mM 1 % ; Sérum de cheval 5 % ; B27 supplément de 2 %, N2 Supplément 1 %, gentamicine 0,2 % ; filtrer la solution à l’aide d’un filtre de 220 nm. À l’aide d’une embase de 1 mL, remplacer également le PBS entourant la puce de ce milieu.
- Mettre la puce dans l’étuve réglée à 37 ° C et 5 % CO2 pendant au moins 5 h (ou toute une nuit) avant de neurone ensemencement.
- Puce à assembler à l’aide de lamelles à motifs
Remarque : Si les lamelles à motifs incluent des objets de référence de résine photosensible positive, effectuez les opérations 4.2.1 à 4.2.9. Dans le cas contraire, effectuer seulement étape 4.2.3, bâton de l’appareil PDMS sur la lamelle de PLO à motifs comme indiqué au point 4.1.2,, mettre milieu de culture à l’intérieur et autour de la puce, puis passez à l’étape 4.2.10.- Déposer une goutte d’eau sur une lame de verre rectangulaire microscope épais et coller la lamelle sur la lame de verre par capillarité (côté non texturés face à la lame de verre). Sous un microscope, marquez l’emplacement des bandes résine photosensible à l’arrière de la lame de verre à l’aide d’un stylo feutre.
- Placez la lamelle de verre modelé sur le support de masque de l’aligneur de masque. S’appuyer sur la marque faite avec un stylo feutre pour centrer les objets de référence de la résine photosensible.
- Exécute l’étape de plasma comme décrit dans 4.1.1 sur la puce PDMS.
- Placez le jeton PDMS sur le porte-substrat mobile (chuck) de l’aligneur de masque.
Remarque : Pour augmenter le contraste optique, placer une tranche de silicium sous la puce PDMS. La plaquette de silicium doit rester fermement attachée sur le mandrin au cours du processus d’alignement (utilisez un ruban adhésif transparent pour le coller sur le mandrin). - Soulevez le mandrin à la limite de contact mécanique pour aligner la puce avec le tableau des bandes de résine photosensible situé sur la lamelle couvre-objet.
- Obtenir un contact mécanique entre la puce et la lamelle en soulevant le mandrin jusqu'à ce que la surface de la PDMS piliers touch la lamelle de verre pour terminer.
- Abaisser le mandrin. Retirer la lamelle maintenant collée à la puce de la titulaire de masque. Puis placer le dispositif dans un plat de Pétri de 35 mm et tout transférer dans le four (température : 70 ° C) pendant 5 à 10 min augmenter la force de liaison.
- Procéder comme au point 4.1.3.
Remarque : si à l’aide de PLO à motif lamelles couvre-objet, aller directement à l’étape 4.2.7 revenir 4.2.9. - Remplacer PLO par le placage moyen qui suit que les procédures décrites aux étapes 4.1.5 (vidange) et 4.1.3 (remplissage). À l’aide d’une embase de 1 mL, remplacer également le PBS entourant la puce de ce milieu.
- Mettre la puce dans l’étuve réglée à 37 ° C et 5 % de CO2 jusqu’au neurone semis, avec un minimum de temps de 5 h d’incubation.
5. culture de neurones
- Préparer 100 mL de milieu (milieu HH) de la dissection en mélangeant 10 mL de HBSS 10 x, 2 mL de Hepes 1 M et 88 mL d’eau stérile dans un flacon en plastique de 200 mL.
NOTE : Le support HH peut être préparé la veille de la culture. - Disséquer hippocampe d’embryon de souris E18 extraite du mère mis à mort par dislocation cervicale (souris C57BL/6J de Charles Rivers). Étapes de la dissection sont par exemple détaillé dans 16.
- Placer hippocampe dans un tube en plastique contenant de la trypsine (0,3 mL de trypsine 2,5 %, sans EDTA dans 2,7 mL de milieu HH) pendant 10 min à 37 ° C afin d’initier la dissociation chimique.
- Jeter presque tout le liquide et le remplacer par 5 à 10 mL de HH à l’aide de pipettes en plastique jetables. Faites-le 3 fois. Pour le dernier remplissage, utiliser 1 mL de milieu au lieu de HH de placage.
- Mécaniquement, se dissocient les tissus avec une embase de 1 mL en aspirant et éjecter le volume complet plusieurs fois, en évitant de faire des bulles et en utilisant les passages de pas plus de 15-20.
- Dans un récipient séparé de 500 µL, préparer une solution à l’aide de 5 µL de suspension cellulaire diluée dans 45 µL de PBS. Prendre 1 µL de cette solution à l’aide d’une pipette µL 10 et insérez la suspension diluée dans un compteur de cellules de Malassez. Les indications fournies dans 17 permet d’estimer le nombre de cellules.
Remarque : Un seul hippocampe offre habituellement environ 0,5 millions de neurones. - Centrifuger à 100 x g pendant 6 min à température ambiante.
- Jeter le surnageant et remplacez-le par le volume de milieu de placage pour atteindre une concentration de 10 millions de cellules/mL. Remettre les cellules par successivement aspirent et éjecter la suspension cellulaire avec une embase de 1 mL.
- Dresser le médium placage présent dans la puce (reportez-vous à l’étape 4.1.5). Prélever 2-3 µL de la solution fraîchement extrait à l’aide d’une pipette de 10 µL et y injecter à l’entrée (reportez-vous à la procédure d’injection décrite à l’étape 4.1.3). Répéter immédiatement la même opération à la sortie.
- Injecter environ le même volume de milieu de placage dans chaque réservoir (reportez-vous à l’étape 4.1.3).
Remarque : Observer rapidement la puce au microscope pour vérifier la densité des cellules. L’ordre de grandeur de la densité cellulaire optimale correspond à environ 5 à 10 cellules au sein de la surface carrée délimité par 4 piliers (0,3 mm2, c'est-à-dire de 15 à 20 cellules / mm2). - Finit par répéter l’étape 5.10, utilisez à la place de 0,5 à 1 µL de la suspension cellulaire pour atteindre la densité des cellules ciblées.
- Placer la puce ensemencée dans une étuve réglée à 37 ° C 5 % CO2.
6. observation d’exclusion fluorescence
- Remplacement du milieu de culture par le moyen d’imagerie.
- Préparer le milieu d’imagerie comme 4.1.7 mais utilisez plutôt MEM dépourvue de rouge de phénol et ajouter dextran fluorescent. Dans ce but, diluer dextran (poids moléculaire 10 000 g/mol, solution mère concentrée à 10 mg/mL dans du PBS) afin d’obtenir une concentration finale de 0,5 à 1 mg/mL dans le milieu de l’imagerie.
Remarque : Utilisation Dextran se conjugue avec des maxima d’Absorption/Emission 496/524 ou 650/668. Préférez la première image de 0,45 µm photosensible positive élevée structures (pour se débarrasser de leur fluorescence auto dans la largeur de bande rouge) et le second aux neurones d’image (moins toxiques). - Vider toutes les entrées à l’aide d’une pipette de 10 µL et les re-remplir complètement avec le milieu de l’imagerie (se référer aux étapes 4.1.3 et 4.1.5 pour la méthodologie précise de remplacement moyen).
- Préparer le milieu d’imagerie comme 4.1.7 mais utilisez plutôt MEM dépourvue de rouge de phénol et ajouter dextran fluorescent. Dans ce but, diluer dextran (poids moléculaire 10 000 g/mol, solution mère concentrée à 10 mg/mL dans du PBS) afin d’obtenir une concentration finale de 0,5 à 1 mg/mL dans le milieu de l’imagerie.
- Imagerie
- Placez le jeton sous un microscope à épifluorescence équipé d’une chambre environnementale réglementée à 37 ° C et 5 % de CO2. Utiliser un 40 X, ouverture numérique (NA) 0,8 sec objective, 30 % de la puissance totale (pleine puissance : 3 W) et 30 ms de temps d’exposition. Acquisition d’images de cellules au foyer (à partir de célibataire à plusieurs images successives dans le cas d’expériences Time-lapse).
- Analyse d’images
- Normaliser les images à l’aide de la routine de réduction de fond mis en œuvre dans un logiciel dédié pour obtenir un fond homogène. Voir 13 pour le détail des mesures incluses dans ce logiciel de traitement d’image. A l’image de sortie une. Format de MAT.
- Convert. Fichier MAT en. Images TIFF 8 bits à l’aide de la routine mises dans des documents supplémentaires (conversion_mattotiff.m, qui appelle à la importfilevol.m).
- Effectuer l’importation > séquence d’images sur ImageJ pour construire une vidéo de la. Images au format TIFF.
- Calculer l’intensité moyenne P d’une zone carrée localisée dans le centre d’un pilier (objet reference) et l’intensité moyenne B d’une zone carrée de la chambre dépourvue de cellules (fond, c'est-à-dire la hauteur zéro). Voir 18 pour obtenir un exemple de méthodologie détaillée du traitement de l’image.
NOTE : La dimension latérale des zones carrées utilisées comme références d’intensité doivent être près de la moitié du diamètre du pilier pour obtenir un nombre suffisant de pixels tout en évitant la pollution lumineuse sur les bords de la borne. - Utilisez les valeurs de P et B pour établir la Loi de conversion linéaire de l’intensité j’ai à hauteur h:

avec hc la hauteur connue de la chambre, et
et
Remarque : PDMS n’affiche aucun autofluorescence détectable. - Sélectionnez une zone autour de la zone d’intérêt, intégrer l’intensité à l’aide de ImageJ (voir 19 pour plus de détails) et appliquer la Loi de conversion obtenue en 6.3.5 pour mesurer le volume d’un compartiment de la cellule.
Remarque : La zone d’intérêt pourrait être sélectionnée basée sur la fluorescence des éléments subcellulaires comme des cônes d’actine, par exemple, pour le choix de la croissance dans les neurones de GFP-LifeAct, sélectionnez la zone d’intérêt dans le canal d’émission de GFP, enregistrer le contour de ce zone à l’aide d’un outil de gestionnaire de ROI, puis mesure le volume cellulaire enfermé dans la même zone dans le canal d’émission de dextran (en rouge).
Résultats
Le résultat du processus de fabrication décrite dans les sections 1 et 2 est illustré par les images de la Figure 1 a-1 b et la courbe de la Figure 1. Le tableau de la Figure 1 affiche les valeurs de rugosité de deux différentes zones représentatives de la puce PDMS, c'est-à-dire dans la centrale et la chambre intermédiaire suivante haut de 20 µm. Une diminution de la rugosité d’un facteur d’environ 7 a été obtenue à l’aide de gravé wafer Si au lieu de photorésine SU-8. Ensuite, FXm a été tout d’abord appliqué sur une bande de résine photosensible de géométrie connue (Figure 2 a) dans une chambre haute de 10 µm. Après traitement de l’image et l’intensité à la conversion de hauteur (voir le graphique de la Figure 2 b), profils de FXm effectuées sur des coupes transversales le long de cette bande (Figure 2) fournissent les profils de la hauteur désirée (Figure 2D). Figure 2D montre la comparaison entre les profils obtenus à l’aide de profilométrie mécanique et méthodes FXm. Ces profils, y compris la valeur de bord et du plateau, sont très semblables, validation de la méthode. Notez que la dispersion des données FXm n’est pas représentative de la résolution ultime de la méthode, tel qu’évaluée dans la Figure 3 et Figure 4, mais découle de la faible intensité employées pour éviter un éventuel effet de la très faible auto-fluorescence de la résine photosensible dans le chenal GFP.
Ensuite, nous avons observé des neurites à 3 µm et 10 chambres haute µm (Figure 3). L’écart du bruit de fond est à environ 18 nm après l’intensité à la conversion et fond la correction de hauteur. Cette valeur est légèrement supérieure à la rugosité physique des surfaces PDMS coulées sur des surfaces de silicium (12 nm, consultez le tableau de la Figure 1) mais beaucoup plus faible que la rugosité mesurée sur PDMS obtenus à partir des moules de SU-8. Ces résultats mettent en évidence la valeur ajoutée de forage de puits dans les plaquettes de silicium au lieu d’ouvrir des trous dans la résine photosensible SU-8 à monter des piliers. Une telle valeur faible permet un haut bruit rapport signal et des images très claires en volume, telles que celle qui est affichée dans la Figure 3 a. Par exemple des données qui peuvent être extraites de ces images, nous avons calculé le volume de 1,6 µm (c.-à-d. 10 pixels) large neurites tranche (voir le graphique de la Figure 3 b). En utilisant dans une première approximation à un ajustement linéaire de ces données donne une valeur de hauteur de neurites moyenne d’environ 400 nm, à comparer avec par exemple le diamètre axonal 500 nm trouvé en 10 jours les petits vieux dans le corps calleux 5. Nous avons également combiné FXm avec micropatterns d’adhérence consistant en série est attenant 2 µm et 6 µm de large bandes de 30 µm de longueur. Notre but était d’étudier l’influence de la largeur des neurites sur sa forme 3D. La figure 3 montre une représentation 3D en fausses couleurs d’une image de neurone tout obtenue dans une chambre haute de 10 µm. Neurites se répandent sur 2 µm et 6 µm larges rayures, tandis que le soma est situé à l’extrémité de la bande plus importante. Profils de hauteur ont été prélevés dans trois sections différentes de la Croix. En cohérence avec le graphique affiché dans la Figure 3 a, la surface intégrées sur l’augmentation de coupes avec la largeur de neurites (Figure 3D).
Aussi, nous nous sommes concentrés sur les structures 3D de croissance cône (GC). Figure 4 A-B affiche deux profils GC différents obtenus dans une chambre forte à 3 µm, qui mettent en valeur leur sous-structure ramifié. En outre, nous avons effectué des expériences en Time-lapse pour suivre la dynamique du volume des catalogues globaux dans une chambre haute de 12 µm. La figure 4 affiche un cycle de rétrécissement et la réactivation d’un GC donnée dans un délai de quelques dizaines de minutes. Grâce à l’utilisation de souris GFP-lifeact, les cônes de croissance ont été localisées dans la longueur d’onde d’émission de GFP (510 nm) de leur concentration élevée d’actine. La surface identifiée à la longueur d’onde était utilisée pour intégrer sur la longueur d’onde d’émission de dextran à 647 nm pour calculer le volume de GC. La figure 4 montre enfin la distribution du volume du GC à des moments différents et l’emplacement sur trois différents neurones, centrés sur une valeur d’environ 6 µm3.

Figure 1 : chambres de FXm PDMS. (A) les plans des quatre principales étapes différentes de microfabrication menant au moule final. L’emplacement de l’entrée, de sortie et de réservoirs sont indiqués. Barreaux de l’échelle : 1 mm. (B) Image de la chambre de PDMS FXm obtenue à l’aide d’un profilomètre optique. Cette image montre la chambre centrale contenant 3 rangées de 10 µm haute piliers et les chambres intermédiaires de 20, 50 et 90 µm de hauteur. Echelle : 500 µm. (C) vue en coupe de la puce sur les deux lignes pointillées tracées au point B. Or/jaune : section transversale sur piliers, bleus : section transversale entre les piliers. (D) signifie que les valeurs de la rugosité PDMS mesurée sur 50 × 50 µm2 zones moulés sur silicium et sur la chambre intermédiaire de la grande SU-8 de 20 µm (voir flèches pour l’emplacement de ces zones). Valeurs moyennes ont été prélevés les mesures des trois domaines différents. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
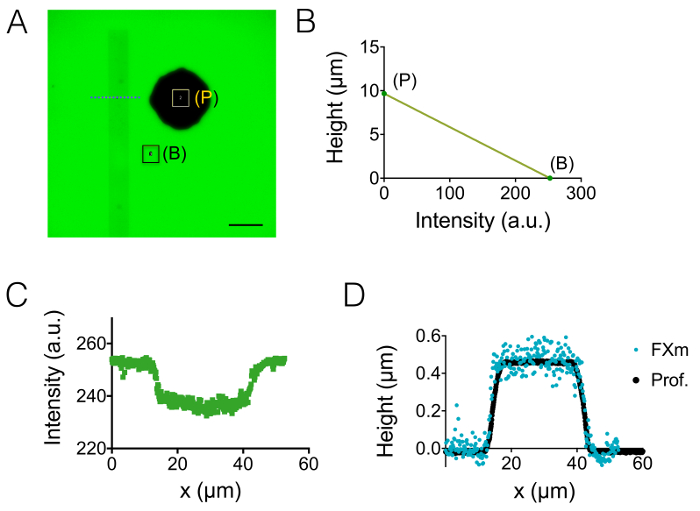
Figure 2 : étalonnage de FXm méthode utilisant une bande de résine photosensible comme objet d’intérêt. (A) image de GFP-fluorescence prise dans une chambre haute de 10 µm remplie de 10 000 dextran MW absorbant à 488 nm à 1 mg/mL. (B: fond, P: pilier). Observation avec un objectif de NA X 0,8 40 sec. Barre d’échelle : 50 µm. (B) Loi d’étalonnage linéaire provenant de l’intensité moyenne des deux rectangles colorés montré dans le profil d’intensité de Fluorescence de (C) a. obtenu au niveau de la ligne bleue pointillée affiché (a), traversant la résine photosensible rayure (0,45 µm haute positive photosensible). (D) comparaison des profils obtenus de mécanique profilomètre (points noirs) et FXm après l’intensité à la conversion de la taille des données de (B) (points bleus). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
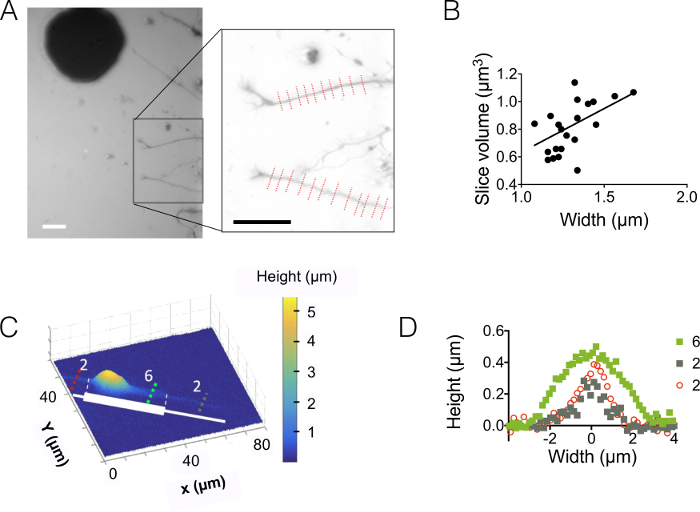
Figure 3 : imagerie volume neurites. (A) neurites s’étendant dans la chambre haute centrale de 3 µm de soma situé dans la chambre intermédiaire ensuite 15 µm. Imagerie effectuée à l’aide de 10 000 dextran MW absorbant à 488 nm et un 40 X, NA 0,8 sec objectif. L’encart obtenu après que l’utilisation de la routine de réduction fond met en évidence les deux neurites et choisi pour tracer le graphique du côté droit. Barreaux de l’échelle : 30 µm. (B) neurites tranche volume en fonction de la largeur des neurites calculé à partir du 22 profils (moyenne sur 10 pixels, c'est-à-dire sur une « tranche de neurites » de 1,6 µm) montrés en (A). La ligne continue représente un ajustement linéaire de pente 0,4 µm en passant par l’origine. (C) image fausses couleurs d’un neurone à motifs sur une bande adhésive fait de successives de 2 µm et souches large de 6 µm (représentées en blanc). Les mesures ont été effectuées dans une chambre haute de 10 µm, remplie de 10 000 dextran MW absorbant à 647 nm et à l’aide d’un NA 40 x 0,8 sec objectif. (D) profils de hauteur correspondant aux couleurs lignes pointillées illustrés (C), conservant le même code de couleur. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 4 : l’imagerie statique et dynamique de croissance cône. (A-B) Profils de hauteur du cône croissance obtenues dans une enceinte haute de 3 µm après l’intensité à la conversion de hauteur le long des lignes jaunes affiché dans les images associées. Observation, effectuée à l’aide d’un remplissage avec 10 000 dextran MW absorbant à 488 nm et un 40 X, NA 0,8 sec objectif. (C) imagerie de neurone ensemble dans une chambre haute de 12 µm remplie de 10 000 dextran MW absorbant à 647 nm. Observations ont été faites dans les deux canaux fluorescent : GFP de localisation de cône de croissance (pointillés jaunes) et CY5 pour calculer le volume de GC de l’exclusion de la fluorescence. La surface incluse par des pointillés jaunes a été utilisée pour calculer le volume de GC. Le graphique présente la variation du volume de la GC dans le temps et associé de morphologies fois GFP et CY5 canaux à deux moments différents représentatifs. Toutes les données ont été recueillies à l’aide d’un NA 40 x 0,8 sec objectif chaque barres d’échelle de 3 min. : 10 µm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
| Étape | Masquer la couche de 1:8 µm | Masque à 02:30 µm de couche | Masque 03:40 µm de couche |
| Type de SU-8 | 2007 | 2025 | 2050 |
| Spincoating | 30 s @ 2000 tr/mn | 30 s à 3050 tr/min | 30 s à 3250 tr/min |
| Cuire au four doux | 3 min à 95 ° C | 2 min à 65 ° C + 6 min à 95 ° C | 3 min à 65 ° C + 7 min à 95 ° C |
| Exposition à l’énergie | 110 mJ/cm2 | 155 mJ/cm2 | 170 mJ/cm2 |
| Cuire au four après l’exposition | 4 min à 95 ° C | 1 min à 65 ° C + 6 min à 95 ° C | 2 min à 65 ° C + 7 min à 95 ° C |
| Développement | 2 min 30 s | 5 min | 6 min |
| Cuire dur (facultatif) | 3-5 min à 200 ° C | 3-5 min à 200 ° C | 3-5 min à 200 ° C |
Tableau 1 : étapes de photolithographie effectuées pour construire un appareil contenant une chambre centrale de 12 μm en hauteur. Hauteurs des chambres intermédiaires : 20, 50 et 90 µm.
| Étape | Masque à 01:10 µm de couche | Masque à 02:30 µm de couche | Masque 03:40 µm de couche |
| Type de SU-8 | 2007 | 2025 | 2050 |
| Spincoating | 30 s à 1 500 tr/min | 30 s à 3050 tr/min | 30 s à 3250 tr/min |
| Cuire au four doux | 3 min à 95 ° C | 2 min à 65 ° C + 6 min à 95 ° C | 3 min à 65 ° C + 7 min à 95 ° C |
| Exposition à l’énergie | 125 mJ/cm2 | 155 mJ/cm2 | 170 mJ/cm2 |
| Cuire au four après l’exposition | 4 min à 95 ° C | 1 min à 65 ° C + 6 min à 95 ° C | 2 min à 65 ° C + 7 min à 95 ° C |
| Développement | 2 min 30 s | 5 min | 6 min |
| Cuire dur (facultatif) | 3-5 min à 200 ° C | 3-5 min à 200 ° C | 3-5 min à 200 ° C |
Tableau 2 : étapes de photolithographie effectuées pour construire un appareil contenant une chambre centrale de 10 μm en hauteur. Hauteurs des chambres intermédiaires : 20, 50 et 90 µm.
| Étape | Masque à 01:12 µm de couche | Masque à 02:32 µm de couche | Masque 03:40 µm de couche |
| Type de SU-8 | 2015 | 2025 | 2050 |
| Spincoating | 30 s à 3250 tr/min | 30 s à 2 500 tr/min | 30 s à 3250 tr/min |
| Cuire au four doux | 3 min à 95 ° C | 2 min à 65 ° C + 5 min à 95 ° C | 3 min à 65 ° C + 7 min à 95 ° C |
| Temps d’exposition | 140 mJ/cm2 | 157 mJ/cm2 | 170 mJ/cm2 |
| Cuire au four après l’exposition | 4 min à 95 ° C | 1 min à 65 ° C + 5 min à 95 ° C | 2 min à 65 ° C + 7 min à 95 ° C |
| Développement | 3 min | 5 min | 6 min |
| Cuire dur (facultatif) | 3-5 min à 200 ° C | 3-5 min à 200 ° C | 3-5 min à 200 ° C |
Tableau 3 : étapes de photolithographie effectuées pour construire un appareil contenant une chambre centrale de 3 μm à hauteur de. Hauteurs des chambres intermédiaires : 18, 50 et 90 µm.
Données supplémentaires 1 : masks_neuron_volume_chips.tiff. Vue schématique des masques utilisés pour fabriquer le dispositif PDMS (DRIE masque et masque 1-3). S’il vous plaît cliquez ici pour télécharger ce fichier.
Des données supplémentaires 2 : file « masks_neuron_volume_chips.dxf ». Fichiers électroniques permettant de fabriquer le masque DRIE et masques 1-3. S’il vous plaît cliquez ici pour télécharger ce fichier.
Des données supplémentaires 3 : « Mask_Photoresist-stripes.dxf ». Fichiers électroniques permettant de fabriquer le masque utilisé pour la photolithographie de bandes de résine photosensible. S’il vous plaît cliquez ici pour télécharger ce fichier.
Des données supplémentaires 4 : file conversion_mattotiff.m s’il vous plaît cliquez ici pour télécharger ce fichier.
Des données supplémentaires 5 : file importfilevol.m s’il vous plaît cliquez ici pour télécharger ce fichier.
Discussion
L’imagerie des neurones de volume constitue un défi pour la technique de FXm en raison les extensions longues et minces de ces cellules. Ce protocole décrit des variantes du même type de dispositif microfluidique dédié à l’imagerie de neurone.
Outre les aspects de la conception de la microfluidique, le choix de l’objectif est fondamental pour l’imagerie de fluorescence exclusion et implique un compromis entre la résolution latérale et la clarté de l’image. Il a été démontré en 13 qu’un NA élevée conduisant à une profondeur de champ plus petit que la hauteur de la chambre n’était pas préjudiciable pour la précision de mesure du volume si l’imagerie s’est déroulée au foyer et si n’est pas une marge suffisante entre le contour de l’objet d’i intérêts et les limites de la surface de l’intégration. Cependant, l’utilisation d’une chambre beaucoup plus élevée que la profondeur de champ porte atteinte à la clarté de l’image en raison de la diffusion de photon, qui adoucit les bords des objets d’intérêt. La fabrication d’une chambre haute de 3 µm réduit ce flou latéral et fourni des images exclusion fluorescent exceptionnellement bien définies, même en utilisant NA haute (0,8) 40 X objectifs pour visualiser les branches neuronales avec haute résolution latérale.
Montage de puce est une étape essentielle, en particulier dans le cas des chambres haut de 3 µm, mais attention manipulation comme décrit au point 4.1.2, permet d’éviter l’effondrement du toit. La surface élevée au rapport de volume lié à ces chambres minces a également soulevé la question de la stabilité de la concentration de Dextran au fil du temps. Nous avons vérifié que l’absorption de surface du Dextran après une nuit d’incubation était négligeable : après avoir remplacé Dextran par PBS, la différence d’intensité entre le pilier et l’arrière-plan était environ 1 pour 1000 du contraste entre intensité initiale ces deux régions en présence de Dextran. Notez que les neurones peuvent adhérer sur la lamelle inférieure et sur le toit PDMS. Cet effet disparaît lorsqu’à l’aide de motifs lamelles couvre-objet (c'est-à-dire lorsque nous incuber pas adhésifs molécules au sein de la chambre PDMS), le revêtement est donc strictement localisée sur le fond de la chambre.
En dehors de leur morphologie difficile, les neurones sont plutôt adaptés à FXm due au fait que l’un de la limitation majeure de la méthode, c'est-à-dire l’endocytose dextran, est très limité dans ces cellules. Nous choisissons un 10 kDa formulation pour supprimer à long range (heures) tout phénomène d’endocytose visible.
En conclusion, la simplicité conceptuelle de FXm est équilibrée par un ensemble de questions expérimentales qui ont été résolus par le présent protocole, tels que la rugosité nanométrique de PDMS et micrométrique chambre au sol ou une correction de fond afin de corriger l’inégalité du le plafond de PDMS entre les piliers. Cependant, l’utilisation d’une chambre étroite microfluidique de confiner le milieu fluorescent donne quelques contraintes spécifiques comme le besoin de piliers de soutien, qui abaisse la surface efficace disponible pour l’adhésion cellulaire, ou la nécessité d’exclure le soma de la centrale chambre d’observer des extensions neuronales avec la clarté plus élevée, qui restreint les régions de la cellule accessible à l’observation de haute résolution. Une évolution possible de cette méthode serait de se débarrasser de ce confinement physique, pour être remplacée par une autre optique. Le nouveau développement de la microscopie de nappe de lumière peut-être être combiné avantageusement avec FXm dans l’avenir.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Les auteurs veulent remercier ChiLab, matériaux et laboratoire de microsystèmes - Politecnico di Torino - DISAT, en la personne du professeur C F Pirri, Dr M Cocuzza et Dr S L Marasso, pour leur aide précieuse dans le processus de développement et la fabrication de dispositifs. Nous remercions Victor Racine de Quantacell de discussion et de soutien dans le traitement de l’image. Nous sommes reconnaissants à Isabelle Grandjean et Manon Chartier de la facilité de l’Animal de l’Institut Curie pour leur soutien à la souris et Pablo Vargas et de Ana-Maria Lennon (Institut Curie) pour nous avoir fourni les souris GFP LifeAct. Nous sommes reconnaissants à Olivier Thouvenin Institut Langevin et Clotilde Cadart, Larisa Venkova et Matthieu Piel de l’Institut Curie - UMR 144, pour leur aide dans la compréhension de l’eXclusion de Fluorescence méthode. Enfin, nous remercions la plate-forme technologique de l’Institut Pierre-Gilles de Gennes (CNRS UMS 3750) pour leur soutien dans la microfabrication. Ce travail a été soutenu en partie par l’européen recherche Conseil Advanced Grant no 321107 « Violoncelle », PSL Université (projet SwithNeuroTrails), avenir de l’Investissement de l’ANR et les IPGG Labex Equipex.
matériels
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| Plasmalab System 100 | Oxford Instruments | To perform DRIE | |
| MJB4 mask aligner | SUSS MicroTec | SU-8 photolithography | |
| NXQ 4006 Mask aligner | Neutronix Quintel | Photolithography associated to DRIE | |
| Plasma cleaner | Diener Electronic | Pico PCCE | |
| Cell culture hood | ADS Laminaire | Optimale 12 | |
| Centrifuge | Thermo Fisher Scientific | Heraeus Multifuge X1R | |
| Incubator 37 °C 5% CO2 | Panasonic | MCO-170AICUVH-PE IncuSafe CO2 Incubator | |
| Epifluorescence microscope | Leica | DMi8 | |
| PDMS Oven between 65 and 80 °C | Memmert | ||
| Mechanical profilometer | Veeco | Dektak 6M Stylus Profiler | |
| Optical profilometer | Veeco | Wyko NT9100 | |
| Vacuum desiccator | Verrerie Villeurbanaise (Kartel Labware) | 230KAR | Diameter 200 mm |
| Ultrasonic bath sonicator | Labo Moderne | SHE1000 | Volume of the bath: 0.8 liter |
| Hotplate | Stuart SD162 | SD162 | |
| Masks | |||
| DRIE | Supplementary data | ||
| Su8 Mask 1 | Supplementary data | ||
| Su8 Mask 2 | Supplementary data | ||
| Su8 Mask 3 | Supplementary data | ||
| S1805 calibration stripes | Supplementary data | ||
| Small laboratory equipment | |||
| 1.5 mm hole puncher | Sigma-Aldrich | 29002519 (US reference) | |
| Scalpel or razor blade | |||
| 9" Stainless Steel Flat Spatula with Spoon | VWR International | 82027-532 | To demold PDMS |
| Top Lip Wafer Handling | VWR International | 63042-096 | |
| Curved tweezer | FST | Dumont #7 Forceps - Standard / Dumoxel | To manipulate glass coverslips |
| Substrates | |||
| Silicon wafer | Prolog Semicor Ltd | ||
| 24x24 mm glass coverslips | VWR | 631-0127 | |
| Photoresists and developpers | |||
| AZ 1518 positive photoresist | Microchemicals GmbH | Before DRIE process, thicknes 1.8 µm | |
| AZ 351B developer | Microchemicals GmbH | To develop positive AZ 1518 photoresist | |
| SU-8 2007 | MicroChem | ||
| SU-8 2025 | MicroChem | ||
| SU-8 2050 | MicroChem | ||
| PGMA developer | Technic | To develop SU-8 negative photoresist | |
| Microposit S1805 resist | Chimie Tech Services | Positive photoresist used to obtain 0.5µm high structures | |
| MF 26A developer | Chimie Tech Services | To develop positive S1805 photoresist | |
| Laboratory consumables | |||
| Disposable plastic pipette 3 mL | LifeTechnologies - ThermoFisher | ||
| P100 Petri dishes | TPP | 93100 | |
| 20 mL syringe | Terumo | SS+20ES1 | |
| Transparent scotch tape | |||
| Square wipes | VWR | 115-2148 | |
| Parafilm | DUTSCHER | 90260 | Plastic paraffin film |
| Chemicals | |||
| (3-Methacryloxypropyl)trichlorosilane | abcr | AB 109004 | |
| PDMS and curing agent Sylgard 184 | Sigma-Aldrich | 761036 | |
| isopropanol | W292907 | ||
| poly-ornithine | Sigma-Aldrich | P4957 - 50 mL | |
| Ethanol absolute | Sigma-aldrich | 02865 | 99.8% |
| 3-methacryloxypropyl-trimethoxysilane | Sigma-aldrich | M6514-25ML | (C4H5O2)-(CH2)3- Si(OCH3)3 |
| acetic acid | Sigma-aldrich | 71251-5ML-F | |
| Dextran 10kW conjugated with Alexa488 | LifeTechnologies - ThermoFisher | D22910 | Absoprtion at 488 nm |
| Dextran 10kW conjugated with Alexa647 | LifeTechnologies - ThermoFisher | D22914 | Absoprtion at 647 nm |
| Culture medium | |||
| MEM | LifeTechnologies - ThermoFisher | 21090-022 | |
| Horse Serum | LifeTechnologies - ThermoFisher | 26050088 | |
| B27 | LifeTechnologies - ThermoFisher | 12587-010 | |
| Glutamax 200 mM | LifeTechnologies - ThermoFisher | 35050-061 | |
| Sodium Pyruvate GIBCO 100 mM | LifeTechnologies - ThermoFisher | 11360-070 | |
| Gentamicin | LifeTechnologies - ThermoFisher | 15710-049 | |
| PBS | Sigma-Aldrich | D8537-500ML | |
| HBSS 10x | LifeTechnologies - ThermoFisher | 14180-046 | |
| Hepes 1M | LifeTechnologies - ThermoFisher | 15630-056 | |
| trypsin-EDTA | Sigma-Aldrich | 59418C-100ML | |
| Neurobasal | LifeTechnologies - ThermoFisher | 21103-049 | |
| Neurobasal without phenol red | LifeTechnologies - ThermoFisher | 12348-017 | |
| Softwares | |||
| Routine in Matlab for background normalization | Quantacell | Contact Victor Racine: victor.racine@quantacell.com | |
| ImageJ | To select specific ROI for image analysis | ||
| Routine | ImageJ | Supplementary data | |
| Routine | Matlab | Supplementary data |
Références
- Jun, S., Taheri-Araghi, S. Cell-size maintenance: universal strategy revealed. Trends Microbiol. 23 (1), 4-6 (2015).
- Zlotek-Zlotkiewicz, E., Monnier, S., Cappello, G., Le Berre, M., Piel, M. Optical volume and mass measurements show that mammalian cells swell during mitosis. J. Cell Biol. 211 (4), 765-774 (2015).
- Kim, G. H., Kosterin, P., Obaid, A. L., Salzberg, B. M. A mechanical spike accompanies the action potential in Mammalian nerve terminals. Biophys J. 92 (9), 3122-3129 (2007).
- Iacono, D., et al. Neuronal Hypertrophy in Asymptomatic Alzheimer Disease. J Neuropathol Exp Neurol. 67 (6), 578-589 (2008).
- Cheli, V. T., et al. Conditional Deletion of the L-Type Calcium Channel Cav1. 2 in Oligodendrocyte Progenitor Cells Affects Postnatal Myelination in Mice. J Neurosci. 36 (42), 10853-10869 (2016).
- Kneynsberg, A., Collier, T. J., Manfredsson, F. P., Kanaan, N. M. Quantitative and semi-quantitative measurements of axonal degeneration in tissue and primary neuron cultures. J Neurosci Methods. 266, 32-41 (2016).
- Fouquet, C., et al. Improving axial resolution in confocal microscopy with new high refractive index mounting media. PloS one. 10 (3), (2015).
- Aguet, F., et al. Membrane dynamics of dividing cells imaged by lattice light-sheet microscopy. Mol Biol Cell. 27 (22), 3418-3435 (2016).
- Roland, A. B., et al. Cannabinoid-induced actomyosin contractility shapes neuronal morphology and growth. eLife. 3, (2014).
- Lu, Y. B., et al. Viscoelastic properties of individual glial cells and neurons in the CNS. Proc Natl Acad Sci USA. 103 (47), 17759-17764 (2006).
- Chen, J., Xue, C., Zhao, Y., Chen, D., Wu, M. H., Wang, J. Microfluidic impedance flow cytometry enabling high-throughput single-cell electrical property characterization. Int J Mol Sci. 16 (5), 9804-9830 (2015).
- Bottier, C., et al. Dynamic measurement of the height and volume of migrating cells by a novel fluorescence microscopy technique. Lab Chip. 11, 3855-3863 (2011).
- Cadart, C., et al. Fluorescence eXclusion Measurement of volume in live cells. Methods Cell Biol. 139, 103-120 (2017).
- Marasso, S. L., et al. A novel graphene based nanocomposite for application in 3D flexible micro-supercapacitors. Materials Research Express. 3 (6), 065001 (2016).
- Welch, C. C., Goodyear, A. L., Wahlbrink, T., Lemme, M. C., Mollenhauer, T. Silicon etch process options for micro-and nanotechnology using inductively coupled plasmas. Microelectronic Engineering. 83 (4), 1170-1173 (2006).
- Fath, T., Ke, Y. D., Gunning, P., Götz, J., Ittner, L. M. Primary support cultures of hippocampal and substantia nigra neurons. Nat. Protoc. 4 (1), 78-85 (2008).
- Getting intensity values from single ROI. Image Intensity Processing Available from: https://imagej.net/Image_Intensity_Processing (2017)
- 19. Tools. ImageJ User Guide Available from: https://imagej.nih.gov/ij/docs/guide/146-19.html (2017)
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon
Nous utilisons des cookies afin d'améliorer votre expérience sur notre site web.
En continuant à utiliser notre site ou en cliquant sur le bouton ''continuer'', vous acceptez l'utilisation de cookies.