Method Article
Alta resolución volumen imágenes de neuronas por la exclusión de uso de fluorescencia método y dispositivos microfluídicos dedicada
En este artículo
Resumen
Volumen es un parámetro importante en cuanto a las características fisiológicas y patológicas de las células. Se describe un método de exclusión fluorescente que permite medición de campo completo en vitro volumen neuronal con sub-micrométrica resolución axial necesaria para el análisis de neuritas y estructuras dinámicas implicadas en el crecimiento neuronal.
Resumen
Volumen es un parámetro importante en cuanto a características fisiológicas y patológicas de las neuronas en diferentes escalas de tiempo. Las neuronas son células bastante singular en cuanto a su morfología ramificada extendida y en consecuencia plantear algunos retos metodológicos para la medición de volumen. En el caso particular de en vitro crecimiento neuronal, la metodología solicitada debe incluir sub-micrométrica resolución axial combinada con la observación de campo completo en escalas de tiempo de minutos a horas o días. A diferencia de otros métodos como en la reconstrucción de la forma de la célula usando proyección de imagen confocal, medidas eléctricamente o microscopía de fuerza atómica, el recientemente desarrollado método de exclusión de fluorescencia (FXm) tiene el potencial para cumplir con estos desafíos. Sin embargo, aunque es simple en su principio, aplicación de una alta resolución FXm para neuronas requiere ajustes múltiples y una metodología dedicada. Aquí presentamos un método basado en la combinación de la exclusión de fluorescencia dispositivos microfluídicos múltiples compartimientos de baja rugosidad y finalmente micropatterning para lograr en vitro las medidas de volumen neuronal local. El proporcionado por el dispositivo de alta resolución nos permite medir el volumen local de procesos neuronales (neuritas) y el volumen de algunas estructuras específicas involucradas en el crecimiento neuronal, tales como conos de crecimiento (GCs).
Introducción
El conocimiento preciso del volumen celular ha atraído mayor atención en los últimos años, impulsado por el tema de la homeostasis del tamaño celular en solos-celled microorganismos 1 y en general en las células mitotic 2. Sin embargo, es pertinente también para las células post mitóticas, para que las neuronas constituyen un ejemplo paradigmático la cuestión del volumen de la célula.
Volumen es una importante firma de eventos fisiológicos y patológicos en diferentes escalas y puntos de tiempo en la vida neuronal, de la deformación axonal transitoria asociada a actividad eléctrica (escala de milisegundos) 3 a la irreversible neuronal de la hinchazón que ocurre durante la fase asintomática de neurodegenerativas enfermedades (años en los seres humanos) 4. Sin embargo, el mayor cambio de volumen se produce en una escala de tiempo intermedio de días o semanas (dependiendo el organismo considerado) durante el crecimiento neuronal. La prolongada y compleja morfología de neuronas plantea cuestiones múltiples, entre los que la regulación del tamaño celular. Diámetro y longitud axonal son de hecho fuertemente regulado en vivo, con valores específicos para cada tipo neuronal 5,6.
Estos temas, complejos de abordar en vivo, también pueden abordarse de manera simplificada in vitro. En ese objetivo, un método dedicado a la medición de volumen rápido lo suficiente como para seguir la dinámica de crecimiento (es decir, en una escala de tiempo de minutos) y compatible con la observación durante horas o días se requiere. Varios métodos se han desarrollado sobre los años para proporcionar un acceso directo o indirecto a volumen celular in vitro. Reconstrucción de la célula de proyección de imagen confocal es uno de ellos, pero este método implica el etiquetado y repetidas exposiciones a la luz al tiempo que muestra una limitada resolución axial de aproximadamente 500 nm 7. Tenga en cuenta que estos dos últimos inconvenientes son parcialmente inundados por un método más sofisticado y reciente llamado enrejado de la microscopia de luz-hoja 8. Microscopía de fuerza atómica ha utilizado 9 pero este método de análisis es por esencia lenta y tediosa. Por otra parte, el contacto físico que requiere la célula podría interferir con la medición teniendo en cuenta la extrema suavidad de neuronas 10. Método indirecto utilizando la impedancia o resonancia han sido empleados para 11tipos de células diferentes, pero son insuficientes para extendido adhesivo células como las neuronas.
Uno de los métodos más prometedores se basa en la medida del volumen excluido de las células en una cámara estrecha llenado de un colorante fluorescente. El método de exclusión de la fluorescencia (FXm) es simple en su principio, ya que no requiere etiquetado y es adecuado para imágenes ópticas rápido, de largo plazo de poblaciones celulares con una resolución axial potencialmente el óptica. Más precisamente, la resolución en z depende de la intensidad de la fluorescencia máxima en el compartimiento de la cultura (es decir, en la región carente de células) dividida por el rango dinámico de la cámara, aunque varias fuentes de ruido de limitan esta última resolución. Este método ha sido muy fuerte el volumen de la migración de células adherentes 12 o para el estudio de cambio de volumen durante la mitosis de células de mamífero, como bien se describe en el 13. Sin embargo, las neuronas constituyen un desafío metodológico para grandes teniendo en cuenta su extensa ramificación en procesos sub-micrométricas.
Aquí presentamos un método que conduce a la fabricación de cámaras grandes lisas para obtener con alta precisión el volumen y la altura de ramas neuronales y estructuras dinámicas en crecimiento neuronal como conos de crecimiento.
Cámaras deben tener alturas similares que el objeto a medir con el fin de optimizar la resolución axial. Por lo tanto, hemos diseñado diferentes dispositivos de FXm caracterizados por cámaras de medición central de tres alturas diferentes. El más delgado (3 μm de altura) se dedica a la medición de neurita: esta altura baja excluye soma, que permanecen en la cámara intermedia alta cerca de 15 μm. Cámaras central más gruesos (10 a 12 μm) son lo suficientemente altas para seguir el crecimiento de células enteras. El dispositivo incluye también dos embalses situados a ambos lados de la cámara central. Cuatro agujeros de inyección (IH) así se implementan y se señalan como sigue: la entrada y salida sirven para introducir las suspensiones celulares en el chip, mientras que los otros dos alimentan a los embalses.
Tenemos primera calibración fabricados cubreobjetos para mediciones de altura con fotoresistencia estructuras de geometría conocida. Entonces hemos reflejado las neuronas cada vez más libres, pero también morfológicamente limitadas neuronas en micropatrones de adhesión.
Protocolo
El estudio se llevó a cabo con arreglo a las directrices comunitarias sobre el cuidado y uso de animales de laboratorio: 86/609/CEE. El propósito de la investigación y el protocolo se describen en el proyecto ético anexo de ERCadg CellO, que fue aprobado y es revisado periódicamente por la AECEI. Institut Curie Animalario ha recibido licencia #C75-05-18, 24/04/2012, reportando al Comité d'Ethique en matière d'expérimentation animale Paris Centre et Sud (número de registro nacional: #59).
1. fabricación del molde
Nota: El molde incluye cámaras central e intermedias conectadas a una entrada y una salida, más dos depósitos (y entradas) situados a ambos lados de la cámara central.
- Utilice unas pinzas planas para manipular obleas de silicio de 51 mm de diámetro y transferirlos de un lugar a otro.
- Molde de silicona de la cámara central
- Llenar una pipeta de plástico de 2 mL con photoresist positivo. Coloque la pipeta en el centro de una oblea de silicio de 51 mm de diámetro y presione en su embalse que cubren alrededor del 75% de la superficie de la oblea con la fotoresistencia. Capa de centrifugado a 3000 rpm por 30 s.
- Transferir la oblea de un recubridor de vuelta a una placa, con una temperatura de 100 ° C, durante 50 s.
- Transferencia de la oblea de la placa al titular del sustrato (chuck) del alineador de la máscara. Exponer a través de la "máscara DRIE" (ver archivos complementarios "masks_neuron_volume_chips.tiff" y "masks_neuron_volume_chips.dxf").
- Retire la oblea de la tirada y luego sumergirse en un vaso de 100 mL, plato que contiene el revelador (dilución 1:4 en agua desionizada) de cristalización. Suelte la oblea y mantenerlo durante 1 min agitando continuamente y suavemente el plato de la cristalización.
Nota: Para guardar reactivos, llene el plato de cristalización a 1 cm de altura como máximo. - Llevar la oblea del desarrollador y sumérjalo en un plato la cristalización de 100 mL con agua desionizada para unos 10 s.Then lugar la oblea sobre un papel absorbente y secar con nitrógeno a presión con una pistola aire.
- Colocar la oblea para 50 s en la placa caliente con una temperatura de 115 ° C.
- Realizar grabado profundo reactiva de Ion (DRIE) con los siguientes parámetros (más información sobre la técnica DRIE puede encontrarse en referencias 14 y 15); Paso de Pasivación: 50 sccm de C4F8, que forro flujo 10 sccm, CP 10 W, inductivo juntado de Plasma (ICP) 1500 W, total presión mbar 24, temperatura de 18 ° C; Paso de grabado: 100 sccm de SF6, que forro flujo 10 sccm, CP 11 W, ICP 1500 W, total presión mbar 24, temperatura de 18 ° C; Tiempo de estabilización: 4 s; Tiempo de grabado: 7 s; Duración del proceso: normalmente 5 min para aprox. 10 μm grabado al agua fuerte profundidad.
- Disolver la fotoresistencia de buceo el sustrato en un plato la cristalización con acetona.
- Sumerja la oblea en una solución de piraña (2/3 de H2O2 (30%) y 1/3 de H2SO4 (95 %)) durante 5 minutos.
PRECAUCION: Siempre agregar H2O2 primero y luego H24 y enjuague por lo menos 3 veces en agua desionizada después de la limpieza.
- Fabricar los moldes correspondientes a cámaras intermedias realizando pasos 1.2.1 a 1.2.11 siguiendo los parámetros enumerados en las tablas 1-3.
Nota: Cada tabla corresponde a un dispositivo caracterizado por una altura específica de la cámara de observación central.
Nota: Realice todo el proceso de uso creciente de materiales fotoresistentes alturas (máscaras de 1 a 3 para cada dispositivo, ver archivos complementarios "masks_neuron_volume_chips.tiff" y "masks_neuron_volume_chips.dxf").- Colocar la oblea de silicio grabado al agua fuerte en el soporte del sustrato spin coater.
- Compruebe que la máquina de pintar del giro funciona correctamente verificando que la aspiración del sustrato es eficaz (el sustrato debe girar y permanecer en el lugar durante la velocidad de rotación nominal).
- La viscosidad de SU-8 aumenta con la altura de la gama del grueso específicas. Siempre utilizar botellas de 20-30 mL para almacenar SU-8 photoresists y viértala en la oblea antes de spin-coating (SU-8 podría ser demasiado viscoso para ser manipulado con una pipeta de plástico).
- Vierta el photoresist negativo tipo de epoxy SU-8 en el centro del sustrato hasta cubrir alrededor del 75% de la oblea de silicio, entonces spin-capa utilizando los parámetros indicados en la fila "Autoensamblados" de las tablas.
- Lugar de la oblea cubierta sobre una placa para la duración y la temperatura indica en fila "suave cuece al horno".
- Monte la oblea del grabado y la adecuada "SU-8 máscara" en el alineador de la máscara.
- Alinee la oblea del grabado al agua fuerte con la máscara con las cruces de alineación dedicada (tamaño típico de estos cruces: 50 μm × 150 μm) diseñado en cada máscara.
Nota: Dos cruces a cada lado del dispositivo son suficiente (uno en la parte inferior izquierda) y uno en la parte superior derecha. - Exponer utilizando la-línea del alineador de la máscara (longitud de onda 365 nm) con la dosis de UV apropiada, como se indica en la fila "Exposición energía".
Nota: El tiempo de exposición se calcula dividiendo del específico de la energía E de exposición para cada fotoresistencia el poder efectivo de la lámpara UV, modulado por la absorción de la mascarilla: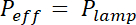 x (1 - absorciónmáscara). La absorción es alrededor del 20% para máscaras flexibles y despreciable para la máscara del cromo duro.
x (1 - absorciónmáscara). La absorción es alrededor del 20% para máscaras flexibles y despreciable para la máscara del cromo duro. - Colocar la oblea revestida sobre una placa para la duración y la temperatura indicada en la fila 'Post bake'.
- Preparar platos de cristalización de vidrio de 100 mL dos, uno que contiene el revelador, la otra vacía. Buceo la oblea en el desarrollador para la duración indicada en fila «Desarrollo». Agite suavemente el plato cristalización a lo largo del desarrollo
- Espolvorear la oblea con isopropanol sobre el plato vacío cristalización para cerca de 5 s. Por último, colocar la oblea sobre un papel absorbente y secar con nitrógeno a presión con una pistola aire.
- Lugar la oblea revestida sobre una placa para la duración y temperatura indicada en la fila "duro cuece al horno" (opcional).
Nota: Este paso es útil para evitar las grietas en la fotoresistencia y proporcionar una superficie plana homogénea para los próximos pasos. - Repita los pasos 1.3.1 - 1.3.10 para completar los procesos enumerados en la tabla 1-3.
- Después el último paso de fotolitografía, silanize el master final del molde compuesto por 3 capas de materiales fotoresistentes negativo mediante el envío de dos gotas de silano Trichloro(1H,1H,2H,2H-perfluoro-octyl) a cada lado de la maestría en un plato de Petri de 100 mm de 100 μl. Sello el disco de Petri con una capa de parafina de plástico e incube 20 min a temperatura ambiente (RT).
Nota: El molde maestro está listo y puede utilizarse varias veces.
- Colocar la oblea de silicio grabado al agua fuerte en el soporte del sustrato spin coater.
2. fabricación de la viruta PDMS
- Vierta 90 g de polímero orgánico basado en silicona (polidimetilsiloxano: PDMS) en un vaso de plástico de 100 mL. Añadir 10 g de su agente endurecedor (1:10 en peso). Revolver la mezcla con una pipeta de plástico durante 2-3 minutos.
Nota: Se mezclan 90 g de PDMS con 10 g de agente para la fabricación de 6 chips endurecedor. - Colocar la mezcla dentro de un desecador de vacío y una bomba durante unos 30 minutos para eliminar las burbujas de aire atrapadas en el PDMS.
- Coloque el molde principal en un plato Petri P100 y verter 15 mL de la mezcla en el molde utilizando una jeringa.
Nota: 15 mL conduce a 1,5 mm de altura total del chip. - Coloque la estructura de PDMS-molde dentro de un desecador de vacío y una bomba hasta que no hay más burbujas de aire que estalla en la superficie de los PDMS.
Nota: Este paso toma unos 30 minutos. - Empuje la oblea en el fondo de la caja Petri con una punta de cono para evitar volumen PDMS muerto debajo de la oblea. Coloque el dispositivo de PDMS-molde en el horno a 70 ° C durante al menos 2 h.
- El bloque PDMS bajo una campana química con una espátula plana de acero inoxidable y por colada isopropanol en el chip del demold. Coloque en el Banco de acero inoxidable de la campana.
- Corte alrededor de la oblea de silicio y la réplica PDMS con un bisturí del demold.
- Corte alrededor (dejar un 2 mm margen) el chip usando un bisturí o una cuchilla de afeitar.
- Perforar las entradas presionando firmemente el punzón de diámetro 1.5 mm y accionar para cortar y tallar el agujero de la entrada. Lo mismo en las cuatro zonas dedicadas del chip donde se inyectará el líquido.
- Limpiar el chip pegado y pelando cinta adhesiva en el lado microestructurado. Espolvorear el isopropanol en ambos lados. Luego secar el chip con nitrógeno a presión con una pistola aire.
3. fabricación de modelado cubreobjetos (24 × 24 mm2)
Nota: Manipular cubreobjetos con la pinza curva.
- Patrones de Poly-ornitina (PLO)
- Aplicar una O2 limpieza plasma en cubreobjetos de vidrio. Parámetros de plasma: presión de bombeo: 0,25 mbar; O2 fuente duración: 3 min; flujo del gas: 10 sccm; desviación máxima: ± 5 sccm; duración de plasma: 3 min; presión: 0,36 mbar; desviación máxima: ±0. 10 mbar; conjunto potencia: 50 W; desviación máxima: 5%; duración de la ventilación: 45 s.
- Mezcla 484 μl de ácido acético y 56 μl de 3-methacryloxypropyl-Isobutiltrimetoxisilano, completo con etanol absoluto para obtener un volumen total de 15 mL.
- Usando un cono de punta de 1 mL, poner una gota de 500 μl de esta solución en cada cubreobjetos, espere 2-3 min en seco usando un limpiador de microfibras para salas blancas.
Nota: Cubreobjetos de vidrio silanizada pueden almacenarse 1 mes a temperatura ambiente en cajas de plástico selladas con una capa de parafina de plástico. - Coloque cada cubreobjetos en un recubridor spin situado en un entorno limpio. Ponga una gota de un photoresist positivo que cubren alrededor del 75% de los cubreobjetos (500 μl) y spin-capa a 4000 rpm durante 30 s para llegar a un espesor final de 0.45 μm.
- Colocar el cubre-objetos durante 1 min sobre una placa con una temperatura de 115 ° C.
- Utilizar un alineador de máscara, exponer cada cubreobjetos en una longitud de onda de 435 nm (línea G) a través de la máscara dedicada según los parámetros del fabricante (dosis de UV sobre 50-60 mJ.cm–1)
- 2 platos de cristalización de vidrio, uno que contiene el promotor (sin dilución), el otro que contiene agua desionizada.
- Buceo uno por uno cada cubreobjetos en el desarrollador para 1 minuto agitando continuamente y suavemente el plato de la cristalización. Luego sumerja la oblea en agua desionizada durante aproximadamente 5 s. Coloque la oblea sobre un papel absorbente y secar con nitrógeno a presión con una pistola aire.
- Aplicar un plasma2 de activación O con los mismos parámetros que en 3.1.1.
- Bajo el capó, deponen cuatro 170 μl gotas de una solución 100 μg/mL PLO por P100 caja de Petri. Coloque el patrón de los cubreobjetos sobre cada una de estas gotas. Sello de la caja Petri con una capa de parafina de plástico para evitar que se sequen. Incubar durante una noche a TA.
Nota: La solución de PLO debe permanecer pegada al cubreobjetos por capilaridad. - Preparar cuatro recipientes (típicamente P60 de Petri), tres de ellos llenan de PBS y el cuarto con agua desionizada. Preparar dos platos de cristalización de vidrio de etanol puro.
- Sacar cada cubreobjetos de las cajas Petri, sumérjalo en el primer baño de PBS para 10-15 s, evacuar el líquido en un tejido absorbente por poner el cubreobjetos verticalmente en el lado, inserte con por ejemplo la cara estampada por dentro de la bañera de etanol.
Nota: Una vez completada la disolución de la fotoprotección, se hace difícil localizar el lado estampado del cubreobjetos. Por lo tanto, es importante en esta etapa para rastrear su ubicación. - Colocar el etanol cristalización plato en un sonicador de baño de ultrasonidos (120 W / 35 kHz) y dejar que la fotoresistencia disolverse durante 3 minutos.
Nota: Cambiar el baño de etanol cada 4 cubre-objetos para limitar la dilución por el PBS que pudiesen afectar la disolución de la fotoresistencia. - Saca el cubreobjetos del baño de etanol, después lo sumergirse varias veces en el segundo baño de PBS. Compruebe el aspecto superficial repetidamente hasta que la grasa como la superficie que resulta de la película líquida restante de etanol desaparece.
- Sumerja durante 5-10 s el cubreobjetos en el tercer baño de PBS. Entonces inmediatamente transferirlo para el baño de agua desionizada. Coloque el cubreobjetos sobre un papel absorbente y secar con nitrógeno a presión con una pistola aire.
Nota: El último enjuague con agua desionizada se utiliza para evitar la formación de cristales de PBS durante la etapa de secado.
- Estructuras de fotoprotección para la calibración de la altura
- Realice sólo pasos 3.1.4. a 3.1.8. usando la máscara dedicada (máscara "Photoresist stripes", vea archivo suplementario "Mask_Photoresist-stripes.dxf").
4. aplicación de montaje y final de la viruta
- Montaje de platos de fondo de vidrio de la viruta
- Poner el chip PDMS y el plato de cristal que será consolidado en la cámara de plasma para la activación de la superficie. Parámetros: presión de bombeo: 0,25 mbar; O2 fuente duración: 3 min; flujo del gas: 10 sccm; desviación máxima: ± 5 sccm; duración de plasma: 30 s; presión: 0.40 mbar; desviación máxima: ±0. 10 mbar; conjunto potencia: 50 W; desviación máxima: 5%; duración de la ventilación: 45 s.
- Suavemente ponga el chip activado de PDMS en contacto con el cubreobjetos de cristal y delicado aplique presión en los bordes de la viruta para el chip para el cubreobjetos. Para aumentar la fuerza de Unión, coloque el dispositivo en el horno a 70 ° C durante 5 a 10 min.
Nota: No presione en piezas que contengan pilares, podría derrumbarse bajo demasiada presión. - Bajo el capó (es decir, a temperatura ambiente) y dentro de 30 minutos después de la Unión, coloque un cono 10 μl llenado con una solución 100 μg/mL PLO en el IHs, y luego inyectar el líquido. Ajustar el volumen para formar una gota en la parte superior de cada IHs. Luego, utilizando un cono de punta de 1 mL, añadir PBS en la caja Petri todo el chip.
- Deje que el chip a temperatura ambiente con un mínimo de tiempo de incubación de 2 h. Para la incubación durante la noche, selle la caja de Petri utilizando una película de plástico de la parafina para evitar que se sequen.
Nota: El líquido no debe derramar fuera de otra manera el chip que el chip debe ser desechado. - Presione ligeramente un cono 10 μL en cada IH y aspirar el exceso de líquido. Entonces stick totalmente el cono dentro de la toma de corriente y elaborar el líquido restante.
- Vuelva a colocar PLO laminina siguiendo las instrucciones dadas en los pasos 4.1.5 (vaciado) y 4.1.3 (relleno). Incubar a temperatura ambiente durante 1 h.
- Reemplazar laminina por el medio de cultivo tras las instrucciones dadas en los pasos 4.1.5 (vaciado) y 4.1.3 (relleno). Composición del medio de cultivo: MEM 81.8%; Piruvato de sodio 100 mM 1%; Glutamax 200 mM 1%; Caballo suero 5%; B27 suplemento 2%, N2 suplemento 1%, gentamicina 0,2%; filtrar la solución con un filtro de 220 nm. Un cono de punta de 1 mL, sustituir también el PBS que rodea el chip por este medio.
- Poner el chip en la incubadora regulada a 37 º C y 5% CO2 para por lo menos 5 horas (o durante la noche) antes de neurona siembra.
- Montaje utilizando cubre-objetos con motivos de la viruta
Nota: Si el cubreobjetos con motivos incluyen objetos de referencia de photoresist positivo, realice pasos 4.2.1 a 4.2.9. De lo contrario, realizar sólo paso 4.2.3, stick, el dispositivo PDMS en el cubreobjetos de PLO con motivos como se indica en el punto 4.1.2, pone medio de cultivo dentro y alrededor del chip entonces vaya al paso 4.2.10.- Depositar una gota de agua sobre un portaobjeto de microscopio gruesa rectangular y pegar el cubreobjetos sobre el portaobjetos de cristal por capilaridad (no-patrón hacia el portaobjetos de cristal). Bajo el microscopio, marque la ubicación de las rayas de la fotoresistencia en la parte trasera de la corredera de vidrio con un rotulador.
- Coloque el cubreobjetos de vidrio modelado en el soporte de la máscara del alineador de la máscara. Dependen de la marca con el rotulador para centrar los objetos de referencia de fotoprotección.
- Realice el paso de plasma como se describe en 4.1.1 en el chip PDMS.
- Coloque el chip PDMS en el soporte de sustrato móvil (chuck) del alineador de la máscara.
Nota: Para aumentar el contraste óptico, colocar una oblea de silicio bajo el chip PDMS. La oblea de silicio debe permanecer atada firmemente en el portabrocas durante el proceso de alineación (uso una cinta transparente para pegarlo a la tirada). - Levante el mandril en el límite de contacto mecánico para alinear el chip con el conjunto de rayas de photoresist en el cubreobjetos.
- Lograr contacto mecánico entre la viruta y el cubreobjetos por terminando de levantar el mandril hasta que la superficie de los PDMS pilares toque el cubreobjetos de vidrio.
- Inferior del mandril. Quite el cubreobjetos ahora adherido al chip del titular de la máscara. Luego coloque el dispositivo en un plato de Petri de 35 mm y transferir todo en el horno (temperatura: 70 ° C) durante 5 a 10 minutos aumentar la fuerza de Unión.
- Realizar como en el paso 4.1.3.
Nota: Si utilizando PLO patrón cubreobjetos, ir directamente de paso 4.2.7 a 4.2.9. - Reemplazar el PLO por el siguiente medio de galjanoplastia que los procedimientos descritos en los pasos 4.1.5 (vaciado) y 4.1.3 (relleno). Un cono de punta de 1 mL, sustituir también el PBS que rodea el chip por este medio.
- Poner el chip en la incubadora regulada a 37 º C y 5% de CO2 hasta la neurona siembra, con un mínimo de tiempo de incubación de 5 h.
5. cultura neurona
- Preparar 100 mL de disección medio (HH) mezclando 10 mL de HBSS 10 x 2 mL de Hepes 1 M y 88 mL de agua estéril en un frasco de plástico de 200 mL.
Nota: El medio HH se puede preparar el día antes de la cultura. - Diseccionar el hipocampo de embriones de ratones E18 extraído de una madre sacrificada por dislocación cervical (ratones C57BL/6J de Charles Rivers). Pasos de disección son por ejemplo, en 16.
- Coloque el hipocampo en un tubo de plástico que contiene tripsina (0,3 mL de tripsina 2.5%, sin EDTA en 2,7 mL de medio de HH) por 10 min a 37 ° C para iniciar la disociación química.
- Elimine casi todo el líquido y reemplazarlo con 5 a 10 mL de HH utilizando pipetas de plástico desechables. Hacerlo 3 veces. Para el último relleno, utilizar 1 mL de medio en lugar de HH de la galjanoplastia.
- Disocian mecánicamente los tejidos mediante un cono portapuntas de 1 mL aspirando y expulsando el volumen completo varias veces, evitando burbujas y uso de no más de 15-20 pasos.
- En un recipiente separado de 500 μl, prepare una solución con 5 μl de la suspensión celular diluido en 45 μl de PBS. Toma 1 μl de esta solución con una pipeta μl 10 e inserte la suspensión diluida en un contador de células de Malassez. Utilice las indicaciones en el 17 para estimar el número de células.
Nota: Un hipocampo solo proporciona generalmente unos 0,5 millones de neuronas. - Centrifugar a 100 x g durante 6 min a TA.
- Deseche el sobrenadante y reemplazarlo por el volumen de medio de placas necesaria para lograr una concentración de 10 millones de células/mL. Resuspenda cuidadosamente los eritrocitos por sucesivamente aspiran y expulsión de la suspensión de células con un cono de punta de 1 mL.
- Elaborar el forro medio presente en el chip (consulte el paso 4.1.5). Recoger 2-3 μl de la solución recién resuspendida utilizando una pipeta de 10 μl e inyectar en la entrada (consulte el procedimiento de inyección se describe en el paso 4.1.3). Inmediatamente repetir la misma operación en la salida.
- Inyección sobre el mismo volumen de medio forro en cada depósito (ver paso 4.1.3).
Nota: Observar rápidamente el chip bajo el microscopio para comprobar la densidad de las células. La orden de la magnitud de la densidad celular óptima corresponde a unos 5-10 células en la superficie del cuadrado delimitado por 4 pilares (sobre 0.3 mm2, es decir, sobre 15-20 células por mm2). - Eventualmente repetir 5.10 usando en su lugar 0.5-1 μl de la suspensión celular para llegar a la densidad celular específica.
- Coloque el chip sembrado en una incubadora regulada a 37 ° C 5% CO2.
6. observación de exclusión fluorescencia
- Sustitución del medio de cultivo por medio de imágenes.
- Preparar el medio de imágenes como en 4.1.7 pero utiliza en su lugar MEM sin rojo de fenol y añadir dextrano fluorescente. En ello, diluir dextrano (peso molecular 10.000 g/mol, solución concentrada a 10 mg/mL en PBS) para alcanzar una concentración final de 0.5-1 mg/mL en el medio de imágenes.
Nota: Uso dextrán se conjuga con los máximos de absorción/emisión 496/524 o 650/668. Prefieren la primera imagen de 0,45 μm photoresist positivo alto estructuras (para deshacerse de su fluorescencia automático en el ancho de banda roja) y el segundo a las neuronas de la imagen (menos tóxico). - Vaciar todas las entradas con una pipeta de 10 μl y volver a llenarlos completamente con el medio de proyección de imagen (consulte los pasos 4.1.3 y 4.1.5 para la metodología precisa de repuesto de la media).
- Preparar el medio de imágenes como en 4.1.7 pero utiliza en su lugar MEM sin rojo de fenol y añadir dextrano fluorescente. En ello, diluir dextrano (peso molecular 10.000 g/mol, solución concentrada a 10 mg/mL en PBS) para alcanzar una concentración final de 0.5-1 mg/mL en el medio de imágenes.
- La proyección de imagen
- Coloque el chip en un microscopio de epifluorescencia equipado con una cámara ambiental regulada en 37 ° C y 5% de CO2. Uso seco de 40 X, apertura numérica (NA) 0.8 objetivo, 30% de potencia total (plena potencia: 3 W) y 30 ms de tiempo de exposición. Adquisición de imágenes de células en el foco (de simple a varias imágenes sucesivas en el caso de Time-lapse experimentos).
- Análisis de imagen
- Normalizar las imágenes usando la rutina de reducción de fondo implementada en software dedicado para conseguir un fondo homogéneo. Ver 13 de los detalles de las medidas incluidas en este software de procesamiento de imágenes. La imagen de salida tiene una. Formato de MAT.
- Convertir. MAT archivo en. Imágenes TIFF 8 bits utilizando la rutina en material complementario (conversion_mattotiff.m, que importfilevol.m).
- Realizar importación > secuencia de imágenes en ImageJ para construir un video del. Imágenes TIFF.
- Calcular la intensidad promedio P de un cuadrado área localizada en el centro de un pilar (objeto de referencia) y la intensidad promedio B de un área cuadrada de la sala desprovista de células (de fondo, es decir, altura cero). Ver 18 un ejemplo de metodología detallado de procesamiento de imágenes.
Nota: La dimensión lateral de las áreas de cuadrados utilizados como referencias de intensidad deben ser aproximadamente la mitad del diámetro del pilar para conseguir un número suficiente de píxeles evitando la contaminación lumínica de los bordes del pilar. - Utilice los valores de P y B para establecer la ley de la conversión lineal de intensidad a altura h:

con hc la altura conocida de la cámara, y
y
Nota: PDMS no muestra detectable autofluorescencia. - Seleccione un área alrededor de la zona de interés, integrar intensidad utilizar ImageJ (ver 19 para más detalles) y aplicar la ley de conversión obtenida en 6.3.5 para medir el volumen de un compartimiento celular.
Nota: La zona de interés podría ser seleccionada basado en la fluorescencia del celulares elementos como conos de actinia, e.g., para la selección de crecimiento en neuronas de GFP-LifeAct, seleccionadas la zona de interés en el canal de emisión de GFP, excepto el contorno de este zona, mediante una herramienta ROI manager, luego medir el volumen de celular incluido dentro de la misma zona en el canal de emisión de dextrano (en rojo).
Resultados
El resultado del proceso de fabricación descrito en las secciones 1 y 2 se ilustra en las imágenes de la figura 1A-1B y la curva de la figura 1. La tabla de la figura 1 muestra los valores de rugosidad de dos diferentes áreas representativas de la viruta PDMS, es decir, en la central y la cámara intermedia de 20 μm de alto siguiente. Una disminución de la rugosidad por un factor de cerca de 7 se ha obtenido mediante grabado oblea de Si en lugar de photoresist SU-8. Entonces, FXm fue aplicado por primera vez en una franja de la fotoresistencia de geometría conocida (figura 2A) dentro de una cámara alta de 10 μm. Después de procesamiento de imágenes y conversión de altura de intensidad (véase el gráfico de la figura 2B), grandes perfiles en secciones transversales a lo largo de esta banda (figura 2) proporcionan los perfiles de altura deseada (Figura 2D). Figura 2D muestra la comparación entre los perfiles obtenidos mediante perfilometría mecánica y métodos de FXm. Estos perfiles, incluyendo el valor del borde y la meseta, son muy similares, validando el método. Tenga en cuenta que la dispersión de los datos de grandes no es representativa de la resolución final del método, como se evaluó en la figura 3 y figura 4, pero los resultados de la baja intensidad emplean para evitar un posible efecto de la muy débil auto-fluorescencia de photoresist en el canal GFP.
Entonces, observamos neurites de 3 μm y 10 μm alta cámaras (figura 3). La desviación estándar del ruido de fondo es cerca de 18 nm después de intensidad para corrección de conversión y de la antecedentes de altura. Este valor es ligeramente superior a la rugosidad de la física de superficies PDMS casted en superficies silicio (12 nm, consulte la tabla de la figura 1) pero mucho menor que la rugosidad medida en PDMS obtenidas de moldes de SU-8. Estos resultados destacan el valor añadido de la perforación de pozos en obleas de silicio en lugar de abrir agujeros en photoresist SU-8 para pilares. Un valor tan bajo permite una alta señal a ruido y obtener imágenes muy claras en el volumen como la que se muestra en la Figura 3A. Como ejemplo de los datos que pueden ser obtenidos de tales imágenes, calcula el volumen de 1,6 μm (es decir, 10 píxeles) amplia neurita cortar (véase el gráfico de la figura 3B). Uso de un ajuste lineal de estos datos en una primera aproximación da un valor de altura de neurita media de cerca de 400 nm, frente a por ejemplo el diámetro axonal 500 nm encontrados en 10 días cachorros viejo dentro del cuerpo calloso 5. También combinamos FXm con micropatrones de adhesión consisten en serie contrapeados μm 2 y 6 μm rayas anchas de 30 μm de longitud. Nuestro objetivo fue estudiar la influencia de la anchura del neurite en forma 3D. Figura 3 muestra una representación 3D en falso color de una imagen de neurona entera obtenida en una cámara alta de 10 μm. Neuritas se extienden sobre 2 μm y rayas anchas de 6 μm, mientras que el soma se encuentra en la extremidad de la banda más grande. Perfiles de altura fueron dibujados en tres secciones diferentes. En coherencia con el gráfico mostrado en la Figura 3A, la superficie integrada sobre el aumento de cortes transversales con la anchura de la neurita (figura 3D).
También nos enfocamos en las estructuras 3D de crecimiento cono (GC). Figura 4 A-B muestra dos diferentes perfiles GC obtenidos en una cámara alta de 3 μm, que resaltan su estructura ramificada de la sub. Además, se realizaron experimentos de lapso de tiempo para seguir la dinámica del volumen de GCs en una cámara alta de 12 μm. Figura 4 muestra un ciclo de contracción y la reactivación de un GC determinado dentro de una escala de tiempo de unas pocas decenas de minutos. Gracias a la utilización de ratones lifeact de GFP, conos de crecimiento fueron localizados en la longitud de onda de emisión de GFP (510 nm) de la concentración de actina alta. La superficie identificada en la longitud de onda se utilizó para integrar sobre la longitud de onda de emisión de dextrano en 647 nm para calcular volumen de GC. Figura 4 muestra finalmente la distribución del volumen de la GC en diferentes puntos temporales y la ubicación de tres neuronas diferentes, centradas en un valor de alrededor de 6 μm3.

Figura 1: cámaras de FXm PDMS. Esquemas (A) de las cuatro etapas principales de microfabricación conduce al molde final. Se indica la ubicación de la entrada, salida y embalses. Barras de escala: 1 m m. (B) imagen de la cámara de PDMS FXm obtenida con un Perfilómetro óptico. Esta imagen muestra la cámara central que contiene 3 filas de 10 μm de altos pilares y los compartimientos intermedios de 20, 50 y 90 μm de altura. Barra de escala: 500 μm. (C) vista transversal de la viruta a lo largo de las dos líneas punteadas dibujadas en (B). Amarillo/oro: sección transversal a lo largo de los pilares, azul: sección entre pilares. (D) significa que los valores de la rugosidad PDMS medición en 50 × 50 μm2 zonas moldeadas en silicona y en la cámara intermedia SU-8 alta 20 μm (ver las flechas para la ubicación de estas áreas). Se obtuvieron valores promedios de las mediciones de tres áreas diferentes. Haga clic aquí para ver una versión más grande de esta figura.
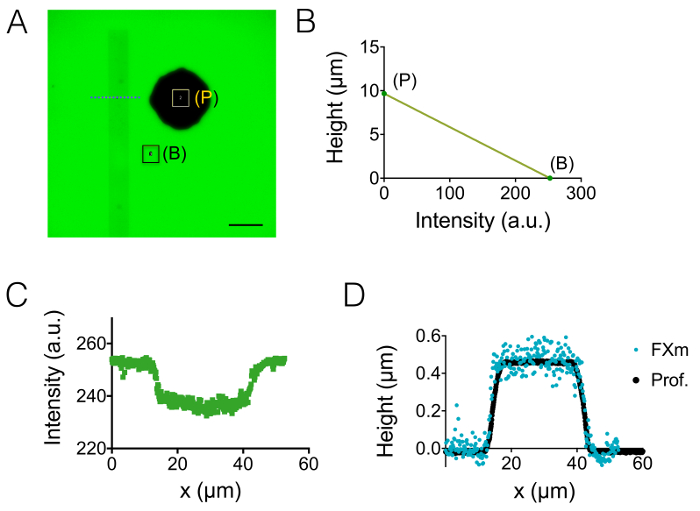
Figura 2: calibración del método de FXm usando una fotoprotector de la raya como objeto de interés. (A) imagen de la fluorescencia de GFP en una cámara alta de 10 μm llena de 10.000 dextrano MW absorbente a 488 nm a 1 mg/mL. (B: fondo, P: Pilar). Observación con objetivo 40 X NA 0.8 seca. Barra de escala: 50 μm. (B) ley de calibración lineal obtenida de la intensidad media de los dos rectángulos coloreados se muestra en el perfil de intensidad de fluorescencia (C) a. obtenido a nivel de la línea discontinua azul en (A), cruzando la fotoresistencia Raya (μm 0.45 alta photoresist positivo). (D) comparación de los perfiles obtenidos de Perfilómetro mecánico (puntos negros) y grandes después de intensidad a la conversión de altura de los datos de (B) (puntos azules). Haga clic aquí para ver una versión más grande de esta figura.
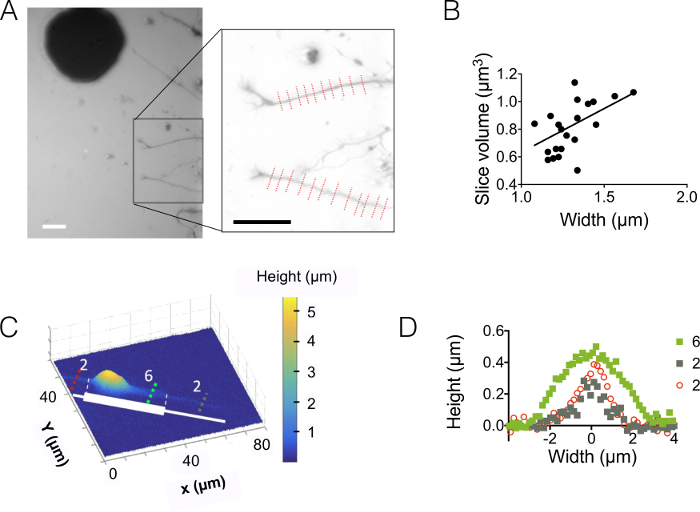
Figura 3: proyección de imagen de volumen neurita. (A) neurita que extendía en la cámara alta central de 3 μm de soma situado en la cámara intermedia siguiente 15 μm. Proyección de imagen realizada con 10.000 dextrano MW absorbente a 488 nm y 40 X, NA 0.8 seca objetivo. El margen obtenido después de que el uso de la rutina de reducción de fondo destaca los dos neurites y elegido para trazar la gráfica de la derecha. Barras de escala: 30 μm. (B) Neurite rebanada volumen como una función de la anchura de la neurita obtenida de los 22 perfiles (promedio 10 píxeles, es decir, en un 1.6 μm "neurite slice") se muestra en (A). La línea continua representa un ajuste lineal de pendiente 0.4 μm pasa por el origen. (C) imagen de color falso de una neurona con motivos en una banda adhesiva hecho de sucesivas μm 2 y 6 μm ancho tocones (representados en blanco). Las medidas fueron hechas en una cámara alta de 10 μm con 10.000 dextrano MW absorbente en 647 nm y utilizando una NA 40 x 0.8 seca objetivo. (D) perfiles de altura correspondiente a las líneas punteadas color que se muestra en (C), manteniendo el mismo código de color. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: proyección de imagen de cono estático y dinámico crecimiento. (A-B) Perfiles de altura de cono de crecimiento obtenidos en una cámara alta de 3 μm después de intensidad a la conversión de altura a lo largo de las líneas amarillas muestran en imágenes asociadas. Observación realizada usando un relleno con 10.000 dextrano MW absorbente a 488 nm y 40 X, NA 0.8 seca objetivo. (C) proyección de imagen de neurona entera en una cámara alta de 12 μm llena de 10.000 dextrano MW absorbente en 647 nm. Observaciones se han hecho en dos canales fluorescentes: GFP para localización del cono de crecimiento (líneas amarillas punteadas) y CY5 para calcular volumen de GC de la exclusión de la fluorescencia. La superficie por líneas amarillas punteadas se utilizó para calcular el volumen de la GC. El gráfico muestra la variación del volumen de la GC en el tiempo y morfologías asociadas en canales de GFP y CY5 en dos momentos diferentes representativos. Todos los datos fueron adquiridos con una NA 40 x 0.8 seca objetivo cada barras de escala de 3 minutos: 10 μm. haga clic aquí para ver una versión más grande de esta figura.
| Paso | Máscara de capa de 1:8 μm | Máscara 2:30 μm capa | Máscara 3:40 μm capa |
| Tipo de SU-8 | 2007 | 2025 | 2050 |
| Autoensamblados | 30 s @ 2000 rpm | 30 s a 3050 rpm | 30 s @ 3250 rpm |
| Cocer al horno suave | 3 min a 95 ° C | 2 min a 65 ° C + 6 min a 95 ° C | 3 min a 65 ° C + 7 min a 95 ° C |
| Energía de la exposición | 110 mJ/cm2 | 155 mJ/cm2 | 170 mJ/cm2 |
| Post-exposición cueza al horno | 4 min a 95 ° C | 1 min a 65 ° C + 6 min a 95 ° C | 2 min a 65 ° C + 7 min a 95 ° C |
| Desarrollo | 2 min 30 s | 5 min | 6 min |
| Hornear duro (opcional) | 3-5 min @ 200 ° C | 3-5 min @ 200 ° C | 3-5 min @ 200 ° C |
Tabla 1: pasos de fotolitografía realizaron para construir un dispositivo que contiene una camara de 12 μm de altura central. Alturas de las cámaras intermedias: 20, 50 y 90 μm.
| Paso | Máscara 1:10 μm capa | Máscara 2:30 μm capa | Máscara 3:40 μm capa |
| Tipo de SU-8 | 2007 | 2025 | 2050 |
| Autoensamblados | 30 s @ 1500 rpm | 30 s a 3050 rpm | 30 s @ 3250 rpm |
| Cocer al horno suave | 3 min a 95 ° C | 2 min a 65 ° C + 6 min a 95 ° C | 3 min a 65 ° C + 7 min a 95 ° C |
| Energía de la exposición | 125 mJ/cm2 | 155 mJ/cm2 | 170 mJ/cm2 |
| Post-exposición cueza al horno | 4 min a 95 ° C | 1 min a 65 ° C + 6 min a 95 ° C | 2 min a 65 ° C + 7 min a 95 ° C |
| Desarrollo | 2 min 30 s | 5 min | 6 min |
| Hornear duro (opcional) | 3-5 min @ 200 ° C | 3-5 min @ 200 ° C | 3-5 min @ 200 ° C |
Tabla 2: Fotolitografía los pasos realizados para construir un dispositivo que contiene una camara de 10 μm de altura central. Alturas de las cámaras intermedias: 20, 50 y 90 μm.
| Paso | Máscara 1:12 μm capa | Máscara 2:32 μm capa | Máscara 3:40 μm capa |
| Tipo de SU-8 | 2015 | 2025 | 2050 |
| Autoensamblados | 30 s @ 3250 rpm | 30 s @ 2500 rpm | 30 s @ 3250 rpm |
| Cocer al horno suave | 3 min a 95 ° C | 2 min a 65 ° C + 5 min a 95 ° C | 3 min a 65 ° C + 7 min a 95 ° C |
| Tiempo de exposición | 140 mJ/cm2 | 157 mJ/cm2 | 170 mJ/cm2 |
| Post-exposición cueza al horno | 4 min a 95 ° C | 1 min a 65 ° C + 5 min a 95 ° C | 2 min a 65 ° C + 7 min a 95 ° C |
| Desarrollo | 3 min | 5 min | 6 min |
| Hornear duro (opcional) | 3-5 min @ 200 ° C | 3-5 min @ 200 ° C | 3-5 min @ 200 ° C |
Tabla 3: Fotolitografía los pasos realizados para construir un dispositivo que contiene una camara de 3 μm de altura central. Alturas de las cámaras intermedias: 18, 50 y 90 μm.
Datos complementarios 1: masks_neuron_volume_chips.tiff. Vista esquemática de las máscaras que se utilizan para fabricar el dispositivo PDMS (DRIE máscara y máscaras 1-3). Haga clic aquí para descargar este archivo.
Información adicional 2: archivo "masks_neuron_volume_chips.dxf". Archivos electrónicos que permiten para fabricar la máscara DRIE y máscaras 1-3. Haga clic aquí para descargar este archivo.
Información adicional 3: "Mask_Photoresist-stripes.dxf". Archivos electrónicos que permiten para fabricar la máscara utilizada para la fotolitografía de photoresist rayas. Haga clic aquí para descargar este archivo.
Información adicional 4: archivo conversion_mattotiff.m haga clic aquí para descargar este archivo.
Datos complementarios 5: importfilevol.m del archivo por favor haga clic aquí para descargar este archivo.
Discusión
Proyección de imagen de volumen de neuronas constituye un reto para la técnica de grandes debido a las extensiones de estas células largas y delgadas. Este protocolo describe las variantes del mismo tipo de dispositivo de microfluidos dedicada a la proyección de imagen de la neurona.
Al lado de los aspectos de diseño microfluídico, la elección del objetivo es fundamental para la proyección de imagen de exclusión de la fluorescencia e implica un compromiso entre la resolución lateral y la claridad de la imagen. Se ha demostrado en el 13 que un NA alta conducen a una profundidad de foco más pequeño que la altura de cámara no era perjudicial para la precisión de la medición de volumen si se realizó la proyección de imagen en foco y si se deja un margen suficiente entre el contorno del objeto del os intereses y los límites de la superficie de integración. Sin embargo, el uso de una cámara muy superior a la profundidad del foco impide la claridad de la imagen debido a la difusión del fotón, que suaviza los bordes de los objetos de interés. La fabricación de una cámara alta de 3 μm redujo este desenfoque lateral y proporciona imágenes excepcionalmente definidas exclusión fluorescente con NA alto (0,8) 40 X objetivos visualizar ramas neuronales con alta resolución lateral.
Chip de montaje es un paso crítico, en particular en el caso de alta cámaras de 3 μm, pero manipulación cuidadosa tal como se describe en el punto 4.1.2 evita el colapso del techo. La superficie alta al cociente del volumen asociado a estas cámaras delgadas también planteó la cuestión de la estabilidad de la concentración de dextranos en el tiempo. Hemos comprobado que la absorción superficial del dextrán después de una noche de incubación era insignificante: después de reemplazar el dextrano por PBS, la diferencia de intensidad entre el pilar y el fondo era de 1 por 1000 del contraste de intensidad inicial entre estas dos regiones en presencia de dextrano. Nota que las neuronas pueden adherirse sobre el cubreobjetos de la parte inferior y en el techo PDMS. Este efecto desaparece cuando usando modelado cubreobjetos (es decir, cuando no incubar adhesivo moléculas dentro de la cámara PDMS), como la capa es por lo tanto estrictamente localizada en la parte inferior de la cámara.
Aparte de su morfología desafiante, las neuronas son más bien adecuadas para grandes debido a que uno de la gran limitación del método, es decir, endocitosis de dextrano, es muy limitado en estas células. Elegimos un 10 kDa formulación para suprimir a largo rango (horas) cualquier fenómeno de endocitosis visible.
En conclusión, la simplicidad conceptual de FXm es balanceada por un conjunto de temas experimentales que han sido resueltos por el presente Protocolo, tales como rugosidad nanométrica de PDMS y altura de cámara micrométrico o corrección de fondo para corregir las irregularidades de el techo de PDMS entre pilares. Sin embargo, el uso de una cámara estrecha microfluídicos para limitar el medio fluorescente produce algunas limitaciones como la necesidad de pilares de apoyo, que reduce la superficie efectiva disponible para la adhesión celular, o la necesidad de excluir el soma de la central cámara para observar extensiones neuronales con la mayor claridad, que limita las regiones de la célula accesible a la observación de alta resolución. Una posible evolución de este método sería deshacerse de este confinamiento físico, para ser reemplazado por una óptica. El nuevo desarrollo de la microscopía de luz de hoja puede combinarse ventajosamente con grandes en el futuro.
Divulgaciones
Los autores no declaran conflicto de intereses.
Agradecimientos
Los autores quieren reconocer ChiLab, materiales y laboratorio de microsistemas - Politecnico di Torino - DISAT, en la persona del Prof. C F Pirri, Dr. M Cocuzza y Dr. S L Marasso, por su precioso apoyo en el proceso de desarrollo y fabricación del dispositivo. Agradecemos a Víctor Racine de Quantacell para la discusión y apoyo en el tratamiento de la imagen. Agradecemos a Isabelle Grandjean y Manon Chartier de la facilidad Animal del Instituto Curie por su apoyo para los ratones y Pablo Vargas y Ana María Lennon (Instituto Curie) por facilitarnos los ratones LifeAct de GFP. Agradecemos a Olivier Thouvenin del Institut Langevin y Clotilde Cadart, Larisa Venkova y Matthieu Piel de Instituto Curie - 144 UMR, por su ayuda en la comprensión de la exclusión de fluorescencia método. Por último, agradecer a la plataforma tecnológica de Institut Pierre-Gilles de Gennes (CNRS UMS 3750) por su apoyo en microfabricación. Este trabajo fue apoyado en parte por el Europeo investigación Consejo Advanced Grant no. 321107 "CellO", Université de PSL (proyecto SwithNeuroTrails), ANR Investissement Avenir y la empresa Labex IPGG y Equipex.
Materiales
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| Plasmalab System 100 | Oxford Instruments | To perform DRIE | |
| MJB4 mask aligner | SUSS MicroTec | SU-8 photolithography | |
| NXQ 4006 Mask aligner | Neutronix Quintel | Photolithography associated to DRIE | |
| Plasma cleaner | Diener Electronic | Pico PCCE | |
| Cell culture hood | ADS Laminaire | Optimale 12 | |
| Centrifuge | Thermo Fisher Scientific | Heraeus Multifuge X1R | |
| Incubator 37 °C 5% CO2 | Panasonic | MCO-170AICUVH-PE IncuSafe CO2 Incubator | |
| Epifluorescence microscope | Leica | DMi8 | |
| PDMS Oven between 65 and 80 °C | Memmert | ||
| Mechanical profilometer | Veeco | Dektak 6M Stylus Profiler | |
| Optical profilometer | Veeco | Wyko NT9100 | |
| Vacuum desiccator | Verrerie Villeurbanaise (Kartel Labware) | 230KAR | Diameter 200 mm |
| Ultrasonic bath sonicator | Labo Moderne | SHE1000 | Volume of the bath: 0.8 liter |
| Hotplate | Stuart SD162 | SD162 | |
| Masks | |||
| DRIE | Supplementary data | ||
| Su8 Mask 1 | Supplementary data | ||
| Su8 Mask 2 | Supplementary data | ||
| Su8 Mask 3 | Supplementary data | ||
| S1805 calibration stripes | Supplementary data | ||
| Small laboratory equipment | |||
| 1.5 mm hole puncher | Sigma-Aldrich | 29002519 (US reference) | |
| Scalpel or razor blade | |||
| 9" Stainless Steel Flat Spatula with Spoon | VWR International | 82027-532 | To demold PDMS |
| Top Lip Wafer Handling | VWR International | 63042-096 | |
| Curved tweezer | FST | Dumont #7 Forceps - Standard / Dumoxel | To manipulate glass coverslips |
| Substrates | |||
| Silicon wafer | Prolog Semicor Ltd | ||
| 24x24 mm glass coverslips | VWR | 631-0127 | |
| Photoresists and developpers | |||
| AZ 1518 positive photoresist | Microchemicals GmbH | Before DRIE process, thicknes 1.8 µm | |
| AZ 351B developer | Microchemicals GmbH | To develop positive AZ 1518 photoresist | |
| SU-8 2007 | MicroChem | ||
| SU-8 2025 | MicroChem | ||
| SU-8 2050 | MicroChem | ||
| PGMA developer | Technic | To develop SU-8 negative photoresist | |
| Microposit S1805 resist | Chimie Tech Services | Positive photoresist used to obtain 0.5µm high structures | |
| MF 26A developer | Chimie Tech Services | To develop positive S1805 photoresist | |
| Laboratory consumables | |||
| Disposable plastic pipette 3 mL | LifeTechnologies - ThermoFisher | ||
| P100 Petri dishes | TPP | 93100 | |
| 20 mL syringe | Terumo | SS+20ES1 | |
| Transparent scotch tape | |||
| Square wipes | VWR | 115-2148 | |
| Parafilm | DUTSCHER | 90260 | Plastic paraffin film |
| Chemicals | |||
| (3-Methacryloxypropyl)trichlorosilane | abcr | AB 109004 | |
| PDMS and curing agent Sylgard 184 | Sigma-Aldrich | 761036 | |
| isopropanol | W292907 | ||
| poly-ornithine | Sigma-Aldrich | P4957 - 50 mL | |
| Ethanol absolute | Sigma-aldrich | 02865 | 99.8% |
| 3-methacryloxypropyl-trimethoxysilane | Sigma-aldrich | M6514-25ML | (C4H5O2)-(CH2)3- Si(OCH3)3 |
| acetic acid | Sigma-aldrich | 71251-5ML-F | |
| Dextran 10kW conjugated with Alexa488 | LifeTechnologies - ThermoFisher | D22910 | Absoprtion at 488 nm |
| Dextran 10kW conjugated with Alexa647 | LifeTechnologies - ThermoFisher | D22914 | Absoprtion at 647 nm |
| Culture medium | |||
| MEM | LifeTechnologies - ThermoFisher | 21090-022 | |
| Horse Serum | LifeTechnologies - ThermoFisher | 26050088 | |
| B27 | LifeTechnologies - ThermoFisher | 12587-010 | |
| Glutamax 200 mM | LifeTechnologies - ThermoFisher | 35050-061 | |
| Sodium Pyruvate GIBCO 100 mM | LifeTechnologies - ThermoFisher | 11360-070 | |
| Gentamicin | LifeTechnologies - ThermoFisher | 15710-049 | |
| PBS | Sigma-Aldrich | D8537-500ML | |
| HBSS 10x | LifeTechnologies - ThermoFisher | 14180-046 | |
| Hepes 1M | LifeTechnologies - ThermoFisher | 15630-056 | |
| trypsin-EDTA | Sigma-Aldrich | 59418C-100ML | |
| Neurobasal | LifeTechnologies - ThermoFisher | 21103-049 | |
| Neurobasal without phenol red | LifeTechnologies - ThermoFisher | 12348-017 | |
| Softwares | |||
| Routine in Matlab for background normalization | Quantacell | Contact Victor Racine: victor.racine@quantacell.com | |
| ImageJ | To select specific ROI for image analysis | ||
| Routine | ImageJ | Supplementary data | |
| Routine | Matlab | Supplementary data |
Referencias
- Jun, S., Taheri-Araghi, S. Cell-size maintenance: universal strategy revealed. Trends Microbiol. 23 (1), 4-6 (2015).
- Zlotek-Zlotkiewicz, E., Monnier, S., Cappello, G., Le Berre, M., Piel, M. Optical volume and mass measurements show that mammalian cells swell during mitosis. J. Cell Biol. 211 (4), 765-774 (2015).
- Kim, G. H., Kosterin, P., Obaid, A. L., Salzberg, B. M. A mechanical spike accompanies the action potential in Mammalian nerve terminals. Biophys J. 92 (9), 3122-3129 (2007).
- Iacono, D., et al. Neuronal Hypertrophy in Asymptomatic Alzheimer Disease. J Neuropathol Exp Neurol. 67 (6), 578-589 (2008).
- Cheli, V. T., et al. Conditional Deletion of the L-Type Calcium Channel Cav1. 2 in Oligodendrocyte Progenitor Cells Affects Postnatal Myelination in Mice. J Neurosci. 36 (42), 10853-10869 (2016).
- Kneynsberg, A., Collier, T. J., Manfredsson, F. P., Kanaan, N. M. Quantitative and semi-quantitative measurements of axonal degeneration in tissue and primary neuron cultures. J Neurosci Methods. 266, 32-41 (2016).
- Fouquet, C., et al. Improving axial resolution in confocal microscopy with new high refractive index mounting media. PloS one. 10 (3), (2015).
- Aguet, F., et al. Membrane dynamics of dividing cells imaged by lattice light-sheet microscopy. Mol Biol Cell. 27 (22), 3418-3435 (2016).
- Roland, A. B., et al. Cannabinoid-induced actomyosin contractility shapes neuronal morphology and growth. eLife. 3, (2014).
- Lu, Y. B., et al. Viscoelastic properties of individual glial cells and neurons in the CNS. Proc Natl Acad Sci USA. 103 (47), 17759-17764 (2006).
- Chen, J., Xue, C., Zhao, Y., Chen, D., Wu, M. H., Wang, J. Microfluidic impedance flow cytometry enabling high-throughput single-cell electrical property characterization. Int J Mol Sci. 16 (5), 9804-9830 (2015).
- Bottier, C., et al. Dynamic measurement of the height and volume of migrating cells by a novel fluorescence microscopy technique. Lab Chip. 11, 3855-3863 (2011).
- Cadart, C., et al. Fluorescence eXclusion Measurement of volume in live cells. Methods Cell Biol. 139, 103-120 (2017).
- Marasso, S. L., et al. A novel graphene based nanocomposite for application in 3D flexible micro-supercapacitors. Materials Research Express. 3 (6), 065001 (2016).
- Welch, C. C., Goodyear, A. L., Wahlbrink, T., Lemme, M. C., Mollenhauer, T. Silicon etch process options for micro-and nanotechnology using inductively coupled plasmas. Microelectronic Engineering. 83 (4), 1170-1173 (2006).
- Fath, T., Ke, Y. D., Gunning, P., Götz, J., Ittner, L. M. Primary support cultures of hippocampal and substantia nigra neurons. Nat. Protoc. 4 (1), 78-85 (2008).
- Getting intensity values from single ROI. Image Intensity Processing Available from: https://imagej.net/Image_Intensity_Processing (2017)
- 19. Tools. ImageJ User Guide Available from: https://imagej.nih.gov/ij/docs/guide/146-19.html (2017)
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados