Method Article
Monitoraggio-cellula-trasmissione degli aggregati della proteina del Prion-come in Drosophila Melanogaster
In questo articolo
Riepilogo
Raccogliendo la prova sostiene l'idea che aggregati proteici patogeni associati con malattie neurodegenerative diffuse tra cellule con proprietà simil-da prioni. Qui, descriviamo un metodo che permette la visualizzazione della cellula--cellula diffusione del prione-come i complessi nell'organismo modello, Drosophila melanogaster.
Abstract
L'aggregazione della proteina è una caratteristica centrale della maggior parte delle malattie neurodegenerative, tra cui il morbo di Alzheimer (annuncio), malattia di Parkinson (MDP), malattia di Huntington (HD) e sclerosi laterale amiotrofica (ALS). Aggregati proteici sono strettamente associati con neuropatologia in queste malattie, anche se non è noto l'esatto meccanismo attraverso il quale l'aggregazione di proteine aberranti sconvolge la normale omeostasi cellulare. Emergendo dati forniscono l'appoggio importante per l'ipotesi che aggregati patogeni in AD, PD, HD e ALS hanno molte somiglianze ai prioni, che sono agenti infettivi sola proteina responsabili per le encefalopatie spongiformi trasmissibili. I prioni auto-replicano di templating la conversione delle versioni piegato in modo nativo della stessa proteina, causando la diffusione del fenotipo aggregazione. Come i prioni e proteine prioniche-like in annuncio, PD, HD e ALS mossa da una cellula a altra è attualmente un'area di intensa ricerca. Qui, è descritto un modello di Drosophila melanogaster che permette il monitoraggio della trasmissione del prion-like, cellula--cellula di aggregati di huntingtina (Htt) connesso con HD. Questo modello si avvale di strumenti potenti per manipolare l'espressione del transgene in molti tessuti differenti di Drosophila e utilizza una proteina citoplasmatica fluorescente contrassegnati al direttamente del prion-come trasferimento del rapporto di mutante Htt aggregati. D'importanza, l'approccio che descriviamo qui può essere utilizzato per identificare nuovi geni e vie che mediano la diffusione degli aggregati della proteina tra cellulari diversi tipi in vivo. Informazioni acquisite da questi studi si espanderà la limitata comprensione dei meccanismi patogenetici che sono alla base di malattie neurodegenerative e rivelare nuove opportunità per intervento terapeutico.
Introduzione
L'ipotesi del prion afferma che l'agente infettivo responsabile per le encefalopatie spongiformi trasmissibili (ad es., malattia di Creutzfeldt – Jakob in esseri umani, scrapie nelle pecore, malattia del deperimento cronico in cervi e alci e "malattia della mucca pazza" nei bovini ) è esclusivamente composto di proteine e privo di acidi nucleici1. In malattie da prioni, la proteina prionica cellulare (PrPC) assume una piega non nativi, stabile (PrPSc) che è altamente beta foglio-ricco e auto-propagarsi convertendo e reclutamento monomerico PrPC molecole in amiloide stabile funzioni di aggregazione. PrPSc aggregati utilizzano questo meccanismo di auto-replica per diffondere tra diverse cellule dell'organismo e anche tra gli organismi individuali2.
Misfolding e aggregazione è anche una caratteristica centrale della maggior parte delle malattie neurodegenerative (morbo di Alzheimer (annuncio), malattia di Parkinson (MDP), malattia di Huntington (HD) e sclerosi laterale amiotrofica (ALS))3. Formazione di assemblee di proteina aggregati intra - o extra - cellulare in queste malattie è strettamente associato con citotossicità4 e progredisce lungo percorsi altamente riproducibili e malattia-specifica attraverso il cervello sopra tempo5, 6. questi modelli di diffusione suggeriscono che aggregati patogeni associati a questi disturbi del prion-come le proprietà. Forte sostegno ora esiste per la trasmissione di prioni-come degli aggregati associati con l'annuncio, PD, HD e ALS - si sono sparsi dalla cellula--cellula e modello il cambiamento conformazionale di monomeriche forme della stessa proteina in cellule precedentemente inalterato7, 8.
La maggior parte degli studi che studiano da prioni-come diffusione di aggregati proteici fino ad oggi è stata effettuata utilizzando modelli di coltura di cellule di mammifero, dove gli aggregati trasferire nel citoplasma delle cellule naive dallo spazio extracellulare o da un'altra cella citoplasma9,10,11,12,13,14,15, o iniettando materiale contenente aggregato nel cervello di topo e monitoraggio aggregare aspetto fuori le iniezione sito16,17,18,19,20,21,22, 23. più recentemente, gli animali transgenici sono stati utilizzati per dimostrare che gli aggregati intracellulari diffondersi ad altre cellule all'interno di cervelli intatti24,25,26,27, 28,29,30. Qui, descriviamo un metodo per la visualizzazione diretta di trasferimento aggregati tra le singole celle nel cervello intatto di Drosophila melanogaster. Quasi due decenni fa in primo luogo sono stati sviluppati in Drosophila modelli di HD/poliglutammina (polyQ) malattie31,32 e hanno intestimabile molti meccanismi patogenetici che sono alla base di questi disordini 33. HD è un disordine neurodegenerative ereditato causato da una mutazione autosomica dominante del gene che codifica per la proteina huntingtina (Htt)34. Questa mutazione provoca dilatazione di un tratto poliQ nei pressi N-terminale di Htt di là di una soglia di patogena di ~ 37 Glutamine, causando la proteina di misfold e aggregazione35,36. Selvaggio-tipo Htt proteine contenenti < 37 Glutamine in questo tratto raggiungere loro ovile nativo, ma possono essere indotte a aggregato al diretto contatto fisico con un Htt aggregazione "seme"12,27,37. Sfruttiamo questo homotypic, aggregazione nucleato di selvaggio-tipo Htt come una lettura per il trasferimento del prion-like e citoplasmico voce di mutante Htt aggregati originari di altre cellule.
Determinare i meccanismi da cui aggregati del prion-come viaggio tra le celle può portare all'identificazione di nuovi bersagli terapeutici per le malattie neurodegenerative incurabili. Prendiamo vantaggio del ciclo di vita rapido, facilità d'uso e trattabilità genetica della Drosophila melanogaster definire meccanismi molecolari per cellula--cellula diffusione del mutante Htt aggregati. La nostra strategia sperimentale utilizza due sistemi di espressione binaria disponibile in Drosophila, la consolidata Gal4 specifici a Monte d'attivazione sequenza (Gal4-UAS) sistema38 e il recentemente sviluppato QF-QUAS sistema39. Questi due sistemi indipendenti di aggancio permette di limitare l'espressione dei transgeni Htt mutanti e wild-type a popolazioni distinte delle cellule all'interno della stessa volare40. Utilizzando questo approccio, esaminiamo da prioni-come diffusione del mutante Htt monitorando la ridistribuzione di citoplasmico Htt di selvaggio-tipo dallo stato normalmente diffusa, solubile a uno stato aggregato, una diretta conseguenza del contatto fisico con un mutante pre-formato Htt aggregazione "seme". Conversione di selvaggio-tipo Htt dal mutante Htt possa essere confermati mediante biochimici o trasferire tecniche biofisiche che segnalano interazioni proteina-proteina, quali l'energia di risonanza di fluorescenza (FRET)9,27,41 .
Soprattutto, possiamo anche accedere un gran numero di strumenti genetici in Drosophila per identificare geni e/o le vie che mediano del prion-come diffusione di aggregati proteici. Recentemente abbiamo utilizzato questo approccio per svelare un ruolo chiave per il recettore delle cellule superficiali dell'organismo saprofago,42,Draper43, nel trasferimento mutante Htt aggregati da un neurone assoni a vicina glia fagocitica in Drosophila centrale sistema nervoso (SNC)27. Così, l'approccio e la formazione immagine-base genetica che descriviamo qui può rivelare importanti informazioni di base biologiche su un fenomeno malattia rilevanti in simple-to-use ma organismo modello potente, Drosophila.
Protocollo
1. giunto Gal4 e QF-ha mediato l'espressione del Htt Transgene in Drosophila
- Raccogliere e/o generare transgenici di Drosophila melanogaster righe contenenti tessuto-specifici Gal4 o QF "driver", come pure le righe contenenti transgeni Htt wild-type o mutanti a valle del Gal4-UAS38 o39di QF-QUAS. Garantire che le proteine che vengono espresse da questi transgeni sono fuse a proteine fluorescenti o epitopo-etichetta per consentire per la differenziazione dei mutanti e wild-type Htt transgene prodotti al volo stesso. Vedere la Figura 1.
Nota: In genere usiamo frammenti dell'esone 1 dell'umano gene Htt27. Tuttavia, transgenici mosche invece possono essere generati per esprimere più Htt frammenti o altri geni se lo si desidera. - Pianificare la strategia genetica tale che transgeni Htt mutanti e wild-type si esprimerà in popolazioni cellulari distinti, non sovrapposte.
- Se si verifica qualsiasi sovrapposizione di espressione, mutanti e wild-type Htt proteine sintetizzate nelle stesse cellule saranno co-aggregazione e impedire il rilevamento di eventi del prion-like. Per evitare questo problema, utilizzare il Gal4 e QF specifici repressori, Gal80 e QS40, rispettivamente (Figura 1). Selezionare i driver Gal4 e/o QF inerente allo sviluppo controllato può aiuto per limitare l'espressione del mutante Htt a cellule post-mitotiche, eliminando la possibilità che la divisione cellulare potrebbe contribuire a diffondere aggregazione di cellula--cellula.
- Utilizzando condizioni di coltura standard, si accoppiano le mosche per generare progenie che esprimono la popolazione di cellule mutante Htt tramite QF (o Gal4) in un "donatore" e popolazione delle cellule di selvaggio-tipo Htt via Gal4 (o QF) in un "destinatario". Vedere la Figura 1 per una schematica che mostra una possibile combinazione genetica.
Nota: I driver QF e Gal4 che sono stati utilizzati per generare i dati riportati nelle figure 1-4 e nella nostra precedente pubblicazione27 includono il driver di neurone (ORN) recettori olfattivi DA1, Or67d-QF e il pan-glial driver, repo-Gal4. - Generare controllo mosche in parallelo che esprimono il selvaggio-tipo Htt in entrambe le popolazioni cellulari di QF - e Gal4-labeled.
- Raccogliere progenie del genotipo desiderato ed età degli animali come appropriato.

Figura 1 . Approccio genetico per accoppiati espressione dei mutanti e wild-type transgeni Htt utilizzando i sistemi di espressione binaria QF-QUAS e Gal4-UAS. Nella "cella A," una proteina Htt mutante mCherry-etichetta contenente un tratto poliQ patogeni-lunghezza (Q91) viene espresso utilizzando un driver QF situato a valle di un promotore tessuto-specifica un ("PA"). In "delle cellule B," un Htt di selvaggio-tipo YFP-etichetta contenente un tratto poliQ normali (Q25) è espresso tramite un driver di Gal4 comandati dal tessuto-specifico promotore B ("PB"). Nella figure 2-4, Or67d-QF è stata usata per guidare QUAS-HttQ91-mCherry espressione in ORNs DA1 e repo-Gal4 è stata usata per esprimere YFP-HttQ25-UAS in tutti glia27. D'importanza, HttQ91-mCherry è espresso solo in cellule che esprimono QF in virtù della sequenza QUAS posizionato a Monte del transgene. Allo stesso modo, HttQ25-YFP si esprime solo tramite Gal4, che riconosce specificamente il UAS. Se qualsiasi sovrapposizione nella distribuzione del tessuto dei driver QF e Gal4 viene rilevata, possono essere introdotto transgeni codifica QS in cellule che esprimono Gal4 e Gal80 in cellule che esprimono QF. Aggiungendo tag proteina fluorescente sul wild-type e mutante Htt consente la differenziazione delle due proteine durante la formazione immagine e la capacità di misurare FRET tra coppie di appropriato donatore/accettore (ad es., CFP/YFP o YFP/mCherry). Clicca qui per visualizzare una versione più grande di questa figura.
2. micro-dissezione e la fissazione del cervello adulto della drosofila
Nota: Questa procedura di dissezione è stata modificata da una precedente pubblicazione44e può essere utilizzata per preparare i cervelli per imaging segnale di fluorescenza diretta da fusioni di proteina Htt-fluorescenti. Modifiche alla procedura che può essere fatto per immunostaining i cervelli sono discussi nella sezione successiva.
- Raccogliere i seguenti materiali e metterli su ghiaccio: tampone fosfato salino contenente 0.03% Triton X-100 (PBS/T); provette per microcentrifuga contenente 970 µ l di soluzione fissante paraformaldeide al 4% (PFA), preparata con l'aggiunta di 200 µ l 20% PFA a 770 µ l PBS/T; un piatto di vetro trasparente contenente un pozzo; una pipetta da trasferimento monouso; forcipe di dissezione due (uno n ° 3 e n. 5).
- Anestetizzare le mosche adulte usando CO2 e trasferirli in un pozzetto del vetro piatto sul ghiaccio.
- Utilizzando una pipetta da trasferimento, aggiungere una piccola quantità (~ 500 µ l) di PBS freddo/T la ben contenente le mosche anche per quanto riguarda un pozzo vuoto. Evitare di introdurre troppe bolle come possono interferire con la dissezione.
- Posizionare il piatto di vetro su una superficie piana sotto un microscopio per dissezione e regolare l'ingrandimento fino a quando il corpo volo riempie il campo visivo ed è a fuoco.
- Posizionare un paio di fonti di luce a collo di cigno in modo che la luce è illuminante entrambi i lati del vetro piatto. Ancorare un tessuto di laboratorio piegato sotto il piatto di vetro per scartare parti corpo/cuticola durante la dissezione.
- Utilizzando le pinze di n ° 3 nella mano non dominante, trasferire un singolo fly nel contenente PBS/T. immobilizzare al volo afferrando in possesso del suo addome con il lato ventrale rivolto verso l'alto.
- Mantenendo al volo completamente sommerso in PBS/T, rimuovere la testa di Mosca con il forcipe di n. 5 nella mano dominante. Inserire una punta di questa pinza sotto la cuticola nel piccolo spazio adiacente la proboscide su un lato della testa vola. Fissare il grip sulla testa pizzicando le pinze per afferrare l'occhio da entrambi i lati.
- Rimuovere la testa di Mosca dal suo corpo tirando le due pinze. Smaltire il corpo volo su un tessuto di laboratorio posizionato nelle vicinanze. Mantenere la pressione sul forcipe di mano dominante affinché la testa di Mosca non è perduta.
- Un suggerimento di posizione delle pinze n. 3 nella non-dominante a mano nello stesso piccolo spazio sotto la cuticola su altro lato della proboscide. Una volta posizionato, pizzicare le pinze per afferrare gli occhi nella stessa posizione su entrambi i lati della testa.
- Una volta fissato il grip sulla cuticola, tirare delicatamente il forcipe apart a 180°. Questa azione si romperà la testa cuticola senza danneggiare il cervello. Eliminare residui di cuticola su un tessuto di laboratorio.
- Anche se ideale, la cuticola testa non venga completamente rimosso in un unico passaggio. In questo caso, rimuovere il cuticola pezzo per pezzo, fino a quando il cervello è completamente esposto. Fare attenzione a non per danneggiare il cervello, evitando il contatto diretto con il forcipe.
- Rimuovere il cervello dissecato dalla PBS/T afferrare ahold di un annesso trachea, oppure aspirando il cervello nello spazio tra le punte di forcipe per azione capillare.
Nota: Rimozione di Trachea è facoltativo, come si tende a non oscurare i lobi antennali sulla superficie anteriore del cervello dove abbiamo immagine in genere. Tuttavia, se la trachea interferisca con imaging, rimuoverli con attenzione così il cervello non è danneggiato da questa manipolazione. - Trasferire il cervello volo in uno dei tubi del microcentrifuge contenente soluzione fissante sul ghiaccio. Assicurarsi che il cervello si stacca dal forcipe e sia immersa nella soluzione fissante.
- Una volta che tutti i cervelli sono stati sezionati, sottoporli a un "breve-fix" inserendo i tubi chiusi su un nutator per ~ 5 min a temperatura ambiente al buio.
Nota: Questo passaggio breve fissazione assicura che i cervelli sono più facili da gestire nei passaggi successivi e non aderiscono ai lati del tubo del microcentrifuge. - Rimuovere la maggior parte della soluzione fissante con una pipetta P1000 e scartarla.
- Evitare di aspirare cervelli dal tubo durante questo e qualsiasi passaggio di lavaggio successivo. Impostare il P1000 a un volume più basso (ad es., 650 µ l) e rimuovere il surnatante in due passaggi. Inoltre, tenere la microcentrifuga in linea con una fonte di luce (ad es. plafoniere) mentre ad aspirazione per visualizzare meglio il cervello.
- Aggiungere 1 mL di PBS/T fresco ai cervelli. Lavare rapidamente (< 1 min) al buio, aspirare il supernatante dal cervello e scartare.
- Ripetere il lavaggio con PBS/T al buio a temperatura ambiente secondo il seguente calendario: 2 lavaggi rapidi (< 1 min), 1 X 5 min, 3 X 20 min e 1 X 1 h lavare. Garantire la tops su microcentrifuga e posizionare i tubi su un nutator tra lavaggi.
- Se si desidera la colorazione DAPI, Sostituisci il lavaggio finale 1h con 1 X 30 min di incubazione con 250 ng/mL DAPI diluito in PBS/T, seguita da 2 X rapida e 1 X 20 min lavaggi.
- Dopo l'ultimo lavaggio, rimuovere la maggior parte del surnatante di tampone di lavaggio, avendo cura di non per disturbare i cervelli e aggiungere 30 µ l di reagente antifade basati su glicerolo ai cervelli. Incubare a 4 ° C al buio senza movimento per almeno 1 h e fino a 24 h.
3. modifiche alla sezione 2 per i cervelli di Immunostaining adulto
Nota: Utilizzare questo protocollo per l'imaging non fluorescente proteine o per fusioni di proteina fluorescente con debole fluorescenza.
- Aumentare la concentrazione di Triton X-100 in PBS/T da 10 volte (PBS + 0,3% Triton X-100) per ogni passo. Questo assicura che abbastanza detersivo è presenta per permeabilize le membrane cellulari e consentire gli anticorpi accedere a spazi intracellulari.
- Difficoltà i cervelli in 4% PFA in PBS/T per 20 min a temperatura ambiente.
- Dopo aver eseguito tutti i lavaggi, incubare i cervelli in tampone bloccante preparata (PBS/T + 5% di siero di capra normale (NGS)) per 30 min a temperatura ambiente al buio.
- Rimuovere e gettare il tampone bloccante. Aggiungere 0,5 mL di soluzione di anticorpo primario per tubo preparato come un mix master diluendo gli anticorpi primari come appropriato in tampone bloccante fresco (Vedi Tabella materiali comunemente utilizzati anticorpi e diluizioni). Incubare i cervelli in anticorpi primari su un nutator per almeno 24 h a 4 ° C al buio.
- Rimuovere la soluzione di anticorpo primario dal cervello utilizzando un P1000 e riservare in una nuova provetta. Aggiungere 1 mL di PBS/T per lavare il cervello.
Nota: La soluzione di anticorpo primario riservato può essere conservata a 4 ° C fino a quattro settimane. Abbiamo correttamente riciclato gli anticorpi primari fino a due volte in esperimenti successivi. - Lavare rapidamente i cervelli (< 1 min) in PBS/T, aspirare il supernatante dal cervello e scartare. Ripetere ancora una volta questo lavaggio rapido.
- Continuare il lavaggio con 1 mL di PBS/T a temperatura ambiente secondo il seguente calendario: 1 X 5 min, 3 X 20 min e 1 X 1 h lavare. Garantire la tops su microcentrifuga e posizionare i tubi su un nutator durante i lavaggi.
- Dopo l'ultimo lavaggio, rimuovere la maggior parte del surnatante PBS/T e aggiungere 0,5 mL di soluzione di anticorpo secondario preparata come un mix master diluendo gli anticorpi come appropriato in tampone bloccante fresco (Vedi Tabella materiali per gli anticorpi comunemente usati e diluizioni). Incubare cervelli in anticorpi secondari per 24 h a 4 ° C al buio.
- Ripetere i passaggi di lavaggio in punti 3.5, 3.6 e 3.7 dopo incubazione con gli anticorpi secondari.
- Dopo il lavaggio finale, rimuovere la maggior parte del tampone di lavaggio e assicurarsi che tutti i cervelli sono situati nella parte inferiore del tubo. Aggiungere 30 µ l di reagente antifade ai cervelli. Incubare a 4 ° C al buio senza movimento per 16-24 h.
4. whole Brain montaggio
- Posizionare un vetrino per microscopio sotto un microscopio per dissezione e un'etichetta con informazioni di identificazione.
Nota: In alternativa, il cervello può essere montato direttamente su un vetro di copertura per accedere a entrambi i lati del cervello durante la formazione immagine. Un protocollo che descrive questa procedura di montaggio è stato precedentemente pubblicati45. - Rimuovere i cervelli da ciascuna provetta per microcentrifuga utilizzando una punta smussata dispensare (preparati utilizzando una lama di rasoio per rimuovere il fondo ~ 1 cm fuori una punta di 1-200 µ l) e trasferirli nel bel mezzo della diapositiva. Trasferimento come reagente antifade poco con i cervelli come possibile.
- Utilizzare pinze per posizionare i cervelli l'orientamento desiderato (ad es., dorsale rivolta verso la parte superiore della diapositiva e superficie anteriore rivolto verso l'alto per il lobo antenne di imaging). Quando orienta i cervelli, considera il percorso della luce del microscopio per essere utilizzato per l'imaging di loro.
- Rimuovere eccesso di reagente antifade dalla diapositiva utilizzando l'angolo aguzzo di un tessuto di laboratorio piegato senza disturbare i cervelli. Far scivolare il sedere per ~ 5-10 minuti al buio per consentire i cervelli di aderire alla diapositiva.
- Circondano i cervelli volare con quattro piccoli pezzi di vetro rotto coperchio inserito su ogni lato del cervello a formare un quadrato che è ~ 19 mm x 19 mm. Posizionare uno spigolo di un vetro di copertura di 22 x 22 mm n. 1.5 appena di fuori di uno di questi piccoli pezzi di vetro e abbassare delicatamente il vetrino coprioggetto sopra i cervelli per completare il ponte-mount.
- Pipettare lentamente fresco antifade reagente per riempire la superficie sotto il vetrino coprioggetti, facendo attenzione a non disturbare il coprioggetto o il cervello. Eliminare qualsiasi eccesso antifade utilizzando l'angolo aguzzo di un tessuto di laboratorio piegato senza contatto diretto con il vetrino coprioggetti.
- Aggiungere una goccia di smalto chiaro, rapido-essiccamento a ciascuno dei quattro angoli del coprivetrino. Lasciare per asciugare per ~ 10 min. Quindi, sigillare i quattro bordi del coprivetrino con smalto per unghie per racchiudere completamente il cervello.
- Cervelli di immagine immediatamente o conservare a 4 ° C fino al momento.
- Se la formazione immagine di fluorescenza intrinseca da fusioni di proteina Htt-fluorescente, immagine il cervello entro 24 h per il miglior segnale.
5. imaging e quantificazione del Prion-come trasmissione di aggregati
- Immagine il cervello montato usando un microscopio confocale, equipaggiato con un 40x o 60/63 X obiettivo olio per raccogliere immagini di fetta z attraverso la regione del cervello dove vengono espressi i driver selezionati Gal4 e QF (Figura 2A, B).
- Eccitare la proteina fluorescente fusioni o immunolabeled proteine usando i laser appropriati (ad es., 488 nm per GFP/YFP/FITC o 552 nm per mCherry/Cy3). Impostare windows di rilevamento che catturano fluorescente massimo segnale eliminando canale cross-talk utilizzando un sistema di rilevazione spettrale o specifici per ogni fluoroforo emissione filtri passa-banda/lungo.
Nota: Essere attento quando imaging aggregati proteici. È facile per pixel al centro di ogni aggregato si impregni, soprattutto in grandi aggregati. Tentare di ridurre al minimo la saturazione dell'immagine, ma essere consapevoli del rischio di perdere il segnale da più piccoli aggregati specie (Figura 2B, C, D). Troppa saturazione aumenterà lo sfondo dell'immagine, rendendo più difficile l'identificazione puncta isolato. Prova diversa impostazioni per ottimizzare prima di prendere le immagini finali in ogni set di dati di imaging.
- Eccitare la proteina fluorescente fusioni o immunolabeled proteine usando i laser appropriati (ad es., 488 nm per GFP/YFP/FITC o 552 nm per mCherry/Cy3). Impostare windows di rilevamento che catturano fluorescente massimo segnale eliminando canale cross-talk utilizzando un sistema di rilevazione spettrale o specifici per ogni fluoroforo emissione filtri passa-banda/lungo.
- Analizzare i dati di quantificare singola puncta sia manualmente spostando attraverso singole z-sezioni (ad esempio, Figura 2C e Figura 4A) o dopo il rendering in 3 dimensioni (Figura 3 le fette di confocale A, B).
Nota: Questo può essere fatto in immagine J o altre software di elaborazione di immagini.- Se gli aggregati sono ben separati e fluorescenza di fondo minimo non esiste, utilizzare software di analisi di immagine per identificare, quantificare e analizzare gli aggregati come oggetti"distinti" o "superfici" in una ricostruzione 3D della pila confocale (sistematicamente Figura 3 B).
Nota: Il Software azioni descritte sono specifiche per lo strumento e il software utilizzato qui (Vedi la Tabella materiali).- Visualizzare una serie z confocale in modalità di visualizzazione 3D. Utilizzare la procedura guidata di "Analisi" per individuare i singoli punti in un canale selezionato (ad es. il canale rosso per HttQ91-mCherry aggrega nella Figura 3). Regolare la soglia e filtri per rappresentare accuratamente tutti gli aggregati di dimensioni eterogeneo come singoli oggetti nell'immagine.
- Abilitare "Dividere gli oggetti" sotto "L'elaborazione pre-filtro binario" per separare strettamente associati aggregati che vengono uniti in modo aberrante dall'algoritmo software. Si noti che il numero totale di oggetti e relative misurazioni associati viene segnalato sotto "Misure."
Nota: Questo metodo per quantificare mutante Htt aggregati non è favorevole per il protocollo di immunostaining perché il centro di aggregati amiloidi, è impenetrabile agli anticorpi. Di conseguenza, gli aggregati vengono visualizzati sul microscopio come anello-come le strutture, e software di analisi di immagine è in grado di identificare accuratamente e distinguere questi singoli "punti".
- Quantificare gli aggregati di selvaggio-tipo Htt manualmente spostando attraverso lo z-stack e segnando aggregati di selvaggio-tipo Htt, assicurandosi che non aggregati sono doppio-segnato se appaiono in più di una fetta (Figura 4A).
Nota: Questo approccio manuale quantificazione può essere utilizzato quando il numero degli aggregati in ogni stack confocale è ragionevole (ad es., 20-50). È anche utile quando software di analisi di immagine non può distinguere singola puncta dal circostante segnale nello stesso canale (ad es., segnale da Htt di selvaggio-tipo diffuso in cellule vicine) (Figura 2B, C e Figura 4 A). quantificare gli aggregati di Htt di selvaggio-tipo può essere difficile anche perché molte strutture normali del cervello appaiono punctate (ad esempio, i processi delle cellule e sinapsi). Co-localizzazione di segnali Htt wild-type e mutanti può essere utilizzato come criterio di selezione; Tuttavia, è possibile che alcuni mutanti Htt "semi" scenda sotto il limite di rilevazione del confocale. Una proteina diversa da Htt che non viene aggregato in cellule del destinatario (ad esempio, GFP) può essere utilizzata per etichettare strutture indipendenti punctate nel cervello.
- Se gli aggregati sono ben separati e fluorescenza di fondo minimo non esiste, utilizzare software di analisi di immagine per identificare, quantificare e analizzare gli aggregati come oggetti"distinti" o "superfici" in una ricostruzione 3D della pila confocale (sistematicamente Figura 3 B).
- Utilizzare software di analisi di immagine per eseguire ulteriore caratterizzazione dei singoli aggregati. Ad esempio, determinare la distribuzione delle dimensioni degli aggregati (Figura 3C), percentuale co-localizzazione tra mutanti e wild type Htt proteine (Figura 4A), o misurare direttamente le interazioni proteina-proteina mediante FRET ( Figura 4 B).
- Determinare la distribuzione di dimensione delle singola puncta utilizzando un algoritmo di rilevamento di spot o superficie che identifica con precisione tutti gli aggregati visibili in un determinato canale. Utilizzare il software di analisi di immagine per prendere le misure pertinenti dei punti o superfici, ad esempio ottenere 'diametro aggregato' (fig. 3 C), 'volume' o informazioni di 'intensità' da 'Misure' nella procedura guidata di "Analisi" del software descritto al punto 5.2.1.
Nota: Nella Figura 3C, segnaliamo la distribuzione dei diametri per tutte le HttQ91-mCherry puncta, identificato in un singolo glomerulo DA1. - Determinare la percentuale co-localizzazione tra HttQ25-YFP e HttQ91-mCherry aggregati spostando manualmente affettare attraverso un z-stack confocale (metodo più preciso). Tuttavia, fare attenzione a non per contare eventuali aggregati due volte. Per evitare questo, contare solo aggregati in una particolare sezione z Se il piano di messa a fuoco è attraverso il centro dell'aggregato (Figura 4A).
- Calcolare efficienza FRET per aggregati HttQ25-YFP indotti con colocalized HttQ91-mCherry segnale utilizzando il metodo di photobleaching accettore.
- In primo luogo, eliminare potenziali cross-talk tra canali YFP e mCherry attraverso la creazione di finestre di rilevamento per segnali mCherry-YFP sola e che non producono nessun segnale in altro canale. Utilizzando queste impostazioni, prendere un 'prima immagine' di singola puncta HttQ25-YFP e loro associato HttQ91-mCherry segnale ( Figura 4B).
- Quindi, photobleach la mCherry segnale regolando il laser rosso (ad es., 552 nm) a 100% intensità e scansione fino a quando il segnale è andato. Ripristinare le impostazioni utilizzate per la ' prima immagine' e prendere una 'immagine dopo' dei puncta stesso (Figura 4B).
- Calcolare misure di intensità di fluorescenza per ogni punctum prima e dopo photobleaching utilizzando software di analisi di immagine.
- Calcolare l'efficienza FRET sottraendo YFP donatore di fluorescenza misurata nella 'prima immagine' (YFPiniziale) da YFP donatore in fluorescenza il 'immagine' (YFPfinale). Dividere questo valore per YFPfinalee moltiplicare per 100.
Nota: Il FRET efficienza può essere calcolata pixel-per-pixel o complessiva per ogni aggregazione HttQ25-YFP (Figura 4B). Accettore photobleaching è una tecnica particolarmente utile per calcolare l'efficienza FRET, quando il segnale di mCherry connesso con ogni HttQ25-YFP punctum è sufficientemente elevato per produrre rilevabile YFP dequenching dopo mCherry photobleaching.
- Determinare la distribuzione di dimensione delle singola puncta utilizzando un algoritmo di rilevamento di spot o superficie che identifica con precisione tutti gli aggregati visibili in un determinato canale. Utilizzare il software di analisi di immagine per prendere le misure pertinenti dei punti o superfici, ad esempio ottenere 'diametro aggregato' (fig. 3 C), 'volume' o informazioni di 'intensità' da 'Misure' nella procedura guidata di "Analisi" del software descritto al punto 5.2.1.
Risultati
I metodi descritti qui produrranno dati affidabile dimostrando del prion-come trasferimento di aggregati proteici Htt dalla popolazione di una cella a altra nel fly intatto CNS. Conversione di selvaggio-tipo Htt da diffusa a punctate è osservata da fluorescenza diretta di questa proteina di fusione di YFP nel destinatario glia come risultato HttQ91-mCherry espressione nel donatore ORNs (Figura 2A-C e Figura 4A, B). Accurati report degli eventi di trasferimento del prion-come tra queste due popolazioni di cellule richiede un'attenta selezione di mosche transgeniche e driver Gal4/QF di produrre livelli di forte espressione di transgeni di Htt mutanti e wild-type senza alcuna sovrapposizione durante sviluppo o in età adulta. Inoltre, disegno premuroso di transgeni fusione proteina fluorescente-Htt possibile attivare potenti analisi a valle. Ad esempio, mutanti e wild type Htt aggregati possono essere quantificati come oggetti punctati sia manualmente (Figura 2C e Figura 4A) o utilizzando software di analisi di immagine (Figura 3A, B), può essere misurata e caratterizzato ulteriormente come aggregazione popolazioni (Figura 3C), possono essere valutati per la co-localizzazione tra mutante e proteine di selvaggio-tipo (Figura 4A) e può essere analizzato per FRET27 (Figura 4 B). Queste analisi richiedono fusione di mutanti e wild-type Htt ai tag di proteina fluorescente con proprietà di fluorescenza sufficientemente separati, ma con abbastanza sovrapposizione spettrale per abilitare FRET tra coppie di donatore e accettore (ad es., CFP/YFP9 o YFP/mCherry27).
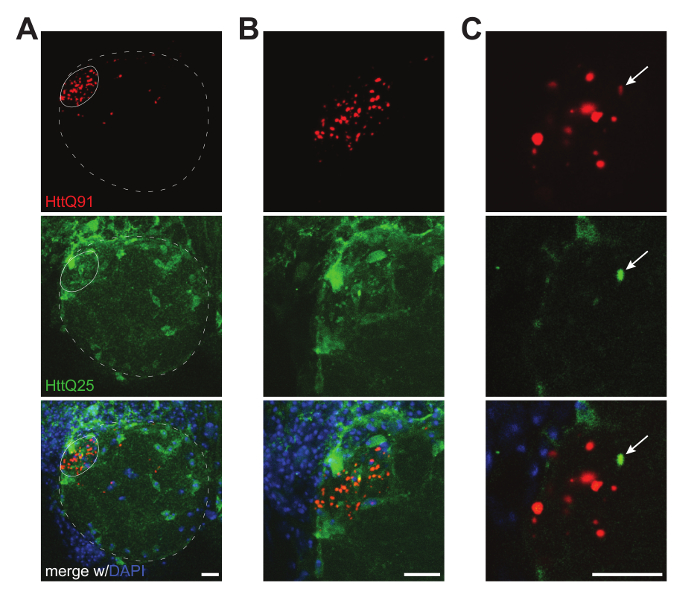
Figura 2 . Immagini confocal del prione-come conversione di glial HttQ25-YFP da un neurone HttQ91-mCherry aggregati. (A) massima proiezione intensità di ~ 30 µm di confocal fette mostrando un lobo antenne da una Mosca maschio esprimendo HttQ91-mCherry (rosso) in assoni DA1 ORN utilizzando Or67d-QF e HttQ25-YFP (verde) in cellule gliali utilizzando repo-Gal4. I confini approssimativi del lobo antenne e DA1 glomerulo, cui assoni DA1 ORN terminano, sono indicati dalle linee punteggiate e solide, rispettivamente. (B) massima proiezione intensità da ~ 20 µm di confocal fette mostrando una vista ingrandita della regione glomerulare DA1 da a. (C) A singola 0.35 µm confocale fetta mostrando una singola puncta HttQ25-YFP e relativo associato (segnale HttQ91-mCherry indicato dalla freccia in ogni canale). Il segnale nel canale rosso è stato migliorato per visualizzare co-localizzazione tra HttQ25-YFP e segnali di HttQ91-mCherry. Tutte le immagini sono state acquisite utilizzando un obiettivo di olio 40 X 1.4NA. Barre di scala = 10 µm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3 . Analisi tridimensionale di HttQ91-mCherry aggregati in assoni DA1 ORN. (A) un 3D rappresentazione degli aggregati di HttQ91-mCherry espresso nel glomerulo DA1 via Or67d-QF utilizzando gli stessi dati mostrati nella Figura 2B. (B) una schermata che Mostra singoli oggetti o "macchie" identificati dai dati grezzi in (A) utilizzando un pacchetto di software di analisi di immagine. Il software identificato 56 oggetti di varie dimensioni in questa canale/immagine. Il punto indicato dalla freccia (B) è stato misurato per hanno un diametro di ~ 1,2 µm. frecce scegliere posizioni dove due oggetti inesatto vengono uniti in uno spot per il software, probabilmente a causa della vicinanza dei puncta individuo. Per ovviare a questo, le impostazioni di soglia diversi devono essere testate nel software e/o macchie Unite devono essere separati manualmente se possibile. Barre di scala = 10 µm. (C) istogramma mostra la distribuzione dei diametri misurati dal software per la HttQ91-mCherry "macchie" illustrate in (B). Clicca qui per visualizzare una versione più grande di questa figura.
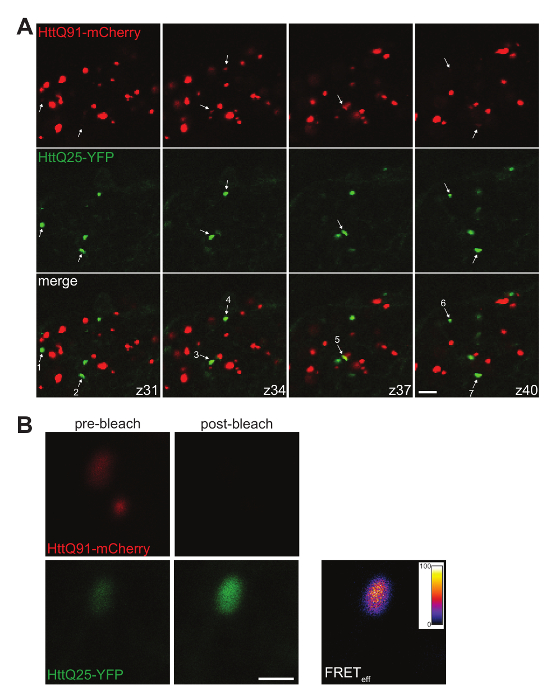
Figura 4 . Co-localizzazione e analisi FRET di indotto aggregati HttQ25-YFP. (A) A montaggio di 4 singoli 0,35 µm confocale z-fette da un cervello maschile volo esprimere HttQ91-mCherry in ORNs DA1 utilizzando Or67d-QF e HttQ25-YFP in glia utilizzando repo-Gal4. I segnali sono stati adeguati affinché anche piccoli aggregati di HttQ91-mCherry sono visibili e indotto HttQ25-YFP aggregati si distinguono dalle circostanti diffusa del segnale. Le fette mostrate sono separate da ~ 1,0 µm (numero di slice indicato all'angolo inferiore destro delle immagini unite) affinché più complessi possono essere osservati. Le frecce indicano puncta HttQ25-YFP che sono stati determinati per essere a o vicino a fuoco in quella particolare z-fetta spostando manualmente tramite lo stack di z. Dei sette HttQ25-YFP puncta indicato qui, sei hanno rilevabile associato HttQ91-mCherry segnale (cioè, 86% degli aggregati HttQ25-YFP co-localizza con HttQ91-mCherry). Si noti che il segnale di mCherry associato HttQ25-YFP puncta è spesso più debole rispetto alla maggior parte dei puncta mCherry-positivi nel glomerulo DA1. Barra della scala = 5 µm. (B) A HttQ25-YFP/HttQ91-mCherry-co-localizzato punctum prima (pannelli a sinistra) e dopo (destra pannelli) mCherry (accettore) photobleaching. L'aumento della fluorescenza YFP (donatore) fu usato per produrre un'immagine di efficienza (FRETFEP) di FRET pixel-per-pixel utilizzando il AccPbFRET plug-in per ImageJ46. Questo particolare aggregato ha un complessivo FRETeff del 61%. Barra della scala = 1 µm. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Come i numeri dei pazienti affetti da malattie neurodegenerative continua ad aumentare, c'è un urgente bisogno di aumentare la comprensione della patogenesi molecolare di queste malattie, in modo che possono essere sviluppate terapie migliori. Qui, descriviamo i metodi che consentono il monitoraggio del prion-come trasmissione degli aggregati della proteina patogena tra diversi tipi di cellule nell'organismo modello, Drosophila melanogaster. Recentemente abbiamo utilizzato questa metodologia per dimostrare da prioni-come trasmissione del mutante Htt aggregati in vivo e per identificare un fagocitico recettore che media la diffusione di questi aggregati dai neuroni a glia27. Il nostro approccio sfrutta diversi vantaggi dell'utilizzo di Drosophila per studiare la malattia genetica umana: il ciclo di vita breve e vasto set di strumenti genetici, che possono accelerare la scoperta di informazioni biologiche di base che sono terapeuticamente rilevanti.
I metodi che descriviamo qui offrono due grandi vantaggi rispetto altri animale esistente e modelli di cultura per la trasmissione di prioni-come cellulare: (1) l'agente causativo aggregante (ad es., mutante Htt) è prodotto in una cella che risiedono in un tessuto intatto e (2) espressione della versione piegata normalmente della stessa proteina (ad es., selvaggio-tipo Htt) in una popolazione di cella separata fornisce prontamente accessibile "reporter" per gli eventi del prion-come. Abbiamo raggiunto espressione del mutante e del selvaggio-tipo Htt proteine in popolazioni di cellule non sovrapposte all'interno dell'organismo stesso utilizzando sofisticati strumenti genetici che si sono affermati in Drosophila40. Poiché molti driver diversi tessuto-specifici Gal4 e QF sono prontamente disponibili, esame del prion-come trasferimento tra essenzialmente qualsiasi tipi distinti delle cellule nel corpo volo è fattibile.
Un componente critico dell'approccio sta ottenendo segregata espressione del mutante e del selvaggio-tipo Htt proteine in popolazioni differenti delle cellule all'interno dello stesso animale. Qualsiasi sovrapposizione di espressione deve essere eliminato in modo che l'aggregazione di Htt wild type in cellule recettive citoplasmico penetrazione del prione-come aggregati originari del donatore cellule9,12,27, segnala in modo preciso 41. Questo è possibile con l'introduzione di ulteriori strumenti genetici (ad es., Gal80 e QS repressori40) per alleviare questo problema. Una volta che il genotipo ideale è progettato e selezionato, è necessario stabilire un metodo sistematico per quantificare puncta. Questo dipenderà in larga misura il numero di cellule che sono etichettati, il numero di aggregazioni che appaiono e il rapporto segnale-rumore del campione. Criteri quali co-localizzazione e/o FRET positivi possono essere utilizzati per analizzare i dati, come abbiamo descritto qui in Figura 4. Tuttavia, limitare la selezione di selvaggio-tipo Htt aggregati sulla base di queste caratteristiche possono portare a sottovalutazione degli eventi del prion-come trasferimento, poiché alcuni semi di aggregazione Htt mutante potrebbero scendere di sotto il limite di rilevamento del microscopio confocale.
L'approccio in vivo qui descritto non è esclusivo per prioni-come comportamento di aggregati associato HD o anche altri disturbi polyQ. Transgenici mosche possono essere sviluppati per esaminare da prioni-come diffusione dell'alfa-synuclein nel PD, tau in annuncio e SOD1 o TDP-43 in ALS utilizzando lo stesso paradigma sperimentale. Per ciascuna di queste proteine, un mutante di aggregazione-incline dovrebbe essere espressi in cellule del donatore e una versione solubile della proteina stessa che aggregati soli quando nucleate dovrebbero essere espresso in cellule recettive. Questo paradigma sperimentale può anche essere utile per investigare l'idea emergente che patogeni proteine associate con differenti malattie potrebbero interagire tramite un meccanismo di cross-semina47. Infine, gli strumenti genetici una miriade disponibili in Drosophila possono essere applicati per studiare e identificare i meccanismi molecolari alla base di citoplasma al citoplasma diffusione di aggregati proteici patogeni associati a queste malattie mortali.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Ringraziamo i membri dei laboratori Kopito, Luo e Pearce per molte discussioni utili durante lo sviluppo di questi metodi. Ringraziamo anche Brian Temsamrit per la lettura critica di questo manoscritto. Questo lavoro è stato sostenuto dall'Università delle scienze e le fondazioni di beneficenza W.W. Smith.
Materiali
| Name | Company | Catalog Number | Comments |
| Phosphate buffered saline (PBS), 10X, pH 7.4 | ThermoFisher Scientific | AM9625 | Dilute to 1X |
| Triton X-100 | Sigma-Aldrich | T9284-1L | |
| Kimwipes | Thomas Scientific | 2904F24 | |
| 20% paraformaldehyde (PFA) | Electron Microscopy Sciences | 15713-S | |
| Normal Goat Serum (NGS), filtered | Lampire Biological Laboratories | 7332500 | Aliquot and freeze upon receipt |
| Chicken anti-GFP | Aves Labs | GFP-1020 | Use at 1:500 dilution |
| Rabbit anti-DsRed | Clontech | 632496 | Use at 1:2000 dilution; can recognize DsRed-based fluorescent proteins (e.g. mCherry, mStrawberry, tdTomato, etc.) |
| Mouse anti-Bruchpilot | Developmental Studies Hybridoma Bank | nc82 | Use at 1:100 dilution; will label active pre-synaptic structures thoughout the fly brain |
| FITC anti-chicken | ThermoFisher Scientific | SA1-7200 | Use at 1:250 dilution |
| Alexa Fluor 568 anti-rabbit | Life Technologies | A11011 | Use at 1:250 dilution |
| Alexa Fluor 647 anti-mouse antibody | Life Technologies | A21235 | Use at 1:250 dilution |
| Slowfade Gold Antifade Reagent | Life Technologies | S36936 | |
| Microscope Slides (25 x 75 x 1.0 mm) | Fisher Scientific | 12-550-143 | |
| Cover Glass (22 x 22 mm) | Globe Scientific | 1404-15 | |
| Dumont Biology Grade Forceps, Style 3 | Ted Pella | 503 | use in non-dominant hand |
| Dumont Biology Grade Forceps, Style 5 | Ted Pella | 505 | use in dominant hand |
| LAS X image analysis software | Leica | ||
| Imaris image analysis software | Bitplane |
Riferimenti
- Prusiner, S. B. Biology and genetics of prions causing neurodegeneration. Annu Rev Genet. 47, 601-623 (2013).
- Haïk, S., Brandel, J. P. Infectious prion diseases in humans: Cannibalism, iatrogenicity and zoonoses. Infect Genet Evol. 26, 303-312 (2014).
- Balch, W. E., Morimoto, R. I., Dillin, A., Kelly, J. W. Adapting Proteostasis for Disease Intervention. Science. 319 (5865), 916-919 (2008).
- Stroo, E., Koopman, M., Nollen, E. A., Mata-Cabana, A. Cellular Regulation of Amyloid Formation in Aging and Disease. Front Neurosci. 11, 64 (2017).
- Brundin, P., Melki, R., Kopito, R. Prion-like transmission of protein aggregates in neurodegenerative diseases. Nat Rev Mol Cell Biol. 11 (4), 301-307 (2010).
- Jucker, M., Walker, L. C. Self-propagation of pathogenic protein aggregates in neurodegenerative diseases. Nature. 501 (7465), 45-51 (2013).
- Stopschinski, B. E., Diamond, M. I. The prion model for progression and diversity of neurodegenerative diseases. Lancet Neurol. 16 (4), 323-332 (2017).
- Walker, L. C., Jucker, M. Neurodegenerative diseases: expanding the prion concept. Annu Rev Neurosci. 38, 87-103 (2015).
- Holmes, B. B., et al. Heparan sulfate proteoglycans mediate internalization and propagation of specific proteopathic seeds. Proc Natl Acad Sci U S A. 110 (33), 3138-3147 (2013).
- Munch, C., O'Brien, J., Bertolotti, A. Prion-like propagation of mutant superoxide dismutase-1 misfolding in neuronal cells. Proc Natl Acad Sci U S A. 108 (9), 3548-3553 (2011).
- Nonaka, T., et al. Prion-like properties of pathological TDP-43 aggregates from diseased brains. Cell Rep. 4 (1), 124-134 (2013).
- Ren, P. H., et al. Cytoplasmic penetration and persistent infection of mammalian cells by polyglutamine aggregates. Nat Cell Biol. 11 (2), 219-225 (2009).
- Trevino, R. S., et al. Fibrillar structure and charge determine the interaction of polyglutamine protein aggregates with the cell surface. J Biol Chem. 287 (35), 29722-29728 (2012).
- Volpicelli-Daley, L. A., et al. Exogenous alpha-synuclein fibrils induce Lewy body pathology leading to synaptic dysfunction and neuron death. Neuron. 72 (1), 57-71 (2011).
- Zeineddine, R., et al. SOD1 protein aggregates stimulate macropinocytosis in neurons to facilitate their propagation. Mol Neurodegener. 10, 57 (2015).
- Ayers, J. I., Fromholt, S. E., O'Neal, V. M., Diamond, J. H., Borchelt, D. R. Prion-like propagation of mutant SOD1 misfolding and motor neuron disease spread along neuroanatomical pathways. Acta Neuropathol. 131 (1), 103-114 (2016).
- Clavaguera, F., et al. Brain homogenates from human tauopathies induce tau inclusions in mouse brain. Proc Natl Acad Sci U S A. 110 (23), 9535-9540 (2013).
- de Calignon, A., et al. Propagation of tau pathology in a model of early Alzheimer's disease. Neuron. 73 (4), 685-697 (2012).
- Eisele, Y. S., et al. Induction of cerebral beta-amyloidosis: intracerebral versus systemic Abeta inoculation. Proc Natl Acad Sci U S A. 106 (31), 12926-12931 (2009).
- Luk, K. C., et al. Pathological alpha-synuclein transmission initiates Parkinson-like neurodegeneration in nontransgenic mice. Science. 338 (6109), 949-953 (2012).
- Meyer-Luehmann, M., et al. Exogenous induction of cerebral beta-amyloidogenesis is governed by agent and host. Science. 313 (5794), 1781-1784 (2006).
- Mougenot, A. L., et al. Prion-like acceleration of a synucleinopathy in a transgenic mouse model. Neurobiol Aging. 33 (9), 2225-2228 (2012).
- Rey, N. L., et al. Widespread transneuronal propagation of alpha-synucleinopathy triggered in olfactory bulb mimics prodromal Parkinson's disease. J Exp Med. 213 (9), 1759-1778 (2016).
- Babcock, D. T., Ganetzky, B. Transcellular spreading of huntingtin aggregates in the Drosophila brain. Proc Natl Acad Sci U S A. 112 (39), 5427-5433 (2015).
- Kim, D. K., et al. Anti-aging treatments slow propagation of synucleinopathy by restoring lysosomal function. Autophagy. 12 (10), 1849-1863 (2016).
- Liu, L., et al. Trans-synaptic spread of tau pathology in vivo. PLoS One. 7 (2), 31302 (2012).
- Pearce, M. M., Spartz, E. J., Hong, W., Luo, L., Kopito, R. R. Prion-like transmission of neuronal huntingtin aggregates to phagocytic glia in the Drosophila brain. Nat Commun. 6, 6768 (2015).
- Pearce, M. M. Prion-like transmission of pathogenic protein aggregates in genetic models of neurodegenerative disease. Curr Opin Genet Dev. 44, 149-155 (2017).
- Pecho-Vrieseling, E., et al. Transneuronal propagation of mutant huntingtin contributes to non-cell autonomous pathology in neurons. Nat Neurosci. 17 (8), 1064-1072 (2014).
- Wu, J. W., et al. Neuronal activity enhances tau propagation and tau pathology in vivo. Nat Neurosci. 19 (8), 1085-1092 (2016).
- Jackson, G. R., et al. Polyglutamine-expanded human huntingtin transgenes induce degeneration of Drosophila photoreceptor neurons. Neuron. 21 (3), 633-642 (1998).
- Warrick, J. M., et al. Expanded polyglutamine protein forms nuclear inclusions and causes neural degeneration in Drosophila. Cell. 93 (6), 939-949 (1998).
- McGurk, L., Berson, A., Bonini, N. M. Drosophila as an In Vivo Model for Human Neurodegenerative Disease. Genetics. 201 (2), 377-402 (2015).
- The Huntington's Disease Collaborative Research Group. A novel gene containing a trinucleotide repeat that is expanded and unstable on Huntington's disease chromosomes. Cell. 72 (6), 971-983 (1993).
- Bates, G. P., et al. Huntington disease. Nat Rev Dis Primers. 1, 15005 (2015).
- Scherzinger, E., et al. Self-assembly of polyglutamine-containing huntingtin fragments into amyloid-like fibrils: implications for Huntington's disease pathology. Proc Natl Acad Sci U S A. 96 (8), 4604-4609 (1999).
- Chen, S., Berthelier, V., Yang, W., Wetzel, R. Polyglutamine aggregation behavior in vitro supports a recruitment mechanism of cytotoxicity. J Mol Biol. 311 (1), 173-182 (2001).
- Brand, A. H., Perrimon, N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes. Development. 118 (2), 401-415 (1993).
- Potter, C. J., Tasic, B., Russler, E. V., Liang, L., Luo, L. The Q system: a repressible binary system for transgene expression, lineage tracing, and mosaic analysis. Cell. 141 (3), 536-548 (2010).
- Riabinina, O., Potter, C. J. The Q-System: A Versatile Expression System for Drosophila. Methods Mol Biol. 1478, 53-78 (2016).
- Costanzo, M., et al. Transfer of polyglutamine aggregates in neuronal cells occurs in tunneling nanotubes. J Cell Sci. 126 (16), 3678-3685 (2013).
- Freeman, M. R., Delrow, J., Kim, J., Johnson, E., Doe, C. Q. Unwrapping glial biology: Gcm target genes regulating glial development, diversification, and function. Neuron. 38 (4), 567-580 (2003).
- MacDonald, J. M., et al. The Drosophila cell corpse engulfment receptor Draper mediates glial clearance of severed axons. Neuron. 50 (6), 869-881 (2006).
- Wu, J. S., Luo, L. A protocol for dissecting Drosophila melanogaster brains for live imaging or immunostaining. Nat Protoc. 1 (4), 2110-2115 (2006).
- Tito, A. J., Cheema, S., Jiang, M., Zhang, S. A Simple One-step Dissection Protocol for Whole-mount Preparation of Adult Drosophila Brains. J Vis Exp. (118), (2016).
- Roszik, J., Szöllosi, J., Vereb, G. AccPbFRET: an ImageJ plugin for semi-automatic, fully corrected analysis of acceptor photobleaching FRET images. BMC Bioinformatics. 9, 346 (2008).
- Spires-Jones, T. L., Attems, J., Thal, D. R. Interactions of pathological proteins in neurodegenerative diseases. Acta Neuropathol. 134 (2), 187-205 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon