Method Article
Pluripotenti indotte generazione di cellule staminali da cellule del sangue Utilizzando Sendai Virus e centrifugazione
In questo articolo
Riepilogo
We propose a protocol for reprogramming peripheral blood mononuclear cells (PBMCs) into induced pluripotent stem cells (iPSCs). By plating the transduced blood cells onto matrix-coated plates with centrifugation, iPSCs are successfully induced from floating cells. This technique suggests a simple and effective reprogramming protocol for cells such as PBMCs and CBMCs.
Abstract
Il recente sviluppo di cellule staminali pluripotenti indotte umane (hiPSCs) ha dimostrato che le cellule somatiche mature possono tornare a una, stato pluripotente indifferenziata. Ora, riprogrammazione avviene con vari tipi di cellule somatiche adulte: cheratinociti, cellule urina, fibroblasti, ecc I primi esperimenti erano solitamente fatto con fibroblasti dermici. Tuttavia, questo richiede una procedura chirurgica invasiva per ottenere fibroblasti da pazienti. Pertanto, cellule in sospensione, come le cellule del sangue e delle urine, sono stati considerati ideali per la riprogrammazione causa della convenienza di ottenere le cellule primarie. Qui, riportiamo un protocollo efficace per la generazione di IPSC dalle cellule mononucleate del sangue periferico (PBMC). Con placcatura le PBMC trasdotte in serie ad un nuovo, piastra con centrifugazione a matrice rivestita, questo protocollo può fornire facilmente colonie IPSC. Questo metodo è applicabile anche per le cellule mononucleate del sangue del cordone ombelicale (CBMCs). Questo studio presenta un prot semplice ed efficienteOcol per la riprogrammazione di PBMC e CBMCs.
Introduzione
Le cellule staminali sono stati uno dei materiali più interessanti nella terapia clinica per gli ultimi decenni 1. Le proprietà interessanti di cellule staminali sono pluripotenza e la capacità di auto-rinnovarsi. Nel 1981, le prime cellule staminali embrionali (ESC) sono stati isolati dal embrione di topo 2. Tuttavia, quando la tecnica è stata applicata agli embrioni umani, ha affrontato diverse questioni etiche.
Nel 2006, quando il dottor Yamanaka e il suo team riprogrammate la prima cella pluripotenti da cellule somatiche del mouse, il campo delle cellule staminali ha riguadagnato la sua possibilità e l'interesse si è riaccesa 3. Offrendo diversi fattori definite, cellule staminali pluripotenti sono state con successo "indotte" dalle cellule somatiche adulte, e furono così chiamati "cellule staminali pluripotenti indotte (iPSCs)." Nel 2007, questa tecnica è stata applicata a cellule umane 4, cedendo le cellule con le caratteristiche esatte della CES, ma nessuno del dibattito etico. Teoricamente, iPSCs può essere generato da qualsiasi tipo cellulare ottenuta da qualsiasi individuo o paziente. iPSCs paziente-specifici sono in aumento come un potenziale strumento in grado di simulare i fenotipi di malattia e le condizioni epigenetici di ogni singolo paziente. Usando la modifica del gene o altri metodi che possono invertire la condizione patogena, iPSCs paziente-specifici possono essere utilizzati anche in medicina personalizzata 5. Inoltre, iPSCs sono meno associati con rigetto immunitario perché hanno la stessa identità immunitario come il donatore, rendendo l'auto-trapianto più fattibile 6. Pertanto, iPSCs sono diventate la piattaforma più promettente nella modellazione della malattia, lo screening di stupefacenti, e le terapie rigenerative. Alla luce di questi vantaggi, protocolli migliorati che possono dare più puro e rendimenti più elevati nel minor tempo possibile dalla sorgente più piccola cellula sono costantemente in fase di sviluppo. Una considerazione importante di trovare il protocollo più efficiente per applicazioni future è il tipo di cellula primaria. La maggior parte della prima generazione proto iPSCCols sono ottimizzati per cellule aderenti dal momento che le linee originali IPSC sono state indotte da fibroblasti cutanei 4. Tuttavia, l'isolamento e la preparazione di queste cellule sono lavoro intensivo. Inoltre, l'isolamento di fibroblasti cutanei comprende procedure chirurgiche invasive che possono diventare una grave lacuna per la più ampia applicazione.
Pertanto, per l'ulteriore uso di iPSCs, è necessaria una fonte cella con acquisizione conveniente. Il sangue è considerato come fonte di cellule ideale in quanto ottenuto attraverso una procedura piuttosto minimamente invasiva 7-9. In questo studio, abbiamo sviluppato una semplice modifica al protocollo generare hiPSCs da cellule mononucleate del sangue periferico (PBMC). Senza il difficile processo di espansione di un tipo di cellula specifica, come le cellule CD34 +, le cellule del sangue intero o PBMC sono stati in serie placcato su piastre rivestite di matrice per centrifugazione dopo trasduzione con il virus di Sendai contenente fattori Yamanaka. Questo metodo riduce il tempo richiesto per lal'attaccamento delle cellule galleggianti trasdotte e diminuito la perdita di cellule riprogrammate che non erano in grado di collegare in proprio.
Protocollo
Etica Dichiarazione: Questo protocollo di studio è stato approvato dal Comitato Etico dell'Università Cattolica di Corea (KC12TISI0861).
1. Isolamento dei monociti cellule dal sangue
- L'isolamento di cellule monociti (Giorno -5)
- Ottenere almeno 10 ml di sangue fresco da un prelievo di sangue in un tubo preparato cellulare (CPT).
- Trasferire il sangue in una nuova provetta conica da 50 ml e diluire con soluzione salina sterile tamponata con fosfato (PBS) in un rapporto 1: 4.
NOTA: un più alto rapporto di diluizione può essere utilizzato per una maggiore purezza. - Aggiungere 10 ml di media gradiente di densità in una nuova provetta conica da 50 ml e strato attentamente il sangue diluito in cima media gradiente di densità. Centrifugare a 750 xg per 30 min a temperatura ambiente (RT) senza freno centrifugazione.
- Trasferire accuratamente lo strato di buffy ad un nuovo tubo conico da 50 ml, aggiungere 30 ml di PBS al tubo, e lavare le cellule.
- Centrifugare le cellule a 515 xg per 5 minuti a temperatura ambiente.
- Eliminare il PBS e risospendere le cellule in 0,5 ml di mezzi delle cellule del sangue.
- Contare le cellule e piastra 1 x 10 6 cellule per pozzetto di una piastra da 24 pozzetti. Aggiungere PBS ai pozzetti circostanti per evitare l'evaporazione.
- Stabilizzare le cellule per 5 giorni a 37 ° C in 5% CO 2 prima trasduzione. Aggiungere un ulteriore 0,5 ml di mezzi delle cellule del sangue fresco nelle giornate 3-4 senza disturbare le cellule.
2. trasduzione da Sendai virus
- Trasduzione (giorno 0)
- Raccogliere e trasferire le cellule del sangue ad un tubo conico da 15 ml e contarli con un emocitometro.
- Preparare 3 x 10 5 cellule per trasduzione e centrifugare le cellule a 515 xg per 5 minuti a temperatura ambiente.
- Eliminare il surnatante tramite aspirazione e risospendere le cellule in 0,5 ml di mezzi di cellule del sangue.
- Trasferire le cellule in un pozzetto di una piastra non rivestiti 24 pozzetti.
- Scongelare la miscela di virus di Sendai in ghiaccio e aggiungerlo al SUcellule trascorsa. Aggiungere Sendai virus alle cellule in base alle raccomandazioni del produttore.
- Sigillare la piastra con un film di sigillatura e centrifugare a 1150 xg per 30 min a 30 ° C.
- Dopo centrifugazione, incubare le cellule a 37 ° C in 5% CO 2 overnight (O / N).
- trasferimento di cellule di matrice alimentatore (1 ° giorno)
- Il giorno successivo, il cappotto di un 24-pozzetti con vitronectina. Diluire la soluzione vitronectin in PBS per una concentrazione finale 5 mg / ml. Aggiungere 1 ml di vitronectina in un pozzetto di una piastra da 24 pozzetti ed incubare a temperatura ambiente per almeno 1 ora. Rimuovere la soluzione di rivestimento prima dell'uso. piastre rivestite possono essere memorizzati in RT per 3 giorni.
- Trasferire tutti i supporti contenenti le cellule e virus al rivestito bene.
- Raccogliere le cellule rimanenti con l'aggiunta di 0,5 ml di mezzi delle cellule del sangue fresco e aggiungerlo alla cella contenente bene.
- Centrifugare la piastra a 1.150 xg per 10 min a 35 ° C.
- dopo centorifugation, rimuovere il surnatante, aggiungere 1 ml di IPSC dei media, e mantenere le cellule a 37 ° C in 5% di CO 2 O / N.
- In secondo luogo il trasferimento di cellule (2 ° giorno)
- Coat pozzetti di una nuova 24-pozzetti con 5 mg / ml vitronectina, come descritto al punto 2.2.1. Utilizzare uno pozzetto della piastra per ogni trasduzione.
- Trasferire la sospensione cellulare dalla prima piastra alla piastra vitronectina appena rivestito.
NOTA: se non necessario, cellule in sospensione possono essere scartati. La procedura di cui al punto 2.3 può essere ripetuta 2-3 volte con le celle di sospensione. Se si ripeteranno i passi, raccogliere le cellule. - Nel frattempo, aggiungere 1 ml di COPSI media per pozzo della prima piastra, per la manutenzione e incubare a 37 ° C in 5% CO 2 O / N.
- Mantenere le cellule attaccate a 37 ° C e 5% CO 2 ed eseguire un cambiamento supporto quotidiano con mezzi COPSI fresco. Le colonie appariranno nei giorni 14-21 dopo trasduzione.
- Centrifuge la piastra appena rivestito contenente le celle di sospensione a 1.150 xg e 35 ° C per 10 min.
- Dopo centrifugazione, incubare le cellule a 37 ° C in 5% CO 2 O / N.
- Il giorno successivo, rimuovere il surnatante e sostituirlo con i media IPSC freschi.
NOTA: La procedura di cui al punto 2.3 può essere ripetuta 2-3 volte con le cellule di sospensione. Se si ripeteranno i passi, raccogliere le cellule del surnatante e ripetere il punto 2.3. - Mantenere le cellule attaccate con i cambiamenti dei media al giorno fino a raggiungere l'80% di confluenza.
3. Manutenzione delle cellule riprogrammato
- manutenzione anticipata dopo la cultura 24-pozzetti
- 7-10 giorni dopo la trasduzione, una volta che le cellule sono confluenti, preparare una, piatto vitronectina in acciaio 60 mm. Diluire soluzione vitronectina in PBS per una concentrazione finale 5 mg / ml. Aggiungere 1 ml di vitronectina al piatto ed incubare a temperatura ambiente per almeno 1 ora.
- lava ilcellule con PBS e aggiungere 1 ml di PBS / 1 mM EDTA per staccare le cellule.
- Incubare a 37 ° C e 5% CO 2 per 2 min.
- Raccogliere le cellule e centrifugare a 250 xg ed RT per 2 min. Rimuovere il surnatante e risospendere le cellule in 3 ml di mezzi freschi IPSC.
- Piastra tutte le cellule risospese sulla nuova rivestito piatto di 60 mm.
- Aggiungere 10 mM chinasi RHO alle cellule e mantenerli a 37 ° C in 5% CO 2 fino a quando le cellule sono 80% confluenti.
- Spalato per l'aspetto delle colonie (Subclonaggio Preparazione)
- Preparare una, piatto vitronectina rivestite da 100 mm, come descritto al punto 3.1.2.
- Lavare le cellule con PBS e aggiungere 1 ml di PBS / 1 mM EDTA per staccare le cellule.
- Incubare a 37 ° C e 5% CO 2 per 2 min.
- Raccogliere le cellule e centrifugare a 250 xg ed RT per 2 min.
- Contare le cellule utilizzando un emocitometro e preparare 1 x 10 4 cellule per piatto.
- Centrifugare le cellule a 250 xg ed RT per 2 min.
- Risospendere 1 x 10 4 cellule in 6 ml di IPSC mezzi, li piatto sulla rivestito piatto da 100 mm, e aggiungere 10 inibitore della chinasi mM RHO ai media.
- Incubare le cellule a 37 ° C in 5% CO 2 per una settimana finché appaiono grandi colonie.
NOTA: Mantenere e ampliare le colonie per subcloning. Subcloning di solito è fatto in meno di 5 passaggi.
- Colony raccolta utilizzando iPSC colonia distacco soluzione
- Una settimana prima colonia di raccolta, di semi di 1 x 10 4 cellule in un piatto vitronectina rivestite da 100 mm, come indicato al punto 3.2.7.
- Preparare un piatto vitronectina rivestito 60 mm aggiungendo 2 ml di soluzione di vitronectina e incubando a temperatura ambiente per almeno 1 ora.
- Osservando al microscopio (40x o ingrandimento 100X), marcare colonie con chiari confini con un pennarello. Rimuovere la soluzione vitronectin dalla nuova piastra realizzato in fase 3.3.2 e aggiungere 6 mldi IPSC multimediale integrato con 10 mM RHO chinasi.
- Rimuovere il terreno di coltura dalle cellule e lavare con 3 ml di PBS.
- Aggiungere 1 ml di soluzione di distacco iPSC colonia e incubare per 30 s a temperatura ambiente.
- Rimuovere la soluzione dalla piastra ed incubare a temperatura ambiente per altri 30 sec.
- Usando una pipetta P200, disegnare 200 ml di mezzi dalla piastra e staccare le colonie mirati pipettando. Trasferire le colonie sparse per il nuovo piatto da 100 mm.
- Incubare e mantenere le cellule a 37 ° C in 5% CO 2.
NOTA: Dopo aver ottenuto un puro colonie IPSC, le cellule sono state mantenute fino a raggiungere il passaggio 10. La caratterizzazione è stata fatta dopo almeno 10 passaggi. Diluizioni Anticorpo e informazioni di primer sono riportati nelle tabelle 1 e 2.
Risultati
Questo protocollo presenta un metodo semplice per riprogrammare PBMC isolate da sangue. Utilizzando la combinazione di rivestimento di serie e centrifugazione, iPSCs sono stati generati con successo. Con questo metodo, iPSCs potrebbero essere generati con una piccola quantità di cellule di sangue intero senza isolare o espandere un tipo di cellula specifico. Abbiamo generato con successo iPSCs da solo 1x10 4 celle in una piccola piastra di coltura cellulare.
Prima di riprogrammazione, le cellule del sangue sono stati isolati utilizzando supporti gradiente di densità. Le cellule del sangue sono state trasdotte 5 giorni dopo l'isolamento. La figura 1A illustra lo schema del metodo riprogrammazione delle cellule in sospensione. Dopo l'isolamento, PBMC sono state trasdotte con il virus di Sendai contenente fattori Yamanaka in un non-rivestito bene. Il giorno dopo, le cellule di sospensione sono state raccolte e placcato su un piatto vitronectina rivestita mediante centrifugazione, mentre le cellule attdoleva per il bene non rivestito sono state scartate. Le cellule attaccate che sono apparsi sulla piastra vitronectin rivestite sono state mantenute e ampliate ulteriormente. le cellule attaccate sono state mantenute con i cambiamenti dei media giornaliera utilizzando iPSC media. Il processo di placcatura con centrifugazione è stata ripetuta per le cellule in sospensione rimanenti.
iPSCs riprogrammati potrebbero essere distinti dai loro distinti morfologia diversi giorni dopo la trasduzione. Tuttavia, durante la riprogrammazione, era difficile per espandere le cellule riprogrammate in una forma pura. Alla fase iniziale di riprogrammazione, i iPSCs sono stati mescolati con le cellule attaccate non riprogrammate differenziati (Figura 2A). La freccia nella Figura 2A mostra i tipi cellulari COPSI-come nell'immagine. Prima di espansione, le iPSCs e di altre cellule erano difficili da discriminare. Pertanto, una colonia pura fu ottenuta soltanto dal processo di prelievo colonia. Con semina le cellule in una bassa densità, colonie che erano large sufficiente per scegliere Si ottennero, come mostrato nella Figura 2B. Dopo aver staccato la colonia, i grumi di cellule sono state dissociate in singole cellule e colta (Figura 2C). Colonie Pure IPSC sono state osservate in seguito (Figura 2D). Dopo i iPSCs puri sono stati espansi, la qualità delle cellule è stato testato. Le cellule sono state colorate con fosfatasi alcalina per confermare stato indifferenziato della iPSCs (Figura 3A). Le colonie derivate da iPSCs isolati tutti hanno mostrato una colorazione positiva. Marker pluripotenti sono state confermate da immunofluorescenza, mostrato nella Figura 3B. Le cellule riprogrammate altamente espresso marcatori pluripotenti, come SSEA4, OCT4, SOX2, TRA-1-81, Klf4, e, soprattutto, TRA-1-60. L'espressione dei marcatori pluripotenti stata confermata mediante RT-PCR, mostrato nella Figura 3C. sono stati rilevati OCT4 e SOX2, come mostrato nei dati di fluorescenza. marcatori aggiuntivi come Nanog, DPPA5, TDGF1 erano positively espresso pure.
Per ulteriori analisi, una analisi del cariotipo è stato fatto. Come mostrato nella figura 4A, le iPSCs generati mostrato un modello normale cromosomico. Per confermare vero pluripotenza, le cellule sono state differenziate in ectoderma, mesoderma, endoderma e. I iPSCs generati differenziati con successo in tutti e tre i foglietti embrionali, si vede in figura 4B. Poiché virus Sendai è noto per la sua struttura di integrazione, la rimozione di DNA virale è mostrato in Figura 4C. Uno su tre cellule tendevano ad avere DNA virale rimangono, anche dopo diversi passaggi. Tuttavia, il gene integrato è rimovibile subcloning processi.
In conclusione, abbiamo dimostrato un protocollo di generare iPSCs da PBMC galleggianti. Il protocollo mostrato elevata efficienza e necessaria solo una piccola quantità di sangue. I iPSCs generati avevano elevato pluripotenza ed evitati VI integrazione ral.
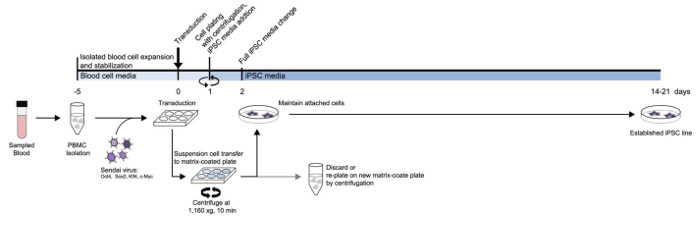
Figura 1: Schema del protocollo iPSC generazione da PBMC. Uno schema dettagliato del protocollo progettato in base al tipo medio e timeline. Clicca qui per vedere una versione più grande di questa figura.
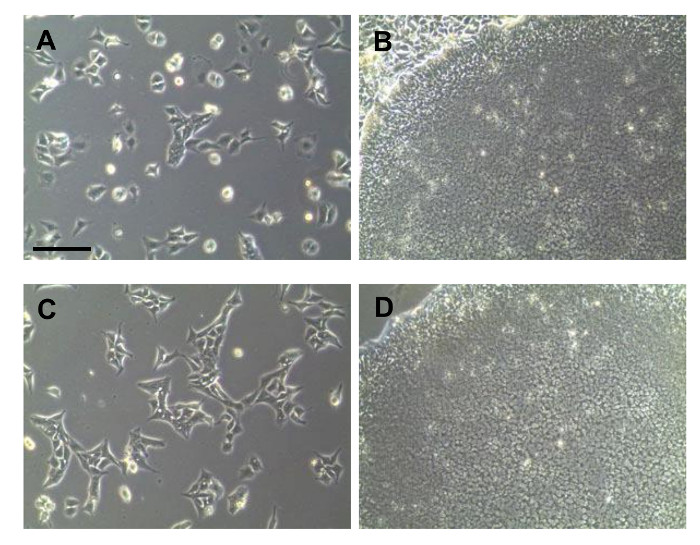
Figura 2: immagine in campo chiaro di iPSCs generati da PBMC. (A) la morfologia precoce di cellule riprogrammate. (B) L'immagine di una colonia prima di scegliere. (C) Le cellule dopo il processo di purificazione colonia prelievo. (D) Morfologia di una colonia pura. Tutte le barre di scala indicano 200 micron. /ecsource.jove.com/files/ftp_upload/54650/54650fig2large.jpg "target =" _ blank "> Clicca qui per vedere una versione più grande di questa figura.
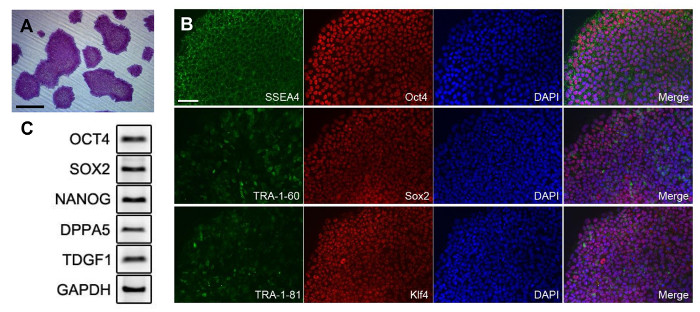
Figura 3: pluripotenza di iPSCs generata da PBMC. (A) Colonie colorate con fosfatasi alcalina. Immagine (B) fluorescenza dei iPSCs generati. Il rapporto di diluizione anticorpo è mostrato in Tabella 1. Analisi (C) PCR di marcatori pluripotenti. Informazioni Primer è fornita in tabella 2. Tutte le barre di scala indicano 200 micron. Clicca qui per vedere una versione più grande di questa figura.
caricare / 54650 / 54650fig4.jpg "/>
Figura 4: Ulteriori analisi delle iPSCs generati. (A) cariogramma normale del IPSC. Immagine (B) fluorescenza delle cellule dopo tre germe differenziazione layer. (C) PCR analisi di Sendai vettori virali. Le cellule non-trasdotte sono state usate come controllo negativo, e le cellule raccolte un giorno dopo trasduzione sono state usate come controllo positivo. Informazioni Primer è fornita in tabella 2. Tutte le barre di scala indicano 200 micron. Clicca qui per vedere una versione più grande di questa figura.
| Nome anticorpo | Concentrazione |
| SSEA4 | 1: 200 |
| Oct3 / 4 | 1: 100 |
| TRA-1-60 | 1: 200 |
| Sox2 | 1: 100 |
| TRA-1-81 | 1: 100 |
| Klf4 | 1: 250 |
| Alexa Fluor 488 capra anti-topo IgG (H + L) anticorpo | 1: 400 |
| Alexa Fluor 594 capra anti-coniglio IgG (H + L) anticorpo | 1: 400 |
Tabella 1. diluizioni di anticorpi.
| Nome destinazione | Direzione | Sequenza Primer | Dimensione |
| Oct3 / 4 | Inoltrare | ACC CCT GGT GCC GTG AA | 190 |
| Inverso | GGC TGA ATA CCT TCC CAUn ATA | ||
| SOX2 | Inoltrare | CAG CGC ATG GAC AGT TAC | 321 |
| Inverso | GGA GTG GGA GGA AGA GGT | ||
| NANOG | Inoltrare | AAA GGC AAA CAA CCC ACT | 270 |
| Inverso | GCT ATT CTT CGG CCA GTT | ||
| LIN28 | Inoltrare | GTT CGG CTT CCT GTC CAT | 122 |
| Inverso | CTG CCT CAC CCT CCT TCA | ||
| DPPB5 | Inoltrare | CGG CTG CTG AAA GCC ATT TT | 215 |
| Inverso | AGT TTG AGC ATC CCT CGC TC | ||
| TDGF1 | Inoltrare | TCC TTC TAC GGA CGG AAC TG | 140 |
| Inverso | AGA AAT GCC TGA GGA AAG CA | ||
| SeV | Inoltrare | GGA TCA CTA GGT GAT ATC GAG C | 181 |
| Inverso | ACC AGA CAA GAG TTT AAG AGA TAT GTA TC | ||
| KOS | Inoltrare | ATG CAC CGC TAC GAC GTG AGC GC | 528 |
| Inverso | ACC TTG ACA ATC CTG ATG TGG | ||
| Klf4 | Inoltrare | TTC CTG CAT GCC AGA GGA GCC C | 410 |
| Inverso | AAT GTA TCG AAG GTG CTC AA |
Tabella 2. Informazioni Primer.
Discussione
Dal momento che le cellule staminali embrionali (CSE) hanno evidenziato diverse lacune, è stato richiesto l'esigenza di uno strumento alternativo. Pertanto, lo sviluppo delle cellule staminali pluripotenti indotte (iPSCs) da Yamanaka è venuto sotto i riflettori internazionali. E 'stato quasi un decennio da quando Yamanaka ha scoperto che pluripotenza può essere indotta con l'aggiunta di soli quattro geni nelle cellule somatiche adulte. Dal momento che iPSCs sono "indotte" dalle cellule somatiche maturi, possono eludere questioni etiche che un tempo era stata la preoccupazione relativa al CES. A differenza di CES, iPSCs possono essere generati da ogni individuo. Pertanto, essi possono essere utilizzati nella ricerca medica personalizzata e altri studi, come screening di farmaci, modelli di malattia o farmaci rigenerativi.
La prima iPSC umana è stata generata da fibroblasti dermici nel 2007. Vari gruppi riprogrammati con successo questo tipo di cellule, ma una procedura chirurgica invasiva è stato richiesto per ottenere questa cellula primaria. Pertanto, non era l'ideafonte l per la generazione di varie iPSCs malattia o la progettazione di medicina rigenerativa. sono stati necessari Un'alternativa e tipi di cellule facilmente ottenute, come cheratinociti, monociti urina e le cellule del sangue. Tuttavia, queste cellule sono difficili da espandersi, e alcuni di loro hanno una maggiore probabilità di contaminazione 10.
In questo studio, abbiamo presentato un protocollo in grado di generare con successo iPSCs dalle cellule di sangue intero, come PBMC. Senza alcun processo di espansione di uno specifico tipo di cellula, questo protocollo suggerisce un metodo relativamente semplice per generare iPSCs, anche da una piccola quantità di globuli primarie. Una volta che le PBMC sono stati isolati dal sangue, le cellule sono state stabilizzate in mezzi globuli per 5 giorni. Il passo di stabilizzazione prima di trasduzione è stato fondamentale in questo protocollo. L'efficienza riprogrammazione era meglio dopo incubazione delle cellule nei mezzi delle cellule del sangue. Questo supporto è stato utilizzato per l'espansione di cellule CD34 +. Tuttavia, quando le cellule di sangue intero sono state mantenute in questo midia per 5 giorni, il numero di cellule era quasi identico al primo conteggio. Utilizzando cellule di sangue intero, è stato difficile per confermare se fossero state ampliate le cellule CD34 +. Tuttavia, la fase di stabilizzazione stesso sembrava essere importante per la riprogrammazione successo.
Dopo trasduzione, siamo in serie placcato le cellule su un piatto vitronectina rivestita per centrifugazione. Originariamente, le cellule riprogrammate andati a fondo del pozzo dopo trasduzione. Aumentando le cellule con forza meccanica o centrifugazione, le cellule trasdotte erano in grado di attaccare e proliferare. Tuttavia, poiché il protocollo utilizzato cellule di sangue intero, metà dei tentativi di prove con diversi campioni comportato vari tipi di morfologie quando espanso. Pertanto, l'isolamento di colonie IPSC puri da colonia raccolta è stato fondamentale in questo protocollo. Le cellule espanse dalla colonia isolato mostrato tutte le caratteristiche di un puro iPSCs.
Nonostante altri benefici, iPSCs hannodiversi ostacoli da superare. Poiché diversi fattori Yamanaka sono noti oncogeni, si teme che iPSCs può svilupparsi in tumori in vivo. Pertanto, è importante che le cellule riprogrammate hanno alcuna espressione genica esogeno rimanente per integrazione. Nei primi anni dopo lo sviluppo di iPSCs, virus, come i retrovirus e lentivirus, sono stati utilizzati per la riprogrammazione. Il tasso di riprogrammazione era successo alto, ma questi virus richiede l'integrazione casuale nel genoma della cellula. Per questa ragione, sono stati sviluppati diversi altri metodi. I virus come virus Sendai sono noti per trasdurre le cellule senza integrazione, e altri metodi che utilizzano piccole molecole e vettori episomiale sono stati sviluppati pure. In questo studio, abbiamo utilizzato il virus Sendai per la riprogrammazione. Il tasso di integrazione è stata relativamente bassa rispetto a lentivirus. Abbiamo anche confermato che ci sono stati alcuni componenti virali rimanenti nelle iPSCs generati. Uno su tre prove ha portato integratione, anche dopo 15-20 passaggi. Tuttavia, questa integrazione è rimovibile subcloning colonie derivate da una singola cellula. Nella nostra esperienza, la densità di ottenere una colonia sana derivata da una singola cellula era 1x10 4 cellule per piatto 100 mm. Le colonie che hanno portato in una forma perfettamente circolare sono stati raccolti e ampliati per la PCR riconferma dei componenti virali rimanenti. Le colonie derivate da cellule non integrati sono stati mantenuti per un ulteriore uso. Una volta eliminate, le cellule mantenute allo stato non integrato.
In questo studio, si consiglia un protocollo per la riprogrammazione delle cellule del sangue. Abbiamo aggiunto diversi passaggi, come placcatura seriale e centrifugazione, per aumentare il tasso di riprogrammazione delle cellule galleggianti. Questo metodo è stato fatto con PBMC e cellule mononucleate del sangue del cordone (CBMCs). Questo protocollo è stato utilizzato per generare iPSCs omozigoti in strutture GMP in Corea del Sud. Il nostro gruppo si attende l'accelerazione del processo di riprogrammazione iPSC utilizzandoil nostro protocollo.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
This work was supported by a grant from the Basic Science Research Program through the National Research Foundation of Korea (NRF), funded by the Ministry of Science, ICT, and Future Planning (2013R1A1A1076125).
Materiali
| Name | Company | Catalog Number | Comments |
| Plasticware | |||
| 100 mm Dish | TPP | 93100 | |
| 6-well Plate | TPP | 92006 | |
| 50 ml Cornical Tube | SPL | 50050 | |
| 15 ml Cornical Tube | SPL | 50015 | |
| 10 ml Disposable Pipette | Falcon | 7551 | |
| 5 ml Disposable Pipette | Falcon | 7543 | |
| 12-well Plate | TPP | 92012 | |
| 24-well Plate | TPP | 92024 | |
| PBMC Isolation Materials | |||
| DPBS | Life Technologies | 14190-144 | |
| Ficoll | GE Healthcare | 17-1440-03 | |
| StemSpan | STEMCELL Technologies | 9805 | Blood cell media |
| CC110 | STEMCELL Technologies | 8697 | Blood cell media supplement (100x) |
| iPSC Generation and Culture Materials | |||
| CytoTune-iPSC Sendai Reprogramming Kit | Life Technologies | A16518 | |
| TeSR-E8 Media | STEMCELL Technologies | 5940 | iPSC media |
| Vitronectin | Life Technologies | A14700 | |
| ROCK Inhibitor | Sigma Aldrich | Y0503 | |
| TrypLE express (TrypLE) | Life Technologies | 12604-039 | |
| ReleSR | STEMCELL Technologies | 12604-039 | Colony detaching solution |
| Quality Control Materials | |||
| 18 mm Cover Glass | Superior | HSU-0111580 | |
| 4% Paraformaldyhyde | Tech & Innovation | BPP-9004 | |
| Triton X-100 | BIOSESANG | 9002-93-1 | |
| Bovine Serum Albumin | Vector Lab | SP-5050 | |
| Anti-SSEA4 Antibody | Millipore | MAB4304 | |
| Anti-Oct4 Antibody | Santa Cruz | SC9081 | |
| Anti-TRA-1-60 Antibody | Millipore | MAB4360 | |
| Anti-Sox2 Antibody | Biolegend | 630801 | |
| Anti-TRA-1-81 Antibody | Millipore | MAB4381 | |
| Anti-Klf4 Antibody | Abcam | ab151733 | |
| Alexa Fluor 488 goat anti-mouse IgG (H+L) antibody | Molecular Probe | A11029 | |
| Alexa Fluor 594 goat anti-rabbit IgG (H+L) antibody | Molecular Probe | A11037 | |
| DAPI | Molecular Probe | D1306 | |
| Prolong gold antifade reagent | Invitrogen | P36934 | |
| Slide Glass, Coated | Hyun Il Lab-Mate | HMA-S9914 | |
| Trizol | Invitrogen | 15596-018 | |
| Chloroform | Sigma Aldrich | 366919 | |
| Isoprypylalcohol | Millipore | 109634 | |
| Ethanol | Duksan | 64-17-5 | |
| RevertAid First Strand cDNA Synthesis kit | Thermo Scientfic | K1622 | |
| i-Taq DNA Polymerase | iNtRON BIOTECH | 25021 | |
| UltraPure 10X TBE Buffer | Life Technologies | 15581-044 | |
| loading star | Dyne Bio | A750 | |
| Agarose | Sigma-Aldrich | 9012-36-6 | |
| 1kb (+) DNA ladder marker | Enzynomics | DM003 | |
| Alkaline Phosphatase | Millipore | SCR004 | |
| Tris base | Fisher Scientific | BP152-1 | Rinse Buffer |
| Sodium Chloride | Duchefa Biochemie | S0520.1000 | Rinse Buffer |
| Tween-20 | BIOSESANG | T1027 | Rinse Buffer |
| Hydrochloric Acid | Duksan | 1129 | Rinse Buffer |
Riferimenti
- Serra, M., Brito, C., Correia, C., Alves, P. M. Process engineering of human pluripotent stem cells for clinical application. Trends Biotechnol. 30 (6), 350-359 (2012).
- Martin, G. R. Isolation of a pluripotent cell line from early mouse embryos cultured in medium conditioned by teratocarcinoma stem cells. Proc Natl Acad Sci U S A. 78 (12), 7634-7638 (1981).
- Takahashi, K., Yamanaka, S. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors. Cell. 126 (4), 663-676 (2006).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Chun, Y. S., Byun, K., Lee, B. Induced pluripotent stem cells and personalized medicine: current progress and future perspectives. Anat Cell Biol. 44 (4), 245-255 (2011).
- Seki, T., Fukuda, K. Methods of induced pluripotent stem cells for clinical application. World J Stem Cells. 7 (1), 116-125 (2015).
- Churko, J. M., Burridge, P. W., Wu, J. C. Generation of human iPSCs from human peripheral blood mononuclear cells using non-integrative Sendai virus in chemically defined conditions. Methods Mol Biol. 1036, 81-88 (2013).
- Loh, Y. H., et al. Reprogramming of T cells from human peripheral blood. Cell Stem Cell. 7 (1), 15-19 (2010).
- Ohmine, S., et al. Induced pluripotent stem cells from GMP-grade hematopoietic progenitor cells and mononuclear myeloid cells. Stem Cell Res Ther. 2 (6), (2011).
- Mae, S., et al. Monitoring and robust induction of nephrogenic intermediate mesoderm from human pluripotent stem cells. Nat Commun. 4, 1367 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon