Method Article
Inducida por la generación de células madre pluripotentes a partir de células de la sangre usando el virus Sendai y centrifugación
En este artículo
Resumen
We propose a protocol for reprogramming peripheral blood mononuclear cells (PBMCs) into induced pluripotent stem cells (iPSCs). By plating the transduced blood cells onto matrix-coated plates with centrifugation, iPSCs are successfully induced from floating cells. This technique suggests a simple and effective reprogramming protocol for cells such as PBMCs and CBMCs.
Resumen
El reciente desarrollo de las células madre pluripotentes inducidas humanas (hiPSCs) demostró que las células somáticas maduras pueden volver a un estado indiferenciado, pluripotente. Ahora, la reprogramación se lleva a cabo con varios tipos de células somáticas adultas: queratinocitos, células de orina, fibroblastos, etc. Los primeros experimentos se hace generalmente con fibroblastos dérmicos. Sin embargo, esto requiere un procedimiento quirúrgico invasivo para obtener fibroblastos de los pacientes. Por lo tanto, las células en suspensión, tales como células de sangre y orina, se considera ideal para la reprogramación debido a la comodidad de la obtención de las células primarias. Aquí, se presenta un protocolo eficiente para la generación de IPSC a partir de células mononucleares de sangre periférica (CMSP). Mediante siembra de las PBMCs transducidas en serie a una nueva placa usando centrifugación matriz recubierta, este protocolo puede proporcionar fácilmente colonias IPSC. Este método es aplicable a células mononucleares de sangre de cordón umbilical (CBMC) también. Este estudio presenta una prot simple y eficienteocol para la reprogramación de las PBMC y CBMC.
Introducción
Las células madre han sido uno de los materiales más atractivos de la terapia clínica desde hace varias décadas 1. Las propiedades atractivas de las células madre son la pluripotencia y la capacidad de auto-renovación. En 1981, se aislaron las primeras células madre embrionarias (CES) a partir del embrión de ratón 2. Sin embargo, cuando la técnica se aplicó a los embriones humanos, se enfrentó a varios problemas éticos.
En 2006, cuando el Dr. Yamanaka y su equipo reprogramar la primera célula pluripotente de las células somáticas de ratón, el campo de células madre recuperó su posibilidad y el interés se reavivó 3. Mediante la entrega de varios factores definidos, las células madre pluripotentes fueron correctamente "inducidos" a partir de células somáticas adultas, y por lo tanto se denominan "células madre pluripotentes inducidas (iPS)." En 2007, esta técnica se aplicó a las células humanas 4, las células con las características exactas de los CES rendimiento pero ninguno de debate ético. Teóricamente, las iPSCs puede generarse a partir de cualquier tipo de célula obtenido de cualquier persona o paciente. iPS específicas de pacientes se están levantando como una herramienta potencial que puede simular los fenotipos de la enfermedad y las condiciones epigenéticas de cada paciente individual. El uso de la edición de genes u otros métodos que pueden revertir la condición patógena, iPSCs específicos del paciente también pueden ser utilizados en la medicina personalizada 5. Por otra parte, iPSCs están menos asociadas con el rechazo inmunológico debido a que tienen la misma identidad inmune como donante, haciendo auto-trasplante más factible 6. Por lo tanto, iPSCs se han convertido en la plataforma más prometedora en el modelado de la enfermedad, la detección de drogas y terapias regenerativas. Teniendo en cuenta estos beneficios, la mejora de los protocolos que se dan más puro y mayores rendimientos en el menor tiempo posible de la fuente de células más pequeñas son constantemente en desarrollo. Una consideración importante de encontrar el protocolo más eficiente para la aplicación en el futuro es el principal tipo de células. La mayor parte de los primeros proto generación de IPSCcols están optimizados para las células adherentes ya que las líneas de IPSC originales fueron inducidas a partir de fibroblastos de la piel 4. Sin embargo, el aislamiento y la preparación de estas células son mano de obra intensiva. Además, el aislamiento de los fibroblastos de la piel incluye procedimientos quirúrgicos invasivos que pueden convertirse en un defecto importante para una aplicación más amplia.
Por lo tanto, para el uso adicional de iPSCs, se requiere una fuente de células con la adquisición conveniente. La sangre se considera como una fuente de células ideal, ya que se obtiene a través de un procedimiento bastante mínimamente invasiva 7-9. En este estudio, hemos desarrollado una simple modificación al protocolo de generación de hiPSCs a partir de células mononucleares de sangre periférica (PBMCs). Sin el proceso de expansión difícil de un tipo específico de célula, como las células CD34 +, las células de sangre entera o PBMCs se sembraron en serie en placas de matriz recubierto por centrifugación después de la transducción con el virus de Sendai que contiene factores de Yamanaka. Este método reduce el tiempo requerido para launión de las células transducidas flotantes y la disminución de la pérdida de células reprogramadas que no eran capaces de unirse por su cuenta.
Protocolo
Declaración de Ética: Este protocolo de estudio fue aprobado por la junta de revisión institucional de la Universidad Católica de Corea (KC12TISI0861).
1. Aislamiento de células monocíticas de la sangre
- El aislamiento de las células monocíticas (Día -5)
- Obtener al menos 10 ml de sangre fresca de una extracción de sangre en un tubo de preparación de células (CPT).
- La transferencia de la sangre a un nuevo tubo cónico de 50 ml y diluir con solución salina tamponada con fosfato estéril (PBS) a una proporción de 1: 4.
NOTA: una mayor proporción de dilución se puede utilizar para una mayor pureza. - Añadir 10 ml de medios de gradiente de densidad a un nuevo tubo cónico de 50 ml y la capa cuidadosamente la sangre diluida en la parte superior de los medios de gradiente de densidad. Centrifugar a 750 xg durante 30 min a temperatura ambiente (RT) sin un freno de centrifugación.
- transferir con cuidado la capa leucocitaria a un nuevo tubo cónico de 50 ml, añadir 30 ml de PBS al tubo y lavar las células.
- Centrifugar las células a 515 xg durante 5 min a TA.
- Desechar el PBS y resuspender las células en 0,5 ml de medio de células sanguíneas.
- Contar las células y la placa 1 x 10 6 células por pocillo de una placa de 24 pocillos. Añadir PBS a los pocillos circundantes para evitar la evaporación.
- Estabilizar las células durante 5 días a 37 ° C en 5% de CO 2 antes de la transducción. Añadir un adicional de 0,5 ml de medio de células de sangre fresca en los días 3-4 sin molestar a las células.
2. La transducción por el virus de Sendai
- Transducción (Día 0)
- Recoger y transferir las células de la sangre a un tubo cónico de 15 ml y contarlos utilizando un hemocitómetro.
- Preparar 3 x 10 5 células por transducción y centrifugar las células a 515 xg durante 5 min a TA.
- Eliminar el sobrenadante por aspiración y resuspender las células en 0,5 ml de medio de células sanguíneas.
- Transferir las células a un pocillo de una placa no recubierta 24 pocillos.
- Descongelar la mezcla de virus Sendai en hielo y añadirlo a la Dospended células. Añadir virus Sendai a las células en base a las recomendaciones del fabricante.
- Sellar la placa con una lámina de sellado y se centrifuga a 1.150 xg que durante 30 minutos a 30 ° C.
- Después de la centrifugación, se incuban las células a 37 ° C en 5% de CO 2 durante la noche (O / N).
- la transferencia de células de matriz de alimentación (Día 1)
- Al día siguiente, recubrir una placa de 24 pocillos con vitronectina. Diluir la solución de vitronectina en PBS para una concentración / ml 5 mg final. Añadir 1 ml de vitronectina a un pocillo de una placa de 24 pocillos y se incuba a TA durante al menos 1 hr. Eliminar la solución de recubrimiento antes de su uso. Las placas recubiertas se pueden almacenar en RT durante 3 días.
- Transferir todos los medios que contienen las células y el virus al pocillo recubierto.
- Recoger las células restantes con un adicional de 0,5 ml de medio de células de la sangre fresca y añadirlo al pozo que contiene células.
- Centrifugar la placa a 1.150 xg durante 10 minutos a 35 ° C.
- después cientorifugation, eliminar el sobrenadante, añadir 1 ml de medio de IPSC, y mantener las células a 37 ° C en 5% de CO 2 O / N.
- En segundo lugar la transferencia de células (Día 2)
- Recubrir los pocillos de una nueva placa de 24 pocillos con 5 mg / ml de vitronectina, como se describe en el paso 2.2.1. Utilice un pocillo de la placa para cada transducción.
- Transferir la suspensión celular de la primera placa a la placa de vitronectina recién recubierto.
NOTA: Si no es necesario, las células en suspensión pueden ser descartados. El procedimiento mencionado en el paso 2.3 se puede repetir 2-3 veces con las células en suspensión. Si se repetirán los pasos, recoger las células. - Mientras tanto, añadir 1 ml de medio de IPSC al pozo de la primera placa, para el mantenimiento, e incubar a 37 ° C en 5% de CO 2 O / N.
- Mantener las células unidas a 37 ° C y 5% de CO2 y realizar un cambio de los medios de comunicación diaria con los medios de IPSC fresco. Las colonias aparecerán en los días 14-21 después de la transducción.
- centrifuge la placa recién recubierta que contiene las células en suspensión en 1.150 xg y 35 ° C durante 10 min.
- Después de la centrifugación, se incuban las células a 37 ° C en 5% de CO 2 O / N.
- Al día siguiente, eliminar el sobrenadante y reemplazarlo con medios frescos IPSC.
NOTA: El procedimiento mencionado en el paso 2.3 se puede repetir 2-3 veces con las células en suspensión. Si se repetirán los pasos, recoger las células en el sobrenadante y repetir el paso 2.3. - Mantener las células unidas con cambios de medio día hasta que se alcanza el 80% de confluencia.
3. Mantenimiento de la célula reprogramadas
- mantenimiento preventivo después del cultivo en placa de 24 pocillos
- 7-10 días después de la transducción, una vez que las células son confluentes, preparar una, antena vitronectina-revestido 60-mm. Diluir la solución de vitronectina en PBS para una concentración / ml 5 mg final. Añadir 1 ml de vitronectina al plato y se incuba que a TA durante al menos 1 hr.
- lava ellas células con PBS y se añade 1 ml de PBS / EDTA 1 mM para separar las células.
- Incuban a 37 ° C y 5% de CO2 durante 2 min.
- Recoger las células y centrifugar ellos en 250 xg y TA durante 2 min. Eliminar el sobrenadante y resuspender las células en 3 ml de medio de IPSC frescas.
- Placa de todas las células se volvieron a suspender en el plato recién recubierto 60-mm.
- Añadir RHO mM inhibidor de quinasa 10 a las células y mantenerlas a 37 ° C en 5% de CO 2 hasta que las células son 80% de confluencia.
- Dividir para el aspecto de colonias (Subclonación Preparación)
- Preparar una, antena vitronectina recubierto de 100-mm, tal como se describe en el paso 3.1.2.
- Se lavan las células con PBS y añadir 1 ml de EDTA PBS / 1 mM para separar las células.
- Incuban a 37 ° C y 5% de CO2 durante 2 min.
- Recoger las células y centrifugar ellos en 250 xg y TA durante 2 min.
- Contar las células utilizando un hemocitómetro y preparar 1 x 10 4 células por placa.
- Centrifugar las células a 250 g y la temperatura ambiente durante 2 min.
- Resuspender 1 x 10 4 células en 6 ml de medio de IPSC, la placa de ellas en el revestido placa de 100 mm, y añaden RHO mM inhibidor de quinasa 10 a los medios de comunicación.
- Se incuban las células a 37 ° C en 5% de CO2 durante una semana hasta que aparezcan grandes colonias.
NOTA: Mantener y ampliar las colonias para la subclonación. Subclonación generalmente se realiza en menos de 5 pasajes.
- Colonia recogiendo el uso de IPSC colonia solución de desprendimiento
- Una semana antes de la recolección de la colonia, las semillas de 1 x 10 4 células en un plato de vitronectina recubiertos de 100 mm, como se menciona en el paso 3.2.7.
- Preparar una, antena vitronectina recubiertas de 60 mm mediante la adición de 2 ml de solución de vitronectina e incubando a temperatura ambiente durante al menos 1 hr.
- Al observar a través del microscopio (40X o 100X), marcar colonias con límites claros utilizando un rotulador. Eliminar la solución de vitronectina de la nueva placa hecha en el paso 3.3.2 y añadir 6 mlIPSC de los medios de comunicación complementado con 10 mM RHO quinasa.
- Retire el medio de cultivo de las células y lavarlos con 3 ml de PBS.
- Añadir 1 ml de solución de desprendimiento IPSC colonia y se incuba durante 30 s a temperatura ambiente.
- Retirar la solución de la placa e incubar que a TA durante 30 sec adicional.
- Con una pipeta P200, dibujar 200 l de los medios de comunicación de la placa y separar las colonias dirigidas por pipeteo. La transferencia de las colonias dispersas a la nueva placa de 100 mm.
- Incubar y mantener las células a 37 ° C en 5% de CO 2.
NOTA: Después de obtener una colonia pura de IPSC, las células se mantuvieron hasta que alcanzaron el paso 10. La caracterización se realiza después de al menos 10 pasajes. Diluciones de anticuerpos y la información de cebadores se muestran en las Tablas 1 y 2.
Resultados
Este protocolo presenta un método simple para reprogramar PBMCs aisladas de la sangre. Utilizando la combinación de las planchas de serie y centrifugación, iPSCs se generaron con éxito. Con este método, iPSCs podrían ser generados con una pequeña cantidad de células de sangre entera sin aislar o expansión de un tipo celular específico. Hemos generado con éxito células iPS a partir de sólo 1x10 4 células en una placa de cultivo de células pequeñas.
Antes de la reprogramación, se aislaron células de la sangre utilizando medios de gradiente de densidad. Las células sanguíneas se transdujeron 5 días después del aislamiento. La Figura 1A ilustra el esquema del método de reprogramación de las células en suspensión. Después del aislamiento, PBMCs fueron transducidas con virus Sendai que contiene factores de Yamanaka en un pozo no recubierto. Al día siguiente, se recogieron las células en suspensión y se sembró en una placa de vitronectina recubierta en centrifugación, mientras que las células attdolía al pozo no recubiertas fueron descartados. Las células unidas que aparecieron en la placa de vitronectina recubiertos se mantuvieron y se expandieron más. Se adjuntan las células se mantuvieron con cambios de medio día utilizando medios de IPSC. El proceso de recubrimiento con centrifugación se repitió para las células en suspensión restantes.
iPS reprogramadas podrían ser distinguidos por sus distintas morfología varios días después de la transducción. Sin embargo, durante la reprogramación, era duro para expandir las células reprogramadas en una forma pura. En la etapa temprana de la reprogramación, las iPSCs se mezclaron con células unidas no reprogramadas diferenciadas (Figura 2A). La flecha en la Figura 2A muestra los tipos de células IPSC-como en la imagen. Antes de la expansión, las células iPS y otras células eran difíciles de discriminar. Por lo tanto, una colonia pura solamente se obtuvo mediante el proceso de picking colonia. Por siembra de las células en una densidad baja, las colonias que eran lse obtuvieron arge suficiente para recoger, como se muestra en la Figura 2B. Después de aislar la colonia, los grupos de células se disociaron en células individuales y se cultivaron (Figura 2C). IPSC colonias puras se observaron después (Figura 2D). Después se expandieron las iPSCs puros, se puso a prueba la calidad de las células. Las células se tiñeron con fosfatasa alcalina para confirmar el estado no diferenciado de la iPSCs (Figura 3A). Colonias derivadas de células iPS aisladas todos mostraron tinción positiva. Marcadores pluripotentes se confirmaron mediante tinción de inmunofluorescencia, que se muestra en la Figura 3B. Las células reprogramadas marcadores pluripotentes tales como SSEA4, Oct4, Sox2, TRA-1-81, KLF4, y, lo más importante, TRA-1-60 altamente expresado. La expresión de marcadores pluripotentes fue confirmada por RT-PCR, que se muestra en la Figura 3C. se detectaron Oct4 y Sox2, como se muestra en los datos de fluorescencia. marcadores adicionales tales como NANOG, DPPA5, TDGF1 eran positivEly expresó también.
Para un análisis más detallado, se realizó un análisis de cariotipo. Como se muestra en la Figura 4A, las iPSCs generadas mostraron un patrón cromosómico normal. Para confirmar verdadera pluripotencia, las células se diferenciaron en ectodermo, mesodermo y endodermo. Las iPSCs generados diferencian con éxito en las tres capas germinales, visto en la figura 4B. Dado que el virus Sendai es conocido por su propiedad libre de la integración, la eliminación de ADN viral se muestra en la Figura 4C. Uno de cada tres células tendían a tener ADN viral siguen siendo, incluso después de varios pasajes. Sin embargo, el gen integrado era extraíble por procesos subclonación.
En conclusión, hemos demostrado un protocolo de generación de células iPS a partir de PBMCs flotantes. El protocolo mostró una alta eficiencia y requiere sólo una pequeña cantidad de sangre. Las células iPS generadas tenían alta pluripotencia y evitaron vi la integración ral.
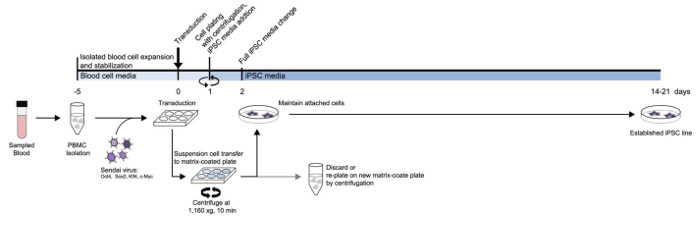
Figura 1: Esquema del protocolo de generación de IPSC de PBMCs. Un diagrama detallado del protocolo diseñado basado en el tipo de medio y línea de tiempo. Haga clic aquí para ver una versión más grande de esta figura.
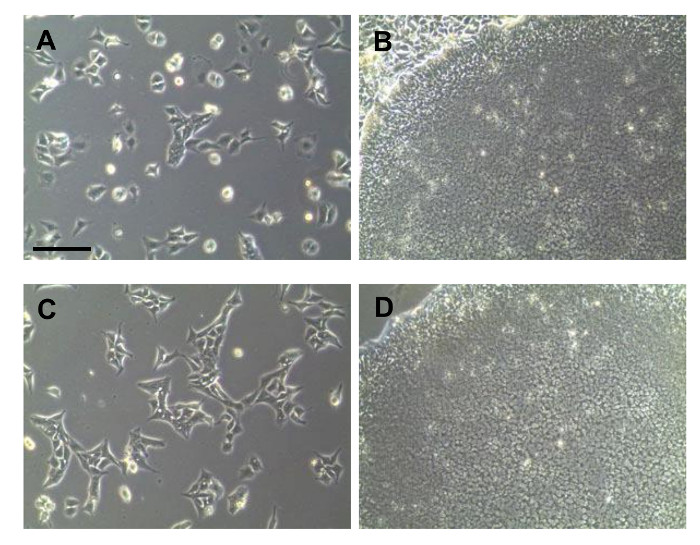
Figura 2: Imagen de campo brillante de iPSCs generadas a partir de PBMCs. (A) la morfología temprana de células reprogramadas. (B) Imagen de una colonia antes de recoger. (C) Las células después del proceso de purificación de colonias picking. (D) Morfología de una colonia pura. Todas las barras de escala métrica que 200 micras. /ecsource.jove.com/files/ftp_upload/54650/54650fig2large.jpg "target =" _ blank "> Haga clic aquí para ver una versión más grande de esta figura.
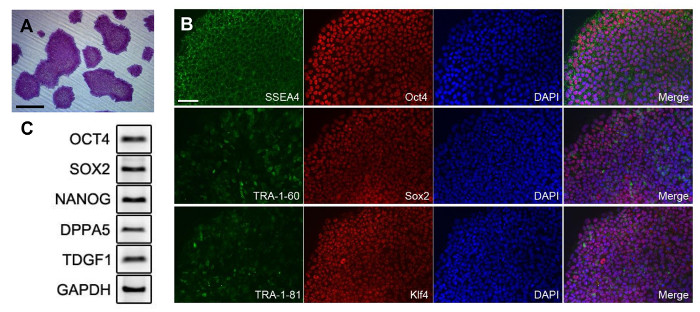
Figura 3: pluripotencia de las células iPS genera a partir de PBMCs. Colonias (A) se tiñeron con fosfatasa alcalina. Imagen (B) de fluorescencia de las iPSCs generados. La relación de dilución de anticuerpos se muestra en la Tabla 1. Análisis (C) PCR de marcadores pluripotentes. Primer información se proporciona en la Tabla 2. Todas las barras de escala métrica que 200 micras. Haga clic aquí para ver una versión más grande de esta figura.
cargar / 54650 / 54650fig4.jpg "/>
Figura 4: Un análisis más detallado de las células iPS generadas. (A) karyogram normal de la IPSC. Imagen (B) de la fluorescencia de las células después de tres germen diferenciación capa. (C) PCR análisis de vectores virales Sendai. Se utilizaron células no transducidas como control negativo, y las células cosechadas un día después de la transducción se utilizaron como control positivo. Primer información se proporciona en la Tabla 2. Todas las barras de escala métrica que 200 micras. Haga clic aquí para ver una versión más grande de esta figura.
| Nombre de anticuerpos | Concentración |
| SSEA4 | 1: 200 |
| Oct3 / 4 | 1: 100 |
| TRA-1-60 | 1: 200 |
| Sox2 | 1: 100 |
| TRA-1-81 | 1: 100 |
| KLF4 | 1: 250 |
| Alexa Fluor 488 de cabra anti-IgG de ratón (H + L) de anticuerpos | 1: 400 |
| Alexa Fluor 594 de IgG de cabra anti-conejo (H + L) de anticuerpos | 1: 400 |
Tabla 1. diluciones de anticuerpo.
| Nombre de destino | Dirección | Secuencia Primer | tamaño |
| OCT3 / 4 | Adelante | ACC CCT GGT GCC GTG AA | 190 |
| Marcha atrás | GGC TGA ATA CCT CCT CAUn ATA | ||
| Sox2 | Adelante | CAG CGC ATG GAC TAC AGT | 321 |
| Marcha atrás | GGA GTG GGA GGA AGA GGT | ||
| NANOG | Adelante | AAA AAA GGC ACT CAA CCC | 270 |
| Marcha atrás | GCT ATT CTT CGG CCA GTT | ||
| Lin28 | Adelante | GTT CGG CTT CCT CAT GTC | 122 |
| Marcha atrás | TCA CTG CCT CAC CCT CCT | ||
| DPPB5 | Adelante | CGG CTG CTG AAA GCC ATT TT | 215 |
| Marcha atrás | AGT TTG AGC ATC CCT CGC TC | ||
| TDGF1 | Adelante | TCC GGA TTC TAC CGG AAC TG | 140 |
| Marcha atrás | AGA AAT GCC TGA GGA AAG CA | ||
| SEV | Adelante | GGA TCA CTA GGT GAT ATC GAG C | 181 |
| Marcha atrás | ACC AGA CAA GAG TTT AAG AGA TAT GTA TC | ||
| KOS | Adelante | GC ATG CAC CGC TAC GAC GTG AGC | 528 |
| Marcha atrás | ACC TTG ACA ATC ATG CTG TGG | ||
| KLF4 | Adelante | TTC CTG AGA CAT CGC GGA GCC C | 410 |
| Marcha atrás | AAT GTA TCG AAG GTG CTC AA |
Tabla 2. Primer información.
Discusión
Dado que las células madre embrionarias (CME) mostraron varias deficiencias, se requiere la necesidad de una herramienta alternativa. Por lo tanto, el desarrollo de las células madre pluripotentes inducidas (iPS) por Yamanaka fue objeto de la atención internacional. Ha sido casi una década desde Yamanaka descubrió que la pluripotencia se puede inducir mediante la adición de sólo cuatro genes en células somáticas adultas. Desde iPSCs son "inducidos" a partir de células somáticas maduras, pueden evadir las cuestiones éticas que una vez había sido la preocupación relativa a los CES. A diferencia de los CES, iPSCs se pueden generar a partir de cada individuo. Por lo tanto, pueden ser utilizados en la investigación médica personalizada y otros estudios, tales como la detección de drogas, el modelado de la enfermedad, o medicamentos regenerativos.
La primera IPSC humano se generó a partir de fibroblastos dérmicos en 2007. Varios grupos reprogramados con éxito este tipo de células, pero se requiere un procedimiento quirúrgico invasivo para obtener esta célula primaria. Por lo tanto, no era la ideal fuente para generar células iPS diversas enfermedades o el diseño de la medicina regenerativa. se necesitaban Una alternativa y tipos de células fácilmente obtenidos, tales como los queratinocitos, monocitos orina y las células sanguíneas. Sin embargo, estas células son difíciles de ampliar, y algunos de ellos tienen una mayor probabilidad de contaminación 10.
En este estudio, presentamos un protocolo que puede generar con éxito células iPS a partir de células de sangre entera como CMSP. Sin ningún tipo de proceso de expansión de un tipo celular específico, este protocolo sugiere un método relativamente simple para generar iPSCs, incluso de una pequeña cantidad de células de la sangre primarios. Una vez que los PBMCs se aislaron de la sangre, se estabilizaron las células en medios de células sanguíneas durante 5 días. La etapa de estabilización antes de la transducción era crítico en este protocolo. La eficiencia de reprogramación era mejor después de la incubación de las células en medios de células sanguíneas. Se utilizó este medio para la expansión de células CD34 +. Sin embargo, cuando las células de sangre entera se mantuvieron en esto media durante 5 días, el número de células era casi idéntica a la primera cuenta. El uso de células de sangre entera, era difícil confirmar si se han ampliado las células CD34 +. Sin embargo, la propia etapa de estabilización parecía ser importante para la reprogramación éxito.
Después de la transducción, se chapada en serie las células sobre una placa revestida de vitronectina por centrifugación. Originalmente, las células reprogramadas hundió hasta el fondo del pozo después de la transducción. Al aumentar las células con la fuerza mecánica o centrifugación, las células transducidas fueron capaces de unir y proliferar. Sin embargo, ya que el protocolo utilizado células de sangre entera, la mitad de los intentos de los ensayos con diferentes muestras resultó en varios tipos de morfologías cuando se expande. Por lo tanto, el aislamiento de colonias IPSC puros mediante la recolección de colonia era crítico en este protocolo. Las células expandidas de la colonia aislada mostraron todas las características de un iPSCs puros.
A pesar de otros beneficios, tienen iPSCsvarios obstáculos que superar. Desde hace varios factores Yamanaka son conocidos oncogenes, existe la preocupación de que iPS pueden convertirse en tumores in vivo. Por lo tanto, es importante que las células reprogramadas no tienen expresión de genes exógenos restante por integración. En los primeros años después del desarrollo de las células iPS, virus, tales como retrovirus y lentivirus, se utilizaron para la reprogramación. La tasa de éxito fue la reprogramación de alto, sin embargo, estos virus requiere la integración aleatoria en el genoma celular. Por esta razón, se han desarrollado varios otros métodos. Los virus como el virus Sendai son conocidos para transducir las células sin la integración, y se han desarrollado otros métodos que utilizan pequeñas moléculas y vectores episomales también. En este estudio, hemos utilizado el virus Sendai para la reprogramación. La tasa de integración fue relativamente baja en comparación con los lentivirus. También confirmó que había algunos componentes virales restantes en las células iPS generadas. Uno de cada tres ensayos resultó en integrationes, incluso después de 15-20 pasajes. Sin embargo, esta integración fue extraíble subclonando las colonias derivadas de una sola célula. En nuestra experiencia, la densidad para obtener una colonia sana derivada de una sola célula era de 1x10 4 células por placa de 100 mm. Las colonias que dieron lugar a una forma circular perfecta se seleccionaron y se expandieron para PCR de re-confirmación de los componentes virales restantes. Las colonias derivadas de células no integradas se mantuvieron para su uso posterior. Una vez borradas, las células mantienen el estado no integrado.
En este estudio, se sugiere un protocolo para la reprogramación de células sanguíneas. Añadimos varios pasos, tales como chapado de serie y centrifugación, para aumentar la tasa de reprogramación de las células flotantes. Este método se realizó con PBMCs y células mononucleares de sangre de cordón (CBMCs). Este protocolo se utiliza para generar células iPS homocigotos en instalaciones GMP en Corea del Sur. Nuestro grupo espera con interés la aceleración del proceso de reprogramación de IPSC usandonuestro protocolo.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
This work was supported by a grant from the Basic Science Research Program through the National Research Foundation of Korea (NRF), funded by the Ministry of Science, ICT, and Future Planning (2013R1A1A1076125).
Materiales
| Name | Company | Catalog Number | Comments |
| Plasticware | |||
| 100 mm Dish | TPP | 93100 | |
| 6-well Plate | TPP | 92006 | |
| 50 ml Cornical Tube | SPL | 50050 | |
| 15 ml Cornical Tube | SPL | 50015 | |
| 10 ml Disposable Pipette | Falcon | 7551 | |
| 5 ml Disposable Pipette | Falcon | 7543 | |
| 12-well Plate | TPP | 92012 | |
| 24-well Plate | TPP | 92024 | |
| PBMC Isolation Materials | |||
| DPBS | Life Technologies | 14190-144 | |
| Ficoll | GE Healthcare | 17-1440-03 | |
| StemSpan | STEMCELL Technologies | 9805 | Blood cell media |
| CC110 | STEMCELL Technologies | 8697 | Blood cell media supplement (100x) |
| iPSC Generation and Culture Materials | |||
| CytoTune-iPSC Sendai Reprogramming Kit | Life Technologies | A16518 | |
| TeSR-E8 Media | STEMCELL Technologies | 5940 | iPSC media |
| Vitronectin | Life Technologies | A14700 | |
| ROCK Inhibitor | Sigma Aldrich | Y0503 | |
| TrypLE express (TrypLE) | Life Technologies | 12604-039 | |
| ReleSR | STEMCELL Technologies | 12604-039 | Colony detaching solution |
| Quality Control Materials | |||
| 18 mm Cover Glass | Superior | HSU-0111580 | |
| 4% Paraformaldyhyde | Tech & Innovation | BPP-9004 | |
| Triton X-100 | BIOSESANG | 9002-93-1 | |
| Bovine Serum Albumin | Vector Lab | SP-5050 | |
| Anti-SSEA4 Antibody | Millipore | MAB4304 | |
| Anti-Oct4 Antibody | Santa Cruz | SC9081 | |
| Anti-TRA-1-60 Antibody | Millipore | MAB4360 | |
| Anti-Sox2 Antibody | Biolegend | 630801 | |
| Anti-TRA-1-81 Antibody | Millipore | MAB4381 | |
| Anti-Klf4 Antibody | Abcam | ab151733 | |
| Alexa Fluor 488 goat anti-mouse IgG (H+L) antibody | Molecular Probe | A11029 | |
| Alexa Fluor 594 goat anti-rabbit IgG (H+L) antibody | Molecular Probe | A11037 | |
| DAPI | Molecular Probe | D1306 | |
| Prolong gold antifade reagent | Invitrogen | P36934 | |
| Slide Glass, Coated | Hyun Il Lab-Mate | HMA-S9914 | |
| Trizol | Invitrogen | 15596-018 | |
| Chloroform | Sigma Aldrich | 366919 | |
| Isoprypylalcohol | Millipore | 109634 | |
| Ethanol | Duksan | 64-17-5 | |
| RevertAid First Strand cDNA Synthesis kit | Thermo Scientfic | K1622 | |
| i-Taq DNA Polymerase | iNtRON BIOTECH | 25021 | |
| UltraPure 10X TBE Buffer | Life Technologies | 15581-044 | |
| loading star | Dyne Bio | A750 | |
| Agarose | Sigma-Aldrich | 9012-36-6 | |
| 1kb (+) DNA ladder marker | Enzynomics | DM003 | |
| Alkaline Phosphatase | Millipore | SCR004 | |
| Tris base | Fisher Scientific | BP152-1 | Rinse Buffer |
| Sodium Chloride | Duchefa Biochemie | S0520.1000 | Rinse Buffer |
| Tween-20 | BIOSESANG | T1027 | Rinse Buffer |
| Hydrochloric Acid | Duksan | 1129 | Rinse Buffer |
Referencias
- Serra, M., Brito, C., Correia, C., Alves, P. M. Process engineering of human pluripotent stem cells for clinical application. Trends Biotechnol. 30 (6), 350-359 (2012).
- Martin, G. R. Isolation of a pluripotent cell line from early mouse embryos cultured in medium conditioned by teratocarcinoma stem cells. Proc Natl Acad Sci U S A. 78 (12), 7634-7638 (1981).
- Takahashi, K., Yamanaka, S. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors. Cell. 126 (4), 663-676 (2006).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Chun, Y. S., Byun, K., Lee, B. Induced pluripotent stem cells and personalized medicine: current progress and future perspectives. Anat Cell Biol. 44 (4), 245-255 (2011).
- Seki, T., Fukuda, K. Methods of induced pluripotent stem cells for clinical application. World J Stem Cells. 7 (1), 116-125 (2015).
- Churko, J. M., Burridge, P. W., Wu, J. C. Generation of human iPSCs from human peripheral blood mononuclear cells using non-integrative Sendai virus in chemically defined conditions. Methods Mol Biol. 1036, 81-88 (2013).
- Loh, Y. H., et al. Reprogramming of T cells from human peripheral blood. Cell Stem Cell. 7 (1), 15-19 (2010).
- Ohmine, S., et al. Induced pluripotent stem cells from GMP-grade hematopoietic progenitor cells and mononuclear myeloid cells. Stem Cell Res Ther. 2 (6), (2011).
- Mae, S., et al. Monitoring and robust induction of nephrogenic intermediate mesoderm from human pluripotent stem cells. Nat Commun. 4, 1367(2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados