Method Article
Rilevamento di RNA-binding proteins by
In questo articolo
Erratum Notice
Riepilogo
Un protocollo di pull-down RNA è ottimizzato qui per il rilevamento delle interazioni tra RNA-binding proteins (RBPs) e non codificante e RNA codificanti. Un frammento di RNA da recettore degli androgeni (AR) è stato usato come esempio per dimostrare come recuperare la sua RBP da lystate degli adipociti bruni primarie.
Abstract
RNA-binding proteins (RBPs) stanno emergendo come uno strato di regolamentazione per lo sviluppo e la funzione del tessuto adiposo. RBPs svolgono un ruolo chiave nella regolazione dell'espressione genica a livello post-trascrizionale che interessano l'efficienza stabilità e traslazionale di mRNA bersaglio. RNA tecnica di pull-down è stato ampiamente utilizzato per studiare l'interazione RNA-proteina, che è necessario per chiarire il meccanismo sottostante funzione di RBPs 'così come lungo RNA non codificanti' (lncRNAs). Tuttavia, la grande abbondanza di lipidi negli adipociti rappresenta una sfida tecnica nella conduzione di questo esperimento. Ecco una dettagliata protocollo di pull-down RNA è ottimizzato per la cultura adipociti primaria. Un frammento di RNA da (AR) 3 'regione non tradotta del recettore degli androgeni (3'UTR) contenente un elementwas ricco adenilato-uridylate-usato come esempio per dimostrare come recuperare il suo partner RBP, proteine HuR, da adipociti lystate. Il metodo qui descritto può essere applicato per rilevare le interazioni traRBPs e RNA non codificanti, così come tra i RBPs e RNA codificanti.
Introduzione
RBPs sono proteine che si legano all'RNA doppia o singola bloccati in cellule e partecipano a formare complessi RNA-proteina. RBPs possono vincolare una varietà di specie di RNA, inclusi mRNA e RNA non codificanti lunghi (lncRNAs), e di esercitare la loro influenza a livello post-trascrizionale. Entrambi RBPs e lncRNAs stanno emergendo come nuovi regolatori nello sviluppo adiposo e funzionano 1,2,3. Per comprendere il meccanismo di RBP- e regolazione lncRNA-mediata vie cellulari, è spesso necessario rilevare l'interazione tra una molecola di RNA specifico e uno o più RBPs, e, a volte, per identificare l'intero spettro di partner proteici di un RNA trascrizione. Tuttavia, l'esperimento può essere difficile a causa dell'elevato contenuto di lipidi in adipociti. L'RNA protocollo tendina qui descritto può essere impiegato per recuperare i partner proteici di un RNA specifico dai lisati adipociti di colture primarie di 4,5.
Motivazioni per questo protoCol è riassunta come segue. Un esca RNA è trascritto in vitro da un modello di DNA, biotina-etichettati e coniugato con biglie magnetiche rivestite di streptavidina 4. L'esca RNA viene incubato con lisati cellulari per permettere la formazione di complessi RNA-proteina, che vengono successivamente abbassate su un supporto magnetico. Più specificamente, il protocollo di pull-down RNA descritto di seguito utilizzare un frammento di RNA da AR 3'UTR come esca per recuperare HuR proteina, un RBP universalmente espressa vincolato al 3'UTR del mRNA 6,7, da theadipocytelysate di coltura primaria. Per testare la specificità di legame di questo saggio, un RNAas non rilevanti e perline streptavidina vuote è incluso come controllo. Questo protocollo è compatibile con western blotting o spettrometria di massa (MS) per confermare la cattura di un RBP specifica o per identificare la piena repertorio di RBPs catturati, rispettivamente 8.
Diverse tecniche sono a disposizione per studiare l'interazione RNA-proteinaS. Per rivelare RNA legati da un dato RBP, RIP (RNA immunoprecipitazione) e CLIP (reticolazione UV e immunoprecipitazione) può essere applicata. Al contrario, per identificare i partner proteici di un dato RNA, RNA pull-down, Chirp (isolamento cromatina da purificazione RNA), CHART (cattura analisi ibridazione degli obiettivi di RNA) e RAP (purificazione RNA antisenso) può essere applicata. In confronto con quelle successive, RNA tecnica discesa richiede meno sforzo di creare. Può essere impiegato per catturare proteine in vitro e in vivo. L'approccio in vivo di RNA pull-down, che è tecnicamente più impegnativo rispetto al suo omologo in vitro, conserva le interazioni RNA-proteina da reticolazione nelle cellule, cattura RNA aptamer-tag di interesse da cellule, e rileva in seguito RBPs rilegati. RNA pull-down può essere utilizzato per arricchire bassi RBPs abbondanti, e per isolare e identificare i complessi RNA-proteina che hanno diversi ruoli funzionali nel controllo della regolazione cellulare 9,10 .
Protocollo
NOTA: L'RNA di interesse nel contesto di questo studio è un frammento di AR 3'UTR.
1. Preparazione di RNA marcato con biotina
- Per ottenere l'RNA, amplificare T7-AR-oligo mediante PCR, seguita da trascrizione in vitro utilizzando un kit commerciale e seguendo le istruzioni del produttore 11.
NOTA: Primer per PCR sono elencate nella Tabella 4: T7-AR-F e T7-AR-R. - Per non specifico di controllo vincolante, amplificare una sequenza parziale di DNA luciferasi di lucciola (FL) mediante PCR da psiCHECK-2 plasmide, seguita da in vitro trascrizione.
NOTA: Primer per PCR sono elencate nella Tabella 4: hluc (+) - T7-probe-F e hluc (+) - Sonda-R Tabella 4 Sequenze di modello e primer per l'amplificazione PCR... - Per un 50 microlitri reazione PCR, mescolare 50 ng del DNA (oligo o plasmide), 4 dNTP microlitri (2,5 mm di ogni singolo dNTP), 5 ml 10x tampone di reazione, 1 ml 2,5 U / & #181; l DNA polimerasi, 2 microlitri 10 micron di primer in avanti, 2 microlitri 10 micron di primer inverso, e acqua priva di nucleasi.
- Eseguire l'amplificazione PCR in un termociclatore utilizzando il seguente programma. STEP1: un ciclo di 95 ° C per 2 min; STEP2: 35 cicli di 95 ° C per 30 sec, 55 ° C per 30 sec e 72 ° C per 30 sec (AR) o 60 sec (FL); Passo 3: un ciclo di 72 ° C per 10 min.
- Sintetizzare RNA biotinilati utilizzando un kit di trascrizione in vitro seguendo le istruzioni del produttore 11. In questo esperimento, utilizzare prodotti di PCR come modelli per la sintesi di RNA. Per un microlitri in vitro reazione 20 trascrizione, mescolare 200 ng PCR DNA amplificato, 2 ml di ogni singolo NTP, 1 ml 10mM biotina-CTP, 2 ml 10x tampone di reazione, e 2 microlitri T7 RNA polimerasi. Incubare la miscela a 37 ° C per 4 ore.
- In seguito la trascrizione in vitro, miscela di reazione trattamento con 1 ml 2 U / ml DNasi a 37 & #176; C per 15 minuti per degradare DNA stampo. Infine, diluire l'RNA biotinilato ad un volume totale di 60 microlitri per successiva purificazione.
- Purificare RNA biotinilati per eliminare i sali e nucleotidi non incorporati utilizzando le colonne di purificazione seguendo le istruzioni del produttore 12. Misurare la concentrazione di RNA e conservare a -80 ° C per le successive analisi di pull-down.
- Per garantire la corretta struttura secondaria per l'RNA di interesse a interagire RNA-proteina, calore 50 microlitri di RNA biotinilato (50 pM) a 90 ° C per 2 min, raffreddamento su ghiaccio per 2 minuti, quindi fornire con 50 tampone RNA struttura microlitri 2x (Tabella 3), e permettere l'RNA pieghevole a temperatura ambiente per 30 min 13,14.
2. Preparazione di perline RNA-coniugato
NOTA: utilizzare un supporto magnetico per la separazione delle sfere magnetiche e surnatante.
- Risospendere le sfere magnetiche rivestite di streptavidina nel flacone originale per breve vortexing seguendo le istruzioni del produttore. Trasferire 150 ml microsfere in sospensione in una provetta da 1,5 ml. Posizionare il tubo su magnete per 1 min. surnatante degli scarti. Mantenere tallone pellet.
NOTA: In RNA saggi di pull-down, quando si dispensa microsfere in sospensione dal flacone originale, utilizzare 50 perle microlitri per un test seguito da western blotting, e 200 perline microlitri per un test seguito da MS. - Lavare perline volta da ri-sospensione tallone pellet con 1 ml di consigliata dal costruttore 1x W & B (vincolanti e lavaggio) tampone (Tabella 3), seguita ruotando la provetta per 5 min a temperatura ambiente in un rotatore. surnatante degli scarti. Mantenere tallone pellet.
- Per inattivare RNase su perline, lavare perline due volte, ogni volta per ri-sospensione tallone pellet con 400 microlitri tampone A. tampone A è una soluzione alcalina contenente 0,1 M NaOH e 0,05 M NaCl (Tabella 3). Ruotare la provetta per 2 minuti a temperatura ambiente in un rotatore. surnatante degli scarti. Mantenere tallone pellet.
- Per rimuovere NaOH da perline, lavare perline due volte, ogni volta per ri-sospensione tallone pellet con 400 microlitri tampone B (Tabella 3), seguita ruotando la provetta per 2 minuti a temperatura ambiente in un rotatore. surnatante degli scarti. Mantenere tallone pellet.
- Risospendere tallone pellet con 300 microlitri 2x W & B tampone, perline divisa in parti uguali e trasferire in tre provette da 1,5 ml (100 perle microlitri per tubo). Fornire i tubi tallone contenenti 100 microlitri RNasi acqua libera, 100 ml biotinilati FL RNA e 100 ml biotinilati AR 3'UTR RNA, rispettivamente.
- Incubare ogni miscela sferette per 1 ora a temperatura ambiente con rotazione. Posizionare i tubi sul magnete per 1 min. Eliminare tutto il surnatante. Mantenere pellet tallone.
- Lavare perline due volte, ogni volta per ri-sospensione ogni pellet tallone con 400 microlitri 1x W & B tampone, seguito da tubi rotante per 5 min a temperatura ambiente in un rotatore.
- Posizionare i tubi sul magnete per 1 min. Eliminare tutto il surnatante. Risospendere ogni pelle tallonet con 50 buffer di isolamento nucleare microlitri (Tabella 3).
3. Preadipocyte Isolamento e adipogenesis induzione
- Come descritto in precedenti pubblicazioni 5, sezionare preadipociti del cuscinetto adiposo bruno interscapolare (topi wild type C57BL6, 3 settimane).
- Crescere e in vitro differenziare le preadipocytes 5. Le cellule sono pronte per l'applicazione a valle 4-5 giorni dopo l'inizio della differenziazione.
4. Preparazione di Cellular lisato da adipociti primari
NOTA: Assicurarsi che tutte le procedure di preparazione lisato cellulare sono realizzati su ghiaccio, e tutti i buffer sono raffreddati su ghiaccio. omogeneizzatori vetro Dounce pre-raffreddamento su ghiaccio.
- Crescere e differenziarsi preadipocytes in 15 cm piatti 5 (vedere paragrafo 3.2). Ogni piatto fornisce cellule sufficienti per una RNA test di pull-down. Il giorno 4 o 5 giorni di differenziazione, mezzo di coltura cellulare di scarto, e lavare ceLLS una volta con PBS 1x.
- Per raccogliere le cellule, aggiungere 10-15 ml PBS 1x per le cellule, raschiare per raccogliere le cellule in una provetta da 50 ml, e le cellule pellet per centrifugazione a 200 xg per 5 minuti a temperatura ambiente. Gettare il surnatante utilizzando 1 ml pipetta.
- Pellet Risospendere cellule in 2 ml di tampone ipotonico (Tabella 3), e incubare le cellule in ghiaccio per 15 min. Trasferire le cellule in un omogeneizzatore di vetro da 15 ml Dounce e cellule taglio meccanicamente su ghiaccio con 20 colpi.
- Pellet nuclei delle cellule per centrifugazione a 3.300 xg per 15 min a 4 ° C. Mantenere pellet nuclei in ghiaccio per un ulteriore uso (vedere paragrafo 4.6). Trasferire il surnatante in provette da 1,5 ml, aggiungere 3M KCl al supernatante ad ottenere una concentrazione finale di 150 mM, e centrifuga a 20.000 xg per 15 min a 4 ° C.
- Dopo la centrifugazione, rimuovere con attenzione lipidlayeron alto utilizzando 1 ml pipetta, e raccogliere il surnatante come il lisato cellulare citoplasmatica.
- Risospendere pellet nuclei (vedere paragrafo 4.4) a 1 ml di tampone di isolamento nucleare. Tnuclei RASFERIMENTO ad un omogeneizzatore di vetro da 15 ml Dounce usando 1 ml pipetta, e nuclei di taglio meccanico sul ghiaccio con 100 colpi. Pellet detriti nucleare per centrifugazione a 20.000 xg per 15 min a 4 ° C, e raccogliere il surnatante come il lisato cellulare nucleare.
- O combinare i lisati cellulari nucleari e citoplasmatici, o utilizzare ogni frazione a parte, per l'applicazione di pull-down a valle. In questo esperimento demo, queste due frazioni di lisato cellulare sono stati combinati in un rapporto in volume di 1: 1.
- Aggiungere inibitore RNase a questo lisato cellulare non frazionata per ottenere una concentrazione finale di 0,5 U / ml. Mettere da parte 100 ml di lisato cellulare come controllo di input per Western Blotting 15.
5. rilegatura e eluizione di RBP
- lisato Split cella in tre aliquote uguali (lisato cellulare 1ml in una provetta da 1,5 ml), e incubare con vuoto, FL RNA-coniugato e perline AR 3'UTR RNA-coniugati (vedi paragrafo 2.8), rispettivamente, per 3 ore a 4 ° Ccon rotazione.
- Posizionare i tubi sul magnete per 1 min. Raccogliere il surnatante con una pipetta 1 ml, seguito da isolamento di RNA da surnatante per confermare l'RNA integralità. Isolamento dell'RNA utilizzando una soluzione di tiocianato di guanidinio-fenolo acido seguendo le istruzioni del produttore (Tabella 2). Valutare l'integrità RNA analizzando 28S e 18S subunità di RNA ribosomiale. Mantenere pellet tallone sul ghiaccio.
- Lavare le perline magnetiche sei volte, ogni volta per risospendere ogni pellet perlina di 1 ml di tampone di isolamento nucleare contenente 40 U RNasi inibitore e 0,25% IGEPAL, seguita da tubi rotante per 2 minuti a temperatura ambiente in un rotatore. Utilizzare un supporto magnetico per la separazione delle sfere magnetiche e surnatante. Eliminare tutto il surnatante. Mantenere pellet tallone sul ghiaccio.
- Utilizzare seguente procedura per eluire complessi RNA-proteina da perline per western blotting. In primo luogo, risospendere ogni pellet tallone in 75 microlitri di buffer di isolamento nucleare; poi, aggiungere 25 ml 4x Western Blot carico buffer (contenente SDS) per ottenere un volume totale di 100 microlitri. Infine, bollire perline in questa soluzione 100 ml a 95 ° C per 5 min.
NOTA: Utilizzare i seguenti passi per eluire complessi RNA-proteina da perline per la SM. STEP1: risospendere ogni pellet tallone in 100 microlitri tampone di eluizione (Tabella 3); STEP2: incubare campioni di perline per 2-3 ore a temperatura ambiente con rotazione; Fase 3: centrifuga e raccogliere il surnatante proteici; Fase 4: concentrato soluzioni di proteine di 20-30 ml prima della presentazione per la SM. - Centrifugare ogni 100 ml di campione perline a 12.000 xg per 30 sec a 4 ° C, e raccogliere il surnatante. Aliquota 15 microlitri surnatante per l'analisi Western Blot (vedere paragrafo 6.1 e 6.2). Sondare la membrana risultante con l'anticorpo monoclonale HuR topo diluito (1: 1000 diluizione), durante la notte.
6. Western Blotting per la verifica di RBP
- RBPs separate mediante SDS-PAGE utilizzando tecniche standard.
- condurre noipoppa assorbente utilizzando tecniche standard sondando per le RBPs connessi con l'RNA di interesse.
NOTA: In questo esperimento demo, HuR anticorpo monoclonale murino è stato utilizzato per la immunolocalizzazione di proteine HuR. - Incubare la membrana risultante con l'anticorpo HuR diluito (1: 1000 diluizione) in 5% w / v senza grassi latte in polvere, 1x TBS, 0,1% Tween - 20 a 4 ° C con dolce agitazione, durante la notte. Lavare membrana tre volte con 1x TBST. Incubare la membrana con l'anticorpo secondario (capra anti-topo IgG-HRP) a temperatura ambiente per 1 ora. membrana di lavaggio. Sviluppare e segnale di registrazione.
Risultati
In questo esperimento demo, un frammento di AR RNA è stato utilizzato come esca per catturare la sua proteina legante HuR. Sia un RNA esca FL che non è rilevante per la proteina HuR, ed una aliquota di coniugati perline streptavidina vuote serviti come controlli negativi. interazioni RNA-proteina possono avvenire sia in nucleo o nel citoplasma, e questo protocollo pull-down può essere applicato a totale o frazionato (nucleare o citoplasmatica) lisati cellulari. Analisi di proteine mediante western blotting mostra che il campione di 15 microlitri lisato cellulare non frazionata (vedi paragrafo 4.8), come ingresso di controllo non-tallone, ha dato un risultato positivo, indicando che la proteina HuR viene rilevato nel lisato adipociti della cultura mouse principale utilizzando l'anticorpo monoclonale. La frazione di eluizione AR RNA ha dato esito positivo, indicando che la proteina HuR viene rilevato nel lisato adipociti base alle AR perline RNA-coniugati per il saggio di pull-down RNA. Sia la frazione RNA eluizione FL e bcampione lank tallone ha dato risultati negativi, indicando che le interazioni non specifiche tra FL frammento di RNA e proteine HuR, nonché tra le perle bianche e proteine HuR non rilevabili da questo approccio pull-down RNA. I risultati di questi due controlli negativi indicano inoltre che una interazione specifica tra il frammento AR RNA e proteine HuR è stato rilevato dal RNA protocollo tendina qui descritto.
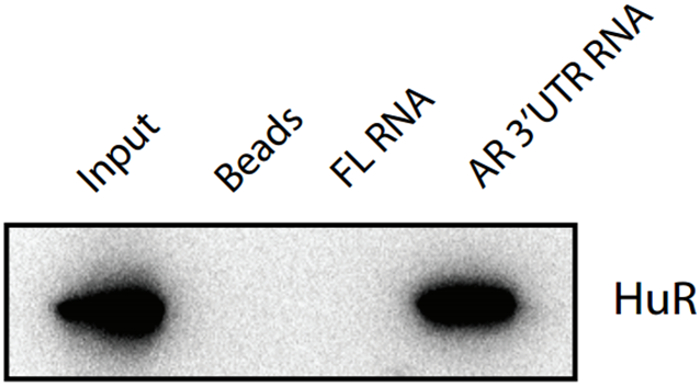
Figura 1: Western Blot verifica di Hur catturate dalla RNA a tendina saggi ingresso: ricostituito lisato cellulare (citoplasmatica + lisato nucleare);. Perle: il campione da sfere magnetiche rivestite di streptavidina vuote; FL RNA: perline streptavidina legati con la FL mRNA; AR RNA 3'UTR: perline streptavidina legati con l'AR 3'UTR dell'mRNA; Anticorpo primario: HuR anticorpo monoclonale murino; anticorpo secondario: capra anti-mouse IgG-HRP; Abbreviazione, FL: luciferasi di lucciola, AR: recettore degli androgeni; Clicca qui per vedere una versione più grande di questa figura.
| Centrifuga |
| termociclatore |
| Spettrofotometro |
| Rotatore |
| gel elettroforesi delle proteine e apparecchi di blotting |
| Magnete |
Tabella 1: principali attrezzature utilizzate in questo studio Le principali attrezzature utilizzate in questo studio..
| Nel kit di trascrizione in vitro |
| RNA biotinilato |
| Spin colonna per il recupero di frammenti di DNA e RNA |
| sfere magnetiche streptavidina |
| HuR del mouse anticorpo monoclonale |
| Capra anti-topo IgG-HRP |
| priva di nucleasi acqua grado biologia molecolare |
| Tampone fosfato salina |
| RNase Inhibitor |
| inibitore della proteasi |
| polimerasi Pfu DNA |
| guanidinio Acid soluzione tiocianato-fenolo |
Tabella 2: Principali reagenti utilizzati in questo studio importante reagenti utilizzati in questo studio..
| Tampone struttura 2x RNA |
| 20 mM Tris-HCl (pH 7,4) |
| 0.2 M KCl |
| 20 mM MgCl2 |
| 2 mM DTT |
| 0.8 U / ml RNasi inibitore |
| tampone A |
| 0,1 M NaOH |
| 0,05 M NaCl |
| Buffer B |
| 0.1 M NaCl |
| 1x W & B (rilegatura e di lavaggio) tampone |
| 5 mM Tris-HCl (pH7.4) |
| 1 M NaCl |
| 1x tampone ipotonica |
| 10 mM Tris-HCl (pH 7,4) |
| 10 mM KCl |
| 2 mM MgCl2 |
| 1 mM DTT |
| 1 mM PMSF |
| inibitore della proteasi 1x |
| Buffer di isolamento 1x nucleare |
| 25 mM Tris-HCl (pH 7,4) |
| 150 mM KCl |
| 2 mM MgCl2 |
| 1 mM DTT |
| 0,5% IGEPAL (o 0,25% IGEPAL per eluizione di RBPs) |
| 1 mM PMSF |
| inibitore della proteasi 1x |
| 0,4 U / inibitore della RNasi ml (o 40 U / ml RNasi inibitore per eluizione di RBPs) |
| 1x tampone di eluizione |
| 2 mM biotina in 1x PBS |
. Tabella 3: Le principali soluzioni utilizzate in questo studio tampone 2x struttura dell'RNA (vedi punto 1.7); Buffer A (vedi paragrafo 2.3); Buffer B (vedi paragrafo 2.4); 1x W & B (rilegatura e di lavaggio) tampone (vedere paragrafi 2.2, 2.5, 2.7); 1x tampone ipotonico (vedere paragrafo 4.3); buffer di isolamento 1x Nucleare (vedere paragrafi 2.8, 4.6, 5.3, 5.4); 1x tampone di eluizione (vedi NOTA seguente paragrafo 5.4).
lways "> Modello e primer sequenze T7-oligo-AR 5'-TCT AAT ACG ACT CAC TAT AGG GCT GGG CTT TTT TTT TCT TCT CTT CTT CTC TCT TTT TCT TCT TCC CTC CCT AGC TTA TGA CCG TGG CAG TCT -3 ' T7-AR-F 5'-CTA ATA CGA CTC ACT ATA G -3 ' T7-AR-R 5'-AGA CTG CCA CGG TCA TAA GC -3 ' hluc (+) - T7-probe-F 5'-TAATACGACTCACTATAGGGAGCCCCTGCTAACGACATTTACAACGAG-3 ' hluc (+) - Sonda-R 5'-CATAATCATAGGGCCGCGCACACAC-3 'Tabella 4: Sequenze di modello e primer per PCR amplificazione T7-AR-oligo:. Stampo di DNA per l'amplificazione PCR (vedere la sezione 1.1); T7-AR-F: forward primer per l'amplificazione PCR (vedi paragrafo 1.1); T7-AR-R: invertire primer per l'amplificazione PCR (vedi paragrafo 1.1); hluc (+) - T7-probe-F: forward primer per l'amplificazione PCR (vedere paragrafo 1.2); hluc (+) - Sonda-R: primer reverse per l'amplificazione PCR (vedi paragrafo 1.2).
Discussione
lncRNAs e RBPs hanno un ruolo vitale nella salute e la malattia, tuttavia i meccanismi molecolari di queste molecole sono poco conosciuti. Identificazione di proteine che interagiscono con le molecole lncRNA è un passo fondamentale verso la comprensione dei meccanismi di regolazione. Arricchimento del RBPs da parte del sistema di analisi a tendina RNA si basa sulla cattura in-soluzione di interagire complesso in modo che le proteine da un lisato cellulare possono essere selettivamente estratti utilizzando esche RNA biotinilati legati alle sfere magnetiche streptavidina. Diversi altri metodi come Chirp, grafico e RAP possono raggiungere lo stesso obiettivo, ma questi metodi vuole uno sforzo maggiore da configurare rispetto al RNA discesa tecnica 16,17,18.
La forza principale di RNA pull-down è che è relativamente semplice protocollo e facile da eseguire. Sia CHIRP e RAP comportano una fase di reticolazione che conserva naturalmente complessi RNA-proteina e la progettazione di una serie completa di oligonucleotidi antisenso (90 Nucleotides a lungo in RAP, e 20 nucleotidi a lungo in Chirp) piastrellatura l'intero bersaglio RNA 16,17,18. RNA pieghevole è un passo fondamentale nei saggi di pull-down di RNA. Il principale limite di questo protocollo è che le RNA bersaglio sono sintetizzati in vitro e non possono essere piegati correttamente nella struttura nativa per consentire la corretta associazione con la sua RBP (s). RNA non-native mal ripiegate possono sia riuscire a formare le interazioni RNA-proteine e invalidare saggi funzionali, o interazioni di forma che si verificano solo in vitro, in condizioni non fisiologici. Anche se il protocollo di pull-down descritto qui adottato un procedimento ben noto per RNA piegatura 13,14 (vedi paragrafo 1.7), non garantisce la corretta piegatura per altri trascritti di RNA.
RNA pull-down è spesso accoppiato con RIP come un approccio complementare per rilevare l'RNA (s) vincolati dal RBP di interesse 19. Un altro passo fondamentale in questo protocollo è la rimozione della frazione lipidica da cellula totalelisato (vedere paragrafo 4.5) in quanto la grande abbondanza di lipidi negli adipociti maturi rappresenta una grande sfida tecnica. Il protocollo qui descritto è particolarmente sviluppata per l'uso con adipociti. Tuttavia, i componenti di base di questo protocollo e il rigore di tutti i tamponi utilizzati possono essere modificati e adattati per l'applicazione di altri modelli di coltura cellulare. Per i tessuti che hanno alti livelli di RNase endogena, possono essere applicati diversi protocolli di pull-down di RNA.
RNA pull-down, che utilizza RNA di interesse per identificare le RBPs associati, è una tecnica efficace ed efficiente per sondare le funzioni cardine e meccanismi di lncRNAs, che hanno una vasta gamma di ruoli in cellule umane. Il futuro sviluppo della cattura RNA-based, tra cui l'RNA pull-down, sarà necessaria per affrontare le sfide con la definizione dei componenti, l'assemblaggio e la funzione di complessi RNA-proteina, così come la generazione RBPs sufficienti dal basso abbondanti complessi RNA-proteina per la SM.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Questo lavoro è stato finanziato dalla Singapore NRF borsa di studio (NRF-2011NRF-NRFF001-025), così come CBRG (NMRC / CBRG / 0070/2014).
Materiali
| Name | Company | Catalog Number | Comments |
| Major materials used in this study. | |||
| MEGAscript kit | Ambion | ||
| Biotin-14-CTP | Invitrogen | ||
| NucAway spin columns | Ambion | ||
| Dynabeads M-280 Streptavidin | Invitrogen | ||
| HuR (3A2) mouse monoclonal antibody (sc-5261) | Santa Cruz Biotechnology | ||
| Goat anti-mouse IgG-HRP(sc-2005) | Santa Cruz Biotechnology | ||
| Hypure Molecular Biology Grade Water (nuclease-free) | HyClone | ||
| Phosphate Buffer Saline (1×PBS) | HyClone | ||
| RNase inhibitor | Bioline | ||
| Protease inhibitor | Sigma | ||
| Pfu Turbo DNA polymerase | Agilent Technologies | ||
| TRIsure | Bioline | ||
| Name | Company | Catalog Number | Comments |
| Major equipment used in this study. | |||
| Sorvall Legend Micro 21R centrifuge | Thermo Scientific | ||
| Sorvall Legend X1R centrifuge | Thermo Scientific | ||
| Bio-Rad T100 thermal cycler | Bio-Rad | ||
| Nanodrop 2000 | Thermo Scientific | ||
| BOECO rotator Multi Bio RS-24 | BOECO,Germany | ||
| Protein gel electrophoresis and blotting apparatus | Bio-Rad | ||
| DynaMag-2 magnet | Life Technologies |
Riferimenti
- Huot, M. É, et al. The Sam68 STAR RNA-binding protein regulates mTOR alternative splicing during adipogenesis. Mol Cell. 46 (2), 187-199 (2012).
- Sun, L., et al. Long noncoding RNAs regulate adipogenesis. Proc Natl Acad Sci U S A. 110 (9), 3387-3392 (2013).
- Engreitz, J. M., et al. The Xist lncRNA exploits three-dimensional genome architecture to spread across the X-chromosome. Science. 341 (6147), 1237973(2013).
- Zhao, X. Y., Li, S., Wang, G. X., Yu, Q., Lin, J. D. A long noncoding RNA transcriptional regulatory circuit drives thermogenic adipocyte differentiation. Mol Cell. 55, 372-382 (2014).
- Alvarez-Dominguez, J. R., et al. De novo reconstruction of adipose tissue transcriptomes reveals long non-coding RNA regulators of brown adipocyte development. Cell Metab. 21, 764-776 (2015).
- Adams, D. J., Beveridge, D. J., van der Weyden, L., Mangs, H., Leedman, P. J., Morris, B. J. HADHB, HuR, and CP1 bind to the distal 3-untranslated region of human renin mRNA and differentially modulate renin expression. J Biol Chem. 278 (45), 44894-44903 (2003).
- Yeap, B. B., et al. Novel binding of HuR and poly(C)-binding protein to a conserved UC-rich motif within the 3´ untranslated region of the androgen receptor messenger RNA. J Biol Chem. 277, 27183-27192 (2002).
- König, J., Zarnack, K., Luscombe, N. M., Ule, J. Protein-RNA interactions: new genomic technologies and perspectives. Nat Rev Genet. 13, 77-83 (2012).
- Ferrè, F., Colantoni, A., Helmer-Citterich, M. Revealing protein-lncRNA interaction. Briefings in Bioinformatics. , 1-11 (2015).
- McHugh, C. A., Russell, P., Guttman, M. Methods for comprehensive experimental identification of RNA-protein interactions. Genome Biol. 15 (1), 203(2014).
- Lee, Y. S., et al. AU-rich element-binding protein negatively regulates CCAAT enhancer binding protein mRNA stability during long term synaptic plasticity in Aplysia. Proc Natl Acad Sci U S A. 109 (38), 15520-15525 (2012).
- Hermann, M., Cermak, T., Voytas, D. F., Pelczar, P. Mouse genome engineering using designer nucleases. J Vis Exp. (86), (2014).
- Tsai, M. C., et al. Long Noncoding RNA as Modular Scaffold of Histone Modification Complexes. Science. 329 (5992), 689-693 (2010).
- Wan, Y., et al. Landscape and variation of RNA secondary structure across the human transcriptome. Nature. 505, 706-709 (2014).
- Li, H., et al. Identification of mRNA binding proteins that regulate the stability of LDL receptor mRNA through AU rich elements. J Lipid Res. 50 (5), 820-831 (2009).
- McHugh, C. A., et al. The Xist lncRNA interacts directly with SHARP to silence transcription through HDAC3. Nature. 521 (7551), 232-236 (2015).
- Chu, C., et al. Systematic Discovery of Xist RNA Binding Proteins. Cell. 161 (2), 404-416 (2015).
- Chu, C., Spitale, R. C., Chang, H. Y. Technologies to probe functions and mechanisms of long noncoding RNAs. Nat Struct Mol Biol. 22 (1), 29-35 (2015).
- Zhao, J., et al. Genome-wide identification of Polycomb-associated RNAs by RIP-seq. Mol Cell. 40 (6), 939-953 (2010).
Erratum
Formal Correction: Erratum: Detection of RNA-binding Proteins by In Vitro RNA Pull-down in Adipocyte Culture
Posted by JoVE Editors on 8/20/2016. Citeable Link.
A correction to the author list was made to: Detection of RNA-binding Proteins by In Vitro RNA Pull-down in Adipocyte Culture.
The author list has been updated from:
Qianfan Bai1*, Zhiqiang Bai1*, Lei Sun1,2
1Cardiovascular and Metabolic Disorders Program, Duke-NUS Medical School
2Institute of Molecular and Cell Biology, Singapore
*These authors contributed equally
to:
Qianfan Bai1*, Zhiqiang Bai1*, Shaohai Xu3, Lei Sun1,2
1Cardiovascular and Metabolic Disorders Program, Duke-NUS Medical School
2Institute of Molecular and Cell Biology, Singapore
3Division of Bioengineering, School of Chemical & Biomedical Engineering, Nanyang Technological University
*These authors contributed equally
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon