Method Article
Nachweis von RNA-bindenden Proteinen durch
In diesem Artikel
Zusammenfassung
Ein RNA-Pull-down-Protokoll wird für den Nachweis von Interaktionen zwischen RNA-Bindeproteine (RBPs) und nicht-codierende sowie kodierenden RNAs hier optimiert. Ein RNA-Fragment von Androgen-Rezeptor (AR) wurde als Beispiel verwendet, zu zeigen, wie ihre RBP aus lystate primärer braunen Fettzellen zu erhalten.
Zusammenfassung
RNA-bindende Proteine (RBPs) entstehen als regulatorischer Ebene in der Entwicklung und Funktion von Fettgewebe. RBPs spielen eine wichtige Rolle bei der Genregulation bei posttranskritionelle Ebenen durch die Stabilität und Translationseffizienz von Ziel-mRNAs beeinflussen. RNA pull-down-Technik weit verbreitet RNA-Protein-Wechselwirkung zu untersuchen ist, die notwendig ist, den Mechanismus zugrunde liegende RBPs 'sowie lange nicht-kodierenden RNAs' (lncRNAs) Funktion aufzuklären. Jedoch stellt die hohe Lipid Fülle in Adipozyten eine technische Herausforderung, dieses Experiment in der Durchführung. Hier ist eine detaillierte RNA Pull-Down-Protokoll wird für die primäre Adipozyten Kultur optimiert. Ein RNA-Fragment von Androgen-Rezeptors (AR) 3 'untranslatierten Region (3'-UTR), die eine Adenylat-uridylat reichen elementwas verwendet als ein Beispiel enthält, zu zeigen, wie ihre RBP Partner HuR Protein von Adipozyten lystate abzurufen. Das hier beschriebene Verfahren kann um die Wechselwirkungen zu detektieren angewendet werden zwischenRBPs und nicht-kodierenden RNAs sowie zwischen RBPs und kodierenden RNAs.
Einleitung
RBPs sind Proteine, die bei der Bildung von RNA-Protein-Komplexe in Zellen zur Doppel- oder Einzelstrang-RNA binden und zu beteiligen. RBPs kann eine Vielzahl von RNA-Spezies binden, einschließlich mRNA und lange nichtkodierende RNAs (lncRNAs) und deren Einfluss auf posttranskriptionales Ebenen ausüben. Beide RBPs und lncRNAs sind , sich als neue Aufsichtsbehörden bei adipösen Entwicklung und Funktion 1,2,3. Um zu verstehen, den Mechanismus der RBP- und lncRNA vermittelte Regulation der zellulären Wege, ist es oft notwendig ist, die Wechselwirkung zwischen einem spezifischen RNA-Moleküls und eines oder mehrere RBPs zu erfassen, und manchmal das gesamte Spektrum von Proteinpartner einer RNA zu identifizieren, Transkript. Jedoch kann der Versuch aufgrund des hohen Gehaltes an Lipiden in Adipozyten schwierig sein. Die RNA - Pull-Down - Protokoll hier beschrieben werden eingesetzt 4,5 die Proteinpartner eines spezifischen RNA aus den Adipozyten Lysaten von Primärkulturen zu erhalten.
Rationale für dieses Protocol wird wie folgt zusammengefasst. Ein RNA - Köder in vitro aus einem DNA - Template transkribiert ist, Biotin-markierte und konjugiert an Streptavidin-beschichteten magnetischen Kügelchen 4. Die RNA Köder wird mit Zelllysaten inkubiert für die Bildung von RNA-Protein-Komplexen zu ermöglichen, die anschließend gezogen werden, auf einem Magnettreten. Mehr die RNA - Pull-Down - Protokoll unten beschrieben speziell, verwenden Sie ein RNA - Fragment von AR 3'UTR als Köder HuR Protein, ein universell ausgedrückt RBP gebunden 3'UTR von mRNAs 6,7, von theadipocytelysate der Primärkultur zu erhalten. Um die Bindungsspezifität dieses Assays testen, wurde eine nicht-relevanten RNAas auch blank Streptavidin-Kügelchen als Kontrolle eingeschlossen. Dieses Protokoll ist kompatibel mit Western - Blot oder Massenspektrometrie (MS) die Erfassung eines bestimmten RBP zu bestätigen oder das gesamte Repertoire der erfassten RBPs bzw. 8 zu identifizieren.
Verschiedene Techniken sind verfügbar RNA-Protein-Wechselwirkung zu untersuchens. Offenbaren RNAs gebunden durch eine gegebene RBP, RIP (RNA Immunpräzipitation) und CLIP (UV-Vernetzung und Immunpräzipitation) angewendet werden kann. Im Gegensatz dazu kann zu identifizieren angewendet werden Proteinpartner eines bestimmten RNA, RNA Pull-Down-Chirp (Chromatin Isolierung von RNA-Reinigung), CHART (Capture-Hybridisierungsanalyse von RNA-Targets) und RAP (RNA-Antisense-Reinigung). Im Vergleich zu den späteren, RNA-Pull-down-Technik dauert weniger Aufwand einzurichten. Es kann zu erfassen Proteine in vitro verwendet werden , und in vivo. Die in - vivo - Ansatz von RNA - Pull-down, die technisch anspruchsvoller als seine in - vitro - Gegenstück ist, RNA-Protein - Interaktionen bewahrt durch in Zellen Vernetzung erfasst Aptamer-markierte RNAs von Interesse von Zellen und erkennt anschließend gebunden RBPs. RNA - Pull-down kann zu bereichern niedrig reichlich RBPs verwendet werden, und die RNA-Protein - Komplexe zu isolieren und zu identifizieren , die bei der Kontrolle der zellulären Regulation 9,10 diversen funktionalen Rollen haben .
Protokoll
HINWEIS: Die RNA von Interesse im Rahmen dieser Studie ist ein Fragment von AR 3'UTR.
1. Herstellung von Biotin-markierter RNA
- Um die RNA zu erhalten, T7-AR-Oligos durch PCR, gefolgt von in vitro Transkription unter Verwendung eines kommerziellen Kits und nach den Anweisungen des Herstellers 11 amplifizieren.
HINWEIS: T7-AR-F und T7-AR-R: Primer für die PCR sind in Tabelle 4 aufgeführt. - Für nicht-spezifische Bindungskontrolle, amplifizieren eine Teilsequenz der Glühwürmchen - Luciferase (FL) DNA durch PCR aus psiCHECK-2 - Plasmid, gefolgt von in vitro Transkription.
HINWEIS: Primer für die PCR sind in Tabelle 4: hluc (+) - T7-probe-F und hluc (+) - Sonde-R Tabelle 4 Sequenzen von Template und Primer für die PCR - Amplifikation... - Für eine 50 & mgr; l PCR-Reaktion, mischen 50 ng DNA (Oligo oder Plasmid), 4 ul dNTPs (2,5 mM jedes einzelnen dNTP), 5 ul 10x Reaktionspuffer, 1 ul 2,5 U / & #181; l DNA-Polymerase, 2 & mgr; l 10 & mgr; M Vorwärtsprimer, 2 ul 10 uM Reverse-Primer und Nuklease-freies Wasser.
- Führen Sie die PCR-Amplifikation in einem Thermocycler das folgende Programm. Schritt 1: ein Zyklus von 95 ° C für 2 min; Schritt 2: 35 Zyklen von 95 ° C für 30 sec, 55 ° C für 30 sec und 72 ° C für 30 sec (AR) oder 60 s (FL); Schritt 3: ein Zyklus von 72 ° C für 10 min.
- Synthetisieren biotinylierten RNAs eine in vitro Transcription Kit folgenden Anweisungen des Herstellers unter Verwendung von 11. In diesem Experiment verwenden PCR-Produkte als Matrizen für die RNA-Synthese. Für eine 20 & mgr; l in vitro Transkriptionsreaktion, Mischungs 200 ng PCR amplifizierte DNA, 2 & mgr; l jedes einzelnen NTP, 1 ul 10 mM Biotin-CTP, 2 ul 10x Reaktionspuffer und 2 & mgr; l T7 - RNA - Polymerase. Inkubieren der Mischung bei 37 ° C für 4 Stunden.
- Folgende in vitro Transkription, treat Reaktionsmischung mit 1 ul 2 U / & mgr; l DNase bei 37 & #176; C für 15 min Templat-DNA abzubauen. Schließlich wird die biotinylierte RNA auf ein Gesamtvolumen von 60 ul zur anschließenden Reinigung verdünnen.
- Reinige biotinylierten RNAs Salze und nicht eingebauten Nukleotide zu entfernen , indem Reinigungssäulen verwendet folgenden Anweisungen des Herstellers 12. Messen Sie RNA-Konzentration und bei -80 ° C für die nachfolgende Pull-down-Assays.
- Um eine ordnungsgemäße Sekundärstruktur für die RNA von Interesse in der RNA-Protein - Wechselwirkung, Wärme 50 ul biotinyliertes RNA (50 pM) bei 90 ° C für 2 min, Chill auf Eis für 2 min, dann liefern mit 50 ul 2x RNA - Struktur - Puffer (Tabelle 3) und ermöglichen die RNA bei RT für 30 min Falten 13,14.
2. Herstellung von RNA-konjugierten Beads
HINWEIS: Verwenden Sie einen Magnetfuß zur Trennung der magnetischen Kügelchen und Überstand.
- Resuspendieren der Streptavidin-beschichteten magnetischen Kügelchen in der Originalflasche durch kurze Vortexing folgenden Anweisungen des Herstellers. Übertragen Sie 150 ul resuspendiert Kügelchen in ein 1,5-ml-Röhrchen. Das Röhrchen wird auf Magneten für 1 min. Überstand verwerfen. Halten Wulst Pellet.
HINWEIS: In der RNA Pull-down-Assays, wenn erneut suspendiert Perlen aus Originalfläschchen Aliquotierung, verwenden 50 ul Perlen für einen Test durch Western-Blot gefolgt, und 200 & mgr; l Perlen für einen Test von MS gefolgt. - Waschen Perlen einmal durch Resuspendieren Wulst Pellet mit 1 ml der empfohlenen 1x W & B des Herstellers (Bindungs- und Wasch) Puffer (Tabelle 3), gefolgt von den Röhrchen 5 Minuten bei Raumtemperatur in einem Rotator dreht. Überstand verwerfen. Halten Wulst Pellet.
- Zu inaktivieren RNase on beads, wasche zweimal Perlen, jeweils durch Resuspendieren bead Pellet mit 400 ul Puffer A Puffer A ist eine alkalische Lösung , die 0,1 M NaOH und 0,05 M NaCl (Tabelle 3). Drehen Sie das Rohr für 2 Minuten bei Raumtemperatur in einem Rotator. Überstand verwerfen. Halten Wulst Pellet.
- Zum Entfernen NaOH aus Perlen, zweimal waschen Perlen, jedes Mal durch Resuspendieren Wulst Pellet mit 400 ul Puffer B (Tabelle 3), gefolgt von der Röhre 2 min bei Raumtemperatur in einem Rotator dreht. Überstand verwerfen. Halten Wulst Pellet.
- Resuspendieren Wulst Pellet mit 300 ul 2x W & B-Puffer, geteilt Perlen gleichmäßig und Transfer in drei 1,5-ml-Röhrchen (100 & mgr; l Kügelchen pro Röhrchen). Versorgen Sie die Kügelchen enthaltende Röhrchen mit 100 & mgr; l RNase-freiem Wasser, biotinyliert 100 ul FL RNA und 100 ul biotinyliertes AR 3'UTR RNA sind.
- Inkubieren jedes Wulstes Mischung für 1 Stunde bei Raumtemperatur unter Rotation. Die Röhrchen auf Magnet für 1 min. Entsorgen Sie alle Überstand. Halten Sie Perle Pellets.
- Zweimal waschen Perlen, jeweils durch jedes Wulstes Pellet mit 400 ul 1x W & B-Puffer erneut suspendiert, gefolgt von Röhrchen für 5 min bei Raumtemperatur in einem Rotator dreht.
- Die Röhrchen auf Magnet für 1 min. Entsorgen Sie alle Überstand. Resuspendieren jede Wulst pellet mit 50 ul Kernisolationspuffer (Tabelle 3).
3. Preadipozyten Isolierung und Adipogenese Induction
- Wie in 5 in früheren Veröffentlichungen beschrieben, sezieren Preadipozyten aus dem interskapulären braunen Fettpolster (C57BL6 Wildtyp - Mäusen, 3 Wochen alt).
- Wachsen und in vitro Differenzierung der Präadipozyten 5. Zellen sind bereit für nachgeschaltete Anwendung 4-5 Tage nach Beginn der Differenzierung.
4. Herstellung von Zellysat von Primary Adipozyten
HINWEIS: Stellen Sie sicher, alle Verfahren von Zelllysat Zubereitung auf Eis, und alle Puffer auf Eis gekühlt. Pre-Chill-Dounce-Glas Homogenisator auf Eis.
- Wachsen und unterscheiden Preadipozyten in 15 cm - Schalen 5 (siehe Abschnitt 3.2). Jedes Gericht bietet ausreichend Zellen für eine RNA-Pull-Down-Assay. Am Tag 4 oder 5. Tag der Differenzierung, der Verwerfungszellkulturmedium, und waschen ceLLS einmal mit 1x PBS.
- Zur Ernte Zellen, fügen Sie 10-15 ml 1x PBS zu den Zellen, kratzen Zellen in einem 50-ml-Röhrchen und Pellet-Zellen durch Zentrifugation bei 200 g für 5 min bei RT zu sammeln. Überstand verwerfen unter Verwendung von 1 ml Pipette.
- Resuspendieren Zellpellet in 2 ml hypotonischen Puffer (Tabelle 3), und für 15 min Zellen auf Eis inkubieren. Transfer-Zellen zu einem 15 ml-Dounce-Glas-Homogenisator und Scherzellen mechanisch auf Eis mit 20 Schlägen.
- Pellet Zellkerne durch Zentrifugation bei 3.300 xg für 15 min bei 4 ° C. Halten Sie Kerne Pellets auf Eis für die weitere Verwendung (siehe Abschnitt 4.6). Überstand in 1,5 ml-Röhrchen, fügen 3M KCl gegen den Überstand zu einer Endkonzentration von 150 mM zu erhalten, und Schleudern bei 20.000 xg für 15 min bei 4 ° C.
- Nach der Zentrifugation vorsichtig entfernen lipidlayeron oben unter Verwendung von 1 ml Pipette und sammeln Überstand als zytoplasmatische Zellysat.
- Resuspendieren Kerne Pellet in 1 ml Kernisolationspuffer (siehe Abschnitt 4.4). Transfer Kerne in ein 15 ml-Dounce-Glas-Homogenisator unter Verwendung von 1 ml-Pipette und Scherkerne mechanisch auf Eis mit 100 Hüben. Kerntrümmer Pellet durch Zentrifugation bei 20.000 xg für 15 min bei 4 ° C und sammle Überstand als Kern zellulären Lysats.
- Entweder verbinden die Kern- und Cytoplasma-Zellysaten oder jede Fraktion getrennt zu verwenden, für die nachfolgende Pull-Down-Anwendung. In diesem Beispiel Experiment wurden diese zwei Fraktionen von Zell-Lysat in einem Volumenverhältnis von 1: 1.
- Hinzufügen RNase Inhibitor zu diesem Lysat unfraktioniertem Zelle zu einer Endkonzentration von 0,5 U / & mgr; l zu erhalten. Beiseite 100 ul Zelllysat als Eingangskontrolle für Western - Blot - 15.
5. Bindung und Elution von RBP
- Split Zelllysat in drei gleiche Aliquots (1 ml Zelllysat in einem 1,5-ml-Röhrchen), und Inkubation mit leeren, FL RNA-konjugierten und AR 3'UTR RNA-konjugierte Kügelchen (siehe Abschnitt 2.8), jeweils für 3 Stunden bei 4 ° Cmit Rotation.
- Die Röhrchen auf Magnet für 1 min. Sammeln Überstand einer 1 ml Pipette, gefolgt von RNA-Isolierung aus dem Überstand RNA Ganzheitlichkeit zu bestätigen. Isolieren RNA durch eine Säure Guanidiniumthiocyanat-Phenol - Lösung nach den Anweisungen des Herstellers verwendet (Tabelle 2). Bewerten Sie die RNA Intakt durch die Analyse von 28S und 18S-Untereinheiten der ribosomalen RNA. Halten Wulst Pellets auf Eis.
- Waschen Sie die magnetischen Kügelchen sechs Mal, jedes Mal durch Resuspendieren jedes Wulstes Pellet mit 1 ml Kernisolierungspuffer, enthaltend 40 U RNase-Inhibitor und 0,25% IGEPAL, gefolgt von Röhrchen für 2 min bei Raumtemperatur in einem Rotator dreht. Verwenden Sie einen Magnetfuß zur Trennung der magnetischen Kügelchen und Überstand. Entsorgen Sie alle Überstand. Halten Wulst Pellets auf Eis.
- Verwenden Sie folgende Schritte, um RNA-Protein-Komplexe, die aus Perlen für Western-Blot-eluieren. Zunächst resuspendieren jeder Wulst Pellet in 75 ul Kernisolationspuffer; Dann fügen Sie 25 ul 4x Western-Blot-Laden buffer (enthaltend SDS) mit einem Gesamtvolumen von 100 ul zu erhalten. Schließlich sieden Perlen in 100 ul dieser Lösung bei 95 ° C für 5 min.
HINWEIS: Verwenden Sie folgende Schritte RNA-Protein-Komplexe, die aus Perlen für MS zu eluieren. Schritt 1: resuspendieren jedes Wulstes Pellet in 100 & mgr; l Elutionspuffer (Tabelle 3); Schritt 2: bead Proben für 2-3 h bei Raumtemperatur unter Rotation inkubieren; Schritt 3: Zentrifuge und sammeln proteinhaltige Überstand; Schritt 4: Proteinlösungen 20-30 ul vor der Vorlage für MS konzentrieren. - Zentrifuge jeweils 100 & mgr; l Kügelchenprobe bei 12.000 xg für 30 sec bei 4 ° C und sammle den Überstand. Aliquot 15 ul Überstand für Western-Blot-Analyse (siehe Abschnitt 6.1 und 6.2). Sonde die resultierende Membran mit dem verdünnten HuR monoklonalen Maus-Antikörper (1: 1000 Verdünnung), über Nacht.
6. Western Blotting für die Überprüfung der RBP
- Separate RBPs durch SDS-PAGE unter Verwendung von Standardtechniken.
- Führen wirVerwendung von Standardtechniken Heck-Blotting durch Sondieren für die RBPs mit der RNA von Interesse verbunden.
Hinweis: In dieser Demo-Experiment, monoklonale HuR Maus-Antikörper für die Immunnachweis von HuR Protein verwendet wurde. - Inkubieren der resultierenden Membran mit dem verdünnten HuR Antikörper (1: 1000 Verdünnung) in 5% w / v fettfreie Trockenmilch, 1x TBS, 0,1% Tween - 20 bei 4 ° C unter leichtem Schütteln über Nacht. Waschen Sie die Membran dreimal mit 1x TBST. Inkubieren Membran mit dem sekundären Antikörper bei Raumtemperatur für 1 Stunde (anti-Maus-IgG-HRP der Ziege). Waschen Sie die Membran. Entwickeln und Aufzeichnungssignal.
Ergebnisse
In dieser Demo-Experiment wurde ein AR-RNA-Fragment als Köder verwendet, um seine bindende Protein HuR zu erfassen. Sowohl ein FL RNA Köder, die HuR Protein und ein Aliquot von unkonjugiertem blank Streptavidinbeads dienten als negative Kontrollen nicht relevant ist. RNA-Protein-Wechselwirkungen können entweder im Kern oder Cytoplasma auftreten, und das Pull-Down-Protokoll kann entweder insgesamt oder fraktionierte (Kern- oder cytoplasmatische) Zelllysate angewendet werden. Analyse von Proteinen durch Western-Blot zeigt, dass die Probe von 15 ul unfraktioniertem Zelllysat, da keine Perleneingangskontrolle ergab ein positives Ergebnis (siehe Abschnitt 4.8), was darauf hinweist, dass HuR-Protein in der Adipozyten-Lysat von Maus Primärkultur erkannt wird durch die Verwendung der monoklonale Antikörper ist. Die AR-RNA Elution Fraktion ergab ein positives Ergebnis, was darauf hinweist, dass HuR Protein mit Hilfe der AR-RNA-konjugierten Perlen für die RNA-Pull-Down-Assay in Adipozyten-Lysat erkannt wird. Sowohl die FL-RNA Elution Fraktion und die blank Wulst Probe ergaben negative Ergebnisse, dass unspezifische Wechselwirkungen zwischen FL-RNA-Fragment und HuR Protein anzeigt, sowie zwischen den leeren Perlen und HuR Protein sind von dieser RNA-Pull-down-Ansatz nicht nachweisbar. Ergebnisse dieser zwei negative Kontrollen zeigen auch, dass eine spezifische Wechselwirkung zwischen dem AR-RNA-Fragment und HuR Protein erfolgreich von der RNA-Pull-down hier beschriebene Protokoll erkannt wird.
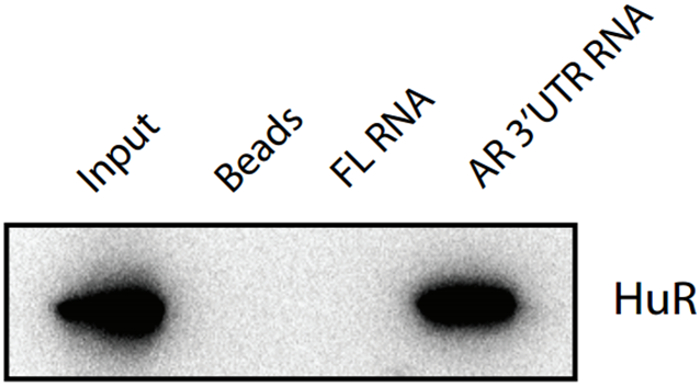
Abbildung 1: Western - Blot - Überprüfung von HuR Captured durch die RNA - Pull-down - Assays Eingang: rekonstruierter Zelllysat (zytoplasmatische + Kern Lysat);. Perlen: die Probe aus leeren Streptavidin-beschichteten magnetischen Kügelchen; FL RNA: Streptavidin mit dem FL-mRNA gebundenen Perlen; AR 3'UTR RNA: Streptavidin-Kügelchen gebunden mit dem AR 3'UTR mRNA; Primäre Antikörper: HuR monoklonalen Maus-Antikörper; Sekundäre Antikörper: Ziege anti-Maus IgG-HRP; Abkürzung, FL: Glühwürmchen - Luciferase, AR: Androgenrezeptor, bitte hier klicken , um eine größere Version dieser Figur zu sehen.
| Zentrifuge |
| Thermocycler |
| Spektrophotometer |
| Rotator |
| Protein-Gel-Elektrophorese und Blotapparatur |
| Magnet |
Tabelle 1: Bedeutende Ausrüstung benutzt in dieser Studie Die wichtigsten Geräte in dieser Studie verwendet..
| In - vitro - Transkription - Kit |
| Biotinylierte RNA |
| Spin-Säule zur Gewinnung von DNA und RNA-Fragmenten |
| Streptavidin magnetische Kügelchen |
| HuR monoklonalen Maus-Antikörper |
| Ziege-anti-Maus-IgG-HRP |
| Nuklease-freies Molekularbiologie Wasser |
| Phosphatpufferkochsalzlösung |
| RNase Inhibitor |
| Protease-Inhibitor |
| Pfu-DNA-Polymerase |
| Säure Guanidiniumthiocyanat-Phenol-Lösung |
Tabelle 2: Die wichtigsten Verwendete Reagenzien in dieser Studienrichtung Reagenzien in dieser Studie verwendet..
| 2x RNA - Struktur - Puffer |
| 20 mM Tris-HCl (pH 7,4) |
| 0,2 M KCl |
| 20 mM MgCl2 |
| 2 mM DTT |
| 0,8 U / ul RNase Inhibitor |
| Puffer A |
| 0,1 M NaOH |
| 0,05 M NaCl |
| Puffer B |
| 0,1 M NaCl |
| 1x W & B (Bindungs- und Wasch) Puffer |
| 5 mM Tris-HCl (pH 7,4) |
| 1 M NaCl |
| 1x hypotonischen Puffer |
| 10 mM Tris-HCl (pH 7,4) |
| 10 mM KCl |
| 2 mM MgCl2 |
| 1 mM DTT |
| 1 mM PMSF |
| 1x Protease-Inhibitor |
| 1x Kernisolationspuffer |
| 25 mM Tris-HCl (pH 7,4) |
| 150 mM KCl |
| 2 mM MgCl2 |
| 1 mM DTT |
| 0,5% IGEPAL (oder 0,25% IGEPAL für die Elution von RBPs) |
| 1 mM PMSF |
| 1x Protease-Inhibitor |
| 0,4 U / ul RNase-Inhibitor (oder 40 U / ml RNase-Inhibitor für die Elution von RBPs) |
| 1x Elutionspuffer |
| 2 mM Biotin in 1x PBS |
. Tabelle 3: Wichtige Verwendete Lösungen in dieser Studie 2x RNA - Struktur - Puffer (siehe Abschnitt 1.7); Puffer A (siehe Abschnitt 2.3); Puffer B (siehe Abschnitt 2.4); 1x W & B (Bindungs- und Wasch) Puffer (siehe Abschnitte 2.2, 2.5, 2.7); 1x hypotonischen Puffer (siehe Abschnitt 4.3); 1x Kernisolationspuffer (siehe Abschnitte 2.8, 4.6, 5.3, 5.4); 1x Elutionspuffer (HINWEIS folgenden siehe Abschnitt 5.4).
mmer "> Template und Primer Sequenzen T7-AR-Oligo 5 'TCT AAT ACG ACT CAC TAT AGG GCT GGG CTT TTT TTT TCT CTT TCT CTC CTT TCT TTT TCT TCT TCC CTC CCT AGC TTA TGA CCG TGG CAG TCT -3' T7-AR-F 5 'CTA ATA CGA CTC ACT ATA G-3' T7-AR-R 5 'AGA CTG CCA CGG TCA TAA GC -3' hluc (+) - T7-probe-F 5'-TAATACGACTCACTATAGGGAGCCCCTGCTAACGACATTTACAACGAG-3 ' hluc (+) - Sonde-R 5'-CATAATCATAGGGCCGCGCACACAC-3 'Tabelle 4: Sequenzen von Template und Primer für die PCR - Amplifikation T7-AR-Oligo. DNA - Matrize für die PCR - Amplifikation (siehe Abschnitt 10,1); T7-AR-F: Vorwärts-Primer für die PCR-Amplifikation (siehe Abschnitt 1.1); T7-AR-R: Rückwärts-Primer für die PCR-Amplifikation (siehe Abschnitt 1.1); hluc (+) - T7-Sonde-F: Vorwärts-Primer für die PCR-Amplifikation (siehe Abschnitt 1.2); hluc (+) - Sonde-R: Rückwärts-Primer für die PCR-Amplifikation (siehe Abschnitt 1.2).
Diskussion
lncRNAs und RBPs haben eine lebenswichtige Rolle in Gesundheit und Krankheit, aber die molekularen Mechanismen dieser Moleküle schlecht verstanden werden. Identifizierung von Proteinen, die mit lncRNA Molekülen interagieren ist ein wichtiger Schritt in Richtung auf die Regulationsmechanismen aufzuklären. Anreicherung von RBPs durch die RNA-Pull-Down-Assay-System wird in-Lösung auf Basis von Erfassung von komplexen interagieren, so dass Proteine, die aus einem Zelllysat selektiv extrahiert werden können biotinylierte RNA Köder gebunden an Streptavidin magnetische Kügelchen verwendet wird. Mehrere andere Verfahren wie Chirp, CHART und RAP kann das gleiche Ziel zu erreichen, aber diese Methoden mehr Aufwand nimmt als die RNA - Pull-down - Technik 16,17,18 einzurichten.
Die Stärke der RNA-Pull-down ist, dass es ein relativ einfaches Protokoll ist und einfach durchzuführen. Sowohl CHIRP und RAP einen Vernetzungsschritt umfassen, die natürlicherweise RNA-Protein-Komplexe und den Aufbau eines vollständigen Satz von Antisense-Oligonukleotiden vorkommende bewahrt (90 nucleotides lang in RAP und 20 Nukleotide lang in Chirp) , um das gesamte Ziel - RNA 16,17,18 Fliesen. RNA-Faltung ist ein kritischer Schritt in der RNA-Pull-down-Assays. Die Hauptbeschränkung dieses Protokolls ist es, dass die Ziel - RNAs in vitro synthetisiert werden und nicht richtig in native Struktur gefaltet werden kann , die richtige zu ermöglichen , mit seiner RBP (en) zu binden. Falsch gefaltete nicht-native RNAs können entweder scheitern RNA-Protein - Wechselwirkungen zu bilden und funktionelle Assays ungültig oder Form Wechselwirkungen , die nur in vitro unter nicht - physiologischen Bedingungen auftreten. Obwohl das Pull-Down - Protokoll hier beschrieben ein gut anerkanntes Verfahren zur RNA angenommen 13,14 Faltung (Abschnitt 1.7 sehen), ist es nicht für andere RNA - Transkripte richtige Faltung garantiert.
RNA - Pull-down wird oft mit RIP als komplementärer Ansatz gekoppelt , um die RNA (s) durch die RBP von Interesse 19 gebunden zu erkennen. Ein weiterer wichtiger Schritt in diesem Protokoll ist die Entfernung von Lipidfraktion aus GesamtzellLysat, da die hohe Lipid Hülle und Fülle in reifen Adipozyten stellt eine große technische Herausforderung dar (siehe Abschnitt 4.5). Das hier beschriebene Protokoll ist besonders für die Verwendung mit Adipozyten entwickelt. Jedoch können die Grundkomponenten dieses Protokolls und der Stringenz aller Puffer verwendet verändert und für die Anwendung auf andere Zellkulturmodellen angepasst werden. Für Gewebe , die hohe endogene RNase haben, verschiedene RNA - Pull-down - Protokolle angewendet werden kann.
RNA-Pull-down, die RNAs von Interesse verwendet die dazugehörigen RBPs zu identifizieren, ist eine effektive und effiziente Technik, um die zentralen Funktionen und Mechanismen von lncRNAs zu sondieren, die eine breite Palette von Rollen in menschlichen Zellen haben. Die zukünftige Entwicklung von RNA-basierten Erfassung, einschließlich RNA Pull-Down wird Herausforderungen benötigt werden, um Adresse mit der Definition der Komponenten, Montage und Funktion von RNA-Protein-Komplexe, sowie ausreichend RBPs von niedrigen reichlich RNA-Protein-Komplexe für MS zu erzeugen.
Offenlegungen
Keine Interessenkonflikte erklärt.
Danksagungen
Diese Arbeit wurde von Singapur NRF Gemeinschaft (NRF-2011NRF-NRFF001-025) sowie CBRG (NMRC / CBRG / 0070/2014) finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Major materials used in this study. | |||
| MEGAscript kit | Ambion | ||
| Biotin-14-CTP | Invitrogen | ||
| NucAway spin columns | Ambion | ||
| Dynabeads M-280 Streptavidin | Invitrogen | ||
| HuR (3A2) mouse monoclonal antibody (sc-5261) | Santa Cruz Biotechnology | ||
| Goat anti-mouse IgG-HRP(sc-2005) | Santa Cruz Biotechnology | ||
| Hypure Molecular Biology Grade Water (nuclease-free) | HyClone | ||
| Phosphate Buffer Saline (1×PBS) | HyClone | ||
| RNase inhibitor | Bioline | ||
| Protease inhibitor | Sigma | ||
| Pfu Turbo DNA polymerase | Agilent Technologies | ||
| TRIsure | Bioline | ||
| Name | Company | Catalog Number | Comments |
| Major equipment used in this study. | |||
| Sorvall Legend Micro 21R centrifuge | Thermo Scientific | ||
| Sorvall Legend X1R centrifuge | Thermo Scientific | ||
| Bio-Rad T100 thermal cycler | Bio-Rad | ||
| Nanodrop 2000 | Thermo Scientific | ||
| BOECO rotator Multi Bio RS-24 | BOECO,Germany | ||
| Protein gel electrophoresis and blotting apparatus | Bio-Rad | ||
| DynaMag-2 magnet | Life Technologies |
Referenzen
- Huot, M. &. #. 2. 0. 1. ;., et al. The Sam68 STAR RNA-binding protein regulates mTOR alternative splicing during adipogenesis. Mol Cell. 46 (2), 187-199 (2012).
- Sun, L., et al. Long noncoding RNAs regulate adipogenesis. Proc Natl Acad Sci U S A. 110 (9), 3387-3392 (2013).
- Engreitz, J. M., et al. The Xist lncRNA exploits three-dimensional genome architecture to spread across the X-chromosome. Science. 341 (6147), 1237973 (2013).
- Zhao, X. Y., Li, S., Wang, G. X., Yu, Q., Lin, J. D. A long noncoding RNA transcriptional regulatory circuit drives thermogenic adipocyte differentiation. Mol Cell. 55, 372-382 (2014).
- Alvarez-Dominguez, J. R., et al. De novo reconstruction of adipose tissue transcriptomes reveals long non-coding RNA regulators of brown adipocyte development. Cell Metab. 21, 764-776 (2015).
- Adams, D. J., Beveridge, D. J., van der Weyden, L., Mangs, H., Leedman, P. J., Morris, B. J. HADHB, HuR, and CP1 bind to the distal 3-untranslated region of human renin mRNA and differentially modulate renin expression. J Biol Chem. 278 (45), 44894-44903 (2003).
- Yeap, B. B., et al. Novel binding of HuR and poly(C)-binding protein to a conserved UC-rich motif within the 3´ untranslated region of the androgen receptor messenger RNA. J Biol Chem. 277, 27183-27192 (2002).
- König, J., Zarnack, K., Luscombe, N. M., Ule, J. Protein-RNA interactions: new genomic technologies and perspectives. Nat Rev Genet. 13, 77-83 (2012).
- Ferrè, F., Colantoni, A., Helmer-Citterich, M. Revealing protein-lncRNA interaction. Briefings in Bioinformatics. , 1-11 (2015).
- McHugh, C. A., Russell, P., Guttman, M. Methods for comprehensive experimental identification of RNA-protein interactions. Genome Biol. 15 (1), 203 (2014).
- Lee, Y. S., et al. AU-rich element-binding protein negatively regulates CCAAT enhancer binding protein mRNA stability during long term synaptic plasticity in Aplysia. Proc Natl Acad Sci U S A. 109 (38), 15520-15525 (2012).
- Hermann, M., Cermak, T., Voytas, D. F., Pelczar, P. Mouse genome engineering using designer nucleases. J Vis Exp. (86), (2014).
- Tsai, M. C., et al. Long Noncoding RNA as Modular Scaffold of Histone Modification Complexes. Science. 329 (5992), 689-693 (2010).
- Wan, Y., et al. Landscape and variation of RNA secondary structure across the human transcriptome. Nature. 505, 706-709 (2014).
- Li, H., et al. Identification of mRNA binding proteins that regulate the stability of LDL receptor mRNA through AU rich elements. J Lipid Res. 50 (5), 820-831 (2009).
- McHugh, C. A., et al. The Xist lncRNA interacts directly with SHARP to silence transcription through HDAC3. Nature. 521 (7551), 232-236 (2015).
- Chu, C., et al. Systematic Discovery of Xist RNA Binding Proteins. Cell. 161 (2), 404-416 (2015).
- Chu, C., Spitale, R. C., Chang, H. Y. Technologies to probe functions and mechanisms of long noncoding RNAs. Nat Struct Mol Biol. 22 (1), 29-35 (2015).
- Zhao, J., et al. Genome-wide identification of Polycomb-associated RNAs by RIP-seq. Mol Cell. 40 (6), 939-953 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten