Method Article
Diretto Evolution Metodo
In questo articolo
Riepilogo
We present a detailed protocol to construct and screen mutant libraries for directed evolution campaigns in Saccharomyces cerevisiae.
Abstract
Evoluzione diretta in Saccharomyces cerevisiae offre molti vantaggi interessanti nella progettazione di enzimi per applicazioni biotecnologiche, un processo che prevede la realizzazione, la clonazione e l'espressione di biblioteche mutante, accoppiato ad alta frequenza omologa ricombinazione del DNA in vivo. Qui, vi presentiamo un protocollo per creare e biblioteche schermo mutanti di lievito in base l'esempio di un fungina aril-alcol ossidasi (AAO) per migliorare la sua attività totale. Due segmenti proteici sono stati sottoposti ad evoluzione focalizzata diretto mediante mutagenesi casuale e in vivo ricombinazione DNA. Le sporgenze di ~ 50 bp fiancheggianti ogni segmento ammessi corretto rimontaggio del gene AAO-fusione in un vettore linearizzato dando luogo ad una completa plasmide autonomamente replicare. Librerie Mutant arricchiti con varianti funzionali AAO sono stati proiettati in S. surnatanti cerevisiae con un saggio ad alta sensibilità basato sulla reazione di Fenton. Il processo generale dicostruzione della libreria a S. cerevisiae qui descritto può essere facilmente applicato ad evolversi molti altri geni eucarioti, evitando reazioni PCR in più, in vitro di ricombinazione del DNA e legatura passi.
Introduzione
Evoluzione molecolare diretto è un metodo affidabile, veloce e affidabile per progettare enzimi 1, 2., Attraverso cicli iterativi di casuali mutazioni, ricombinazione e lo screening, le versioni migliorate di enzimi possono essere generati che agiscono sui nuovi substrati, nelle reazioni romanzo, a non naturale ambienti, o anche per assistere alla cella di raggiungere nuovi obiettivi metabolici 3-5. Tra i padroni di casa utilizzate in evoluzione diretta, di birra lievito Saccharomyces cerevisiae offre un repertorio di soluzioni per l'espressione funzionale di proteine eucariotiche complesse che non sono altrimenti disponibili in controparti procarioti 6,7.
Utilizzato esaustivamente in studi di biologia cellulare, questo piccolo modello eucariote ha molti vantaggi in termini di modificazioni post-traduzionali, la facilità di manipolazione e trasformazione di efficienza, che sono tutti tratti importanti per progettare enzimi dalla evoluzione diretta 8. Inoltre, l'alta frequenzadi omologa ricombinazione del DNA a S. cerevisiae accoppiato al suo apparato a prova di lettura efficiente apre una vasta gamma di possibilità per la creazione di una biblioteca e di assemblaggio genica in vivo, favorendo l'evoluzione dei sistemi diversi da singoli enzimi per vie artificiali complessi 9-12. Il nostro laboratorio ha trascorso gli ultimi dieci anni la progettazione di strumenti e strategie per l'evoluzione molecolare di diverse ligninases nel lievito (ossidoriduttasi coinvolti nella degradazione della lignina durante il decadimento legno naturale) 13-14. In questa comunicazione, vi presentiamo un protocollo dettagliato per la preparazione e le librerie schermo mutanti in S. cerevisiae per un modello flavooxidase, arile-alcol ossidasi (AAO 15) -, che può essere facilmente tradotto a molti altri enzimi. Il protocollo prevede un metodo di evoluzione mirata-diretto (MORPHING: mutageni organizzata ricombinazione omologa processo da in vivo raggruppamento) assistito dall'apparato cellula di lievito 16, unda test di screening molto sensibile basato sulla reazione di Fenton per rilevare l'attività all'AAO secreta nel brodo di coltura 17.
Protocollo
1. Mutant Biblioteca Edilizia
- Visita le regioni da sottoporre a MORPHING con l'aiuto di algoritmi di calcolo basati sui modelli di struttura di cristallo o di omologia disponibili 18.
- Qui, indirizzare due regioni di AAO da Pleurotus eryngii per mutagenesi casuale e ricombinazione (Met [α1] -Val109, Phe392-Gln566), mentre amplificando il resto del gene (844 bp) di alta fedeltà PCR (Figura 1).
Nota: Diversi segmenti possono essere studiati morphing in modo indipendente o combinata 16.
- Qui, indirizzare due regioni di AAO da Pleurotus eryngii per mutagenesi casuale e ricombinazione (Met [α1] -Val109, Phe392-Gln566), mentre amplificando il resto del gene (844 bp) di alta fedeltà PCR (Figura 1).
- Amplificare le zone interessate dal mutageno PCR. Creare zone tra i segmenti (~ 50 bp ciascuno) si sovrappongono sovrapponendo reazioni PCR delle regioni definite.
- Preparare mutageno PCR di segmenti target in un volume finale di 50 ml contenenti DNA stampo (0,92 ng / mL), 90 nM oligo senso (RMLN per segmento MI e AAO-BP per il segmento M-II), 90 nM di unntisense Primer (AAO-92C per il segmento MI e RMLC per il segmento M-II), 0,3 mM dNTP (0,075 mM ciascuno), 3% (v / v) dimetilsolfossido (DMSO), 1,5 mM MgCl 2, 0,05 mM MnCl 2 e 0,05 U / ml Taq DNA polimerasi. Primer sequenze sono descritti in Figura 1.
- Utilizzare il seguente programma PCR: 95 ° C per 2 min (1 ciclo); 95 ° C per 45 sec, 50 ° C per 45 sec, 74 ° C per 45 sec (28 cicli); e 74 ° C per 10 min (1 ciclo).
- Amplificare le regioni non mutageni con ultra-alto polimerasi fedeltà e comprende anche le zone di sovrapposizione corrispondenti segmenti mutageni e / o sbalzi vettore linearizzati.
- Preparare miscele di reazione in un volume finale di 50 ml contenente: template DNA (0,2 ng / ml), 250 nM oligo senso HFF, 250 nM oligo antisenso HFR, 0,8 mM dNTP (0,2 mM ciascuno), 3% (v / v) dimetilsolfossido (DMSO) e 0,02 U / ml iProof DNA polimerasi. Primer sequenze sono descritti in Figura 1.
- Utilizzare il seguente programma PCR: 98 ° C per 30 sec (1 ciclo); 98 ° C per 10 sec, 55 ° C per 25 sec, 72 ° C per 45 sec (28 cicli); e 72 ° C per 10 min (1 ciclo).
Nota: Con le condizioni descritte in 1.2 e 1.3 sovrapposizioni di 43 bp (regione plasmide-M1); 46 bp (M1 regione regione-HF); 47 bp (HF regione-M2 regione) e 61 bp (M2 plasmide regione-) sono progettate (Figura 1) per favorire in vivo splicing nel lievito. - Purificare tutti i frammenti di PCR (mutageni e non mutageni) con un kit di estrazione gel commerciale secondo il protocollo del produttore.
- Linearizzare il vettore tale che regioni fiancheggianti di circa 50 bp sono creati che sono omologhi ai 5'- e 3'-estremità del gene bersaglio.
- Preparare una miscela di reazione linearizzazione contenente 2 mg di DNA, 7.5 U Bam HI, 7.5 U Xho I, 20 mg BSA e 2 ml di tampone Bam HI 10x in un volume finale di 20 ul.
- Incubare la miscela di reazione a 37 ° C per 2 ore e 40 min. Successivamente, procedere con inattivazione a 80 ° C per 20 min.
- Purificare il vettore linearizzato mediante estrazione gel di agarosio per evitare la contaminazione con il plasmide circolare residua (Figura 2).
- Caricare la miscela di reazione di digestione nel mega-well di un basso punto di fusione gel semi-preparativa (0,75%, w: v) e un'aliquota (5 ml) della miscela di reazione nella adiacente e reporter.
- Run DNA elettroforesi (5 V / cm tra gli elettrodi, 4 ᵒC) e separare il gel corrispondente al mega-bene e conservarlo a 4 ᵒC in 1x TAE.
- Macchia la corsia con la scala peso molecolare e il giornalista. Visualizzare le bande sotto la luce UV. Nick la posizione in cui i luoghi linearizzate vettore.
Nota: Poiché la qualità del vettore linearizzato purificato è un critiFattore cal per ricombinazione di successo e di assemblaggio nel lievito, evitare di macchiare il gel per semi-preparativa elettroforesi del DNA. L'uso di coloranti e raggi UV per l'estrazione gel può influenzare la stabilità del vettore di DNA, compromettere l'efficienza vivo ricombinazione in. In alternativa ai coloranti EtBr tossici, Gel Rosso e coloranti SYBR sono comunemente usati per il gel di colorazione. - In assenza di luce UV, di individuare il vettore linearizzato nel frammento mega-pozzetto utilizzando la guida dei nick nella corsia giornalista macchiato in modo che possa essere isolato.
- Estrarre il vettore linearizzato da agarosio e purificarla con un kit di estrazione gel commerciale secondo il protocollo del produttore.
Nota: utilizzare ad alta copia vettori navetta episomiale con marcatori di antibiotici e auxotrofia: In questo esempio abbiamo impiegato il uracile indipendente e ampicillina resistenza pJRoC30 vettore, sotto il controllo del promotore di lievito GAL1.
- Preparare un mi equimolarexture dei frammenti PCR e mescolare con il vettore linearizzato in un rapporto 2: 1, con non meno di 100 ng di plasmide linearizzato (prova differenti rapporti di biblioteca equimolare / vettore aperta per ottenere buone rese di trasformazione).
- Misurare l'assorbanza dei frammenti di PCR e vettore linearizzato a 260 nm e 280 nm per determinare la loro concentrazione e purezza.
- Trasformare cellule competenti di lievito con la miscela di DNA utilizzando un kit di lievito di trasformazione commerciale (vedi tabella per le forniture) in base alle istruzioni del produttore.
- Qui, usare una proteasi carente e URA3 - dipendente S. ceppo cerevisiae, BJ5465. Trasformare le cellule con il vettore circolarizzato parentale come standard interno durante lo screening (vedi sotto). Inoltre, controllare il fondo trasformando il vettore linearizzato in assenza di frammenti di PCR.
Nota: In caso di rilevamento di livelli iniziali di secrezione bassi, usare S.cerevisiae proteasi ceppi carenti come BJ5465 per favorire l'accumulo di proteina attiva in sovranatanti. Se l'enzima bersaglio subisce hyperglycosylation, l'uso di ceppi glicosilazione-deficienti (ad esempio, Δ kre2 che è solo in grado di associare piccoli oligomeri mannosio) potrebbe essere una scelta adatta.
- Qui, usare una proteasi carente e URA3 - dipendente S. ceppo cerevisiae, BJ5465. Trasformare le cellule con il vettore circolarizzato parentale come standard interno durante lo screening (vedi sotto). Inoltre, controllare il fondo trasformando il vettore linearizzato in assenza di frammenti di PCR.
- Piastra le cellule trasformate su SC piastre di abbandono e incubare a 30 ° C per tre giorni. Piastra (su SC piastre di abbandono integrato con uracile) URA3 - S. cellule cerevisiae prive plasmide come controllo negativo per lo screening (vedi sotto).
2. Alta-Throughput Screening Assay (Figura 3)
- Riempire un numero appropriato di sterili piastre da 96 pozzetti (23 piastre per analizzare una libreria di cloni 2.000) con 50 microlitri terreno minimo per pozzetto con l'aiuto di un robot pipettamento.
- Scegli singole colonie dalla SC-drop out piatti e trasferirli alle piastre a 96 pozzetti.
- In ogni piatto, inoculare numero di colonna 6 con il tipo parentale come standard interno e ben H1 con URA3 - S. cellule cerevisiae (in media SC supplementato con uracile) senza plasmide come controllo negativo.
Nota: Beh H1 è riempito in particolare con i media di abbandono integrati con uracile. Un supporto vuoto pozzetto contenente senza cellule può essere preparata anche come controllo di sterilità aggiuntivo.
- In ogni piatto, inoculare numero di colonna 6 con il tipo parentale come standard interno e ben H1 con URA3 - S. cellule cerevisiae (in media SC supplementato con uracile) senza plasmide come controllo negativo.
- Coprire le piastre con i loro coperchi e avvolgerli in Parafilm. Incubare le piastre per 48 ore a 30 ° C, 225 rpm e 80% di umidità relativa in un agitatore umido.
- Rimuovere il Parafilm, aggiungere 160 ml di mezzo di espressione ad ogni pozzetto con l'aiuto del robot pipettaggio, sigillare le piastre e incubare per altre 24 ore.
Nota: terreno minimo e medio espressione sono preparati come riportato altrove 19. livelli di secrezione possono variare a seconda del gene in esame e, di conseguenza, tha incubazione devono essere ottimizzati in ogni caso a sincronizzare la crescita cellulare in tutti i pozzetti. - Centrifuga i piatti (master) a 2.800 x g per 10 minuti a 4 ° C.
- Trasferire 20 ml di surnatante dai pozzetti della piastra master piastra replica utilizzando una movimentazione multistazione robotico liquido.
Nota: Per favorire la secrezione di enzimi si consiglia di sostituire il segnale peptide nativo della proteina bersaglio da peptidi segnali comunemente utilizzati per l'espressione eterologa in lievito (ad esempio, il fattore α prepro-leader, il leader del K 1 Killer tossina da S. cerevisiae, o anche versioni chimeriche di entrambi i peptidi 13). In alternativa, il peptide segnale nativo può essere evoluto esclusivamente per la secrezione nel lievito. - Aggiungere 20 ml di 2 mM p -methoxybenzylalcohol a 100 mM sodio fosfato tampone pH 6.0 con l'aiuto del robot pipettamento. Mescolare le piastre brevemente con un 96-noill mixer piastra e incubare per 30 minuti a RT.
- Con il robot pipettaggio, aggiungere 160 ml di reagente FOX per ciascuna piastra replica e mescolare brevemente con il mixer (concentrazione finale di miscela FOX nel pozzo: 100 mM xilenolo arancio, 250 mM Fe (NH 4) 2 (SO 4) 2 e 25 mM H 2 SO 4).
- Aggiungere più additivi al reagente per migliorare la sensibilità, come co-solventi organici (DMSO, etanolo, metanolo) o sorbitolo 17. Qui, amplificare la risposta con l'aggiunta di sorbitolo ad una concentrazione finale di 100 mM (Figura 4).
- Leggere le piastre (modalità end-point, t 0) a 560 nm su un lettore di piastre.
- Incubare le piastre a temperatura ambiente fino a quando il colore si sviluppa e misurare nuovamente l'assorbimento (t 1).
- Calcolare l'attività relativa dalla differenza tra il valore Abs dopo incubazione e quella della misurazione iniziale normalizzati al pareTipo ntale per ogni piastra (Dt 1 - t 0).
- Sottoporre i migliori successi mutanti a due ri-proiezioni consecutive per escludere falsi positivi.
Nota: Tipicamente, ri-proiezioni includono isolamento plasmidico dal lievito, amplificazione e purificazione in Escherichia coli, seguita da trasformazione di cellule di lievito fresco con il plasmide 19. Ogni clone selezionato è ri-proiettato in pentaplicate.
Risultati
AAO da P. eryngii è un flavooxidase extracellulare che fornisce perossidasi fungine con H 2 O 2 per iniziare attaccare lignina. Due segmenti di AAO stati sottoposti a evoluzione focalizzata-diretto da MORPHING al fine di migliorare la sua attività e la sua espressione in S. cerevisiae 19. Indipendentemente dalle enzimi stranieri nutriti da S. cerevisiae, la questione più critica quando la costruzione di librerie di mutanti nel lievito riguarda la progettazione di specifiche regioni sovrapposte per favorire la giunzione tra i frammenti e la loro clonazione nel vettore linearizzato. Nell'esempio attuale, per ciascuna reazione di PCR, tutti i frammenti avevano sporgenze di circa 50 bp promuovere in splicing vivo nel lievito. Il numero di eventi di ricombinazione dipende dal numero di segmenti da assemblare e clonato con il vettore linearizzato (cioè, due eventi di crossover avvenuti tra latre segmenti PCR -il due segmenti mutageni che fiancheggiano il segment non mutagenizzato più due crossover supplementari con il vettore linearizzato; Figura 1). Secondo la nostra esperienza, le sequenze di sovrapposizione di lunghezza superiore a 50 bp diminuire la probabilità di ricombinazione interna mentre non migliorano l'efficienza di trasformazione.
Carichi mutazionali stati adeguati campionando librerie di mutanti con paesaggi diversi, calcolando il numero di cloni con <10% dell'attività dell'enzima parentale, e in seguito li controllo sequenziando un campione casuale di varianti attivi e non attivi (Figura 5A). Per la determinazione del coefficiente di variazione S. cellule cerevisiae sono state trasformate con l'AAO genitori e placcato su SC-drop out piatti. colonie individuali sono stati raccolti e inoculati in 96 a micropiastre e l'attività dei cloni è stato valutato dai preparati freschi. campione mutageni2 (Taq / MnCl 2 0,05 mm) è stato scelto come punto di partenza per la costruzione di una biblioteca e di screening.
Poiché l'attività biologica del AAO aumenta la concentrazione di H 2 O 2 nel mezzo di reazione, abbiamo cercato un saggio sensibile e preciso per quantificare lievi modifiche H 2 O 2. FOX è un metodo chimico basato sulla reazione di Fenton 20, per cui l'ossidazione di H 2 O 2 azionamenti la reazione di Fe 3+ con xilenolo arancio per formare un complesso blu-viola (o -cresolsulfone-phthalein 3 ', 3' '- bis (methylimino) diacetato ε 560 = 1,5 x 10 4 M -1 cm -1). La fase di ossidazione ferrosi è stato amplificato con l'aggiunta di sorbitolo per aumentare la sensibilità del saggio, aumentando la propagazione di radicali con ε apparente 560 = 2,25 x 10 5 M -1 cm -1 (Fifigura 4).
Il limite di rilevazione di questo dosaggio (nell'intervallo mM) è stata calcolata con il metodo determinazione bianco in una piastra a 96 pozzetti con standard in triplice copia (0, 0,5, 1, 1,5, 2, 2,5, 3 e 4 mM H 2 O 2 ) e utilizzando diversi surnatanti da S. cerevisiae privo della URA3 - plasmide (Figura 5B). Il saggio era lineare in presenza di sorbitolo (fino a 8 mM di H 2 O 2), anche se la linearità era più persistente in assenza di tale zucchero (almeno fino a 30 mM di H 2 O 2) la risposta era più debole (ad esempio, a 6 mM di H 2 O 2, un miglioramento di 4 volte è stato ottenuto in presenza di sorbitolo -Deep porpora da un'assorbanza di 0,24 in sua assenza -Dark arancione (Figura 5B)). Il rapporto tra Abs e la concentrazione AAO è stata valutata con quantità crescenti di eè stata osservata nzyme (da surnatanti lievito) e una risposta lineare; R 2 = 0,997 (Figura 5C).
È da notare che il segnale FOX era stabile per molte ore senza alcuna interferenza apparente dai diversi elementi del brodo di coltura. La sensibilità stimata di FOX era ~ 0,4 mM di H 2 O 2 prodotta dall'AAO nel surnatante in presenza di sorbitolo, e ~ 2 mM in sua assenza.
Una libreria mutante di 2.000 cloni è stato costruito e proiettato con questa analisi. Diversi mutanti AAO sono stati identificati con notevolmente migliorato la secrezione e l'attività contro l'alcool p -methoxybenzyl (Figura 5D) 19.

Figura 1.. MORPHING Protocollo per AAO Evolution due diverse regioni del AAO sono stati presi di mira per mutagenesi casuale e ricombinazione: M1 (blu, 590 bp) che include il peptide segnale (SP); M2 (giallo, 528 bp). La regione HF (grigio, 844 bp) è stato amplificato con polimerasi ad alta fedeltà. Regioni mutageni sono stati mappati nella struttura cristallina di AAO (PDB ID: 3FIM). Cliccate qui per vedere una versione più grande di questa figura.
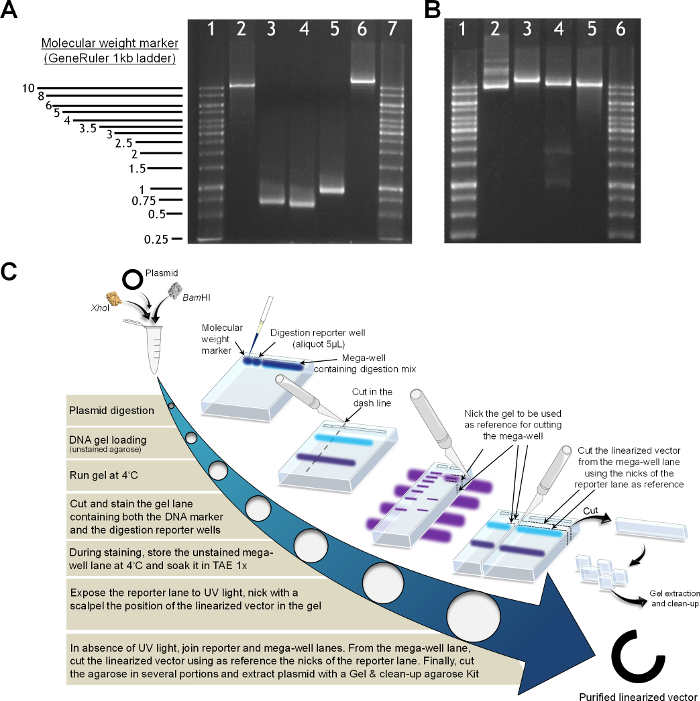
Figura 2. Preparazione di prodotti di PCR e il vettore linearizzato (A) Analytical gel (1% w: v). Contenente un indicatore del peso molecolare (1 kb scala) in corsie 1 e 7; il vettore linearizzato Bam HI e Xho I, corsia 2; PCR segmento M1, corsia 3; PCR segmento M2, corsia 4; PCR segmento HF, corsia 5; l'in vivo rimontato vettore linearizzato con Nhe I (contenente il gene completo AAO con le regioni MI, HF e M-II), corsia 6. (B) Vector linearizzazione, corsie 1 e 6 standard molecolari, 1 Kb scala; plasmide miniprep, corsia 2; plasmide linearizzato con Nhe I, corsia 3; plasmide linearizzato con Bam HI e Xho I, corsia 4; plasmide linearizzato ottenuto per estrazione gel e clean-up dopo la digestione, corsia 5. (C) Protocollo per la purificazione del plasmide. Cliccate qui per vedere una versione più grande di questa figura.

Figura 3. High-Throughput Screening protocollo. Panoramica del processo. Cliccate qui per visualizzare un vers più grandiione di questa figura.

Figura 4. Il metodo FOX. Funghi bianco-rot attaccare la parete cellulare del legno attraverso una reazione di Fenton che produce radicale ossidrile OH •. Il metodo coppie FOX questa reazione xilenolo arancio (XO), e l'assorbanza del complesso XO-Fe 3+ è misurata a 560 nm. Ferroso ossidazione viene amplificato con l'aggiunta di sorbitolo alla miscela dei reagenti. Si prega di cliccare qui per vedere una versione più grande di questa figura.

Figura 5. mutageni paesaggi per MORPHING librerie utilizzando diverso soggetto a errori di PCR Condizioni e validazione dei test di screening. (A ng>) paesaggi di morphing. linea orizzontale continua mostra l'attività del tipo parentale nel saggio mentre le linee tratteggiate indicano il coefficiente di variazione del dosaggio. Le percentuali indicano il numero di cloni con meno del 10% dell'attività enzimatica parentale. Le attività sono tracciate in ordine decrescente. (B) Il limite di rilevamento FOX è stata valutata con concentrazioni crescenti di H 2 O 2 in presenza (cerchi neri) e assenza (quadrati bianchi) di sorbitolo. Correlazione (C) lineare tra la concentrazione AAO (surnatanti trasformanti) e Abs 560nm. Ogni punto corrisponde alla media di 8 esperimenti e comprende la deviazione standard. (D) mutante paesaggio biblioteca. Le varianti selezionate (quadrati in ombra) sono stati nuovo controllo come riportato altrove 19. linea continua mostra l'attività di tipo parentale AAO.> Clicca qui per vedere una versione più grande di questa figura.
Discussione
In questo articolo, abbiamo riassunto la maggior parte dei suggerimenti e trucchi impiegati nel nostro laboratorio per progettare enzimi dalla evoluzione diretta a S. cerevisiae (utilizzando AAO come esempio) in modo che possano essere adattati per l'uso con molti altri sistemi enzimatici eucariotica semplicemente seguendo l'approccio comune descritti qui.
In termini di creazione di biblioteca, Morphing è un metodo rapido one-pot di introdurre e ricombinare le mutazioni casuali a piccoli tratti di proteine, lasciando le restanti regioni della proteina inalterata 16. Le librerie con diversi carichi mutazionali possono essere facilmente preparati e ricombinati in vivo, insieme con il plasmide linearizzato, per generare un vettore completo autonomamente replicare. E 'fondamentale che le sequenze che si sovrappongono fiancheggiano ogni tratto per consentire i frammenti del gene completo per essere rimontati attraverso la ricombinazione in vivo, evitando reazioni aggiuntivi PCR e nei passaggi legatura in vitro. In questo pROTOCOLLO, la frequenza di eventi di crossover tra frammenti di PCR può essere aumentata riducendo le dimensioni delle zone di sovrapposizione, anche se questo può compromettere l'efficienza di trasformazione. Indipendentemente dalle DNA polimerasi utilizzati per mutageno PCR, i carichi mutazionali possono essere regolati in precedenza costruendo e analizzando piccoli paesaggi libreria mutante (Figura 5A). Se si utilizza il kit GeneMorph II, è ancora consigliabile seguire questo approccio, poiché in vivo DNA ricombinazione può notevolmente modificare i carichi mutazionali stimato dal fabbricante. In termini generali, paesaggi mutanti in cui 35 - 50% del totale dei cloni proiettati hanno meno del 10% dell'attività parentale sono adatti per campagne evoluzione diretta, anche se questo numero varia in funzione della proteina bersaglio e la sua attività. In genere, l'analisi delle biblioteche mutante paesaggi sono ulteriormente verificata da sequenziamento del DNA di un campione casuale di mutanti. Nell'esempio attuale, la Taqpolimerasi DNA è stato utilizzato per il suo elevato tasso di errore, che è legata alla mancanza di 3'→ 5'proof lettura attività esonucleasi. I carichi mutazionali nelle librerie Taq sono stati modificati mediante l'aggiunta di diverse concentrazioni di MnCl 2, ma l'uso di dNTP sbilanciate e / o la riduzione delle concentrazioni di template gene sono opzioni adatte. limiti intrinseci di MORPHING provenire dal numero di segmenti da ricombinati. Secondo la nostra esperienza, fino a quattro blocchi di proteine (cinque eventi di crossover contando le aree di ricombinazione con il vettore linearizzato) possono essere assemblati in parallelo con buone rese di trasformazione (~ 10 5 cloni per reazione di trasformazione). Questo metodo può essere facilmente modificato per eseguito mutagenesi sito multiple-saturazione (ad esempio, utilizzando NDT primer degenerati o la creazione di degenerazione per 22 codoni uniche) per esplorare diverse posizioni contemporaneamente riducendo in modo significativo gli sforzi di screening 21,22.
Il diretta "cieco" protocollo di screening per AAO è estremamente sensibile ed affidabile (basato sulla rilevazione diretta di H 2 O 2 indipendentemente dal substrato dall'enzima), che rappresenta un saggio complementare ad altri ben consolidata indiretta protocolli per rilevare perossidi (per lo più perossidasi accoppiamento con substrati colorimetrici). Infatti, il dosaggio FOX è stato regolarmente impiegato per misurare H 2 O 2 in fluidi biologici, e può ora essere facilmente tradotto in protocolli di evolvere AAO e qualsiasi altro H 2 O 2 produrre enzimi (ad esempio, ossidasi di glucosio, deidrogenasi cellobiosio, gliossale ossidasi, metanolo ossidasi), in particolare per l'attività su substrati non naturali in cui le risposte sono altrimenti difficili da individuare.
S. cerevisiae è l'ospite più adeguata per evoluzione diretta di geni eucariotici poiché offre elevate efficienze di trasformazione (fino a 1 x 106 trasformanti / ug di DNA), si esegue l'elaborazione complessa e modificazioni post-traduzionali (compresi N- ed elaborazione C-terminale, e glicosilazione) ed esporta proteine estranee nel brodo di coltura tramite una via secretoria. Inoltre, ben consolidati strumenti di biologia molecolare, sono a disposizione per lavorare con questo lievito, compresi episomiale uni-o bi-direzionale (non integrativo) vettori navetta sotto il controllo di promotori di diversi punti di forza. Ultimo ma non meno importante, la sua alta frequenza di ricombinazione omologa DNA ha permesso una serie di metodi da sviluppare per ottenere la diversità DNA che sono attualmente in uso per evolvere singole proteine, nonché percorsi enzimatici più complessi 8, 12, 13, 23. il vivo riparazione lacuna e il dispositivo a prova di lettura di questo lievito possono anche essere impiegati per creare chimere quando i geni diversi (con circa. 60% di identità di sequenza del DNA), come pure per mischiare migliori prole / mutazioni da un directed campagna di evoluzione, o di riunire in vitro e in metodi di ricombinazione in vivo su un giro di evoluzione, arricchendo così le biblioteche mutante in termini di pieghevolezza e funzione.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
This work was supported by the European Commission project Indox-FP7-KBBE-2013-7-613549; a Cost-Action CM1303-Systems Biocatalysis; and the National Projects Dewry [BIO201343407-R] and Cambios [RTC-2014-1777-3].
Materiali
| Name | Company | Catalog Number | Comments |
| 1. Culture media | |||
| Ampicillin sodium salt | Sigma-Aldrich | A0166 | CAS Nº 69-52-3 M.W. 371.39 |
| Bacto Agar | Difco | 214010 | |
| Cloramphenicol | Sigma-Aldrich | C0378 | CAS Nº 56-75-7 M.W. 323.13 |
| D-(+)-Galactose | Sigma-Aldrich | G0750 | CAS Nº 59-23-4 M.W. 180.16 |
| D-(+)-Glucose | Sigma-Aldrich | G5767 | CAS Nº 50-99-7 M.W. 180.16 |
| D-(+)-Raffinose pentahydrate | Sigma-Aldrich | 83400 | CAS Nº 17629-30-0 M.W. 594.51 |
| Peptone | Difco | 211677 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P0662 | CAS Nº 7778-77-0 M.W. 136.09 |
| Uracil | Sigma Aldrich | U1128 | |
| Yeast Extract | Difco | 212750 | |
| Yeast Nitrogen Base without Amino Acids | Difco | 291940 | |
| Yeast Synthetic Drop-out Medium Supplements without uracil | Sigma-Aldrich | Y1501 | |
| Name | Company | Catalog Number | Comments |
| 2. PCR Reactions | |||
| dNTP Mix | Agilent genomics | 200415-51 | 25 mM each |
| iProof High-Fidelity DNA polymerase | Bio-rad | 172-5301 | |
| Manganese(II) chloride tetrahydrate | Sigma-Aldrich | M8054 | CAS Nº 13446-34-9 M.W. 197.91 |
| Taq DNA Polymerase | Sigma-Aldrich | D4545 | For error prone PCR |
| Name | Company | Catalog Number | Comments |
| 3. Plasmid linearization | |||
| BamHI restriction enzyme | New England Biolabs | R0136S | |
| Bovine Serum Albumin | New England Biolabs | B9001S | |
| XhoI restriction enzyme | New England Biolabs | R0146S | |
| Not I restriction enzyme | New England Biolabs | R0189S | |
| Gel Red | Biotium | 41003 | For staining DNA |
| Name | Company | Catalog Number | Comments |
| 4. FOX assays | |||
| Ammonium iron(II) sulfate hexahydrate | Sigma-Aldrich | F3754 | CAS Nº 7783-85-9 M.W. 392.14 |
| Anysil Alcohol | Sigma Aldrich | W209902 | CAS Nº 105-13-5 M.W. 138.16 |
| D-Sorbitol | Sigma-Aldrich | S1876 | CAS Nº 50-70-4 M.W. 182.17 |
| Hydrogen peroxide 30% | Merck Millipore | 1072090250 | FOX standard curve |
| Xylenol Orange disodium salt | Sigma-Aldrich | 52097 | CAS Nº 1611-35-4 M.W. 716.62 |
| Name | Company | Catalog Number | Comments |
| 5. Agarose gel stuff | |||
| Agarose | Norgen | 28035 | CAS Nº 9012-36-6 |
| Gel Red | Biotium | 41003 | DNA analysis dye |
| GeneRuler 1kb Ladder | Thermo Scientific | SM0311 | DNA M.W. standard |
| Loading Dye 6x | Thermo Scientific | R0611 | |
| Low-melting temperature agarose | Bio-rad | 161-3112 | CAS Nº 39346-81-1 |
| Name | Company | Catalog Number | Comments |
| 6. Kits and cells | |||
| S. cerevisiae strain BJ5465 | LGC Promochem | ATTC 208289 | Protease deficient strain with genotype: MATα ura3-52 trp1 leu2-delta1 his3-delta200 pep4::HIS3 prb1-delta1.6R can1 GAL |
| E. coli XL2-Blue competent cells | Agilent genomics | 200150 | For plasmid purification and amplification |
| NucleoSpin Gel and PCR Clean-up Kit | Macherey-Nagel | 740,609,250 | DNA gel extraction |
| NucleoSpin Plasmid Kit | Macherey-Nagel | 740,588,250 | Column miniprep Kit |
| Yeast Transformation Kit | Sigma-Aldrich | YEAST1-1KT | Included DNA carrier (Salmon testes) |
| Zymoprep yeast plasmid miniprep I | Zymo research | D2001 | Plasmid extraction from yeast |
| Name | Company | Catalog Number | Comments |
| 7. Plates | |||
| 96-well plates | Greiner Bio-One | 655101 | Clear, non-sterile, polystyrene (for activity measurements) |
| 96-well plates | Greiner Bio-One | 655161 | Clear, sterile, polystyrene (for microfermentations) |
| 96-well plate lid | Greiner Bio-One | 656171 | Clear, sterile, polystyrene (for microfermentations) |
Riferimenti
- Jäckel, C., Hilvert, D. Biocatalysts by evolution. Curr. Opin. Biotechnol. 21 (6), 753-759 (2010).
- Bornscheuer, U. T. Engineering the third wave of biocatalysis. Nature. 485 (7397), 185-194 (2012).
- Renata, H., Wang, Z. W., Arnold, F. H. Expanding the enzyme universe: accessing non-natural reactions by mechanism-guided directed evolution. Angew. Chem. Int. Ed. 54 (11), 3351-3367 (2015).
- Cobb, R. E., Chao, R., Zhao, H. Directed evolution: past, present and future. AIChE J. 59 (5), 1432-1440 (2013).
- Abatemarco, J., Hill, A., Alper, H. S. Expanding the metabolic engineering toolbox with directed evolution. Biotechnol. J. 8 (12), 1397-1410 (2013).
- Pourmir, A., Johannes, T. W. Directed evolution: selection of the host organism. Comput Struct Biotechnol J. 2 (3), e201209012(2012).
- Krivoruchko, A., Siewers, V., Nielsen, J. Opportunities for yeast metabolic engineering: lessons from synthetic biology. Biotechnol J. 6 (3), 262-276 (2011).
- Gonzalez-Perez, D., Garcia-Ruiz, E., Alcalde, M. Saccharomyces cerevisiae in directed evolution: an efficient tool to improve enzymes. Bioeng Bugs. 3, 172-177 (2012).
- Alcalde, M. Mutagenesis protocols in Saccharomyces cerevisiae by In Vivo Overlap Extension. Methods Mol. Biol. 634, 3-14 (2010).
- Bulter, T., Alcalde, M. Preparing libraries in Saccharomyces cerevisiae. Methods. Mol. Biol. 231, 17-22 (2003).
- Ostrov, N., Wingler, L. M., Cornish, W. Gene assembly and combinatorial libraries in S. cerevisiae via reiterative recombination. Methods. Mol. Biol. 978, 187-203 (2013).
- Shao, Z., Zhao, H., Zhao, H. DNA assembler, an in vivo genetic method for rapid construction of biochemical pathways. Nucleic Acids Res. 37 (2), e16(2009).
- Alcalde, M. Engineering the ligninolytic enzyme consortium. Trends Biotechnol. 33 (3), 155-162 (2015).
- Garcia-Ruiz, E. Directed evolution of ligninolytic oxidoreductases: from functional expression to stabilization and beyond. Cascade Biocatalysis: integrating stereoselective and environmentally friendly reactions. , Wiley-VCH. 1-22 (2014).
- Hernandez-Ortega, A., Ferreira, P., Martinez, A. T. Fungal aryl-alcohol oxidase: a peroxide-producing flavoenzyme involved in lignin degradation. Appl. Microbiol. Biotechnol. 93 (4), 1395-1410 (2012).
- Gonzalez-Perez, D., Molina-Espeja, P., Garcia-Ruiz, E., Alcalde, M. Mutagenic organized recombination process by homologous in vivo grouping (MORPHING) for directed enzyme evolution. PLoS One. 9, e90919(2014).
- Rhee, S. G., Chang, T., Jeong, W., Kang, D. Methods for Detection and Measurement of Hydrogen Peroxide Inside and Outside of Cells. Mol. Cells. 29 (6), 539-549 (2010).
- Sebestova, E., Bendl, J., Brezovsky, J., Damborsky, J. Computational tools for designing smart libraries. Methods. Mol. Biol. 1179, 291-314 (2014).
- Viña-Gonzalez, J., Gonzalez-Perez, D., Ferreira, P., Martinez, A. T., Alcalde, M. Focused directed evolution of aryl-alcohol oxidase in yeast using chimeric signal peptides. Appl. Environ. Microbiol. , (2015).
- Gay, C., Collins, J., Gebicki, J. M. Hydroperoxide Assay with the Ferric-Xylenol orange Complex. Anal. Biochem. 273 (2), 149-155 (1999).
- Reetz, M. T. Biocatalysis in organic chemistry and biotechnology: Past, present, and future. J. Am. Chem. Soc. 135 (34), 12480-12496 (2013).
- Mate, D. M., Gonzalez-Perez, D., Mateljak, I., Gomez de Santos, P., Vicente, A. I., Alcalde, M. The pocket manual of directed evolution: Tips and tricks. Biotechnology of Microbial Enzymes: Production, Biocatalysis and Industrial Applications. , Elsevier. Forthcoming Forthcoming.
- Chao, R., Yuan, Y., Zhao, H. Recent advances in DNA assembly technologies. FEMS Yeast Res. 15, 1-9 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon
Utilizziamo i cookies per migliorare la tua esperienza sul nostro sito web.
Continuando a utilizzare il nostro sito web o cliccando “Continua”, accetti l'utilizzo dei cookies.