Method Article
Dirigida Método Evolución en
En este artículo
Resumen
We present a detailed protocol to construct and screen mutant libraries for directed evolution campaigns in Saccharomyces cerevisiae.
Resumen
La evolución dirigida en Saccharomyces cerevisiae ofrece muchas ventajas atractivas en el diseño de enzimas para aplicaciones biotecnológicas, un proceso que consiste en la construcción, la clonación y la expresión de bibliotecas de mutantes, junto a la alta frecuencia de recombinación homóloga de ADN in vivo. A continuación, se presenta un protocolo para crear y bibliotecas mutantes de levadura en pantalla basado en el ejemplo de un fungicida aril-alcohol oxidasa (AAO) para mejorar su actividad total. Dos segmentos de proteínas se sometieron a la evolución dirigida-centrado por mutagénesis aleatoria y recombinación de ADN in vivo. Voladizos de ~ 50 pb que flanquean cada segmento permite el correcto montaje del gen AAO-fusión en un vector linealizado que da lugar a un plásmido de replicación autónoma completa. Bibliotecas mutantes enriquecidos con variantes funcionales AAO se rastrearon en S. sobrenadantes cerevisiae con un ensayo de alto rendimiento sensible basado en la reacción de Fenton. El proceso general deconstrucción de la biblioteca en S. cerevisiae se describe aquí se puede aplicar fácilmente a evolucionar muchos otros genes eucarióticos, evitando reacciones adicionales de PCR, en la recombinación de ADN y la ligadura pasos in vitro.
Introducción
Evolución molecular dirigida es un método robusto, rápido y fiable para diseñar enzimas 1, 2. A través de rondas iterativas de mutación al azar, recombinación y análisis, versiones mejoradas de las enzimas se pueden generar que actúan sobre los nuevos sustratos, en las reacciones novedosas, en no natural ambientes, o incluso para ayudar a la célula para alcanzar nuevos objetivos metabólicos 3-5. Entre los anfitriones utilizados en la evolución dirigida, levadura Saccharomyces cerevisiae de cerveza ofrece un repertorio de soluciones para la expresión funcional de las proteínas eucariotas complejos que no son de otra manera disponible en homólogos procariotas 6,7.
Se utiliza exhaustivamente en estudios de biología celular, este pequeño modelo eucariota tiene muchas ventajas en términos de las modificaciones post-traduccionales, la facilidad de manipulación y la eficiencia de transformación, todos los cuales son rasgos importantes para diseñar enzimas por evolución dirigida 8. Por otra parte, la alta frecuenciade la recombinación del ADN homólogo en S. cerevisiae acoplado a su aparato de prueba de lectura eficiente abre una amplia gama de posibilidades para la creación de la biblioteca y el conjunto de genes in vivo, el fomento de la evolución de los diferentes sistemas de enzimas individuales a vías artificiales complejos 9-12. Nuestro laboratorio ha pasado la última década el diseño de herramientas y estrategias para la evolución molecular de diferentes ligninasas en la levadura (oxidorreductasas implicadas en la degradación de la lignina durante la descomposición de la madera natural) 13-14. En esta comunicación, presentamos un protocolo detallado para preparar y bibliotecas mutantes pantalla en S. cerevisiae para un modelo flavooxidase, alcohol-oxidasa-arilo (AAO 15) -, que se puede traducir fácilmente a muchas otras enzimas. El protocolo consiste en un método de evolución dirigida centrado (MORPHING: Proceso mutagénico Organizada recombinación homóloga in vivo por la Agrupación), asistido por el aparato de célula de levadura 16, unada ensayo de cribado muy sensible basado en la reacción de Fenton con el fin de detectar la actividad de AAO secretada en el caldo de cultivo 17.
Protocolo
1. Mutante de construcción de biblioteca
- Elegir las regiones para ser sometidos al de transformación con la ayuda de algoritmos de cálculo en base a la estructura cristalina o de homología de 18 modelos disponibles.
- Aquí, el objetivo de dos regiones de AAO de Pleurotus eryngii para la mutagénesis al azar y la recombinación (Met [α1] -Val109, Phe392-Gln566), mientras que la amplificación de la resto del gen (844 bp) por PCR de alta fidelidad (Figura 1).
Nota: Varios segmentos pueden ser estudiados por MORPHING de manera independiente o combinada 16.
- Aquí, el objetivo de dos regiones de AAO de Pleurotus eryngii para la mutagénesis al azar y la recombinación (Met [α1] -Val109, Phe392-Gln566), mientras que la amplificación de la resto del gen (844 bp) por PCR de alta fidelidad (Figura 1).
- Amplificar las zonas seleccionadas por PCR mutagénica. Crear áreas entre segmentos (~ 50 pb cada uno) se superponen mediante la superposición de las reacciones de PCR de las regiones definidas.
- Preparar PCR mutagénica de los segmentos objetivo en un volumen final de 50 l que contenía ADN molde (0.92 ng / l), 90 nM sentido oligo (RMLN para el segmento de MI y AAO-BP para el segmento M-II), 90 nM unacebador ntisense (AAO-92C para el segmento de MI y RMLC para el segmento M-II), dNTPs 0,3 mM (0,075 mM cada uno), 3% (v / v) sulfóxido de dimetilo (DMSO), 1,5 mM MgCl 2, MnCl 0,05 mM 2 y 0,05 U / l de Taq ADN polimerasa. Secuencias de cebadores se detalla en la Figura 1.
- Utiliza el siguiente programa de PCR: 95ºC durante 2 min (1 ciclo); 95 ° C durante 45 s, 50ºC durante 45 s, 74ºC durante 45 segundos (28 ciclos); y 74 ° C durante 10 min (1 ciclo).
- Amplificar las regiones no mutagénicos con ultra alta fidelidad de la polimerasa e incluyen las áreas correspondientes se solapan los segmentos mutagénicos y / o salientes vector linealizado.
- Preparar las mezclas de reacción en un volumen final de 50 l que contenía: plantilla de ADN (0,2 ng / l), 250 nM sentido oligo HFF, 250 nM HFR oligo antisentido, mM dNTPs 0,8 (0,2 mM cada uno), 3% (v / v) de dimetilsulfóxido (DMSO) y 0,02 U / l iproof ADN polimerasa. secuencias de los cebadores se detallan en Figura 1.
- Utiliza el siguiente programa de PCR: 98 ° C durante 30 segundos (1 ciclo); 98 ° C durante 10 seg, 55 ° C durante 25 seg, 72 ° C durante 45 seg (28 ciclos); y 72 ° C durante 10 min (1 ciclo).
Nota: Con las condiciones descritas en el punto 1.2 y 1.3 solapamientos de 43 pb (región de plásmido-M1); 46 pb (M1 región por región HF); 47 pb (región-región M2 HF) y 61 pb (M2 plásmido región) se han diseñado (Figura 1) para favorecer el empalme en vivo en la levadura. - Purificar todos los fragmentos de PCR (mutagénicos y no mutagénicos) con un kit de extracción de gel comercial de acuerdo con el protocolo del fabricante.
- Linealizar el vector de modo que se crean regiones de aproximadamente 50 pares de bases que flanquean que son homólogos a los extremos 5 'y 3' extremos del gen diana.
- Preparar una mezcla de reacción que contiene linealización 2 g de ADN, 7,5 U Bam HI, 7,5 U XhoI, 20 g de BSA y 2 l de tampón de Bam HI 10x en un volumen final de 20 l.
- Incubar la mezcla de reacción a 37 ° C durante 2 horas y 40 min. A continuación, proceder a la inactivación a 80 ° C durante 20 minutos.
- Se purifica el vector linealizado mediante extracción en gel de agarosa para evitar la contaminación con el plásmido circular residual (Figura 2).
- Cargar la mezcla de reacción de digestión en el mega-pocillo de una bajo punto de fusión en gel de agarosa semi-preparativa (0,75%, w: v), así como una parte alícuota (5 l) de la mezcla de reacción en el adyacente así como un reportero.
- Ejecutar electroforesis de ADN (5 V / cm entre los electrodos, 4 ᵒC) y separar el gel de agarosa correspondiente a la mega bien y que lo conserve en 4 ᵒC en 1x TAE.
- Manchar el carril con la escala de peso molecular y el reportero. Visualizar las bandas bajo luz UV. Nick la posición en los lugares vector linealizado.
Nota: Como la calidad del vector linealizado purificado es un critifactor de cal para la recombinación exitosa y montaje en la levadura, para evitar mancharse gel de electroforesis de ADN semi-preparativa. El uso de colorantes y la exposición UV para la extracción de gel puede afectar a la estabilidad del vector de ADN, comprometer la eficacia de recombinación in vivo en. Como alternativa a los colorantes EtBr tóxicos, colorantes SYBR Gel Red y se utilizan comúnmente para la tinción de gel. - En la ausencia de luz UV, identificar el vector linealizado en el fragmento de mega-bien utilizando la guía de las muescas en el carril de reportero manchado de modo que pueda ser aislado.
- Extraer el vector linealizado a partir de agarosa y se purifica con un kit de extracción de gel comercial de acuerdo con el protocolo del fabricante.
Nota: Use vectores lanzadera episomal de alta copia con marcadores de antibióticos y auxotrofía: En este ejemplo se empleó el uracilo independiente y ampicilina resistencia pJRoC30 vector, bajo el control del promotor de levadura GAL1.
- Preparar un IM equimolarxture de los fragmentos de PCR y se mezcla con el vector linealizado en una proporción de 2: 1, con no menos de 100 ng de plásmido linealizado (prueba de diferentes proporciones de biblioteca equimolar / vector abierta para lograr buenos rendimientos de transformación).
- Medir la absorbancia de los fragmentos de PCR y el vector linealizado en 260 nm y 280 nm para determinar su concentración y pureza.
- Transformar células competentes de levadura con la mezcla de ADN utilizando un kit de transformación de levadura comercial (véase la Tabla de suministros) de acuerdo con las instrucciones del fabricante.
- A continuación, utilice una proteasa deficiente y URA3 - S. dependientes cerevisiae cepa, BJ5465. Transformar las células con el vector parental circularizado como un estándar interno durante la selección (véase a continuación). Además, compruebe el fondo mediante la transformación del vector linealizado en ausencia de fragmentos de PCR.
Nota: En caso de detectar niveles iniciales bajos de secreción, use S.cerevisiae cepas deficientes en proteasa como BJ5465 de favorecer la acumulación de proteína activa en sobrenadantes de cultivo. Si la enzima diana se somete a hiperglicosilación, el uso de cepas deficiente en glicosilación (por ejemplo, Δ kre2 que sólo es capaz de fijar oligómeros de manosa más pequeñas) podría ser una opción adecuada.
- A continuación, utilice una proteasa deficiente y URA3 - S. dependientes cerevisiae cepa, BJ5465. Transformar las células con el vector parental circularizado como un estándar interno durante la selección (véase a continuación). Además, compruebe el fondo mediante la transformación del vector linealizado en ausencia de fragmentos de PCR.
- Placa de las células transformadas en placas de deserción SC y los incuban a 30 ° C durante tres días. Placa (en placas de deserción SC suplementado con uracilo) URA3 - S. células cerevisiae que carecen de plásmido como control negativo para la detección (ver abajo).
2. Alta-Throughput Screening de ensayo (Figura 3)
- Llenar un número apropiado de placas de 96 pocillos estériles (23 placas para analizar una biblioteca de 2.000 clones) con 50 l de medio mínimo por pocillo con la ayuda de un robot de pipeteado.
- Recoger las colonias individuales de la SC-abandono placas y transferirlos a las placas de 96 pocillos.
- En cada placa, inocular número de columna 6 con el tipo parental como patrón interno y así H1 con URA3 - S. células cerevisiae (en medio SC suplementado con uracilo) sin plásmido como control negativo.
Nota: Bueno H1 está llena específicamente con los medios de deserción suplementado con uracilo. Un medio bien contiene en blanco sin células también puede prepararse como un control de esterilidad adicional.
- En cada placa, inocular número de columna 6 con el tipo parental como patrón interno y así H1 con URA3 - S. células cerevisiae (en medio SC suplementado con uracilo) sin plásmido como control negativo.
- Se cubren las placas con sus tapas y envolverlos en Parafilm. Incubar las placas durante 48 horas a 30 ° C, 225 rpm y 80% de humedad relativa en un agitador húmedo.
- Retire el Parafilm, añadir 160 l de medio de expresión a cada pocillo con la ayuda del robot de pipeteado, vuelva a sellar las placas y se incuban durante 24 horas.
Nota: medio mínimo y medio de expresión se preparan como informado en otras partes 19. los niveles de secreción pueden variar en función del gen bajo estudio y, en consecuencia, tél incubación veces deben ser optimizados en cada caso para sincronizar el crecimiento de las células en todos los pocillos. - Centrifugar las placas (placas maestras) a 2800 x g durante 10 min a 4 ° C.
- Transferencia de 20 l del sobrenadante de los pocillos de la placa principal a la placa de réplica usando un manejo multiestación robótico líquido.
Nota: Para favorecer la secreción de enzimas, es aconsejable sustituir el péptido señal nativa de la proteína diana mediante péptidos señal comúnmente utilizados para la expresión heteróloga en la levadura (por ejemplo, el factor α prepro-líder, el líder de la K 1 Killer toxina de S. cerevisiae, o incluso versiones quiméricas de ambos péptidos 13). Alternativamente, el péptido señal nativo puede ser desarrollado exclusivamente para la secreción en la levadura. - Añadir 20 l de p -methoxybenzylalcohol 2 mM en tampón de fosfato de sodio 100 mM pH 6,0 con la ayuda del robot de pipeteado. Agitar las placas brevemente con un 96-nosll mezclador de placas y se incuba durante 30 min a TA.
- Con el robot de pipeteado, añadir 160 l de reactivo FOX a cada placa de réplica y agitar brevemente con el mezclador (concentración final de la mezcla de FOX en el pozo: 100 M xilenol naranja, 250 M Fe (NH4) 2 (SO4) 2 y 25 mm de H 2 SO 4).
- Añadir varios aditivos al reactivo para mejorar la sensibilidad, tales como co-disolventes orgánicos (DMSO, etanol, metanol) o sorbitol 17. Aquí, amplificar la respuesta mediante la adición de sorbitol a una concentración final de 100 mM (Figura 4).
- Leer las placas (modo de punto final, t 0) a 560 nm en un lector de placas.
- Incubar las placas a temperatura ambiente hasta que se desarrolle el color y medir la absorción de nuevo (t 1).
- Calcular la actividad relativa de la diferencia entre el valor Abs después de la incubación y el de la medición inicial normalizado a la pareTipo ntal para cada placa (Dt 1 - t 0).
- Someter los mejores éxitos mutantes a dos re-exámenes consecutivos para descartar falsos positivos.
Nota: Por lo general, re-proyecciones incluyen el aislamiento del plásmido de la levadura, la amplificación y purificación en Escherichia coli, seguido de la transformación de células de levadura fresca con el plásmido 19. Cada clon seleccionado se vuelve a proyectará en pentaplicate.
Resultados
AAO de P. Eryngii es un flavooxidase extracelular que suministra peroxidasas fúngicas con H 2 O 2 a empezar a atacar la lignina. Dos segmentos de AAO se sometieron a la evolución dirigida centrado-morphing con el fin de mejorar su actividad y su expresión en S. 19 cerevisiae. Independientemente de las enzimas extranjeros albergadas por S. cerevisiae, el problema más crítico en la construcción de bibliotecas de mutantes en la levadura se refiere a la ingeniería de las regiones superpuestas específicas para favorecer el empalme entre los fragmentos y su clonación en el vector linealizado. En el ejemplo actual, para cada reacción de PCR, todos los fragmentos tenían salientes de aproximadamente 50 pares de bases para promover en el empalme vivo en la levadura. El número de eventos de recombinación depende de la cantidad de segmentos a ser montado y se clonó con el vector linealizado (es decir, dos eventos de cruce se llevaron a cabo entre eltres segmentos de PCR -los dos segmentos mutagénicos que flanquean el segmentados no mutagenizado más dos cruces adicionales con el vector linealizado; Figura 1). De acuerdo con nuestra experiencia, la superposición de secuencias de más de 50 pb disminuyen la probabilidad de recombinación interna, mientras que no mejoran la eficiencia de transformación.
Cargas mutacionales se ajustaron mediante el muestreo de bibliotecas de mutantes con diferentes paisajes, calcular el número de clones con <10% de la actividad de la enzima parental, y aún más control de ellos mediante secuenciación de una muestra aleatoria de variantes activas y no activas (Figura 5A). Para la determinación del coeficiente de varianza S. cerevisiae células se transformaron con la AAO los padres y chapada en SC-abandono placas. Las colonias individuales se seleccionaron y se inocularon en una placa de pocillos 96 y se evaluó la actividad de los clones a partir de preparaciones frescas. muestra mutagénico2 (Taq / MnCl2 0,05 mM) fue elegido como el punto de partida para la construcción de bibliotecas y cribado.
A medida que la actividad biológica de AAO aumenta la concentración de H 2 O 2 en el medio de reacción, se realizaron búsquedas de un ensayo sensible y preciso para cuantificar los cambios de menor importancia en H 2 O 2. FOX es un método químico basado en la reacción de Fenton 20, en el que la oxidación por H 2 O 2 unidades de la reacción de Fe 3+ con naranja de xilenol para formar un complejo azul-púrpura (o -cresolsulfone-ftaleína 3 ', 3' '- bis (metilimino) diacetato ε 560 = 1,5 x 10 4 M -1 cm -1). La etapa de oxidación ferroso se amplificó mediante la adición de sorbitol para mejorar la sensibilidad del ensayo, el aumento de la propagación de radicales con un ε aparente 560 = 2,25 x 10 5 M -1 cm -1 (Fifigura 4).
El límite de detección de este ensayo (en el rango mu M) se calculó por el método de determinación en blanco en una placa de 96 pocillos con las normas, por triplicado, (0, 0,5, 1, 1,5, 2, 2,5, 3 y 4 mM H 2 O 2 ) y el uso de varios sobrenadantes de S. cerevisiae que carecen de la URA3 - plásmido (Figura 5B). El ensayo fue lineal en presencia de sorbitol (hasta 8 M de H 2 O 2), y aunque la linealidad fue más persistente en ausencia de este azúcar (al menos hasta 30 M de H 2 O 2) la respuesta fue más débil (por ejemplo, en 6 M de H 2 O 2, una mejora de 4 veces se obtuvo en presencia de sorbitol -Profunda morado de una absorbancia de 0,24 en su ausencia -dark naranja (Figura 5B)). La relación entre el ABS y la concentración AAO se evaluó con cantidades crecientes de correose observó nzyme (a partir de sobrenadantes de levadura) y una respuesta lineal; R 2 = 0,997 (Figura 5C).
Es notable que la señal de FOX fue estable durante varias horas sin ninguna interferencia aparente por los diferentes elementos en el caldo de cultivo. La sensibilidad estimada de FOX era ~ 0,4 M de H 2 O 2 producido por la AAO en el sobrenadante en presencia de sorbitol, y ~ 2 M en su ausencia.
Una biblioteca de mutantes de 2.000 clones se construyó y se exploró con este ensayo. Varios mutantes AAO se identificaron con la mejora notablemente la secreción y la actividad contra el alcohol p metoxibencilo (Figura 5D) 19.

Figura 1.. MORPHING Protocolo para la AAO Evolución Dos regiones diferentes de AAO fueron objeto de mutagénesis al azar y recombinación: M1 (azul, 590 pb) que incluye el péptido señal (SP); M2 (amarillo, 528 pb). La región de alta frecuencia (gris, 844 pb) se amplificó con polimerasas de alta fidelidad. Regiones mutagénicos fueron asignadas en la estructura cristalina de la AAO (AP ID: 3FIM). Por favor, haga clic aquí para ver una versión más grande de esta figura.
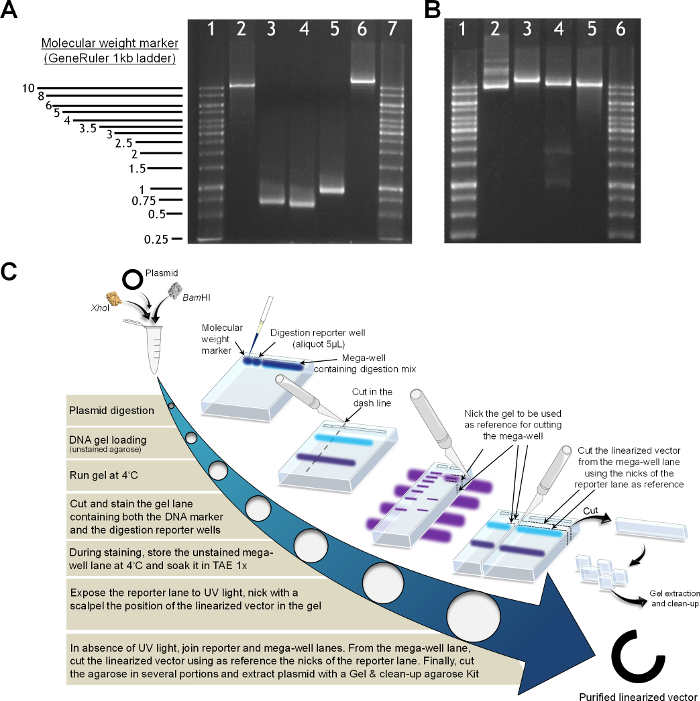
Figura 2. Preparación de los productos de PCR y el vector linealizado (A) en gel de agarosa analítico (1% w: v). Que contiene un marcador de peso molecular (escalera de 1 kb) en los carriles 1 y 7; el vector linealizado con Bam HI y Xho I, el carril 2; PCR segmento M1, calle 3; PCR segmento M2, carril 4; PCR HF segmento, carril 5; v en elivo vuelto a montar vector linealizado con NheI (que contiene el gen completo AAO con las regiones MI, HF y H-II), carril 6. (B) linealización del vector, carriles 1 y 6 estándares moleculares, escalera de 1 Kb; plásmido miniprep, carril 2; plásmido linealizado con Nhe I, calle 3; plásmido linealizado con Bam HI y Xho I, carril 4; plásmido linealizado obtenido por extracción de gel y la limpieza después de la digestión, 5. Protocolo de carril (C) para la purificación del plásmido. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3. De Alto Rendimiento protocolo para la detección. Descripción general del proceso. Haga clic aquí para conocer el vers más grandesión de esta figura.

Figura 4. El método de FOX. Hongos de podredumbre blanca atacar a la pared celular de la madera a través de una reacción de Fenton que produce radical hidroxilo OH •. Las parejas método FOX esta reacción de xilenol naranja (XO), y la absorbancia del complejo de la XO-Fe 3+ se mide a 560 nm. La oxidación ferroso se amplifica por la adición de sorbitol a la mezcla de reactivos. Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 5. mutagénicos paisajes morphing Bibliotecas Empleo de los distintos propenso a errores condiciones de PCR y Validación del Ensayo de selección. (A > ng) MORPHING paisajes. línea horizontal continua muestra la actividad de tipo parental en el ensayo, mientras que las líneas discontinuas indican el coeficiente de variación del ensayo. Los porcentajes indican el número de clones con menos de 10% de la actividad de la enzima parental. Las actividades se representan en orden descendente. (B) Se evaluó el límite de detección FOX con concentraciones crecientes de H 2 O 2 en presencia (círculos negros) y ausencia (cuadrados blancos) de sorbitol. Correlación (C) lineal entre la concentración de AAO (sobrenadantes transformantes) y 560 nm Abs. Cada punto corresponde a la media de 8 experimentos e incluye la desviación estándar. (D) Mutant paisaje biblioteca. Las variantes seleccionadas (cuadrados sombreada) se volvieron a seleccionar como se informó en otro lugar 19. La línea continua muestra la actividad de AAO tipo parental.> Haga clic aquí para ver una versión más grande de esta figura.
Discusión
En este artículo, hemos resumido la mayor parte de los consejos y trucos empleados en nuestro laboratorio para diseñar enzimas por evolución dirigida en S. cerevisiae (usando AAO como ejemplo) para que puedan ser adaptados para su uso con muchos otros sistemas de enzimas eucariotas simplemente siguiendo el enfoque común se describe aquí.
En cuanto a la creación de la biblioteca, el morphing es un método rápido de un solo recipiente para introducir y recombinar las mutaciones al azar en pequeños tramos de proteínas, dejando el resto de regiones de la proteína alterada 16. Las bibliotecas con varias cargas de mutaciones se pueden preparar fácilmente y recombinados in vivo, junto con el plásmido linealizado, para generar un vector de replicación autónoma completa. Es fundamental que las secuencias superpuestas flanquean cada tramo para permitir que los fragmentos del gen completo al volver a montar a través de la recombinación in vivo, evitando reacciones adicionales de PCR y en los pasos de ligación in vitro. En este pROTOCOLO, la frecuencia de eventos de cruce entre los fragmentos de PCR se puede incrementar mediante la reducción del tamaño de las zonas de solapamiento, aunque esto puede comprometer la eficiencia de transformación. Independientemente de las polimerasas de ADN utilizados para la PCR mutagénica, las cargas de mutaciones se pueden ajustar mediante la construcción y el análisis de pequeños paisajes biblioteca de mutantes (Figura 5A) anteriormente. Si se utiliza el Kit GeneMorph II, todavía es aconsejable seguir este enfoque ya que en vivo de recombinación de ADN se puede modificar notablemente las cargas mutacionales estimado por el fabricante. En términos generales, paisajes mutantes en las que 35 - 50% de los clones totales examinados tienen menos de 10% de la actividad de los padres son apropiados para las campañas de evolución dirigida, aunque este número varía en función de la proteína diana y su actividad. Por lo general, el análisis de bibliotecas mutantes paisajes se verificó adicionalmente por secuenciación del ADN de una muestra aleatoria de mutantes. En el ejemplo actual, la TaqADN polimerasa se utilizó debido a su alta tasa de error, que está vinculada a la falta de 3 '→ 5'proof de lectura de actividad de exonucleasa. Las cargas de mutaciones en las bibliotecas de Taq fueron modificados por la adición de diferentes concentraciones de MnCl 2, pero el uso de dNTPs desequilibrados y / o la reducción de las concentraciones de la plantilla de genes también son opciones adecuadas. limitaciones inherentes de MORPHING llegado a partir del número de segmentos a recombinar. De acuerdo con nuestra experiencia, hasta cuatro bloques de proteínas (cinco eventos de cruce contando las áreas de recombinación con el vector linealizado) puede ser empalmado con buenos rendimientos de transformación (~ 10 5 clones por reacción de transformación). Este método puede ser fácilmente modificado para realizado mutagénesis múltiple saturación de sitio (por ejemplo, utilizando cebadores degenerados NDT o la creación de degeneración durante 22 codones únicos) para explorar varias posiciones simultáneamente al tiempo que reduce significativamente los esfuerzos de detección 21,22.
El protocolo de selección directa "a ciegas" para AAO es extremadamente sensible y fiable (basado en la detección directa de H 2 O 2 independientemente del sustrato utilizado por la enzima), lo que representa un ensayo complementario a otro bien establecido indirecta protocolos para detectar peróxidos (en su mayoría peroxidasas acoplamiento con sustratos colorimétricos). De hecho, el ensayo FOX se ha empleado habitualmente para medir H 2 O 2 en fluidos biológicos, y ahora se puede traducir fácilmente en los protocolos de evolucionar AAO y cualquier otra H 2 O 2 productores de enzimas (por ejemplo, glucosa oxidasas, deshidrogenasas celobiosa, glioxal oxidasas, oxidasas metanol), en particular para la actividad en sustratos no naturales donde las respuestas son de otra manera difícil de detectar.
S. cerevisiae es el anfitrión más adecuada para la evolución dirigida de genes eucariotas ya que ofrece alta eficiencia de transformación (hasta 1 x 106 transformantes / mg de ADN), que realiza complejo de procesamiento y las modificaciones después de la traducción (incluyendo N- y procesamiento C-terminal, y glicosilación) y exporta proteínas extrañas en el caldo de cultivo a través de una vía secretora. Además, las herramientas de biología molecular bien establecidos están disponibles para trabajar con esta levadura, incluyendo vectores lanzadera episomal uni o bi-direccional (no integradora) bajo el control de promotores de diferentes potencias. Por último, pero no menos importante, su alta frecuencia de recombinación de ADN homóloga ha permitido una serie de métodos para ser desarrollado para obtener diversidad de ADN que actualmente se utiliza para evolucionar proteínas individuales, así como vías enzimáticas más complejas 8, 12, 13, 23. el vivo de reparación de vacío en y el dispositivo de corrección de pruebas de esta levadura se pueden también emplearse para crear quimeras cuando recombinación de genes diferentes (con aprox. 60% de identidad de secuencia de ADN), así como para mezclar mejores descendiente / mutaciones de un directed campaña de la evolución, o para reunir in vitro e in vivo en métodos de recombinación de una ronda de evolución, enriqueciendo de esta manera las bibliotecas mutantes en cuanto a la capacidad de plegado y la función.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
This work was supported by the European Commission project Indox-FP7-KBBE-2013-7-613549; a Cost-Action CM1303-Systems Biocatalysis; and the National Projects Dewry [BIO201343407-R] and Cambios [RTC-2014-1777-3].
Materiales
| Name | Company | Catalog Number | Comments |
| 1. Culture media | |||
| Ampicillin sodium salt | Sigma-Aldrich | A0166 | CAS Nº 69-52-3 M.W. 371.39 |
| Bacto Agar | Difco | 214010 | |
| Cloramphenicol | Sigma-Aldrich | C0378 | CAS Nº 56-75-7 M.W. 323.13 |
| D-(+)-Galactose | Sigma-Aldrich | G0750 | CAS Nº 59-23-4 M.W. 180.16 |
| D-(+)-Glucose | Sigma-Aldrich | G5767 | CAS Nº 50-99-7 M.W. 180.16 |
| D-(+)-Raffinose pentahydrate | Sigma-Aldrich | 83400 | CAS Nº 17629-30-0 M.W. 594.51 |
| Peptone | Difco | 211677 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P0662 | CAS Nº 7778-77-0 M.W. 136.09 |
| Uracil | Sigma Aldrich | U1128 | |
| Yeast Extract | Difco | 212750 | |
| Yeast Nitrogen Base without Amino Acids | Difco | 291940 | |
| Yeast Synthetic Drop-out Medium Supplements without uracil | Sigma-Aldrich | Y1501 | |
| Name | Company | Catalog Number | Comments |
| 2. PCR Reactions | |||
| dNTP Mix | Agilent genomics | 200415-51 | 25 mM each |
| iProof High-Fidelity DNA polymerase | Bio-rad | 172-5301 | |
| Manganese(II) chloride tetrahydrate | Sigma-Aldrich | M8054 | CAS Nº 13446-34-9 M.W. 197.91 |
| Taq DNA Polymerase | Sigma-Aldrich | D4545 | For error prone PCR |
| Name | Company | Catalog Number | Comments |
| 3. Plasmid linearization | |||
| BamHI restriction enzyme | New England Biolabs | R0136S | |
| Bovine Serum Albumin | New England Biolabs | B9001S | |
| XhoI restriction enzyme | New England Biolabs | R0146S | |
| Not I restriction enzyme | New England Biolabs | R0189S | |
| Gel Red | Biotium | 41003 | For staining DNA |
| Name | Company | Catalog Number | Comments |
| 4. FOX assays | |||
| Ammonium iron(II) sulfate hexahydrate | Sigma-Aldrich | F3754 | CAS Nº 7783-85-9 M.W. 392.14 |
| Anysil Alcohol | Sigma Aldrich | W209902 | CAS Nº 105-13-5 M.W. 138.16 |
| D-Sorbitol | Sigma-Aldrich | S1876 | CAS Nº 50-70-4 M.W. 182.17 |
| Hydrogen peroxide 30% | Merck Millipore | 1072090250 | FOX standard curve |
| Xylenol Orange disodium salt | Sigma-Aldrich | 52097 | CAS Nº 1611-35-4 M.W. 716.62 |
| Name | Company | Catalog Number | Comments |
| 5. Agarose gel stuff | |||
| Agarose | Norgen | 28035 | CAS Nº 9012-36-6 |
| Gel Red | Biotium | 41003 | DNA analysis dye |
| GeneRuler 1kb Ladder | Thermo Scientific | SM0311 | DNA M.W. standard |
| Loading Dye 6x | Thermo Scientific | R0611 | |
| Low-melting temperature agarose | Bio-rad | 161-3112 | CAS Nº 39346-81-1 |
| Name | Company | Catalog Number | Comments |
| 6. Kits and cells | |||
| S. cerevisiae strain BJ5465 | LGC Promochem | ATTC 208289 | Protease deficient strain with genotype: MATα ura3-52 trp1 leu2-delta1 his3-delta200 pep4::HIS3 prb1-delta1.6R can1 GAL |
| E. coli XL2-Blue competent cells | Agilent genomics | 200150 | For plasmid purification and amplification |
| NucleoSpin Gel and PCR Clean-up Kit | Macherey-Nagel | 740,609,250 | DNA gel extraction |
| NucleoSpin Plasmid Kit | Macherey-Nagel | 740,588,250 | Column miniprep Kit |
| Yeast Transformation Kit | Sigma-Aldrich | YEAST1-1KT | Included DNA carrier (Salmon testes) |
| Zymoprep yeast plasmid miniprep I | Zymo research | D2001 | Plasmid extraction from yeast |
| Name | Company | Catalog Number | Comments |
| 7. Plates | |||
| 96-well plates | Greiner Bio-One | 655101 | Clear, non-sterile, polystyrene (for activity measurements) |
| 96-well plates | Greiner Bio-One | 655161 | Clear, sterile, polystyrene (for microfermentations) |
| 96-well plate lid | Greiner Bio-One | 656171 | Clear, sterile, polystyrene (for microfermentations) |
Referencias
- Jäckel, C., Hilvert, D. Biocatalysts by evolution. Curr. Opin. Biotechnol. 21 (6), 753-759 (2010).
- Bornscheuer, U. T. Engineering the third wave of biocatalysis. Nature. 485 (7397), 185-194 (2012).
- Renata, H., Wang, Z. W., Arnold, F. H. Expanding the enzyme universe: accessing non-natural reactions by mechanism-guided directed evolution. Angew. Chem. Int. Ed. 54 (11), 3351-3367 (2015).
- Cobb, R. E., Chao, R., Zhao, H. Directed evolution: past, present and future. AIChE J. 59 (5), 1432-1440 (2013).
- Abatemarco, J., Hill, A., Alper, H. S. Expanding the metabolic engineering toolbox with directed evolution. Biotechnol. J. 8 (12), 1397-1410 (2013).
- Pourmir, A., Johannes, T. W. Directed evolution: selection of the host organism. Comput Struct Biotechnol J. 2 (3), e201209012(2012).
- Krivoruchko, A., Siewers, V., Nielsen, J. Opportunities for yeast metabolic engineering: lessons from synthetic biology. Biotechnol J. 6 (3), 262-276 (2011).
- Gonzalez-Perez, D., Garcia-Ruiz, E., Alcalde, M. Saccharomyces cerevisiae in directed evolution: an efficient tool to improve enzymes. Bioeng Bugs. 3, 172-177 (2012).
- Alcalde, M. Mutagenesis protocols in Saccharomyces cerevisiae by In Vivo Overlap Extension. Methods Mol. Biol. 634, 3-14 (2010).
- Bulter, T., Alcalde, M. Preparing libraries in Saccharomyces cerevisiae. Methods. Mol. Biol. 231, 17-22 (2003).
- Ostrov, N., Wingler, L. M., Cornish, W. Gene assembly and combinatorial libraries in S. cerevisiae via reiterative recombination. Methods. Mol. Biol. 978, 187-203 (2013).
- Shao, Z., Zhao, H., Zhao, H. DNA assembler, an in vivo genetic method for rapid construction of biochemical pathways. Nucleic Acids Res. 37 (2), e16(2009).
- Alcalde, M. Engineering the ligninolytic enzyme consortium. Trends Biotechnol. 33 (3), 155-162 (2015).
- Garcia-Ruiz, E. Directed evolution of ligninolytic oxidoreductases: from functional expression to stabilization and beyond. Cascade Biocatalysis: integrating stereoselective and environmentally friendly reactions. , Wiley-VCH. 1-22 (2014).
- Hernandez-Ortega, A., Ferreira, P., Martinez, A. T. Fungal aryl-alcohol oxidase: a peroxide-producing flavoenzyme involved in lignin degradation. Appl. Microbiol. Biotechnol. 93 (4), 1395-1410 (2012).
- Gonzalez-Perez, D., Molina-Espeja, P., Garcia-Ruiz, E., Alcalde, M. Mutagenic organized recombination process by homologous in vivo grouping (MORPHING) for directed enzyme evolution. PLoS One. 9, e90919(2014).
- Rhee, S. G., Chang, T., Jeong, W., Kang, D. Methods for Detection and Measurement of Hydrogen Peroxide Inside and Outside of Cells. Mol. Cells. 29 (6), 539-549 (2010).
- Sebestova, E., Bendl, J., Brezovsky, J., Damborsky, J. Computational tools for designing smart libraries. Methods. Mol. Biol. 1179, 291-314 (2014).
- Viña-Gonzalez, J., Gonzalez-Perez, D., Ferreira, P., Martinez, A. T., Alcalde, M. Focused directed evolution of aryl-alcohol oxidase in yeast using chimeric signal peptides. Appl. Environ. Microbiol. , (2015).
- Gay, C., Collins, J., Gebicki, J. M. Hydroperoxide Assay with the Ferric-Xylenol orange Complex. Anal. Biochem. 273 (2), 149-155 (1999).
- Reetz, M. T. Biocatalysis in organic chemistry and biotechnology: Past, present, and future. J. Am. Chem. Soc. 135 (34), 12480-12496 (2013).
- Mate, D. M., Gonzalez-Perez, D., Mateljak, I., Gomez de Santos, P., Vicente, A. I., Alcalde, M. The pocket manual of directed evolution: Tips and tricks. Biotechnology of Microbial Enzymes: Production, Biocatalysis and Industrial Applications. , Elsevier. Forthcoming Forthcoming.
- Chao, R., Yuan, Y., Zhao, H. Recent advances in DNA assembly technologies. FEMS Yeast Res. 15, 1-9 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados