Method Article
Réalisé Evolution Méthode
Dans cet article
Résumé
We present a detailed protocol to construct and screen mutant libraries for directed evolution campaigns in Saccharomyces cerevisiae.
Résumé
L' évolution dirigée dans Saccharomyces cerevisiae offre de nombreux avantages attrayants lors de la conception d' enzymes pour des applications biotechnologiques, un processus qui implique la construction, le clonage et l' expression de bibliothèques de mutants, couplé à haute fréquence recombinaison homologue de l' ADN in vivo. Ici, nous présentons un protocole pour créer et bibliothèques de mutants d'écran dans la levure basé sur l'exemple d'un champignon aryl-alcool oxydase (AAO) pour renforcer son activité totale. Deux segments de protéines ont été soumis à l' évolution concentrée dirigée par mutagenèse aléatoire et in vivo la recombinaison de l' ADN. Surplombs de ~ 50 pb flanquant chaque segment a permis au réassemblage correcte du gène AAO-fusion dans un vecteur linéarisé donnant naissance à un plasmide à réplication autonome complète. Bibliothèques de mutants fonctionnels enrichis avec des variantes AAO ont été criblés dans S. Les surnageants cerevisiae avec un dosage à haut débit sensible à la base de la réaction de Fenton. Le processus général deconstruction d'une banque de S. cerevisiae décrit ici peut être facilement appliquée à évoluer beaucoup d' autres gènes eucaryotes, en évitant des réactions PCR supplémentaires, in vitro recombinaison de l' ADN et ligature étapes.
Introduction
Évolution moléculaire dirigée est une méthode robuste, rapide et fiable pour concevoir des enzymes 1, 2. Par séries itératives de mutation aléatoire, recombinaison et de criblage, des versions améliorées des enzymes peuvent être générés qui agissent sur de nouveaux substrats, dans de nouvelles réactions, dans la non-naturelle environnements, ou même pour aider la cellule à atteindre de nouveaux objectifs métaboliques 3-5. Parmi les hôtes utilisés dans l' évolution dirigée, la levure Saccharomyces cerevisiae du brasseur propose un répertoire de solutions pour l'expression fonctionnelle des protéines eucaryotes complexes qui ne sont pas autrement disponibles dans homologues procaryotes 6,7.
Utilisé de façon exhaustive dans les études de biologie cellulaire, ce petit modèle eucaryote présente de nombreux avantages en termes de modifications post-traductionnelles, la facilité de manipulation et de transformation d' efficacité, sont tous les traits qui importants à l' ingénieur enzymes par évolution dirigée 8. En outre, la haute fréquencede recombinaison homologue de l' ADN dans S. cerevisiae couplé à son appareil de relecture efficace ouvre un large éventail de possibilités de création de la bibliothèque et de l' assemblage de gènes in vivo, de favoriser l'évolution des différents systèmes d' enzymes uniques aux voies artificielles complexes 9-12. Notre laboratoire a passé la dernière décennie la conception d' outils et de stratégies pour l'évolution moléculaire des différentes ligninases dans la levure (des oxydoréductases impliquées dans la dégradation de la lignine lors de la pourriture du bois naturel) 13-14. Dans cette communication, nous présentons un protocole détaillé pour préparer et bibliothèques de mutants d'écran dans S. cerevisiae pour un modèle flavooxidase, -aryl-alcool oxydase (AAO 15) -, qui peut être facilement traduits à de nombreuses autres enzymes. Le protocole implique une méthode d'évolution ciblée dirigée (MORPHING: Mutagène organisée par recombinaison homologue Process in vivo Regroupement) assistée par l'appareil de cellule de levure 16,da test de criblage très sensible basé sur la réaction de Fenton, afin de détecter l' activité AAO sécrété dans le bouillon de culture 17.
Protocole
1. Mutant Library Construction
- Choisissez les régions à soumettre à MORPHING avec l'aide d'algorithmes de calcul sur la base des modèles disponibles sur la structure cristalline ou homologie 18.
- Ici, cibler deux régions de AAO de Pleurote du panicaut pour la mutagenèse aléatoire et la recombinaison (Met [α1] -Val109, Phe392-Gln566), tout en amplifiant le reste du gène (844 pb) par PCR haute fidélité (Figure 1).
Note: Plusieurs segments peuvent être étudiés par MORPHING de manière indépendante ou combinée 16.
- Ici, cibler deux régions de AAO de Pleurote du panicaut pour la mutagenèse aléatoire et la recombinaison (Met [α1] -Val109, Phe392-Gln566), tout en amplifiant le reste du gène (844 pb) par PCR haute fidélité (Figure 1).
- Amplifier les zones ciblées par PCR mutagène. Créer des zones de chevauchement entre des segments (~ 50 pb chacun) en superposant des réactions PCR des régions définies.
- Préparer une PCR mutagene des segments ciblés dans un volume final de 50 ul contenant une matrice d'ADN (0,92 ng / ul), 90 nM de détection de l'oligo (pour le segment de RMLN IM et AAO-BP pour le segment M-II), 90 nM d'unntisense amorce (AAO-92C pour le segment IM et RMLC pour le segment M-II), mM dNTP 0,3 (0,075 mM chacun), 3% (v / v) de diméthylsulfoxyde (DMSO), 1,5 mM de MgCl2, 0,05 mM de MnCl2 et 0,05 U / pl de Taq ADN polymerase. Amorces séquences sont décrites en détail dans la figure 1.
- Utilisez le programme de PCR suivant: 95 ° C pendant 2 min (1 cycle); 95 ° C pendant 45 s, 50 ° C pendant 45 s, 74 ° C pendant 45 s (28 cycles); et 74 ° C pendant 10 min (1 cycle).
- Amplifier les régions non-mutagène avec ultra-haute polymérase fidélité et inclure les zones correspondantes se chevauchent les segments mutagènes et / ou des surplombs de vecteur linéarisé.
- Préparer des mélanges réactionnels dans un volume final de 50 ul contenant: modèle d'ADN (0,2 ng / pl), 250 nM d'oligonucleotide sens HFF, 250 nM d'oligo antisens HFR, 0,8 mM de dNTP (0,2 mM chacun), 3% (v / v) de sulfoxyde de diméthyle (DMSO) et 0,02 U / pl d' ADN polymerase iProof. Amorces séquences sont détaillées dans Figure 1.
- Utilisez le programme de PCR suivant: 98 ° C pendant 30 secondes (1 cycle); 98 ° C pendant 10 s, 55 ° C pendant 25 s, 72 ° C pendant 45 s (28 cycles); et 72 ° C pendant 10 min (1 cycle).
Remarque: Avec les conditions décrites en 1.2 et 1.3 chevauchements de 43 pb (région plasmidique-M1); 46 pb (M1 region-HF); 47 pb (HF région M2 région) et 61 pb (M2 plasmidique de région) sont conçus (Figure 1) pour favoriser dans l' épissage in vivo dans la levure. - On purifie tous les fragments de PCR (mutagènes et non mutagènes) avec un kit d'extraction de gel du commerce, selon le protocole du fabricant.
- Linéariser le vecteur de telle sorte que les régions d'environ 50 pb adjacentes sont créées, qui sont homologues aux extrémités 5 'et 3' extrémités du gène cible.
- Préparer un mélange réactionnel contenant de linéarisation 2 pg d' ADN, 7,5 U Bam HI Xho I 7,5 U, 20 ug de BSA et 2 pi de tampon de Bam HI 10x dans un volume final de 20 ul.
- Incuber le mélange réactionnel à 37 ° C pendant 2 h et 40 min. Ensuite, procéder à l'inactivation à 80 ° C pendant 20 min.
- Purifier le vecteur linéarisé par extraction sur gel d' agarose afin d' éviter toute contamination avec le plasmide circulaire résiduel (figure 2).
- Charger le mélange de réaction de digestion dans le méga-puits d'un gel semi-préparative bas point de fusion d'agarose (0,75%, w: v), ainsi que d'une partie aliquote (5 pi) du mélange de réaction dans le puits adjacent en tant que journaliste.
- Run ADN électrophorèse (5 V / cm entre les électrodes, 4 ᵒC) et séparer le gel d'agarose correspondant à la méga-puits et le stocker à 4 ᵒC dans 1x TAE.
- Colorer la voie avec l'échelle de poids moléculaire et le journaliste. Visualisez les bandes sous lumière UV. Nick la position où les places de vecteurs linéarisés.
Remarque: Comme la qualité du vecteur linéarisé purifié est un critifacteur cal pour la recombinaison avec succès et de l'assemblage dans la levure, éviter la coloration de gel d'électrophorèse d'ADN semi-préparative. L'utilisation de colorants et de l' exposition aux UV pour l' extraction de gel peut affecter la stabilité du vecteur d'ADN, ce qui compromet l'efficacité in vivo de recombinaison. Comme alternative aux colorants de EtBr toxiques gel rouge et des colorants SYBR sont couramment utilisés pour la coloration du gel. - En l'absence de lumière UV, d'identifier le vecteur linéarisé dans le fragment méga-puits en utilisant la direction des entailles dans la voie indicatrice colorée de manière à pouvoir être isolée.
- Extraire le vecteur linéarisé à partir d'agarose et on purifie avec un kit d'extraction de gel du commerce, selon le protocole du fabricant.
Remarque: Utilisez haute copie vecteurs navettes épisomique avec des marqueurs antibiotiques et auxotrophie: Dans cet exemple , nous avons utilisé l'uracile indépendant et résistance à l' ampicilline pJRoC30 vecteur, sous le contrôle du promoteur de levure GAL1.
- Préparer une mi équimolairexture des fragments de PCR et le mélanger avec le vecteur linéarisé à un ratio de 2: 1, avec pas moins de 100 ng du plasmide linéarisé (essai différents rapports de bibliothèque équimolaire / vecteur ouvert à obtenir de bons rendements de transformation).
- Mesurer l'absorbance des fragments de PCR et le vecteur linéarisé à 260 nm et 280 nm afin de déterminer leur concentration et leur pureté.
- Transformer des cellules compétentes de levure avec le mélange d'ADN en utilisant un kit de transformation de levure commerciale (voir le tableau pour les fournitures) selon les instructions du fabricant.
- Ici, utiliser une protéase déficiente et URA3 - dépendante S. cerevisiae, BJ5465. Transformer les cellules avec le vecteur circularise parental comme standard interne durant la sélection (voir ci-dessous). En outre, vérifier les antécédents en transformant le vecteur linéarisé en l'absence de fragments de PCR.
Remarque: En cas de détection de niveaux initiaux de sécrétion faible, utilisez S.cerevisiae souches déficientes en protease comme BJ5465 pour favoriser l'accumulation de protéine active dans les surnageants de culture. Si l'enzyme cible est soumis à une hyperglycosylation, l'utilisation de souches de glycosylation déficiente (par exemple Δ kre2 qui est seulement capable de se fixer oligomères plus petits mannose) pourrait être une option appropriée.
- Ici, utiliser une protéase déficiente et URA3 - dépendante S. cerevisiae, BJ5465. Transformer les cellules avec le vecteur circularise parental comme standard interne durant la sélection (voir ci-dessous). En outre, vérifier les antécédents en transformant le vecteur linéarisé en l'absence de fragments de PCR.
- Plaque les cellules transformées sur SC plaques d'abandon et les incuber à 30 ° C pendant trois jours. Plate (sur SC plaques d' abandon complété par l' uracile) URA3 - S. cellules cerevisiae dépourvues du plasmide comme témoin négatif pour le dépistage (voir ci - dessous).
2. Haut-Throughput Screening Assay (Figure 3)
- Remplir un nombre approprié de plaques à 96 puits stériles (23 plaques pour analyser une banque de 2.000 clones) avec 50 ul par puits de milieu minimal à l'aide d'un robot de pipetage.
- Prélever des colonies individuelles de la SC-goutte sur les plaques et les transférer sur les plaques à 96 puits.
- Dans chaque assiette, inoculer le numéro de la colonne 6 avec le type parental comme étalon interne et bien H1 avec URA3 - S. cerevisiae (cellules dans du milieu SC complété avec de l' uracile) sans plasmide en tant que témoin négatif.
Remarque: Eh bien H1 est rempli spécifiquement avec les médias d'abandon complété par l' uracile. Un média vierge puits contenant sans cellules peut également être préparé comme un contrôle de stérilité supplémentaire.
- Dans chaque assiette, inoculer le numéro de la colonne 6 avec le type parental comme étalon interne et bien H1 avec URA3 - S. cerevisiae (cellules dans du milieu SC complété avec de l' uracile) sans plasmide en tant que témoin négatif.
- Couvrir les plaques avec leurs couvercles et les envelopper dans Parafilm. Incuber les plaques pendant 48 heures à 30 ° C, 225 tpm et 80% d'humidité relative dans un shaker humide.
- Retirer le parafilm, ajouter 160 ul de milieu d'expression dans chaque puits à l'aide du robot de pipetage, et de reboucher les plaques incuber pendant encore 24 heures.
Note: moyenne minimale et moyenne d'expression sont préparés comme rapporté ailleurs 19. Les taux de sécrétion peuvent varier selon le gène à l'étude et, par conséquent, le til l'incubation fois doivent être optimisées dans chaque cas pour synchroniser la croissance des cellules dans tous les puits. - Centrifuger les plaques (plaques de base) à 2800 x g pendant 10 min à 4 ° C.
- Transférer 20 pl du surnageant des puits de la plaque maîtresse à la plaque de réplique à l'aide d'un robot de manipulation multiposte liquide.
Remarque: Pour favoriser la sécrétion de l' enzyme , il est conseillé de remplacer le peptide signal natif de la protéine cible par des peptides de signal couramment utilisés pour l' expression hétérologue dans la levure (par exemple, le facteur α prépro-chef, le chef de la K 1 tueur toxine de S. cerevisiae, ou même des versions chimères de deux peptides 13). En variante, le peptide signal natif peut être évolué exclusivement pour la sécrétion dans la levure. - Ajouter 20 pi de 2 mM de p -methoxybenzylalcohol dans 100 mM de tampon phosphate de sodium à pH 6,0 à l'aide du robot de pipetage. Incorporer les plaques brièvement avec un nous 96ll mélangeur de plaque et les incuber pendant 30 min à température ambiante.
- Avec le robot de pipetage, ajouter 160 ul du réactif FOX à chaque plaque de réplique et remuer brièvement avec le mélangeur (concentration finale de mélange FOX dans le puits: 100 uM xylénol orange, 250 uM Fe (NH 4) 2 (SO 4) 2 et 25 mM de H 2 SO 4).
- Ajouter plusieurs additifs au réactif pour améliorer la sensibilité, tels que des co-solvants organiques (DMSO, l' éthanol, le méthanol ou le sorbitol) 17. Ici, amplifier la réaction par addition de sorbitol à une concentration finale de 100 mM (figure 4).
- Lire les plaques (mode point final, t 0) à 560 nm sur un lecteur de plaque.
- Incuber les plaques à température ambiante jusqu'à ce que la couleur se développe et mesurer l'absorption à nouveau (t 1).
- Calculer l'activité relative de la différence entre la valeur d'ABS après incubation et celui de la mesure initiale normalisée à la éplucherType ntal pour chaque plaque (1 At - t 0).
- Soumettre les meilleurs tubes mutants à deux re-projections consécutives pour exclure les faux positifs.
Remarque: En général, re-projections comprennent l' isolement du plasmide à partir de levure, d' amplification et de purification dans Escherichia coli, suivie d' une transformation de cellules de levure fraîche avec le plasmide 19. Chaque clone sélectionné est re-projeté en pentaplicate.
Résultats
AAO de P. eryngii est un flavooxidase extracellulaire qui fournit des peroxydases fongiques par H 2 O 2 pour commencer à attaquer la lignine. Deux segments de AAO ont été soumis à l' évolution centrée dirigée par MORPHING afin d'améliorer son activité et son expression dans S. cerevisiae 19. Indépendamment des enzymes étrangers hébergés par S. cerevisiae, la question la plus critique lors de la construction des bibliothèques mutantes dans la levure concerne l'ingénierie des régions qui se chevauchent spécifiques pour favoriser l'épissage entre les fragments et leur clonage dans le vecteur linéarisé. Dans l'exemple actuel, pour chaque réaction PCR, tous les fragments avaient des surplombs d'environ 50 pb à favoriser dans épissage in vivo dans la levure. Le nombre d'événements de recombinaison dépend du nombre de segments à assembler et cloné avec le vecteur linéarisé (ie, deux événements de croisement ont eu lieu entre letrois segments PCR -les deux segments mutagène flanquant le segment- non mutagénisé et deux croisements supplémentaires avec le vecteur linéarisé; Figure 1). Selon notre expérience, les séquences qui se chevauchent de plus de 50 pb diminuent la probabilité de recombinaison interne alors qu'ils n'améliorer l'efficacité de la transformation.
Charges mutationnelles ont été ajustées en échantillonnant les bibliothèques mutantes avec des paysages différents, calculer le nombre de clones avec <10% de l'activité de l' enzyme parentale, et en outre les vérifier par séquençage d' un échantillon aléatoire de variants actifs et non-actifs (figure 5A). Pour la détermination du coefficient de variance S. cellules cerevisiae ont été transformées avec l'AAO parental et étalées sur SC-goutte sur les plaques. Des colonies individuelles ont été prélevées et inoculées dans une plaque à 96 puits, et l'activité des clones a été évaluée à partir de préparations fraîches. échantillon Mutagène2 (Taq / MnCl2 0,05 mM) a été choisi comme le point pour la construction et le criblage de banques départ.
En tant que l'activité biologique des AAO augmente la concentration en H 2 O 2 dans le milieu réactionnel, nous avons cherché un dosage précis et sensible pour quantifier des changements mineurs dans H 2 O 2. FOX est un procédé chimique basé sur la réaction de Fenton 20, dans lequel l' oxydation par H 2 O 2 entraîne la réaction de Fe 3+ à l' orange de xylénol pour former un complexe bleu-violet (o-phtaléine -cresolsulfone 3 ', 3' '- bis (méthylimino) diacétate ε 560 = 1,5 x 10 4 M -1 cm -1). L'étape d'oxydation ferreux a été amplifiée par l' addition de sorbitol pour améliorer la sensibilité de l'essai, ce qui augmente la propagation des radicaux avec une apparente ε = 2,25 x 560 10 5 M -1 cm -1 (Figurer 4).
La limite de détection de cet essai (dans la gamme pM) a été calculée par la méthode de détermination de vide dans une plaque à 96 puits avec les normes en triple exemplaire (0, 0,5, 1, 1,5, 2, 2,5, 3 et 4 uM de H 2 O 2 ) et l' utilisation de plusieurs surnageants de S. cerevisiae manquant URA3 - plasmide (figure 5B). Le dosage était linéaire en présence de sorbitol (jusqu'à 8 uM de H 2 O 2), et bien que la linéarité est plus persistant en l'absence de ce sucre (au moins jusqu'à 30 uM de H 2 O 2) , la réponse est plus faible (par exemple à 6 uM de H 2 O 2, une amélioration de quatre fois a été obtenue en présence de sorbitol à partir d' une -deep pourpre absorbance de 0,24 en l'absence -Dark orangée (figure 5B)). La relation entre Abs et la concentration AAO a été évaluée avec des quantités croissantes de enzyme (à partir de surnageants des levures) et une réponse linéaire a été observée; R 2 = 0,997 (figure 5C).
Il est à noter que le signal FOX était stable pendant plusieurs heures sans aucune interférence apparente par les différents éléments dans le bouillon de culture. La sensibilité estimée de FOX était d' environ 0,4 pM de H 2 O 2 produit par l'AAO dans le surnageant en présence de sorbitol et ~ 2 uM en son absence.
Une banque de mutants de 2.000 clones a été construite et criblée avec cet essai. Plusieurs mutants AAO ont été identifiés avec notamment l' amélioration de la sécrétion et l' activité contre p - méthoxybenzyle alcool (Figure 5D) 19.

Figure 1.. MORPHING Protocole pour AAO Evolution Deux régions différentes de AAO ont été ciblés pour la mutagenèse aléatoire et la recombinaison: M1 (bleu, 590 pb) qui comprend le peptide signal (SP); M2 (jaune, 528 pb). La région de HF (gris, 844 pb) a été amplifié avec polymérases haute fidélité. Régions mutagéniques ont été cartographiés dans la structure cristalline de AAO (PDB ID: 3FIM). S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
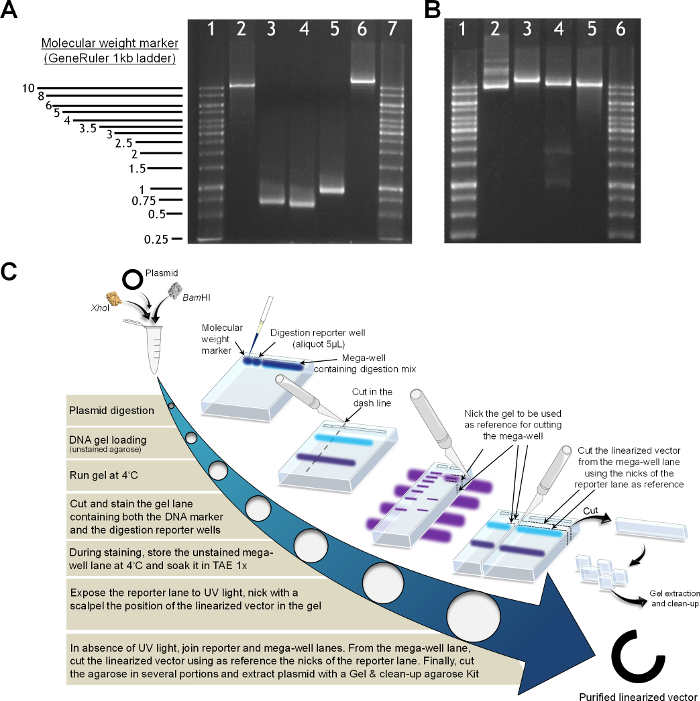
Figure 2. Préparation de produits de PCR et le vecteur linéarisé (A) sur gel d' agarose analytique (1% p: v). Contenant un marqueur de poids moléculaire (1 kb ladder) dans les pistes 1 et 7; le vecteur linéarisé Bam HI et Xho I, ligne 2; PCR le segment M1, piste 3; PCR le segment M2, piste 4; PCR segment de HF, piste 5; l'en vivo réassemblé vecteur linéarisé avec Nhe I (contenant le gène complet AAO avec les régions MI, HF et M-II), couloir 6. (B) Vector linéarisation, pistes 1 et 6 normes moléculaires, 1 Kb échelle; plasmide miniprep, piste 2; plasmide linéarisé avec Nhel, piste 3; le plasmide linéarisé avec Bam HI et Xho I, la piste 4; plasmide linéarisé obtenu par extraction de gel et de nettoyage après la digestion, la voie 5. (C) Protocole pour la purification de plasmide. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.

Figure 3. Protocole de criblage à haut-débit. Vue d' ensemble du processus. S'il vous plaît cliquer ici pour voir un plus grand version de ce chiffre.

Figure 4. La méthode de Fox. Champignon de la pourriture blanche attaque la paroi cellulaire du bois par une réaction de Fenton , qui produit un radical hydroxyle OH •. La méthode FOX couples cette réaction à xylénol orange (XO), et l'absorbance du complexe du XO-Fe est mesurée à 560 nm. Oxydation ferreux est amplifiée par l'addition de sorbitol au mélange réactif. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.

Figure 5. Mutagène Paysages pour MORPHING Bibliothèques Utilisation Différent Erreur Conditions PCR Prone et validation du test de dépistage. (A ng>) paysages MORPHING. ligne horizontale montre l'activité du type parental dans l'essai, tandis que les lignes en pointillés indiquent le coefficient de variation du dosage. Les pourcentages indiquent le nombre de clones ayant moins de 10% de l'activité de l'enzyme parentale. Les activités sont tracés dans l'ordre décroissant. (B) La limite de détection FOX a été évaluée avec des concentrations croissantes de H 2 O 2 en présence (cercles noirs) et de l' absence (carrés blancs) de sorbitol. Corrélation (C) linéaire entre la concentration AAO (surnageants transformantes) et Abs 560nm. Chaque point correspond à la moyenne des 8 expériences et comprend l'écart-type. (D) Mutant du paysage de la bibliothèque. Les variantes sélectionnées (carré ombré) ont été de nouveau criblées comme rapporté ailleurs 19. La ligne continue montre l'activité de type parental AAO.> S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Discussion
Dans cet article, nous avons résumé la plupart des trucs et astuces employées dans notre laboratoire pour concevoir des enzymes par l' évolution dirigée dans S. cerevisiae ( en utilisant AAO comme exemple) afin qu'ils puissent être adaptés pour une utilisation avec de nombreux autres systèmes enzymatiques eucaryote en suivant simplement l'approche commune décrite ici.
En termes de création de la bibliothèque, MORPHING est une méthode en un seul pot rapide à introduire et à recombiner des mutations aléatoires dans les petites étendues de protéines tout en laissant les autres régions de la protéine non modifiée 16. Les bibliothèques avec plusieurs charges mutationnelles peuvent être facilement préparées et recombinés in vivo, ainsi que le plasmide linéarisé, pour générer un vecteur à replication autonome complète. Il est essentiel que les séquences qui se chevauchent flanquent chaque étirement pour permettre aux fragments du gène complet réassemblés par recombinaison in vivo, ce qui évite les réactions de PCR supplémentaires et dans les étapes de ligature in vitro. Dans cette protocoledel, la fréquence des événements de croisement entre les fragments de PCR peut être augmentée en réduisant la taille des régions qui se chevauchent, bien que cela puisse compromettre l'efficacité de la transformation. Quelles que soient les polymérases d'ADN utilisées pour la PCR mutagene, les charges mutationnelles peuvent être ajustées en construisant et en analysant au préalable les petits paysages de la bibliothèque de mutants (figure 5A). Si le kit GeneMorph II est utilisé, il est toujours conseillé de suivre cette approche car in vivo recombinaison de l' ADN peut notamment modifier les charges mutationnelles estimées par le fabricant. En termes généraux, les paysages mutantes dans lesquelles 35 - 50% du total des clones sélectionnés ont moins de 10% de l'activité parentale sont appropriés pour des campagnes d'évolution dirigée, bien que ce nombre varie en fonction de la protéine cible et son activité. En règle générale, l'analyse des bibliothèques mutantes paysages sont en outre vérifiée par séquençage de l'ADN d'un échantillon aléatoire de mutants. Dans l'exemple actuel, la TaqL'ADN polymérase a été utilisé en raison de son taux élevé d'erreurs, qui est liée à l'absence de 3'→ 5'proof lecture activité exonucléase. Les charges mutationnelles dans les banques de Taq ont été modifiées par l'addition de différentes concentrations de MnCl 2, mais l'utilisation de dNTP asymétriques et / ou la réduction des concentrations de modèles génétiques sont également des options appropriées. limitations inhérentes à MORPHING proviennent du nombre de segments à recombiner. Selon notre expérience, jusqu'à quatre blocs de protéines (cinq événements de croisement en comptant les zones de recombinaison avec le vecteur linéarisé) peut être épissé avec de bons rendements de transformation (~ 10 5 clones par réaction de transformation). Cette méthode peut être facilement modifié pour effectué mutagénèse saturation multiple (par exemple, en utilisant des amorces dégénérées NDT ou la création de dégénération pour 22 codons uniques) pour explorer plusieurs positions simultanément tout en réduisant considérablement les efforts de dépistage 21,22.
Le protocole de criblage direct "aveugle" pour AAO est extrêmement sensible et fiable (basé sur la détection directe de H 2 O 2 quel que soit le substrat utilisé par l'enzyme), ce qui représente un test complémentaire à d' autres bien établie indirecte protocoles pour détecter les peroxydes (principalement des peroxydases de couplage avec des substrats colorimétriques). En effet, le dosage FOX a été couramment utilisé pour mesurer H 2 O 2 dans les fluides biologiques, et elle peut maintenant être facilement traduits dans des protocoles d'évoluer AAO et toute autre H 2 O 2 produisant des enzymes (par exemple, glucose oxydases, déshydrogénases cellobiose, le glyoxal oxydases, méthanol oxydases), en particulier pour l'activité sur des substrats non-naturels où les réponses sont par ailleurs difficiles à détecter.
S. cerevisiae est l'hôte le plus approprié pour l' évolution dirigée des gènes eucaryotes car il offre des efficacités de transformation élevées (jusqu'à 1 x 106 transformants / ug d'ADN), il exécute le traitement et les modifications post-traductionnelles complexes (y compris les N- et C-terminaux de traitement, et la glycosylation) et exporte des protéines étrangères dans le bouillon de culture par l' intermédiaire d' une voie sécrétoire. En outre, des outils de biologie moléculaire bien établies sont disponibles pour travailler avec cette levure, y compris épisomique unidirectionnelle ou bidirectionnelle (non intégratif) vecteurs navettes sous le contrôle des promoteurs de différentes forces. Last but not least, sa haute fréquence de recombinaison homologue de l' ADN a permis une gamme de méthodes à développer pour obtenir une diversité d'ADN qui sont actuellement utilisés pour évoluer protéines simples, ainsi que les voies d'enzymes plus complexes 8, 12, 13, 23. la vivo réparation de fente et le dispositif de relecture de cette levure peuvent également être utilisés pour créer des chimères quand recombiner gènes différents (avec env. 60% d'identité de séquence d'ADN), ainsi que de mélanger les meilleurs descendants / mutations d'une directed campagne d'évolution, ou de réunir in vitro et dans les méthodes de recombinaison in vivo dans un tour de l' évolution, enrichissant ainsi les bibliothèques mutantes en termes de pliabilité et la fonction.
Déclarations de divulgation
Les auteurs ont rien à révéler.
Remerciements
This work was supported by the European Commission project Indox-FP7-KBBE-2013-7-613549; a Cost-Action CM1303-Systems Biocatalysis; and the National Projects Dewry [BIO201343407-R] and Cambios [RTC-2014-1777-3].
matériels
| Name | Company | Catalog Number | Comments |
| 1. Culture media | |||
| Ampicillin sodium salt | Sigma-Aldrich | A0166 | CAS Nº 69-52-3 M.W. 371.39 |
| Bacto Agar | Difco | 214010 | |
| Cloramphenicol | Sigma-Aldrich | C0378 | CAS Nº 56-75-7 M.W. 323.13 |
| D-(+)-Galactose | Sigma-Aldrich | G0750 | CAS Nº 59-23-4 M.W. 180.16 |
| D-(+)-Glucose | Sigma-Aldrich | G5767 | CAS Nº 50-99-7 M.W. 180.16 |
| D-(+)-Raffinose pentahydrate | Sigma-Aldrich | 83400 | CAS Nº 17629-30-0 M.W. 594.51 |
| Peptone | Difco | 211677 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P0662 | CAS Nº 7778-77-0 M.W. 136.09 |
| Uracil | Sigma Aldrich | U1128 | |
| Yeast Extract | Difco | 212750 | |
| Yeast Nitrogen Base without Amino Acids | Difco | 291940 | |
| Yeast Synthetic Drop-out Medium Supplements without uracil | Sigma-Aldrich | Y1501 | |
| Name | Company | Catalog Number | Comments |
| 2. PCR Reactions | |||
| dNTP Mix | Agilent genomics | 200415-51 | 25 mM each |
| iProof High-Fidelity DNA polymerase | Bio-rad | 172-5301 | |
| Manganese(II) chloride tetrahydrate | Sigma-Aldrich | M8054 | CAS Nº 13446-34-9 M.W. 197.91 |
| Taq DNA Polymerase | Sigma-Aldrich | D4545 | For error prone PCR |
| Name | Company | Catalog Number | Comments |
| 3. Plasmid linearization | |||
| BamHI restriction enzyme | New England Biolabs | R0136S | |
| Bovine Serum Albumin | New England Biolabs | B9001S | |
| XhoI restriction enzyme | New England Biolabs | R0146S | |
| Not I restriction enzyme | New England Biolabs | R0189S | |
| Gel Red | Biotium | 41003 | For staining DNA |
| Name | Company | Catalog Number | Comments |
| 4. FOX assays | |||
| Ammonium iron(II) sulfate hexahydrate | Sigma-Aldrich | F3754 | CAS Nº 7783-85-9 M.W. 392.14 |
| Anysil Alcohol | Sigma Aldrich | W209902 | CAS Nº 105-13-5 M.W. 138.16 |
| D-Sorbitol | Sigma-Aldrich | S1876 | CAS Nº 50-70-4 M.W. 182.17 |
| Hydrogen peroxide 30% | Merck Millipore | 1072090250 | FOX standard curve |
| Xylenol Orange disodium salt | Sigma-Aldrich | 52097 | CAS Nº 1611-35-4 M.W. 716.62 |
| Name | Company | Catalog Number | Comments |
| 5. Agarose gel stuff | |||
| Agarose | Norgen | 28035 | CAS Nº 9012-36-6 |
| Gel Red | Biotium | 41003 | DNA analysis dye |
| GeneRuler 1kb Ladder | Thermo Scientific | SM0311 | DNA M.W. standard |
| Loading Dye 6x | Thermo Scientific | R0611 | |
| Low-melting temperature agarose | Bio-rad | 161-3112 | CAS Nº 39346-81-1 |
| Name | Company | Catalog Number | Comments |
| 6. Kits and cells | |||
| S. cerevisiae strain BJ5465 | LGC Promochem | ATTC 208289 | Protease deficient strain with genotype: MATα ura3-52 trp1 leu2-delta1 his3-delta200 pep4::HIS3 prb1-delta1.6R can1 GAL |
| E. coli XL2-Blue competent cells | Agilent genomics | 200150 | For plasmid purification and amplification |
| NucleoSpin Gel and PCR Clean-up Kit | Macherey-Nagel | 740,609,250 | DNA gel extraction |
| NucleoSpin Plasmid Kit | Macherey-Nagel | 740,588,250 | Column miniprep Kit |
| Yeast Transformation Kit | Sigma-Aldrich | YEAST1-1KT | Included DNA carrier (Salmon testes) |
| Zymoprep yeast plasmid miniprep I | Zymo research | D2001 | Plasmid extraction from yeast |
| Name | Company | Catalog Number | Comments |
| 7. Plates | |||
| 96-well plates | Greiner Bio-One | 655101 | Clear, non-sterile, polystyrene (for activity measurements) |
| 96-well plates | Greiner Bio-One | 655161 | Clear, sterile, polystyrene (for microfermentations) |
| 96-well plate lid | Greiner Bio-One | 656171 | Clear, sterile, polystyrene (for microfermentations) |
Références
- Jäckel, C., Hilvert, D. Biocatalysts by evolution. Curr. Opin. Biotechnol. 21 (6), 753-759 (2010).
- Bornscheuer, U. T. Engineering the third wave of biocatalysis. Nature. 485 (7397), 185-194 (2012).
- Renata, H., Wang, Z. W., Arnold, F. H. Expanding the enzyme universe: accessing non-natural reactions by mechanism-guided directed evolution. Angew. Chem. Int. Ed. 54 (11), 3351-3367 (2015).
- Cobb, R. E., Chao, R., Zhao, H. Directed evolution: past, present and future. AIChE J. 59 (5), 1432-1440 (2013).
- Abatemarco, J., Hill, A., Alper, H. S. Expanding the metabolic engineering toolbox with directed evolution. Biotechnol. J. 8 (12), 1397-1410 (2013).
- Pourmir, A., Johannes, T. W. Directed evolution: selection of the host organism. Comput Struct Biotechnol J. 2 (3), e201209012(2012).
- Krivoruchko, A., Siewers, V., Nielsen, J. Opportunities for yeast metabolic engineering: lessons from synthetic biology. Biotechnol J. 6 (3), 262-276 (2011).
- Gonzalez-Perez, D., Garcia-Ruiz, E., Alcalde, M. Saccharomyces cerevisiae in directed evolution: an efficient tool to improve enzymes. Bioeng Bugs. 3, 172-177 (2012).
- Alcalde, M. Mutagenesis protocols in Saccharomyces cerevisiae by In Vivo Overlap Extension. Methods Mol. Biol. 634, 3-14 (2010).
- Bulter, T., Alcalde, M. Preparing libraries in Saccharomyces cerevisiae. Methods. Mol. Biol. 231, 17-22 (2003).
- Ostrov, N., Wingler, L. M., Cornish, W. Gene assembly and combinatorial libraries in S. cerevisiae via reiterative recombination. Methods. Mol. Biol. 978, 187-203 (2013).
- Shao, Z., Zhao, H., Zhao, H. DNA assembler, an in vivo genetic method for rapid construction of biochemical pathways. Nucleic Acids Res. 37 (2), e16(2009).
- Alcalde, M. Engineering the ligninolytic enzyme consortium. Trends Biotechnol. 33 (3), 155-162 (2015).
- Garcia-Ruiz, E. Directed evolution of ligninolytic oxidoreductases: from functional expression to stabilization and beyond. Cascade Biocatalysis: integrating stereoselective and environmentally friendly reactions. , Wiley-VCH. 1-22 (2014).
- Hernandez-Ortega, A., Ferreira, P., Martinez, A. T. Fungal aryl-alcohol oxidase: a peroxide-producing flavoenzyme involved in lignin degradation. Appl. Microbiol. Biotechnol. 93 (4), 1395-1410 (2012).
- Gonzalez-Perez, D., Molina-Espeja, P., Garcia-Ruiz, E., Alcalde, M. Mutagenic organized recombination process by homologous in vivo grouping (MORPHING) for directed enzyme evolution. PLoS One. 9, e90919(2014).
- Rhee, S. G., Chang, T., Jeong, W., Kang, D. Methods for Detection and Measurement of Hydrogen Peroxide Inside and Outside of Cells. Mol. Cells. 29 (6), 539-549 (2010).
- Sebestova, E., Bendl, J., Brezovsky, J., Damborsky, J. Computational tools for designing smart libraries. Methods. Mol. Biol. 1179, 291-314 (2014).
- Viña-Gonzalez, J., Gonzalez-Perez, D., Ferreira, P., Martinez, A. T., Alcalde, M. Focused directed evolution of aryl-alcohol oxidase in yeast using chimeric signal peptides. Appl. Environ. Microbiol. , (2015).
- Gay, C., Collins, J., Gebicki, J. M. Hydroperoxide Assay with the Ferric-Xylenol orange Complex. Anal. Biochem. 273 (2), 149-155 (1999).
- Reetz, M. T. Biocatalysis in organic chemistry and biotechnology: Past, present, and future. J. Am. Chem. Soc. 135 (34), 12480-12496 (2013).
- Mate, D. M., Gonzalez-Perez, D., Mateljak, I., Gomez de Santos, P., Vicente, A. I., Alcalde, M. The pocket manual of directed evolution: Tips and tricks. Biotechnology of Microbial Enzymes: Production, Biocatalysis and Industrial Applications. , Elsevier. Forthcoming Forthcoming.
- Chao, R., Yuan, Y., Zhao, H. Recent advances in DNA assembly technologies. FEMS Yeast Res. 15, 1-9 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon