Method Article
Un semplice e rapido protocollo non-enzimaticamente dissociare freschi tessuti umani per l'analisi di linfociti infiltranti
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
This protocol describes the rapid non-enzymatic dissociation of fresh human tissue fragments for qualitative and quantitative assessment of CD45+ cells (lymphocytes/leukocytes) present in various normal and malignant human tissues. Additionally, the supernatant obtained from the primary tissue homogenate can be collected and stored for further analysis or experimentation.
Abstract
La capacità delle cellule maligne di eludere il sistema immunitario, caratterizzata dalla fuga del tumore da entrambe le risposte immunitarie innate e adattative, è ormai accettato come un importante segno distintivo di cancro. La nostra ricerca sul cancro al seno si concentra sul ruolo attivo che tumorali infiltranti linfociti svolgono nella progressione tumorale e outcome dei pazienti. Verso questo obiettivo, abbiamo sviluppato una metodologia per la rapida isolamento delle cellule linfoidi intatte dai tessuti normali e anormali nel tentativo di valutarli in prossimità al loro stato originario. Omogenati preparati utilizzando un dissociatore spettacolo meccanico sia aumentata la vitalità delle cellule e di recupero, mantenendo l'espressione del recettore di superficie rispetto tessuti enzima-digerite. Inoltre, la digestione enzimatica del materiale insolubile restante non ha recuperato CD45 + cellule supplementari indicano che le misurazioni quantitative e qualitative nella omogeneizzato primario probabile riflettano effettivamente sottopopolazioni infiltrano nel Fragm tessutient. Le cellule linfoidi in questi omogenati possono essere facilmente caratterizzata usando immunologica (fenotipo, proliferazione, etc.) o molecolare (DNA, RNA e / o proteine) approcci. Cellule CD45 + possono essere utilizzati anche per la purificazione sottopopolazione, l'espansione in vitro o crioconservazione. Un ulteriore vantaggio di questo approccio è che il supernatante tessuto primaria da omogenati può essere utilizzato per caratterizzare e confrontare citochine, chemochine, immunoglobuline e antigeni presenti in tessuti normali e maligni. Questa funzione di protocollo estremamente bene per i tessuti mammari umani e dovrebbero essere applicate a una vasta gamma di tessuti normali e anormali.
Introduzione
The tumor microenvironment is composed of various cell types with numerous studies showing they each play distinct and important roles in tumorigenesis1,2. These include, but are not limited to, infiltrating immune cells, stromal cells, endothelial cells and tumor cells3. Ex vivo studies of tumor infiltrating lymphocytes (TIL; CD45+ cells or leukocytes, which are predominantly lymphocytes in breast tumors) from fresh human tissue samples is made difficult by their low frequency, the small sample sizes often available for research and the potential for loss of viability during extraction. Because immune cells infiltrating tumors are usually present as passengers rather than permanent residents in general they are easier to release from the tissue matrix.
Dissociating tumor tissue while maintaining cellular integrity is technically challenging and has traditionally been performed using a combination of mechanical and enzymatic steps to prepare single cell suspensions4-8. This approach involves lengthy incubation periods and is associated with a significant reduction in cell viability as well as the loss of cell surface receptors by enzymatic cleavage. High quality flow cytometric studies characterizing TIL in the tumor microenvironment as well as clean purifications of CD45+ subpopulations by flow cytometry or antibody-coated beads are more difficult to achieve from enzyme-digested tumor tissue. In addition, the supernatant (SN) from the resulting tumor homogenate is not amenable to further analysis including quantification of secreted proteins (cytokines, chemokines, immunoglobulins or tumor antigens) or experimental treatment of normal cells, because of the potential for protein degradation in the enzymatic digests.
In our search for a method to prepare single cell homogenates from breast tissues [including tumor, non-adjacent non-tumor (NANT) and normal (from mammary reductions) breast tissues] without enzymatic digestion, we tested a variety of mechanical homogenization techniques. Homogenates prepared using a mechanical dissociator had increased cell viability (2-fold) and total cell recovery (2-fold) while preserving surface receptor expression. Enzymatic digestion of the remaining insoluble material did not recover additional CD45+ cells suggesting they were all released in the initial homogenate. Thus, this rapid and simple approach allows both qualitative and quantitative assessment of the CD45+ subpopulations present in various normal and malignant human tissues. An added advantage of this approach is that the SN from the initial homogenate (primary tissue SN) can be collected and stored for further analysis or experimentation.
Protocollo
NOTA: Tutti i campioni sono stati acquisiti utilizzando un protocollo approvato dal Comitato Etico Medico dell'Istituto Jules Bordet con il consenso informato scritto ottenuto da ogni paziente.
1. Preparazione del tessuto omogenato
- Sezionare tessuti asportati (tessuto maligno e normale resezione dalla sala operatoria) sono nel laboratorio di patologia da personale qualificato per il ritiro immediato. Tumore, NANT (presa la distanza maggiore dal tumore possibile) e frammenti di tessuto normale sono comunemente trattati nell'ambito 1 - 3 ore di escissione chirurgica in un laboratorio BSL2 utilizzando le procedure di biosicurezza standard per tessuti umani. Un diagramma di flusso del protocollo è illustrato in Figura 1.
- Pesare tutti i frammenti di tessuto (normali, NANT e tumorali) e misurare la lunghezza, la larghezza e l'altezza (lunghezza x larghezza x altezza). Questo è un passo importante per la successiva normalizzazione delle sottopopolazioni di cellule, RNA estratto, etc.
NOTE: La gamma di dimensione del campione è di 100 a 10.000 mm 3 senza grassi, se possibile. - Colophon il frammento tumorale su un vetrino per H & E colorazione per verificare che il tessuto è in realtà parte del tumore.
- Fare questo premendo un microscopio vetrino sul frammento tumore e applicando una leggera pressione con le dita per qualche secondo.
- Fissare la diapositiva con isopropanolo per 2 min seguita da una fase di lavaggio in acqua. Controcolorare il tessuto per 30 secondi con ematossilina di Mayer.
- Lavare il vetrino in sei vasche di acqua. Colorare in Floxina B 2% per 15 sec.
- Lavare in un bagno di acqua seguita da quattro bagni di isopropanolo e finire con un bagno di acqua.
- Incubare in isopropanolo per 1 min e scarico. Chiaro in due bagni di xilene.
- Montare con mezzo di montaggio a base di xilene. Esaminate per cellularità tumorale (Figura 2).
NOTA: Principalmente, le cellule tumorali attaccano alla diapositiva impresso - stromale, linfoide o annuncioipose cellule rimangono raramente, lasciando spazi tra le cellule tumorali (Figura 2).
- Posizionare il frammento di tessuto in un piccolo piatto di coltura contenente 1 ml di, mezzo di cellule ematopoietiche senza siero chimica definita (di seguito denominato medio) a temperatura ambiente e tagliare a dadini in piccoli pezzi (~ 1 - 2 mm 2) con una sterile bisturi.
- Trasferire tutto (frammenti di tessuto + media) per un tubo meccanico dissociatore C.
- Lavare la piastra di Petri e bisturi con 2 ml di terreno con una pipetta Pasteur e aggiungere questo al tubo C (volume del mezzo per la dissociazione = 3 ml).
- Utilizzare il A.01 meccanica programma dissociatore per tubi C (il programma più delicato) per rendere omogenei i frammenti di tessuto in una sospensione di cellule singole. Posizionare il tubo C nell'apparato ed eseguire il programma due volte in successione (un ciclo = 25 sec).
NOTA: Questa procedura omogeneizzazione è stata stabilita e convalidato per il tessuto mammario umano, altro tumo o tessuto tipi può essere necessario utilizzare un programma diverso e dovrebbe essere testato prima. - Rimuovere il tubo C dall'apparato e decantare l'omogenato direttamente in una cella di filtro 40 micron seduto su un tubo da 50 ml. Utilizzando la stessa pipetta Pasteur come al punto 1.6, trasferire eventuali residui di liquido nel tubo C al filtro cella.
- Trasferire il liquido filtrato in un tubo da 15 ml con una punta micropipetta 1 ml. Mantenere temporaneamente il filtro delle cellule e il suo tubo da 50 ml.
- Risciacquare il tubo C con ulteriori 3 ml di terreno e trasferire questo, sempre con la stessa pipetta Pasteur come al punto 1.6, al filtro cella ancora seduto sul tubo 50 ml. Spremere un importo massimo del liquido residuo intrappolato nel tessuto unhomogenized nel tubo 50 ml muovendo delicatamente attorno al filtro con una pipetta Pasteur pulita o 1 ml punta che viene successivamente gettato via per evitare di contaminare l'eluato.
- Posizionare il filtro cellulare a testa in giù su tegli tubo C originale e lavare con 3 ml di terreno in modo che il tessuto unhomogenized scende nel tubo C.
- Re-omogeneizzare come al punto 1.7 per due cicli del programma A.01.
- Versare questa seconda omogenato attraverso il filtro cellulare seduto sul tubo 50 ml, risciacquare il tubo C nuovo con 3 medio ml (passo 1.10) e trasferire con la pipetta Pasteur al filtro cellulare seduto sul tubo 50 ml nuovo spremitura massimo quantità di liquido dal tessuto connettivo residua rimasta intrappolata nel filtro cella.
- A questo punto, un volume di 2,5 ml è ~ nel tubo 15 ml e ~ 9 ml nel tubo 50 ml.
2. Separazione del surnatante tessuti e cellule
- Centrifugare omogenati nei 15 ml e 50 ml provette per 15 min a 600 xg a temperatura ambiente.
- Decantare il SN dal tubo 15 ml in una provetta pulita e memorizzare temporaneamente a 4 ° C. Questo surnatante = tumore primario, o ti NANT normaleSSUE SN (volume finale di 2,5 ml) viene successivamente chiarito e aliquotati prima di conservazione a -80 ° C per le analisi future (vedi sotto).
- Eliminare il surnatante dal tubo 50 ml.
- Risospendere delicatamente entrambi pellet di cellule in un volume finale di 1 ml di mezzo. Brevemente, prima delicatamente rompere il pellet cellulare in entrambi i tubi (toccando il tubo su una superficie dura). Risospendere il pellet di cellule sciolto in 50 ml con 500 ml di medio e trasferire la sospensione cellulare al tubo 15 ml per risospendere il pellet secondo. Ripetere questo passaggio una volta con il secondo 500 ml di media per il massimo recupero di cellule.
- Trasferire 10 ml di sospensione cellulare per un piccolo tubo, mescolare con 10 ml di trypan blu (diluizione 1: 1) e contare il numero di cellule vitali mediante un emocitometro.
NOTA: A questo punto una frazione della sospensione cellulare può essere analizzata mediante citometria di flusso per valutare la dimensione della cella, granularità ed eventualmente un numero limitato di marcatori sottopopolazione per maggiori precvalutazione ise della distribuzione cella relativa nel omogeneizzato prima vasta analisi o sperimentazioni. Tutte le analisi di citometria a flusso incorporano etichettatura CD45 per la normalizzazione delle sottopopolazioni. - Agglomerare le cellule per centrifugazione a 300 xg per 10 min a temperatura ambiente. Le cellule del tumore, NANT, o tessuti normali sono ora pronti per essere ulteriormente purificati o analisi. Questi passaggi aggiuntivi sono migliori quando eseguite nello stesso giorno come la chirurgia.
NOTA: Per l'analisi di citometria di flusso, ma non di separazione delle cellule dei globuli rossi residue dovrebbero essere lisati dopo etichettatura anticorpo aggiungendo 0,4 ml di tampone di lisi dei globuli rossi per il pellet, immediatamente vortex per 1 sec e incubando un minimo di 10 min a camera temperatura (protetto dalla luce) prima dell'analisi.
3. Precisazione del surnatante Tissue
- Centrifugare le provette da 1,5 ml con SN tessuto a 15.000 xg per 15 minuti a 4 ° C.
- Rimuovere accuratamente tegli surnatante senza toccare o disturbare il pellet. Trasferire in una provetta pulita (o tubi) a seconda del numero e del volume di aliquote desiderati.
- Conservare il supernatante a -80 ° C per un uso futuro.
4. Il sangue del paziente
- Raccogliere un campione di sangue da ogni paziente come un controllo mediante prelievo venoso in provette eparinizzate il giorno prima dell'intervento. Centrifugare il sangue a 400 xg per 10 min a 20 ° C con il freno fuori per ottenere plasma e un buffy coat.
- Rimuovere il plasma e chiarire che per centrifugazione a 10.000 xg per 15 min a 20 ° C. Aliquotare e conservare plasma a -80 ° C per un uso futuro. Diluire il buffy coat in mezzo
- Separare le cellule mononucleari utilizzando standard di Ficoll-Hypaque gradiente centrifugazione prima dell'analisi immediata, isolamento sottopopolazione, DNA / RNA estrazione / proteine o crioconservazione.
5. Citometria a flusso
- Cellule etichetta secondo la produzioneistruzioni di r a 4 ° C, al riparo dalla luce. Lisare globuli rossi in sospensioni cellulari da frammenti di tessuto dopo l'etichettatura di anticorpi con l'aggiunta di 0,4 ml di tampone di lisi cellulare al pellet.
- Vortex immediatamente per 1 sec e incubare un minimo di 10 min a temperatura ambiente, protetto dalla luce. Passare le cellule marcate in un citofluorimetro per l'acquisizione dei dati senza lavaggio.
Risultati
La digestione enzimatica di frammenti di tessuto sia con soluzioni disponibili in commercio dissociazione tessuto o varie miscele di laboratorio di collagenasi, DNasi e / o inibitori ialuronidasi, unirà un'ampia varietà di recettori presenti sulla superficie delle cellule. I nostri studi, inizialmente concentrati sulle cellule CD4 + T infiltranti i tumori al seno, sono stati rapidamente presentati con un grave problema tecnico dovuto alla scissione dei recettori CD4 superficie utilizzando enzimatici standard di protocolli di digestione 4-8. Abbiamo testato una varietà di enzimi collagenasi con o senza DNasi e inibitori ialuronidasi, trovando che collagenasi I e II completamente rimosso CD4 dalla superficie cellulare, nonostante la vitalità dei linfociti alto (Figura 3A). Un effetto moderato sulla CD4 è stato osservato con collagenasi IV in tempi brevi di incubazione (1 - 2 hr) con etichettatura anticorpi CD4 inferiore identificati nel linfonodo digerito e tessuto tumorale rispetto al sangue non digerito e midollo osseo dello stesso paziente (Figura 3B; notare che a causa di limitazioni sul tessuto che riceviamo non siamo riusciti a confrontare linfonodo digerito e digerito e tessuto tumorale). Questa perdita di CD4 è stato confermato essere un sottoprodotto tecnica di collagenasi IV digestione confrontando cellule digerito e digerito CD4 + T isolate dal sangue di donatori sani (dati non mostrati). Mentre queste cellule CD4 lo T possono essere isolate da frammenti di tessuto IV-digerito collagenasi utilizzando sfere magnetiche, le popolazioni purificate spesso contengono quantità variabili di contaminazione da parte di altre cellule del microambiente tumorale e quindi erano non ottimale per i nostri scopi sperimentali.
In una continua ricerca di un approccio migliore, nel 2008, abbiamo testato un nuovo dissociatore meccanico senza l'utilizzo di enzimi. Questo apparecchio ha prodotto omogenati di tessuto del seno in modo rapido e con integrità recettore di superficie mantenuto 9. Flusso rappresentante citometria dot plots del tumore omogenati cellule following dissociazione e l'etichettatura con anticorpi specifici contro le cellule epiteliali (EpCAM) e dei leucociti (CD45) sono illustrati nella Figura 3C. Queste immagini sono tipiche osservazioni di routine per omogenati di tumore al seno. Questo protocollo non modifica in modo significativo la vitalità delle cellule CD45 + (cellule morte 4%); Tuttavia, vi è una notevole perdita di EpCAM + cellule vitali (cellule morte 38% nel tumore indicati, varia tra tumori). Nonostante questa perdita, il vitale EpCAM + e CD45 + sottoinsiemi possono essere suddivise in base a dimensioni e struttura in grandi cellule epiteliali (= cellule tumorali), piccole cellule epiteliali e grandi cellule CD45 + e CD45 + piccole cellule (la maggioranza delle cellule , che sono principalmente i linfociti; Figura 3C).
Il significativo incremento CD45 + cellule vitali ottenute con questo approccio consente l'acquisizione di multicolor riproducibili citometria a flusso di dati (mediantefino a dieci colori) per caratterizzare più pienamente i sottoinsiemi TIL nel cancro al seno. Flusso rappresentante citometria dot plots delle principali sottoinsiemi TIL sono mostrati in Figura 4, con i recenti esperimenti dimostrando che è anche possibile rilevare e quantificare minori cellule T e B sottoinsiemi tumore infiltrante 10-12, compresa colorazione intracellulare per canonica T e di trascrizione cellule B Fattori 12. Strategie di gating linfociti sono basati su cellule CD45 + vitali identificati con una macchia vitalità (nota:. Tumori possono variare notevolmente nella quantità di detriti cellulari e cellule epiteliali morte seconda del sottotipo BC, grado, etc). Questo approccio è stato utilizzato con successo per purificare sottopopolazioni linfocitarie dall'omogeneizzato con perline magnetiche o di separazione delle cellule per l'analisi dell'espressione genica mediante microarray o qRT-PCR 9. Mediatori solubili nel SN, compresi citochine e immunoglobuline, sono stati quantificati con un branello- immunodosaggio base ai dati risultanti normalizzati alla dimensione del frammento di tessuto. Un confronto di citochine (un pool Th1 / Th2 / Th7 / Th9 / Th17) o totali immunoglobuline (IgA, IgE, IgG, IgM) in BC SN con SN da tessuto mammario normale rivela incrementi sia associati al tessuto tumorale (Figura 5). Questi tessuti primario SN sono anche stati effettivamente analizzati per antigeni 10-12 e usato per trattare sperimentalmente linfociti da donatori sani, riproducendo così il fenotipo TIL in cellule normali 9.
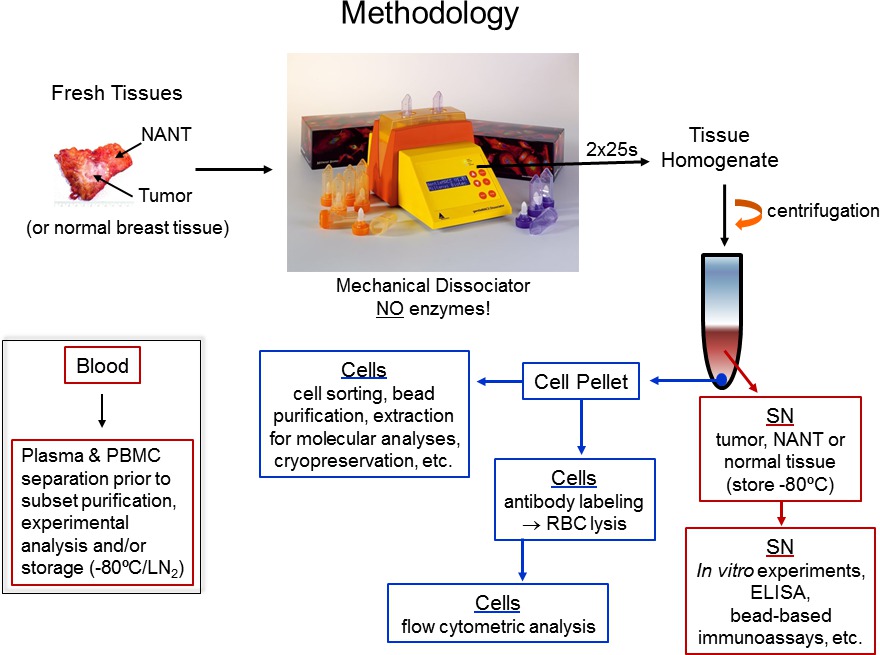
Sono mostrati Figura 1. Il diagramma di flusso protocollo. La procedura per la lavorazione di tessuti umani freschi e alcuni approcci analitici che possono poi essere utilizzati per valutare linfociti infiltranti i tessuti umani.

Figura 2. H & E macchiato l'immagine di una impronta tessuto tumorale mammario. Questa impronta, tratto da un frammento di tessuto tumorale mammario fresco, mostra la presenza di cellule tumorali con gli spazi aperti che riflettono le aree che non sono stati trasferiti. Cliccate qui per vedere una versione più grande di questa figura.

. Figura 3. Ottimizzazione di dissociazione tessuto del seno per l'analisi dei linfociti analisi citofluorimetrica di: (A) CD4 espressione di superficie su linfociti di non digerito e Collagenase I / II digerito tessuto tumorale; (B) l'espressione CD4 sui linfociti di linfonodi e tumori dei tessuti digerito with Collagenasi IV e rispetto al sangue e midollo osseo cellule non digerito dallo stesso paziente; e (C) Analisi di leucociti totali (CD45 +) e cellule epiteliali (EpCAM +) da tessuto tumorale rapidamente dissociato meccanicamente utilizzando il protocollo qui descritto. La vitalità delle cellule epiteliali e CD45 + cellule usando il nostro protocollo è valutata mediante l'incorporazione di risolvibile redditività tintura. Clicca qui per vedere una versione più grande di questa figura.

Figura 4. tumore infiltrante sottopopolazioni linfocitarie nel omogeneizzato. Multicolor citometria a flusso è stata effettuata il giorno dell'intervento dopo dissociazione meccanica utilizzando il protocollo descritto qui. Il cellule vitali e importanti sottopopolazioni linfocitarie unre mostrato: CD3 è un marker delle cellule T pan, CD4 e CD8 sono importanti marcatori subset di cellule T e CD19 è un marker delle cellule B padella. Cliccate qui per vedere una versione più grande di questa figura.
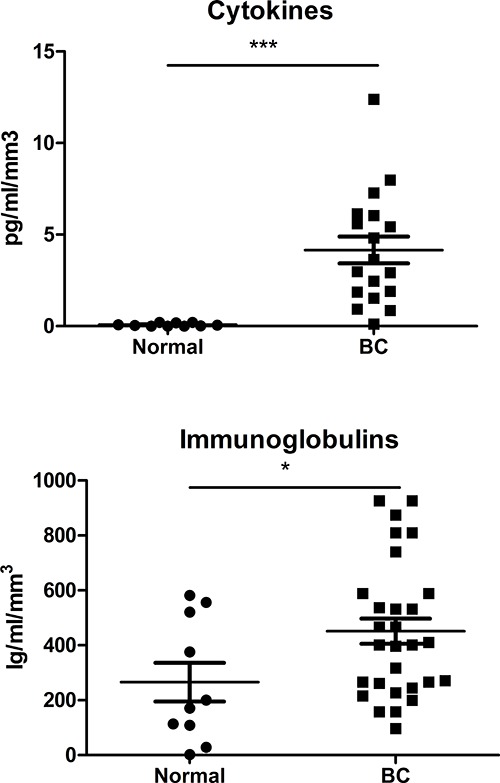
Figura 5. mediatori solubili nel surnatante. Un gruppo di citochine (Th1 / Th2 / Th7 / Th9 / Th17) e immunoglobuline (IgA, IgE, IgG, IgM) sono stati valutati mediante saggi immunologici basati perline-per analizzare SN derivato dal normale e BC tessuti utilizzando il protocollo descritto qui.
Discussione
Questo studio descrive un metodo ottimizzato per la rapida preparazione di normali e maligne omogenati di tessuto mammario senza digestione enzimatica per la successiva separazione delle cellule, l'estrazione, la crioconservazione e / o l'analisi fenotipica di CD45 + sottopopolazioni. L'obiettivo di questo approccio sperimentale è quello di produrre immagini della TIL che riflettono strettamente il loro stato in vivo e confrontarle con tessuti normali con la manipolazione minima dei tessuti freschi dalla sala operatoria. Ad oggi, il nostro laboratorio ha utilizzato questo protocollo per analizzare> 250 nuovi tessuti BC (tumorali e NANT),> 35 tessuti normali del seno di riduzione mammaria. In linea di principio, questo protocollo dovrebbe essere direttamente applicabile ad altri tipi di tumore o di tessuti; Tuttavia, alcuni ottimizzazione può essere necessario (cioè, il programma meccanico di dissociazione, numero di cicli, volume dei supporti, ecc). Per i tessuti a bassa infiltrazione linfocitaria, utilizzando un frammento di tessuto più grande può essere necessary, che è quello che facciamo per i tessuti normali, in cui la conta dei linfociti è bassa. In alternativa, un passo centrifugazione in gradiente di densità potrebbe essere aggiunto per arricchire la sospensione leucociti. Questa analisi di T e B sottopopolazioni di cellule umane infiltranti i tumori della mammella comprende una valutazione di> 100 marcatori di citometria di flusso (citometria a flusso di dati per 74 marcatori è disponibile nei dati supplementari di Gu-Trantien, et al. 9) e la purificazione di specifica di linfociti sottopopolazioni per le successive analisi immunologiche e molecolari. Inoltre, abbiamo valutato citochine, chemochine, immunoglobuline e antigeni tumorali nel SN tumore primario (rispetto agli NANT e normale SN tessuti) 10,11 come un riflesso di questi mediatori molecolari nel microambiente tumorale. Abbiamo testato anche l'effetto di trattare i linfociti del sangue periferico da donatori sani con tumore SN e dimostrato che molti dei cambiamenti di espressione genica identificati nella TIL possono essere specificamente riprodotto 9.
Nella loro microambiente, le cellule tumorali e stromali sono più strettamente tenuti nella matrice del tessuto rispetto alle cellule immunitarie, che sono tipicamente migratori. Il metodo qui descritto fornisce un mezzo semplice e rapida per isolare e analizzare TIL senza digestione enzimatica. I nostri numerosi tentativi di partecipare a questo approccio omogeneizzazione meccanica con una breve digestione enzimatica per la produzione di cellule vitali e del recettore linfoidi positivo e epiteliali dallo stesso frammento tumore non sono riusciti. Tissue omogeneizzazione rilascia efficiente linfociti, ma si traduce in una perdita significativa della vitalità delle cellule tumorali. Breve digestione enzimatica del tessuto libera un cellule tumorali vitali (Il numero totale aumenta in funzione del tempo di digestione) ma ha la conseguenza di una parallela diminuzione l'espressione di molti recettori di superficie, particolarmente evidente sui linfociti. Così, per l'analisi comparativa delle cellule tumorali e TIL dal medesimo tumoppure è ancora necessario elaborare separatamente due frammenti tumorali sequenziali.
Attualmente, la maggior parte degli studi volti a caratterizzare TIL far assumere digestione enzimatica (ore a O / N) spesso accoppiato con una qualche forma di dissezione meccanica 4-8. Ampliamento del TIL per ulteriori analisi o terapia prevede generalmente la successiva ex vivo coltivazione di TIL o frammenti di tessuto tumorale con agenti stimolanti (giorni o settimane) 13-15. Il metodo rapido, non enzimatica per la dissociazione tissutale qui descritto fornisce un mezzo semplice e riproducibili per l'estrazione CD45 + cellule intatte da frammenti di tessuto normale e anormale prima della loro espansione o analisi. Questa rapida acquisizione di cellule CD45 + da pazienti sottoposti tumorectomia potrebbe essere sviluppato per l'uso in un test di biomarker prognostico. Inoltre, essa può produrre TIL con maggiore potenziale di espansione ex vivo prima adottive immunotheRapy.
Divulgazioni
The authors declare that no conflict of interest exists.
Riconoscimenti
Questo lavoro è stato sostenuto da sovvenzioni fromthe Fondo belga per la Ricerca Scientifica (FNRS), Les Amis de l'Institut Bordet, FNRS-Opération Télévie, Piano di cancro del Belgio, Fonds Lambeau-Marteaux, Fonds JC Heuson e Fonds Barsy.
Materiali
| Name | Company | Catalog Number | Comments |
| GentleMacs Dissociator | Miltenyi Biotec | 130-093-235 | BD Medimachine is somewhat equivalent |
| Centrifuge 5810 R | Eppendorf | or other standard table top centrifuge | |
| Centrifuge 5417 R | Eppendorf | or other standard microcentrifuge | |
| Esco Class II A2 Biosafety Cabinet | ESCO global | or other standard BSL2 hood | |
| Inverted Microscope | Nikon eclipse TS100 | or other microscope compatible for a hemacytometer | |
| Bürker Chamber | Marienfield | 640210 | or other standard hemacytometer |
| Navios Flow Cytometer | Beckman Coulter | or other flow cytometer (8-10 color recommended) | |
| GentleMacs C-Tube | Miltenyi Biotec | 130-096-344 | BD Medimachine uses Filcon |
| Cell Culture Dish | Sarstedt | 72,710 | or other non-pyrogenic plasticware |
| Disposable Scalpel | Swann-Morton | 0510 | or standard single use sterile scalpel |
| BD Cell Strainer 40 µm | Becton Dickinson | 734-0002 | or other non-pyrogenic plasticware |
| BD Falcon Tube 50 ml | Becton Dickinson | 352070 | or other non-pyrogenic plasticware |
| BD Falcon Tube 15 ml | Becton Dickinson | 352097 | or other non-pyrogenic plasticware |
| BD FACS Tube 5 ml | Becton Dickinson | 352008 | or other non-pyrogenic plasticware |
| Sterile Pasteur Pipette 5 ml | VWR | 612-1685 | or other non-pyrogenic plasticware |
| Microfuge Tube 1.5 ml | Eppendorf | 7805-00 | or other non-pyrogenic plasticware |
| X-Vivo 20 | Lonza | BE04-448Q | serum-free medium recommended |
| Phosphate buffered saline | Lonza | BE17-516F | standard physiological PBS |
| Trypan blue | VWR | 17942E | or other vital stain |
| VersaLyse | Beckman Coulter | A09777 | for flow cytometry experiments |
| Fixable viability Dye eFluor 780 | eBioscience | 65-0865-14 | for flow cytometry experiments |
| anti-CD3 FITC | BD Biosciences | 345763 | for flow cytometry experiments |
| anti-CD3 Vio Blue | Miltenyi Biotec | 130-094-363 | for flow cytometry experiments |
| anti-CD4 PE | BD Biosciences | 345769 | for flow cytometry experiments |
| anti-CD4 APC | Miltenyi Biotec | 130-091-232 | for flow cytometry experiments |
| anti-CD8 ECD | Beckman Coulter | 737659 | for flow cytometry experiments |
| anti-CD8 PerCP | BD Biosciences | 345774 | for flow cytometry experiments |
| anti-CD19 APC-Vio770 | Miltenyi Biotec | 130-096-643 | for flow cytometry experiments |
| anti-CD45 VioGreen | Miltenyi Biotec | 130-096-906 | for flow cytometry experiments |
Riferimenti
- Chen, D. S., Mellman, I. Oncology meets immunology: the cancer-immunity cycle. Immunity. 39 (1), 1-10 (2013).
- Boudreau, A., van't Veer, J. L., Bissell, M. J. An 'elite hacker': breast tumors exploit the normal microenvironment program to instruct their progression and biological diversity. Cell Adh Migr. 6 (3), 236-248 (2012).
- Gajewski, T. F., Schreiber, H., Fu, Y. X. Innate and adaptive immune cells in the tumor microenvironment. Nat Immunol. 14 (10), 1014-1022 (2013).
- Quezada, S. A., et al. Limited tumor infiltration by activated T effector cells restricts the therapeutic activity of regulatory T cell depletion against established melanoma. J Exp Med. 205 (9), 2125-2138 (2008).
- Grange, C., et al. Phenotypic characterization and functional analysis of human tumor immune infiltration after mechanical and enzymatic disaggregation. J Immunol Methods. 372 (1-2), 119-126 (2011).
- McCauley, H. A., Guasch, G. Serial orthotopic transplantation of epithelial tumors in single-cell suspension. Methods Mol Biol. 1035, 231-245 (2013).
- Gros, A., et al. Myeloid cells obtained from the blood but not from the tumor can suppress T-cell proliferation in patients with melanoma. Clin Cancer Res. 18 (19), 5212-5223 (2012).
- Zirakzadeh, A. A., Marits, P., Sherif, A., Winqvist, O. Multiplex B cell characterization in blood, lymph nodes, and tumors from patients with malignancies. J Immunol. 190 (11), 5847-5855 (2013).
- Gu-Trantien, C., et al. CD4(+) follicular helper T cell infiltration predicts breast cancer survival. J Clin Invest. 123 (7), 2873-2892 (2013).
- Buisseret, L., et al. Lymphocytes Infiltrating Breast Cancer : Density, Composition And Organization. Annals of Oncology. 25 (1), 17 (2014).
- Garaud, S., et al. Characterization of B Cells Infiltrating Human Breast Cancer. Annals of Oncology. 25 (1), 18 (2014).
- Gu-Trantien, C., et al. Cxcl13-Producing Follicular Helper T Cells In Human Breast Cancer. Annals of Oncology. 25 (1), 17 (2014).
- Yee, C. The use of endogenous T cells for adoptive transfer. Immunol Rev. 257 (1), 250-263 (2014).
- Butler, M. O., et al. Ex vivo expansion of human CD8+ T cells using autologous CD4+ T cell help. PLoS One. 7 (1), 30229 (2012).
- Ye, Q., et al. Engineered artificial antigen presenting cells facilitate direct and efficient expansion of tumor infiltrating lymphocytes. J Transl Med. 9, 131 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon