Method Article
Isolement de populations de cellules myéloïdes et épithéliales vivantes à partir du poumon de souris
Dans cet article
Résumé
Ce protocole détaille l’isolement de populations immunes et non immunes vivantes du poumon de souris à l’état d’équilibre et après une infection grippale. Il fournit également des stratégies de contrôle pour identifier les sous-ensembles de cellules épithéliales et myéloïdes.
Résumé
Les poumons sont continuellement exposés à des agents pathogènes et à d’autres stimuli environnementaux nocifs, ce qui les rend vulnérables aux dommages, aux dysfonctionnements et au développement de maladies. Des études utilisant des modèles murins d’infection respiratoire, d’allergie, de fibrose et de cancer ont été essentielles pour révéler les mécanismes de progression de la maladie et identifier des cibles thérapeutiques. Cependant, la plupart des études axées sur le poumon de souris privilégient l’isolement des cellules immunitaires ou des cellules épithéliales, plutôt que des deux populations simultanément. Ici, nous décrivons une méthode de préparation d’une suspension unicellulaire complète de populations immunitaires et non immunitaires adaptée à la cytométrie en flux et au tri cellulaire activé par fluorescence. Ces populations comprennent des cellules épithéliales, des cellules endothéliales, des fibroblastes et une variété de sous-ensembles de cellules myéloïdes. Ce protocole implique un lavage broncho-alvéolaire et un gonflement ultérieur des poumons avec de la dispase. Les poumons sont ensuite digérés dans un mélange de libérase. Cette méthode de traitement libère une variété de types de cellules divers et permet d’obtenir une suspension unicellulaire qui ne nécessite pas de dissociation manuelle contre un filtre, ce qui favorise la survie cellulaire et produit un grand nombre de cellules vivantes pour les analyses en aval. Dans ce protocole, nous définissons également des schémas de contrôle pour les sous-ensembles de cellules épithéliales et myéloïdes dans les poumons naïfs et infectés par la grippe. L’isolement simultané de cellules immunitaires et non immunitaires vivantes est essentiel pour interroger la diaphonie intercellulaire et acquérir une compréhension plus approfondie de la biologie pulmonaire dans la santé et la maladie.
Introduction
Le poumon est composé des voies respiratoires, des alvéoles et de l’interstitium. Les cellules immunitaires et non immunitaires résident dans ces compartiments pour contribuer à la fois à la fonction pulmonaire homéostatique (échange gazeux) et à la défense de l’hôte contre les agressions environnementales, telles que les infections virales. Les grandes et petites voies respiratoires, ou les bronches et les bronchioles, sont tapissées de cellules épithéliales. Les cellules épithéliales prédominantes dans ces régions sont les cellules massues et ciliées qui sont responsables de la sécrétion de molécules protectrices et de la facilitation de la clairance mucociliaire1. Les alvéoles sont les structures les plus distales du poumon, tapissées de deux types de cellules épithéliales, les cellules alvéolaires de type I (ATI) et les cellules alvéolaires de type II (ATII). Les ATI sont responsables des échanges gazeux, et les ATII sécrètent et recyclent les tensioactifs pour assurer une tension superficielle appropriée 2,3. Les ATII se renouvellent d’eux-mêmes et peuvent également se différencier en ATI, un rôle particulièrement pertinent après des lésions pulmonaires4. De plus, les ATII fournissent une niche de soutien pour le principal type de cellules immunitaires qui peuplent la niche alvéolaire, les macrophages alvéolaires (MA)5,6. Au-delà de l’épithélium, les fibroblastes, les cellules endothéliales et les macrophages interstitiels (MI) (qui peuvent être associés à la fois aux nerfs et aux vaisseaux) composent l’interstitium 7,8,9,10. En réponse à une infection et à une lésion, de nombreuses cellules pulmonaires meurent et les cellules immunitaires, y compris les monocytes et les neutrophiles, s’infiltrent dans les tissus11,12. Les monocytes infiltrant les poumons se différencient en macrophages et peuvent contribuer au compartiment des macrophages à long terme13.
Les méthodes actuelles de préparation de suspensions unicellulaires à partir du poumon de souris sont généralement basées sur la collagénase et nécessitent une dissociation physique des tissus14. Cela peut entraîner un faible nombre de populations de cellules non immunitaires viables. Certains protocoles d’isolement des cellules épithéliales sont basés sur la dispase et produisent des proportions plus élevées de cellules épithéliales vivantes ; Cependant, ces protocoles n’étudient généralement pas les rendements et la viabilité des cellules immunitaires 15,16,17. La cytométrie en flux est une méthode couramment utilisée pour distinguer les populations cellulaires au sein d’un tissu digéré. Au départ, la cytométrie en flux pour les MA, les IM, les monocytes et les neutrophiles est clairement délimitée. Cependant, au cours de l’inflammation, le processus de déclenchement devient variable et difficile à interpréter en raison du continuum d’expression des marqueurs de surface des monocytes infiltrants se différenciant en macrophages. Par conséquent, le protocole présenté ici décrit également des stratégies de contrôle pour identifier les populations de cellules myéloïdes d’intérêt après une infection.
La dissociation robuste des cellules épithéliales pulmonaires et myéloïdes est essentielle pour discerner leurs fonctions homéostatiques et inflammatoires. Une méthode permettant d’isoler ces compartiments cellulaires en parallèle permettra d’analyser en aval les principaux types de cellules qui maintiennent la santé et stimulent la maladie. Une vue d’ensemble schématique du flux de travail de ce protocole se trouve à la figure 1.
Protocole
Ce protocole est conforme aux directives de l’Institutional Animal Care and Use Committee de la Harvard Medical School (numéros de subvention : R35GM150816 et P30DK043351). Des souris femelles C57BL/6J âgées de 8 à 12 semaines ont été utilisées pour les expériences. Ce protocole convient également aux souris mâles. Les détails des réactifs et de l’équipement utilisés dans cette étude sont fournis dans la table des matériaux.
1. Préparation des matériaux
- Décongelez les enzymes nécessaires sur de la glace, y compris la dispase, la libérase et la DNAse.
- Préparez d’autres réactifs nécessaires. Remplissez une seringue de 10 ml avec 5 ml d’EDTA de 2 mM dans du DPBS et ajustez-la avec une aiguille de 27 G. Remplissez la seringue de 1 ml avec du DPBS et fixez le cathéter.
2. Prélèvement des poumons
- Euthanasier la souris par injection intrapéritonéale de 400 à 500 mg/kg et de 25 à 100 mg/kg de mélange de kétamine et de xylazine (selon les protocoles approuvés par l’établissement). Procédez à la dissection une fois que la souris ne répond plus au pincement du coussinet du pied (~5 min).
REMARQUE : Un mélange de kétamine / xylazine est utilisé pour l’euthanasie au lieu du dioxyde de carbone pour prévenir l’hémorragie induite par la suffocation et l’infiltration des cellules immunitaires qui peuvent fausser les résultats. - Vaporisez la souris avec de l’éthanol à 70 %. Disséquez la souris à l’aide de ciseaux chirurgicaux fins et d’une pince #7. Faites une incision dans l’abdomen à l’aide de ciseaux et coupez latéralement à travers le péritoine.
- Faites une petite entaille dans le diaphragme pour libérer le vide. Coupez le diaphragme et la cage thoracique inférieure de la cavité corporelle pour exposer les poumons et le cœur.
- Perfuser les poumons avec 5 mL d’EDTA 2 mM dans du DPBS dans une seringue de 10 mL munie d’une aiguille de 27 G. La perfusion est réalisée en pénétrant dans la base du cœur avec l’aiguille, dirigée de la droite vers le ventricule gauche, et en distribuant lentement le liquide de la seringue18.
REMARQUE : La perfusion doit être effectuée lentement pour éviter la rupture du système vasculaire. - Ouvrez la cage thoracique au niveau du sternum en coupant les côtés de la cage thoracique, puis coupez verticalement à travers le sternum pour exposer la trachée19. Coupez autant de muscle et de tissu conjonctif que possible à l’aide de ciseaux fins et d’une pince #7 sans percer la trachée ou les poumons.
- Enroulez la trachée dans une suture (taille 2-0) comme pour l’attacher mais sans serrer. Entaillez la trachée latéralement et insérez le cathéter. Fixez fermement la suture autour du cathéter en tirant fermement sur la suture. N’insérez pas le cathéter à plus de 1 cm dans le poumon.
- Gonflez les poumons avec 1 mL de DPBS. Retirez le DPBS en tirant lentement sur la seringue. Regonflez les poumons avec le même DPBS et tirez à nouveau sur la seringue pour prélever le liquide de lavage broncho-alvéolaire (BALF).
- Débranchez la seringue du cathéter, en laissant le cathéter inséré. Versez BALF dans un tube de microcentrifugation.
- Remplissez la même seringue de 1 ml avec 1 ml de dispase et remettez-la en place dans le cathéter. Distribuer la seringue pour gonfler les poumons avec de la dispase. Tout en retirant le cathéter, resserrez la suture autour de la trachée pour éviter les fuites de dispase.
- Coupez la trachée latéralement. Retirez les poumons de la cavité corporelle en coupant le tissu conjonctif le long de l’arrière de la cage thoracique tout en utilisant des pinces pour tirer les poumons et la trachée vers le haut.
- Retirez le cœur des poumons. Placez les poumons dans 5 mL de DPBS sur de la glace.
- À cette étape, prenez le BALF séparé et essorez-le à 400 x g pendant 5 min à 4 °C. Le surnageant peut être aliquote et stocké selon les besoins. Il doit y avoir une pastille cellulaire visible qui peut être remise en suspension dans 100 μL de RPMI.
REMARQUE : On peut attendre jusqu’à 2 heures pour procéder à la digestion, ce qui permet de récolter plusieurs souris à la fois.
3. Digérer les poumons
- Préparez le mélange de digestion. Ajouter 83 μg/mL de libérase (solution mère de 50 μL) et 100 μg/mL d’ADN (solution mère de 15 μL) dans 3 mL d’IMRP par poumon.
- Disséquez les cinq lobes des poumons et jetez tout tissu conjonctif 19,20. Hachez le poumon avec des ciseaux pendant 1 min sur une lame de verre.
- Transférez le poumon haché dans un tube à centrifuger de 50 ml à l’aide de ciseaux de dissection. Ajouter la pastille de cellule récupérée du BALF et remise en suspension dans le RPMI dans le tube de centrifugation. Ajouter 3 mL de mélange de digestion dans le tube contenant l’échantillon à l’aide d’une pipette sérologique de 5 mL.
- Pipetez le mélange de digestion pulmonaire de haut en bas 2 à 3 fois. Placer à 37 °C dans un agitateur orbital à 140 tr/min avec les tubes de digestion placés à un angle de 45 degrés pendant 40 min.
4. Préparation de la suspension monocellulaire
- Retirez les tubes à centrifuger de 50 ml de l’agitateur orbital et placez-les sur de la glace. Pipette le mélange de digestion pulmonaire de haut en bas 5 à 6 fois. Filtrer à travers une crépine à cellules de 70 μm dans un nouveau tube de 50 mL. Lavez toutes les cellules restantes à travers le filtre et neutralisez les enzymes avec 10 ml de FBS à 5 % dans RPMI.
- Essorage pendant 5 min 4 °C à 600 x g. Aspirer le surnageant à l’aide d’un aspirateur ou d’une pipette. Remettre en suspension dans 1 mL de tampon de lyse ACK et incuber pendant 3 minutes à température ambiante pour lyser les globules rouges (GR). Ajouter 9 ml de PBS et pipeter de haut en bas.
- Essorer pendant 5 min à 4 °C à 600 x g. Aspirer le surnageant à l’aide d’un aspirateur ou d’une pipette. Remettre en suspension dans 1 mL de tampon FACS (1 % FBS dans PBS) et filtrer à travers une maille de 64 μm dans un tube de microcentrifugation à l’aide d’une pipette p1000.
5. Cytométrie en flux et analyses en aval
- Si vous effectuez une cytométrie en flux ou un tri cellulaire activé par fluorescence dans une plaque de 96 puits, colorez environ 1 à 2 millions de cellules par puits, soit une fraction de 8 % de la suspension pulmonaire.
- Faites tourner les cellules vers le bas. Tous les centrages dans une plaque à 96 puits doivent être effectués pendant 2,5 min (4 °C) à 600 x g. Retirez le surnageant et lavez les cellules une fois en les remettant en suspension dans 200 μL de DPBS. Tournez vers le bas et effleurez-vous.
- Si des cellules doivent être colorées pour l’analyse par cytométrie en flux, procédez à la procédure de coloration suivante.
- Remettre en suspension les cellules dans une coloration zombie vivante/morte (1:150) et un bloc Fc (1:250) dans 25 μL de DPBS pendant 10 min à température ambiante dans l’obscurité.
- Préparez le mélange de coloration des anticorps à une concentration de 2x dans le tampon FACS.
REMARQUE : Un mélange de coloration doit inclure tous les marqueurs du panel myéloïde (tableau 1) ou du panel épithélial (tableau 2) à leurs concentrations indiquées. - Ajouter 25 μL de mélange colorant à 25 μL de suspension pulmonaire dans une assiette et incuber sur de la glace pendant 30 minutes dans l’obscurité. Le volume total de coloration doit maintenant être de 50 μL et les anticorps primaires doivent être à leur concentration finale de 1x la concentration.
- Ajoutez 150 μL de tampon FACS, tournez pendant 2,5 min à 4 °C à 600 x g et retirez le surnageant. Laver deux fois dans 200 μL de tampon FACS.
- Si une coloration intracellulaire est nécessaire, fixez les cellules à l’aide d’un kit de fixation intracellulaire. Si une coloration intracellulaire n’est pas nécessaire, les cellules peuvent être fixées pendant 20 min dans un PFA à 4 % dans le DPBS.
- Lavez trois fois le fixateur avec 200 μL de tampon FACS. Les échantillons peuvent être conservés en suspension dans 200 μL de tampon FACS à 4 °C dans l’obscurité pendant 2 à 3 jours. Avant d’analyser les échantillons, des billes de comptage peuvent être ajoutées pour quantifier le nombre de cellules.
- Si les cellules doivent être triées, procédez à la procédure de coloration suivante.
- Remettre les cellules en suspension dans 1x mélange d’anticorps de coloration avec bloc Fc (1:250). Incuber pendant 30 min sur de la glace dans l’obscurité.
- Ajoutez 150 μL de tampon FACS, tournez pendant 2,5 min à 4 °C à 600 x g et retirez le surnageant. Laver trois fois avec 200 μL de tampon FACS et remettre en suspension dans 200 μL de tampon FACS avant de trier.
REMARQUE : Il est recommandé d’ajouter un colorant de viabilité, tel que DAPI, aux cellules avant le tri.
Résultats
Une digestion réussie se traduira par environ 20 à 25 millions de cellules avec une viabilité de 90 à 95 %. Si l’on ajoute environ 25 000 billes de comptage à une fraction de 8 % du poumon, les billes devraient compromettre 1 à 3 % des événements collectés. Après le gate sur les singulets, environ 90 % à 95 % des cellules doivent être négatives pour Zombie Aqua (indiquant la viabilité) (Figure 2A, Figure 3A).
Parmi les cellules CD45+, les cellules CD64+F4/80+ sont définies comme des macrophages (Figure 2B). Au départ, 80 à 90 % de tous les macrophages du poumon sont définis comme des MA (Siglec-F+CD11c+), et le reste peut être classé comme IM (Siglec-F-CD11c-) (Figure 2C). Cependant, lors d’une infection et d’une inflammation, cette définition change, et le gating IM contiendra des macrophages dérivés de monocytes (moMacs) en plus des IM (Siglec-F-CD11clo/hi). De plus, le ratio passe à environ 4 % de MI et 90 % de MI/moMac 9 jours après l’infection (ppp) (Figure 2C).
Les cellules CD64-F4/80+ peuvent être contrôlées par Siglec-F et Ly6C. Les cellules Siglec-F+ sont des éosinophiles (Figure 2D). Les cellules Ly6C+Siglec-F-CD11b+ peuvent être identifiées comme des monocytes (Figure 2D). Les cellules qui sont négatives pour CD64 et F4/80 (figure 2B) peuvent être davantage fermées comme CD11b+Ly6G+ pour identifier les neutrophiles (figure 2E).
Pour bloquer les cellules épithéliales, les premières cellules CD45+ doivent être exclues (Figure 3). Ensuite, les cellules endothéliales (CD31+) et les fibroblastes (Pdgfra+) peuvent être identifiés (Figure 3B). Par la suite, les cellules CD31-Pdgfra- qui sont EpCAM+ sont identifiées comme des cellules épithéliales (Figure 3C). Les cellules épithéliales peuvent être subdivisées en ATII (CMH-II+CD104-), cellules des voies respiratoires (cellules club et cellules ciliées) (CD104+) et autres cellules épithéliales (CMH-II-CD104-) (Figure 3D). La proportion de cellules ATII diminue au cours de l’infection.
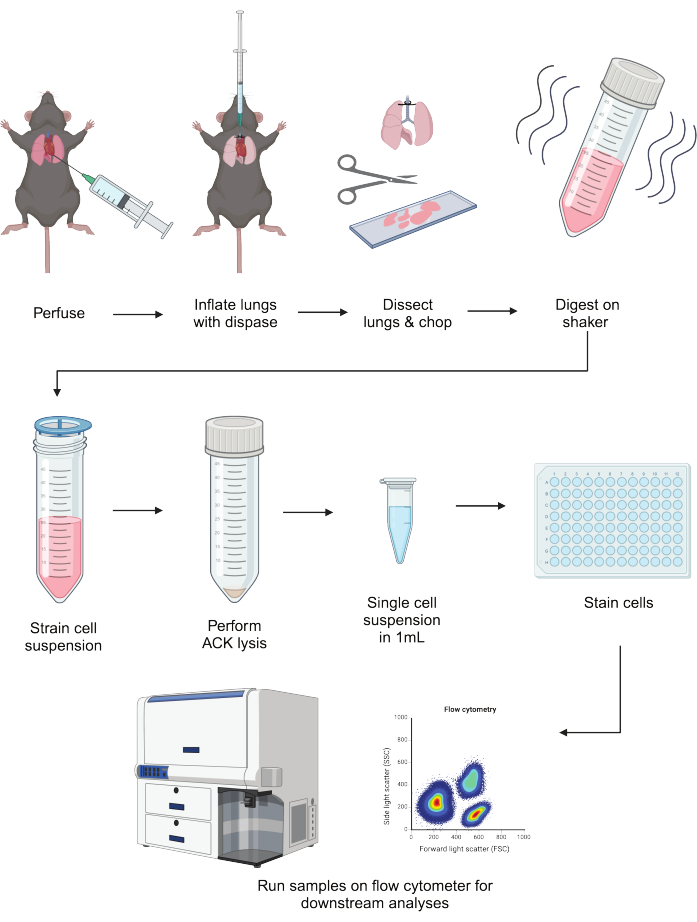
Figure 1 : Représentation schématique du protocole. Les poumons de souris sont perfusés, gonflés avec de la dispase et disséqués. Après les avoir hachés, ils sont digérés dans un mélange libérase/DNAse en secouant pendant 40 min. Les digestions sont passées dans une crépine cellulaire et les globules rouges sont lysés. Après remise en suspension dans le tampon FACS, les cellules peuvent être plaquées et colorées avant les applications en aval, y compris la cytométrie en flux. Cette figurine a été créée à l’aide de BioRender. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Stratégie de déclenchement des cellules myéloïdes. Déclenchement représentatif des analyses par cytométrie en flux de poumons de souris non infectés, 9 jours ou 14 jours après l’infection (ppp) par la grippe A/Porto Rico/8/34 (H1N1). (A) Déclenchement des cellules CD45+ vivantes. (B) Cellules CD45+ fermées CD64 par F4/80. (C) cellules CD64+F4/80+ (macrophages) contrôlées en AM et IMs/moMac à l’aide de Siglec-F par CD11c. (D) cellules CD64-F4/80+ contrôlées en éosinophiles et monocytes. (E) Cellules CD64-F4/80- contrôlées pour identifier les neutrophiles. Veuillez cliquer ici pour voir une version agrandie de cette figure.
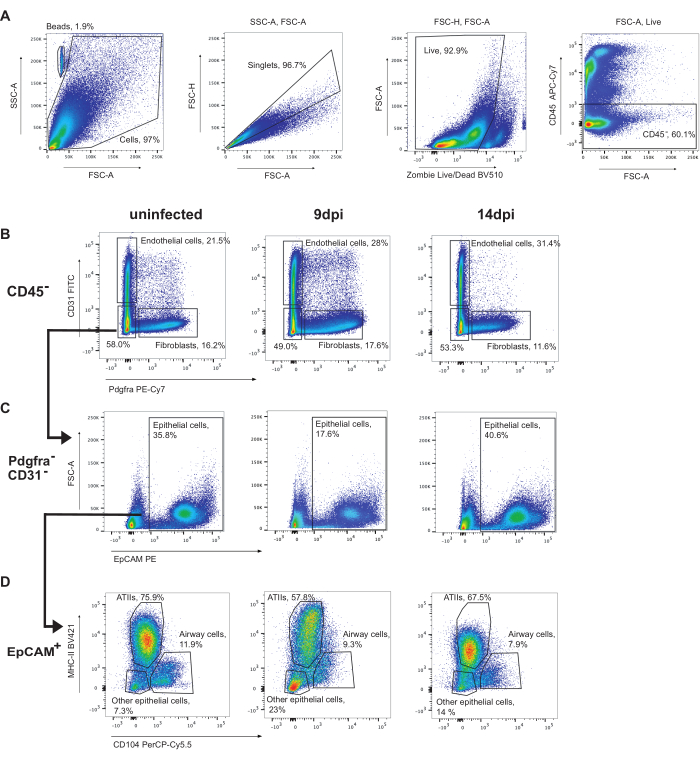
Figure 3 : Stratégie de déclenchement des cellules épithéliales. Déclenchement représentatif des analyses par cytométrie en flux de poumons de souris non infectés, 9 dpi ou 14 dpi avec la grippe A/Porto Rico/8/34 (H1N1). (A) Déclenchement de cellules CD45- vivantes. (B) Cellules CD45 bloquées sur CD31 (cellules endothéliales) par Pdgfra (fibroblastes). (C) Les cellules CD31-Pdgfra-EpCAM+ sont des cellules épithéliales. (D) Les cellules épithéliales de contrôle CMH-II par CD104 identifient les ATII (CMH-II+, CD104-), les cellules épithéliales des voies respiratoires (y compris les cellules ciliées et les cellules club) (CD104+) et d’autres cellules épithéliales (CMH-II-CD104-). Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Anticorps | Fluorophore | Concentration de coloration | Clone |
| anti-souris CD11c | AF-488 | 1:300 | N418 |
| anti-souris CD170 (Siglec-F) | PE | 1:200 | 1RNM44N |
| anti-souris CD45 | BV711 | 1:200 | 30-F11 |
| anti-souris CD64 (FcγRI) | APC | 1:200 | X54-5/7.1 |
| anti-souris F4/80 | PE/Cy7 | 1:100 | BM8 |
| anti-souris Ly-6C | BV605 | 1:500 | HK1.4 |
| anti-souris Ly-6G | PerCP/Cyanine5.5 | 1:300 | 1A8 |
| anti-souris/humain CD11b | BV421 | 1:800 | M1/70 |
| Taches vivantes/mortes | BV510 | 1:150 | N.A. |
Tableau 1 : Indice d’anticorps pour le panel myéloïde. Anticorps utilisés pour la stratégie de déclenchement myéloïde.
| Anticorps | Fluorophore | Concentration de coloration | Clone |
| anti-souris CD104 | PerCP/Cyanine5.5 | 1:200 | Référence 346-11A |
| anti-souris CD140a (Pdgfra) | PE/Cyanine7 | 1:200 | L’APA5 |
| anti-souris CD31 | FITC | 1:200 | 390 |
| anti-souris CD326 (Ep-CAM) | PE | 1:200 | G8.8 |
| anti-souris CD45 | APC/Cyanine7 | 1:200 | 30-F11 |
| anti-souris I-A/I-E (MHC-II) | BV421 | 1:400 | M5/114.15.2 |
| Taches vivantes/mortes | BV510 | 1:150 | N.A. |
Tableau 2 : Indice d’anticorps pour le panel épithélial. Anticorps utilisés pour la stratégie de déclenchement épithélial.
Discussion
Ce protocole décrit une digestion pulmonaire de souris qui isole environ 20 à 25 millions de cellules par souris avec une viabilité de 90 à 95 %. Il permet en outre de collecter des BALF pour une analyse plus approfondie. La suspension cellulaire résultante est compatible avec de multiples techniques de laboratoire, notamment la cytométrie en flux et le tri cellulaire activé par fluorescence pour isoler les cellules pour le séquençage ou la culture cellulaire. Brièvement, après la perfusion, le BALF est collecté et les poumons sont gonflés avec de la dispase. Les poumons sont ensuite hachés et digérés dans une solution de libérase/DNase. Après la lyse et le filtrage, on peut passer à des applications en aval, y compris l’analyse par cytométrie en flux de sous-ensembles myéloïdes et épithéliaux lors d’une infection par le virus de la grippe.
Si le BALF ne peut pas être récupéré et/ou si les poumons ne restent pas correctement gonflés, cela peut être dû à un mauvais placement du cathéter. Si le cathéter est placé trop loin dans la trachée/bronches, il peut percer le tissu jusqu’au péritoine. Cela se traduira par un manque d’inflation et une incapacité à récupérer le BALF. Si les poumons ont gonflé mais que le liquide ne peut pas être extrait d’un ou de plusieurs lobes du poumon, cela peut être dû au fait que le cathéter est dirigé dans une bronche. Tirer le cathéter légèrement plus loin devrait remédier à ce problème. Une perfusion suffisante est nécessaire pour éliminer la contamination des cellules immunitaires circulantes21. Dans les poumons gravement endommagés, les hémorragies et la diminution de l’intégrité de la barrière peuvent rendre les alvéoles plus susceptibles d’éclater sous l’effet de la pression de perfusion22. Par conséquent, il est essentiel de perfuser très lentement les souris dont les poumons sont endommagés.
Vous trouverez ci-dessous quelques lignes directrices sur les meilleures pratiques en matière de coloration par cytométrie en flux. En ce qui concerne la porte myéloïde, si une population Siglec-F+CD11c- est présente dans la porte des macrophages (CD45+CD64+F4/80+), il s’agit probablement d’une contamination par des éosinophiles. Nous avons constaté que les éosinophiles sont autofluorescents dans les canaux PE et BV605 - il est donc essentiel d’éviter d’utiliser un anticorps CD64 conjugué à ces fluorophores. De plus, bien que cette méthode de digestion puisse isoler les lymphocytes vivants, de nombreux marqueurs de surface spécifiques aux lymphocytes, tels que CD4 et CD8, sont clivés au cours du processus. Par conséquent, ce protocole n’est pas adapté à l’analyse par cytométrie en flux des populations de lymphocytes T. Dans de tels cas, une approche basée sur la collagénase peut être une alternative plus efficace23,24.
De nombreuses questions expérimentales nécessitent l’analyse des fractions immunitaires et non immunitaires d’une seule souris25,26. Par exemple, lors de l’étude des interactions entre différents types de cellules, il est essentiel de collecter et d’analyser les populations d’une seule souris afin de minimiser les artefacts potentiels qui peuvent survenir en raison de la variabilité biologique entre les animaux individuels. De plus, la comparaison des populations de cellules immunitaires et non immunitaires du même microenvironnement permet une compréhension plus complète de la dynamique des tissus et de leurs rôles dans le maintien de l’homéostasie et dans les processus pathologiques.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu en partie par des subventions des National Institutes of Health (R35GM150816 et P30DK043351), de la Fondation Charles H. Hood et du Harvard Stem Cell Institute. Nous remercions Alexander Mann et tous les autres membres du laboratoire Franklin pour leur aide et leurs conseils dans la conception et l’amélioration des schémas de contrôle et d’analyse de la cytométrie en flux. Nous remercions également la plateforme de cytométrie en flux d’immunologie de la Harvard Medical School. L’analyse par cytométrie en flux a été réalisée à l’aide de FlowJo. Les schémas des figures ont été créés à l’aide de BioRender.
matériels
| Name | Company | Catalog Number | Comments |
| 1 mL syringe with Slip Tip | VWR | BD309659 | |
| 1.7 mL microcentrifuge tube | DOT Scientific | RN1700-GMT | |
| 10 mL pipettes (disposable) | Fisher Scientific | 12-567-603 | |
| 10 mL Syringe with BD Luer-Lok Tip | VWR | 75846-756 | |
| 123 count eBeads Counting Beads | Thermo Scientific | 01-1234-42 | |
| 12-channel pipette (30-300ul) | USA Scientific | 7112-3300 | |
| 16% paraformaldehyde | VWR | 100503-917 | |
| 23 G needle with regular bevel | VWR | 305194 | |
| 27 G needle with regular bevel | VWR | BD305109 | |
| 5 mL pipettes (disposable) | Thermo Fisher Scientific | 170373 | |
| 50 mL centrifuge tubes | Olympus | 28-108 | |
| 96-well round bottom plate | Corning | 3797 | |
| ACK lysing buffer | Gibco | A100492-01 | |
| Alexa Fluor 488 anti-mouse CD11c | BioLegend | 117311 | |
| Anti-F4/80 Rat Monoclonal Antibody (PE (Phycoerythrin)/Cy7) | BioLegend | 123114 | |
| APC anti-mouse CD64 (FcγRI) | BioLegend | 139306 | |
| APC/Cyanine7 anti-mouse CD45 | BioLegend | 103115 | |
| BD Insyte Autoguard Shielded IV Catheters | VWR | 381423 | |
| Brilliant Violet 421 anti-mouse I-A/I-E (MHC-II) | BioLegend | 107632 | |
| Brilliant Violet 421 anti-mouse/human CD11b | BioLegend | 101235 | |
| Brilliant Violet 605 anti-mouse Ly-6C | BioLegend | 128036 | |
| Brilliant Violet 711 anti-mouse CD45 | BioLegend | 103147 | |
| C57BL/6J mice | Jackson Laboratories | ||
| Cd140a (PDGFRA) Monoclonal Antibody (APA5), PE-Cyanine7, eBioscience | Life Technologies | 25-1401-82 | |
| CD170 (Siglec F) Monoclonal Antibody (1RNM44N), PE | Life Technologies | 12170280 | |
| Cell strainers | Corning | 352350 | |
| Centrifuge | Eppenodorf | Centrifuge 5910R | |
| Deoxyribonuclease I from bovine pancreas (DNase) | Millipore Sigma | DN25-100MG | Reconstituted at 20 mg/mL in DPBS as stock solution stored at -20 °C |
| Dispase | VWR | 76176-668 | Thawed once and stored as 1mL aliquots at -20 °C |
| Dissection forceps (Dumont #7) | Fine Science Tools | 11297-00 | |
| Dissection scissors | Fine Science Tools | 14060-09 | |
| DPBS | Thermo Fisher Scientific | 14190250 | |
| eBioscience fixation kit | Life Technologies | 00-5523-00 | |
| EDTA | Life Technologies | AM9260G | |
| Ethanol | VWR | TX89125170HU | |
| FBS | GeminiBio | 100-106 | Thawed once and heat-inactivated before long-term storage as aliquots at -20 °C |
| FITC anti-mouse CD31 Antibody | BioLegend | 102406 | |
| Gibco RPMI 1640 Medium | Fisher Scientific | 11-875-093 | |
| Glass slides | Fisher Scientific | 12-552-3 | |
| graduated reservoir | USA Scientific | 1930-2235 | |
| Ice bucket | Corning | 432128 | |
| Ketamine hydrocholoride injection (100 mg/mL) | Dechra | Ketamine and xyalazine euthanization mixture can be kept at 30 mg/mL ketamine hydrochloride and 4.5mg/mL xylazine in sterile DPBS for up to one month. | |
| Liberase | Millipore Sigma | 5401119001 | Reconstituted at 5 mg/mL in DPBS as stock solution stored at -20 °C |
| Lids for 96-well plates | Fisher Scientific | 07-201-731 | |
| Orbital Incubator Shaker | Barnstead Lab-Line | SHKE4000 | |
| p1000 pipette | Eppenodorf | 3123000063 | |
| p1000 tips | USA Scientific | 1122-1830 | |
| p200 pipette | Eppenodorf | 3123000055 | |
| p200 tips | USA Scientific | 1110-1700 | |
| PE anti-mouse CD326 (Ep-CAM) | BioLegend | 118206 | |
| PerCP/Cyanine5.5 anti-mouse CD104 Antibody | BioLegend | 123614 | |
| PerCP/Cyanine5.5 anti-mouse Ly-6G | BioLegend | 127616 | |
| Pipet-Aid | Drummond | 4-000-101 | |
| Purified anti-mouse CD16/32 | BioLegend | 101302 | Referred to as "Fc block" in text |
| Spray bottle | VWR | 23609-182 | |
| Suture (Size 2-0) | VWR | 100190-026 | |
| Underpads | VWR | 56617-014 | |
| Xysed (xylazine 100mg/mL) | Pivetal | See ketamine hydrocholoride notes above. | |
| Zombie Aqua Fixable Viability Kit | BioLegend | 423102 |
Références
- Wanner, A., Salathé, M., O'Riordan, T. G. Mucociliary clearance in the airways. Am J Respir Crit Care Med. 154 (6), 1868-1902 (1996).
- Mason, R. J., Williams, M. C. Type II alveolar cell. Defender of the alveolus. Am Rev Respir Dis. 115 (1), 81-91 (1977).
- Fehrenbach, H. Alveolar epithelial type II cell: defender of the alveolus revisited. Respir Res. 2 (1), 33-46 (2001).
- Basil, M. C., Alysandratos, K. -. D., Kotton, D. N., Morrisey, E. E. Lung repair and regeneration: Advanced models and insights into human disease. Cell Stem Cell. 31 (4), 439-454 (2024).
- Hussell, T., Bell, T. J. Alveolar macrophages: plasticity in a tissue-specific context. Nat Rev Immunol. 14 (2), 81-93 (2014).
- Gschwend, J., et al. Alveolar macrophages rely on GM-CSF from alveolar epithelial type 2 cells before and after birth. J Exp Med. 218 (1), e20210745 (2021).
- Gillich, A., et al. Capillary cell-type specialization in the alveolus. Nature. 586 (7831), 785-789 (2020).
- Schyns, J., et al. Non-classical tissue monocytes and two functionally distinct populations of interstitial macrophages populate the mouse lung. Nat Commun. 10 (1), 3964 (2019).
- Chakarov, S., et al. Two distinct interstitial macrophage populations coexist across tissues in specific subtissular niches. Science. 363 (6432), eaat3773 (2019).
- Tsukui, T., et al. Collagen-producing lung cell atlas identifies multiple subsets with distinct localization and relevance to fibrosis. Nat Commun. 11 (1), 1920 (1920).
- Aegerter, H., et al. Influenza-induced monocyte-derived alveolar macrophages confer prolonged antibacterial protection. Nat Immunol. 21 (2), 145-157 (2020).
- Johansson, C., Kirsebom, F. C. M. Neutrophils in respiratory viral infections. Mucosal Immunol. 14 (4), 815-827 (2021).
- Li, F., et al. Monocyte-derived alveolar macrophages autonomously determine severe outcome of respiratory viral infection. Sci Immunol. 7 (71), eabj5761 (2022).
- Moll, H. P., et al. Orthotopic transplantation of syngeneic lung adenocarcinoma cells to study PD-L1 expression. J Vis Exp. (143), e58101 (2019).
- Warshamana, G. S., Corti, M., Brody, A. R. TNF-α, PDGF, TGF-β1 expression by primary mouse bronchiolar-alveolar epithelial and mesenchymal cells: TNF-α induces TGF-β1. Exp Mol Pathol. 71 (1), 13-33 (2001).
- Corti, M., Brody, A. R., Harrison, J. H. Isolation and primary culture of murine alveolar type II cells. Am J Respir Cell Mol Biol. 14 (3), 309-315 (1996).
- Quantius, J., et al. Influenza virus infects epithelial stem/progenitor cells of the distal lung: Impact on Fgfr2b-driven epithelial repair. PLoS Pathog. 12 (5), e1005544 (2016).
- Wu, J., et al. Transcardiac perfusion of the mouse for brain tissue dissection and fixation. BIO Protoc. 11 (8), e3988 (2021).
- Morton, J., Snider, T. A. Guidelines for collection and processing of lungs from aged mice for histological studies. Pathobiol Aging Age Relat Dis. 7 (1), 1313676 (2017).
- Bijgaart, R. J. E., van den Kong, N., Maynard, C., Plaks, V. Ex vivo live imaging of lung metastasis and their microenvironment. J Vis Exp. (108), e53741 (2016).
- Shi, W., et al. Isolation and purification of immune cells from the liver. Int Immunopharmacol. 85, 106632 (2020).
- Short, K. R., et al. Influenza virus damages the alveolar barrier by disrupting epithelial cell tight junctions. Eur Respir J. 47 (3), 954-966 (2016).
- Laidlaw, B. J., et al. CD4+ T cell help guides formation of CD103+ lung-resident memory CD8+ T cells during influenza viral infection. Immunity. 41 (4), 633-645 (2014).
- D'Agostino, M. R., et al. Protocol for isolation and characterization of lung tissue-resident memory T cells and airway-trained innate immunity after intranasal vaccination in mice. STAR Protoc. 3 (2), 101652 (2022).
- Waal, A. M., de Hiemstra, P. S., Ottenhoff, T. H., Joosten, S. A., vander Does, A. M. Lung epithelial cells interact with immune cells and bacteria to shape the microenvironment in tuberculosis. Thorax. 77 (4), 408-416 (2022).
- Bhattacharya, J., Westphalen, K. Macrophage-epithelial interactions in pulmonary alveoli. Semin Immunopathol. 38 (4), 461-469 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon