Method Article
Suivi à long terme des taux de consommation d’oxygène dans des cultures épithéliales pigmentaires rétiniennes hautement différenciées et polarisées
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Nous présentons un nouvel appareil pour mesurer les taux de consommation d’oxygène (OCR) dans les cultures d’épithélium pigmentaire rétinien (EPR). L’appareil peut mesurer l’OCR pendant des semaines sur des EPR cultivés sur des plaques de culture cellulaire standard avec des milieux standard pendant que les plaques sont dans un incubateur de culture cellulaire standard.
Résumé
Le métabolisme mitochondrial est essentiel au fonctionnement normal de l’épithélium pigmentaire rétinien (EPR), une monocouche de cellules de la rétine importante pour la survie des photorécepteurs. Le dysfonctionnement mitochondrial de l’EPR est une caractéristique de la dégénérescence maculaire liée à l’âge (DMLA), la principale cause de cécité irréversible dans les pays développés, et de la vitréorétinopathie proliférative (PVR), une complication cécitante des décollements de rétine. Les conditions dégénératives de l’EPR ont été bien modélisées par des systèmes de culture d’EPR qui sont hautement différenciés et polarisés pour imiter l’EPR in vivo . Cependant, la surveillance des taux de consommation d’oxygène (OCR), un indicateur de la fonction mitochondriale, a été difficile dans de tels systèmes de culture, car les conditions qui favorisent la polarisation et la différenciation idéales de l’EPR ne permettent pas de mesurer facilement l’OCR.
Ici, nous présentons un nouveau système, Resipher, pour surveiller l’OCR pendant des semaines à la fois dans des cultures d’EPR bien différenciées tout en maintenant l’EPR sur des substrats de croissance optimaux et des milieux de culture physiologiques dans un incubateur de culture cellulaire standard. Ce système calcule l’OCR en mesurant le gradient de concentration en oxygène présent dans les milieux au-dessus des cellules. Nous discutons des avantages de ce système par rapport aux autres méthodes de détection de l’OCR et de la manière de mettre en place le système de mesure de l’OCR dans les cultures d’EPR. Nous abordons les principaux conseils et astuces pour l’utilisation du système, la prudence dans l’interprétation des données et les instructions pour résoudre les problèmes de résultats inattendus.
Nous fournissons également un calculateur en ligne pour extrapoler le niveau d’hypoxie, de normoxie ou d’hyperoxie ressenti par les cultures d’EPR en fonction du gradient d’oxygène dans le milieu au-dessus des cellules détectées par le système. Enfin, nous passons en revue deux applications du système, en mesurant l’état métabolique des cellules EPR dans un modèle PVR et en comprenant comment l’EPR s’adapte métaboliquement à l’hypoxie. Nous prévoyons que l’utilisation de ce système sur des cultures d’EPR hautement polarisées et différenciées améliorera notre compréhension du métabolisme mitochondrial de l’EPR à la fois dans des états physiologiques et pathologiques.
Introduction
L’épithélium pigmentaire rétinien (EPR) est une monocouche de cellules épithéliales fonctionnellement postmitotiques et hautement polarisées qui forment une barrière entre les photorécepteurs sensibles à la lumière de la rétine et leur circulation sanguine, un lit capillaire appelé choriocapillaire. Comme le rôle des neurones de soutien de la glie, l’EPR remplit une myriade de fonctions pour soutenir les photorécepteurs, y compris la phagocytose des segments externes des photorécepteurs, le transport des nutriments et le soutien métabolique des photorécepteurs, et la sécrétion de facteurs de croissance essentiels, tous essentiels au maintien de la fonction visuelle.
La dégénérescence de l’EPR est à l’origine de plusieurs troubles dégénératifs courants de la vision. Dans la dégénérescence maculaire liée à l’âge (DMLA), l’une des causes les plus courantes de perte de vision incurable dans le monde, l’EPR meurt, et les photorécepteurs sus-jacents souffrent donc d’une dégénérescence secondaire. Dans la vitréorétinopathie proliférative (PVR), l’EPR sort de son état postmitotique normalement quiescent, proliférant et se dédifférenciant en un état mésenchymateux (une transition dite épithéliale à mésenchymateuse [EMT]) avec des altérations de son métabolisme. La dédifférenciation de l’EPR entraîne une perte de support de l’EPR vers les photorécepteurs tout en déclenchant un état plus fibreux. Cela entraîne à la fois une dégénérescence des photorécepteurs et des cicatrices induites par l’EPR, qui déclenchent toutes deux une perte de vision 1,2.
Une grande partie du soutien de l’EPR aux photorécepteurs est métabolique, et la dérégulation métabolique est un facteur critique dans de nombreuses maladies rétiniennes, y compris la DMLA et la PVR. L’EPR sert de barrière régulatrice entre les photorécepteurs et leur source d’oxygène et de nutriments, le choriocapillaire. Ainsi, ce que l’EPR choisit de métaboliser par rapport à ce qu’il choisit de passer du choriocapillaire aux photorécepteurs régit fortement le métabolisme et la survie des photorécepteurs. De nombreuses études ont montré que l’EPR dépend fortement du métabolisme mitochondrial pour sa santé normale, et que les photorécepteurs dépendent fortement de la glycolyse3. Cela a introduit le concept d’états métaboliques complémentaires et entrelacés entre les photorécepteurs et l’EPR. Plus précisément, l’EPR réduit son métabolisme des substrats métaboliques préférés des photorécepteurs et utilise à la place les sous-produits du métabolisme des photorécepteurs combinés aux métabolites que les photorécepteurs ne consomment pas. Dans des maladies telles que la PVR et la DMLA, les preuves suggèrent fortement que l’EPR devient plus glycolytique et moins dépendant du métabolisme mitochondrial ; ce changement vers la glycolyse de l’EPR peut priver les photorécepteurs des métabolites dont ils ont besoin, déclenchant une dégénérescence 4,5,6. Étant donné à quel point le métabolisme de l’EPR et des photorécepteurs est interdépendant et à quel point le métabolisme altéré sous-tend la maladie rétinienne, il existe un fort intérêt pour la modélisation et la manipulation du métabolisme de l’EPR à des fins thérapeutiques.
Bien que l’étude du métabolisme mitochondrial de l’EPR in vivo soit idéale, de nombreux aspects du métabolisme mitochondrial de l’EPR ne peuvent être sondés pratiquement que dans un système de culture in vitro. Des progrès significatifs ont été réalisés au cours des dernières décennies vers des cultures d’EPR haute fidélité, au point que les cultures d’EPR les plus soigneusement préparées sont maintenant utilisées pour la thérapie de remplacement cellulaire dans les essais cliniques sur l’homme7. Pour maintenir de telles cultures haute fidélité, l’EPR doit être cultivé sur des substrats particuliers dans des milieux particuliers pendant des mois avant l’expérimentation. Dans ces conditions, les cultures d’EPR sont différenciées et polarisées au maximum, ce qui se rapproche de l’EPR in vivo. Malheureusement, il n’existe actuellement aucun équipement capable de mesurer le métabolisme mitochondrial spécifiquement à partir de l’EPR in vivo. Bien que la surveillance de l’oxygène du réseau capillaire rétinien ait été réalisée in vivo à l’aide de l’oxymétrie par résonance paramagnétique électronique (EPR)8, cela n’est pas possible pour l’analyse RPE. Les différences entre le métabolisme de l’EPR in vivo et in vitro ne sont pas bien décrites, mais il a été démontré que les cultures d’EPR ont une activité mitochondriale élevée, similaire à l’EPR in vivo 3,9, ce qui suggère que des informations significatives sur le métabolisme mitochondrial de l’EPR peuvent être obtenues en utilisant des cultures EPR haute fidélité.
Comme tout métabolisme mitochondrial conduit à la consommation d’oxygène, la mesure des taux de consommation d’oxygène (OCR) de l’EPR est un indicateur fidèle du métabolisme mitochondrial. La mesure de l’OCR dans les cultures d’EPR a été difficile, car les conditions qui favorisent la polarisation et la différenciation maximales de l’EPR empêchent souvent des mesures OCR précises à long terme avec les techniques actuellement disponibles, telles que l’analyseur Seahorse. Dans cet article sur les méthodes, un nouveau dispositif, appelé Resipher (ci-après dénommé « le système »), est présenté, qui permet une mesure continue de l’OCR pendant des semaines dans des EPR cultivés dans des conditions qui favorisent au maximum la polarisation et la différenciation. La facilité avec laquelle l’OCR peut être mesurée par ce système dans des conditions de culture d’EPR qui favorisent au maximum la différenciation et la polarisation de l’EPR est unique parmi les appareils de mesure OCR existants.
Cet article fournit des conseils et des astuces pour l’utilisation du système avec des cultures RPE, suivis d’une démonstration de deux applications particulières. Tout d’abord, l’EPR EMT, imitant la PVR, est déclenchée par l’exposition au facteur de croissance transformant-bêta (TGFβ)1,10,11,12. Le système est utilisé pour surveiller l’évolution du métabolisme de l’EPR au cours du processus EMT. Deuxièmement, le rôle de l’hypoxie dans le métabolisme de l’EPR est exploré à l’aide de ce système. L’hypoxie est un contributeur important à la pathogenèse de la DMLA, car le choriocapillaire s’amincit avec l’âge de13 ou 14 ans. La combinaison de ce système avec des chambres d’hypoxie permet de modéliser le métabolisme mitochondrial de l’EPR altéré avec l’hypoxie subtile qui accompagne le vieillissement. Enfin, un calculateur en ligne utilisant les données Resipher est introduit pour permettre de déterminer si les cultures d’EPR sont dans des conditions hypoxiques, normoxiques ou hyperoxiques. Ces informations sont essentielles pour tirer des conclusions sur le métabolisme de l’EPR à partir d’études de culture in vitro sur l’EPR.
Protocole
Pour les protocoles permettant d’établir des cultures primaires humaines ou iPSC-RPE, voir les références suivantes 15,16,17,18. L’acquisition et l’utilisation de tissus humains pour ces protocoles ont été examinées et autorisées par le Conseil d’examen institutionnel de l’Université du Michigan (HUM00105486).
1. Application générale du système à la culture de l’EPR
- Plaque de cellules souches pluripotentes induites humaines (iPSC) dérivées de l’EPR ou cellules EPR humaines primaires dans des plaques à 96 puits compatibles avec le système.
- En supposant que des cultures matures déjà existantes soient cultivées sur des plaques de culture cellulaire à 24 puits, lavez les cellules avec une solution saline tamponnée au phosphate (PBS) une fois, puis ajoutez 500 μL de trypsine-EDTA à 0,25 % (Table des matériaux). Incuber pendant 10 à 40 minutes dans un incubateur de culture cellulaire, en vérifiant toutes les 5 à 10 minutes jusqu’à ce que les cellules soient arrondies et presque prêtes à se détacher.
- Pipeter doucement le milieu sur les cellules pour les détacher du plastique de culture cellulaire, puis transférer dans 3 fois le volume du milieu de culture cellulaire (1 500 μL par 24 puits), puis faire tourner dans une centrifugeuse à 250 × g pendant 5 min à température ambiante.
REMARQUE : La recette des milieux de culture cellulaire EPR est détaillée dans le tableau 1 et disponible dans les références9, 15, 16, 17 et 18 publiées précédemment. - Réintroduisez des cellules dans des milieux de culture cellulaire EPR avec 15 % de sérum de veau fœtal (FBS) et comptez les cellules à l’aide d’un hémocytomètre.
- Amorcez 74 000 cellules pour chaque puits d’une plaque de 96 puits (généralement 225-300 × 105 cellules/cm2), après revêtement avec des substrats de revêtement de matrice extracellulaire hautement spécialisés (tableau des matériaux) en suivant les instructions du fabricant.
REMARQUE : Les cellules d’amorçage se trouvent uniquement sur une plaque compatible avec Resipher (tableau des matériaux) et uniquement dans des puits correspondant au réseau de sondes sur le couvercle de détection (colonnes 3, 4, 9 et 10 pour le couvercle à 32 canaux ; voir tableau des matériaux). Le couvercle de détection du système est disponible avec des capteurs à 4, 32 ou 96 canaux (voir l’emplacement des capteurs dans la Figure 1A-D), et une gamme de plaques de culture cellulaire standard est compatible. - Étant donné que les puits de bordure sont sujets à l’évaporation, ce qui affecte considérablement la disponibilité de l’oxygène, et par conséquent, les lectures OCR, évitez d’ensemencer des cellules sur tous les puits de bordure (rangées A et H d’une plaque de 96 puits). De plus, ajoutez 200 μL d’eau stérile dans chacun des puits vides de la plaque à 96 puits pour réduire les effets d’évaporation.
- Gardez la plaque sur une surface de table stable pendant 10 minutes pour permettre aux cellules de se déposer ; puis retournez-le dans l’incubateur. Changer le milieu après 24 à 72 h, en le remplaçant par un milieu de culture EPR standard avec 5 % de FBS. Culture pendant au moins 4 semaines, en changeant de support 2 fois par semaine.
REMARQUE : Les cellules sont prêtes pour l’expérimentation lorsqu’elles sont pigmentées, pavées et très compactes (Figure 1E).
- Configuration et acquisition de données avec le système
- Placez le couvercle de détection sur une plaque réceptrice vide de 96 puits, puis remettez-la dans l’incubateur de culture cellulaire. Alignez et montez l’appareil sur le couvercle de détection et connectez-le au Hub via le câble USB fourni. Cela crée un « sandwich » Resipher (Figure 2A).
- Accédez à l’application du site Web Lucid lab (https://lab.lucidsci.com/) et cliquez sur le bouton Nouvelle expérience dans le coin supérieur droit pour créer une nouvelle expérience.
- Nommez le titre de l’expérience et entrez toutes les notes expérimentales pertinentes pour l’étude particulière (par exemple, le numéro de passage de l’iPSC-RPE que l’on utilise).
- Créez des conditions de santé et des groupes de traitement. Par exemple, si vous testez les effets de différentes concentrations sériques dans le milieu sur l’OCR, sélectionnez Sérum dans le nom du groupe, puis entrez les concentrations sériques du milieu à utiliser, en ajoutant d’autres valeurs de pourcentage sérique en cliquant sur le bouton + . Attribuez les puits qui recevront un pourcentage sérique particulier et sélectionnez un motif de couleur pour ces puits. Ajoutez d’autres groupes (par exemple, une variable expérimentale différente à tester) si nécessaire en cliquant sur le bouton Ajouter un groupe .
- Définissez la configuration de la plaque et sélectionnez le périphérique et le style de plaque correspondants. Cliquez sur le bouton Ajouter une plaque et choisissez Appareil ; Sélectionnez le type de plaque. Sélectionnez le traitement et cliquez sur le puits correspondant.
- Cliquez sur le bouton Démarrer maintenant pour démarrer l’expérience. Vérifiez que le voyant sur le site Web et la LED sur le Hub sont verts fixes.
- Laissez le système fonctionner pendant 15 à 60 minutes pour vous assurer que chaque capteur est correctement calibré et détecte avec précision l’oxygène atmosphérique, qui doit être à une concentration de ~200 μM dans un incubateur de culture cellulaire entièrement humidifié avec 5 % de CO2 (Figure 2Bi).
REMARQUE : Si l’un des capteurs présente une réduction de plus de 20 % par rapport à la moyenne des autres capteurs d’une configuration d’incubateur de culture cellulaire standard, envisagez d’exclure ce puits de l’analyse des données, car le capteur est défectueux (Figure 2Bii).
REMARQUE : Si tous les capteurs sont à plus de 20 % de la moyenne des autres capteurs et de l’O2 atmosphérique attendu signalé comme étant de 200 μM dans une configuration d’incubateur de culture cellulaire standard, le couvercle de détection a été utilisé trop de fois ; le remplacer par un nouveau couvercle de détection (Figure 2Biii).
REMARQUE : L’obtention des lectures d’O2 dans l’air avant de commencer une expérience améliore considérablement le dépannage par la suite. Si un puits particulier apparaît comme une valeur aberrante après qu’une expérience a été réalisée et que ce puits avait un capteur qui n’était pas étalonné, le problème peut provenir du capteur, et non de la réplique biologique. - Retirez l’appareil du couvercle de détection (mais ne débranchez pas le câble USB de l’appareil). Placez l’appareil à l’envers dans l’incubateur pour permettre au moteur de l’appareil de se réinitialiser. Ne réutilisez pas l’appareil tant que tous les sons du moteur n’ont pas cessé (environ 20-30 s).
REMARQUE : Il est important de garder l’appareil connecté au Hub via le câble USB à tout moment pendant une expérience, même lorsque l’appareil n’est pas sur le couvercle de détection. Cela permet à l’appareil de surveiller et de générer des rapports en permanence sur l’environnement de l’incubateur de culture cellulaire. - Replacez la plaque avec le couvercle de détection dans le capot de culture cellulaire, avec une plaque à 96 puits contenant les cultures RPE. Modifiez le milieu de la plaque avec des cultures EPR avec le même milieu et le même volume pour tous les puits afin d’obtenir des valeurs OCR de référence pour chaque puits. Assurez-vous de remplir tous les puits sans EPR avec de l’eau stérile pour aider à prévenir l’évaporation.
REMARQUE : En général, nous recommandons 60 à 100 μL de milieux de culture EPR par 96 puits, d’après les données démontrant que des volumes de milieux plus faibles entraînent un épuisement rapide des nutriments, mais que des volumes de milieux plus élevés entraînent une hypoxie involontaire9. - Transférez le couvercle de détection sur la plaque contenant les cultures cellulaires et le couvercle standard (non Resipher) sur la plaque contenant les cultures cellulaires sur la plaque réceptrice vide à 96 puits pour maintenir la stérilité du couvercle standard, ce qui sera nécessaire à divers moments de l’expérience.
- Remettez la plaque contenant les cultures cellulaires et le couvercle de détection dans l’incubateur de culture cellulaire. Une fois de plus, alignez et montez l’appareil sur le couvercle de détection et connectez-le au Hub via le câble USB fourni (en assemblant le « sandwich »). Vérifiez que les indicateurs de l’interface du site Web et du Hub sont tous verts fixes.
REMARQUE : Les données de concentration d’O2 s’afficheront immédiatement, tandis que les données OCR n’apparaîtront qu’après la collecte de suffisamment de données d’O2 , soit environ 1 h. - Laissez l’appareil mesurer l’OCR sur chaque puits pendant au moins 12 à 24 h pour capturer un OCR de base.
REMARQUE : L’OCR de base diffère en fonction de nombreux facteurs, notamment du type de support appliqué aux cellules. Dans la figure 2C, les milieux de culture RPE standard avec différentes quantités de FBS (0 %, 5 %, 15 %) ont des valeurs OCR légèrement différentes. De plus, les cultures avec plus de FBS peuvent maintenir l’activité mitochondriale plus longtemps après l’application du milieu sur les cellules avant que l’OCR ne chute en raison de l’épuisement des métabolites mitochondriaux (côté droit de la courbe). - Une fois qu’une OCR de base est obtenue, répétez les étapes 1.2.8 à 1.2.12, mais changez le milieu sur la plaque avec des cultures EPR dans les conditions expérimentales (généralement, ± un médicament ou une comparaison de différentes conditions de milieu).
REMARQUE : Les modifications de support de routine peuvent également être gérées de la même manière. À chaque fois que l’incubateur de culture cellulaire est ouvert, attendez-vous à une perturbation importante des lectures OCR, car elles dépendent de la température, de l’humidité, des concentrations de CO2 et d’autres facteurs qui sont tous temporairement perturbés par l’ouverture de la porte de l’incubateur. Lors du retrait de l’appareil du couvercle de détection, il n’est pas nécessaire de mettre l’expérience en pause en ligne. Une fois que la porte de l’incubateur de culture cellulaire est ouverte ou que le milieu a été changé sur la plaque sondée, il faut généralement 2 à 4 heures pour rétablir le gradient d’oxygène et commencer à mesurer un OCR précis. - Une fois les données obtenues, soustrayez l’OCR de base pour chaque puits de l’OCR après l’application de la condition expérimentale, afin de déterminer l’OCR Delta déclenché par le traitement.
- Une fois l’expérience terminée, stérilisez et réutilisez le couvercle de détection (qui peut être réutilisé 3 à 5 fois, bien que les performances se dégradent avec une utilisation répétée). Pour stériliser le couvercle de détection, plongez l’ensemble du couvercle dans de l’éthanol à 70 % pendant 10 minutes dans une hotte de culture cellulaire, puis retirez-le de l’immersion et laissez-le reposer dans l’armoire, côté sonde vers le haut (évitez de toucher les pointes délicates de la sonde), et laissez l’éthanol et l’eau s’évaporer totalement avant de placer le couvercle de détection sur une nouvelle plaque réceptrice à 96 puits.
- Prenez des images en fond clair pour normaliser les valeurs OCR en fonction du numéro de cellule.
REMARQUE : L’accumulation de mélanine dans l’EPR facilite la normalisation de l’OCR sur la base d’un simple comptage du nombre de cellules dans des cultures vivantes à l’aide d’un microscope à fond clair.- Retirez l’appareil et placez-le à l’envers, comme à l’étape 1.2.8. Dans le capot de culture cellulaire, remplacez le couvercle de détection sur la plaque de culture RPE par un couvercle standard à 96 puits.
- À l’aide d’un microscope inversé standard, prenez des images en fond clair de chaque puits.
REMARQUE : Gardez la zone d’une image cohérente entre les puits (emplacement relatif dans le puits et grossissement objectif). - Dans la hotte de culture cellulaire, remplacez le couvercle standard à 96 puits de la plaque de culture EPR par le couvercle de détection et remettez-le dans l’incubateur pour une surveillance plus approfondie.
- Comptez le numéro de cellule dans chaque puits à l’aide d’ImageJ ou d’un autre logiciel.
- Normalisez l’OCR en fonction du nombre de cellules dans les différents groupes expérimentaux. Le système signale l’OCR en unités de fmol∙(mm2)-1∙s-1-la consommation par unité de section transversale. Par conséquent, pour normaliser l’OCR par cellule, divisez l’OCR par le nombre de cellules par mm2 (plutôt que par le nombre de cellules par puits). De manière équivalente, multipliez l’OCR rapporté par le système par la section transversale du puits (environ 31 mm2 pour une plaque standard de 96 puits) pour obtenir l’OCR en unités de fmol∙s-1∙well-1.
- Contrôles pour la détermination des paramètres bioénergétiques de l’EPR.
REMARQUE : Pour s’assurer que le système et les cultures d’EPR réagissent aux manipulations mitochondriales de manière prévisible, on peut tester certaines petites molécules bien établies pour cibler des étapes particulières de la phosphorylation oxydative mitochondriale. Ces tests sont analogues aux étapes effectuées dans un test de stress mitochondrial à l’aide de l’analyseur Seahorse10 et fournissent un profil bioénergétique pour les cultures d’EPR.- Cultivez des cellules EPR dans 65 μL de milieux de culture EPR standard avec 5 % de FBS et mesurez l’OCR pendant 24 h pour établir une base de référence.
- Aspirer le milieu de culture et ajouter 65 μL de milieu de culture EPR standard avec 5 % de FBS et avec des découpleurs mitochondriaux (3 μM de cyanure de carbonyle-p-trifluorométhoxyphénylhydrazone [FCCP] ou 500 nM de Bam15, tableau des matériaux). Incuber toute la nuit.
REMARQUE : Les découpleurs mitochondriaux devraient augmenter considérablement l’OCR. S’assurer que l’OCR augmenté n’est pas égal ou proche de l’OCR maximal théorique limité à la diffusion d’environ 275 fmol∙(mm2)-1∙s-1 pour 65 μL de milieu dans des conditions de culture standard. L’OCR maximal pour les différents volumes de médias couramment utilisés est disponible dans la section Discussion. - Après une nuit d’incubation, aspirer le milieu à l’aide de découpleurs mitochondriaux et le remplacer par 65 μL de milieu de culture EPR standard à 5 % de FBS. Observez la plaque pendant encore 24 heures pour déterminer la récupération de l’OCR de l’EPR après l’élimination des médicaments.
- Calculez les effets des découpleurs sur l’OCR en soustrayant l’OCR obtenu après traitement de l’OCR obtenu avant traitement (Figure 3).
REMARQUE : L’augmentation de l’OCR avec l’ajout de découpleurs mitochondriaux signifie la capacité respiratoire mitochondriale de réserve de la cellule19. - Pour mesurer d’autres paramètres mitochondriaux généralement capturés lors du test de stress mitochondrial avec un analyseur Seahorse, effectuez les mêmes étapes ci-dessus avec les composés suivants (Figure 4).
- Utilisez l’oligomycine à 1 μM pour évaluer la respiration liée à l’ATP.
- Utilisez de l’antimycine A et de la roténone à 1 μM pour évaluer toute respiration non mitochondriale.
REMARQUE : Le FCCP, l’oligomycine, l’antimycine A et la roténone peuvent être toxiques pour l’EPR. S’ils induisent une mort cellulaire importante, les mesures OCR ne seront pas précises. Ainsi, les mesures OCR ne doivent être obtenues qu’à un moment où il n’y a pas de mort cellulaire évidente, généralement dans les premières heures suivant l’ajout des médicaments (Figure 4).
2. Mesurer les changements dans le métabolisme mitochondrial dans l’EPR subissant une EMT
- Suivez le même protocole que celui décrit à la section 1.2.
- Pour induire l’EMT après l’obtention des mesures OCR de base, ajoutez du TGF-β2 (10 ng/mL) aux cultures d’EPR et surveillez l’OCR pendant 3 semaines. Rafraîchissez le support avec du TGFβ2 frais tous les 2 à 3 jours (Figure 5).
- Au moins 2 fois par semaine, obtenez des images en fond clair comme indiqué à la section 1.3 pour vous assurer que le nombre de cellules ne change pas. Si le nombre change, normalisez les valeurs OCR en fonction du numéro de cellule dans chaque puits.
REMARQUE : Le même protocole peut être utilisé pour d’autres inducteurs de l’EMT afin de surveiller les changements à long terme de l’OCR tout au long de la période de culture pendant que les cellules sont dans l’incubateur.
3. Mesurer les changements dans le métabolisme mitochondrial dans l’EPR hypoxique
REMARQUE : L’application du système dans des conditions hypoxiques, normoxiques ou hyperoxiques est la même que celle de la section 1.2, à l’exception du maintien du « sandwich » dans une chambre d’hypoxie (table des matériaux) placée dans un incubateur de culture cellulaire standard.
- Percez un trou sur le couvercle de la chambre d’hypoxie pour y monter le câble USB de type c et scellez-le avec du silicone ou du mastic (Figure 6A).
- Assemblez le couvercle de détection avec la plaque de culture cellulaire dans le capot et transférez-les dans la chambre d’hypoxie.
- Installez le « sandwich » (assiette, couvercle de détection, appareil) dans la chambre d’hypoxie. Placez également dans la chambre d’hypoxie une boîte de Pétri avec de l’eau stérile pour aider à maintenir l’humidité. Enfin, placez un capteur portable O2 dans la chambre d’hypoxie (Table des matériaux). La configuration est illustrée à la Figure 6A.
- Scellez la chambre et connectez-la à une bouteille de gaz avec une concentration hypoxique de O2 et une concentration standard de culture cellulaire de CO2 (par exemple, 1 % d’O2 et 5 % de CO2).
- Échangez lentement l’air dans la chambre d’hypoxie avec l’air 1 % O2/5 % CO2 dans la bouteille jusqu’à ce que le capteur d’oxygène indique la concentration d’O2 souhaitée, généralement entre 1 % et 10 %.
REMARQUE : Le capteur portable O2 nécessite du temps pour s’équilibrer, donc le gaz de la bouteille doit être ajouté lentement dans la chambre d’hypoxie. - Fermez les vannes d’entrée et de sortie de la chambre d’hypoxie et transférez toute la chambre dans l’incubateur.
- Configurez et démarrez l’expérience comme indiqué à la section 1.2.
REMARQUE : Le prééquilibrage du milieu dans la chambre d’hypoxie avant l’ajout aux cellules peut réduire le temps nécessaire à la monocouche cellulaire pour subir une véritable hypoxie (Figure 6B).
4. Calcul de la concentration d’O2 au niveau de la monocouche d’EPR pour déterminer si les cellules sont dans des conditions hypoxiques, normoxiques ou hyperoxiques
REMARQUE : Par défaut, le système mesure la concentration d’O2 entre environ 1 et 1,5 mm au-dessus du fond du puits, en supposant qu’une plaque standard est utilisée avec le couvercle de détection recommandé pour la culture monocouche (voir https://lucidsci.com/docs/LucidScientific_Sensing_Lid_Selection_Guide.pdf). Bien que la concentration d’O2 au niveau de la monocouche cellulaire ne soit pas mesurée directement, les données du système peuvent être utilisées pour estimer la concentration d’O2 au niveau de l’EPR. Plus précisément, sachant qu’il existe un gradient d’oxygène entre le haut de la colonne de support, où l’O2 est disponible, et le bas de la colonne du milieu, où l’O2 est consommé, les lois de la diffusion de Fick peuvent être combinées avec le taux d’OCR mesuré pour extrapoler les niveaux d’O2 au niveau de la monocouche cellulaire. Un calculateur pour cette estimation est fourni en ligne : https://lucidsci.com/notes?entry=oxygen_diffusion (et sous la forme d’un cahier interactif open-source à l’https://observablehq.com/@lucid/oxygen-diffusion-and-flux-in-cell-culture, le code source de ce calculateur peut être trouvé à https://github.com/lucidsci/oxygen-diffusion-calculator).
- Une fois que vous avez accédé à la calculatrice, entrez le volume de support en microlitres (par exemple, 100 μL).
- Entrez la concentration d’oxygène saturé à l’interface air-liquide (le haut de la colonne de média au-dessus des cellules). Cette valeur est de ~185 μM à pression atmosphérique standard avec 5% O2 et 95% d’humidité.
REMARQUE : Cela peut également être déterminé à partir de mesures O2 par des sondes système dans des puits de fluide uniquement (sans cellules au fond du puits). - Dans l’entrée Flux, entrez le Flux/OCR signalé par le système.
- Sur la base de ces valeurs, la concentration d’oxygène à l’état d’équilibre à chaque hauteur de la colonne de milieu est calculée et indiquée dans le graphique (figure 7). L’axe des x de la parcelle est la hauteur en mm au-dessus du fond du puits. L’axe des y est la concentration calculée en O2 à cette hauteur (en μM).
- La concentration en O2 au fond du puits (où se trouvent les cellules) est indiquée sur la ligne située sous le graphique.
REMARQUE : En général, on estime que l’EPR in vivo présente une concentration d’O2 de 4 à 9 %. Chaque 10 μM d’O2 dans des conditions atmosphériques normales correspond à environ 1 % d’O2. Ainsi, la normoxie au niveau des cellules est d’environ 40-90 μM de O2. Plus la colonne de support est élevée, plus le O2 sera bas au niveau de la monocouche RPE9. Ainsi, si les cellules semblent être dans des conditions hypoxiques, le volume du milieu peut être réduit. Si les cellules semblent être dans des conditions hyperoxiques, le volume du milieu peut être augmenté.
Résultats
La configuration « sandwich » de l’expérience Resipher est illustrée à la figure 2A. Les couvercles de détection avec 32 sondes correspondant aux colonnes 3, 4, 9 et 10 sur la plaque à 96 puits se trouvent entre la plaque de la cellule et le dispositif. Après s’être connecté au Hub, l’appareil active des moteurs pour déplacer le couvercle de détection vers le haut et vers le bas, en mesurant la concentration de O2 dans la colonne de support à une plage de hauteurs au-dessus de la monocouche de cellule (généralement 1-1,5 mm). Le gradient d’O2 est donc mesuré en continu en enregistrant les concentrations d’O2 à ces différentes hauteurs au-dessus de la monocouche. À partir de mesures de gradient et de l’application des lois de diffusion de Fick, l’appareil calcule automatiquement le flux d’oxygène d’au-dessus de la plage de hauteur de la sonde vers en dessous, qui est le taux de consommation d’oxygène (OCR) de la monocouche de cellule. Le couvercle de détection détecte O2, qu’un puits soit vide (air uniquement), rempli de fluides mais pas de cellules (fluides uniquement) ou que des fluides recouvrent une monocouche de cellules (milieux + cellules). Pour vérifier la qualité du fonctionnement des capteurs dans le couvercle de détection, le couvercle peut d’abord être placé sur une plaque vide (à air uniquement) de 96 puits. Si chaque capteur de chaque puits fonctionne, l’O2 rapporté doit être cohérent les uns avec les autres et proche de 200 μM, ce qui est proche de l’O2 attendu dans l’atmosphère dans des conditions d’incubateur de culture cellulaire standard. La figure 2Bi montre les lectures d’O2 de huit sondes dans l’air atmosphérique (signalées comme la concentration en μM puisque la conversion d’unité par défaut suppose que les sondes sont dans un liquide à 37 °C). Ces lectures sont serrées (±5 %) et proches de 200 μM, ce qui signifie que les huit capteurs fonctionnent bien. Dans la figure 2Bii, le même couvercle de détection que dans la figure 2Bi est utilisé, mais un dispositif différent est déployé. Ici, deux capteurs de l’appareil défectueux sont détectés sur la base de courbes aberrantes (en rouge). Cela suggère que le dispositif est problématique et doit être remplacé (ou que les puits correspondant à des capteurs défectueux doivent être exclus de l’expérience). Dans la figure 2Biii, le même dispositif que la figure 2Bi est utilisé, mais deux couvercles de détection différents sont utilisés. Le couvercle1 (côté gauche) présente une grande variabilité entre les sondes, ce qui suggère que le matériau de détection du couvercle est dégradé, généralement parce que le couvercle a été utilisé trop de fois. Le Lid2 (côté droit) est nouveau, montrant des lectures serrées et regroupées autour de 200 μM. À l’aide du système, on peut analyser l’effet de la supplémentation sérique dans le milieu sur le métabolisme mitochondrial de l’EPR. Comme le montre la figure 2C, l’augmentation des niveaux de supplémentation sérique a déclenché des niveaux d’OCR RPE subtilement plus élevés. Il est important de noter qu’une supplémentation sérique plus élevée a également permis au métabolisme mitochondrial d’être maintenu pendant une période plus longue. Même sans aucune supplémentation sérique, l’OCR est maintenue pendant environ 90 heures avant que l’épuisement des substrats métaboliques mitochondriaux ne déclenche une baisse de l’OCR. Ainsi, des changements de milieu avec 100 μL par 96 puits tous les 3,5 jours (deux fois par semaine) sont suffisants, même avec des milieux sans sérum, pour éviter l’épuisement des nutriments dans les cultures d’EPR matures, hautement différenciées et polarisées.
Les taux d’OCR peuvent varier considérablement d’un puits à l’autre, même dans les cultures où tous les puits semblent avoir le même nombre de cellules et la même morphologie. Ainsi, pour déterminer si les augmentations de l’OCR sont attribuables à une condition expérimentale ou simplement à des différences de base entre les puits, une normalisation est nécessaire. La première étape de la normalisation consiste à s’assurer que le nombre de cellules entre les puits est uniforme, comme il est décrit à la section 1.3 du protocole. Si le nombre de cellules varie d’un puits à l’autre, l’OCR du puits doit être normalisé en fonction du nombre de cellules. En général, pour les cultures d’EPR hautement matures et polarisées, le nombre de cellules entre les puits est remarquablement cohérent9. Même dans de tels scénarios, l’OCR de base entre les puits peut varier, comme le montre la figure 3A. Ici, l’OCR de base, avant tout traitement, varie jusqu’à 100 fmol/mm2/sec entre les puits. En mesurant l’OCR avant le traitement, on peut ensuite déterminer l’OCR Delta déclenché par le traitement expérimental et l’isoler de toute différence d’OCR de base entre les puits. Dans ce cas, les traitements comparent deux découpleurs mitochondriaux (FCCP vs BAM15) avec contrôle du véhicule. Une fois que l’OCR de base est utilisée pour la normalisation, les effets spécifiques du traitement pour les découpleurs par rapport au contrôle deviennent évidents (comparer la figure 3B et la figure 3C). En règle générale, les expériences susceptibles de modifier l’OCR doivent être divisées en au moins deux phases : l’OCR initiale pour établir un OCR de base et une phase de traitement pour déterminer l’OCR Delta.
Pour confirmer que les cultures d’EPR répondent de la manière attendue à la manipulation mitochondriale et établir un profil bioénergétique pour l’EPR, les outils classiques à petites molécules utilisés dans le test de stress mitochondrial sur l’analyseur Seahorse d’Agilent peuvent être utilisés avec le système. Tout d’abord, la capacité mitochondriale de rechange peut être mesurée à l’aide d’un découpleur mitochondrial19. Dans les cultures d’EPR, le découplage mitochondrial BAM15 présente une augmentation beaucoup plus robuste et soutenue de l’OCR par rapport au FCCP (Figure 3C,D), sans induire de toxicité. D’autres paramètres disponibles sur le test de stress mitochondrial de l’analyseur Seahorse, notamment la respiration basale, la respiration liée à l’ATP, les fuites de protons et la capacité respiratoire non mitochondriale, peuvent être calculés à l’aide du système. Alors que l’analyseur Seahorse s’appuie sur des ports d’injection pour délivrer séquentiellement des toxines qui inhibent des complexes de phosphorylation oxydative spécifiques afin de déduire les paramètres ci-dessus, Resipher n’a pas cette capacité. Cependant, chaque toxine mitochondriale peut être ajoutée à des puits séparés, et différents paramètres respiratoires peuvent être calculés en fonction du changement de pli sur les puits traités avec véhicule. La figure 4A compare les différences d’OCR entre les puits traités avec le découplage mitochondrial BAM15, l’inhibiteur de l’ATP synthase oligomycine, l’inhibiteur des complexes I et III antimycine/roténone et le contrôle du véhicule (DMSO). Comme ces toxines peuvent induire la mort cellulaire sur de plus longues périodes et que la mort cellulaire affectera l’OCR, les lectures OCR ont été prises après seulement quelques heures de traitement. La figure 4B montre les différences d’OCR entre le véhicule et les puits traités avec chaque petite molécule mitochondrial active sous forme de graphique à barres, extrapolées à partir des derniers points temporels de la figure 4A. Les valeurs OCR de la figure 4B peuvent ensuite être utilisées pour calculer les paramètres bioénergétiques de la culture d’EPR, comme le montre la figure 4C. Chaque valeur codée en couleur dans le profil bioénergétique de la figure 4C provient de la même valeur codée en couleur sur la figure 4B. Ainsi, des paramètres tels que la respiration liée à l’ATP, la fuite de protons, la capacité respiratoire mitochondriale maximale et la capacité respiratoire non mitochondriale peuvent être estimés à l’aide du système OCR. Un résumé du mode d’action de chaque petite molécule mitochondrial active est présenté à la figure 4D.
Après avoir démontré le protocole de ce système sur des cultures d’EPR et des expériences de contrôle pour tester les profils bioénergétiques des EPR, on peut explorer les applications expérimentales du système à la biologie et à la pathologie de l’EPR. Dans la vitréorétinopathie proliférative (PVR), une affection qui survient après un décollement de la rétine ou un traumatisme oculaire, l’EPR subit une transformation spectaculaire connue sous le nom de transition épithéliale à mésenchymateuse (EMT) par laquelle l’EPR très régulière et semblable à un pavé perd ses adhérences cellule-cellule et se transdifférencie en cellules mésenchymateuses en forme de fuseau contractiles et mobiles 1,2. Cela déclenche des forces contractiles tangentielles sur la rétine qui peuvent induire un décollement de la rétine. Les inducteurs classiques de l’EMT dans l’EPR comprennent le facteur de croissance transformant-bêta (TGFβ) et le facteur de nécrose tumorale-alpha (TNFα)10,11,12. Parmi les trois isoformes du TGFβ chez les mammifères, le TGFβ2 est l’inducteur le plus important et le plus puissant de l’EMT dans la rétine1, provoquant une élongation cellulaire distincte des cellules EPR hexagonales caractéristiques et une expression accrue des marqueurs mésenchymateux. La réponse EMT induite par le TGFβ2 dans l’EPR s’accompagne d’une suppression de la respiration mitochondriale et d’une augmentation ultérieure de la capacité glycolytique, comme démontré précédemment à l’aide d’un Seahorse XFe9610. Bien que le Seahorse fournisse des profils bioénergétiques en temps réel, il ne permet pas de surveillance OCR à long terme. Ainsi, dans la première application du système OCR dans cette étude (Figure 5), la respiration mitochondriale a été suivie pendant trois semaines alors que l’EMT était induite avec le TGFβ2 dans l’EPR, permettant une modélisation in vitro de la reprogrammation métabolique qui se produit pendant la PVR. Contrairement à l’analyse ponctuelle de l’EPR EMT à l’aide d’un analyseur Seahorse, l’état métabolique de l’EPR subissant une EMT peut être suivi pendant plusieurs semaines. Il est évident que l’exposition répétée de l’EPR au TGFβ2 provoque un déclin progressif continu du métabolisme mitochondrial. Des données antérieures utilisant l’analyseur Seahorse XF ont indiqué que le TGFβ2 réduisait significativement la capacité respiratoire maximale à 24h et 72h, mais n’affectait pas les niveaux ocr basaux10. L’OCR basal inchangé avec TGFβ2 jusqu’à 72 h est corroboré par le système OCR utilisé dans cette étude (Figure 5). Cependant, les données à plus long terme du système OCR dans cette étude révèlent qu’après 5 jours d’exposition au TGFβ2, les niveaux d’OCR basaux commencent à diminuer, la baisse de l’OCR basal devenant la plus évidente à 20 jours. Ces résultats mettent en évidence des changements à long terme dans l’OCR basal qui n’ont pas été capturés avec les points temporels précédents de Seahorse.
Dans une deuxième application, le système peut être utilisé pour surveiller le métabolisme mitochondrial de l’EPR dans des conditions hypoxiques. Comme la DMLA est liée à l’amincissement de la choriocapillaire et à l’hypoxie13,20, la compréhension des ajustements de l’EPR à l’hypoxie sous-toxique devrait fournir des informations sur la pathogenèse de la DMLA. Pour soumettre les cultures d’EPR à une hypoxie contrôlée tout en surveillant le métabolisme mitochondrial, le système peut être placé dans une chambre d’hypoxie qui, à son tour, est placée dans un incubateur de culture cellulaire. Pour faciliter cette configuration, un petit trou est percé dans le couvercle d’une chambre d’hypoxie, permettant au câble USB du système d’atteindre l’intérieur de la chambre d’hypoxie. Le trou est scellé avec du mastic ou du silicone (Figure 6A). Un capteur d’O2 portable peut être placé dans la chambre d’hypoxie pour surveiller les niveaux atmosphériques d’O2 et s’assurer que l’étanchéité de la chambre d’hypoxie reste intacte. À l’aide de ce système, le taux d’équilibre entre la concentration atmosphérique en O2 et la concentration en O2 dans le milieu peut être déterminé. Sur la figure 6B, un milieu équilibré avec l’O2 atmosphérique est placé dans des puits individuels sur une plaque de 96 puits, sans aucune cellule, et introduit dans une chambre d’hypoxie contenant seulement 5 % d’O2 (~50 μM O2). Au fil du temps, le milieu de chaque puits s’équilibre avec la nouvelle concentration atmosphérique. Plus la hauteur de la colonne de support est élevée (plus le volume est important), plus l’équilibrage prend du temps. Avec 65 μL de milieu dans un puits d’une plaque de 96 puits, l’équilibre avec l’O2 atmosphérique prend environ 5 h. Cependant, le temps d’équilibre prend plus de 10 h lorsque le volume du support est de 100 à 200 μL. Ces résultats soulignent l’importance de pré-équilibrer les milieux avec la concentration atmosphérique souhaitée d’O2 avant d’appliquer le milieu sur des cellules lors d’une expérience d’hypoxie.
Pour les expériences d’hypoxie, il est important de s’assurer que les niveaux d’O2 dans la chambre d’hypoxie restent stables. Le fait de garder l’un des puits de bord de la plaque à 96 puits exempt de cellules ou de milieux permettra à l’un des capteurs du couvercle de détection de surveiller en permanence l’O2 atmosphérique. Dans la figure 6C, une telle surveillance avec un seul puits exposé à l’air montre que la chambre d’hypoxie a une fuite lente, de sorte qu’au bout de 30 heures, les cellules sont proches des concentrations atmosphériques d’O2 . Comme contrôle final pour les expériences d’hypoxie, il est important de déterminer si la solubilité de l’O2 diffère entre les milieux « frais » et « conditionnés ». Si la solubilité diffère entre les milieux initialement placés sur les cellules et les milieux qui sont sur les cellules depuis un certain temps, alors le gradient d’O2 dans la colonne des milieux au-dessus des cellules différera en tant que « conditions » des milieux avec le temps sur les cellules. Ceci, à son tour, affecte le calcul de l’OCR. Sur la figure 6D, une plaque de 96 puits avec des puits vides (« air »), des puits sans cellules mais avec des milieux frais (« nouveaux milieux »), et des puits sans cellules mais avec des milieux préalablement incubés sur des cultures EPR pendant 48 h (« milieux conditionnés ») a d’abord été placée dans l’O2 atmosphérique pour permettre une équilibrage complet. La plaque a ensuite été placée dans une chambre d’hypoxie à environ 3-4% O2 (30-40 μM) et laissée s’équilibrer. Enfin, la plaque a de nouveau été ramenée à des concentrations atmosphériques d’O2 . Les courbes de concentration en O2 pour les milieux neufs et les milieux conditionnés sont identiques, ce qui démontre que la solubilité de l’O2 entre les milieux neufs et conditionnés est identique, ce qui confirme que les changements dans la composition des milieux au fil du temps à mesure que les nutriments sont consommés et que les sous-produits sont sécrétés dans les milieux ne modifient pas de manière significative la solubilité de l’O2 et n’affectent donc pas par inadvertance l’OCR.
Comme la hauteur de la colonne de média au-dessus des cellules affecte considérablement la disponibilité de l’O2 au niveau de la monocouche9 de l’EPR, il est important de déterminer si un volume de média particulier induit une hypoxie, une normoxie ou une hyperoxie au niveau de la monocouche de l’EPR. À l’aide des mesures du système, combinées à une calculatrice en ligne (https://lucidsci.com/notes?entry=oxygen_diffusion (et sous la forme d’un cahier interactif open-source à https://observablehq.com/@lucid/oxygen-diffusion-and-flux-in-cell-culture) - code source de cette calculatrice à https://github.com/lucidsci/oxygen-diffusion-calculator), la concentration d’oxygène au niveau de la monocouche EPR peut être estimée. La figure 7 montre une capture d’écran de la calculatrice interactive. L’EPR in vivo voit généralement une concentration d’O2 de 4 à 9 %, ce qui se traduit par une concentration molaire d’O2 de ~40 à 90 μM.

Figure 1 : Emplacement des capteurs sur différents couvercles de détection, disposition des plaques pour le couvercle de détection à 32 canaux et morphologie de l’EPR. Puits qui correspondent à l’emplacement des capteurs dans les différents couvercles de détection pour les options de couvercle (A) 4 canaux, (B) 32 canaux et (C) 96 canaux. (D) Disposition de plaque recommandée pour le couvercle du capteur à 32 canaux. En raison des effets de bord, les cellules ne doivent pas être ensemencées dans les puits de bord et à la place, le milieu doit être placé dans ces quatre puits (violet). Les cellules sont ensemencées dans des puits orange. Les puits restants doivent être remplis d’eau stérile (bleu) pour éviter les effets d’évaporation. (E) Culture d’EPR mature avec des cellules pigmentées hexagonales, serrées. Barre d’échelle = 20 μm. Abréviations : EPR = épithélium pigmentaire rétinien. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Configuration du système Resipher « sandwich » et contrôle de la qualité. (A) Le couvercle de détection remplace un couvercle à plaque standard à 96 puits et ses sondes sont insérées dans les puits correspondants. L’appareil en noir repose fermement sur le couvercle de détection via des aimants. Les images sur le côté gauche de la figure montrent l’appareil et le couvercle de détection dans leur orientation correcte. Cependant, pour les photos du côté droit, l’appareil et le couvercle de détection ont été tournés sur leurs côtés pour mieux afficher leur sous-profil. (B) Contrôle de la qualité des couvercles de détection et du dispositif. (i,ii) Le même couvercle de détection est placé dans une plaque réceptrice vide de 96 puits (air uniquement), et deux dispositifs différents sont placés séquentiellement sur le couvercle de détection. (i) Le premier dispositif présente des données provenant de huit sondes avec une faible variabilité et des lectures d’O2 ~ 200 μM, attendues pour l’O2 atmosphérique. (ii) Le deuxième dispositif présente les données de huit sondes dans lesquelles deux sondes lisent de manière anormale (points rouges). (iii) Un dispositif a été utilisé avec une plaque vide de 96 puits (air seulement), mais avec deux couvercles de détection différents testés séquentiellement. Les données de 16 sondes ont été tracées, démontrant une grande variabilité pour Lid1 (côté gauche) et une faible variabilité pour Lid2 (côté droit). (C) Des parcelles OCR de cultures d’EPR dans des milieux de 100 μL avec différentes quantités de sérum ont été surveillées pendant 120 h. Les cellules EPR dans les milieux libres de sérum (magenta) ont un OCR plus faible, l’OCR diminuant plus tôt que les milieux contenant des quantités plus élevées de sérum. Abréviations : OCR = taux de consommation d’oxygène ; EPR = épithélium pigmentaire rétinien. Veuillez cliquer ici pour voir une version agrandie de cette figure.
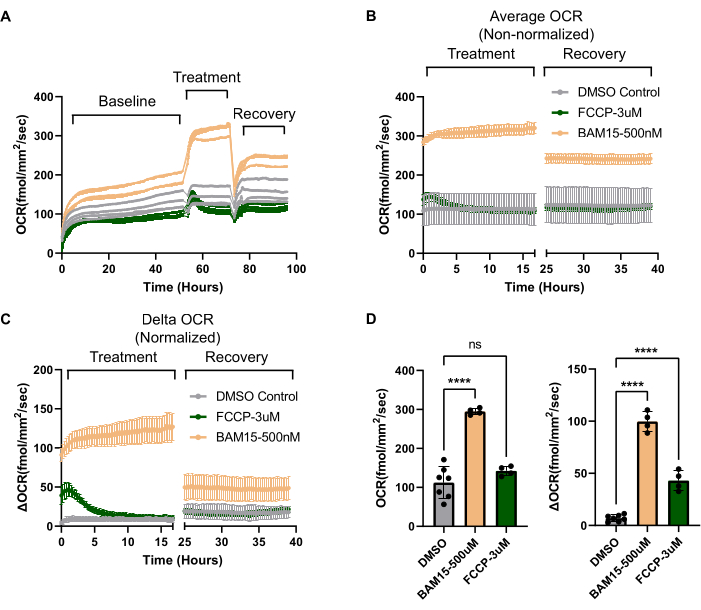
Figure 3 : Effets à long terme des découpleurs mitochondriaux démontrés de manière unique avec le système OCR. (A) Données OCR brutes au fil du temps (heures) mettant en évidence l’OCR initial (établi sur 50 h), la phase de traitement (~20 h après l’ajout des médicaments) et la phase de récupération (~20 h après l’élimination du médicament). L’EPR a été traitée avec deux découpleurs mitochondriaux différents, FCCP (3 μM, N = 4) ou BAM15 (500 nM, N = 4) et comparée au contrôle du véhicule DMSO (0,6 %, N = 7). Chaque trace représente un seul puits. (B) OCR moyen de tous les puits pour chaque condition en (A) tracé pour les phases de traitement et de récupération. Sans normalisation (l’axe Y est l’OCR absolue), il est difficile de voir l’ampleur de l’effet du traitement avec FCCP. (C) Lorsque chaque trace OCR est soustraite de son OCR avant le traitement, un Delta OCR peut être calculé (tracé sur l’axe Y). Cela permet d’isoler les effets du traitement sur l’OCR, malgré la variabilité de l’OCR de base entre les puits. En utilisant Delta OCR, l’effet de FCCP devient évident. Il devient également évident à quel point l’effet de la FCCP sur le découplage mitochondrial est de courte durée, par rapport à BAM15. Enfin, il devient évident que, par rapport au FCCP, l’exposition à BAM15, même après que le médicament a été lavé, crée un nouvel état « adapté » d’OCR plus élevé et soutenu. (D) Les tracés en (B) et (C) sont affichés sous forme de graphique à barres, ce qui permet une comparaison directe entre les données non normalisées (graphique de gauche, à partir de (B)) et normalisées (Delta OCR, graphique de droite, à partir de (C)). Abréviations : OCR = taux de consommation d’oxygène ; EPR = épithélium pigmentaire rétinien ; FCCP = cyanure de carbonyle-p-trifluorométhoxyphénylhydrazone ; DMSO = diméthylsulfoxyde de méthyle. Veuillez cliquer ici pour voir une version agrandie de cette figure.
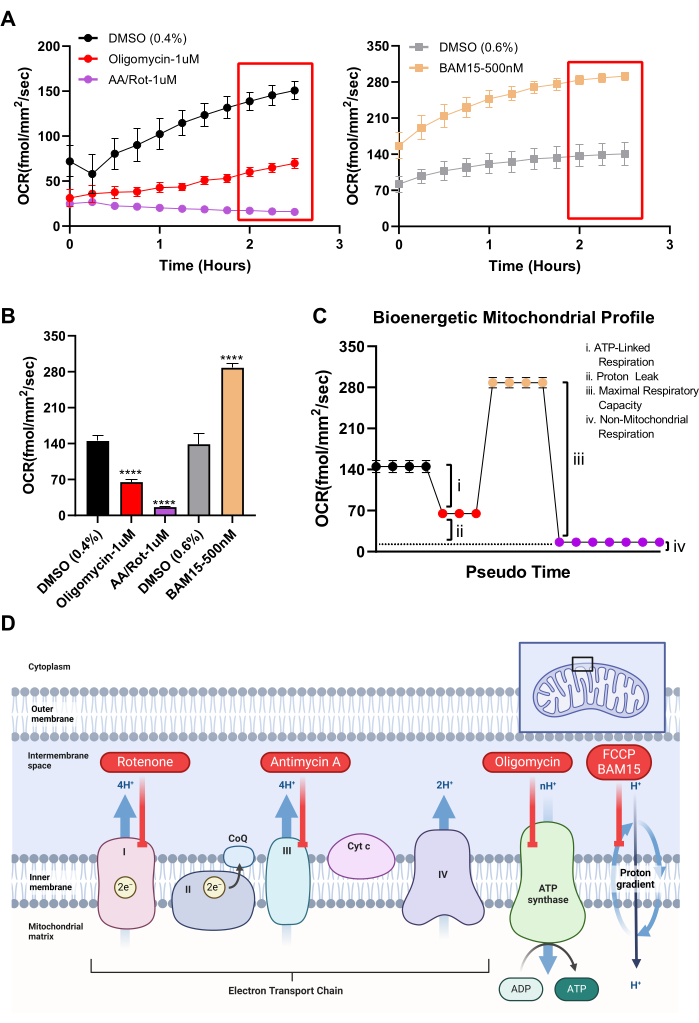
Figure 4 : Utilisation du système OCR pour calculer les paramètres de la bioénergétique mitochondriale. (A) En utilisant les mêmes petites molécules qui font partie du test de stress mitochondrial de l’analyseur Seahorse, l’OCR sur les 2,5 premières heures de la phase de traitement est illustré pour les cultures RPE. (Graphique de gauche) L’inhibiteur de l’ATP-synthase oligomycine (N = 3), les inhibiteurs complexes I et III antimycine A/roténone (N = 7), ou véhicule, DMSO 0,4 % (N = 4). (Graphique de droite) Le découpleur mitochondrial BAM15 (N = 4) ou véhicule DMSO 0,6% (N = 4). (B) Moyenne des trois derniers points temporels pour chaque groupe de traitement (cases rouges en (A)). (C) Représentation d’un profil bioénergétique simulé du test de stress mitochondrial de l’analyseur Seahorse basé sur les données Resiper. Les points de données codés en couleur sur le graphique simulé correspondent aux graphiques à barres codés en couleur dans (B). Tous les paramètres respiratoires peuvent être calculés à l’aide des données Resipher et de la sélection de petites molécules. (D) Schéma du mode d’action des réactifs utilisés à la figure 3 et à la figure 4. Abréviations : OCR = taux de consommation d’oxygène ; EPR = épithélium pigmentaire rétinien ; DMSO = sulfoxyde de diméthyle ; AA = antimycine A ; Pourriture = roténone. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Suivi OCR à long terme de l’EPR subissant une transition épithéliale à mésenchymateuse stimulée par le TGFβ2. Des cultures primaires d’EPR humaines matures ont été traitées avec 10 ng/mL de TGFβ2 ou un témoin de véhicule tous les 2 à 3 jours. (A) Les mesures OCR en temps réel ont été surveillées pendant 3 semaines avec (B) Quantification de Delta OCR au fil du temps (Jour 19-Jour 6). N = 6-7 puits par condition, test t non apparié, * P < 0,05. Abréviations : OCR = taux de consommation d’oxygène ; EPR = épithélium pigmentaire rétinien ; TGFβ2 = facteur de croissance transformant bêta-2. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 6 : Surveillance de la réponse mitochondriale de l’EPR à l’hypoxie. (A) Le « sandwich » du système OCR a été assemblé dans une chambre d’hypoxie et le câble USB a été connecté au Hub via un trou percé dans le couvercle et scellé avec de la graisse ou du mastic silicone. Également dans la chambre d’hypoxie se trouve une boîte de Pétri d’eau stérile pour maintenir l’humidité et un capteur portable O2. L’ensemble du système a été laissé dans un incubateur de culture cellulaire après que l’atmosphère de la chambre d’hypoxie ait été remplacée par une concentration plus faible d’O2. (B) Le milieu met du temps à s’équilibrer aux concentrations d’O2 au-dessus de l’interface air-liquide. Différents volumes de milieux EPR équilibrés à l’O2 atmosphérique ont été ajoutés dans des puits vides (sans cellules) d’une plaque de 96 puits. La chambre d’hypoxie a ensuite été ajustée à 5 % (50 μM) d’O2. La concentration d’O2 dans le milieu de chaque puits a été surveillée pendant 20 h. Les puits avec des volumes de média plus élevés s’équilibrent à 5 % d’O2 plus lentement que les volumes de média plus faibles. N = 8. (C) Le système peut être utilisé pour détecter les fuites d’air dans la chambre d’hypoxie. La chambre d’hypoxie a été configurée avec 1 % d’O2, et le couvercle de détection a été placé au-dessus de puits vides (à l’air seulement). Les orifices d’entrée et de sortie de la chambre d’hypoxie ont été laissés ouverts, et l’atmosphère de la chambre d’hypoxie s’est rééquilibrée aux niveaux atmosphériques d’O2 sur ~90 h. (D) Les nouveaux milieux et les surnageants (« milieux conditionnés ») ont une solubilité O2 similaire. Cela élimine le problème de la solubilité différentielle de l’O2 dans les milieux lorsque sa composition change pendant la culture, ce qui aurait un impact sur la cohérence des lectures OCR au fil du temps. Le même volume (100 μL) de milieux frais (« nouveaux milieux ») ou de milieux incubés pendant 48 h sur des cellules (« milieux conditionnés ») a été ajouté dans des puits d’une plaque sans cellules. Le système OCR « sandwich » a d’abord été soumis à des niveaux atmosphériques d’O2, puis transféré dans la chambre d’hypoxie (3-5 % d’O2 ou 30-50 μM), puis transféré à nouveau à des niveaux atmosphériques d’O2. Les niveaux d’O2 dans l’air ont été surveillés par des sondes dans des puits vides (uniquement à l’air). La variation de la concentration en O2 lors de chaque transition entre les deux types de milieux est identique, ce qui montre que la solubilité de l’O2 entre les deux types de milieux est identique. N = 6. Abréviations : OCR = taux de consommation d’oxygène ; EPR = épithélium pigmentaire rétinien. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 7 : Concentrations d’O2 au niveau de la monocouche EPR déterminées à l’aide d’un calculateur en ligne. Capture d’écran de la calculatrice en ligne (https://observablehq.com/@lucid/oxygen-diffusion-and-flux-in-cell-culture). Voir le texte pour plus de détails sur la façon dont le calculateur peut être utilisé pour déterminer la quantité d’oxygène disponible sur la monocouche EPR. Abréviations : EPR = épithélium pigmentaire rétinien. Veuillez cliquer ici pour voir une version agrandie de cette figure.
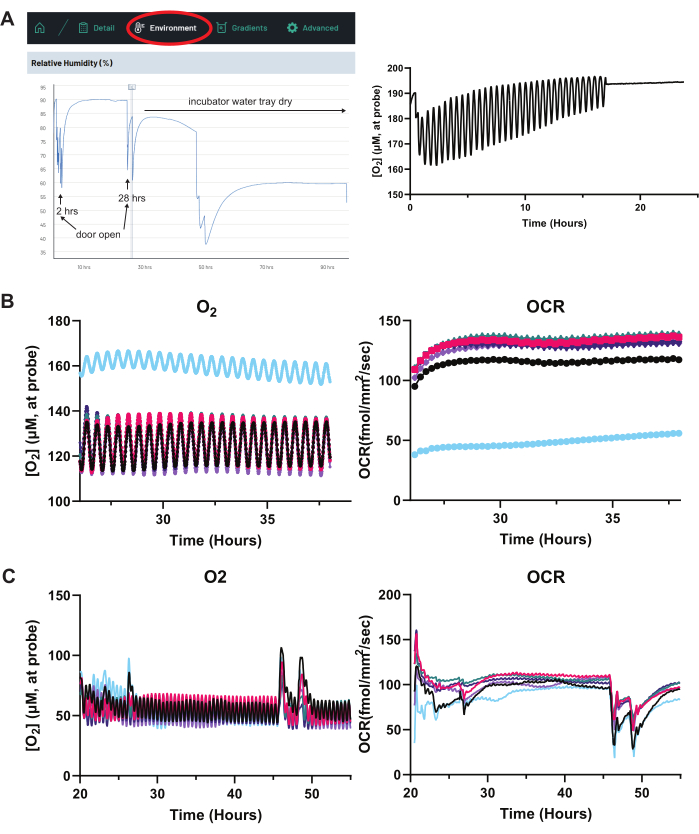
Figure 8 : Expériences de dépannage : évaporation, cellules malsaines et fluctuations excessives de l’OCR. (A) Évaporation excessive. En cas d’irrégularité dans la trace OCR d’un puits, commencez par cliquer sur l’onglet Environnement de l’interface en ligne du système (à gauche). Ici, l’humidité, la température et la pression atmosphérique peuvent être surveillées. L’ouverture de l’incubateur de culture cellulaire perturbera temporairement l’humidité (voir 2 h et 28 h sur la trace). Cependant, à partir de 30 h, la trace d’humidité diminue constamment avec le temps, même sans ouverture de porte. Cela suggère que le bac à eau de l’incubateur de culture cellulaire est sec. Toute perturbation de l’humidité ou de la température modifiera les lectures OCR. (À droite) Une faible humidité exacerbe l’évaporation. Dans la trace de droite, un puits subissant une évaporation rapide présente des différences de concentration en O2 de plus en plus faibles entre le haut et le bas de l’excursion de la sonde du capteur (1 mm à 1,5 mm au-dessus de la monocouche EPR). Cela suggère que l’extrémité de la sonde est proche de l’interface air-média. Lorsque la concentration d’O2 s’aplatit à une valeur proche des concentrations atmosphériques connues d’O2 (~200 μM), cela suggère que l’extrémité de la sonde est entièrement hors de la colonne de support et n’échantillonne que de l’air. Les relevés OCR ne sont plus valides et ces puits doivent être changés immédiatement. (B) Cellules malsaines. Le tracé de la trace d’O2 et d’OCR de chaque puits individuel permet d’identifier un puits aberrant, généralement causé par le fait que les cellules sont en mauvaise santé. Ici, le puits désigné par la trace bleue est malsain. N = 6 puits. (C) Ouvertures des portes de l’incubateur et changement de support. Les changements d’humidité atmosphérique, de température, de pression et de CO2 lors de l’ouverture de la porte de l’incubateur, ainsi que tout changement de média, perturbent temporairement le gradient d’équilibre O2 établi dans le puits, provoquant des pics aberrants d’O2 et d’OCR au début et à la fin de ces graphiques. N = 6 puits. Abréviations : OCR = taux de consommation d’oxygène. Veuillez cliquer ici pour voir une version agrandie de cette figure.
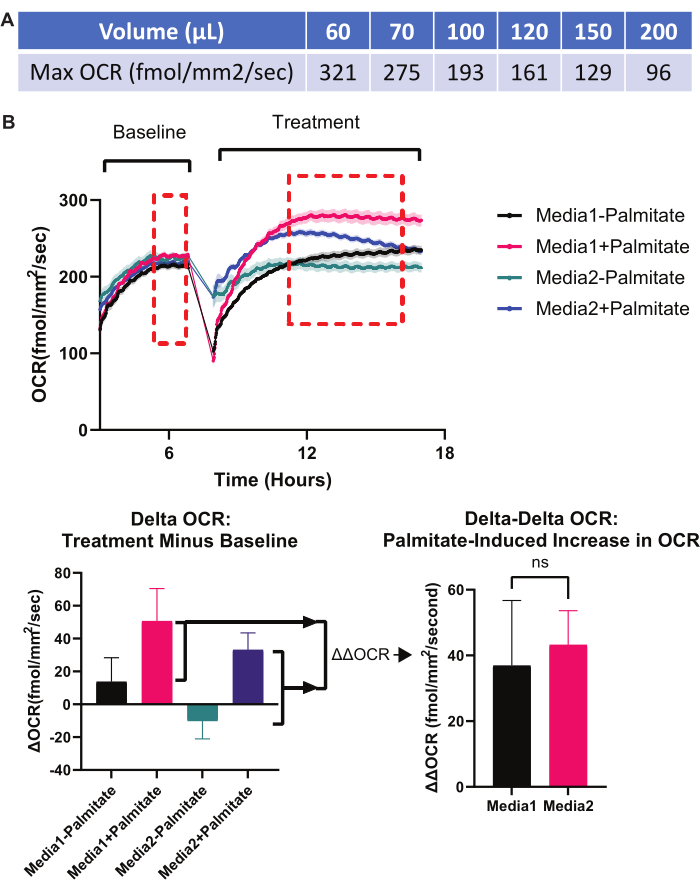
Figure 9 : Expériences de dépannage : volume de support et normalisation. (A) Le volume du support limite l’OCR maximum. Étant donné que des volumes de support plus élevés entraînent une disponibilité plus faible de l’O2 au niveau de la monocouche RPE, chaque volume de support dans un seul puits d’une plaque de 96 puits a un OCR maximal. Au-dessus de cet OCR,l’O2 au niveau des cellules est essentiellement de 0% (anoxique). Ainsi, si l’on est proche de l’OCR théorique maximum pour un volume de support donné, il est utile de réduire le volume du support, de sorte qu’aucun « effet plafond » OCR n’est observé au cours de l’expérience. L’OCR théorique maximal réalisable pour chaque volume de support (dans une plaque standard de 96 puits) est indiqué. Toutes les valeurs ont été calculées à l’aide du calculateur en ligne dont il est question à la section 4 du protocole et qui est illustré à la figure 7. (B) Normalisation appropriée. Pour les expériences où l’on s’intéresse à l’effet d’une variable sur la capacité d’une autre variable à induire le métabolisme mitochondrial, un dispositif expérimental OCR Delta-Delta est idéal. Dans la configuration en (B), la capacité de deux milieux différents à favoriser la β-oxydation du palmitate est testée. En cas de β-oxydation de l’acide gras, il devrait y avoir une augmentation significative de l’OCR mitochondrial. Certains milieux peuvent favoriser la β-oxydation du palmitate mieux que d’autres. Sur le côté gauche du graphique OCR, deux milieux différents (Média 1 vs. Média 2) sont introduits dans des puits parallèles de cultures EPR. L’OCR de base est obtenu. Ensuite, le palmitate est ajouté à la moitié des puits contenant chaque type de milieu. Cela crée quatre conditions : Média 1 - palmitate, Média 1 + palmitate, Média 2 - palmitate, Média 2 + palmitate. La réponse de l’OCR à chacune de ces nouvelles conditions médiatiques est enregistrée. Ensuite, les valeurs OCR après les traitements expérimentaux (moyenne de la case en pointillés rouges sous la phase de traitement) sont soustraites des valeurs OCR de base avant le traitement (moyenne de la case en pointillés rouges sous la phase de base), créant ainsi des graphiques à barres Delta OCR (en bas à gauche). Enfin, la capacité du média 1 par rapport au média 2 à favoriser la β-oxydation est déterminée dans un graphique à barres OCR Delta-Delta (en bas à droite). Ici, la différence entre « Delta OCR du média 1 + palmitate » et « Delta OCR du média 1 - palmitate » est calculée et comparée à la différence entre « Delta OCR du média 2 + palmitate » et « Delta OCR du média 2 - palmitate ». Ce type de configuration expérimentale et de normalisation isole les effets du Média 1 par rapport au Média 2 sur la promotion de la β-oxydation ; la structure générale de cette normalisation est applicable à toute expérience où l’on mesure l’effet d’une variable sur la capacité d’une autre variable à modifier l’OCR. Abréviations : OCR = taux de consommation d’oxygène ; EPR = épithélium pigmentaire rétinien. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : La recette des milieux de culture cellulaire EPR. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
Le métabolisme mitochondrial de l’EPR joue un rôle essentiel dans la pathogenèse des maladies rétiniennes cécitantes courantes, notamment la DMLA et la PVR. La modélisation in vitro du métabolisme mitochondrial de l’EPR permet d’isoler son état métabolique de ceux des tissus environnants, tout en soumettant le tissu à différentes agressions simulant la maladie de manière contrôlée. Une telle modélisation in vitro du métabolisme mitochondrial de l’EPR a été facilitée par l’avènement de cultures primaires humaines de haute fidélité et de cultures iPSC-EPR qui atteignent l’état de différenciation et la polarité nécessaires pour se rapprocher étroitement de l’EPR in vivo. Bien que la surveillance du taux de consommation d’oxygène (OCR) dans de telles cultures soit une méthode idéale pour surveiller le métabolisme mitochondrial, la configuration de la surveillance OCR empêche souvent la culture de cultures dans les conditions nécessaires à l’EPR hautement polarisée et entièrement différenciée. Ici, nous décrivons Resipher comme un nouveau système de surveillance du métabolisme mitochondrial de l’EPR par le biais d’une évaluation en temps réel et à long terme de l’OCR dans des cultures d’EPR hautement différenciées et polarisées.
D’autres méthodes ont été utilisées pour surveiller l’OCR dans l’EPR. L’analyseur Seahorse XF est conçu pour surveiller l’OCR sur de courtes périodes de temps avec des manipulations aiguës de la fonction mitochondriale. Cependant, les dosages Seahorse nécessitent que les cellules soient cultivées sur des plaques personnalisées, ce qui n’est pas propice à la prise en charge d’une polarité et d’une différenciation élevées de l’EPR21. Calton, Vollrath et leurs collègues ont adapté le Seahorse pour l’évaluation des cultures d’EPR hautement différenciées cultivées sur des supports microporeux en coupant la membrane microporeuse en morceaux et en les plaçant dans la plaque Seahorse, mais cette méthode ne fonctionne toujours que pour la surveillance à court terme des cultures, car les cellules n’ont pas accès au CO2 Au cours de l’essai, l’essai ne peut être effectué que dans des conditions non stériles, et la coupe du support microporeux induira la mort et le dysfonctionnement de la partie de bord du support microporeux au fil du temps22. Le Barofuse est un système de perfusion sans pompe dans lequel des bandes d’EPR cultivées sur des supports microporeux peuvent être chargées dans le système de perfusion, ce qui permet de surveiller l’OCR en temps réel23. À l’instar de la configuration modifiée pour le Seahorse par Calton, Vollrath et leurs collègues, les principales limites de l’évaluation de l’EPR avec Barofuse sont la nécessité de couper les supports microporeux et la nature à court terme et finale de l’essai. L’Oxygraph est également conçu pour les changements cinétiques rapides de l’OCR en réponse aux manipulations métaboliques24. Il n’a pas été utilisé pour les cultures EPR et souffrirait des mêmes limites pour l’évaluation des cultures EPR hautement polarisées et différenciées que l’hippocampe et le Barofuse.
Contrairement à ces autres méthodes, le système OCR décrit ici permet de surveiller l’OCR pendant que l’EPR est cultivé dans des plaques/substrats de culture cellulaire standard dans des conditions standard avec des milieux EPR standard dans un incubateur de CO2 de culture cellulaire standard, ce qui permet de sonder facilement et facilement les cultures d’EPR hautement différenciées et polarisées. En plus de générer des données sur les modifications de l’OCR en temps réel par le système, ces mêmes cellules peuvent être utilisées pour des analyses supplémentaires à la fin de la période de surveillance de l’OCR. Cela permet de réaliser des expériences très souples où l’effet d’une intervention sur l’OCR est testé à plusieurs reprises sur une période de plusieurs semaines (par exemple, l’induction de l’EMT RPE dans la figure 5) ou où la récupération à long terme d’une intervention peut être testée (par exemple, la récupération après le découplage mitochondrial dans la figure 3) ou où les changements métaboliques en cascade en réponse à une intervention initiale peuvent être surveillés (par exemple, - effets du sérum sur l’OCR une fois que les nutriments du milieu sont épuisés dans la figure 2). Le système OCR de cet article est particulièrement pratique pour mesurer l’OCR dans les cultures d’EPR hautement polarisées et différenciées. De telles cultures sont fonctionnellement postmitotiques, avec un tassement serré et une forte pigmentation. Cela signifie que chaque puits contient des numérosde cellule 9 presque identiques, évitant ainsi la nécessité de normaliser l’OCR en numéro de cellule pour la plupart des expériences. Même lorsqu’il est nécessaire de normaliser le nombre de cellules, la forte pigmentation de l’EPR et les bordures cellulaires bien délimitées permettent à la normalisation de se produire avec un comptage cellulaire simple non invasif basé sur l’imagerie en fond clair. Pour les expériences impliquant une prolifération indifférenciée de l’EPR et de l’EPR, comme les expériences EMT de la figure 5, il devient nécessaire de normaliser l’OCR par puits en fonction du nombre de cellules pour savoir ce qu’est l’OCR « par cellule ».
En dehors du domaine rétinien, le système a été utilisé pour étudier la respiration cellulaire et l’activité métabolique dans divers types de cellules, y compris les cellules des îlots pancréatiques25, le muscle squelettique26, le cancer de l’ovaire27, les cardiomyocytes28 et même des organismes entiers, tels que C. elegans29. La surveillance en temps réel et à long terme de l’OCR à l’aide du système OCR décrit ici a révélé des informations clés sur les changements bioénergétiques et le dysfonctionnement mitochondrial dans ces divers modèles de maladies.
Le système Resipher comporte des limitations. La méthode de détermination de l’OCR est fondamentalement différente de celle de l’analyseur Seahorse, du BaroFuse ou de l’Oxygraph. Il nécessite l’établissement d’un gradient O2 pour calculer l’OCR, et l’établissement du gradient peut prendre des heures. Ainsi, les expériences cinétiques rapides, possibles dans les autres systèmes, ne sont pas possibles avec ce système. De plus, contrairement aux autres systèmes, Resipher ne dispose pas de ports d’injection pour l’administration rapide de petites molécules ou la collecte de métabolites. Au lieu de cela, le couvercle de détection doit être retiré dans une hotte de culture cellulaire, les médicaments doivent être ajoutés ou les milieux remplacés, ainsi que la collecte manuelle des métabolites à analyser.
Pour établir et résoudre les problèmes d’une expérience à l’aide du système OCR décrit dans ce document, il est recommandé de procéder comme suit. Tout d’abord, minimisez l’évaporation. Rappelez-vous que le gradient O2 dans la colonne média au-dessus de la monocouche RPE dépend fortement de la hauteur de la colonne9 et que le système calcule l’OCR en fonction du gradient O2 . Ainsi, s’il y a un abaissement marqué de la colonne de média pendant l’expérience en raison de l’évaporation, le gradient O2 changera, affectant les lectures OCR. Il est essentiel de garder le bac à eau de l’incubateur plein, mais il est également suggéré de placer de l’eau dans tous les puits inutilisés de la plaque de 96 puits. De plus, les puits au bord de la plaque ont des taux d’évaporation plus élevés, de sorte que ces puits sont laissés vides de cellules dans les expériences typiques présentées ci-dessus. Le capteur d’humidité du système OCR peut aider à surveiller l’humidité de l’incubateur. Sur la figure 8A, la baisse d’humidité pendant l’expérience suggère que le bac à eau de l’incubateur est vide. À mesure que l’évaporation s’accélère dans le puits, les lectures de concentration d’O2 pour le puits augmentent et finissent par « stagner » à la concentration atmosphérique d’O2 (~200 μM) (figure 8A).
Deuxièmement, vérifiez que le « sandwich » du système OCR est correctement assemblé. Les points de contact métalliques au bas de l’appareil et en haut du couvercle de détection doivent être propres, sans débris pour empêcher un contact complet. L’appareil et le couvercle de détection doivent être fixés complètement au ras et alignés uniformément sur la plaque à 96 puits sans aucune inclinaison. Le câble USB reliant l’appareil au concentrateur ne doit pas être sous tension, ce qui peut entraîner un couple du « sandwich » et une restriction du mouvement correct de l’appareil.
Troisièmement, assurez-vous que l’appareil et les capteurs du couvercle de détection sont calibrés. La figure 2B explique comment vérifier rapidement le bon fonctionnement du capteur au début de chaque expérience. Quatrièmement, assurez-vous qu’un puits particulier ne présente pas une OCR aberrante avant la manipulation expérimentale. Après s’être assuré que les capteurs fonctionnent, la détermination des valeurs OCR de référence pour tous les puits contenant des cellules peut aider à déterminer si certains puits contiennent des cellules malsaines. La figure 8B montre plusieurs puits faisant l’objet d’une évaluation OCR de base, dans laquelle le puits de couleur bleue démontre clairement un OCR plus faible (et donc une concentration élevée d’O2 ). Ce puits est une valeur aberrante et devrait être exclu de la manipulation expérimentale.
Cinquièmement, minimisez les manipulations environnementales lors des lectures OCR actives. Le gradient O2 est très sensible aux changements de température, d’humidité, de niveaux de CO2 dans l’incubateur et de pression atmosphérique. L’ouverture de la porte de l’incubateur affecte considérablement ces facteurs, provoquant des interruptions temporaires des lectures OCR jusqu’à ce que le gradient O2 de la colonne de média revienne à l’équilibre (Figure 8C). Les paramètres affectant l’OCR peuvent être surveillés dans l’interface en ligne du système sous l’onglet « Environnement » (Figure 8A).
Sixièmement, déterminez le volume de support optimal pour l’expérience. Alors qu’un volume de média insuffisant peut créer des artefacts dus à une évaporation excessive (voir la première mesure de dépannage ci-dessus) ou à un épuisement prématuré des nutriments dans le milieu, un volume de média excessif limite la disponibilité de l’O2 à la monocouche EPR, déclenchant l’hypoxie9. La figure 7 est une capture d’écran du calculateur en ligne permettant de déterminer l’effet du volume du support sur la concentration en O2 au niveau de la monocouche EPR. L’OCR maximal possible pour un volume de support donné est illustré à la Figure 9A. Si le taux d’OCR pour une expérience donnée utilisant un volume donné se rapproche du taux affiché à la figure 9A, alors la concentration d’O2 au niveau de la monocouche EPR approche l’anoxie (0 %) et un volume de support plus faible est nécessaire. En général, les expériences impliquant des processus intensifs mitochondriaux dans la culture d’EPR ne doivent pas avoir des volumes de milieu supérieurs à 75 μL par 96 puits.
Septièmement, effectuer une normalisation appropriée pour isoler les effets d’une intervention expérimentale sur l’OCR. Étant donné que différents puits peuvent avoir un OCR de base différent, il est essentiel d’obtenir des lectures OCR de base pour chaque puits avant une intervention expérimentale. Soustrayez l’OCR de base de l’OCR après l’intervention expérimentale pour obtenir l'« OCR Delta », qui démontre l’effet de l’intervention expérimentale sur l’OCR. La méthode d’obtention de l’OCR Delta est illustrée à la figure 3 et à la figure 9B. Si l’on s’intéresse aux effets d’une intervention expérimentale sur la capacité d’une autre intervention expérimentale à induire l’OCR, une étape de normalisation supplémentaire, l'« OCR Delta-Delta », est nécessaire. Un tel exemple est illustré à la figure 9B, où la capacité du palmitate (une intervention expérimentale) à induire une augmentation de l’OCR (via l’induction de la β-oxydation) dépend du type de milieu pour la culture de l’EPR (une deuxième intervention expérimentale).
Enfin, minimisez le risque de contamination. Le système surveille l’OCR en introduisant un capteur d’oxygène dans le milieu au-dessus des cellules. Cela augmente le risque d’infection. Des baisses inattendues de l’OCR pendant l’expérience ou un faible OCR inattendu au début de l’expérience pourraient être des signes d’infection. Les plaques doivent être surveillées activement pour détecter toute infection à l’aide d’un microscope de culture cellulaire. Une contamination croisée entre des plaques à 96 puits peut se produire si un couvercle de détection est réutilisé. Pour minimiser une telle contamination croisée, il faut immerger tout le couvercle dans de l’éthanol à 70 % pendant 20 minutes pour le décontaminer après la réalisation d’une expérience, avant de réutiliser le couvercle de détection dans une nouvelle expérience (voir la section 1.2.15 du protocole).
En conclusion, un protocole est présenté pour la surveillance en temps réel et à long terme du métabolisme mitochondrial dans des cultures d’EPR haute fidélité via l’évaluation de l’OCR à l’aide d’un nouveau système appelé Resipher. Il peut être utilisé pour de nombreuses applications sur des cultures d’EPR hautement polarisées et différenciées, en particulier, pour sonder les propriétés métaboliques physiologiques des EPR isolées ainsi que le métabolisme altéré de l’EPR dans des modèles in vitro de maladies rétiniennes, y compris la DMLA et la PVR.
Déclarations de divulgation
Richard A. Bryan et Kin Lo sont des employés de Lucid Scientific, qui fabrique le système Resiper.
Remerciements
Nous remercions les Drs Daniel Hass et Jim Hurley d’avoir eu l’idée de tester la solubilité de l’O2 dans des milieux nouveaux par rapport à des milieux conditionnés à titre de contrôle. Nous remercions la Dre Magali Saint-Geniez pour sa contribution rédactionnelle au manuscrit. Nous remercions Scott Szalay de l’Instrument and Electronic Services Core, Kellogg Eye Center, pour avoir équipé la chambre d’hypoxie du câble USB Resiopher. Aucun financement fédéral n’a été utilisé pour la recherche sur le HFT. Le noyau de services électroniques est soutenu par le P30 EY007003 du National Eye Institute. Ce travail est soutenu par une subvention départementale sans restriction de la Recherche pour prévenir la cécité (RPB). J.M.L.M. est soutenu par l’Initiative James Grosfeld pour la dégénérescence maculaire liée à l’âge sec, la Fondation E. Matilda Ziegler pour les aveugles, une subvention de la banque d’yeux Eversight, une subvention K08EY033420 du National Eye Institute et le soutien de Dee et Dickson Brown ainsi que de la Fondation David et Lisa Drews Discovering Hope. D.Y.S. est soutenu par le programme Scientia de l’UNSW. L.A.K. est soutenu par le prix Iraty, Monte J. Wallace, Michel Plantevin, une subvention R01EY027739 du National Eye Institute et l’activité d’acquisition de la recherche médicale de l’armée du ministère de la Défense VR220059.
matériels
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco | #25-200-056 | |
| 3,3',5-triiodo-L-thyronine sodium salt | Sigma | T5516 | |
| 32-channel Resipher lid | Lucid Scientific | NS32-101A for Falcon | |
| Antimycin A from streptomyces sp. | Sigma | A8674-25MG | Inhibitor of Complex III of the electron transport chain |
| BAM15 | Sigma | SML1760-5MG | Uncoupling agent to increase mitochondrial respiration |
| DMSO, cell culture grade | Sigma-aldrich | D4540-100ML | Vehicle for reconstituting mitochondrial drugs |
| Extracellular matrix coating substrates: Synthemax II-SC | Corning | #3535 | Extracellular matrix for hfRPE |
| Extracellular matrix coating substrates: Vitronectin | Gibco | A14700 | Extracellular matrix for iPSC-RPE |
| FCCP | Sigma | C2920-10MG | Uncoupling agent to increase mitochondrial respiration |
| Fetal Bovine Serum (Bio-Techne S11550H) | Bio-Techne | S11550H | |
| Hydrocortisone-Cyclodextrin | Sigma | H0396 | |
| Hypoxia chamber | Embrient Inc. | MIC-101 | |
| N1 Media Supplement | Sigma | N6530 | |
| Non-Essential Amino Acids Solution | Gibco | 11140050 | |
| O2 sensor | Sensit technology or Forensics Detectors | P100 or FD-90A-O2 | |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| Recombinant human TGFβ2 | Peprotech | 100-35B | Transforming growth factor beta-2 to induce epithelial-mesenchymal transition |
| Rotenone | Sigma | R8875-1G | Inhibitor of Complex I of the electron transport chain |
| System-compatible plate | Corning | #353072 | |
| Taurine | Sigma | T8691 | |
| αMEM (Alpha Modification of Eagle's Media) | Corning | 15-012-CV |
Références
- Shu, D. Y., Butcher, E., Saint-Geniez, M. EMT and EndMT: emerging roles in age-related macular degeneration. Int J Mol Sci. 21 (12), 4271(2020).
- Shu, D. Y., Lovicu, F. J. Myofibroblast transdifferentiation: the dark force in ocular wound healing and fibrosis. Prog Retin Eye Res. 60, 44-65 (2017).
- Hurley, J. B. Retina metabolism and metabolism in the pigmented epithelium: a busy intersection. Annu Rev Vis Sci. 7, 665-692 (2021).
- Wu, W., et al. Deficient RPE mitochondria energetics leads to subretinal fibrosis in age-related macular degeneration. Invest Ophthalmol Vis Sci. 64 (8), 2420(2023).
- Hansman, D. S., et al. Metabolic reprogramming of the retinal pigment epithelium by cytokines associated with age-related macular degeneration. Biosci Rep. 44 (4), BSR20231904BSR20231904(2024).
- Li, K. X., et al. Modulation of pyruvate kinase M2 activity as a therapy in a primary RPE cell culture model of proliferative vitreoretinopathy. Invest Ophthalmol Vis Sci. 62 (8), 2213(2021).
- Gupta, S., et al. Progress in stem cells-based replacement therapy for retinal pigment epithelium: in vitro differentiation to in vivo delivery. Stem Cells Transl Med. 12 (8), 536-552 (2023).
- Wilson, D. F., et al. Oxygen distribution and vascular injury in the mouse eye measured by phosphorescence-lifetime imaging. Appl Opt. 44 (25), 5239-5248 (2005).
- Hass, D. T., et al. Medium depth influences O2 availability and metabolism in human RPE cultures. Invest Ophthalmol Vis Sci. 64 (14), 4(2023).
- Shu, D. Y., Butcher, E. R., Saint-Geniez, M. Suppression of PGC-1α drives metabolic dysfunction in TGFβ2-Induced EMT of retinal pigment epithelial cells. Int J Mol Sci. 22 (9), 4701(2021).
- Shu, D. Y., et al. Dimethyl fumarate blocks tumor necrosis factor-alpha-driven inflammation and metabolic rewiring in the retinal pigment epithelium. Front Mol Neurosci. 15, 896786(2022).
- Ng, P. Q., Saint-Geniez, M., Kim, L. A., Shu, D. Y. Divergent metabolomic signatures of TGFβ2 and TNFα in the induction of retinal epithelial-mesenchymal transition. Metabolites. 13 (2), 213(2023).
- Kar, D., et al. Choriocapillaris impairment is associated with delayed rod-mediated dark adaptation in age-related macular degeneration. Invest Ophthalmol Vis Sci. 64 (12), 41(2023).
- Curcio, C. A., Kar, D., Owsley, C., Sloan, K. R., Ach, T. Age-related macular degeneration, a mathematically tractable disease. Invest Ophthalmol Vis Sci. 65 (3), 4(2024).
- Maminishkis, A., et al. Confluent monolayers of cultured human fetal retinal pigment epithelium exhibit morphology and physiology of native tissue. Invest Ophthalmol Vis Sci. 47 (8), 3612-3624 (2006).
- Maminishkis, A., Miller, S. S. Experimental models for study of retinal pigment epithelial physiology and pathophysiology. J Vis Exp: JoVE. (45), e2032(2010).
- Zhang, Q., et al. A platform for assessing outer segment fate in primary human fetal RPE cultures. Exp Eye Res. 178, 212-222 (2019).
- Sharma, R., Bose, D., Montford, J., Ortolan, D., Bharti, K. Triphasic developmentally guided protocol to generate retinal pigment epithelium from induced pluripotent stem cells. STAR Protoc. 3 (3), 101582(2022).
- Marchetti, P., Fovez, Q., Germain, N., Khamari, R., Kluza, J. Mitochondrial spare respiratory capacity: Mechanisms, regulation, and significance in non-transformed and cancer cells. FASEB J. 34 (10), 13106-13124 (2020).
- Rosenfeld, P. J., Trivizki, O., Gregori, G., Wang, R. K. An update on the hemodynamic model of age-related macular degeneration. Am J Ophthalmol. 235, 291-299 (2022).
- Fitch, T. C., et al. Real-time analysis of bioenergetics in primary human retinal pigment epithelial cells using high-resolution respirometry. J Vis Exp: JoVE. (192), (2023).
- Calton, M. A., Beaulieu, M. O., Benchorin, G., Vollrath, D. Method for measuring extracellular flux from intact polarized epithelial monolayers. Mol Vis. 24, 425-433 (2018).
- Grumbine, M. K., et al. Maintaining and assessing various tissue and cell types of the eye using a novel pumpless fluidics system. J Vis Exp: JoVE. (197), (2023).
- Mu, C., Shearer, J. Protocol for measuring respiratory function of mitochondria in frozen colon tissue from rats. STAR Protoc. 4 (4), 102560(2023).
- Rocha, D. S., Manucci, A. C., Bruni-Cardoso, A., Kowaltowski, A. J., Vilas-Boas, E. A. A practical and robust method to evaluate metabolic fluxes in primary pancreatic islets. Mol Metab. 83, 101922(2024).
- Kanaan, M. N., et al. Cystine/glutamate antiporter xCT controls skeletal muscle glutathione redox, bioenergetics and differentiation. Redox Biol. 73, 103213(2024).
- da Veiga Moreira, J., et al. Methylene blue metabolic therapy restrains in vivo ovarian tumor growth. Cancers. 16 (2), 355(2024).
- McNally, L. A., Altamimi, T. R., Fulghum, K., Hill, B. G. Considerations for using isolated cell systems to understand cardiac metabolism and biology. J Mol Cell Cardiol. 153, 26-41 (2021).
- Misare, K. R., et al. The consequences of tetraploidy on Caenorhabditis elegans physiology and sensitivity to chemotherapeutics. Sci Rep. 13 (1), 18125(2023).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon