Method Article
Langzeitüberwachung des Sauerstoffverbrauchs in hochdifferenzierten und polarisierten retinalen Pigmentepithelkulturen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Wir stellen ein neuartiges Gerät zur Messung des Sauerstoffverbrauchs (OCR) in retinalen Pigmentepithelkulturen (RPE) vor. Das Gerät kann die OCR wochenlang an RPE messen, das auf Standard-Zellkulturplatten mit Standardmedien gezüchtet wurde, während sich die Platten in einem Standard-Zellkulturinkubator befinden.
Zusammenfassung
Der mitochondriale Stoffwechsel ist entscheidend für die normale Funktion des retinalen Pigmentepithels (RPE), einer Monoschicht von Zellen in der Netzhaut, die für das Überleben der Photorezeptoren wichtig ist. Die mitochondriale Dysfunktion von RPE ist ein Kennzeichen der altersbedingten Makuladegeneration (AMD), der Hauptursache für irreversible Erblindung in den Industrieländern, und der proliferativen Vitreoretinopathie (PVR), einer erblindenden Komplikation von Netzhautablösungen. RPE-degenerative Erkrankungen wurden durch RPE-Kultursysteme, die hochdifferenziert und polarisiert sind, um in vivo RPE nachzuahmen, gut modelliert. Die Überwachung der Sauerstoffverbrauchsraten (OCR), eines Indikators für die mitochondriale Funktion, erwies sich in solchen Kultursystemen jedoch als schwierig, da die Bedingungen, die eine ideale RPE-Polarisation und -Differenzierung begünstigen, keine einfachen OCR-Messungen ermöglichen.
Hier stellen wir ein neuartiges System, Resipher, vor, um OCR wochenlang in gut differenzierten RPE-Kulturen zu überwachen, während das RPE auf optimalen Wachstumssubstraten und physiologischen Kulturmedien in einem Standard-Zellkultur-Inkubator gehalten wird. Dieses System berechnet die OCR, indem es den Sauerstoffkonzentrationsgradienten misst, der in den Medien über den Zellen vorhanden ist. Wir diskutieren die Vorteile dieses Systems gegenüber anderen Methoden zur Erkennung von OCR und wie man das System zur Messung von OCR in RPE-Kulturen einrichtet. Wir behandeln wichtige Tipps und Tricks für die Verwendung des Systems, Vorsicht bei der Interpretation der Daten und Richtlinien für die Fehlerbehebung bei unerwarteten Ergebnissen.
Wir bieten auch einen Online-Rechner zur Extrapolation des Hypoxie-, Normoxie- oder Hyperoxie-RPE-Niveaus an, die auf dem Sauerstoffgradienten in den Medien über den vom System erkannten Zellen basieren. Schließlich untersuchen wir zwei Anwendungen des Systems, die Messung des Stoffwechselzustands von RPE-Zellen in einem PVR-Modell und das Verständnis, wie sich das RPE metabolisch an Hypoxie anpasst. Wir gehen davon aus, dass die Verwendung dieses Systems an hochpolarisierten und differenzierten RPE-Kulturen unser Verständnis des mitochondrialen RPE-Stoffwechsels sowohl unter physiologischen als auch unter Krankheitszuständen verbessern wird.
Einleitung
Das retinale Pigmentepithel (RPE) ist eine Monoschicht aus funktionell postmitotischen, hochpolarisierten Epithelzellen, die eine Barriere zwischen lichtempfindlichen Photorezeptoren in der Netzhaut und deren Durchblutung bilden, ein Kapillarbett, das als Choriokapillaris bezeichnet wird. Wie die Rolle der Glia-unterstützenden Neuronen erfüllt das RPE unzählige Funktionen zur Unterstützung von Photorezeptoren, einschließlich der Phagozytose der äußeren Segmente der Photorezeptoren, des Transports von Nährstoffen und der metabolischen Unterstützung der Photorezeptoren sowie der Sekretion essentieller Wachstumsfaktoren, die alle für die Aufrechterhaltung der Sehfunktion entscheidend sind.
Die Degeneration des RPE liegt mehreren häufigen degenerativen Erkrankungen des Sehvermögens zugrunde. Bei der altersbedingten Makuladegeneration (AMD), einer der häufigsten Ursachen für unheilbaren Sehverlust weltweit, stirbt das RPE ab, und die darüber liegenden Photorezeptoren erleiden daher eine sekundäre Degeneration. Bei der proliferativen Vitreoretinopathie (PVR) verlässt das RPE stattdessen seinen normalerweise ruhenden postmitotischen Zustand, proliferiert sich und dedifferenziert sich in einen mesenchymalen Zustand (einen sogenannten epithelial-mesenchymalen Übergang [EMT]) mit Veränderungen in seinem Stoffwechsel. Die RPE-Dedifferenzierung führt zu einem Verlust der RPE-Unterstützung für Photorezeptoren und löst gleichzeitig einen fibrotischeren Zustand aus. Dies führt sowohl zu einer Degeneration der Photorezeptoren als auch zu einer RPE-induzierten Narbenbildung, die beide einen Sehverlust auslösen 1,2.
Ein großer Teil der Unterstützung der Photorezeptoren durch RPE ist metabolisch, und metabolische Dysregulation ist ein kritischer Faktor bei zahlreichen Netzhauterkrankungen, einschließlich AMD und PVR. Das RPE dient als regulatorische Barriere zwischen den Photorezeptoren und ihrer Sauerstoff- und Nährstoffquelle, der Choriokapillaris. Daher bestimmt das, was das RPE metabolisiert, im Vergleich zu dem, was das RPE von der Choriokapillaris zu den Photorezeptoren durchdringt, stark den Photorezeptorstoffwechsel und das Überleben. Zahlreiche Studien haben gezeigt, dass das RPE für seine normale Gesundheit stark vom mitochondrialen Stoffwechsel abhängig ist und dass die Photorezeptoren stattdessen stark auf die Glykolyse angewiesensind 3. Dadurch wurde das Konzept komplementärer, miteinander verflochtener Stoffwechselzustände zwischen Photorezeptoren und dem RPE eingeführt. Insbesondere reduziert das RPE seinen Metabolismus von bevorzugten Photorezeptor-Stoffwechselsubstraten und nutzt stattdessen die Nebenprodukte des Photorezeptorstoffwechsels in Kombination mit den Metaboliten, die Photorezeptoren nicht verbrauchen. Bei Krankheiten wie PVR und AMD deuten Hinweise stark darauf hin, dass das RPE glykolytischer und weniger abhängig vom mitochondrialen Stoffwechsel wird; Diese Verschiebung hin zur RPE-Glykolyse kann dazu führen, dass die Photorezeptoren die benötigten Metaboliten verlieren, was zu einer Degeneration führt 4,5,6. Angesichts der gegenseitigen Abhängigkeit von RPE und Photorezeptorstoffwechsel und der Tatsache, wie stark ein veränderter Stoffwechsel der Netzhauterkrankung zugrunde liegt, besteht ein starkes Interesse an der Modellierung und Manipulation des RPE-Stoffwechsels für therapeutische Zwecke.
Während die Untersuchung des mitochondrialen RPE-Stoffwechsels in vivo ideal ist, können viele Aspekte des mitochondrialen RPE-Stoffwechsels nur in einem in vitro-Kultursystem praktisch untersucht werden. In den letzten Jahrzehnten wurden erhebliche Fortschritte auf dem Weg zu High-Fidelity-RPE-Kulturen erzielt, so dass die am sorgfältigsten gepflegten RPE-Kulturen heute in klinischen Studien am Menschen für die Zellersatztherapie verwendet werden7. Um solche High-Fidelity-Kulturen zu erhalten, muss das RPE vor dem Experimentieren monatelang auf bestimmten Substraten in bestimmten Medien gezüchtet werden. Unter diesen Bedingungen sind RPE-Kulturen maximal differenziert und polarisiert, was sich dem in vivo RPE annähert. Leider gibt es derzeit keine Geräte, die den mitochondrialen Stoffwechsel spezifisch aus dem RPE in vivo messen können. Während die Sauerstoffüberwachung des retinalen Kapillarnetzwerks in vivo mit Hilfe der Elektronen-Paramagnetischen Resonanz (EPR)-Oximetrie8 durchgeführt wurde, ist dies bei der RPE-Analyse nicht möglich. Unterschiede zwischen dem RPE-Metabolismus in vivo und in vitro sind nicht gut beschrieben, aber es wurde gezeigt, dass RPE-Kulturen eine hohe mitochondriale Aktivität aufweisen, ähnlich wie RPE in vivo 3,9, was darauf hindeutet, dass mit Hilfe von High-Fidelity-RPE-Kulturen signifikante Einblicke in den mitochondrialen RPE-Stoffwechsel gewonnen werden können.
Da jeder mitochondriale Stoffwechsel zu einem Sauerstoffverbrauch führt, ist die Messung der RPE-Sauerstoffverbrauchsraten (OCR) ein zuverlässiger Indikator für den mitochondrialen Stoffwechsel. Die Messung von OCR in RPE-Kulturen erwies sich als schwierig, da die Bedingungen, die eine maximale RPE-Polarisation und -Differenzierung begünstigen, mit derzeit verfügbaren Techniken wie dem Seahorse Analyzer oft langfristige genaue OCR-Messungen ausschließen. In diesem Methodenpapier wird ein neuartiges Gerät, der Resipher (im Folgenden als "das System" bezeichnet), vorgestellt, das eine kontinuierliche Messung der OCR über Wochen in RPE ermöglicht, die unter Bedingungen angebaut werden, die die Polarisation und Differenzierung maximal fördern. Die Leichtigkeit, mit der OCR von diesem System unter RPE-Kulturbedingungen gemessen werden kann, die die RPE-Differenzierung und Polarisation maximal fördern, ist einzigartig unter den bestehenden OCR-Messgeräten.
Dieses Dokument enthält Tipps und Tricks für die Verwendung des Systems mit RPE-Kulturen, gefolgt von einer Demonstration von zwei speziellen Anwendungen. Erstens wird die RPE-EMT, die den PVR nachahmt, durch das Engagement gegenüber dem transformierenden Wachstumsfaktor-beta (TGFβ)1,10,11,12 ausgelöst. Das System wird verwendet, um zu überwachen, wie sich der RPE-Stoffwechsel während des EMT-Prozesses entwickelt. Zweitens wird die Rolle von Hypoxie im RPE-Stoffwechsel anhand dieses Systems untersucht. Hypoxie ist ein wichtiger Faktor für die Pathogenese der AMD, da die Choriokapillaris mit dem Altervon 13,14 Jahren dünner wird. Die Kombination dieses Systems mit Hypoxiekammern ermöglicht es, den veränderten mitochondrialen RPE-Stoffwechsel mit der subtilen Hypoxie, die mit dem Altern einhergeht, zu modellieren. Schließlich wird ein Online-Rechner mit Resipher-Daten eingeführt, mit dem man bestimmen kann, ob sich RPE-Kulturen in hypoxischen, normoxischen oder hyperoxischen Bedingungen befinden. Solche Informationen sind entscheidend, um Rückschlüsse auf den RPE-Metabolismus aus In-vitro-RPE-Kulturstudien zu ziehen.
Protokoll
Für Protokolle zur Etablierung von humanen Primär- oder iPSC-RPE-Kulturen siehe die folgenden Referenzen 15,16,17,18. Die Gewinnung und Verwendung von menschlichem Gewebe für diese Protokolle wurde vom Institutional Review Board (HUM00105486 der University of Michigan geprüft und genehmigt.
1. Allgemeine Anwendung des Systems auf die RPE-Kultur
- Platte humane induzierte pluripotente Stammzellen (iPSC), abgeleitete RPE-Zellen oder primäre humane RPE-Zellen in systemkompatiblen 96-Well-Platten.
- Unter der Annahme bereits vorhandener reifer Kulturen, die auf 24-Well-Zellkulturplatten gezüchtet wurden, waschen Sie die Zellen einmal mit phosphatgepufferter Kochsalzlösung (PBS) und fügen Sie dann 500 μl 0,25 % Trypsin-EDTA hinzu (Table of Materials). Inkubieren Sie 10-40 Minuten in einem Zellkultur-Inkubator und kontrollieren Sie alle 5-10 Minuten, bis die Zellen rund und fast bereit zum Ablösen sind.
- Pipettieren Sie das Medium vorsichtig über die Zellen, um sie vom Zellkulturkunststoff zu lösen, und übertragen Sie es anschließend auf das 3-fache Volumen des Zellkulturmediums (1.500 μl pro 24-Well) und schleudern Sie es in einer Zentrifuge bei 250 × g für 5 Minuten bei Raumtemperatur.
HINWEIS: Das Rezept für RPE-Zellkulturmedien ist in Tabelle 1 aufgeführt und in den zuvor veröffentlichten Referenzen 9,15,16,17,18 verfügbar. - Resuspendieren Sie die Zellen in RPE-Zellkulturmedien mit 15 % fötalem Rinderserum (FBS) und zählen Sie die Zellen mit einem Hämozytometer.
- 74.000 Zellen für jede Vertiefung einer 96-Well-Platte (in der Regel 225-300 × 105 Zellen/cm2) aussäen, nachdem sie gemäß den Anweisungen des Herstellers mit hochspezialisierten extrazellulären Matrix-Beschichtungssubstraten (Materialtabelle) beschichtet wurden.
HINWEIS: Keimzellen nur auf einer Resipher-kompatiblen Platte (Materialtabelle) und nur in Vertiefungen, die dem Sondenarray auf dem Messdeckel entsprechen (Spalten 3, 4, 9 und 10 für den 32-Kanal-Deckel; siehe Materialtabelle). Der Sensordeckel des Systems ist mit 4-, 32- oder 96-Kanal-Sensoren erhältlich (siehe Sensorpositionen in Abbildung 1A-D), und eine Reihe von Standard-Zellkulturplatten sind kompatibel. - Da Randvertiefungen anfällig für Verdunstung sind, was sich dramatisch auf die Sauerstoffverfügbarkeit und damit auf die OCR-Messwerte auswirkt, ist das Aussäen von Zellen in allen Randvertiefungen (Reihen A und H einer 96-Well-Platte) zu vermeiden. Geben Sie außerdem 200 μl steriles Wasser in jede der leeren Vertiefungen der 96-Well-Platte, um Verdunstungseffekte zu reduzieren.
- Halten Sie die Platte 10 Minuten lang auf einer stabilen Tischfläche, damit sich die Zellen absetzen können. Bringen Sie es dann wieder in den Inkubator zurück. Wechseln Sie das Medium nach 24-72 h und ersetzen Sie es durch Standard-RPE-Nährmedien mit 5 % FBS. Kultur für mindestens 4 Wochen, 2x pro Woche Medienwechsel.
HINWEIS: Zellen sind bereit für Experimente, wenn sie pigmentiert, kopfsteingepflastert und sehr kompakt sind (Abbildung 1E).
- Einrichten und Erfassen von Daten mit dem System
- Setzen Sie den Sensordeckel auf eine leere 96-Well-Empfängerplatte und setzen Sie diese dann wieder in den Zellkultur-Inkubator ein. Richten Sie das Gerät aus, montieren Sie es auf dem Sensordeckel und verbinden Sie es über das mitgelieferte USB-Kabel mit dem Hub. Dadurch entsteht ein Resipher-"Sandwich" (Abbildung 2A).
- Gehen Sie zur Lucid-Lab-Website-Anwendung (https://lab.lucidsci.com/) und klicken Sie auf die Schaltfläche Neues Experiment in der rechten oberen Ecke, um ein neues Experiment zu erstellen.
- Nennen Sie den Titel des Experiments und geben Sie alle relevanten experimentellen Notizen für die jeweilige Studie ein (z. B. die Durchgangsnummer des iPSC-RPE, das verwendet wird).
- Erstellen Sie Brunnenbedingungen und Behandlungsgruppen. Wenn Sie beispielsweise die Auswirkungen verschiedener Serumkonzentrationen in den Medien mit OCR testen, wählen Sie Serum im Gruppennamen aus, geben Sie dann die zu verwendenden Serumkonzentrationen des Mediums ein und fügen Sie weitere Serumprozentwerte hinzu, indem Sie auf die Schaltfläche + klicken. Weisen Sie zu, welche Vertiefungen einen bestimmten Serumprozentsatz erhalten, und wählen Sie ein Farbmuster für diese Vertiefungen aus. Fügen Sie bei Bedarf weitere Gruppen hinzu (z. B. eine andere experimentelle Variable zum Testen), indem Sie auf die Schaltfläche Gruppe hinzufügen klicken.
- Definieren Sie die Platteneinrichtung und wählen Sie das entsprechende Gerät und den Plattenstil aus. Klicken Sie auf die Schaltfläche Platte hinzufügen und wählen Sie Gerät; Wählen Sie den Plattentyp aus. Wählen Sie die Behandlung aus und klicken Sie auf die entsprechende Vertiefung.
- Klicken Sie auf die Schaltfläche Jetzt starten , um das Experiment zu starten. Vergewissern Sie sich, dass die Anzeige auf der Website und die LED am Hub durchgehend grün leuchten.
- Lassen Sie das System 15 bis 60 Minuten laufen, um sicherzustellen, dass jeder Sensor ordnungsgemäß kalibriert ist und den Luftsauerstoff genau erkennt, der in einem vollständig befeuchteten Zellkultur-Inkubator mit 5 % CO2 in einer Konzentration von ~200 μM liegen sollte (Abbildung 2Bi).
HINWEIS: Wenn einer der Sensoren mehr als 20 % vom Durchschnitt der anderen Sensoren in einem Standard-Zellkultur-Inkubator abweicht, sollten Sie dies bei der Datenanalyse ausschließen, da der Sensor fehlerhaft ist (Abbildung 2Bii).
HINWEIS: Wenn alle Sensoren mehr als 20 % vom Durchschnitt der anderen Sensoren und von dem erwarteten atmosphärischen O2 abweichen, der in einem Standard-Zellkultur-Inkubator als 200 μM angegeben wird, wurde der Sensordeckel zu oft verwendet. Ersetzen Sie ihn durch einen neuen Sensordeckel (Abbildung 2Biii).
HINWEIS: Das Abrufen der O2-Messwerte in der Luft vor Beginn eines Experiments verbessert die Fehlerbehebung danach erheblich. Wenn ein bestimmtes Bohrloch nach Abschluss eines Experiments als Ausreißer auftritt und dieses Bohrloch einen Sensor hatte, der nicht kalibriert war, liegt das Problem möglicherweise am Sensor und nicht am biologischen Replikat. - Nehmen Sie das Gerät vom Sensordeckel ab (lösen Sie jedoch nicht das USB-Kabel vom Gerät). Legen Sie das Gerät kopfüber in den Inkubator, damit der Motor des Geräts zurückgesetzt werden kann. Verwenden Sie das Gerät erst wieder, wenn alle Motorgeräusche verstummt sind (ca. 20-30 s).
HINWEIS: Es ist wichtig, dass das Gerät während eines Experiments immer über das USB-Kabel mit dem Hub verbunden ist, auch wenn das Gerät nicht am Sensordeckel befestigt ist. Auf diese Weise kann das Gerät die Umgebung des Zellkultur-Inkubators kontinuierlich überwachen und Berichte darüber erstellen. - Setzen Sie die Platte mit dem Sensordeckel wieder in die Zellkulturhaube ein, zusammen mit einer 96-Well-Platte, die die RPE-Kulturen enthält. Ändern Sie das Medium in der Platte mit RPE-Kulturen mit demselben Medium und demselben Volumen für alle Wells, um OCR-Basiswerte für jedes Well zu erhalten. Achten Sie darauf, alle Vertiefungen ohne RPE mit sterilem Wasser zu füllen, um eine Verdunstung zu verhindern.
HINWEIS: Im Allgemeinen empfehlen wir 60-100 μl RPE-Nährmedien pro 96-Well, basierend auf Daten, die zeigen, dass ein geringeres Medienvolumen zu einem schnellen Nährstoffmangel führt, ein höheres Medienvolumen jedoch zu unbeabsichtigter Hypoxie führt9. - Übertragen Sie den Sensordeckel auf die Platte mit den Zellkulturen und den Standarddeckel (ohne Resipher) auf der Platte mit den Zellkulturen auf die leere 96-Well-Empfängerplatte, um die Sterilität des Standarddeckels zu erhalten, die an verschiedenen Stellen während des Experiments benötigt wird.
- Setzen Sie die Platte mit den Zellkulturen und den Sensordeckel wieder in den Zellkultur-Inkubator ein. Richten Sie das Gerät erneut aus, montieren Sie es auf dem Sensordeckel und verbinden Sie es über das mitgelieferte USB-Kabel mit dem Hub (Zusammenbau des "Sandwiches"). Vergewissern Sie sich, dass die Anzeigen auf der Website-Oberfläche und im Hub grün leuchten.
HINWEIS: Die O 2-Konzentrationsdaten werden sofort angezeigt, während die OCR-Daten erst angezeigt werden, nachdem genügend O2 -Daten gesammelt wurden, etwa 1 Stunde. - Lassen Sie das Gerät die OCR an jedem Well mindestens 12 bis 24 Stunden lang messen, um eine OCR zu erfassen.
HINWEIS: Die Basis-OCR hängt von vielen Faktoren ab, einschließlich der Art des Mediums, das auf Zellen aufgebracht wird. In Abbildung 2C weisen Standard-RPE-Nährmedien mit unterschiedlichen FBS-Mengen (0 %, 5 %, 15 %) leicht unterschiedliche OCR-Werte auf. Darüber hinaus können Kulturen mit mehr FBS die mitochondriale Aktivität länger aufrechterhalten, nachdem das Medium auf die Zellen aufgetragen wurde, bevor die OCR aufgrund einer Depletion der mitochondrialen Metaboliten abfällt (rechte Seite der Kurve). - Sobald eine OCR zu Studienbeginn erhalten wurde, wiederholen Sie die Schritte 1.2.8 bis 1.2.12, aber ändern Sie das Medium auf der Platte mit RPE-Kulturen an die experimentellen Bedingungen (in der Regel ± einem Medikament oder einem Vergleich verschiedener Medienbedingungen).
HINWEIS: Routinemäßige Medienwechsel können ebenfalls auf die gleiche Weise gehandhabt werden. Jedes Mal, wenn der Zellkultur-Inkubator geöffnet wird, ist mit erheblichen Unterbrechungen der OCR-Messwerte zu rechnen, da diese von Temperatur, Luftfeuchtigkeit, CO2 - Konzentrationen und anderen Faktoren abhängen, die alle vorübergehend durch das Öffnen der Inkubatortür gestört werden. Während der Entnahme des Geräts aus dem Sensordeckel ist es nicht erforderlich, das Experiment online zu unterbrechen. Nachdem die Tür des Zellkultur-Inkubators geöffnet oder das Medium auf der zu sondierenden Platte gewechselt wurde, dauert es in der Regel 2-4 Stunden, bis der Sauerstoffgradient wieder hergestellt ist und mit der Messung einer genauen OCR begonnen wird. - Nachdem die Daten erhalten wurden, subtrahieren Sie die OCR zu Studienbeginn für jede Vertiefung von der OCR, nachdem die Versuchsbedingung angewendet wurde, um die durch die Behandlung ausgelöste Delta-OCR zu bestimmen.
- Nachdem das Experiment abgeschlossen ist, sterilisieren Sie den Sensordeckel und verwenden Sie ihn wieder (der 3-5x wiederverwendet werden kann, obwohl die Leistung bei wiederholtem Gebrauch nachlässt). Um den Messdeckel zu sterilisieren, tauchen Sie den gesamten Deckel 10 Minuten lang in 70 % Ethanol in eine Zellkulturhaube, nehmen Sie ihn dann aus dem Eintauchen und legen Sie ihn mit der Sondenseite nach oben in den Schrank (vermeiden Sie es, die empfindlichen Sondenspitzen zu berühren) und lassen Sie Ethanol und Wasser vollständig verdampfen, bevor Sie den Messdeckel auf eine neue 96-Well-Empfängerplatte setzen.
- Nehmen Sie Hellfeldbilder auf, um OCR-Werte auf die Zellennummer zu normalisieren.
HINWEIS: Die Melaninakkumulation in RPE erleichtert die Normalisierung der OCR auf der Grundlage einer einfachen Zählung der Zellzahlen in lebenden Kulturen mit einem Hellfeldmikroskop.- Entfernen Sie das Gerät und stellen Sie es auf den Kopf, wie in Schritt 1.2.8 beschrieben. Tauschen Sie in der Zellkulturhaube den Sensordeckel auf der RPE-Kulturplatte gegen einen Standarddeckel mit 96 Vertiefungen aus.
- Nehmen Sie mit einem inversen Standardmikroskop Hellfeldbilder von jeder Vertiefung auf.
HINWEIS: Halten Sie den Bereich auf dem Bild zwischen den Vertiefungen konsistent (relative Position in der Vertiefung und Objektivvergrößerung). - Ersetzen Sie in der Zellkulturhaube den standardmäßigen 96-Well-Deckel auf der RPE-Kulturplatte durch den Sensordeckel und setzen Sie ihn zur weiteren Überwachung wieder in den Inkubator ein.
- Zählen Sie die Zellzahl in jeder Vertiefung mit ImageJ oder einer anderen Software.
- Normalisieren Sie OCR auf die Zellzahl in den verschiedenen Versuchsgruppen. Das System meldet OCR in Einheiten von fmol∙(mm2)-1∙s-1-dem Verbrauch pro Querschnittseinheit. Um die OCR pro Zelle zu normalisieren, dividieren Sie die OCR daher durch die Zellzahl pro mm2 (und nicht durch die Zellzahl pro Well). Äquivalent multiplizieren Sie die vom System gemeldete OCR mit der Querschnittsfläche des Wells (ca. 31 mm2 für eine Standardplatte mit 96 Wells), um OCR in den Einheiten fmol∙s-1∙well-1 zu erhalten.
- Kontrollen zur Bestimmung bioenergetischer RPE-Parameter.
HINWEIS: Um sicherzustellen, dass das System und die RPE-Kulturen auf vorhersehbare Weise auf mitochondriale Manipulationen reagieren, können bestimmte kleine Moleküle getestet werden, die gut etabliert sind, um bestimmte Schritte der mitochondrialen oxidativen Phosphorylierung zu erreichen. Diese Tests sind analog zu den Schritten, die in einem mitochondrialen Stresstest mit dem Seahorse Analyzer10 durchgeführt werden, und liefern ein bioenergetisches Profil für RPE-Kulturen.- Kultivieren Sie RPE-Zellen in 65 μl Standard-RPE-Kulturmedien mit 5 % FBS und messen Sie OCR für 24 Stunden, um eine Ausgangsbasis zu schaffen.
- Aspirieren Sie kultivierte Medien und fügen Sie 65 μl Standard-RPE-Kulturmedien mit 5 % FBS und mitochondrialen Entkopplern (3 μM Carbonylcyanid-p-trifluormethoxyphenylhydrazon [FCCP] oder 500 nM Bam15, Materialtabelle) hinzu. Über Nacht inkubieren.
HINWEIS: Mitochondriale Entkoppler sollten die OCR signifikant erhöhen. Stellen Sie sicher, dass die erhöhte OCR nicht auf oder in der Nähe der theoretischen maximalen diffusionsbegrenzten OCR von ca. 275 fmol∙(mm2)-1∙s-1 für 65 μl Medien unter Standardkulturbedingungen liegt. Max OCR für verschiedene häufig verwendete Medienvolumes ist im Abschnitt Diskussion verfügbar. - Nach der Inkubation über Nacht wird das Medium mit mitochondrialen Entkopplern aspiriert und durch 65 μl Standard-RPE-Kulturmedien mit 5 % FBS ersetzt. Beobachten Sie die Platte für weitere 24 Stunden, um die Wiederfindung der RPE-OCR nach der Entfernung der Medikamente zu bestimmen.
- Berechnen Sie die Auswirkungen von Entkopplern auf die OCR, indem Sie die nach der Behandlung erhaltene OCR von der vor der Behandlung erhaltenen OCR subtrahieren (Abbildung 3).
HINWEIS: Die Erhöhung der OCR durch Zugabe von mitochondrialen Entkopplern bedeutet die mitochondriale Atmungskapazität der Zelle19. - Um andere mitochondriale Parameter zu messen, die typischerweise während des mitochondrialen Stresstests mit einem Seahorse Analyzer erfasst werden, führen Sie die gleichen Schritte wie oben mit den folgenden Verbindungen durch (Abbildung 4).
- Verwenden Sie Oligomycin bei 1 μM, um die ATP-gebundene Atmung zu beurteilen.
- Verwenden Sie Antimycin A und Rotenon bei 1 μM, um eine nicht-mitochondriale Atmung zu beurteilen.
HINWEIS: FCCP, Oligomycin, Antimycin A und Rotenon können für RPE toxisch sein. Wenn sie zu einem signifikanten Zelltod führen, sind OCR-Messungen nicht genau. Daher sollten OCR-Messungen nur zu einem Zeitpunkt durchgeführt werden, zu dem es keinen offensichtlichen Zelltod gibt, in der Regel innerhalb der ersten Stunden nach der Zugabe der Medikamente (Abbildung 4).
2. Messung von Veränderungen des mitochondrialen Stoffwechsels bei RPE unter EMT
- Befolgen Sie das gleiche Protokoll wie in Abschnitt 1.2 beschrieben.
- Um eine EMT nach OCR-Ausgangsmessungen zu induzieren, fügen Sie TGF-β2 (10 ng/ml) zu RPE-Kulturen hinzu und überwachen Sie die OCR über einen Zeitraum von 3 Wochen. Aktualisieren Sie das Medium alle 2 bis 3 Tage mit frischem TGFβ2 (Abbildung 5).
- Mindestens 2x pro Woche Hellfeldbilder wie in Abschnitt 1.3 aufnehmen, um sicherzustellen, dass sich die Zellzahl nicht verändert. Wenn sich die Anzahl ändert, normalisieren Sie die OCR-Werte auf die Zellennummer in jedem Well.
HINWEIS: Das gleiche Protokoll kann für andere EMT-Induktoren verwendet werden, um langfristige OCR-Veränderungen während des gesamten Kulturzeitraums zu überwachen, während sich die Zellen im Inkubator befinden.
3. Messung von Veränderungen im mitochondrialen Stoffwechsel bei hypoxischem RPE
HINWEIS: Die Anwendung des Systems unter hypoxischen, normoxischen oder hyperoxischen Bedingungen ist die gleiche wie in Abschnitt 1.2, mit der Ausnahme, dass das "Sandwich" in einer Hypoxiekammer (Materialtabelle) in einem Standard-Zellkultur-Inkubator aufbewahrt wird.
- Bohren Sie ein Loch in den Deckel der Hypoxiekammer, durch das das USB-Typ-C-Kabel geführt wird, und versiegeln Sie es mit Silikon oder Kitt (Abbildung 6A).
- Montieren Sie den Sensordeckel mit der Zellkulturplatte in der Haube und übertragen Sie sie in die Hypoxiekammer.
- Stellen Sie das "Sandwich" (Platte, Sensordeckel, Gerät) in der Hypoxiekammer auf. Stellen Sie auch eine Petrischale mit sterilem Wasser in die Hypoxiekammer, um die Luftfeuchtigkeit aufrechtzuerhalten. Platzieren Sie abschließend einen tragbaren O2 -Sensor in der Hypoxiekammer (Materialtabelle). Das Setup ist in Abbildung 6A dargestellt.
- Verschließen Sie die Kammer und schließen Sie sie an eine Gasflasche mit einer hypoxischen Konzentration von O2 und der Standard-Zellkulturkonzentration von CO2 an (z. B. 1 % O2 und 5 % CO2) an.
- Tauschen Sie die Luft in der Hypoxiekammer langsam gegen die 1 % O2/5 % CO2 -Luft in der Flasche aus, bis der Sauerstoffsensor die gewünschte O2 -Konzentration anzeigt, normalerweise irgendwo zwischen 1 % und 10 %.
HINWEIS: Der tragbare O2 -Sensor benötigt Zeit, um sich auszugleichen, daher sollte das Gas aus der Flasche langsam in die Hypoxiekammer gegeben werden. - Schließen Sie die Einlass- und Auslassventile der Hypoxiekammer und übertragen Sie die gesamte Kammer in den Inkubator.
- Richten Sie das Experiment wie in Abschnitt 1.2 ein und starten Sie es.
HINWEIS: Das Voräquilibrieren von Medien in der Hypoxiekammer vor der Zugabe zu Zellen kann die Zeit verkürzen, die die Zellmonoschicht benötigt, um eine echte Hypoxie zu erleben (Abbildung 6B).
4. Berechnung derO2-Konzentration an der RPE-Monoschicht, um zu bestimmen, ob sich Zellen in hypoxischen, normoxischen oder hyperoxischen Bedingungen befinden
HINWEIS: Das System misst die O2 -Konzentration standardmäßig zwischen ca. 1 und 1,5 mm über dem Boden der Vertiefung, vorausgesetzt, es wird eine Standardplatte mit dem entsprechenden empfohlenen Messdeckel für Monolayer-Kulturen verwendet (siehe https://lucidsci.com/docs/LucidScientific_Sensing_Lid_Selection_Guide.pdf). Während dieO2-Konzentration an der Zellmonoschicht nicht direkt gemessen wird, können die Daten des Systems verwendet werden, um dieO2-Konzentration auf der Ebene des RPE abzuschätzen. Insbesondere in dem Wissen, dass zwischen dem oberen Teil der Mediensäule, wo O2 verfügbar ist, und dem unteren Teil der Mediensäule, wo O2 verbraucht wird, ein Sauerstoffgradient besteht, können die Fickschen Diffusionsgesetze mit der gemessenen OCR-Rate kombiniert werden, um die O2 -Spiegel in der Zellmonoschicht zu extrapolieren. Ein Rechner für diese Schätzung wird online zur Verfügung gestellt: https://lucidsci.com/notes?entry=oxygen_diffusion (und in Form eines interaktiven Open-Source-Notizbuchs bei https://observablehq.com/@lucid/oxygen-diffusion-and-flux-in-cell-culture, der Quellcode dieses Rechners ist unter https://github.com/lucidsci/oxygen-diffusion-calculator zu finden).
- Sobald der Rechner aufgerufen ist, geben Sie das Medienvolumen in Mikrolitern ein (z. B. 100 μL).
- Geben Sie die Konzentration des gesättigten Sauerstoffs an der Grenzfläche zwischen Luft und Flüssigkeit (oben in der Mediensäule über den Zellen) ein. Dieser Wert beträgt ~185 μM bei normalem Atmosphärendruck mit 5 % O2 und 95 % Luftfeuchtigkeit.
HINWEIS: Dies kann auch anhand von O2-Messungen mit Systemsonden in reinen Medienvertiefungen (ohne Zellen am Boden der Vertiefung) bestimmt werden. - Geben Sie in der Eingabe Flux den vom System gemeldeten Flux/OCR ein.
- Basierend auf diesen Werten wird die stationäre Sauerstoffkonzentration in jeder Höhe in der Medienspalte berechnet und im Diagramm dargestellt (Abbildung 7). Die x-Achse des Diagramms ist die Höhe in mm über dem Boden der Vertiefung. Die y-Achse ist die berechnete O2 -Konzentration in dieser Höhe (in μM).
- Die O2 -Konzentration am Boden der Vertiefung (wo sich die Zellen befinden) wird in der Zeile unter dem Diagramm angegeben.
HINWEIS: Im Allgemeinen wird geschätzt, dass RPE in vivo eineO2-Konzentration von 4-9% aufweist. Jeweils 10 μM O2 unter normalen atmosphärischen Bedingungen entsprechen etwa 1 % O2. Somit beträgt die Normoxie auf der Ebene der Zellen etwa 40-90 μM O2. Je höher die Mediensäule ist, desto niedriger befindet sich O2 auf dem Niveau der RPE-Monoschicht9. Wenn sich die Zellen also in hypoxischen Bedingungen zu befinden scheinen, kann das Medienvolumen reduziert werden. Wenn sich Zellen in hyperoxischen Bedingungen zu befinden scheinen, kann das Medienvolumen erhöht werden.
Ergebnisse
Der "Sandwich"-Aufbau für das Resipher-Experiment ist in Abbildung 2A dargestellt. Sensordeckel mit 32 Sonden, die den Spalten 3, 4, 9 und 10 auf der 96-Well-Platte entsprechen, befinden sich zwischen der Zellplatte und dem Gerät. Nach dem Anschließen an die Nabe aktiviert das Gerät Motoren, um den Sensordeckel auf und ab zu bewegen und dieO2-Konzentration in der Mediensäule in einem Höhenbereich über der Zellen-Monoschicht (typischerweise 1-1,5 mm) zu messen. DerO2-Gradient wird daher kontinuierlich gemessen, indem dieO2-Konzentrationen in diesen verschiedenen Höhen über der Monoschicht aufgezeichnet werden. Aus Gradientenmessungen und der Anwendung der Fickschen Diffusionsgesetze berechnet das Gerät automatisch den Sauerstofffluss von oberhalb des Sondenhöhenbereichs bis darunter, d. h. die Sauerstoffverbrauchsrate (OCR) der Zellmonoschicht. Der Messdeckel erkennt O2 unabhängig davon, ob eine Vertiefung leer ist (nur Luft), mit Medien, aber ohne Zellen gefüllt ist (nur Medien) oder ob Medien über einer Zellmonoschicht liegen (Medien + Zellen). Als Qualitätskontrolle für die Funktion der Sensoren im Messdeckel kann der Deckel zunächst über eine leere (nur luftgefüllte) 96-Well-Platte gelegt werden. Wenn jeder Sensor für jede Vertiefung funktioniert, sollten die angegebenen O2 -Werte konsistent miteinander sein und nahe bei 200 μM liegen, was nahe dem erwarteten O2 in der Atmosphäre unter den Standardbedingungen eines Zellkultur-Inkubators liegt. Abbildung 2Bi zeigt O2 -Messwerte von acht Sonden in atmosphärischer Luft (angegeben als Konzentration in μM, da bei der Standardeinheitenumrechnung davon ausgegangen wird, dass sich die Sonden in einer 37 °C warmen Flüssigkeit befinden). Diese Messwerte sind knapp (±5 %) und liegen bei 200 μM, was bedeutet, dass alle acht Sensoren gut funktionieren. In Abbildung 2Bii wird derselbe Sensordeckel wie in Abbildung 2Bi verwendet, aber es wird ein anderes Gerät eingesetzt. Hier werden zwei fehlerhafte Gerätesensoren anhand von Ausreißerkurven (in rot) aufgegriffen. Dies deutet darauf hin, dass das Gerät problematisch ist und ausgetauscht werden muss (oder die Vertiefungen, die fehlerhaften Sensoren entsprechen, sollten aus dem Experiment ausgeschlossen werden). In Figur 2Biii wird die gleiche Vorrichtung wie in Figur 2Bi verwendet, aber es werden zwei verschiedene Sensordeckel verwendet. Deckel1 (linke Seite) weist eine hohe Variabilität zwischen den Sonden auf, was darauf hindeutet, dass das Sensormaterial des Deckels abgebaut ist, in der Regel, weil der Deckel zu oft verwendet wurde. Lid2 (rechte Seite) ist neu und zeigt Messwerte, die eng sind und um 200 μM liegen. Mit dem System kann man die Wirkung einer Serumsupplementierung in den Medien auf den mitochondrialen RPE-Stoffwechsel analysieren. Wie in Abbildung 2C zu sehen ist, führte eine Erhöhung der Serumsupplementierung zu leicht höheren RPE-OCR-Spiegeln. Wichtig ist, dass eine höhere Serumsupplementierung auch eine längere Aufrechterhaltung des mitochondrialen Stoffwechsels ermöglichte. Auch ohne Serumsupplementierung wird die OCR etwa 90 Stunden lang aufrechterhalten, bevor die Erschöpfung der mitochondrialen Stoffwechselsubstrate einen Abfall der OCR auslöst. Daher ist ein Medienwechsel mit 100 μL pro 96-Well-Anwendung alle 3,5 Tage (zweimal pro Woche) auch bei serumfreien Medien ausreichend, um einen Nährstoffmangel in reifen, hochdifferenzierten und polarisierten RPE-Kulturen zu vermeiden.
Die OCR-Raten können von Vertiefung zu Vertiefung erheblich variieren, selbst in Kulturen, in denen alle Vertiefungen die gleiche Zellzahl und Morphologie zu haben scheinen. Um zu isolieren, ob der Anstieg der OCR auf eine experimentelle Bedingung oder einfach auf Basisunterschiede in der OCR zwischen den Bohrlöchern zurückzuführen ist, ist eine Normalisierung erforderlich. Der erste Schritt bei der Normalisierung besteht darin, sicherzustellen, dass die Zellzahlen zwischen den Wells konsistent sind, wie in Protokollabschnitt 1.3 beschrieben. Wenn die Zellzahlen zwischen den Vertiefungen variieren, muss die OCR für die Vertiefung auf die Zellzahl normalisiert werden. Im Allgemeinen sind die Zellzahlen zwischen den Wells bei hochreifen und polarisierten RPE-Kulturen bemerkenswert konsistent9. Selbst in solchen Szenarien kann die Basis-OCR zwischen den Bohrlöchern variieren, wie in Abbildung 3A gezeigt. Hier variiert die OCR zu Studienbeginn vor jeder Behandlung zwischen den Vertiefungen um bis zu 100 fmol/mm2/s. Durch die Messung der OCR vor der Behandlung kann man dann die durch die experimentelle Behandlung ausgelöste Delta-OCR bestimmen und diese von etwaigen Unterschieden in der OCR zu Studienbeginn zwischen den Wells isolieren. In diesem Fall vergleichen die Behandlungen zwei mitochondriale Entkoppler (FCCP vs. BAM15) mit der Vehikelkontrolle. Sobald die OCR zu Studienbeginn für die Normalisierung verwendet wird, werden die Behandlungseffekte, die für die Entkoppler im Vergleich zur Kontrolle spezifisch sind, offensichtlich (vgl. Abbildung 3B vs. Abbildung 3C). Im Allgemeinen sollten Experimente, die die OCR verändern können, in mindestens zwei Phasen unterteilt werden: die anfängliche OCR, um eine Ausgangs-OCR zu etablieren, und eine Behandlungsphase, um die Delta-OCR zu bestimmen.
Um zu bestätigen, dass RPE-Kulturen in erwarteter Weise auf mitochondriale Manipulation reagieren, und um ein bioenergetisches Profil für RPE zu erstellen, können die klassischen niedermolekularen Werkzeuge, die im mitochondrialen Stresstest auf dem Seahorse Analyzer von Agilent verwendet werden, mit dem System verwendet werden. Zunächst kann die freie mitochondriale Kapazität mit Hilfe eines mitochondrialen Entkopplers19 gemessen werden. In RPE-Kulturen zeigt der mitochondriale Entkoppler BAM15 einen deutlich robusteren und nachhaltigeren Anstieg der OCR im Vergleich zu FCCP (Abbildung 3C,D), ohne Toxizität zu induzieren. Andere Parameter, die im mitochondrialen Stresstest des Seahorse Analyzer verfügbar sind, einschließlich der basalen Atmung, der ATP-gebundenen Atmung, des Protonenlecks und der nicht-mitochondrialen Atmungskapazität, können mit dem System berechnet werden. Während der Seahorse Analyzer auf Injektionsöffnungen angewiesen ist, um nacheinander Toxine abzugeben, die spezifische oxidative Phosphorylierungskomplexe hemmen, um die oben genannten Parameter abzuleiten, verfügt Resipher nicht über diese Fähigkeit. Jedes mitochondriale Toxin kann jedoch in separate Vertiefungen gegeben werden, und verschiedene Atmungsparameter können auf der Grundlage der Faltenänderung über Vertiefungen berechnet werden, die mit Vehikel behandelt wurden. In Abbildung 4A werden die Unterschiede in der OCR zwischen Vertiefungen, die mit dem mitochondrialen Entkoppler BAM15, dem ATP-Synthase-Inhibitor Oligomycin, den Komplex-I- und -III-Inhibitoren Antimycin/Rotenon und Vehikelkontrolle (DMSO) behandelt wurden, verglichen. Da diese Toxine über längere Zeiträume den Zelltod induzieren können und der Zelltod die OCR beeinflusst, wurden die OCR-Messungen nach nur wenigen Stunden Behandlung durchgeführt. Abbildung 4B zeigt die Unterschiede in der OCR zwischen Vehikel und Wells, die mit jedem mitochondrial aktiven kleinen Molekül in Form eines Balkendiagramms behandelt wurden, extrapoliert von den letzten Zeitpunkten in Abbildung 4A. Die OCR-Werte in Abbildung 4B können dann verwendet werden, um bioenergetische Parameter für die RPE-Kultur zu berechnen, wie in Abbildung 4C zu sehen ist. Jeder farbcodierte Wert im bioenergetischen Profil in Abbildung 4C stammt von demselben farbcodierten Wert in Abbildung 4B. So können Parameter wie ATP-gebundene Atmung, Protonenleck, maximale mitochondriale Atmungskapazität und nicht-mitochondriale Atmungskapazität mit dem OCR-System geschätzt werden. Eine Zusammenfassung der Wirkungsweise jedes mitochondrial aktiven kleinen Moleküls ist in Abbildung 4D dargestellt.
Nachdem das Protokoll für dieses System an RPE-Kulturen und Kontrollexperimenten zum Assay auf bioenergetische RPE-Profile demonstriert wurde, kann man experimentelle Anwendungen des Systems in der RPE-Biologie und -Pathologie untersuchen. Bei der proliferativen Vitreoretinopathie (PVR), einer Erkrankung, die nach einer Netzhautablösung oder einem Augentrauma auftritt, erfährt die RPE eine dramatische Transformation, die als epithelial-mesenchymale Transition (EMT) bekannt ist, wobei die hochregelmäßigen und kopfsteinpflasterartigen RPE ihre Zell-Zell-Adhäsionen verlieren und sich in spindelförmige mesenchymale Zellen transdifferenzieren, die kontraktil und beweglich sind 1,2. Dies löst tangentiale kontraktile Kräfte auf der Netzhaut aus, die eine Netzhautablösung induzieren können. Zu den klassischen Induktoren der EMT bei RPE gehören der transformierende Wachstumsfaktor-beta (TGFβ) und der Tumornekrosefaktor-alpha (TNFα)10,11,12. Von den drei TGFβ-Isoformen der Säugetiere ist TGFβ2 der prominenteste und potenteste Induktor von EMT in der Netzhaut1, was zu einer deutlichen zellulären Elongation der charakteristisch hexagonalen RPE-Zellen und einer erhöhten Expression mesenchymaler Marker führt. Die durch TGFβ2 induzierte EMT-Antwort bei RPE geht mit einer Unterdrückung der mitochondrialen Atmung und einer anschließenden Erhöhung der glykolytischen Kapazität einher, wie zuvor mit einem Seahorse XFe9610 gezeigt wurde. Der Seahorse liefert zwar bioenergetische Profile in Echtzeit, ermöglicht aber keine langfristige OCR-Überwachung. So wurde in der ersten Anwendung des OCR-Systems in dieser Studie (Abbildung 5) die mitochondriale Atmung über drei Wochen verfolgt, während EMT mit TGFβ2 in RPE induziert wurde, was eine In-vitro-Modellierung der metabolischen Reprogrammierung ermöglicht, die während der PVR stattfindet. Im Gegensatz zur Einzelpunktanalyse von RPE-EMT mit einem Seahorse Analyzer kann der Stoffwechselzustand von RPE, das sich einer EMT unterzieht, über mehrere Wochen verfolgt werden. Es ist offensichtlich, dass die wiederholte Exposition von RPE gegenüber TGFβ2 zu einem anhaltenden fortschreitenden Rückgang des mitochondrialen Stoffwechsels führt. Frühere Daten mit dem Seahorse XF Analyzer zeigten, dass TGFβ2 die maximale Atmungskapazität nach 24 Stunden und 72 Stunden signifikant reduzierte, aber die basalen OCR-Werte nichtbeeinflusste 10. Die unveränderte basale OCR mit TGFβ2 bis zu 72 h wird durch das in dieser Studie verwendete OCR-System bestätigt (Abbildung 5). Die längerfristigen Daten des OCR-Systems in dieser Studie zeigen jedoch, dass nach 5 Tagen Exposition gegenüber TGFβ2 die basalen OCR-Werte zu sinken beginnen, wobei ein Abfall der basalen OCR nach 20 Tagen am deutlichsten wird. Diese Ergebnisse unterstreichen langfristige Veränderungen der basalen OCR, die mit früheren Seahorse-Zeitpunkten nicht erfasst wurden.
In einer zweiten Anwendung kann das System zur Überwachung des mitochondrialen RPE-Stoffwechsels unter hypoxischen Bedingungen eingesetzt werden. Da AMD mit einer Ausdünnung der Choriokapillaris und Hypoxie verbunden ist13,20, sollte das Verständnis der Anpassungen des RPE an subtoxische Hypoxie Einblicke in die Pathogenese der AMD geben. Um RPE-Kulturen einer kontrollierten Hypoxie zu unterziehen und gleichzeitig den mitochondrialen Stoffwechsel zu überwachen, kann das System in eine Hypoxiekammer gebracht werden, die wiederum in einem Zellkultur-Inkubator platziert wird. Um diese Einrichtung zu erleichtern, wird ein kleines Loch in den Deckel einer Hypoxiekammer gebohrt, durch das das USB-Kabel des Systems in die Hypoxiekammer gelangen kann. Das Loch wird mit Kitt oder Silikon verschlossen (Abbildung 6A). Ein tragbarer O2 -Sensor kann in der Hypoxiekammer platziert werden, um die atmosphärischen O2 -Werte zu überwachen und sicherzustellen, dass die Dichtung der Hypoxiekammer intakt bleibt. Mit diesem Aufbau kann die Äquilibrierungsrate zwischen der atmosphärischenO2-Konzentration und derO2-Konzentration des Mediums bestimmt werden. In Abbildung 6B werden mit atmosphärischem O2 äquilibrierte Medien in einzelnen Vertiefungen auf einer 96-Well-Platte ohne Zellen platziert und in eine Hypoxiekammer eingeführt, die nur 5 % O2 (~50 μM O2) enthält. Im Laufe der Zeit gleicht sich das Medium in jeder Vertiefung mit der neuen atmosphärischen Konzentration aus. Je höher die Höhe der Mediensäule (mehr Volumen), desto länger dauert die Äquilibrierung. Mit 65 μl Medium in einer Vertiefung einer 96-Well-Platte dauert das Gleichgewicht mit atmosphärischem O2 etwa 5 Stunden. Die Gleichgewichtszeit dauert jedoch mehr als 10 Stunden, wenn das Medienvolumen 100-200 μL beträgt. Diese Ergebnisse unterstreichen, wie wichtig es ist, Medien mit der gewünschten atmosphärischenO2-Konzentration vorauszugleichen, bevor das Medium während eines Hypoxie-Experiments auf Zellen aufgetragen wird.
Bei Hypoxie-Experimenten ist es wichtig, sicherzustellen, dass die O2 - Spiegel in der Hypoxiekammer stabil bleiben. Wenn eine der Randvertiefungen auf der 96-Well-Platte frei von Zellen oder Medien gehalten wird, kann einer der Sensordeckelsensoren das atmosphärische O2 kontinuierlich überwachen. In Abbildung 6C zeigt eine solche Überwachung mit einer einzelnen Vertiefung, die Luft ausgesetzt war, dass die Hypoxiekammer ein langsames Leck aufweist, so dass die Zellen nach 30 Stunden nahe an den atmosphärischenO2-Konzentrationen liegen. Als abschließende Kontrolle für Hypoxie-Experimente ist es wichtig zu bestimmen, ob sich die O2 -Löslichkeit zwischen "frischen" und "konditionierten" Medien unterscheidet. Wenn sich die Löslichkeit zwischen Medien, die ursprünglich auf Zellen platziert wurden, und Medien, die sich über eine signifikante Zeitspanne auf Zellen befunden haben, unterscheidet, dann unterscheidet sich derO2-Gradient in der Medienspalte über den Zellen als Medien-"Bedingungen" mit der Zeit über den Zellen. Dies wirkt sich wiederum auf die Berechnung der OCR aus. In Abbildung 6D wurde eine 96-Well-Platte mit leeren Wells ("Luft"), Wells ohne Zellen, aber mit frischem Medium ("neues Medium") und Wells ohne Zellen, aber mit Medien, die zuvor 48 Stunden lang über RPE-Kulturen inkubiert wurden ("konditioniertes Medium"), zuerst in atmosphärisches O2 gelegt, um eine vollständige Äquilibrierung zu ermöglichen. Die Platte wurde dann in eine Hypoxiekammer bei etwa 3-4% O2 (30-40 μM) gebracht und äquilibrieren gelassen. Schließlich wurde die Platte wieder auf atmosphärischeO2 -Konzentrationen gebracht. DieO2-Konzentrationskurven für neue und konditionierte Medien sind identisch und zeigen, dass dieO2-Löslichkeit zwischen neuen und konditionierten Medien identisch ist, was bestätigt, dass Änderungen in der Medienzusammensetzung im Laufe der Zeit, wenn Nährstoffe verbraucht und Nebenprodukte in das Medium abgegeben werden, die Löslichkeit von O2 nicht signifikant verändern und daher die OCR nicht versehentlich beeinflussen.
Da die Höhe der Mediensäule über den Zellen dieO2-Verfügbarkeit in der RPE-Monoschicht9 dramatisch beeinflusst, ist es wichtig zu bestimmen, ob ein bestimmtes Medienvolumen Hypoxie, Normoxie oder Hyperoxie an der RPE-Monoschicht induziert. Anhand von Messungen aus dem System, kombiniert mit einem Online-Rechner (https://lucidsci.com/notes?entry=oxygen_diffusion (und in Form eines interaktiven Open-Source-Notizbuchs unter https://observablehq.com/@lucid/oxygen-diffusion-and-flux-in-cell-culture) - Quellcode dieses Rechners bei https://github.com/lucidsci/oxygen-diffusion-calculator), kann die Sauerstoffkonzentration an der RPE-Monoschicht abgeschätzt werden. Abbildung 7 zeigt einen Screenshot des interaktiven Rechners. RPE in vivo weist typischerweiseeine O2-Konzentration von 4-9 % auf, was zueiner molaren O2-Konzentration von ~40-90 μM führt.

Abbildung 1: Position der Sensoren auf verschiedenen Sensordeckeln, Plattenlayout für den 32-Kanal-Sensordeckel und RPE-Morphologie. Vertiefungen, die der Position der Sensoren in den verschiedenen Sensordeckeln für die Deckeloptionen (A) 4-Kanal, (B) 32-Kanal und (C) 96-Kanal-Deckel entsprechen. (D) Empfohlene Plattenanordnung für den 32-Kanal-Sensordeckel. Aufgrund von Kanteneffekten sollten Zellen nicht in die Randvertiefungen gesät werden, sondern Medien müssen in diese vier Vertiefungen (violett) gelegt werden. Die Zellen werden in orangefarbene Vertiefungen ausgesät. Die restlichen Vertiefungen sollten mit sterilem Wasser (blau) gefüllt werden, um Verdunstungseffekte zu vermeiden. (E) Reife RPE-Kultur mit hexagonalen, dicht gepackten, pigmentierten Zellen. Maßstabsleiste = 20 μm. Abkürzungen: RPE = retinales Pigmentepithel. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Aufbau des "Sandwiches" des Resipher-Systems und Qualitätskontrolle. (A) Der Messdeckel ersetzt einen standardmäßigen 96-Well-Plattendeckel und wird mit seinen Sonden in die entsprechenden Wells eingesetzt. Das Gerät in Schwarz sitzt über Magnete fest auf dem Sensordeckel. Die Bilder auf der linken Seite der Abbildung zeigen das Gerät und den Sensordeckel in ihrer korrekten Ausrichtung. Für die Fotos auf der rechten Seite wurden das Gerät und der Sensordeckel jedoch zur Seite gedreht, um das Unterprofil besser sichtbar zu machen. (B) Qualitätskontrolle für die Sensordeckel und das Gerät. (i,ii) Derselbe Sensordeckel wird in eine leere 96-Well-Empfängerplatte (nur Luft) eingesetzt, und zwei verschiedene Geräte werden nacheinander auf dem Sensordeckel platziert. (i) Das erste Gerät demonstriert Daten von acht Sonden mit geringer Variabilität und O2 -Messwerten von ~200 μM, die für atmosphärisches O2 erwartet werden. (ii) Das zweite Gerät zeigt Daten von acht Sonden, bei denen zwei Sonden anomal lesen (rote Punkte). (iii) Ein Gerät wurde mit einer leeren 96-Well-Platte (nur Luft) verwendet, aber mit zwei verschiedenen Sensordeckeln, die nacheinander getestet wurden. Es wurden Daten von 16 Sonden aufgetragen, die eine große Variabilität für Lid1 (linke Seite) und eine geringe Variabilität für Lid2 (rechte Seite) zeigen. (C) OCR-Plots von RPE-Kulturen in 100-μl-Medien mit unterschiedlichen Serummengen wurden 120 Stunden lang überwacht. RPE-Zellen in serumfreien Medien (magenta) haben eine niedrigere OCR, wobei die OCR früher abfällt als bei Medien mit höheren Serummengen. Abkürzungen: OCR = Sauerstoffverbrauchsrate; RPE = retinales Pigmentepithel. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
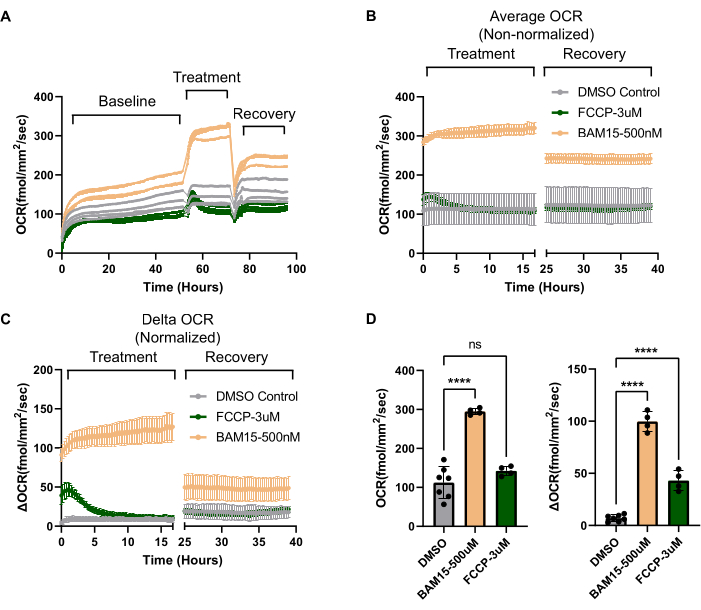
Abbildung 3: Langzeitwirkungen von mitochondrialen Entkopplern, die mit dem OCR-System eindeutig demonstriert wurden. (A) OCR-Rohdaten über die Zeit (Stunden), die die anfängliche OCR (etabliert über 50 Stunden), die Behandlungsphase (~20 h nach der Zugabe von Medikamenten) und die Erholungsphase (~20 Stunden nach dem Auswaschen des Arzneimittels) hervorheben. RPE wurde mit zwei verschiedenen mitochondrialen Entkopplern, FCCP (3 μM, N = 4) oder BAM15 (500 nM, N = 4) behandelt und mit der DMSO-Vehikelkontrolle (0,6%, N = 7) verglichen. Jede Spur stellt eine einzelne Vertiefung dar. (B) Durchschnittliche OCR aller Wells für jede Erkrankung in (A), dargestellt für die Behandlungs- und Erholungsphasen. Ohne Normalisierung (Y-Achse ist absolute OCR) ist es schwierig, das Ausmaß des Behandlungseffekts mit FCCP zu erkennen. (C) Wenn jede OCR-Spur vor der Behandlung von ihrer OCR subtrahiert wird, kann eine Delta-OCR berechnet werden (dargestellt auf der Y-Achse). Dies ermöglicht es, die Auswirkungen der Behandlung auf die OCR zu isolieren, trotz der Variabilität der OCR zu Studienbeginn zwischen den Wells. Mittels Delta OCR wird die Wirkung von FCCP deutlich. Es wird auch deutlich, wie kurzlebig die Wirkung von FCCP auf die mitochondriale Entkopplung im Vergleich zu BAM15 ist. Schließlich wird deutlich, dass im Vergleich zum FCCP die Exposition gegenüber BAM15, selbst nachdem das Medikament abgewaschen wurde, einen neuen "angepassten" Zustand mit anhaltend höherer OCR erzeugt. (D) Die Diagramme in (B) und (C) werden in Form von Balkendiagrammen angezeigt, was einen direkten Vergleich zwischen nicht normalisierten (linkes Diagramm, von (B)) und normalisierten (Delta OCR, rechtes Diagramm, von (C)) Daten ermöglicht. Abkürzungen: OCR = Sauerstoffverbrauchsrate; RPE = retinales Pigmentepithel; FCCP = Carbonylcyanid-p-trifluormethoxyphenylhydrazon; DMSO = Dimethylsulfoxid. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
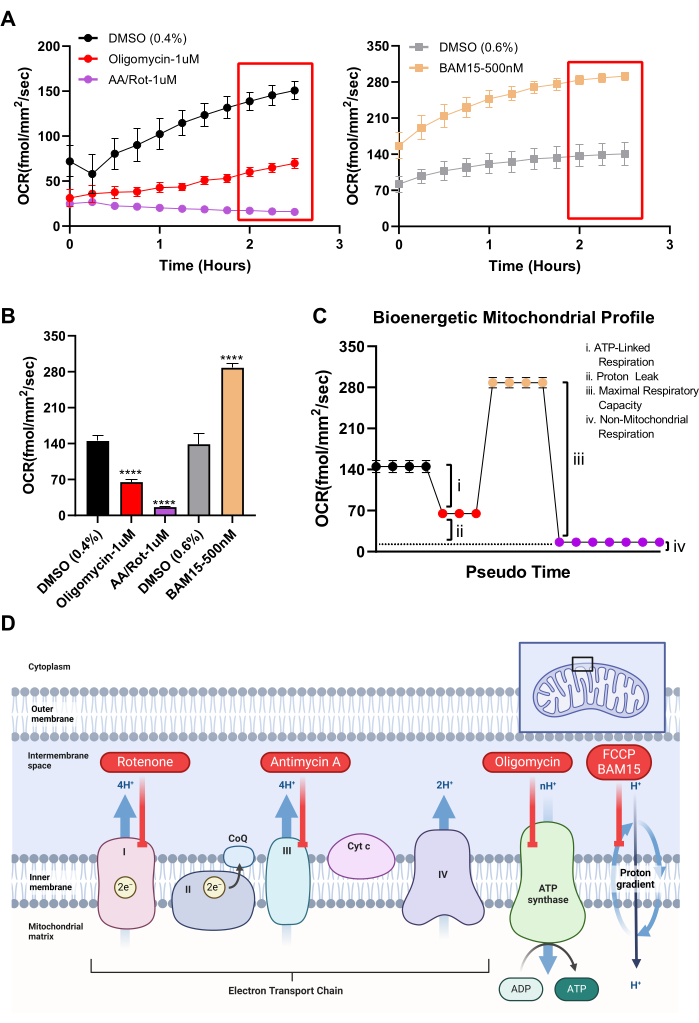
Abbildung 4: Verwendung eines OCR-Systems zur Berechnung von Parametern der mitochondrialen Bioenergetik. (A) Unter Verwendung der gleichen kleinen Moleküle, die Teil des mitochondrialen Stresstests des Seahorse Analyzer sind, wird die OCR über die ersten 2,5 Stunden der Behandlungsphase für RPE-Kulturen dargestellt. (Linke Grafik) Der ATP-Synthase-Inhibitor Oligomycin (N = 3), die Complex-I- und III-Inhibitoren Antimycin A/Rotenon (N = 7) oder Vehikel, DMSO 0,4% (N = 4). (Rechtes Diagramm) Der mitochondriale Entkoppler BAM15 (N = 4) oder Vehikel DMSO 0,6% (N = 4). (B) Durchschnitt der letzten drei Zeitpunkte für jede Behandlungsgruppe (rote Kästchen in (A)). (C) Darstellung eines simulierten bioenergetischen Profils des Seahorse Analyzer Mitochondrial Stress Tests auf der Grundlage von Resipher-Daten. Die farbcodierten Datenpunkte im simulierten Diagramm entsprechen den farbcodierten Balkendiagrammen in (B). Alle respiratorischen Parameter können mit Resipher-Daten und ausgewählten kleinen Molekülen berechnet werden. (D) Diagramm der Wirkungsweise der in den Abbildungen 3 und 4 verwendeten Reagenzien.Abkürzungen: OCR = Sauerstoffverbrauchsrate; RPE = retinales Pigmentepithel; DMSO = Dimethylsulfoxid; AA = Antimycin A; Fäulnis = Rotenon. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Langzeit-OCR-Überwachung von RPE im Übergang von Epithel zu Mesenchym, stimuliert durch TGFβ2. Reife primäre humane RPE-Kulturen wurden alle 2-3 Tage mit 10 ng/ml TGFβ2 oder Vehikelkontrolle behandelt. (A) Echtzeit-OCR-Messungen wurden 3 Wochen lang mit ( B) Quantifizierung der Delta-OCR über die Zeit (Tag 19-Tag 6) überwacht. N = 6-7 Wells pro Bedingung, ungepaarter t-Test, * P < 0,05. Abkürzungen: OCR = Sauerstoffverbrauchsrate; RPE = retinales Pigmentepithel; TGFβ2 = transformierender Wachstumsfaktor beta-2. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6: Überwachung der mitochondrialen Reaktion von RPE auf Hypoxie. (A) Das OCR-System "Sandwich" wurde in einer Hypoxiekammer montiert und das USB-Kabel über ein in den Deckel gebohrtes Loch mit dem Hub verbunden und mit Silikonfett oder Kitt verschlossen. Ebenfalls in der Hypoxiekammer befindet sich eine Petrischale mit sterilem Wasser zur Aufrechterhaltung der Luftfeuchtigkeit und ein tragbarer O2 -Sensor. Das gesamte System wurde in einem Zellkultur-Inkubator belassen, nachdem die Atmosphäre der Hypoxiekammer durch eine niedrigereO2-Konzentration ersetzt worden war. (B) Das Medium benötigt Zeit, um sich auf O2 - Konzentrationen oberhalb der Grenzfläche zwischen Luft und Flüssigkeit auszugleichen. Unterschiedliche Volumina von RPE-Medien, die mit atmosphärischem O2 äquivalent sind, wurden in leeren Vertiefungen (keine Zellen) einer 96-Well-Platte zugegeben. Die Hypoxiekammer wurde dann auf 5% (50 μM) O2 eingestellt. DieO2-Konzentration in den Medien jeder Vertiefung wurde 20 Stunden lang überwacht. Vertiefungen mit höheren Medienvolumina äquilibrierten langsamer auf 5 % O2 als niedrigere Medienvolumina. N = 8. (C) Das System kann zur Erkennung von Luftlecks in der Hypoxiekammer verwendet werden. Die Hypoxiekammer wurde mit 1 % O2 aufgebaut und der Messdeckel über leere Vertiefungen (nur Luft) gelegt. Die Einlass- und Auslassöffnungen der Hypoxiekammer wurden offen gelassen und die Atmosphäre der Hypoxiekammer über ~90 h wieder auf atmosphärische O2 -Werte ausgeglichen. (D) Neue Medien und Überstände ("konditionierte Medien") haben eine ähnliche O2 -Löslichkeit. Dadurch wird die Sorge um die unterschiedliche Löslichkeit vonO2 -Medien beseitigt, da sich ihre Zusammensetzung während der Kultur ändert, was sich auf die Konsistenz der OCR-Messwerte im Laufe der Zeit auswirken würde. Das gleiche Volumen (100 μl) frisches Medium ("neues Medium") oder Medium, das 48 h lang über Zellen inkubiert wurde ("konditioniertes Medium"), wurde in Vertiefungen einer Platte ohne Zellen gegeben. Das OCR-System "Sandwich" wurde zunächst atmosphärischenO2-Niveaus ausgesetzt, dann in die Hypoxiekammer (3-5% O2 oder 30-50 μM) und dann wieder in atmosphärischeO2-Niveaus überführt. Die Konzentrationen von O2 in der Luft wurden durch Sonden in leeren (nur lufthaltigen) Brunnen überwacht. Die Änderung derO2-Konzentration bei jedem Übergang zwischen den beiden Medientypen ist identisch, was zeigt, dass dieO2-Löslichkeit zwischen den beiden Medientypen identisch ist. N = 6. Abkürzungen: OCR = Sauerstoffverbrauchsrate; RPE = retinales Pigmentepithel. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 7: O2 -Konzentrationen an der RPE-Monoschicht, bestimmt mit einem Online-Rechner. Screenshot des Online-Rechners (https://observablehq.com/@lucid/oxygen-diffusion-and-flux-in-cell-culture). Siehe Text für Details, wie der Rechner verwendet werden kann, um die Menge an Sauerstoff zu bestimmen, die an der RPE-Monoschicht verfügbar ist. Abkürzungen: RPE = retinales Pigmentepithel. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
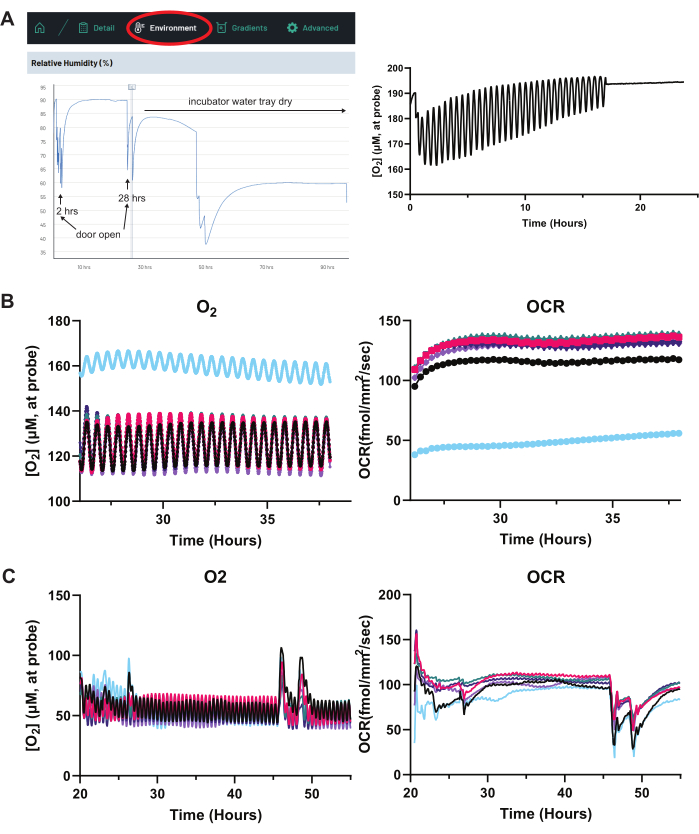
Abbildung 8: Fehlerbehebung bei Experimenten: Verdunstung, ungesunde Zellen und übermäßige OCR-Schwankungen. (A) Übermäßige Verdunstung. Wenn es Unregelmäßigkeiten in der OCR-Ablaufverfolgung für ein Bohrloch gibt, klicken Sie zunächst auf die Registerkarte Umgebung in der Online-Oberfläche des Systems (links). Hier können Luftfeuchtigkeit, Temperatur und Luftdruck überwacht werden. Durch das Öffnen des Zellkultur-Inkubators wird die Luftfeuchtigkeit vorübergehend gestört (siehe 2 h und 28 h auf der Spur). Ab 30 h sinkt die Feuchtekurve jedoch im Laufe der Zeit stetig ab, auch ohne Türöffnung. Dies deutet darauf hin, dass die Wasserschale des Zellkultur-Inkubators trocken ist. Jede Störung der Luftfeuchtigkeit oder Temperatur verändert die OCR-Messwerte. (Rechts) Niedrige Luftfeuchtigkeit verstärkt die Verdunstung. In der Spur auf der rechten Seite zeigt ein Bohrloch, das einer schnellen Verdampfung unterzogen wird, zunehmend kleinere Unterschiede in derO2-Konzentration zwischen der Ober- und Unterseite der Sensorsondenauslenkung (1 mm bis 1,5 mm über der RPE-Monoschicht). Dies deutet darauf hin, dass sich die Sondenspitze in der Nähe der Luft-Medien-Schnittstelle befindet. Wenn derO2-Konzentrationswert bei einem Wert nahe den bekannten atmosphärischenO2-Konzentrationen (~200 μM) "flacht", deutet dies darauf hin, dass die Sondenspitze vollständig außerhalb der Mediensäule liegt und nur Luft probenimmt. OCR-Messwerte sind nicht mehr gültig, und solche Bohrlöcher müssen sofort in den Medien geändert werden. (B) Ungesunde Zellen. Die Darstellung derO2- und OCR-Spur jedes einzelnen Wells ermöglicht die Identifizierung eines Ausreißer-Wells, der in der Regel durch fehlerhafte Zellen verursacht wird. Hier ist das durch die blaue Spur gekennzeichnete Gut fehlerhaft. N = 6 Vertiefungen. (C) Türöffnungen des Inkubators und Medienwechsel. Änderungen der Luftfeuchtigkeit, der Temperatur, des Drucks und des CO2 -Gehalts während des Öffnens der Inkubatortür sowie alle Medienwechsel stören vorübergehend den im Bohrloch etablierten O2-Gleichgewichtsgradienten, was zu abweichendenO2- und OCR-Spitzen zu Beginn und Ende dieser Diagramme führt. N = 6 Vertiefungen. Abkürzungen: OCR = Sauerstoffverbrauchsrate. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
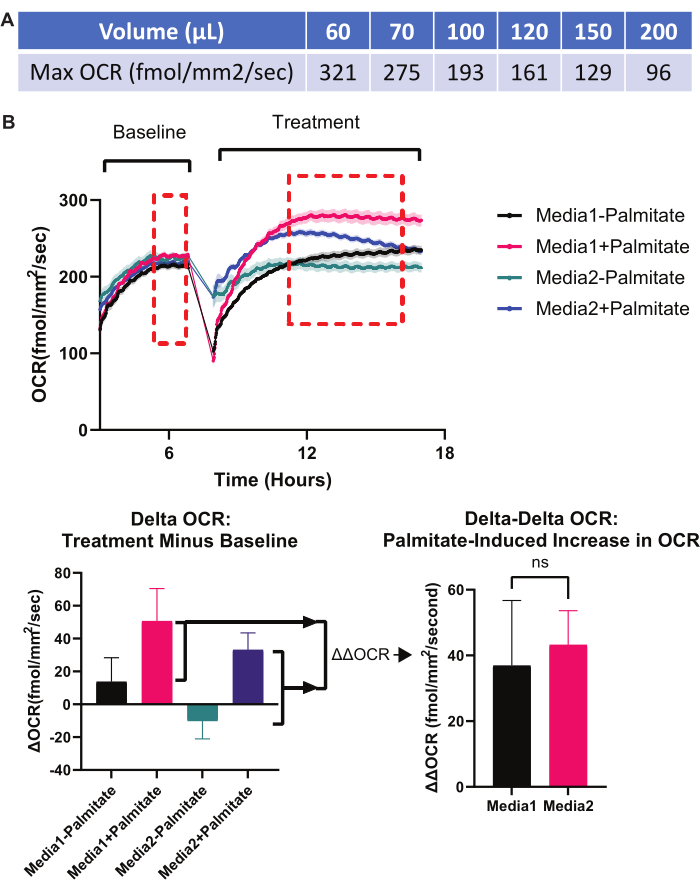
Abbildung 9: Problembehandlung bei Experimenten: Medienvolumen und Normalisierung. (A) Die Medienlautstärke begrenzt die maximal mögliche OCR. Da höhere Medienvolumina zu einer geringerenO2-Verfügbarkeit am RPE-Monolayer führen, hat jedes Medienvolumen in einem einzelnen Well einer 96-Well-Platte eine maximale OCR. Oberhalb dieser OCR liegt derO2-Gehalt auf der Ebene der Zellen im Wesentlichen bei 0 % (anoxisch). Wenn man also nahe an der maximalen theoretischen OCR für ein bestimmtes Medienvolumen liegt, ist es sinnvoll, das Medienvolumen zu verringern, so dass im Verlauf des Experiments kein OCR-"Deckeneffekt" zu sehen ist. Die maximale theoretische OCR, die bei jedem Medienvolumen (in einer standardmäßigen 96-Well-Platte) erreichbar ist, wird angezeigt. Alle Werte wurden mit dem Online-Rechner berechnet, der in Protokollabschnitt 4 besprochen und in Abbildung 7 dargestellt ist. (B) Angemessene Normalisierung. Für Experimente, bei denen man sich für die Wirkung einer Variablen auf die Fähigkeit einer anderen Variablen interessiert, den mitochondrialen Stoffwechsel zu induzieren, ist ein Delta-Delta-OCR-Versuchsaufbau ideal. Im Aufbau in (B) wird die Fähigkeit von zwei verschiedenen Medien getestet, die β-Oxidation von Palmitat zu fördern. Kommt es zu einer β-Oxidation der Fettsäure, sollte es zu einem signifikanten Anstieg der mitochondrialen OCR kommen. Einige Medien können die Palmitat-β-Oxidation besser fördern als andere Medien. Auf der linken Seite des OCR-Diagramms werden zwei verschiedene Medien (Medien 1 vs. Medien 2) in parallele Vertiefungen von RPE-Kulturen eingeführt. Die OCR zu Baseline wird erhalten. Als nächstes wird Palmitat in die Hälfte der Vertiefungen gegeben, die jeden Medientyp enthalten. Dadurch entstehen vier Bedingungen: Medium 1 - Palmitat, Medium 1 + Palmitat, Medium 2 - Palmitat, Medium 2 + Palmitat. Die OCR-Reaktion auf jede dieser neuen Medienbedingungen wird aufgezeichnet. Als Nächstes werden die OCR-Werte nach experimentellen Behandlungen (Durchschnitt des roten gestrichelten Kästchens in der Behandlungsphase) von den OCR-Ausgangswerten vor der Behandlung subtrahiert (Durchschnitt des roten gestrichelten Kästchens in der Ausgangsphase), wodurch Delta-OCR-Balkendiagramme (unten links) erstellt werden. Schließlich wird die Fähigkeit von Medium 1 vs. Medium 2, die β-Oxidation zu fördern, in einem Delta-Delta-OCR-Balkendiagramm (unten rechts) bestimmt. Hier wird die Differenz zwischen "Delta OCR von Medium 1 + Palmitat" und "Delta OCR von Medium 1 - Palmitat" berechnet und mit der Differenz zwischen "Delta OCR von Medium 2 + Palmitat" und "Delta OCR von Medium 2 - Palmitat" verglichen. Diese Art des Versuchsaufbaus und der Normalisierung isoliert die Effekte von Medium 1 vs. Medium 2 auf die Förderung der β-Oxidation; Die allgemeine Struktur dieser Normalisierung ist auf jedes Experiment anwendbar, bei dem die Wirkung einer Variablen auf die Fähigkeit einer anderen Variablen, die OCR zu verändern, gemessen wird. Abkürzungen: OCR = Sauerstoffverbrauchsrate; RPE = retinales Pigmentepithel. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Das Rezept für RPE-Zellkulturmedien. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Der mitochondriale Metabolismus des RPE spielt eine entscheidende Rolle bei der Pathogenese häufiger erblindender Netzhauterkrankungen, einschließlich AMD und PVR. Die Modellierung des mitochondrialen RPE-Stoffwechsels in vitro ermöglicht es, seinen Stoffwechselzustand von dem des umgebenden Gewebes zu isolieren und das Gewebe auf kontrollierte Weise verschiedenen krankheitssimulierenden Beleidigungen auszusetzen. Eine solche in vitro-Modellierung des mitochondrialen RPE-Stoffwechsels wurde durch das Aufkommen von humanen Primär- und iPSC-RPE-Kulturen mit hoher Genauigkeit erleichtert, die den richtigen Differenzierungszustand und die richtige Polarität erreichen, die für eine enge Annäherung an RPE in vivo erforderlich sind. Während die Überwachung des Sauerstoffverbrauchs (OCR) in solchen Kulturen eine ideale Methode zur Überwachung des mitochondrialen Stoffwechsels ist, schließt das Setup für die OCR-Überwachung oft aus, Kulturen unter den Bedingungen zu züchten, die für eine stark polarisierte und vollständig differenzierte RPE erforderlich sind. Hier beschreiben wir Resipher als ein neuartiges System zur Überwachung des mitochondrialen RPE-Stoffwechsels durch Echtzeit-Langzeitbewertung der OCR in hochdifferenzierten und polarisierten RPE-Kulturen.
Andere Methoden wurden verwendet, um OCR in RPE zu überwachen. Der Seahorse XF Analyzer wurde entwickelt, um OCR über kurze Zeiträume mit akuten Manipulationen der Mitochondrienfunktion zu überwachen. Seahorse-Assays erfordern jedoch, dass Zellen auf speziellen Platten gezüchtet werden, die nicht förderlich für die Unterstützung einer hohen RPE-Polarität und -Differenzierung sind21. Calton, Vollrath und Kollegen adaptierten das Seahorse für die Beurteilung von hochdifferenzierten RPE-Kulturen, die auf mikroporösen Trägern gezüchtet wurden, indem sie die mikroporöse Membran in Stücke schnitten und in die Seahorse-Platte legten, aber diese Methode funktioniert immer noch nur für die kurzfristige Endpunktüberwachung von Kulturen, da die Zellen keinen Zugang zu CO2 haben Während des Assays kann der Assay nur unter unsterilen Bedingungen durchgeführt werden, und das Schneiden des mikroporösen Trägers führt im Laufe der Zeit zum Tod und zur Funktionsstörung des Randteils des mikroporösen Trägers22. Die Barofuse ist ein pumpenloses Perfusionssystem, bei dem RPE-Streifen, die auf mikroporösen Trägern gewachsen sind, in das Perfusionssystem geladen werden können, wodurch die OCR in Echtzeit überwacht werden kann23. Ähnlich wie bei dem modifizierten Aufbau für das Seahorse von Calton, Vollrath und Kollegen sind die Haupteinschränkungen der RPE-Bewertung mit Barofuse die Notwendigkeit, die mikroporösen Stützen zu schneiden, und der kurzfristige Endpunktcharakter des Assays. Der Oxygraph ist auch für schnelle kinetische Veränderungen der OCR als Reaktion auf metabolische Manipulationen ausgelegt24. Es wurde bisher nicht für RPE-Kulturen verwendet und würde bei der Bewertung stark polarisierter und differenzierter RPE-Kulturen die gleichen Einschränkungen aufweisen wie das Seepferdchen und das Barofuse-Kulturen.
Im Gegensatz zu diesen anderen Verfahren ermöglicht das hier beschriebene OCR-System die Überwachung der OCR, während RPE in Standard-Zellkulturplatten/-substraten unter Standardbedingungen mit Standard-RPE-Medien in einem Standard-Zellkultur-CO 2-Inkubator kultiviert wird, was eine einfache und bequeme Sondierung von hochdifferenzierten und polarisierten RPE-Kulturen ermöglicht. Zusätzlich zur Generierung von Daten über Echtzeit-OCR-Änderungen durch das System können dieselben Zellen für zusätzliche Assays am Ende des OCR-Überwachungszeitraums verwendet werden. Dies ermöglicht hochflexible Experimente, bei denen die Wirkung einer Intervention auf die OCR über einen Zeitraum von Wochen wiederholt getestet wird (z. B. - RPE-EMT-Induktion in Abbildung 5) oder bei denen die langfristige Erholung von einer Intervention getestet werden kann (z. B. - Erholung von der mitochondrialen Entkopplung in Abbildung 3) oder bei denen kaskadierende metabolische Veränderungen als Reaktion auf eine erste Intervention überwacht werden können (z. B. - Auswirkungen des Serums auf die OCR, sobald die Mediennährstoffe erschöpft sind in Abbildung 2). Das OCR-System in diesem Artikel eignet sich besonders gut für die Messung von OCR in stark polarisierten und differenzierten RPE-Kulturen. Solche Kulturen sind funktionell postmitotisch, mit dichter Packung und starker Pigmentierung. Das bedeutet, dass jede Vertiefung nahezu identische Zellnummern9 enthält, wodurch eine Normalisierung der OCR auf die Zellzahl für die meisten Experimente entfällt. Selbst wenn eine Normalisierung auf die Zellzahl erforderlich ist, ermöglichen die starke RPE-Pigmentierung und die gut abgegrenzten Zellgrenzen die Normalisierung mit einer nicht-invasiven einfachen Zellzählung auf der Grundlage von Hellfeld-Bildgebung. Bei Experimenten mit undifferenziertem RPE und RPE-Proliferation, wie z. B. den EMT-Experimenten in Abbildung 5, ist eine Normalisierung der Per-Well-OCR auf die Zellzahl erforderlich, um zu wissen, was die OCR "pro Zelle" ist.
Außerhalb des retinalen Bereichs wurde das System eingesetzt, um die Zellatmung und die Stoffwechselaktivität in verschiedenen Zelltypen zu untersuchen, darunter Pankreas-Inselzellen25, Skelettmuskulatur26, Eierstockkrebs27, Kardiomyozyten28 und sogar ganze Organismen wie C. elegans29. Die Echtzeit-Langzeitüberwachung der OCR mit dem hier beschriebenen OCR-System lieferte wichtige Erkenntnisse über bioenergetische Verschiebungen und mitochondriale Dysfunktion in diesen verschiedenen Krankheitsmodellen.
Das Resipher-System hat Einschränkungen. Die Methode zur Bestimmung der OCR unterscheidet sich grundlegend von der des Seahorse Analyzers, BaroFuse oder Oxygraph. Es erfordert die Einrichtung eines O2 -Gradienten, um die OCR zu berechnen, und es kann Stunden dauern, bis der Gradient festgelegt ist. Schnelle kinetische Experimente, wie sie in den anderen Systemen möglich sind, sind mit diesem System daher nicht möglich. Darüber hinaus verfügt Resipher im Gegensatz zu den anderen Systemen über keine Injektionsöffnungen für die schnelle Abgabe kleiner Moleküle oder die Sammlung von Metaboliten. Stattdessen muss der Sensordeckel in einer Zellkulturhaube entfernt werden, wobei die Medikamente eingespießt oder das Medium ausgetauscht werden muss, zusammen mit der manuellen Entnahme der zu analysierenden Metaboliten.
Zum Einrichten und Beheben von Problemen mit dem in diesem Dokument beschriebenen OCR-System wird Folgendes empfohlen. Erstens: Minimieren Sie die Verdunstung. Denken Sie daran, dass derO2-Gradient in der Medienspalte über der RPE-Monoschicht stark von der Höhe der Spalte9 abhängt und das System die OCR basierend auf demO2-Gradienten berechnet. Wenn es also während des Experiments zu einer deutlichen Absenkung der Mediensäule aufgrund von Verdunstung kommt, ändert sich derO2-Gradient, was sich auf die OCR-Messwerte auswirkt. Es ist wichtig, die Wasserschale für den Inkubator voll zu halten, aber es wird auch empfohlen, Wasser in alle ungenutzten Vertiefungen der 96-Well-Platte zu geben. Darüber hinaus weisen die Vertiefungen am Rand der Platte höhere Verdampfungsraten auf, so dass diese Vertiefungen in typischen Experimenten, die oben vorgestellt wurden, leer von Zellen bleiben. Der Feuchtigkeitssensor des OCR-Systems kann bei der Überwachung der Luftfeuchtigkeit des Inkubators helfen. In Abbildung 8A deutet der Abfall der Luftfeuchtigkeit während des Experiments darauf hin, dass die Wasserschale des Inkubators leer ist. Wenn sich die Verdunstung im Bohrloch beschleunigt, steigen die O2-Konzentrationsmesswerte für das Bohrloch an und "flachen" schließlich bei der atmosphärischenO2-Konzentration (~200 μM) ab (Abbildung 8A).
Vergewissern Sie sich zweitens, dass das "Sandwich" des OCR-Systems ordnungsgemäß zusammengebaut ist. Die Metallkontaktpunkte an der Unterseite des Geräts und an der Oberseite des Sensordeckels sollten sauber sein, ohne Schmutz, der den vollen Kontakt behindert. Das Gerät und der Sensordeckel sollten vollständig bündig und gleichmäßig ausgerichtet auf der 96-Well-Platte angebracht werden, ohne dass es zu Neigungen kommt. Das USB-Kabel, das das Gerät mit dem Hub verbindet, sollte nicht unter Spannung stehen, was zu einem Drehmoment des "Sandwiches" und einer Einschränkung der ordnungsgemäßen Bewegung des Geräts führen kann.
Drittens: Stellen Sie sicher, dass die Sensoren des Geräts und des Sensordeckels kalibriert sind. Abbildung 2B zeigt, wie zu Beginn jedes Experiments schnell die korrekte Sensorfunktion überprüft werden kann. Viertens: Stellen Sie sicher, dass ein bestimmtes Bohrloch vor der experimentellen Manipulation keinen Ausreißer-OCR aufweist. Nachdem sichergestellt wurde, dass die Sensoren funktionieren, kann die Bestimmung der OCR-Ausgangswerte für alle Wells mit Zellen dabei helfen, festzustellen, ob bestimmte Wells ungesunde Zellen enthalten. Abbildung 8B zeigt mehrere Bohrlöcher, die einer OCR-Basisbewertung unterzogen werden, wobei die blau gefärbte Bohrung eindeutig eine niedrigere OCR (und damit eine hoheO2-Konzentration ) aufweist. Diese Vertiefung ist ein Ausreißer und sollte von experimenteller Manipulation ausgeschlossen werden.
Fünftens: Minimieren Sie Umgebungsmanipulationen während aktiver OCR-Messungen. DerO2-Gradient reagiert sehr empfindlich auf Änderungen der Temperatur, der Luftfeuchtigkeit, des CO2 - Gehalts des Inkubators und des Atmosphärendrucks. Das Öffnen der Inkubatortür beeinflusst diese Faktoren erheblich und führt zu vorübergehenden Unterbrechungen der OCR-Messwerte, bis derO2-Gradient der Mediensäule wieder ins Gleichgewicht zurückkehrt (Abbildung 8C). Parameter, die sich auf die OCR auswirken, können in der Online-Oberfläche des Systems unter der Registerkarte "Umgebung" überwacht werden (Abbildung 8A).
Sechstens: Bestimmen Sie das optimale Medienvolumen für das Experiment. Während ein unzureichendes Medienvolumen Artefakte durch übermäßige Verdunstung (siehe erste Fehlerbehebungsmaßnahme oben) oder vorzeitige Erschöpfung der Nährstoffe im Medium erzeugen kann, begrenzt ein übermäßiges Medienvolumen die Verfügbarkeit von O2 in der RPE-Monoschicht und löst Hypoxieaus 9. Abbildung 7 ist ein Screenshot des Online-Rechners, der zur Bestimmung des Einflusses des Medienvolumens auf dieO2-Konzentration an der RPE-Monoschicht zur Verfügung steht. Die maximal mögliche OCR für ein bestimmtes Medienvolumen ist in Abbildung 9A dargestellt. Wenn die OCR-Rate für ein gegebenes Experiment mit einem gegebenen Volumen annähernd der in Abbildung 9A gezeigten Rate entspricht, dann nähert sich dieO2-Konzentration in der RPE-Monoschicht der Anoxie (0%) und ein geringeres Medienvolumen ist erforderlich. Im Allgemeinen sollten Experimente mit mitochondrienintensiven Prozessen in RPE-Kulturen Medienvolumina von nicht mehr als 75 μl pro 96-Well-Kultur aufweisen.
Siebtens: Führen Sie eine geeignete Normalisierung durch, um die Auswirkungen eines experimentellen Eingriffs auf die OCR zu isolieren. Da verschiedene Wells unterschiedliche OCR-Ausgangswerte aufweisen können, ist es wichtig, vor einem experimentellen Eingriff für jedes Well OCR-Basiswerte zu erhalten. Subtrahieren Sie die OCR zu Studienbeginn von der OCR nach der experimentellen Intervention, um die "Delta-OCR" zu erhalten, die die Wirkung der experimentellen Intervention auf die OCR zeigt. Die Methode zum Abrufen von "Delta OCR" ist in Abbildung 3 und Abbildung 9B dargestellt. Wenn man sich für die Auswirkungen einer experimentellen Intervention auf die Fähigkeit einer anderen experimentellen Intervention zur Induktion von OCR interessiert, ist ein zusätzlicher Normalisierungsschritt, die "Delta-Delta-OCR", erforderlich. Ein solches Beispiel ist in Abbildung 9B dargestellt, wo die Fähigkeit von Palmitat (eine experimentelle Intervention), eine erhöhte OCR (durch Induktion der β-Oxidation) zu induzieren, von der Art des Mediums für die RPE-Kultur (eine zweite experimentelle Intervention) abhängt.
Minimieren Sie schließlich das Risiko einer Kontamination. Das System überwacht die OCR, indem es einen Sauerstoffsensor in das Medium über den Zellen einführt. Dadurch steigt die Ansteckungsgefahr. Unerwartete Einbrüche der OCR während des Experiments oder unerwartet niedrige OCR zu Beginn des Experiments könnten Anzeichen für eine Infektion sein. Die Platten müssen unter einem Zellkulturmikroskop aktiv auf Infektionen überwacht werden. Eine Kreuzkontamination zwischen 96-Well-Platten kann auftreten, wenn ein Sensordeckel wiederverwendet wird. Um eine solche Kreuzkontamination zu minimieren, sollte man den gesamten Deckel nach Abschluss eines Experiments 20 Minuten lang in 70%iges Ethanol zur Dekontamination tauchen, bevor der Messdeckel in einem neuen Experiment wiederverwendet wird (siehe Protokollabschnitt 1.2.15).
Abschließend wird ein Protokoll für die Echtzeit-Langzeitüberwachung des mitochondrialen Metabolismus in High-Fidelity-RPE-Kulturen durch Bewertung der OCR unter Verwendung eines neuartigen Systems namens Resipher vorgestellt. Es kann für zahlreiche Anwendungen an hochpolarisierten und differenzierten RPE-Kulturen eingesetzt werden, insbesondere zur Untersuchung physiologischer metabolischer Eigenschaften von isoliertem RPE sowie eines veränderten RPE-Metabolismus in in vitro-Modellen von Netzhauterkrankungen, einschließlich AMD und PVR.
Offenlegungen
Richard A. Bryan und Kin Lo sind Mitarbeiter von Lucid Scientific, dem Hersteller des Resipher-Systems.
Danksagungen
Wir danken Dr. Daniel Hass und Jim Hurley für die Idee, die Löslichkeit von O2 in neuen versus konditionierten Medien als Kontrolle zu testen. Wir danken Dr. Magali Saint-Geniez für die redaktionelle Mitarbeit an dem Manuskript. Wir danken Scott Szalay von Instrument and Electronic Services Core, Kellogg Eye Center, für die Nachrüstung der Hypoxiekammer mit dem Resipher USB-Kabel. Für die HFT-Forschung wurden keine Bundesmittel verwendet. Der Electronic Services Core wird durch P30 EY007003 des National Eye Institute unterstützt. Diese Arbeit wird durch ein uneingeschränktes Department-Grant von Research to Prevent Blindness (RPB) unterstützt. J.M.L.M. wird unterstützt von der James Grosfeld Initiative for Dry Age-Related Macular Degeneration, der E. Matilda Ziegler Foundation for the Blind, einem Eversight Eye-Bank Grant, einem K08EY033420 Grant des National Eye Institute und Unterstützung von Dee und Dickson Brown sowie der David and Lisa Drews Discovering Hope Foundation. D.Y.S. wird vom UNSW Scientia Programm unterstützt. L.A.K. wird unterstützt durch den Iraty Award, Monte J. Wallace, Michel Plantevin, ein R01EY027739 Stipendium des National Eye Institute und das Department of Defense Army Medical Research Acquisition Activity VR220059.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco | #25-200-056 | |
| 3,3',5-triiodo-L-thyronine sodium salt | Sigma | T5516 | |
| 32-channel Resipher lid | Lucid Scientific | NS32-101A for Falcon | |
| Antimycin A from streptomyces sp. | Sigma | A8674-25MG | Inhibitor of Complex III of the electron transport chain |
| BAM15 | Sigma | SML1760-5MG | Uncoupling agent to increase mitochondrial respiration |
| DMSO, cell culture grade | Sigma-aldrich | D4540-100ML | Vehicle for reconstituting mitochondrial drugs |
| Extracellular matrix coating substrates: Synthemax II-SC | Corning | #3535 | Extracellular matrix for hfRPE |
| Extracellular matrix coating substrates: Vitronectin | Gibco | A14700 | Extracellular matrix for iPSC-RPE |
| FCCP | Sigma | C2920-10MG | Uncoupling agent to increase mitochondrial respiration |
| Fetal Bovine Serum (Bio-Techne S11550H) | Bio-Techne | S11550H | |
| Hydrocortisone-Cyclodextrin | Sigma | H0396 | |
| Hypoxia chamber | Embrient Inc. | MIC-101 | |
| N1 Media Supplement | Sigma | N6530 | |
| Non-Essential Amino Acids Solution | Gibco | 11140050 | |
| O2 sensor | Sensit technology or Forensics Detectors | P100 or FD-90A-O2 | |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| Recombinant human TGFβ2 | Peprotech | 100-35B | Transforming growth factor beta-2 to induce epithelial-mesenchymal transition |
| Rotenone | Sigma | R8875-1G | Inhibitor of Complex I of the electron transport chain |
| System-compatible plate | Corning | #353072 | |
| Taurine | Sigma | T8691 | |
| αMEM (Alpha Modification of Eagle's Media) | Corning | 15-012-CV |
Referenzen
- Shu, D. Y., Butcher, E., Saint-Geniez, M. EMT and EndMT: emerging roles in age-related macular degeneration. Int J Mol Sci. 21 (12), 4271 (2020).
- Shu, D. Y., Lovicu, F. J. Myofibroblast transdifferentiation: the dark force in ocular wound healing and fibrosis. Prog Retin Eye Res. 60, 44-65 (2017).
- Hurley, J. B. Retina metabolism and metabolism in the pigmented epithelium: a busy intersection. Annu Rev Vis Sci. 7, 665-692 (2021).
- Wu, W., et al. Deficient RPE mitochondria energetics leads to subretinal fibrosis in age-related macular degeneration. Invest Ophthalmol Vis Sci. 64 (8), 2420 (2023).
- Hansman, D. S., et al. Metabolic reprogramming of the retinal pigment epithelium by cytokines associated with age-related macular degeneration. Biosci Rep. 44 (4), BSR20231904BSR20231904 (2024).
- Li, K. X., et al. Modulation of pyruvate kinase M2 activity as a therapy in a primary RPE cell culture model of proliferative vitreoretinopathy. Invest Ophthalmol Vis Sci. 62 (8), 2213 (2021).
- Gupta, S., et al. Progress in stem cells-based replacement therapy for retinal pigment epithelium: in vitro differentiation to in vivo delivery. Stem Cells Transl Med. 12 (8), 536-552 (2023).
- Wilson, D. F., et al. Oxygen distribution and vascular injury in the mouse eye measured by phosphorescence-lifetime imaging. Appl Opt. 44 (25), 5239-5248 (2005).
- Hass, D. T., et al. Medium depth influences O2 availability and metabolism in human RPE cultures. Invest Ophthalmol Vis Sci. 64 (14), 4 (2023).
- Shu, D. Y., Butcher, E. R., Saint-Geniez, M. Suppression of PGC-1α drives metabolic dysfunction in TGFβ2-Induced EMT of retinal pigment epithelial cells. Int J Mol Sci. 22 (9), 4701 (2021).
- Shu, D. Y., et al. Dimethyl fumarate blocks tumor necrosis factor-alpha-driven inflammation and metabolic rewiring in the retinal pigment epithelium. Front Mol Neurosci. 15, 896786 (2022).
- Ng, P. Q., Saint-Geniez, M., Kim, L. A., Shu, D. Y. Divergent metabolomic signatures of TGFβ2 and TNFα in the induction of retinal epithelial-mesenchymal transition. Metabolites. 13 (2), 213 (2023).
- Kar, D., et al. Choriocapillaris impairment is associated with delayed rod-mediated dark adaptation in age-related macular degeneration. Invest Ophthalmol Vis Sci. 64 (12), 41 (2023).
- Curcio, C. A., Kar, D., Owsley, C., Sloan, K. R., Ach, T. Age-related macular degeneration, a mathematically tractable disease. Invest Ophthalmol Vis Sci. 65 (3), 4 (2024).
- Maminishkis, A., et al. Confluent monolayers of cultured human fetal retinal pigment epithelium exhibit morphology and physiology of native tissue. Invest Ophthalmol Vis Sci. 47 (8), 3612-3624 (2006).
- Maminishkis, A., Miller, S. S. Experimental models for study of retinal pigment epithelial physiology and pathophysiology. J Vis Exp: JoVE. (45), e2032 (2010).
- Zhang, Q., et al. A platform for assessing outer segment fate in primary human fetal RPE cultures. Exp Eye Res. 178, 212-222 (2019).
- Sharma, R., Bose, D., Montford, J., Ortolan, D., Bharti, K. Triphasic developmentally guided protocol to generate retinal pigment epithelium from induced pluripotent stem cells. STAR Protoc. 3 (3), 101582 (2022).
- Marchetti, P., Fovez, Q., Germain, N., Khamari, R., Kluza, J. Mitochondrial spare respiratory capacity: Mechanisms, regulation, and significance in non-transformed and cancer cells. FASEB J. 34 (10), 13106-13124 (2020).
- Rosenfeld, P. J., Trivizki, O., Gregori, G., Wang, R. K. An update on the hemodynamic model of age-related macular degeneration. Am J Ophthalmol. 235, 291-299 (2022).
- Fitch, T. C., et al. Real-time analysis of bioenergetics in primary human retinal pigment epithelial cells using high-resolution respirometry. J Vis Exp: JoVE. (192), (2023).
- Calton, M. A., Beaulieu, M. O., Benchorin, G., Vollrath, D. Method for measuring extracellular flux from intact polarized epithelial monolayers. Mol Vis. 24, 425-433 (2018).
- Grumbine, M. K., et al. Maintaining and assessing various tissue and cell types of the eye using a novel pumpless fluidics system. J Vis Exp: JoVE. (197), (2023).
- Mu, C., Shearer, J. Protocol for measuring respiratory function of mitochondria in frozen colon tissue from rats. STAR Protoc. 4 (4), 102560 (2023).
- Rocha, D. S., Manucci, A. C., Bruni-Cardoso, A., Kowaltowski, A. J., Vilas-Boas, E. A. A practical and robust method to evaluate metabolic fluxes in primary pancreatic islets. Mol Metab. 83, 101922 (2024).
- Kanaan, M. N., et al. Cystine/glutamate antiporter xCT controls skeletal muscle glutathione redox, bioenergetics and differentiation. Redox Biol. 73, 103213 (2024).
- da Veiga Moreira, J., et al. Methylene blue metabolic therapy restrains in vivo ovarian tumor growth. Cancers. 16 (2), 355 (2024).
- McNally, L. A., Altamimi, T. R., Fulghum, K., Hill, B. G. Considerations for using isolated cell systems to understand cardiac metabolism and biology. J Mol Cell Cardiol. 153, 26-41 (2021).
- Misare, K. R., et al. The consequences of tetraploidy on Caenorhabditis elegans physiology and sensitivity to chemotherapeutics. Sci Rep. 13 (1), 18125 (2023).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten