Method Article
Purification et identification de protéines spécifiques de type cellulaire à partir de tissus complexes à l’aide d’une lignée de souris mutante d’ARNt synthétase de méthionine
Dans cet article
Résumé
Ce protocole décrit comment effectuer le marquage de protéines spécifiques au type cellulaire avec l’azidonorleucine (ANL) à l’aide d’une lignée de souris exprimant une synthétase d’ARNt L274G-méthionine mutante (MetRS*) et les étapes nécessaires pour isoler des protéines marquées spécifiques au type cellulaire. Nous décrivons deux voies d’administration possibles d’ANL chez des souris vivantes par (1) l’eau potable et (2) les injections intrapéritonéales.
Résumé
Comprendre l’homéostasie des protéines in vivo est essentiel pour savoir comment les cellules fonctionnent dans des conditions physiologiques et pathologiques. Le présent protocole décrit le marquage in vivo et la purification ultérieure de protéines nouvellement synthétisées à l’aide d’une lignée de souris modifiée pour diriger le marquage des protéines vers des populations cellulaires spécifiques. Il s’agit d’une lignée inductible par l’expression de la Cre recombinase de L274G-Methionine tRNA synthetase (MetRS*), permettant l’incorporation d’azidonorleucine (ANL) aux protéines, ce qui ne se produirait pas autrement. En utilisant la méthode décrite ici, il est possible de purifier des protéomes spécifiques au type cellulaire marqués in vivo et de détecter des changements subtils dans la teneur en protéines dus à la réduction de la complexité de l’échantillon.
Introduction
L’homéostasie protéique aberrante est causée par un déséquilibre dans la synthèse et la dégradation des protéines. Plusieurs maladies sont liées à des altérations de l’homéostasie des protéines. La caractéristique de certaines maladies est la présence d’agrégats dans différents endroits subcellulaires et zones du cerveau. L’homéostasie des protéines est non seulement importante dans la maladie, mais joue également un rôle crucial dans la fonction organique et cellulaire normale1. Par exemple, la synthèse des protéines est nécessaire pour de nombreuses formes de plasticité neuronale2,3, telle que déterminée par l’utilisation d’inhibiteurs chimiques qui bloquent la synthèse des protéines4. Cependant, il n’est pas clair dans quels types de cellules le protéome est modifié pour soutenir l’apprentissage et la mémoire, ni quelles protéines spécifiques dans chaque type cellulaire augmentent ou diminuent dans leur synthèse ou leur dégradation. Ainsi, une étude approfondie de l’homéostasie des protéines nécessite la capacité de différencier les protéomes provenant de types cellulaires spécifiques. En effet, l’identification de protéomes spécifiques au type cellulaire pour étudier les processus cellulaires se produisant dans un environnement multicellulaire a été un obstacle important en protéomique. Pour cette raison, nous avons développé une technique utilisant l’expression MetRS* combinée à des méthodes bio-orthogonales qui s’est avérée être un moyen efficace d’identifier et de purifier des protéomes spécifiques de type cellulaire, comblant cette lacune 5,6,7.
L’expression d’un mutant MetRS* (MetRS L274G) permet le chargement de l’analogue non canonique de la méthionine ANL dans l’ARNt 8,9 correspondant et son incorporation ultérieure dans les protéines. Lorsque l’expression de MetRS* est régulée par un promoteur spécifique au type cellulaire, l’acide aminé non canonique sera incorporé dans les protéines de manière sélective des cellules. Une fois que l’ANL est incorporée dans les protéines, elle peut être fonctionnalisée sélectivement par la chimie du clic et ensuite visualisée par imagerie (FUNCAT) ou par Western Immunoblot (BONCAT). Alternativement, les protéines peuvent être purifiées sélectivement et identifiées par spectrométrie de masse (MS). Grâce à cette technologie, nous avons créé une lignée de souris exprimant la protéine MetRS* sous le contrôle de la Cre recombinase. Compte tenu du nombre croissant de lignées Cre-mouse disponibles, le système MetRS* peut être utilisé dans n’importe quel domaine pour étudier n’importe quel type de cellule à partir de n’importe quel tissu pour lequel il existe une Cre-line. Le marquage des protéines avec ANL est possible in vitro ou in vivo, et ne modifie pas le comportement de la souris ou l’intégrité des protéines6. La durée de marquage peut être adaptée à la question scientifique de chaque chercheur, en marquant des protéines nouvellement synthétisées (temps de marquage plus courts) ou des protéomes entiers (temps de marquage plus longs). L’utilisation de cette technique est limitée par le nombre de cellules du type que le chercheur est prêt à étudier; Par conséquent, l’isolement des protéines à partir de types cellulaires à faible nombre ou à faible taux métabolique n’est pas possible par cette méthode. L’objectif de la méthode présentée est d’identifier des protéines/protéomes spécifiques au type cellulaire marqués in vivo. Dans ce protocole, nous décrivons comment marquer des protéomes spécifiques au type cellulaire avec ANL chez des souris vivantes et purifier les protéines marquées. Après purification, les protéines peuvent être identifiées par des protocoles de spectrométrie de massede routine 5,10. La réduction de la complexité de l’échantillon obtenue dans cette méthode par la purification sélective de protéines provenant de populations cellulaires spécifiques permet à l’expérimentateur de détecter des changements subtils dans les protéomes, par exemple, en réponse à des changements environnementaux. La purification des protéines marquées peut être réalisée en ~10 jours, sans compter l’analyse MS ou la période de marquage. Ici, nous décrivons deux méthodes d’administration d’ANL à des souris exprimant MetRS*, à savoir (1) l’ajout d’acide aminé dans l’eau potable et (2) l’introduction de LNA par injections intrapéritonéales. Quelle que soit la méthode choisie pour l’administration de l’ANL, les étapes d’isolement et de purification sont les mêmes (à partir de l’étape 2).
Protocole
Toutes les expériences sur les animaux ont été réalisées avec l’autorisation des bureaux du gouvernement local en Allemagne (RP Darmstadt; protocoles: V54-19c20/15-F122/14, V54-19c20/15-F126/1012) ou en Espagne (Comité d’expérimentation animale de l’UCM et de conseil environnemental de la Comunidad de Madrid, numéro de protocole: PROEX 005.0/21) et sont conformes aux règles de la Société Max Planck et aux réglementations espagnoles et suivent les directives de l’UE pour le bien-être animal.
1. Marquage métabolique in vivo avec ANL
- Eau potable:
- Dissoudre ANL et maltose dans l’eau potable ordinaire des souris. La concentration recommandée de maltose est de 0,7 % (poids/vol). La quantité maximale d’ANL ajoutée à l’eau potable est de 1 % (poids/vol). Une fois le mélange prêt, stérilisez-le par filtration et stockez-le à 4 °C.
- Fournir le mélange préparé à l’étape 1.1 aux souris. Changez les bouteilles fréquemment (p. ex., tous les 3 jours) pour éviter les contaminations et vérifiez-les tous les jours pour détecter toute contamination ou obstruction possible. Surveillez quotidiennement la quantité d’eau bue en la pesant pour connaître l’apport en ANL des souris.
REMARQUE : La période maximale d’étiquetage évaluée ici est de 3 semaines en utilisant une concentration de LNA de 1 % dans l’eau potable, ce qui a permis d’obtenir un marquage bien détectable dans le cerveau. Un bon étiquetage peut également être réalisé en 2 semaines. Ni des temps d’étiquetage plus courts, ni des points de temps plus longs, ni d’autres quantités d’ANL n’ont été étudiés par nous en utilisant cette méthode d’administration, ce qui ne signifie pas que ce qui précède ne fonctionnerait pas. Les taux d’incorporation des LNA peuvent varier selon le tissu ou le type de cellule étudié. Le moment de l’étiquetage peut varier en fonction de la question scientifique; Par exemple, si les protéomes devaient être marqués, le temps de marquage devrait être calculé en fonction des demi-vies moyennes des protéines dans le tissu étudié. Si l’intérêt est seulement d’identifier les protéines nouvellement synthétisées, les temps peuvent être raccourcis. Les périodes d’étiquetage et les doses de LNA doivent être testées empiriquement pour chaque condition expérimentale et type de cellule d’intérêt.
- Administration intrapéritonéale :
- Dissoudre ANL dans une solution physiologique de NaCl à 400 mM, un tampon saline phosphate (PBS) ou de l’eau.
- Assurez-vous que l’osmolarité de la solution se situe dans la plage physiologique. Ici, 10 mL/kg de poids corporel de solution ANL sont administrés aux souris.
REMARQUE: Un bon marquage dans les neurones excitateurs du cerveau est obtenu avec succès par une injection intrapéritonéale (IP) quotidienne avec 400 mM ANL pendant 1 semaine. Ni des concentrations plus faibles de LNA ni d’autres périodes de marquage par injections IP n’ont été testées dans cette étude, ce qui ne signifie pas qu’elles ne fonctionneraient pas. ANL est fourni sous forme de chlorhydrate par certaines entreprises. Dans ce cas, le pH des solutions doit être ajusté au pH adéquat (en fonction de la voie d’administration). ANL peut également être synthétisé selon la méthode publiée par Link et al.11 avec quelques modifications5. Le marquage ANL peut être effectué en utilisant d’autres concentrations, durées et voies d’administration ANL. Pour les tissus/types de cellules à faible taux métabolique, un régime à faible teneur en méthionine peut être utilisé, toujours en commençant 1 semaine avant l’administration de LNA. Lorsque 400 mM d’ANL sont utilisés, il peut précipiter lorsqu’il est stocké à 4 °C; un chauffage jusqu’à 37 °C ramène l’ANL dans la solution. Laisser atteindre la température ambiante avant l’injection. Dans la plupart des régions du monde, l’administration d’ANL à des souris est une expérimentation animale et doit être approuvée par les autorités compétentes.
2. Prélèvement tissulaire, lyse et extraction de protéines
- Dissection tissulaire : Disséquer la zone de tissu contenant le type cellulaire d’intérêt et noter le poids du morceau de tissu recueilli.
NOTE: Les échantillons peuvent être conservés à -80 °C pendant plusieurs mois (point d’arrêt). Dans le protocole actuel, le cortex et l’hippocampe des souris ont été disséqués. - Lyse tissulaire: Homogénéiser le tissu à température ambiante en ajoutant un volume 12-15 fois le poids humide du tissu du tampon de lyse (PBS pH 7,4, 1% poids / vol SDS, 1% (vol/vol) Triton X-100, benzonase (1:1000 vol/vol), un inhibiteur de protéase (IP) (EDTA libre 1:4000)). Par exemple, pour un morceau de tissu de 20 mg, ajoutez 240-300 μL de tampon de lyse. Triturer le tissu jusqu’à ce qu’il soit homogénéisé.
NOTE: Les échantillons peuvent être conservés à -80 °C pendant plusieurs mois (point d’arrêt). Pour l’homogénéisation directe dans des tubes de 1,5 mL, l’utilisation d’un homogénéisateur portatif à piles est recommandée, en particulier lorsque de petits morceaux de tissu sont traités. Pour les pièces plus grandes, un homogénéisateur Dounce peut être utilisé. À chaque tampon où des inhibiteurs de la protéase (IP) sont inclus, ils doivent être ajoutés immédiatement avant l’utilisation et non plus tôt. - Dénaturation des protéines : Chauffer l’homogénat à 75 °C pendant 15 min et centrifuger à 17 000 x g pendant 15 min à 10 °C. Transférer le surnageant dans un nouveau tube.
- Mesure de la teneur en protéines: Mesurer la concentration en protéines de chaque échantillon et ajuster tous les échantillons pour les comparer à la même concentration en protéines; Ajustez la concentration en ajoutant un tampon de lyse. Une plage de concentration optimale se situe entre 2 et 4 μg/μL.
NOTE: Les échantillons peuvent être conservés à -80 °C pendant plusieurs mois (point d’arrêt). - Alkylation
- Diluer les échantillons homogénéisés de 2 à 3 fois dans du PBS (pH 7,8) + PI (EDTA libre 1:4000).
- Ajouter l’iodoacétamide (IAA) à une concentration finale de 20 mM (solution mère 500 mM).
- Laisser les échantillons pendant 1-2 h à 20 °C dans l’obscurité. Effectuez les étapes 2.5.2 à 2.5.3 deux fois.
REMARQUE: Pour certains tissus, si le clic non spécifique dans le contrôle négatif reste élevé, il peut être nécessaire d’effectuer une étape de réduction avant l’alkylation12. Préparez le stock IAA fraîchement juste avant de l’ajouter aux échantillons. Les échantillons peuvent être conservés à -80 °C pendant plusieurs mois (point d’arrêt).
- Échange de tampon : Équilibrer les colonnes d’échange de tampons à l’aide du tampon d’échange de chimie de clic (PBS pH 7,8, 0,04 % (poids/vol) SDS, 0,08 % (vol/vol) Triton X-100 et PI (1:4000)). Suivez les instructions du fabricant. Échangez tous les échantillons alkylés.
NOTE: Les échantillons élués peuvent être conservés à -80 °C pendant plusieurs mois (point d’arrêt). Dans cette étape, l’IAA est éliminé et le tampon est échangé par le tampon de chimie de clic. Les protéines peuvent être mesurées à nouveau après l’étape d’échange tampon pour s’assurer que les protéines n’ont pas été perdues pendant le processus d’échange tampon. Selon le type de colonnes utilisées pour l’échange de tampons, les protéines peuvent être massivement perdues. Utilisez les colonnes recommandées pour éviter la perte de protéines. Pour le stockage des échantillons, la préparation d’aliquotes est recommandée. - Réaction de clic pour évaluer l’incorporation de LNA dans l’expérience
- Prélever 40 μL des échantillons élués à l’étape 2.6 et ajouter du PBS (pH 7,8) jusqu’à un volume final de 120 μL.
- Pour configurer la réaction de chimie de clic, ajoutez les réactifs suivants dans l’ordre et le vortex donnés pendant 20 s après chaque ajout: 1,5 μL de ligand triazole (stock 40 mM), 1,5 μL de biotine alcyne (stock 5 mM) et 1 μL de bromure de Cu(I) (stock 10 mg / mL, dissous dans le DMSO par pipetage soigneux de haut en bas, ne pas vortex). Ajouter les réactifs le plus rapidement possible.
- Incuber les échantillons pendant une nuit dans l’obscurité à 4 °C en rotation continue.
- Centrifuger les échantillons à 4 °C pendant 5 min à 17 000 x g. Une pastille légèrement turquoise sera visible. Transférer le surnageant dans un nouveau tube.
NOTA : Préparer le bromure de Cu(I) immédiatement avant utilisation pour éviter l’oxydation du cuivre. Il est essentiel de maintenir constant le rapport des volumes entre l’échantillon lysé et le PBS entre les échantillons pour assurer la même concentration finale de détergent dans chaque tube. Pour accélérer l’assemblage de la réaction chimique de clic, un vortexer de table avec des racks pour tubes est recommandé. Conservez les échantillons cliqués à -80 °C pendant plusieurs mois, ou à -20 °C pendant quelques semaines jusqu’à ce qu’ils soient traités (point d’arrêt).
- Analyse Western blot pour évaluer l’incorporation de la LNA dans l’expérience
- Exécutez une SDS-PAGE avec 20-40 μL du surnageant à partir de la réaction chimique de clic (étape 2.7). Utilisez des gels d’acrylamide à 12%, utilisez des FDS à 1%, 250 mM de Tris-HCl, 2 mM de glycine. Courez jusqu’à ce que l’avant soit sorti du gel.
- Transférer les protéines sur une membrane de nitrocellulose ou de PVDF13.
- Incuber la membrane pendant une nuit à 4 °C avec l’anticorps GFP (1:500, la protéine GFP est co-traduite avec MetRS*5) et l’anticorps biotine (1:1000) pour la détection des alcynes cliqués, qui est conjugué à la biotine.
- Lavez l’anticorps primaire 2 fois pendant 10 min et ajoutez l’anticorps secondaire pendant 1,5 h.
- Laver l’anticorps secondaire 2 fois pendant 10 minutes et scanner (si vous utilisez des anticorps conjugués au fluorophore) ou exposer à des films (si vous utilisez des anticorps conjugués à la peroxydase de raifort).
- Quantifier le signal de biotine à l’aide d’ImageJ ou d’un logiciel similaire, évaluer l’étiquetage général de chaque échantillon de l’expérience et détecter les valeurs aberrantes possibles. Évaluer le rapport signal sur bruit (étiquetage de l’échantillon ANL à l’arrière-plan des témoins négatifs) de l’expérience et détecter les animaux possibles qui n’ont pas adopté / incorporé ANL.
REMARQUE : Pour les échantillons à forte incorporation de LNA, la purification des protéines est théoriquement possible à l’aide d’alcynes non clivables comme celui utilisé dans cette section pour l’évaluation de l’incorporation de LNA. En utilisant des échantillons de cerveau, le bruit de fond obtenu chez les témoins négatifs par la SEP après purification des protéines à l’aide d’alcynes non clivables est trop élevé14. Par conséquent, seuls les alcynes non clivables et plus faciles à manipuler ont été utilisés pour un premier échantillon général et une évaluation expérimentale. Quel que soit le type d’alcyne destiné à la purification des protéines, il est conseillé d’effectuer les étapes d’optimisation du dosage des alcynes décrites dans la section suivante (étape 2.9).
- Réaction de clic pour l’optimisation du dosage d’alcyne clivable
- Préparer quatre tubes avec 40 μL d’un échantillon représentatif (obtenu à l’étape 2.6 et évalué à l’étape 2.8) et ajouter du PBS (pH 7,8) jusqu’à un volume final de 120 μL.
- Configurez la réaction chimique de clic comme expliqué à l’étape 2.7, mais cette fois, ajoutez quatre quantités différentes de DST-alcyne. Pour ledit alcyne, il est recommandé de tester 5 μM, 15 μM, 30 μM et 60 μM.
- Répétez l’étape 2.8 pour décider quel est le dosage d’alcyne le plus approprié pour la question expérimentale spécifique à l’étude, en fonction de l’efficacité d’étiquetage la plus élevée avec le signal de fond le plus faible.
REMARQUE: Avant de soumettre les échantillons restants à une réaction de clic, la quantité optimale d’alcyne doit être déterminée. Il existe plusieurs options d’alcynes disponibles dans le commerce qui peuvent être utilisées. Ils diffèrent par le clivage (par exemple, les agents légers ou réducteurs). La chimie du clic sans cuivre utilisant des réactifs favorisés par la déformation (par exemple, les réactifs DBCO) est également possible. De minuscules changements dans la quantité d’alcynes ou de réactifs DBCO augmentent souvent le signal dans le contrôle négatif (arrière-plan) de manière significative. Ici, la purification protéique à l’aide d’un alcyne avec un pont disulfure clivable par des agents réducteurs (disulfure biotine alcyne ou DST-alcyne15) est décrite. Lors de l’utilisation du DST-alcyne, pour exécuter le SDS-PAGE (étape 8.1), évitez de charger les tampons avec des agents réducteurs puissants tels que le DTT ou le β-Mercaptoéthanol pour éviter le clivage des alcynes. Dans cette étude, le chauffage à 72 °C, pendant 5 min, en utilisant 5 mM de N-éthylmaléimide (NEM) dans le tampon de chargement n’affecte pas la stabilité du DST-alcyne. L’étape 2.9.2 peut être répétée, en testant des doses plus fines dans la gamme des meilleures observées.
- Réaction de clic préparative pour la purification des protéines
- Cliquez sur tous les échantillons obtenus à l’étape 2.6 avec la dose optimale d’alcyne déterminée à l’étape 2.9 et analysez une fraction par SDS-PAGE et Western blot (étape 2.8).
REMARQUE: Assurez-vous que les pipettes sont correctement étalonnées pour assurer la précision de la mise à l’échelle de l’échantillon. Les échantillons cliqués peuvent être conservés à -80 °C pendant plusieurs mois (point d’arrêt).
- Cliquez sur tous les échantillons obtenus à l’étape 2.6 avec la dose optimale d’alcyne déterminée à l’étape 2.9 et analysez une fraction par SDS-PAGE et Western blot (étape 2.8).
3. Purification des protéines
- Échange de tampon
- Soumettre tous les échantillons cliqués à une procédure d’échange de tampon comme décrit à l’étape 2.6, mais utiliser le tampon de liaison à la neutravidine comme tampon d’équilibre (PBS pH 7,4, FDS 0,15% (p/vol), 1% (vol/vol) Triton X-100 et PI (1:2000)).
NOTE: Les échantillons élués peuvent être conservés à -80 °C pendant plusieurs mois (point d’arrêt). Dans cette étape, l’alcyne libre non cliqué est éliminé et le tampon est échangé par le tampon de liaison à la neutravidine.
- Soumettre tous les échantillons cliqués à une procédure d’échange de tampon comme décrit à l’étape 2.6, mais utiliser le tampon de liaison à la neutravidine comme tampon d’équilibre (PBS pH 7,4, FDS 0,15% (p/vol), 1% (vol/vol) Triton X-100 et PI (1:2000)).
- Liaison aux perles de Neutravidin
- Laver les billes de haute capacité Neutravidin avec le tampon de liaison Neutravidin (voir étape 3.1); Mélanger les billes avec le tampon et centrifuger le mélange pendant 5 min à 3000 x g, et jeter le surnageant. Répétez trois fois.
- Préparez une suspension 1:1 de billes de Neutravidin en ajoutant le même volume de billes sèches et de tampon de liaison Neutravidin.
- Réserver 20 à 40 μL de chaque échantillon comme lysat pré-purifié et le conserver à -20 °C.
- Mesurer la concentration en protéines (voir étape 2.4) et mélanger 100 μg de protéines avec 4 μL de boue de billes obtenue à l’étape 3.2.2.
- Incuber le mélange pendant une nuit à 4 °C avec une rotation continue, permettant aux protéines marquées de se lier aux billes.
REMARQUE: En ce qui concerne la réaction chimique de clic, la réaction de purification d’affinité de base décrite à l’étape 3.2.4 peut être mise à l’échelle. Par exemple, pour la purification des protéines des neurones excitateurs de l’hippocampe, 1 mg de lysat de protéines est mélangé avec 40 μL de billes de Neutravidin (suspension 1: 1). La quantité de billes de Neutravidin peut être augmentée ou réduite en fonction de la quantité de protéines marquées par mg de protéines totales, qui dépendra du type de cellule choisi et de la période de marquage et doit être déterminée empiriquement.
- Lavage des perles de neutravidine et élution des protéines
- Recueillir les surnageants par centrifugation (voir étape 3.2.1). Mettre de côté une partie aliquote de 20 à 40 μl de chaque surnageant pour une analyse ultérieure et congeler le reste à -80 °C.
- Lavez les billes avec le tampon de lavage de Neutravidin réfrigéré 1 (PBS pH 7,4, 0,2% (p/vol) SDS, 1% (vol/vol) Triton X-100 et PI (1:2000)) en ajoutant le tampon, en sédimentant les billes et en jetant le surnageant. Répétez cette étape trois fois.
- Ensuite, ajoutez le même tampon et incuber les billes pendant 10 min en rotation continue à 4 °C avant de jeter le surnageant. Répétez cette étape trois fois.
- Lavez les billes comme expliqué aux étapes 3.3.2 à 3.3.3, mais en utilisant le tampon de lavage Neutravidin 2 (1x PBS, pH 7,4 et PI 1:2000).
- Laver les billes comme expliqué à l’étape 3.3.2-3.3.3 mais en utilisant le tampon de lavage Neutravidin 3 (bicarbonate d’ammonium 50 mM et PI 1:2000).
- Éluer les protéines cliquetées en incubant les billes pendant 30 min à 20 °C avec un volume de tampon d’élution de la Neutravidine (5 % (vol/vol) β-mercaptoéthanol et 0,03 % (poids/vol) SDS) correspondant à un volume des billes sèches utilisées.
- Maintenir les billes en suspension pendant l’élution par agitation continue (1000 tr/min) dans un agitateur thermobloc. Éluer deux fois et combiner les deux éluats.
NOTA: La centrifugation mise en place pour la séparation de la fraction solide de la boue (billes) et de la phase aqueuse (surnageant), comme expliqué à l’étape 3.2.1, s’applique aux étapes 3.3.1 à 3.3.7. La quantité de FDS peut être augmentée si l’élution des protéines n’est pas complète. Cependant, avec des quantités supérieures à 0,08% à 0,1% de SDS, les protéines non spécifiquement liées aux billes commencent à être éluées. β-mercaptoéthanol est volatil et toxique; L’utilisation d’une hotte pour les étapes 3.3.6-3.3.7 est conseillée. Les échantillons prélevés aux étapes 3.2.3 et 3.3.1 doivent être analysés par SDS-PAGE et évalués par transfert Western (voir étape 2.8) pour évaluer l’efficacité de la purification par affinité (étape 3.3).
- Évaluation des protéines éluées
- Charger 1/3 des échantillons élués sur une FDS-PAGE (voir étape 2.8.1).
- Colorez le gel pour visualiser les protéines à l’aide d’une méthode à haute sensibilité, telle que la coloration à l’argent ou une méthode fluorescente.
- Si les échantillons ont la qualité attendue, ce qui signifie qu’il y a 3 à 4 fois plus de protéines dans les échantillons marqués ANL par rapport au témoin négatif, les protéines éluées peuvent être identifiées par des méthodes de spectrométrie de masse de routine comme celle expliquée dans la référence5.
Résultats
Après le protocole décrit (résumé à la figure 1), l’ANL a été administrée à des souris soit par injections intrapéritonéales quotidiennes (400 mM ANL 10 mL/kg, Nex-Cre::MetRS*) pendant 7 jours, soit par eau potable (0,7 % de Maltose, 1 % ANL, CamkII-Cre::MetRS*) pendant 21 jours. Après l’étiquetage, les zones cérébrales correspondantes ont été disséquées, lysées, alkylées et cliquées. Les réactions aux clics ont été analysées par SDS-PAGE et Western Immunoblot. Des images représentatives des expériences sont présentées à la figure 2 pour l’administration de LNA par injection IP et à la figure 3 pour l’administration de LNA par eau potable. Notez que le but des figures est de montrer que les deux protocoles d’étiquetage fonctionnent, pas de les comparer. La comparaison n’est pas possible car différentes populations neuronales sont étiquetées dans les deux expériences montrées (neurones excitateurs de l’hippocampe et du cortex, et neurones de Purkinje du cervelet).
Une autre expérience fournit un exemple pour la détermination de la concentration optimale d’alcyne clivable au DST (Figure 4). Dans cet exemple, le changement de pli entre l’échantillon marqué et le témoin est le plus élevé à une concentration d’alcyne de 14 μM. Cette concentration d’alcyne est ensuite appliquée à tous les échantillons. Après vérification du marquage de chaque échantillon de l’expérience avec la quantité d’alcyne choisie, tous les échantillons ont été soumis à la purification de protéines marquées ANL/cliquées sur la biotine par liaison par affinité à l’aide de billes de Neutravidin (Figure 5). Dans cette étape, les protéines sont liées aux billes, lavées, puis éluées en réduisant le pont disulfure présent dans le DST-alcyne. Après cette dernière étape, la biotine et une partie de l’alcyne restent liées aux billes. Les protéines ANL (liées au reste de l’alcyne) sont récupérées dans le tampon d’élution. Pour évaluer l’efficacité de l’étape d’élution, un tiers du volume d’éluat est chargé sur un gel SDS-PAGE et visualisé à l’aide d’une méthode sensible de coloration des protéines totales. Une différence d’intensité d’au moins 3 fois la coloration totale des protéines entre les témoins négatifs et les échantillons marqués ANL doit être observée pour obtenir des résultats interprétables avec la spectrométrie de masse. Toutes les étapes décrites dans le présent protocole seront suivies de la préparation, de l’acquisition et de l’analyse des échantillons de SM5. Bien qu’il n’y ait pas d’exigences particulières pour la SEP (chaque laboratoire peut utiliser son protocole MSde routine 5,10), il faut garder à l’esprit que la quantité de protéines purifiées sera généralement faible (de l’ordre du nanogramme). La figure 6 fournit un exemple de résultats de SEP avec un enrichissement clair dans l’échantillon marqué ANL par rapport au témoin (figure 6A). Cette différence était déjà visible dans la coloration totale des protéines illustrée à la figure 5. Outre les changements dans l’intensité peptidique, il existe également des protéines uniques trouvées dans les deux échantillons (Figure 6B).
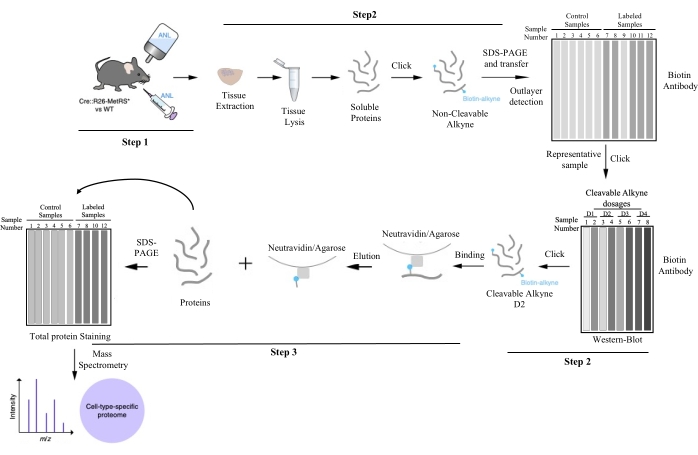
Figure 1 : Pipeline de travail pour la purification de protéines spécifiques au type cellulaire par BONCAT. Après le marquage des protéines avec ANL, le tissu d’intérêt est disséqué et lysé, marqué avec de la biotine par chimie de clic, et la quantité d’étiquetage pour chaque échantillon est évaluée par BONCAT. Les valeurs aberrantes (qui n’ont pas incorporé les LNA) et les échantillons représentatifs peuvent être différenciés à cette étape. L’un des échantillons représentatifs est cliqué à nouveau pour trouver le dosage optimisé d’alcyne clivable pour la purification ultérieure des protéines. Le dosage d’alcyne permettant d’obtenir le meilleur rapport signal/bruit est appliqué à chaque répétition biologique. Les protéines spécifiques au type cellulaire sont obtenues par purification par affinité. Les échantillons purifiés sont étudiés par spectrométrie de masse et les protéines sont identifiées. Cette figure a été modifiée à partir d’Alvarez-Castelao et al. (2017)6. Veuillez cliquer ici pour voir une version agrandie de cette figure.
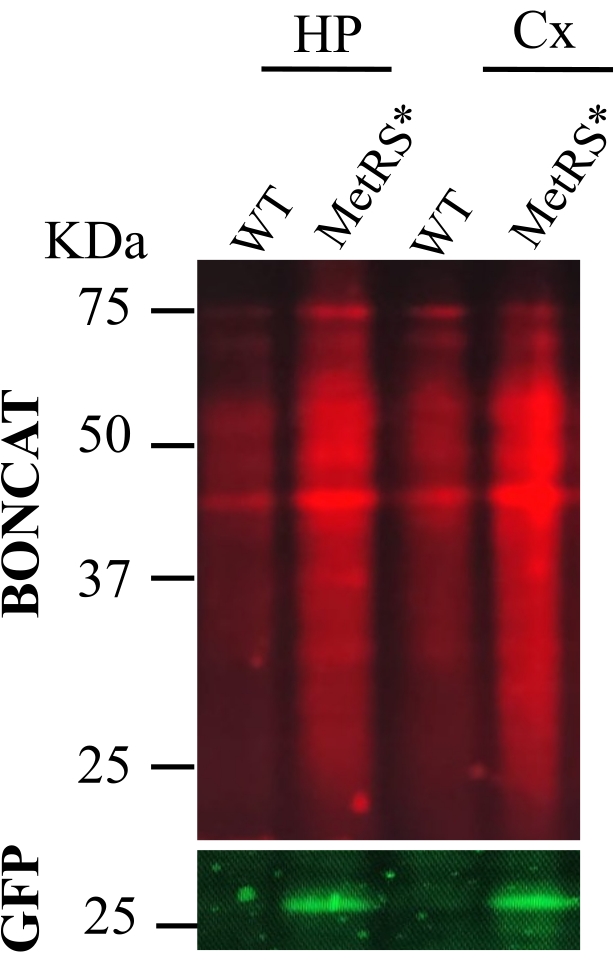
Figure 2 : Administration de LNA par injections intrapéritonéales (IP). BONCAT a été réalisé pour évaluer le marquage des protéines dans l’hippocampe (HP) et le cortex (CX) après marquage métabolique des protéines avec ANL par injections IP quotidiennes pendant 7 jours. Des échantillons de souris de type sauvage en tant que contrôle négatif (poids) ont été cliqués parallèlement aux échantillons étiquetés (MetRS*). Les protéines marquées proviennent des neurones excitateurs utilisant une lignée Nex-Cre::MetRS*16. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Administration de LNA par l’eau potable. BONCAT a été réalisé pour évaluer le marquage des protéines dans les neurones de Purkinje du cervelet après administration d’ANL à des souris GAD-Cre::MetRS* via de l’eau potable pendant 21 jours. La figure montre un contrôle négatif (wt) et un échantillon étiqueté (MetRS*) lysés et cliqués en parallèle. Cette figure a été modifiée à partir d’Alvarez-Castelao et al. (2017)6. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Titrage posologique du DST-alcyne. Une répétition biologique par expérience est choisie comme échantillon représentatif pour titrer la concentration optimale d’alcyne. Trois concentrations (14, 28 et 56 μM) de l’alcyne ont été testées ici dans la réaction de clic pour BONCAT. Le rapport d’étiquetage entre l’échantillon marqué ANL (MetRS*) et le témoin négatif (wt) détermine le rapport signal sur bruit (illustré dans le graphique). 14 μM est la meilleure concentration d’alcyne obtenue dans cette expérience. Le tissu cortex de la lignée de souris Nex::MetRS* marquée ANL a été utilisé pour cette expérience. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Protéines purifiées. Les protéines marquées des neurones de Purkinje ont été purifiées et colorées à l’aide de SYPRO Ruby. Cette figure a été modifiée à partir d’Alvarez-Castelao et al. (2017)6. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 6 : Identification et quantification des protéines. Le protéome cellulaire de Purkinje a été obtenu par spectrométrie de masse en utilisant le cervelet d’une lignée de souris GAD-Cre::MetRS* comme matériau de départ. (A) montre une abondance accrue (intensité peptidique) des protéines identifiées dans les échantillons marqués ANL par rapport aux souris de type sauvage (wt). (B) Le protéome de Purkinje a été obtenu en regroupant des protéines enrichies dans les échantillons marqués ANL dans A (définies par des intensités >3 fois chez les souris MetRS* par rapport à la wt) et des protéines uniques trouvées chez les souris exprimant MetRS*. Cette figure a été modifiée à partir d’Alvarez-Castelao et al. (2017)6. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Les aspects critiques du protocole sont les suivants : l’inclusion de témoins négatifs, ayant suffisamment de réplicats biologiques, la voie d’administration ANL, la quantité et la durée, l’alkylation des échantillons, la concentration d’alcyne et l’élimination du β-mercaptoéthanol lors de l’utilisation de DST-alcyne.
Il est essentiel d’inclure des échantillons témoins négatifs provenant d’animaux marqués ANL sans pilote Cre et donc sans expression MetRS*. Ces échantillons doivent être soumis à toutes les étapes décrites dans le protocole parallèlement aux échantillons de souris MetRS* induites par Cre-induced MetRS* marquées ANL. Les échantillons non cliqués ne sont pas des contrôles valides, car les résultats obtenus en utilisant uniquement ce contrôle peuvent être dus à un clic non spécifique. Nous n’avons pas observé d’incorporation de LNA dans les cellules n’exprimant pas le gène MetRS*, ni d’expression de MetRS* dans les cellules sans Cre ; ainsi, les animaux en humidité étiquetés avec ANL peuvent être utilisés comme témoin négatif.
Étant donné que le protocole décrit ici comporte de nombreuses étapes au cours desquelles des échantillons peuvent être perdus, il est recommandé de calculer les répétitions biologiques en tenant compte des défaillances techniques. Pour les échantillons de cerveau, il y a environ 30% de perte de réplication.
Nous décrivons ici deux voies et durées d’administration de l’ANL. Cela n’exclut pas que d’autres voies d’administration (p. ex., l’ajout d’ANL à l’aliment) fonctionnent aussi bien. En ce qui concerne la quantité de LNA administrée, les expériences présentées ici ont été réalisées en utilisant des quantités relativement élevées de LNA. L’étiquetage avec des quantités inférieures est également possible en fonction de la configuration expérimentale. La période la plus courte de l’étiquetage ANL rapportée dans ce protocole est de 1 semaine et la plus longue de 21 jours. Des périodes d’étiquetage plus courtes ou plus longues pourraient être appliquées, mais nous ne l’avons pas déterminée. Les chercheurs devraient déterminer la voie d’administration de la LNA, la posologie de la LNA et la période de marquage de la LNA adéquates pour la question expérimentale spécifique à l’étude en tenant compte des propriétés métaboliques du tissu, du type de cellule d’intérêt et de la question expérimentale à l’étude.
L’alkylation des échantillons, également connue sous le nom de capsulage, est une étape importante pour éviter le clic d’arrière-plan17. Lorsque l’alkylation est effectuée correctement, le clic non spécifique surveillé par les échantillons témoins est réduit, et la différence entre les échantillons témoins négatifs et les échantillons marqués ANL est plus grande. L’élimination de l’AIA libre après alkylation est également une étape clé. La présence de petites quantités d’IAA lors de la réaction de clic limitera son efficacité. Parfois, l’étape de dessalage qui élimine également l’AIA (étape 2.6), doit être répétée pour assurer l’élimination correcte de l’AAI.
Il existe plusieurs réactifs biotine-alcynes et -DBCO disponibles dans le commerce auprès de diverses sociétés. Les principales différences entre eux concernent la longueur de la chaîne de liaison en polyéthylèneglycol (PEG) et l’absence, la présence et le type de groupes clivables. Quel que soit le type utilisé, des quantités excessives d’alcyne conduisent à un clic non spécifique vers des protéines ne contenant que des acides aminés naturels. Cela peut être facilement évité par un titrage précis et minutieux de la quantité d’alcyne. Comme décrit, la meilleure façon d’y parvenir est d’utiliser les échantillons lysés et alkylés réels à utiliser dans les étapes ultérieures de purification des protéines (étape 2.6) et l’analyse par spectrométrie de masse.
Lorsque les alcynes DST sont utilisés dans la réaction de clic, le β-mercaptoéthanol réduit le pont disulfure et dissocie les protéines marquées des billes à l’étape d’élution (étape 3.3). β-mercaptoéthanol doit être retiré des échantillons pour permettre la digestion enzymatique nécessaire à la SEP. Il y a plusieurs façons de le faire (par exemple, lyophilisation18, excision d’un gel19 ou nettoyage à l’aide de colonnes S-Trap20), et le choix doit être fait par le laboratoire MS qui traite les échantillons.
Les modifications et le dépannage de la méthode doivent être dirigés pour optimiser les étapes critiques mentionnées dans le protocole. Par exemple, s’il y a trop de variabilité, ajoutez plus de réplications biologiques; si le marquage est trop faible, augmenter la concentration de LNA, utiliser des durées de marquage plus longues ou tester d’autres voies d’administration de LNA qui pourraient être plus efficaces pour le tissu étudié. Pour l’alkylation des protéines, une étape de réduction préalable à l’ajout d’IAA pourrait être mise en œuvre.
La principale limitation de la méthode est la purification des protéomes provenant de petites populations cellulaires, même si une petite zone tissulaire est disséquée. Les protéomes de populations cellulaires plus nombreuses qui sont, cependant, répartis sur de plus grandes zones tissulaires sont également difficiles d’accès. Néanmoins, la technique de marquage in vivo décrite ici peut toujours être appliquée pour évaluer les types de cellules référencés par imagerie (par exemple, avec FUNCAT ou FUNCAT-PLA21).
Cette méthode est actuellement la seule méthode disponible pour l’étude in vivo de protéomes spécifiques au type cellulaire basé sur des protéines pleine longueur et pour la visualisation in situ de protéines complètes spécifiques au type cellulaire. D’autres acides aminés non canoniques tels que l’azidohomoalanine (AHA) peuvent également être utilisés pour le marquage des protéines in vivo et la purification des protéomes, mais manquent de spécificité cellulaire22,23. D’autres approches telles que la puromycine conviennent pour fournir une image fixe de la synthèse des protéines24,25. Néanmoins, il est possible d’utiliser des acides aminés non canoniques pendant de plus longues périodes de marquage, reflétant également la dégradation des protéines et montrant un protéome cellulaire plus précis. Les méthodes basées sur BioID sont utilisées pour identifier les protéines dans des régions subcellulaires spécifiques, quel que soit leur moment / lieu de synthèse26.
Dans la lignée de souris que nous avons établie (disponible auprès de Jackson Lab, stock no. 028071), la méthionine synthétase mutante (MetRS*) qui permet l’incorporation de l’ANL dans les cellules est exprimée de manière Cre dépendante. Avec cette ligne de souris comme outil, il est possible d’exprimer exclusivement le MetRS* dans les types de cellules pour lesquels des lignes Cre-driver sont disponibles. Cet arrangement confère à la méthode une polyvalence considérable, ce qui la rend utile dans presque tous les domaines de la recherche biomédicale.
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt financier concurrent.
Remerciements
B.A-C est financé par le ministère espagnol de la Science et de l’Innovation (Ramón y Cajal-RYC2018-024435-I), par la Communauté autonome de Madrid (Atracción de Talento-2019T1/BMD-14057) et par les subventions MICINN (PID2020-113270RA-I00). R. A-P est financé par la Communauté autonome de Madrid (Atracción de Talento-2019T1/BMD-14057). E.M.S. est financé par la Société Max Planck, une bourse de chercheur avancé du Conseil européen de la recherche (subvention 743216), DFG CRC 1080: Mécanismes moléculaires et cellulaires de l’homéostasie neurale, et DFG CRC 902: Principes moléculaires de régulation basée sur l’ARN. Nous remercions D.C Dieterich et P. Landgraf pour leurs conseils techniques et la synthèse du DST-Alcyne. Nous remercions E. Northrup, S. Zeissler, S. Gil Mast et l’animalerie du MPI for Brain Research pour leur excellent soutien. Nous remercions Sandra Goebbels d’avoir partagé la gamme de souris Nex-Cre. Nous remercions Antonio G. Carroggio pour son aide avec l’édition anglaise. B.A-C. conçu, mené et analysé des expériences. B.N-A, D.O.C, R.A-P, C. E. et S. t. D. a mené et analysé des expériences. B.A-C et E.M.S. ont conçu des expériences et supervisé le projet, B.A-C a écrit l’article. Tous les auteurs ont édité l’article.
matériels
| Name | Company | Catalog Number | Comments |
| 12% Acrylamide gels | GenScript | SurePAGE, Bis-Tris, 10 x 8, 12% | |
| β-Mercaptoethanol | Sigma | M6250 | Toxic; use a lab coat, gloves and a fume hood. |
| Ammonium bicarbonate | Sigma | 9830 | Toxic; use a lab coat, gloves and a fume hood |
| ANL | Synthesized as described previously for AHA (see references 5 and 11) | ||
| ANL-HCl | IrishBotech | HAA1625.0500 | |
| Benzonase | Sigma | E1014 | |
| Biotin alkyne | Thermo, | B10185 | |
| Chicken antibody anti-GFP | Aves | 1020 | |
| Complete EDTA-free protease inhibitor | Roche | 4693132001 | Toxic; use a lab coat and gloves. |
| Copper (I) bromide | Sigma | 254185 | 99.999% (wt/wt) |
| Disulfide tag (DST)-alkyne | Synthesized as reported in reference number 15, in which it is referred to as probe 20 | ||
| DMSO | Sigma | 276855 | |
| Filters | Merk | SCGP00525 | |
| Iodo acetamide (IAA) | Sigma | I1149 | |
| IR anti chicken 800 | LI-COR | IRDye 800CW | Donkey anti-Chicken Secondary Antibody |
| IR anti rabit 680 | LI-COR | IRDye 680RD | Goat anti-Rabbit IgG Secondary Antibody |
| Maltose | Sigma | M9171 | |
| Manual Mixer | BioSpec Products | 1083 | |
| NaCl | Sigma | S9888 | |
| N-ethylmaleimide | Sigma | 4259 | Toxic; use a lab coat, gloves and a fume hood. |
| NeutrAvidin beads | Pierce | 29200 | |
| Nitrocellulose membrane | Bio-rad | 1620112 | |
| PBS 1X | Thermo | J62036.K2 | |
| PBS 1X pH 7.8 | Preparation described in reference number 5 | ||
| PD SpinTrap G-25 columns | GE Healthcare | Buffer exchange | |
| Pierce BCA Protein Assay Kit | Thermo, | 23225 | Reagents in the Pierce BCA Protein Assay Kit are toxic to aquatic life. |
| Polyclonal rabbit anti-biotin antibody | Cell Signaling | 5597 | |
| PVDF membrane | Millipore | IPVH00010 | |
| SDS 10% | Sigma | 71736 | |
| SDS-PAGE Running buffer MOPS | GenScript | M00138 | |
| SYPRO Ruby stain | Sigma | S4942 | |
| Table automatic Vortexer | Eppendorf | Mixmate | |
| Triazole ligand | Sigma | 678937 | |
| Triton X-100 | Sigma | T9284 | |
| Water | Sigma | W4502 | Molecular biology grade |
Références
- Alvarez-Castelao, B., Schuman, E. M. The regulation of synaptic protein turnover. Journal of Biological Chemistry. 290 (48), 28623-28630 (2015).
- Sutton, M. A., Schuman, E. M. Dendritic protein synthesis, synaptic plasticity, and memory. Cell. 127 (1), 49-58 (2006).
- Dorrbaum, A. R., Alvarez-Castelao, B., Nassim-Assir, B., Langer, J. D., Schuman, E. M. Proteome dynamics during homeostatic scaling in cultured neurons. Elife. 9, 52939(2020).
- Flexner, J. B., Flexner, L. B., Stellar, E. Memory in mice as affected by intracerebral puromycin. Science. 141 (3575), 57-59 (1963).
- Alvarez-Castelao, B., Schanzenbacher, C. T., Langer, J. D., Schuman, E. M. Cell-type-specific metabolic labeling, detection and identification of nascent proteomes in vivo. Nature Protocols. 14 (2), 556-575 (2019).
- Alvarez-Castelao, B., et al. Cell-type-specific metabolic labeling of nascent proteomes in vivo. Nature Biotechnology. 35 (12), 1196-1201 (2017).
- Ngo, J. T., et al. Cell-selective metabolic labeling of proteins. Nature Chemical Biology. 5 (10), 715-717 (2009).
- Mahdavi, A., et al. Engineered aminoacyl-tRNA synthetase for cell-selective analysis of mammalian protein synthesis. Journal of the American Chemical Society. 138 (13), 4278-4281 (2016).
- Yuet, K. P., et al. Cell-specific proteomic analysis in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 112 (9), 2705-2710 (2015).
- Patel, V. J., et al. A comparison of labeling and label-free mass spectrometry-based proteomics approaches. Journal of Proteome Research. 8 (7), 3752-3759 (2009).
- Link, A. J., Vink, M. K., Tirrell, D. A. Preparation of the functionalizable methionine surrogate azidohomoalanine via copper-catalyzed diazo transfer. Nature Protocols. 2 (8), 1879-1883 (2007).
- Landgraf, P., Antileo, E. R., Schuman, E. M., Dieterich, D. C. BONCAT: Metabolic labeling, click chemistry, and affinity purification of newly synthesized proteomes. Methods in Molecular Biology. 1266, Clifton, N.J. 199-215 (2015).
- Kielkopf, C. L., Bauer, W., Urbatsch, I. L. Analysis of proteins by immunoblotting. Cold Spring Harbor Protocols. 2021 (12), (2021).
- Mellacheruvu, D., et al. The CRAPome: a contaminant repository for affinity purification-mass spectrometry data. Nature Methods. 10 (8), 730-736 (2013).
- Szychowski, J., et al. Cleavable biotin probes for labeling of biomolecules via azide-alkyne cycloaddition. Journal of the American Chemical Society. 132 (51), 18351-18360 (2010).
- Goebbels, S., et al. Genetic targeting of principal neurons in neocortex and hippocampus of NEX-Cre mice. Genesis. 44 (12), 611-621 (2006).
- van Geel, R., Pruijn, G. J., van Delft, F. L., Boelens, W. C. Preventing thiol-yne addition improves the specificity of strain-promoted azide-alkyne cycloaddition. Bioconjugate Chemistry. 23 (3), 392-398 (2012).
- O'Fagain, C. Lyophilization of proteins. Methods in Molecular Biology. 244, Clifton, N.J. 309-321 (2004).
- Shevchenko, A., Tomas, H., Havlis, J., Olsen, J. V., Mann, M. In-gel digestion for mass spectrometric characterization of proteins and proteomes. Nature Protocols. 1 (6), 2856-2860 (2006).
- Elinger, D., Gabashvili, A., Levin, Y. Suspension trapping (S-Trap) is compatible with typical protein extraction buffers and detergents for bottom-up proteomics. Journal of Proteome Research. 18 (3), 1441-1445 (2019).
- tom Dieck, S., et al. Direct visualization of newly synthesized target proteins in situ. Nature Methods. 12 (5), 411-414 (2015).
- McShane, E., et al. Kinetic analysis of protein stability reveals age-dependent degradation. Cell. 167 (3), 803-815 (2016).
- Calve, S., Witten, A. J., Ocken, A. R., Kinzer-Ursem, T. L. Incorporation of non-canonical amino acids into the developing murine proteome. Scientific Reports. 6, 32377(2016).
- David, A., et al. Nuclear translation visualized by ribosome-bound nascent chain puromycylation. Journal of Cell Biology. 197 (1), 45-57 (2012).
- Schmidt, E. K., Clavarino, G., Ceppi, M., Pierre, P. SUnSET, a nonradioactive method to monitor protein synthesis. Nature Methods. 6 (4), 275-277 (2009).
- Roux, K. J., Kim, D. I., Burke, B., May, D. G. BioID: A screen for protein-protein interactions. Current Protocols in Protein Science. 91, 11-15 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon