Method Article
Purificación e identificación de proteínas específicas de tipo celular a partir de tejidos complejos utilizando una línea de ratón de ARNt sintetasa de metionina mutante
En este artículo
Resumen
Este protocolo describe cómo realizar el marcado de proteínas específicas del tipo celular con azidonorleucina (ANL) utilizando una línea de ratón que expresa una sintetasa de ARNt de L274G-Metionina mutante (MetRS*) y los pasos necesarios para el aislamiento de proteínas específicas de tipo celular marcadas. Describimos dos posibles vías de administración de ANL en ratones vivos mediante (1) agua potable y (2) inyecciones intraperitoneales.
Resumen
Comprender la homeostasis de proteínas in vivo es clave para saber cómo funcionan las células tanto en condiciones fisiológicas como de enfermedad. El presente protocolo describe el etiquetado in vivo y la posterior purificación de proteínas recién sintetizadas utilizando una línea de ratón diseñada para dirigir el etiquetado de proteínas a poblaciones celulares específicas. Es una línea inducible por la expresión de Cre recombinasa de L274G-Metionina ARNt sintetasa (MetRS*), lo que permite la incorporación de azidonorleucina (ANL) a las proteínas, que de otro modo no ocurrirá. Utilizando el método descrito aquí, es posible purificar proteomas específicos de tipo celular marcados in vivo y detectar cambios sutiles en el contenido de proteínas debido a la reducción de la complejidad de la muestra.
Introducción
La homeostasis aberrante de proteínas es causada por un desequilibrio en la síntesis y degradación de proteínas. Varias enfermedades están relacionadas con alteraciones en la homeostasis de proteínas. El sello distintivo de algunas enfermedades es la presencia de agregados en diferentes ubicaciones subcelulares y áreas cerebrales. La homeostasis proteica no sólo es importante en la enfermedad, sino que también desempeña un papel crucial en la función normal de los órganos y las células1. Por ejemplo, la síntesis de proteínas es necesaria para muchas formas de plasticidad neuronal2,3, determinada por el uso de inhibidores químicos que bloquean la síntesis de proteínas4. Sin embargo, no está claro en qué tipos de células se altera el proteoma para apoyar el aprendizaje y la memoria, ni se entiende qué proteínas específicas en cada tipo de célula aumentan o disminuyen en su síntesis o degradación. Por lo tanto, un estudio exhaustivo de la homeostasis de proteínas requiere la capacidad de diferenciar proteomas provenientes de tipos celulares específicos. De hecho, la identificación de proteomas específicos de tipo celular para estudiar los procesos celulares que ocurren en un entorno multicelular ha sido un obstáculo importante en proteómica. Por esta razón, desarrollamos una técnica que utiliza la expresión de MetRS* combinada con métodos bio-ortogonales que ha demostrado ser una forma efectiva de identificar y purificar proteomas específicos de tipo celular, llenando este vacío 5,6,7.
La expresión de un mutante MetRS* (MetRS L274G) permite la carga del anl analfa de metionina no canónico en el ARNt 8,9 correspondiente y su posterior incorporación a proteínas. Cuando la expresión de MetRS* está regulada por un promotor específico de tipo celular, el aminoácido no canónico se incorporará a las proteínas de manera selectiva de la célula. Una vez que se incorpora ANL en las proteínas, puede ser funcionalizada selectivamente por química de clics y posteriormente visualizada por imágenes (FUNCAT) o por Western Immunoblot (BONCAT). Alternativamente, las proteínas pueden purificarse selectivamente e identificarse mediante espectrometría de masas (EM). Usando esta tecnología, creamos una línea de ratón que expresa la proteína MetRS* bajo el control de la recombinasa Cre. Teniendo en cuenta el creciente número de líneas de ratón Cre disponibles, el sistema MetRS* se puede utilizar en cualquier campo para estudiar cualquier tipo de célula de cualquier tejido para el que exista una línea Cre. El marcaje de proteínas con ANL es posible in vitro o in vivo, y no altera el comportamiento del ratón ni la integridad de las proteínas6. El período de tiempo de etiquetado se puede adaptar a la pregunta científica de cada investigador, etiquetando proteínas recién sintetizadas (tiempos de etiquetado más cortos) o proteomas completos (tiempos de etiquetado más largos). El uso de esta técnica está limitado por el número de células del tipo que el investigador está dispuesto a estudiar; Por lo tanto, el aislamiento de proteínas de tipos celulares con números bajos o bajas tasas metabólicas no es posible por este método. El objetivo del método presentado es identificar proteínas/proteomas específicos del tipo celular marcados in vivo. En este protocolo, describimos cómo etiquetar proteomas específicos de tipo celular con ANL en ratones vivos y purificar las proteínas marcadas. Después de la purificación, las proteínas pueden ser identificadas por protocolos de espectrometría de masas de rutina 5,10. La reducción de la complejidad de la muestra lograda en este método mediante la purificación selectiva de proteínas de poblaciones celulares específicas permite al experimentador detectar cambios sutiles en los proteomas, por ejemplo, en respuesta a cambios ambientales. La purificación de las proteínas marcadas se puede lograr en ~ 10 días, sin incluir el análisis de MS o el período de etiquetado. Aquí, describimos dos métodos para la administración de ANL a ratones que expresan MetRS*, a saber, (1) agregar el aminoácido en el agua potable y (2) introducir ANL mediante inyecciones intraperitoneales. Independientemente del método elegido para la administración de ANL, los pasos de aislamiento y purificación son los mismos (a partir del paso 2).
Protocolo
Todos los experimentos con animales se realizaron con permiso de las oficinas gubernamentales locales en Alemania (RP Darmstadt; protocolos: V54-19c20/15-F122/14, V54-19c20/15-F126/1012) o España (Comité de Experimentación Animal de la UCM y Consejería Ambiental de la Comunidad de Madrid, número de protocolo: PROEX 005.0/21) y cumplen con las normas de la Sociedad Max Planck y la normativa española y siguen las directrices de la UE para el bienestar animal.
1. Etiquetado metabólico in vivo con ANL
- Agua potable:
- Disuelva ANL y maltosa en el agua potable regular de los ratones. La concentración de maltosa recomendada es de 0,7% (peso / vol). La cantidad máxima de ANL añadida al agua potable es del 1% (peso / vol). Una vez que la mezcla esté lista, esterilizarla por filtración y almacenarla a 4 °C.
- Proporcionar la mezcla preparada en el paso 1.1 a los ratones. Cambie las botellas con frecuencia (por ejemplo, cada 3 días) para evitar contaminaciones y revíselas todos los días para buscar posibles contaminaciones u obstrucciones. Controle la cantidad de agua potable diariamente pesándola, para conocer la ingesta de ANL de los ratones.
NOTA: El período máximo de etiquetado evaluado aquí es de 3 semanas utilizando una concentración de ANL del 1% en el agua potable, lo que resultó en un etiquetado bien detectable en el cerebro. Un buen etiquetado también se puede lograr en 2 semanas. Ni tiempos de etiquetado más cortos, ni puntos de tiempo más largos, ni otras cantidades de ANL han sido estudiadas por nosotros utilizando este método de administración, lo que no implica que lo anterior no funcionaría. Las tasas de incorporación de ANL pueden variar según el tejido estudiado o el tipo de célula. El tiempo de etiquetado puede variar dependiendo de la pregunta científica; Por ejemplo, si los proteomas fueran a ser marcados, entonces el tiempo de etiquetado debe calcularse en función de las vidas medias promedio de las proteínas en el tejido estudiado. Si el interés es solo identificar proteínas recién sintetizadas, los tiempos pueden acortarse. Los períodos de etiquetado y las dosis de ANL deben probarse empíricamente para cada condición experimental y tipo de célula de interés.
- Administración intraperitoneal:
- Disuelva el ANL en solución fisiológica de NaCl a 400 mM, tampón salino fosfato (PBS) o agua.
- Asegúrese de que la osmolaridad de la solución esté dentro del rango fisiológico. Aquí, se administran 10 ml / kg de peso corporal de solución de ANL a los ratones.
NOTA: Un buen etiquetado en las neuronas excitadoras del cerebro se obtiene con éxito mediante una inyección intraperitoneal (IP) diaria con 400 mM ANL durante 1 semana. En este estudio no se han probado concentraciones más bajas de ANL ni otros períodos de etiquetado mediante inyecciones IP, lo que no implica que no funcionen. ANL es suministrado como clorhidrato por algunas compañías. En este caso, el pH de las soluciones debe ajustarse al pH adecuado (dependiendo de la vía de administración). La ANL también puede ser sintetizada siguiendo el método publicado por Link et al.11 con algunas modificaciones5. El etiquetado de ANL se puede realizar utilizando otras concentraciones de ANL, períodos de tiempo y rutas de administración. Para tejidos/tipos de células con bajas tasas metabólicas, se puede utilizar una dieta de metionina de bajo contenido, siempre comenzando 1 semana antes de la administración de ANL. Cuando se utiliza 400 mM ANL, puede precipitar mientras se almacena a 4 °C; el calentamiento hasta 37 °C devuelve el ANL a la solución. Deje que alcance la temperatura ambiente antes de la inyección. En la mayoría de las regiones del mundo, la administración de ANL a ratones es un experimento animal y debe ser aprobado por las autoridades pertinentes.
2. Recolección de tejidos, lisis y extracción de proteínas
- Disección de tejido: disecciona el área de tejido que contiene el tipo de célula de interés y observa el peso de la pieza de tejido recolectada.
NOTA: Las muestras pueden almacenarse a -80 °C durante varios meses (punto de parada). En el protocolo actual, se diseccionaron la corteza y el hipocampo de los ratones. - Lisis tisular: Homogeneizar el tejido a temperatura ambiente añadiendo un volumen 12-15 veces el peso húmedo del tejido del tampón de lisis (PBS pH 7.4, 1% peso / vol SDS, 1% (vol / vol) Triton X-100, Benzonasa (1: 1000 vol / vol), un inhibidor de la proteasa (PI) (EDTA libre 1: 4000)). Por ejemplo, para una pieza de tejido de 20 mg, agregue 240-300 μL de tampón de lisis. Triturar el tejido hasta homogeneizarlo.
NOTA: Las muestras pueden almacenarse a -80 °C durante varios meses (punto de parada). Para la homogeneización directa en tubos de 1,5 ml, se recomienda el uso de un homogeneizador portátil que funcione con baterías, especialmente cuando se procesan pequeños trozos de tejido. Para piezas más grandes, se puede usar un homogeneizador Dounce. A cada tampón donde se incluyen inhibidores de la proteasa (IP), deben agregarse inmediatamente antes de su uso y no antes. - Desnaturalización de proteínas: Calentar el homogeneizado a 75 °C durante 15 min y centrifugar a 17.000 x g durante 15 min a 10 °C. Transfiera el sobrenadante a un tubo nuevo.
- Medición del contenido de proteína: Mida la concentración de proteína de cada muestra y ajuste todas las muestras para compararlas con la misma concentración de proteína; Ajuste la concentración agregando tampón de lisis. Un rango de concentración óptimo está entre 2-4 μg/μL.
NOTA: Las muestras pueden almacenarse a -80 °C durante varios meses (punto de parada). - Alquilación
- Diluir las muestras homogeneizadas 2-3 veces en PBS (pH 7.8) + PI (libre de EDTA 1:4000).
- Añadir yodoacetamida (IAA) a una concentración final de 20 mM (solución madre 500 mM).
- Dejar las muestras durante 1-2 h a 20 °C en la oscuridad. Realice los pasos 2.5.2-2.5.3 dos veces.
NOTA: Para algunos tejidos, si el clic no específico en el control negativo permanece alto, podría ser necesario realizar un paso de reducción antes de la alquilación12. Prepare el stock de IAA recién hecho justo antes de agregarlo a las muestras. Las muestras pueden almacenarse a -80 °C durante varios meses (punto de parada).
- Intercambio de búfer: equilibre las columnas de intercambio de búfer utilizando el búfer de intercambio químico de clic (PBS pH 7.8, 0.04% (peso / vol) SDS, 0.08% (vol / vol) Triton X-100 y PI (1: 4000)). Siga las instrucciones del fabricante. Intercambie todas las muestras alquiladas.
NOTA: Las muestras elutadas pueden almacenarse a -80 °C durante varios meses (punto de parada). En este paso, se elimina el IAA y el búfer de química de clic intercambia el búfer. La proteína se puede medir nuevamente después del paso de intercambio de tampón para asegurarse de que las proteínas no se perdieron durante el proceso de intercambio de tampón. Dependiendo del tipo de columnas utilizadas para el intercambio de tampón, la proteína puede perderse masivamente. Utilice las columnas recomendadas para evitar la pérdida de proteínas. Para el almacenamiento de muestras, se recomienda la preparación de alícuotas. - Haga clic en la reacción para evaluar la incorporación de ANL en el experimento
- Tomar 40 μL de las muestras eluidas en el paso 2.6 y añadir PBS (pH 7,8) a un volumen final de 120 μL.
- Para configurar la reacción química de clic, agregue los siguientes reactivos en el orden y vórtice dados durante 20 s después de cada adición: 1.5 μL de ligando triazol (stock 40 mM), 1.5 μL de biotina alquino (stock 5 mM) y 1 μL de bromuro de Cu(I) (stock 10 mg/mL, disuelto en DMSO por pipeteo cuidadoso hacia arriba y hacia abajo, no vórtice). Agregue los reactivos lo más rápido posible.
- Incubar las muestras durante la noche en la oscuridad a 4 °C con rotación continua.
- Centrifugar las muestras a 4 °C durante 5 min a 17.000 x g. Una bolita ligeramente turquesa será visible. Transfiera el sobrenadante a un tubo nuevo.
NOTA: Prepare el caldo de bromuro de Cu (I) inmediatamente antes de usarlo para evitar la oxidación del cobre. Mantener constante la relación de volúmenes entre la muestra lisada y el PBS entre muestras es esencial para garantizar la misma concentración final de detergente en cada tubo. Para acelerar el montaje de la reacción química de clic, se recomienda un vórtice de mesa con bastidores para tubos. Almacene las muestras en las que se ha hecho clic a -80 °C durante varios meses, o a -20 °C durante un par de semanas hasta su posterior procesamiento (punto de parada).
- Análisis de Western blot para evaluar la incorporación de ANL en el experimento
- Ejecute una SDS-PAGE con 20-40 μL del sobrenadante de la reacción química de clic (paso 2.7). Use geles de acrilamida al 12%, funcione en SDS al 1%, Tris-HCl al 250 mM, glicina al 2 mM. Corra hasta que la parte delantera esté fuera del gel.
- Transferir las proteínas a una membrana de nitrocelulosa o PVDF13.
- Incubar la membrana durante la noche a 4 °C con el anticuerpo GFP (1:500, la proteína GFP se cotraduce con MetRS*5) y el anticuerpo de biotina (1:1000) para la detección de alquinos clic, que se conjuga con la biotina.
- Lave el anticuerpo primario 2 veces durante 10 minutos y agregue el anticuerpo secundario durante 1,5 h.
- Lave el anticuerpo secundario 2 veces durante 10 minutos y escanee (si usa anticuerpos conjugados con fluoróforos) o exponerlo a películas (si usa anticuerpos conjugados con peroxidasa de rábano picante).
- Cuantificar la señal de biotina utilizando ImageJ o software similar, evaluar el etiquetado general de cada muestra del experimento y detectar posibles valores atípicos. Evaluar la relación señal-ruido (etiquetado de la muestra ANL al fondo de los controles negativos) del experimento y detectar posibles animales que no pudieron incorporar/incorporar ANL.
NOTA: Para muestras con alta incorporación de ANL, la purificación de proteínas es teóricamente posible utilizando alquinos no escindibles como el utilizado en esta sección para la evaluación de la incorporación de ANL. Usando muestras cerebrales, el fondo obtenido en los controles negativos por EM después de la purificación de proteínas utilizando alquinos no escindibles es demasiado alto14. Por lo tanto, solo se utilizaron los alquinos no escindibles, más fáciles de manejar y no escindibles, para una primera muestra general y evaluación del experimento. Independientemente del tipo de alquino destinado a la purificación de proteínas, es aconsejable realizar los pasos para la optimización de la dosificación de alquinos descritos en la siguiente sección (paso 2.9).
- Reacción de clic para la optimización de la dosis de alquino escindible
- Preparar cuatro tubos con 40 μL de una muestra representativa (obtenida en la etapa 2.6 y evaluada en la etapa 2.8) y añadir PBS (pH 7,8) a un volumen final de 120 μL.
- Configure la reacción química de clic como se explica en el paso 2.7, pero esta vez, agregue cuatro cantidades diferentes del DST-alquino. Para dicho alquino, se recomienda probar 5 μM, 15 μM, 30 μM y 60 μM.
- Repita el paso 2.8 para decidir cuál es la dosis de alquino más adecuada para la pregunta experimental específica en estudio, en función de la mayor eficiencia de etiquetado con la señal de fondo más baja.
NOTA: Antes de someter las muestras restantes a una reacción de clic, se debe determinar la cantidad óptima de cualquier alquino. Hay varias opciones de alquinos disponibles comercialmente que se pueden utilizar. Se diferencian en la forma de escisión (por ejemplo, agentes ligeros o reductores). También es posible la química de clic sin cobre utilizando reactivos promovidos por deformación (por ejemplo, reactivos DBCO). Los pequeños cambios en la cantidad de alquinos o reactivos DBCO a menudo aumentan significativamente la señal en el control negativo (fondo). Aquí, se describe la purificación de proteínas utilizando un alquino con un puente disulfuro escindible por agentes reductores (disulfuro biotina alquino o DST-alquino15). Cuando utilice el DST-alquino, para ejecutar el SDS-PAGE (paso 8.1), evite cargar tampones con agentes reductores fuertes como DTT o β-mercaptoetanol para evitar la escisión de alquinos. En este estudio, el calentamiento a 72 °C, durante 5 min, utilizando 5 mM de N-etilmaleimida (NEM) en el tampón de carga no afecta la estabilidad del DST-alquino. El paso 2.9.2 se puede repetir, probando dosis más finas en el rango de la mejor observada.
- Reacción de clic preparativa para la purificación de proteínas
- Haga clic en todas las muestras obtenidas en el paso 2.6 con la dosis óptima de alquino determinada en el paso 2.9 y analice una fracción por SDS-PAGE y Western blot (paso 2.8).
NOTA: Asegúrese de que las pipetas estén calibradas correctamente para garantizar la precisión de la ampliación de la muestra. Las muestras en las que se hace clic se pueden almacenar a -80 °C durante varios meses (punto de parada).
- Haga clic en todas las muestras obtenidas en el paso 2.6 con la dosis óptima de alquino determinada en el paso 2.9 y analice una fracción por SDS-PAGE y Western blot (paso 2.8).
3. Purificación de proteínas
- Intercambio de búfer
- Sujete todas las muestras en las que se haga clic a un procedimiento de intercambio de tampón como se describe en el paso 2.6, pero utilice el tampón de unión de neutravidina como tampón de equilibrio (PBS pH 7.4, 0.15% (peso / vol) SDS, 1% (vol / vol) Triton X-100 y PI (1: 2000)).
NOTA: Las muestras elutadas pueden almacenarse a -80 °C durante varios meses (punto de parada). En este paso, se elimina el alquino libre sin clic y el tampón de unión de neutravidina intercambia el tampón.
- Sujete todas las muestras en las que se haga clic a un procedimiento de intercambio de tampón como se describe en el paso 2.6, pero utilice el tampón de unión de neutravidina como tampón de equilibrio (PBS pH 7.4, 0.15% (peso / vol) SDS, 1% (vol / vol) Triton X-100 y PI (1: 2000)).
- Unión a perlas de neutravidina
- Lave las perlas de alta capacidad de Neutravidin con el tampón de unión de Neutravidin (ver paso 3.1); Mezclar las perlas con el tampón y centrifugar la mezcla durante 5 min a 3000 x g, y desechar el sobrenadante. Repita tres veces.
- Prepare una suspensión 1:1 de perlas de Neutravidin agregando el mismo volumen de perlas secas y tampón de unión de Neutravidina.
- Reservar 20-40 μL de cada muestra como lisado prepurificado y almacenarlo a -20 °C.
- Medir la concentración de proteínas (ver paso 2.4) y mezclar 100 μg de proteína con 4 μL de la suspensión de perlas obtenida en el paso 3.2.2.
- Incubar la mezcla durante la noche a 4 °C con rotación continua, permitiendo que las proteínas marcadas se unan a las perlas.
NOTA: En cuanto a la reacción química de clic, la reacción básica de purificación de afinidad descrita en el paso 3.2.4 se puede ampliar. Por ejemplo, para la purificación de proteínas de las neuronas excitadoras del hipocampo, se mezcla 1 mg de lisado de proteínas con 40 μL de perlas de Neutravidin (suspensión 1: 1). La cantidad de perlas de Neutravidin se puede aumentar o reducir de acuerdo con la cantidad de proteínas marcadas por mg de proteína total, que dependerá del tipo de célula elegido y el período de etiquetado y debe determinarse empíricamente.
- Lavados de perlas de neutravidina y elución de proteínas
- Recoger los sobrenadantes por centrifugación (véase el paso 3.2.1). Reservar una alícuota de 20-40 μl de cada sobrenadante para su posterior análisis y congelar el resto a -80 °C.
- Lave las perlas con el tampón de lavado 1 de Neutravidin refrigerado (PBS pH 7.4, 0.2% (peso / vol) SDS, 1% (vol / vol) Triton X-100 y PI (1: 2000)) agregando el amortiguador, sedimentando las perlas y desechando el sobrenadante. Repita este paso tres veces.
- A continuación, añadir el mismo tampón e incubar las perlas durante 10 minutos bajo rotación continua a 4 °C antes de desechar el sobrenadante. Repita este paso tres veces.
- Lave las perlas como se explica en los pasos 3.3.2-3.3.3 pero utilizando el tampón de lavado 2 de Neutravidin (1x PBS, pH 7.4 y PI 1:2000).
- Lavar las perlas como se explica en el paso 3.3.2-3.3.3 pero utilizando el tampón de lavado Neutravidin 3 (bicarbonato de amonio 50 mM y PI 1:2000).
- Eluya las proteínas clicadas incubando las perlas durante 30 min a 20 °C con un volumen de tampón de elución de Neutravidin (5% (vol/vol) β-mercaptoetanol y 0,03% (peso/vol) SDS) correspondiente a un volumen de las perlas secas utilizadas.
- Mantener las perlas en suspensión durante la elución por agitación continua (1000 rpm) en un agitador termobloque. Eluye dos veces y combine ambos eluidos.
NOTA: La centrifugación establecida para la separación de la fracción sólida del purín (perlas) y la fase acuosa (sobrenadante), como se explica en el paso 3.2.1, se aplica a los pasos 3.3.1-3.3.7. La cantidad de SDS se puede aumentar si la elución de proteínas no es completa. Sin embargo, con cantidades superiores al 0,08%-0,1% de SDS, las proteínas no unidas específicamente a las perlas comienzan a ser eluyidas. β-mercaptoetanol es volátil y tóxico; Se recomienda el uso de una capucha para los pasos 3.3.6-3.3.7. Las muestras recogidas en los pasos 3.2.3 y 3.3.1 deben ser ejecutadas por SDS-PAGE y evaluadas por Western blot (ver paso 2.8) para evaluar la eficiencia de la purificación de afinidad (paso 3.3).
- Evaluación de proteínas eluidas
- Cargue 1/3 de las muestras eluyidas en una SDS-PAGE (consulte el paso 2.8.1).
- Tiñe el gel para visualizar proteínas usando un método de alta sensibilidad, como la tinción de plata o un método fluorescente.
- Si las muestras tienen la calidad esperada, lo que significa que hay 3-4 veces más proteína en las muestras marcadas con ANL en comparación con el control negativo, las proteínas eluyentes pueden identificarse mediante métodos de espectrometría de masas de rutina como el explicado en la referencia5.
Resultados
Siguiendo el protocolo descrito (resumido en la Figura 1), se administró ANL a ratones mediante inyecciones intraperitoneales diarias (400 mM ANL 10 mL/kg, Nex-Cre::MetRS*) durante 7 días, o a través de agua potable (0,7% maltosa, 1% ANL, CamkII-Cre::MetRS*) durante 21 días. Después del etiquetado, las áreas cerebrales correspondientes fueron diseccionadas, lisadas, alquiladas y clicadas. Las reacciones de clic fueron analizadas por SDS-PAGE y Western Immunoblot. Las imágenes representativas de los experimentos se muestran en la Figura 2 para la administración de ANL por inyección IP y en la Figura 3 para la administración de ANL a través del agua potable. Tenga en cuenta que el objetivo de las cifras es mostrar que ambos protocolos de etiquetado funcionan, no compararlos. La comparación no es posible porque diferentes poblaciones neuronales están etiquetadas en los dos experimentos mostrados (neuronas excitatorias del hipocampo y la corteza, y las neuronas de Purkinje del cerebelo).
Otro experimento proporciona un ejemplo para la determinación de la concentración óptima de alquino escindible DST (Figura 4). En este ejemplo, el cambio de pliegue entre la muestra marcada y el control es más alto a una concentración de alquino de 14 μM. Esta concentración de alquino se aplica a todas las muestras. Después de la verificación del etiquetado de cada muestra en el experimento con la cantidad de alquino elegida, todas las muestras se sometieron a la purificación de proteínas marcadas con ANL / biotina por unión de afinidad utilizando perlas de Neutravidin (Figura 5). En este paso, las proteínas se unen a las perlas, se lavan y posteriormente se eluyen reduciendo el puente disulfuro presente en el DST-alquino. Después de este último paso, la biotina y parte del alquino permanecen unidas a las cuentas. Las proteínas que contienen ANL (unidas al resto del alquino) se recuperan en el tampón de elución. Para evaluar la eficiencia de la etapa de elución, un tercio del volumen de eluido se carga en un gel SDS-PAGE y se visualiza utilizando un método de tinción de proteína total sensible. Se debe observar al menos una diferencia de intensidad de 3 veces de la tinción total de proteínas entre los controles negativos y las muestras marcadas con ANL para lograr resultados interpretables con espectrometría de masas. La finalización de todos los pasos descritos en este protocolo será seguida por la preparación, adquisición y análisis de muestras de MS5. Aunque no hay requisitos especiales para la EM (cada laboratorio puede utilizar su protocolo rutinario de EM 5,10), debe tenerse en cuenta que la cantidad de proteínas purificadas será generalmente baja (del orden de nanogramos). La Figura 6 proporciona un ejemplo de resultados de EM con un enriquecimiento claro en la muestra marcada con ANL en comparación con el control (Figura 6A). Esta diferencia ya era visible en la tinción de proteína total que se muestra en la Figura 5. Además de los cambios en la intensidad del péptido, también hay proteínas únicas que se encuentran en ambas muestras (Figura 6B).
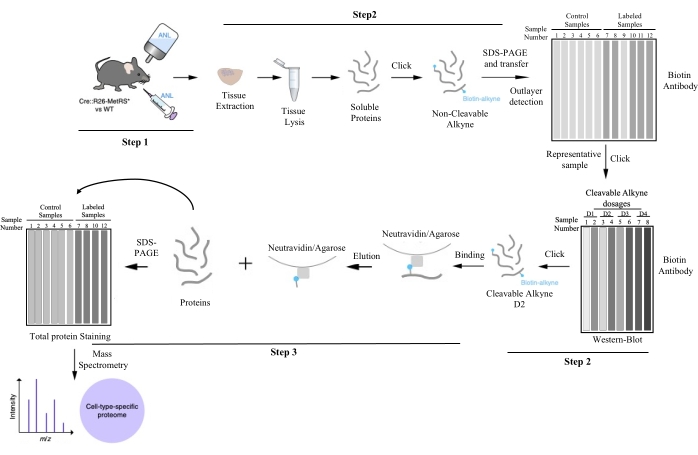
Figura 1: Cartera de trabajo para la purificación de proteínas específicas del tipo celular mediante BONCAT. Después del etiquetado de proteínas con ANL, el tejido de interés se disecciona y lisa, se marca con biotina mediante química de clic, y la cantidad de etiquetado para cada muestra es evaluada por BONCAT. Los valores atípicos (que no incorporaron ANL) y las muestras representativas se pueden diferenciar en este paso. Una de las muestras representativas se hace clic de nuevo para encontrar la dosis de alquino escindible optimizada para la posterior purificación de proteínas. La dosis de alquino que logra la mejor relación señal-ruido se aplica a cada réplica biológica. Las proteínas específicas del tipo celular se obtienen por purificación de afinidad. Las muestras purificadas se estudian por espectrometría de masas y se identifican las proteínas. Esta cifra ha sido modificada de Alvarez-Castelao et al. (2017)6. Haga clic aquí para ver una versión más grande de esta figura.
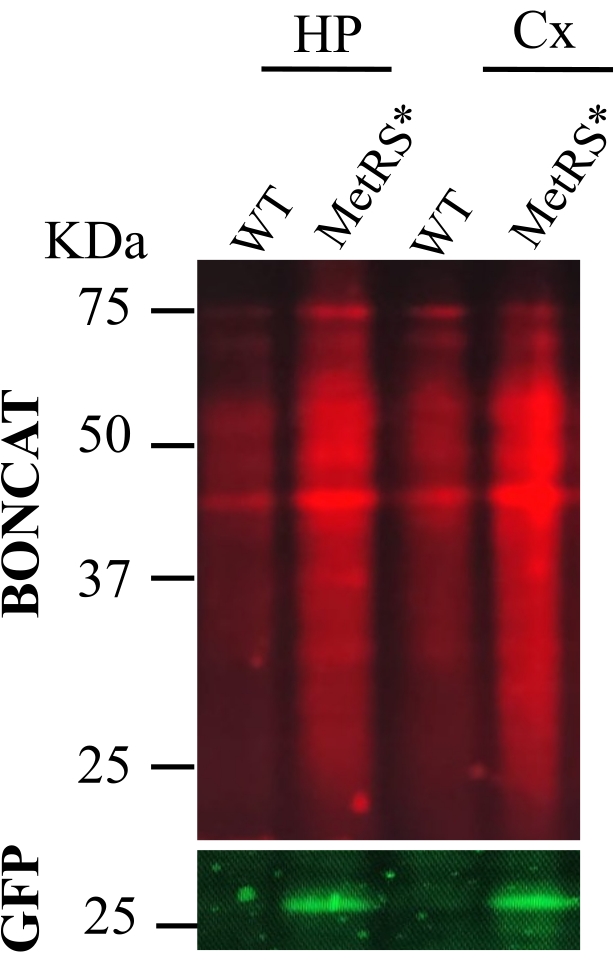
Figura 2: Administración de ANL por inyecciones intraperitoneales (IP). BONCAT se realizó para evaluar el marcado de proteínas en el hipocampo (HP) y la corteza (CX) después del marcado metabólico de proteínas con ANL mediante inyecciones IP diarias durante 7 días. Se hizo clic en muestras de ratones de tipo salvaje como control negativo (wt) en paralelo a las muestras etiquetadas (MetRS*). Las proteínas marcadas son de las neuronas excitadoras usando una línea Nex-Cre::MetRS*16. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Administración de ANL por agua potable. BONCAT se realizó para evaluar el marcado de proteínas en las neuronas de Purkinje del cerebelo después de la administración de ANL a ratones GAD-Cre::MetRS* a través de agua potable durante 21 días. La figura muestra un control negativo (wt) y una muestra etiquetada (MetRS*) lisada y clicada en paralelo. Esta cifra ha sido modificada de Alvarez-Castelao et al. (2017)6. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Titulación de la dosis de DST-alquino. Se elige una réplica biológica por experimento como muestra representativa para valorar la concentración óptima de alquinos. Aquí se probaron tres concentraciones (14, 28 y 56 μM) del alquino en la reacción de clic para BONCAT. La relación de etiquetado entre la muestra marcada con ANL (MetRS*) y el control negativo (wt) determina la relación señal-ruido (que se muestra en el gráfico). 14 μM es la mejor concentración de alquino obtenida en este experimento. Para este experimento se utilizó tejido de corteza de la línea de ratón Nex::MetRS* marcada con ANL. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Proteínas purificadas. Las proteínas marcadas de las neuronas de Purkinje se purificaron y tiñeron con SYPRO Ruby. Esta cifra ha sido modificada de Alvarez-Castelao et al. (2017)6. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Identificación y cuantificación de proteínas. El proteoma de células de Purkinje se obtuvo por espectrometría de masas utilizando cerebelo de una línea de ratón GAD-Cre::MetRS* como material de partida. (A) muestra una mayor abundancia (intensidad peptídica) de las proteínas identificadas en muestras marcadas con ANL en comparación con ratones de tipo salvaje (wt). (B) El proteoma de Purkinje se obtuvo agrupando proteínas enriquecidas en las muestras marcadas con ANL en A (definidas por >intensidades de 3 veces en ratones MetRS* en comparación con wt), y proteínas únicas encontradas en los ratones que expresan MetRS*. Esta cifra ha sido modificada de Alvarez-Castelao et al. (2017)6. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Los aspectos críticos del protocolo son; la inclusión de controles negativos, tener suficientes réplicas biológicas, la vía de administración de ANL, la cantidad y la duración, la alquilación de las muestras, la concentración de alquinos y la eliminación de β-mercaptoetanol cuando se usa DST-alquino.
Es clave incluir muestras de control negativo procedentes de animales marcados con ANL sin conductor Cre y, por lo tanto, sin expresión de MetRS*. Estas muestras deben someterse a todos los pasos descritos en el protocolo en paralelo a las muestras de ratones MetRS* inducidos por Cre marcados con ANL. Las muestras en las que no se hace clic no son controles válidos, ya que cualquier resultado obtenido utilizando solo este control puede deberse a un clic no específico. No hemos observado incorporación de ANL en células que no expresan el gen MetRS*, ni expresión de MetRS* en células sin Cre; por lo tanto, los animales de peso etiquetados con ANL se pueden utilizar como control negativo.
Dado que el protocolo aquí descrito consta de muchos pasos en los que se pueden perder muestras, se recomienda calcular las réplicas biológicas teniendo en cuenta la falla técnica. Para las muestras de cerebro, hay aproximadamente un 30% de pérdida de replicación.
Describimos aquí dos rutas y duraciones de administración de ANL. Esto no excluye que otras vías de administración (por ejemplo, agregar ANL a los alimentos) funcionen igualmente bien. Con respecto a la cantidad de ANL administrada, los experimentos que se muestran aquí se realizaron utilizando cantidades relativamente altas de ANL. El etiquetado con cantidades más bajas también es posible dependiendo de la configuración experimental. El lapso de tiempo más corto de etiquetado de ANL reportado en este protocolo es de 1 semana, y el más largo de 21 días. Se podrían aplicar períodos de etiquetado más cortos o más largos, pero no lo hemos determinado. Los investigadores deben determinar la ruta de administración de ANL, la dosis de ANL y el período de etiquetado de ANL adecuado para la pregunta experimental específica en estudio considerando las propiedades metabólicas del tejido, el tipo de célula de interés y la pregunta experimental en estudio.
La alquilación de muestra, también conocida como tapado, es un paso importante para evitar el clic17 en segundo plano. Cuando la alquilación se realiza correctamente, el clic no específico monitoreado por las muestras de control se reduce, y la diferencia entre el control negativo y las muestras marcadas con ANL es mayor. La eliminación de IAA libre después de la alquilación también es un paso clave. La presencia de pequeñas cantidades de IAA durante la reacción de clic limitará su eficiencia. Ocasionalmente, el paso de desalinización que también elimina IAA (paso 2.6), debe repetirse para garantizar la eliminación adecuada de IAA.
Hay varios reactivos de biotina-alquinos y -DBCO disponibles comercialmente de una variedad de compañías. Las principales diferencias entre ellos se refieren a la longitud de la cadena de encadenamiento de polietilenglicol (PEG) y la ausencia, presencia y tipo de grupos escindibles. Independientemente del tipo utilizado, las cantidades excesivas de alquino conducen a un clic no específico a proteínas que contienen solo aminoácidos naturales. Esto se puede prevenir fácilmente mediante una titulación precisa y cuidadosa de la cantidad de alquinos. Como se ha descrito, la mejor manera de lograr esto es utilizando las muestras lisadas y alquiladas reales que se utilizarán en los pasos posteriores de purificación de proteínas (paso 2.6) y el análisis de espectrometría de masas.
Cuando se utilizan alquinos DST en la reacción de clic, el β-mercaptoetanol reduce el puente disulfuro y disocia las proteínas marcadas de las perlas en la etapa de elución (paso 3.3). β-mercaptoetanol debe eliminarse de las muestras para permitir la digestión enzimática necesaria para la EM. Hay varias maneras de hacer esto (por ejemplo, liofilización18, escisión de un gel19 o limpieza con columnas S-Trap20), y la elección debe ser hecha por el laboratorio de EM que procesa las muestras.
Las modificaciones y la solución de problemas del método deben dirigirse para optimizar los pasos críticos mencionados en el protocolo. Por ejemplo, si hay demasiada variabilidad, agregue más réplicas biológicas; si el etiquetado es demasiado bajo, aumente la concentración de ANL, use períodos de tiempo de etiquetado más largos o pruebe otras rutas de administración de ANL que podrían ser más eficientes para el tejido estudiado. Para la alquilación de proteínas, se podría implementar un paso de reducción previo a la adición de IAA.
La principal limitación del método es la purificación de proteomas que surgen de poblaciones de células pequeñas, incluso si se disecciona un área de tejido pequeño. Los proteomas de poblaciones celulares más numerosas que, sin embargo, están dispersos en áreas de tejido más grandes también son difíciles de acceder. Sin embargo, la técnica de etiquetado in vivo descrita aquí todavía se puede aplicar para evaluar los tipos de células referidas mediante imágenes (por ejemplo, con FUNCAT o FUNCAT-PLA21).
Este método es actualmente el único método disponible para el estudio in vivo de proteomas específicos de tipo celular basados en proteínas de longitud completa y para la visualización in situ de proteínas completas específicas de tipo celular. Otros aminoácidos no canónicos como la azidohomoalanina (AHA) también pueden ser utilizados para el marcado in vivo de proteínas y la purificación de proteomas, pero carecen de especificidad de tipo celular22,23. Otros enfoques, como la puromicina, son adecuados para proporcionar una imagen fija de la síntesis de proteínas24,25. Sin embargo, es posible usar aminoácidos no canónicos durante períodos más largos de etiquetado, lo que también refleja la degradación de proteínas y muestra un proteoma celular más preciso. Los métodos basados en BioID se utilizan para identificar proteínas en regiones subcelulares específicas, independientemente de su momento/lugar de síntesis26.
En la línea de ratones que establecimos (disponible en Jackson Lab, stock no. 028071), la metionina mutante ARNt sintetasa (MetRS*) que permite la incorporación de ANL en las células se expresa de una manera dependiente de Cre. Con esta línea de ratón como herramienta, es posible expresar exclusivamente el MetRS* en tipos de celda para los que están disponibles líneas Cre-driver. Esta disposición dota al método de una versatilidad considerable, por lo que es útil en casi todas las áreas de la investigación biomédica.
Divulgaciones
Los autores declaran que no hay intereses financieros contrapuestos.
Agradecimientos
B.A-C está financiado por el Ministerio de Ciencia e Innovación (Ramón y Cajal-RYC2018-024435-I), por la Comunidad de Madrid (Atracción de Talento-2019T1/BMD-14057) y las ayudas MICINN (PID2020-113270RA-I00). R. A-P está financiado por la Comunidad de Madrid (Atracción de Talento-2019T1/BMD-14057). E.M.S. está financiado por la Sociedad Max Planck, un premio Advanced Investigator del Consejo Europeo de Investigación (subvención 743216), DFG CRC 1080: Mecanismos moleculares y celulares de la homeostasis neuronal, y DFG CRC 902: Principios moleculares de la regulación basada en ARN. Agradecemos a D.C Dieterich y P. Landgraf por su asesoramiento técnico y la síntesis del DST-Alkyne. Agradecemos a E. Northrup, S. Zeissler, S. Gil Mast y a la instalación animal del MPI for Brain Research por su excelente apoyo. Agradecemos a Sandra Goebbels por compartir la línea de ratones Nex-Cre. Agradecemos a Antonio G. Carroggio por su ayuda con la edición en inglés. B.A-C. Diseñé, conduje y analicé experimentos. B.N-A, D.O.C, R.A-P, C. E. y S. t. D. realizó y analizó experimentos. B.A-C y E.M.S. diseñaron experimentos, y supervisaron el proyecto, B.A-C escribió el artículo. Todos los autores editaron el artículo.
Materiales
| Name | Company | Catalog Number | Comments |
| 12% Acrylamide gels | GenScript | SurePAGE, Bis-Tris, 10 x 8, 12% | |
| β-Mercaptoethanol | Sigma | M6250 | Toxic; use a lab coat, gloves and a fume hood. |
| Ammonium bicarbonate | Sigma | 9830 | Toxic; use a lab coat, gloves and a fume hood |
| ANL | Synthesized as described previously for AHA (see references 5 and 11) | ||
| ANL-HCl | IrishBotech | HAA1625.0500 | |
| Benzonase | Sigma | E1014 | |
| Biotin alkyne | Thermo, | B10185 | |
| Chicken antibody anti-GFP | Aves | 1020 | |
| Complete EDTA-free protease inhibitor | Roche | 4693132001 | Toxic; use a lab coat and gloves. |
| Copper (I) bromide | Sigma | 254185 | 99.999% (wt/wt) |
| Disulfide tag (DST)-alkyne | Synthesized as reported in reference number 15, in which it is referred to as probe 20 | ||
| DMSO | Sigma | 276855 | |
| Filters | Merk | SCGP00525 | |
| Iodo acetamide (IAA) | Sigma | I1149 | |
| IR anti chicken 800 | LI-COR | IRDye 800CW | Donkey anti-Chicken Secondary Antibody |
| IR anti rabit 680 | LI-COR | IRDye 680RD | Goat anti-Rabbit IgG Secondary Antibody |
| Maltose | Sigma | M9171 | |
| Manual Mixer | BioSpec Products | 1083 | |
| NaCl | Sigma | S9888 | |
| N-ethylmaleimide | Sigma | 4259 | Toxic; use a lab coat, gloves and a fume hood. |
| NeutrAvidin beads | Pierce | 29200 | |
| Nitrocellulose membrane | Bio-rad | 1620112 | |
| PBS 1X | Thermo | J62036.K2 | |
| PBS 1X pH 7.8 | Preparation described in reference number 5 | ||
| PD SpinTrap G-25 columns | GE Healthcare | Buffer exchange | |
| Pierce BCA Protein Assay Kit | Thermo, | 23225 | Reagents in the Pierce BCA Protein Assay Kit are toxic to aquatic life. |
| Polyclonal rabbit anti-biotin antibody | Cell Signaling | 5597 | |
| PVDF membrane | Millipore | IPVH00010 | |
| SDS 10% | Sigma | 71736 | |
| SDS-PAGE Running buffer MOPS | GenScript | M00138 | |
| SYPRO Ruby stain | Sigma | S4942 | |
| Table automatic Vortexer | Eppendorf | Mixmate | |
| Triazole ligand | Sigma | 678937 | |
| Triton X-100 | Sigma | T9284 | |
| Water | Sigma | W4502 | Molecular biology grade |
Referencias
- Alvarez-Castelao, B., Schuman, E. M. The regulation of synaptic protein turnover. Journal of Biological Chemistry. 290 (48), 28623-28630 (2015).
- Sutton, M. A., Schuman, E. M. Dendritic protein synthesis, synaptic plasticity, and memory. Cell. 127 (1), 49-58 (2006).
- Dorrbaum, A. R., Alvarez-Castelao, B., Nassim-Assir, B., Langer, J. D., Schuman, E. M. Proteome dynamics during homeostatic scaling in cultured neurons. Elife. 9, 52939(2020).
- Flexner, J. B., Flexner, L. B., Stellar, E. Memory in mice as affected by intracerebral puromycin. Science. 141 (3575), 57-59 (1963).
- Alvarez-Castelao, B., Schanzenbacher, C. T., Langer, J. D., Schuman, E. M. Cell-type-specific metabolic labeling, detection and identification of nascent proteomes in vivo. Nature Protocols. 14 (2), 556-575 (2019).
- Alvarez-Castelao, B., et al. Cell-type-specific metabolic labeling of nascent proteomes in vivo. Nature Biotechnology. 35 (12), 1196-1201 (2017).
- Ngo, J. T., et al. Cell-selective metabolic labeling of proteins. Nature Chemical Biology. 5 (10), 715-717 (2009).
- Mahdavi, A., et al. Engineered aminoacyl-tRNA synthetase for cell-selective analysis of mammalian protein synthesis. Journal of the American Chemical Society. 138 (13), 4278-4281 (2016).
- Yuet, K. P., et al. Cell-specific proteomic analysis in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 112 (9), 2705-2710 (2015).
- Patel, V. J., et al. A comparison of labeling and label-free mass spectrometry-based proteomics approaches. Journal of Proteome Research. 8 (7), 3752-3759 (2009).
- Link, A. J., Vink, M. K., Tirrell, D. A. Preparation of the functionalizable methionine surrogate azidohomoalanine via copper-catalyzed diazo transfer. Nature Protocols. 2 (8), 1879-1883 (2007).
- Landgraf, P., Antileo, E. R., Schuman, E. M., Dieterich, D. C. BONCAT: Metabolic labeling, click chemistry, and affinity purification of newly synthesized proteomes. Methods in Molecular Biology. 1266, Clifton, N.J. 199-215 (2015).
- Kielkopf, C. L., Bauer, W., Urbatsch, I. L. Analysis of proteins by immunoblotting. Cold Spring Harbor Protocols. 2021 (12), (2021).
- Mellacheruvu, D., et al. The CRAPome: a contaminant repository for affinity purification-mass spectrometry data. Nature Methods. 10 (8), 730-736 (2013).
- Szychowski, J., et al. Cleavable biotin probes for labeling of biomolecules via azide-alkyne cycloaddition. Journal of the American Chemical Society. 132 (51), 18351-18360 (2010).
- Goebbels, S., et al. Genetic targeting of principal neurons in neocortex and hippocampus of NEX-Cre mice. Genesis. 44 (12), 611-621 (2006).
- van Geel, R., Pruijn, G. J., van Delft, F. L., Boelens, W. C. Preventing thiol-yne addition improves the specificity of strain-promoted azide-alkyne cycloaddition. Bioconjugate Chemistry. 23 (3), 392-398 (2012).
- O'Fagain, C. Lyophilization of proteins. Methods in Molecular Biology. 244, Clifton, N.J. 309-321 (2004).
- Shevchenko, A., Tomas, H., Havlis, J., Olsen, J. V., Mann, M. In-gel digestion for mass spectrometric characterization of proteins and proteomes. Nature Protocols. 1 (6), 2856-2860 (2006).
- Elinger, D., Gabashvili, A., Levin, Y. Suspension trapping (S-Trap) is compatible with typical protein extraction buffers and detergents for bottom-up proteomics. Journal of Proteome Research. 18 (3), 1441-1445 (2019).
- tom Dieck, S., et al. Direct visualization of newly synthesized target proteins in situ. Nature Methods. 12 (5), 411-414 (2015).
- McShane, E., et al. Kinetic analysis of protein stability reveals age-dependent degradation. Cell. 167 (3), 803-815 (2016).
- Calve, S., Witten, A. J., Ocken, A. R., Kinzer-Ursem, T. L. Incorporation of non-canonical amino acids into the developing murine proteome. Scientific Reports. 6, 32377(2016).
- David, A., et al. Nuclear translation visualized by ribosome-bound nascent chain puromycylation. Journal of Cell Biology. 197 (1), 45-57 (2012).
- Schmidt, E. K., Clavarino, G., Ceppi, M., Pierre, P. SUnSET, a nonradioactive method to monitor protein synthesis. Nature Methods. 6 (4), 275-277 (2009).
- Roux, K. J., Kim, D. I., Burke, B., May, D. G. BioID: A screen for protein-protein interactions. Current Protocols in Protein Science. 91, 11-15 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados