Method Article
Visualiser le cerveau en développement dans le poisson zèbre vivant à l’aide de Brainbow et Time-lapse Confocal Imaging
Dans cet article
Résumé
L’imagerie in vivo est un outil puissant qui peut être utilisé pour étudier les mécanismes cellulaires sous-jacents au développement du système nerveux. Ici, nous décrivons une technique pour utiliser la microscopie confocale en time-lapse pour visualiser un grand nombre de cellules multicolores étiquetées Brainbow en temps réel dans le système nerveux du poisson zèbre en développement.
Résumé
Le développement du système nerveux vertébré nécessite une coordination précise des comportements cellulaires complexes et des interactions. L’utilisation de techniques d’imagerie in vivo à haute résolution peut fournir une fenêtre claire sur ces processus dans l’organisme vivant. Par exemple, la division des cellules et de leur progéniture peut être suivie en temps réel au fur et à mesure que le système nerveux se forme. Ces dernières années, les progrès techniques dans les techniques multicolores ont élargi les types de questions qui peuvent être étudiées. L’approche multicolore Brainbow peut être utilisée non seulement pour distinguer entre les cellules comme, mais aussi pour code couleur plusieurs clones différents de cellules connexes que chacun dérive d’une cellule progénitrice. Cela permet une analyse de lignée multiplexe de nombreux clones différents et leurs comportements simultanément pendant le développement. Ici, nous décrivons une technique pour utiliser la microscopie confocale en time-lapse pour visualiser un grand nombre de cellules multicolores étiquetées Brainbow sur le temps réel dans le système nerveux du poisson zèbre en développement. Ceci est particulièrement utile pour suivre les interactions cellulaires entre les cellules comme, qui sont difficiles à étiqueter différentiellement en utilisant des couleurs traditionnelles axées sur les promoteurs. Notre approche peut être utilisée pour suivre les relations de lignée entre plusieurs clones différents simultanément. Les grands ensembles de données générés à l’aide de cette technique fournissent des informations riches qui peuvent être comparées quantitativement à travers les manipulations génétiques ou pharmacologiques. En fin de compte, les résultats générés peuvent aider à répondre à des questions systématiques sur la façon dont le système nerveux se développe.
Introduction
Dans les premières phases du développement, les pools de cellules progénitrices spécialisées se divisent à plusieurs reprises dans les zones de prolifération, produisant divers éventails de cellules filles. Les cellules nées au cours de cette période de développement se différencieront alors et se déplaceront pour former les organes naissants. Dans le système nerveux, les ancêtres tels que les gliales radiales donnent lieu à des neurones immatures dans les zones ventriculaires. Comme les neurones migrent loin des ventricules et mûrissent, le tissu en expansion forme finalement les structures très complexes du cerveau1,2,3,4,5,6. La coordination entre la division des progéniteurs et la différenciation et la migration des neurones déterminera la taille, la forme et donc la fonction éventuelle du cerveau, ayant un impact direct sur le comportement7,8,9,10. Bien qu’un contrôle serré sur ces processus soit clairement crucial pour le développement normal du cerveau, les mécanismes mondiaux qui régulent ces dynamiques ne sont pas bien compris. Ici, nous décrivons un outil pour étudier le développement du système nerveux à une résolution cellulaire, permettant aux chercheurs de visualiser les cellules progénitrices et les neurones in vivo dans le cerveau du poisson zèbre en développement avec Brainbow et suivre leur comportement au fil du temps via la microscopie confocale en time-lapse11. L’approche peut également être adaptée pour visualiser d’autres parties de l’embryon en développement.
Pour observer et distinguer entre les cellules du cerveau du poisson zèbre en développement, nous avons adapté la technique d’étiquetage cellulaire Brainbow11. Brainbow utilise l’expression combinatoire déterminée au hasard de trois protéines fluorescentes distinctes (FPs) pour étiqueter une population de cellules. Tandis que l’expression par défaut pour l’expression de Brainbow est le dTomato rouge de FP, la recombinaination par l’enzyme Cre recombinase résulte dans l’expression du mcerulean (protéine fluorescente cyane, CFP) ou protéine fluorescente jaune (YFP)12,13. La quantité combinée de chaque FP exprimée dans une cellule lui donne une teinte unique, permettant une distinction visuelle claire des cellules voisines. En outre, quand une cellule progénitrice se divise, chaque cellule de fille héritera de la couleur de sa cellule mère, produisant des clones à code couleur et permettant aux chercheurs de tracer la lignée cellulaire11,14. Bien qu’à l’origine utilisé pour analyser les circuits neuronaux chez la souris12, Brainbow a depuis été exprimé dans une grande variété d’organismes modèles, y compris le poisson zèbre15.
Notre technique s’appuie sur les méthodes précédentes d’étiquetage et d’imagerie multicolores pour imager directement plusieurs clones à code couleur au fil du temps dans les poissons zèbres vivants. En raison de leur transparence optique en tant qu’embryons, le poisson zèbre sont bien adaptés aux expériences d’imagerie16, et des études précédentes ont utilisé Brainbow dans le poisson zèbre pour étudier une variété de tissus, y compris le système nerveux11,15,17,18,19,20,21,22,23,24,25, 26,27. La capacité d’imager directement dans l’organisme vivant, avec leur développement ex-utérus rapide, font du poisson zèbre un modèle précieux de développement des vertébrés. Contrairement au cerveau des mammifères, toute la zone de prolifération du cerveau postérieur du poisson zèbre est facilement disponible pour l’imagerie sans perturber son environnement endogène6. Cela permet de mener des expériences dans l’organisme vivant, plutôt que dans les préparations de tissus in vitro ou fixes. Contrairement aux expériences d’imagerie fixe, les études in vivo permettent une conception longitudinale, produisant des heures de données qui peuvent être analysées pour les modèles, augmentant ainsi la probabilité d’observer des événements relativement rares. Selon la vitesse et la durée des événements d’intérêt, les chercheurs peuvent choisir d’effectuer des expériences d’imagerie courte (1 à 2 h) ou longues (jusqu’à 16 h). En utilisant le promoteur de choc thermique du poisson zèbre 70 (hsp70, hsp), l’expression Brainbow peut être contrôlé temporellement28,29. En outre, l’expression mosaïque induite par ce promoteur est bien adaptée à l’étiquetage et le suivi de nombreux clones11.
La capacité d’identifier visuellement plusieurs clones dans le cerveau vivant est un avantage de cette méthode. D’importantes études antérieures qui ont étudié le rôle des clones dans le développement du système nerveux ont utilisé des vecteurs rétroviraux pour étiqueter une seule cellule progénitrice et sa progéniture à l’aide d’un seul FP ou d’une autre protéine facilement visualisée. Un tel étiquetage permet aux chercheurs d’observer un seul clone au fil du temps, in vitro ou in vivo2,30,31,32,33,34,35,36,37,38. Contrairement aux méthodes de suivi du comportement des cellules au sein d’un clone, les couleurs distinctes de Brainbow permettent aux chercheurs d’observer la dynamique parmi les clones. En outre, en utilisant Brainbow pour étiqueter de nombreux clones dans le cerveau, des données supplémentaires sur le comportement clonal sont recueillies par rapport aux techniques qui étiquetent un seul clone11. Fait important, les approches décrites ici peuvent être élargies pour générer des comparaisons de développement entre les poissons qui ont subi différentes manipulations génétiques ou pharmacologiques18. Dans l’ensemble, ces avantages rendent l’imagerie confocale en vivo du poisson zèbre Brainbow-exprimant idéal pour les chercheurs explorant le développement du système nerveux vertébré, en particulier ceux qui s’intéressent au rôle des clones.
Protocole
Les procédures relatives aux sujets animaux ont été approuvées par le Comité institutionnel de soins et d’utilisation des animaux (IACUC) du Lewis and Clark College.
1. Microinjection d’embryons de poissons zèbres
- Installez le type sauvage, le poisson zèbre adulte dans les cuves d’accouplement sex-ségréguées l’après-midi avant d’effectuer des microinjections39,40.
- Préparer la solution d’ADN le matin des microinjections. Dilute hsp:Zebrabow11 plasmid DNA to a concentration of '10 ng/'l in 0.1 mM KCl, along with 2.5% phenol red and 3.75U Cre recombinase enzyme.
- Effectuez des microinjections de solution d’ADN dans des embryons de poisson zèbre à une cellule dans un rayon de 45 min de fécondation39,41. Injecter environ 4,2 nL de cette solution dans chaque embryon, soit l’équivalent de 42 pg d’ADN plasmide.
REMARQUE: L’étape de microinjection peut être omise si une ligne transgénique, Brainbow-exprimant le poisson zèbre est accouplée à la place, comme Tg (ubi:Zebrabow)15 et Tg (neurod:Zebrabow)18. Effectuer des microinjections peut être avantageux car il permet aux chercheurs de titrer le numéro de copie et la densité d’étiquetage. En outre, une deuxième construction d’ADN qui étiquette ou manipule un produit génétique spécifique peut être injecté aux côtés de Brainbow si désiré (par exemple, une protéine fluorescente rouge foncé qui complète Brainbow18). - Maintenir les embryons injectés dans les boîtes Petri de l’E3 moyen39 dans un incubateur de 28 oC pendant 24 h.
2. Choc thermique pour induire l’expression Brainbow
REMARQUE: Si l’ADN plasmide injecté ou le transgène exprimé n’utilise pas le promoteur hsp70 pour conduire l’expression, l’étape de choc thermique peut être ignorée, et les embryons sains doivent être immédiatement transférés à la phénylthiourea (PTU) à 24 h après la fécondation (hpf).
- À 24 hpf, abattre les embryons morts et déformés du groupe d’embryons injectés. Ensuite, transférer les embryons sains dans un tube de 50 ml, avec jusqu’à 20 embryons/tube.
- Remplir les tubes de 10 ml d’E3. Placer un bouchon sur chaque tube mais ne fermez pas bien.
- Placer la grille à tube contenant les tubes de 50 ml à la verticale dans un bain d’eau de 37 oC. Assurez-vous que le niveau d’eau dans le bain est plus élevé que le niveau de l’E3 dans les tubes et laissez-les de 80 à 90 min.
- Retirer la grille à tube avec les embryons du bain d’eau et retourner à l’incubateur de 28 oC à la verticale. Laisser refroidir jusqu’à 1 h pour que l’E3 refroidisse et que les embryons s’acclimatent progressivement à la température. Ensuite, transférer les embryons dans des plats Petri de 0,2 mM PTU en E3 réchauffés dans l’incubateur pour prévenir la pigmentation des embryons.
CAUTION: PTU est un produit chimique toxique qui doit être manipulé avec soin et éliminé de façon appropriée. Le port de gants est suggéré.
3. Dépistage des embryons pour l’expression Brainbow
- À 2 à 4 h après le choc thermique, examinez les embryons sous un microscope standard de dissection de fluorescence pour l’expression de CFP ou YFP, qui indique la recombinaison réussie de Brainbow (de l’expression par défaut de dTomato).
REMARQUE: Si de multiples options de filtre à fluorescence sont disponibles, le dépistage de la PCP est le moyen le plus efficace d’identifier les poissons bien recombinés. Si les filtres CFP et YFP ne sont pas disponibles, YFP peut également être faiblement visualisé sous les filtres de protéines fluorescentes vertes (GFP) en raison du chevauchement de leurs profils spectrals. - Sélectionnez des embryons avec une expression FP robuste tout au long et transférer dans un plat séparé avec PTU. Imagez les embryons à 1 jour après la fécondation (dpf; 28 hpf ou plus tard) ou maintenez les embryons dans PTU et image à 2 ou 3 dpf.
4. Montage d’embryons pour l’imagerie In Vivo
-
Avant le jour de l’expérience, préparez la chambre d’imagerie et l’outil de manipulation d’embryons.
- Préparer la chambre d’imagerie en supercollant soigneusement un anneau en plastique au centre d’un plat Petri de 60 mm.
- Optionnellement, préparez un manipulateur d’embryons sur mesure pour déplacer les embryons dans le plat et orienter les embryons tout en montant. Construisez le manipulateur en supergluant une petite ligne de pêche en nylon (1/2 à l’extrémité de 6 lb) jusqu’à l’extrémité d’un bâton de coton en bois qui a été coupé à une longueur de 4 euros.
- Si nécessaire (c.-à-d., imagerie à 2 dpf ou plus tôt), avant de monter les embryons déchorionate à l’aide de seringues sous un microscope à dissection.
-
Anesthésiez les embryons de poissons zèbres.
- Remplissez un plat Petri de 60 mm à mi-chemin avec de l’E3 et ajoutez 5 à 6 gouttes de 4 mg/mL MS-222 Tricaine-S à une concentration finale de 0,2 mM. Tourbillonnez pour mélanger.
CAUTION: Tricaine doit être manipulé avec soin et éliminé de façon appropriée. - Transférer les embryons de la PTU à la solution tricaine, en veillant à ce que le moins de PTU soit transféré le plus possible. Alors que les réflexes de poisson doivent cesser après 1-2 min, laissez les embryons en tricaine jusqu’à 10 min pour assurer une anesthésie approfondie pour les expériences d’imagerie en time-lapse.
- Confirmez que les embryons sont correctement anesthésiés. Évaluez le réflexe de sursaut en touchant doucement les queues de l’embryon avec le manipulateur d’embryons. Si un mouvement réflexe est observé, ajoutez une goutte supplémentaire de tricaine au plat aussi loin que possible des embryons et tourbillonnez pour mélanger. Observez la fréquence cardiaque de l’embryon sous une portée de dissection. S’il est anormalement lent, ajoutez des gouttes supplémentaires d’E3 au plat pour réduire la concentration de tricaine.
- Remplissez un plat Petri de 60 mm à mi-chemin avec de l’E3 et ajoutez 5 à 6 gouttes de 4 mg/mL MS-222 Tricaine-S à une concentration finale de 0,2 mM. Tourbillonnez pour mélanger.
-
Monter des embryons anesthésiés dans l’agarose.
- Transférer le poisson au centre de l’anneau en plastique dans la chambre d’imagerie et enlever autant d’excès d’E3 que possible à l’aide d’une pipette de transfert à pointe fine.
- À l’aide d’une pipette de transfert propre, recouvrir le poisson d’une finerach (LMA) à faible fondre (LMA) dans l’E3 entreposée à 42 oC et remplissez l’anneau en plastique entier d’une fine couche d’agarose. Utilisez une pipette de transfert pour tirer doucement les embryons vers le haut dans la pointe de la pipette et de nouveau dans l’agarose sans introduire des bulles d’air.
- Avant que l’agarose durcisse, utilisez rapidement un manipulateur d’embryons pour orienter le poisson de manière appropriée. Si l’imagerie avec un microscope droit, placez les embryons aussi près que possible de la surface supérieure de l’agarose. Placez les embryons parallèlement au fond de la chambre d’imagerie avec la queue droite.
REMARQUE: Pour l’imagerie par le cerveau postérieur, les embryons peuvent être placés dans une vue dorsale ou sagittale. Il faut veiller à ce que les têtes des embryons ne coulent pas pendant que l’agarose est encore liquide. D’autres orientations peuvent être appropriées pour cibler d’autres tissus en développement pour l’imagerie. Pour les microscopes inversés, le montage serait fait différemment, généralement en positionnant le poisson près du fond d’un plat Petri à fond de verre.
- Attendez que l’agarose durcisse, puis remplissez la chambre d’imagerie avec L’E3. Ajouter autant d’E3 que possible pour tenir compte de l’évaporation au cours de l’imagerie.
5. Imagerie confocale time-lapse du poisson-poisson-zèbre en développement
-
Placez la chambre d’imagerie sur le microscope confocal en vue de l’imagerie.
- Placez la chambre d’imagerie avec le poisson et l’E3 au microscope confocal.
- Sélectionnez un objectif avec une ouverture numérique élevée et une longue distance de travail, comme un objectif d’immersion en eau 20x (1,0 NA).
- Trouvez une région avec un étiquetage convenablement dense et lumineux du type de cellule(s) d’intérêt.
REMARQUE: Un stade/appareil à température contrôlée au microscope peut également être utilisé pour réguler la température et l’humidité pendant de longues séances d’imagerie. Cela peut diminuer l’évaporation.
-
Configurez les paramètres d’acquisition pour l’imagerie Brainbow.
- Image de chaque canal FP séquentiellement. Si vous utilisez un logiciel commercial (par exemple, le logiciel Zen), cela se fait en préparant trois « pistes ». Utilisez un laser Argon pour exciter la PCP à 458 nm et YFP à 514 nm. Utilisez un laser DPSS 561 nm pour exciter dTomato. Collectez les émissions entre 463-509 nm pour CFP, 519-555 nm pour YFP, et 566-691 nm pour dTomato.
- Pour l’affichage à l’écran, codeR CFP comme bleu, YFP comme jaune, et dTomato comme rouge.
REMARQUE: Ces paramètres varient en fonction des lignes laser et autres caractéristiques disponibles au microscope confocal. Pour augmenter la vitesse de l’imagerie, les canaux CFP et dTomato peuvent être photographiés simultanément sans saignement appréciable. Si un FP séparé est également photographié, comme un FP rouge lointain, cela peut être photographié séquentiellement ou simultanément avec YFP. - Configurez les paramètres d’imagerie générale pour prendre des images 16 bits avec une résolution d’au moins 1024 x 1024 et une moyenne de deux fois. Ajuster le zoom en fonction de la région et le type d’intérêt cellulaire (c.-à-d., pour l’imagerie postérieure avec un objectif 20x le zoom variera de 1,0 à 2,5). Maximisez le champ de vision pour permettre la croissance du poisson au fil du temps.
- Affichage d’une piste à la fois (et éteindre d’autres lasers pour empêcher le photobleaching), optimiser les paramètres d’acquisition pour chaque FP individuellement (p. ex., puissance laser, taille de trou d’épingle, gain de tube photomultiplier, etc.).
- Sélectionnez la gamme Z-stack à l’image.
REMARQUE: Pour de longues séances d’imagerie (2 h), prenez en considération que le poisson continuera à croître tout au long de l’imagerie, de ce fait que le champ XY et la gamme Z-stack devraient en tenir compte avec de la place supplémentaire. Si l’imagerie du cerveau arrière, inclure l’espace dans le champ pour le tissu de se déplacer rostrally pendant la période d’imagerie. L’espace doit également être inclus pour la croissance dans la dimension Z. Inclure dans l’image environ 10 à 20 m au-dessus de la région d’intérêt, que le poisson soit positionné pour une vue sagittale ou dorsale.
-
Configurez des paramètres pour l’imagerie par time-lapse.
- Sélectionnez un intervalle de temps. La longueur d’intervalle varie entre 10-30 min pour suivre les événements mitotiques et apoptotiques dans le cerveau postérieur en développement. La longueur de l’intervalle varie en fonction de la vitesse des événements d’intérêt et du temps total qu’il faut pour capturer une seule pile Z.
- Sélectionnez la durée de la séance d’imagerie. Les séances d’imagerie en accéléré se produisent généralement pendant la nuit et peuvent durer au moins 16 h.
-
Dirigez l’expérience.
REMARQUE : Le poisson peut être euthanasié immédiatement après l’imagerie ou soigneusement disséqué de l’agarose à l’aide de seringues et retourné à l’E3 pour récupérer. Les poissons récupérés peuvent ensuite être photographiés à nouveau à un moment ultérieur, bien que la position et la profondeur cellulaires se déplacent au cours de cette période en raison de la croissance globale.
6. Gestion des fichiers time-lapse
-
Importer le fichier d’image dans le logiciel d’analyse.
- Si vous utilisez un logiciel Zen, enregistrez le fichier en format .czi une fois l’acquisition d’images terminée. Assurez-vous d’enregistrer des données brutes dans un format compatible avec Fidji42 et/ou d’autres logiciels.
- Importer dans le logiciel Fidji à l’aide de BioFormats Importer.
REMARQUE: Les fichiers time-lapse seront importants et peuvent nécessiter beaucoup de temps et de mémoire pour ouvrir pour analyse. Ainsi, il est utile d’être stratégique dans la façon dont ils sont sauvés et ouverts. Il peut être utile d’enregistrer une version de moindre qualité qui peut être ouverte plus facilement à la recherche d’événements d’intérêt à l’œil.
- Si vous utilisez Fidji, créez des sous-ensembles plus petits et plus gérables de fichiers time-lapse. Générer des sous-ensembles soit par le temps (p. ex., en visualisant la pile Z complète au premier moment) ou par profondeur (p. ex., en visualisant les 50 premières sections Z à chaque moment) en cliquant sur Image( Piles Outils Faire Substack.
7. Analyse quantitative des couleurs clonales des images Brainbow
- Mesurer les valeurs d’intensité moyennes rouge, verte et bleue (RGB) pour les cellules d’intérêt aux Fidji.
REMARQUE : Ces instructions sont spécifiques à l’analyse d’image aux Fidji. Cependant, les chercheurs peuvent préférer obtenir des valeurs d’intensité moyenne RGB dans un logiciel alternatif.- Assurez-vous que le fichier est correctement recadré de sorte qu’il n’y a pas d’espace noir sur les bords de l’image. L’espace noir permettra d’abaisser artificiellement les mesures d’intensité minimales nécessaires à l’analyse. Si le fichier doit être recadré, sélectionnez le bouton de sélection Rectangle et dessinez une zone d’intérêt (ROI) autour du champ de vision, à l’exclusion de tout espace noir. Cliquez sur l’image(fr) Récolte.
- Définissez l’outil de mesure pour prendre des mesures de valeur moyenne, minimale et maximale de la valeur grise. Cliquez sur Analysede l’analyse Définir les mesures. Assurez-vous que les cases à cocher pour la valeur de min et max Gray et la valeur de gris moyen sont tous deux sélectionnés.
- Affichage des fichiers de données brutes ou subsetted Z-stacks aux Fidji, trouver des cellules d’intérêt pour l’analyse des couleurs clonales. Dans le cerveau arrière, les clones putatifs peuvent être identifiés visuellement par leur teinte commune ainsi que leur orientation radiale. Ne sélectionnez pas les cellules qui sont en contact étroit avec, et donc difficile à résoudre à partir de, cellules voisines d’une autre couleur. Il peut être difficile de quantifier leur teinte.
- Trouvez le plan Z central de la cellule d’intérêt à l’aide de la barre de Z. Cliquez à droite sur le bouton de sélection ovale et sélectionnez le bouton de sélection elliptique. D’autres outils de sélection seront appropriés pour différents types de cellules. Dessinez un retour sur investissement elliptique autour du centre de la cellule.
- À l’aide de la barre de C,sélectionnez le canal rouge (dTomato). Prenez la mesure d’intensité moyenne pour ce canal en sélectionnant Analyze. Mesure. Répétez l’opération avec le même plan de retour sur investissement et plan focal pour les canaux vert (YFP) et bleu (CFP) et économisez toutes les valeurs d’intensité moyenne.
- Cliquez n’importe où sur l’image pour supprimer le retour sur investissement elliptique dans la cellule d’intérêt.
- Pour mesurer les niveaux de fond pour la normalisation, utilisez la barre de défilement C et sélectionnez le canal rouge (dTomato). Prenez la mesure d’intensité minimale pour l’ensemble du champ dans ce canal en sélectionnant Analyze. Mesure. Répétez l’opération avec le même plan focal pour les canaux vert (YFP) et bleu (CFP) et économisez toutes les valeurs d’intensité minimales.
- Calculez les poids relatifs des canaux RGB pour les cellules d’intérêt.
REMARQUE : Le calcul des poids relatifs des canaux RGB à partir de ces valeurs d’intensité peut être effectué dans une variété de logiciels, tels que Microsoft Excel ou R43.- Normaliser la mesure d’intensité moyenne du retour sur investissement de la cellule pour chaque canal en soustrayant l’intensité minimale de l’ensemble du champ dans ce canal et le plan focal.
- Résumez les valeurs d’intensité moyenne normalisées de RGB de chaque canal pour trouver l’intensité totale normalisée de RGB pour la cellule.
- Calculez le poids relatif du canal pour chaque canal en divisant la valeur d’intensité moyenne normalisée pour ce canal par l’intensité totale normalisée de RGB.
- Afficher les poids relatifs du canal RGB comme parcelles ternaires utilisant le paquet sternaire pour R43,44. Les parcelles ternaires sont utiles pour visualiser le regroupement de couleurs similaires dans l’espace RGB 3D.
- Calculez le coefficient de propagation de couleur pour quantifier la différence entre les couleurs cellulaires.
- Calculez la distance Euclidean entre les deux couleurs dans l’espace RGB 3D à l’aide de l’équation suivante :
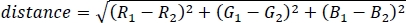
où R, G et B sont les poids relatifs du canal RGB calculés en 7,2. - Pour faciliter la compréhension, normaliser la distance de couleur en divisant par le 2, la distance maximale possible entre les couleurs, pour obtenir un coefficient de diffusion de couleur entre 0 et 1, où 0 indique exactement la même couleur et 1 indique des couleurs complètement différentes (par exemple, rouge pur et bleu pur).
- Calculez la distance Euclidean entre les deux couleurs dans l’espace RGB 3D à l’aide de l’équation suivante :
Résultats
Cette section illustre des exemples de résultats qui peuvent être obtenus à l’aide de l’approche d’imagerie multicolore in vivo décrite ici. Nous montrons que Brainbow clones codés en couleur de cellules dans la zone ventriculaire proliférée de l’arrière-poisson zèbre en développement14 (figure 1).
Typiquement, lorsque les cellules étiquetées Brainbow ont été disposées le long d’une fibre radiale particulière, elles partageaient la même couleur(figure 1D), qui pourrait être quantifiée comme les poids relatifs du canal RGB(figure 1E). Ceci suggère que ces groupes radiaux étaient des clones des cellules de division et que leur couleur semblable pourrait être employée pour les identifier comme étant clonally related, comme observé dans des études précédentes dans le poisson zèbre, la souris, et le poussin14,15. Lorsque la densité d’étiquetage Brainbow est relativement clairsemée, ce codage des couleurs peut être utilisé pour visualiser clairement et suivre de nombreux clones radiaux distincts dans les régions proliférales du cerveau vertébré vivant.
Une analyse quantitative des couleurs a montré que les cellules de fille exprimaient la même couleur que leur cellule mère (progéniteur)(figure 1F), mais que les grappes radiales voisines de cellules peuvent être distinguées les unes des autres11 (figure 1E). Cela signifie que de nombreux clones de cellules apparentées peuvent être suivis simultanément pendant des heures in vivo(figure 2), permettant une analyse et une comparaison de lignées multiplexes. Quantification de l’expression des couleurs dans les clones à 2 dpf, puis à nouveau à 3 dpf (figure 2A-B) a montré que l’expression Brainbow est également resté relativement constante de 2-3 jours in vivo11. Les cellules individuelles peuvent ainsi être identifiées et suivies en temps réel pendant des périodes relativement longues pendant le développement.
L’imagerie en time-lapse a révélé de nombreuses cellules subissant la migration nucléaire interkinétique et la division cellulaire au cours de la période de 1 à 2 dpf(figure 2C-F)11, permettant l’étude du cycle cellulaire. En utilisant un intervalle de time-lapse de 30 min, nous n’avons pas toujours capturé la figure mitotique pendant la division cellulaire. Ceci a introduit une erreur potentielle allant jusqu’à 30 min au temps assigné de division cellulaire. De plus, nous avons observé certains clones qui semblent contenir deux cellules progénitrices (p. ex., figure 2F). Dans ces contraintes, la longueur du cycle cellulaire peut être mesurée. En suivant les progéniteurs individuels étiquetés Brainbow au fil du temps, nous avons calculé un cycle cellulaire moyen de 8,4 à 1,5 h, comparable aux mesures précédentes chez le poisson zèbre1,45,46. En outre, en utilisant la technique d’imagerie Brainbow time-lapse, nous avons également pu observer les cellules individuelles subissant les changements morphologiques stéréotypés associés à l’apoptose (figure 3)11, tels que le blebbing de membrane et la fragmentation cellulaire47,48.
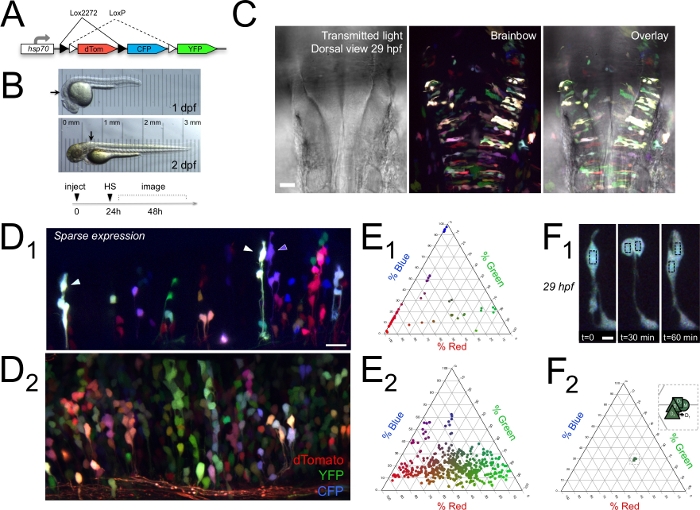
Figure 1 : Brainbow étiquetés groupes clonally liés de cellules de division dans le fond de l’arrière-poisson zèbre en développement. (A) Schéma de hsp:Zebrabow (Brainbow) ADN transitoirement exprimé aux clones de code de couleur. (B) In vivo a transmis des images lumineuses de poissons zèbres en développement à 1 et 2 dpf; les flèches indiquent la région générale de l’arrière-cour cible pour l’imagerie. La translucidité des embryons est démontrée par un micromètre placé sous chaque poisson. Chronologie expérimentale montrant les injections à un stade cellulaire, le choc thermique (HS) à 24 hpf, et l’imagerie de 1-3 dpf. (C) Dorsal vue de 29 hpf zèbres hindbrain étiqueté avec Brainbow. Le canal de lumière transmis a montré la morphologie du cerveau arrière et du ventricule recouverts d’une projection d’intensité maximale de clones étiquetés Brainbow représentant 165 m. Rostral est en place. (D) In vivo Brainbow expression dans le cerveau postérieur, montré dans les projections d’intensité maximale représentant 41 m dans 51 poissons zèbres de fèbres peu étiquetés (D1), et 81 m dans 63,5 hpf zèbres (D2). Dans les deux panneaux, le dorsal est en place et rostral est à gauche. (E) La couleur des cellules en D1 et D2 a été quantifiée comme poids relatifs de canal dans les parcelles ternaires correspondantes (n - 54 cellules E1; 461 cellules E2). (F) La couleur de cellules est restée relativement constante dans les cellules de fille suivant la division cellulaire. Série de points de temps montrant des événements mitotiques in vivo dans le cerveau postérieur de 29 poissons zèbres hpf Brainbow-étiquetés plus de 1 h (F1, projections d’intensité maximale, 30 'm de profondeur). La couleur des cellules mères et filles, indiquée par les boîtes noires en F1, est représentée comme poids relatifs de canal dans la parcelle ternaire dans F2. L’encart montre un zoom de la même intrigue pour montrer des couleurs cellulaires étroitement regroupées (M est mère; D1 et D2 sont des filles à 30 min/carrés, et 60 min/triangles). Barres d’échelle de 30 m (C), 20 m (D1), 16 m (D2)et 5 m (F1). Dans C, D et F, dTomato est codé comme rouge, YFP est codé comme vert, et CFP est codé comme bleu. Images réimprimées avec la permission de Brockway et coll.11. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 2 : L’imagerie in vivo time-lapse combinée à Brainbow color-coding a distingué plusieurs clones voisins de cellules subissant la migration et la division interkinétiques nucléaires. (A) Clusters radiaux à code couleur étiquetés dans le cerveau arrière d’un poisson exprimant Brainbow à 2 dpf (55 hpf; A1) et 3 dpf (71 hpf; A2); projections d’intensité maximale représentant respectivement 62 m et 47 m. Trois amas radiaux codés en couleur sont identifiés avec des pointes de flèche à chaque point de temps. (B) La couleur de toutes les cellules étiquetées Brainbow dans A1 et A2 a été quantifiée comme poids relatifs de canal dans les parcelles ternaires correspondantes dans B1 et B2 (n - 106 cellules, 119 cellules). (C) Le cerveau postérieur du poisson zèbre à 35 hpf étiqueté avec Brainbow (projection d’intensité maximale montrant 24 m); l’encart indiqué avec la boîte blanche pointillée est le premier point de temps affiché dans D. (D) Série de points de temps pris à 30 minutes d’intervalle dans le cerveau arrière de C. Une période de 9,5 h est montrée. Les cellules étiquetées ont subi la migration nucléaire interkinétique ; pointes de flèche blanches indiquent une cellule à la surface apicale. L’arrondissement du soma cellulaire à la surface apicale et une augmentation correspondante du nombre de clones (p. ex., globule rouge à 5,5 h, puis 8 h) ont été considérés comme un événement mitotique. (E) Dorsolateral view of zebrafish hindbrain at 30.5 hpf labeled with Brainbow, where the white dashed-and-dotted line shows the approxmate boundary of the head, and the white dashed box indicates the inset shown in the first time point of F. (F) Series of time points taken at 30 min intervals in the hindbrain from E. Sur une période de 1,5 h, deux cellules bleues indiquées par les pointes blanches de flèche ont pu être observées se déplaçant à la surface apicale et subissant la mitose, suggérant un clone contenant deux cellules progénitrices. Barre d’échelle de 20 m (A), 25 m (C) et 20 m (F). Dans A-D, le dorsal est en place et rostral est à gauche. En E et F, le rostral est à gauche. Dans toutes les images, dTomato est codé comme rouge, YFP est codé comme vert, et CFP est codé comme bleu. Images réimprimées avec la permission de Brockway et coll.11. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 3 : L’imagerie Brainbow en accéléré montre que les cellules de la zone ventriculaire subissent la mort cellulaire in vivo. (A) Le cerveau postérieur du poisson zèbre à 31 hpf étiqueté avec Brainbow (projection d’intensité maximale représentant 36 m). Boîte pointillée blanche indique l’encart montré dans le premier point de temps dans B. (B) Série de points de temps montrant le cerveau arrière dans A à 30 minutes d’intervalle. La pointe de flèche blanche dans le premier panneau montre une cellule de lavande qui a subi l’apoptose comme indiqué dans les panneaux suivants, indiqué par la fragmentation de cellules suivies du dégagement progressif des corps apoptotiques. Les cellules étiquetées voisines semblaient saines tout au long. Dorsal est en place et rostral est à gauche. Barre d’échelle de 20 m (B). Dans toutes les images, dTomato est codé comme rouge, YFP est codé comme vert, et CFP est codé comme bleu. Images réimprimées avec la permission de Brockway et coll.11. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
Discussion
Ce protocole décrit une méthode pour visualiser les clones de cellules progénitrices et de neurones dans le cerveau postérieur du poisson zèbre en développement et les suivre in vivo à l’aide de Brainbow et de microscopie confocale en time-lapse11. Le principal avantage de ce protocole par rapport aux études in vitro ou ex vivo est la capacité d’observer directement la zone de prolifération du cerveau vertébré dans son milieu naturel au fil du temps. Cette technique s’appuie sur des études antérieures qui ont étiqueté un seul clone à l’aide de vecteurs rétroviraux. En revanche, l’utilisation du hsp:Zebrabow construire des codes de couleur de nombreux clones simultanément, permettant le traçage de lignée multiplexe et un accent sur la dynamique entre les clones11. Ce protocole peut être modifié pour se concentrer sur le développement dans d’autres systèmes ou types de cellules. Par exemple, différentes constructions Brainbow peuvent être exprimées dans les embryons de poissons zèbres. Utilisation du promoteur de hsp70 de poisson zèbre dans hsp: Zebrabow se traduira par l’étiquetage de mosaïque d’une variété de types de cellules suivant le choc thermique, y compris les progéniteurs neuronaux et les neurones11, et donne aux chercheurs le contrôle temporel sur l’expressiongénique 29. Il convient de noter que l’utilisation du promoteur de choc thermique étiquette systématiquement les clones à code couleur, tandis que d’autres promoteurs ne peuvent pas clairement délimiter la lignée cellulaire de cette manière. Lorsque l’identité clonale n’est pas un facteur d’intérêt, les constructions utilisant d’autres promoteurs peuvent être utilisées pour étiqueter des types de cellules spécifiques. Par exemple, le promoteur neuroD peut être utilisé pour étiqueter les neurones immatures18,49. En outre, au lieu d’effectuer des microinjections, les chercheurs peuvent choisir d’utiliser transgéniquesFish zébré Brainbow, y compris des lignes telles que Tg (ubi:Zebrabow)15 et Tg (neurod:Zebrabow)18. Dans ce cas, traverse les lignes exprimant Cre recombinase, tels que Tg (hsp:Crea134)15, produire des embryons non injects qui sont recueillis et maintenus de la même manière. Bien que ce protocole soit conçu pour imager les poissons entre 1-3 dpf, afin de coïncider avec la période majeure de neurogenèse développementale50, le poisson zèbre peut être maintenu plus longtemps selon le stade de développement de l’intérêt. Cependant, le choc thermique pour induire l’expression Brainbow avant 24 hpf peut être plus préjudiciable aux embryons51. Selon l’âge et le tissu d’intérêt, différentes stratégies de montage seront appropriées pour avoir le tissu ciblé orienté correctement.
Il y a un certain nombre d’étapes qui peuvent nécessiter le dépannage, en particulier si des modifications sont apportées au protocole. La concentration d’ADN, la taille du bolus injecté et la quantité totale d’ADN délivrée par embryon peuvent toutes être ajustées si l’expression de Brainbow est faible ou clairsemée ou si les embryons ne semblent pas en bonne santé. En outre, la longueur et le moment de l’étape de choc thermique peuvent être modifiés si l’expression souhaitée n’est pas atteinte. Les paramètres d’acquisition utilisés dans l’imagerie varieront en fonction des lignes laser et des filtres disponibles ainsi que de la luminosité de l’expression, du montage et du type d’analyse souhaitée. En outre, les paramètres time-lapse doivent être ajustés en fonction de la vitesse des événements d’intérêt et du temps nécessaire pour acquérir une seule pile Z. L’une des étapes les plus critiques du protocole est le montage approprié du poisson dans l’agarose : si l’angle du poisson est inapproprié ou si le poisson est couvert par trop d’agarose, une imagerie optimale ne sera pas possible. L’optimisation de cette technique peut prendre une certaine pratique. En outre, il est souvent utile de monter plusieurs poissons avant l’imagerie car il peut être difficile de déterminer si le montage est approprié avant de voir l’expression FP sur le microscope confocal lui-même. Une autre étape critique est le dépistage et la sélection des poissons spécifiques à imaginer. Si l’exécution des microinjections, la luminosité, la densité d’étiquetage, et le nombre de copie Brainbow peuvent tous varier considérablement entre les poissons. Les images prises dans les poissons avec l’étiquetage faible, l’étiquetage trop dense, ou un nombre de copie faible résultant en peu de couleurs peuvent être plus difficiles à analyser.
Avec ce protocole, nous avons été en mesure d’image en permanence vivant poissons zèbres jusqu’à 16 h11. Cette durée peut être limitée par l’évaporation E3 de la chambre d’imagerie, la croissance du poisson hors du plan d’imagerie, la mort ou le mouvement du poisson, ou les contraintes de l’utilisateur sur le temps de microscope confocal. L’évaporation pourrait être diminuée par l’utilisation d’un accessoire à température contrôlée au stade du microscope. Il convient de noter que les ensembles de données 5D générés à l’aide de cette approche ont tendance à être de grands fichiers qui nécessitent un espace de disque dur important (par exemple, jusqu’à 100 Go pour un time-lapse de nuit) et une puissance de calcul adéquate à analyser.
Ce protocole guide les chercheurs à visualiser les clones à code couleur au sein d’une population d’ancêtres neuronaux neuronaux neuronaux neuronaux neuronaux et de filles dans le cerveau du poisson zèbre en développement et de suivre leur dynamique au fil du temps. Une expansion possible est la combinaison de Brainbow time-lapse imagerie avec le marquage FP far-rouge pour évaluer les rôles de gènes spécifiques dans le développement18. De cette façon, les clones exprimant des gènes manipulés peuvent être visualisés dans une couleur distincte, suivis au fil du temps, et comparés aux clones voisins non manipulés et étiquetés Par Brainbow. L’imagerie multicolore in vivo peut être utilisée pour répondre à d’importantes questions mécanistes sur la façon dont le système nerveux se forme et fonctionne.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Y. A. Pan, J. Livet et Z. Tobias pour leurs contributions techniques et intellectuelles. Ce travail a été soutenu par la National Science Foundation (Award 1553764) et le M.J. Murdock Charitable Trust.
matériels
| Name | Company | Catalog Number | Comments |
| 1.5 mL transfer pipette (fine tip) | Globe Scientific, Inc. | 134020 | |
| 1-phenyl-2-thiourea (PTU) | Alfa Aesar | L06690 | Diluted to 0.2 mM in E3 to prevent embryo pigmentation |
| 50 mL conical tubes | Corning | 352070 | For heat shocking embryos |
| 6 lb nylon fishing line | SecureLine | NMT250 | For making embryo manipulators |
| 7.5 mL transfer pipet | Globe Scientific, Inc. | 135010 | |
| CaCl2 | Sigma | C3881 | For E3 |
| Cotton swabs | Puritan | 867-WC NO GLUE | For making embryo manipulators |
| Cre recombinase | New England Biolabs | M0298M | |
| Digital dry bath | Genemate | 490016-616 | Used to store LMA at 42 °C |
| Epifluorescence dissection scope | |||
| Glass capillary tubes | World Precision Instruments | TW100F-4 | |
| Incubator | Forma Scientific | 3158 | To maintain embryos at 28 °C |
| Injection plate molds | Adaptive Science Tools | TU-1 | |
| Isotemp water bath | Fisher Scientific | 2320 | For heat shocking embryos |
| KCl | AMRESCO | 0395 | For E3 and for DNA solution for injections |
| Laser-scanning confocal microscope | Zeiss | LSM710 | |
| LE agarose | Genemate | E3120 | To create agarose injection plates |
| Low-melt agarose (LMA) | AMRESCO | J234 | |
| Mating tanks | Aquaneering, Inc. | ZHCT100 | |
| Methylene blue | Sigma | M9140 | For E3 |
| MgSO4 | Sigma | 9397 | For E3 |
| Micromanipulator | World Precision Instruments | M3301 | |
| Micropipette Puller | Sutter Instrument Co. | P-97 | |
| MS-222 Tricaine-S | Western Chemical, Inc. | Stock made at 4 mg/mL in reverse osmosis (RO) water, then added dropwise to E3 to final concentration of 0.2 mM to anesthetize embryos | |
| NaCl | J.T. Baker | 4058-01 | For E3 |
| Petri dishes (90 mm, 60 mm) | Genesee Scientific | 32-107G | To house embryos and create imaging chamber (60 mm) |
| Phenol red | Sigma | P0290 | |
| Soft stitch ring markers | Clover Needlecraft, Inc. | 354 | For creating imaging chamber with Petri dish |
| Super glue (Ultra gel control) | Loctite | 1363589 | For making embryo manipulators |
| Syringe needles | Beckton Dickinson | BD329412 | For dechorionating embryos |
Références
- Lyons, D. A., Guy, A. T., Clarke, J. D. W. Monitoring neural progenitor fate through multiple rounds of division in an intact vertebrate brain. Development. 130, 3427-3436 (2003).
- Noctor, S. C., Flint, A. C., Weissman, T. A., Dammerman, R. S., Kriegstein, A. R. Neurons derived from radial glial cells establish radial units in neocortex. Nature. 409 (6821), 714-720 (2001).
- Kriegstein, A., Alvarez-Buylla, A. The glial nature of embryonic and adult neural stem cells. Annual Review of Neuroscience. 32, 149-184 (2009).
- Florio, M., Huttner, W. B. Neural progenitors, neurogenesis and the evolution of the neocortex. Development. 141, 2182-2194 (2014).
- Mcconnell, S. K. Constructing the Cerebral Cortex: Neurogenesis and Fate Determination Review. Neuron. 15, 761-768 (1995).
- Schmidt, R., Strähle, U., Scholpp, S. Neurogenesis in zebrafish - from embryo to adult. Neural Development. 8, 3(2013).
- Chenn, A., Walsh, C. Regulation of Cerebral Cortical Size by Control of Cell Cycle Exit in Neural Precursors. Science. 297 (5580), 365-369 (2002).
- Lui, J. H., Hansen, D. V., Kriegstein, A. R. Development and Evolution of the Human Neocortex. Cell. 146 (1), 18-36 (2011).
- Nonaka-Kinoshita, M., et al. Regulation of cerebral cortex size and folding by expansion of basal progenitors. The EMBO Journal. 32, 1817-1828 (2013).
- Homem, C. C. F., Repic, M., Knoblich, J. A. Proliferation control in neural stem and progenitor cells. Nature Reviews Neuroscience. 16 (11), 647-659 (2015).
- Brockway, N. L., et al. Multicolor lineage tracing using in vivo time-lapse imaging reveals coordinated death of clonally related cells in the developing vertebrate brain. Developmental Biology. 453 (2), 130-140 (2019).
- Livet, J., et al. Transgenic strategies for combinatorial expression of fluorescent proteins in the nervous system. Nature. 450 (7166), 56-62 (2007).
- Weissman, T. A., Pan, Y. A. Brainbow: New resources and emerging biological applications for multicolor genetic labeling and analysis. Genetics. 199 (2), 293-306 (2015).
- Loulier, K., et al. Multiplex Cell and Lineage Tracking with Combinatorial Labels. Neuron. 81 (3), 505-520 (2014).
- Pan, Y. A., et al. Zebrabow: multispectral cell labeling for cell tracing and lineage analysis in zebrafish. Development. 140, 2835-2846 (2013).
- Ko, S. K., Chen, X., Yoon, J., Shin, I. Zebrafish as a good vertebrate model for molecular imaging using fluorescent probes. Chemical Society Reviews. 40, 2120(2011).
- Kesavan, G., Hammer, J., Hans, S., Brand, M. Targeted knock-in of CreERT2 in zebrafish using CRISPR/Cas9. Cell and Tissue Research. 372, 41-50 (2018).
- Cook, Z. T., et al. Combining near-infrared fluorescence with Brainbow to visualize expression of specific genes within a multicolor context. Molecular Biology of the Cell. 30 (4), 491-505 (2019).
- Kuwata, M., Nikaido, M., Hatta, K. Local heat-shock mediated multi-color labeling visualizing behaviors of enteric neural crest cells associated with division and neurogenesis in zebrafish gut. Developmental Dynamics. 248, 437-448 (2019).
- Kinkhabwala, A., et al. A structural and functional ground plan for neurons in the hindbrain of zebrafish. Proceedings of the National Academy of Sciences. 108 (3), 1164-1169 (2011).
- Heap, L. A., Goh, C. C., Kassahn, K. S., Scott, E. K. Cerebellar Output in Zebrafish: An Analysis of Spatial Patterns and Topography in Eurydendroid Cell Projections. Frontiers in Neural Circuits. 7, 53(2013).
- Robles, E., Filosa, A., Baier, H. Precise Lamination of Retinal Axons Generates Multiple Parallel Input Pathways in the Tectum. The Journal of Neuroscience. 33 (11), 5027-5039 (2013).
- Dirian, L., et al. Spatial Regionalization and Heterochrony in the Formation of Adult Pallial Neural Stem Cells. Developmental Cell. 30, 123-136 (2014).
- Chen, X. A., et al. Behavioral/Cognitive QRFP and Its Receptors Regulate Locomotor Activity and Sleep in Zebrafish. The Journal of Neuroscience. 36 (6), 1823-1840 (2016).
- Albadri, S., De Santis, F., Di Donato, V., Del Bene, F. CRISPR/Cas9-Mediated Knockin and Knockout in Zebrafish. Genome Editing in Neurosciences. Jaenisch, R., Zhang, F., Gage, F. , Springer. Cham, Switzerland. 41-49 (2017).
- Furlan, G., et al. Life-Long Neurogenic Activity of Individual Neural Stem Cells and Continuous Growth Establish an Outside-In Architecture in the Teleost Pallium. Current Biology. 27, 3288-3301 (2017).
- Herget, U., Arturo Gutierrez-Triana, J., Salazar Thula, O., Knerr, B., Ryu, S. Single-cell reconstruction of oxytocinergic neurons reveals separate hypophysiotropic and encephalotropic subtypes in larval zebrafish Brainbow-guided morphology of oxytocinergic cells. eNeuro. 4 (1), (2017).
- Halloran, M. C., et al. Laser-targeted gene expression. Development. 127, 1953-1960 (2000).
- Shoji, W., Sato-Maeda, M. Application of heat shock promoter in transgenic zebrafish. Development, Growth & Differentiation. 50, 401-406 (2008).
- Luskin, M. B., Pearlman, A. L., Sanes, J. R. Cell lineage in the cerebral cortex of the mouse studied in vivo and in vitro with a Recombinant Retrovirus. Neuron. 1 (8), 635-647 (1988).
- Price, J., Thurlow, L. Cell lineage in the rat cerebral cortex: a study using retroviral-mediated gene transfer. Development. 104, 473-482 (1988).
- Walsh, C., Cepko, C. L. Widespread dispersion of neuronal clones across functional regions of the cerebral cortex. Science. 255 (5043), 434-441 (1992).
- Walsh, C., Cepko, C. L. Clonal dispersion in proliferative layers of developing cerebral cortex. Nature. 362 (6421), 632-635 (1993).
- Cai, L., Hayes, N. L., Nowakowski, R. S. Synchrony of Clonal Cell Proliferation and Contiguity of Clonally Related Cells: Production of Mosaicism in the Ventricular Zone of Developing Mouse Neocortex. The Journal of Neuroscience. 17 (6), 2088-2100 (1997).
- Reznikov, K., Acklin, S. E., Van Der Kooy, D. Clonal Heterogeneity in the Early Embryonic Rodent Cortical Germinal Zone and the Separation of Subventricular From Ventricular Zone Lineages. Developmental Dynamics. 210, 328-343 (1997).
- Qian, X., Goderie, S., Shen, Q., Stern, J., Temple, S. Intrinsic programs of patterned cell lineages in isolated vertebrate CNS ventricular zone cells. Development. 125, 3143-3152 (1998).
- McCarthy, M., Turnbull, D. H., Walsh, C. A., Fishell, G. Telencephalic Neural Progenitors Appear to Be Restricted to Regional and Glial Fates before the Onset of Neurogenesis. The Journal of Neuroscience. 21 (17), 6772-6781 (2001).
- Yu, Y. C., Bultje, R. S., Wang, X., Shi, S. H. Specific synapses develop preferentially among sister excitatory neurons in the neocortex. Nature. 458 (7237), 501-504 (2009).
- Nusslein-Volhard, C., Dahm, R. Zebrafish. , Oxford University Press. Oxford, UK. (2002).
- Nasiadka, A., Clark, M. D. Zebrafish Breeding in the Laboratory Environment. ILAR Journal. 53 (2), 161-168 (2012).
- Rosen, J. N., Sweeney, M. F., Mably, J. D. Microinjection of Zebrafish Embryos to Analyze Gene Function. Journal of Visualized Experiments. 25, e1115(2009).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- R Core Team. R: A language and environment for statistical computing. , (2017).
- Smith, M. R. Ternary: an R package to generate ternary plots. , (2017).
- Baye, L. M., Link, B. A. Development/Plasticity/Repair Interkinetic Nuclear Migration and the Selection of Neurogenic Cell Divisions during Vertebrate Retinogenesis. The Journal of Neuroscience. 27 (38), 10143-10152 (2007).
- Leung, L., Klopper, A. V., Grill, S. W., Harris, W. A., Norden, C. Apical migration of nuclei during G2 is a prerequisite for all nuclear motion in zebrafish neuroepithelia. Development. 139, 2635(2012).
- Kerr, J. F. R., Wyllie, A. H., Curriet, A. R. Apoptosis: A Basic Biological Phenomenon with Wide-ranging Implications in Tissue Kinetics. British Journal of Cancer. 26, 239(1972).
- Cole, L. K., Ross, L. S. Apoptosis in the Developing Zebrafish Embryo. Developmental Biology. 240, 123-142 (2001).
- Liao, J., He, J., Yan, T., Korzh, V., Gong, Z. A Class of NeuroD-Related Basic Helix-Loop-Helix Transcription Factors Expressed in Developing Central Nervous System in Zebrafish. DNA and Cell Biology. 18 (4), 333-344 (1999).
- Trevarrow, B., Marks, D. L., Kimmel, C. B. Organization of hindbrain segments in the zebrafish embryo. Neuron. 4 (5), 669-679 (1990).
- Yabu, T., Todoriki, S., Yamashita, M. Stress-induced apoptosis by heat shock, UV and gamma-ray irradiation in zebrafish embryos detected by increased caspase activity and whole-mount TUNEL staining. Fisheries Science. 67, 333-340 (2001).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon