Method Article
Visualización del cerebro en desarrollo en el pez cebra vivo mediante imágenes confocales de barco iris y lapso de tiempo
En este artículo
Resumen
La imagen in vivo es una poderosa herramienta que se puede utilizar para investigar los mecanismos celulares subyacentes al desarrollo del sistema nervioso. Aquí describimos una técnica para el uso de la microscopía confocal de lapso de tiempo para visualizar un gran número de células etiquetadas en Brainbow multicolor en tiempo real dentro del sistema nervioso de pez cebra en desarrollo.
Resumen
El desarrollo del sistema nervioso vertebrado requiere una coordinación precisa de comportamientos e interacciones celulares complejos. El uso de técnicas de imagen in vivo de alta resolución puede proporcionar una ventana clara a estos procesos en el organismo vivo. Por ejemplo, las células divisorias y su progenie se pueden seguir en tiempo real a medida que se forma el sistema nervioso. En los últimos años, los avances técnicos en técnicas multicolor han ampliado los tipos de preguntas que se pueden investigar. El enfoque brainbow multicolor se puede utilizar no sólo para distinguir entre celdas similares, sino también para codificar por colores varios clones diferentes de celdas relacionadas que cada uno deriva de una celda progenitora. Esto permite un análisis de linaje multiplex de muchos clones diferentes y sus comportamientos simultáneamente durante el desarrollo. Aquí describimos una técnica para el uso de la microscopía confocal de lapso de tiempo para visualizar un gran número de células etiquetadas en Brainbow multicolor es en tiempo real dentro del sistema nervioso de pez cebra en desarrollo. Esto es particularmente útil para seguir las interacciones celulares entre células similares, que son difíciles de etiquetar diferencialmente utilizando colores tradicionales impulsados por el promotor. Nuestro enfoque se puede utilizar para rastrear las relaciones de linaje entre varios clones diferentes simultáneamente. Los grandes conjuntos de datos generados con esta técnica proporcionan información rica que se puede comparar cuantitativamente a través de manipulaciones genéticas o farmacológicas. En última instancia, los resultados generados pueden ayudar a responder preguntas sistemáticas sobre cómo se desarrolla el sistema nervioso.
Introducción
En las primeras fases del desarrollo, las piscinas de células progenitoras especializadas se dividen repetidamente en zonas proliferativas, produciendo diversos arreglos de células hijas. Las células nacidas durante este período de desarrollo se diferenciarán y viajarán para formar los órganos nacientes. En el sistema nervioso, los progenitores como la glia radial dan lugar a neuronas inmaduras en zonas ventriculares. A medida que las neuronas se alejan de los ventrículos y maduran, el tejido en expansión eventualmente forma las estructuras altamente complejas del cerebro1,2,3,4,5,6. La coordinación entre la división de los progenitores y la diferenciación y migración de las neuronas determinará el tamaño, la forma y, por tanto, la función del cerebro, afectando directamente al comportamiento7,8,9,10. Si bien el control estricto sobre estos procesos es claramente crucial para el desarrollo normal del cerebro, los mecanismos globales que regulan estas dinámicas no se entienden bien. Aquí describimos una herramienta para estudiar el desarrollo del sistema nervioso a una resolución celular, permitiendo a los investigadores visualizar las células progenitoras y las neuronas in vivo en el cerebro de pez cebra en desarrollo con Brainbow y realizar un seguimiento de su comportamiento a lo largo del tiempo a través de la microscopía confocal de lapso de tiempo11. El enfoque también se puede adaptar para visualizar otras partes del embrión en desarrollo.
Para observar y distinguir entre las células en el cerebro de pez cebra en desarrollo, hemos adaptado la técnica de etiquetado celular Brainbow11. Brainbow utiliza la expresión combinatoria determinada aleatoriamente de tres proteínas fluorescentes distintas (FP) para etiquetar una población de células. Mientras que la expresión predeterminada para la expresión de Brainbow es el FP rojo dTomato, la recombinación por la enzima Cre recombinase da como resultado la expresión de mCerulean (proteína fluorescente cian, CFP) o proteína fluorescente amarilla (YFP)12,13. La cantidad combinada de cada FP expresada en una celda le da un matiz único, lo que permite una clara distinción visual de las celdas vecinas. Además, cuando una célula progenitora se divide, cada célula hija heredará el color de su célula madre, produciendo clones codificados por colores y permitiendo a los investigadores rastrear el linaje celular11,14. Aunque originalmente se utiliza bañó para analizar circuitos neuronales en ratones12,Brainbow se ha expresado desde entonces en una amplia variedad de organismos modelo, incluyendo el pez cebra15.
Nuestra técnica se basa en métodos anteriores de etiquetado y imagen multicolor para crear imágenes directamente de varios clones codificados por colores a lo largo del tiempo en peces cebra vivos. Debido a su transparencia óptica como embriones, los peces cebra son muy adecuados para los experimentos de imagen16,y estudios anteriores han utilizado Brainbow en el pez cebra para estudiar una variedad de tejidos, incluyendo el sistema nervioso11,15,17,18,19,20,21,22,23,24,25, 26,27. La capacidad de imagen directa en el organismo vivo, junto con su rápido desarrollo ex utero, hacen del pez cebra un modelo valioso de desarrollo de vertebrados. A diferencia del cerebro de los mamíferos, toda la zona proliferativa del cerebro trasero del pez cebra está fácilmente disponible para la toma de imágenes sin interrupción de su entorno endógeno6. Esto permite que los experimentos se lleven a cabo en el organismo vivo, en lugar de en preparaciones de tejido sin vitro o fijos. A diferencia de los experimentos de imágenes fijas, los estudios in vivo permiten un diseño longitudinal, produciendo horas de datos que se pueden analizar en busca de patrones, aumentando así la probabilidad de observar eventos relativamente raros. Dependiendo de la velocidad y la duración de los eventos de interés, los investigadores pueden optar por realizar experimentos cortos (1-2 h) o largos (hasta 16 h) de imágenes de lapso de tiempo. Mediante el uso del promotor de choque térmico de pez cebra 70 (hsp70, hsp), la expresión de Brainbow se puede controlar temporalmente28,29. Además, la expresión de mosaico inducida por este promotor es muy adecuada para etiquetar y rastrear muchos clones11.
La capacidad de identificar visualmente múltiples clones dentro del cerebro vivo es una ventaja de este método. Importantes estudios previos que investigaron el papel de los clones dentro del desarrollo del sistema nervioso utilizaron vectores retrovirales para etiquetar una sola célula progenitora y su progenie usando una sola FP u otra proteína fácilmente visualizada. Dicho etiquetado permite a los investigadores observar un solo clon a lo largo del tiempo, ya sea in vitro o in vivo2,,30,31,32,33,34,35,36,37,38. A diferencia de los métodos para rastrear el comportamiento de las células dentro de un clon, los distintos colores de Brainbow permiten a los investigadores observar la dinámica entre los clones. Además, al usar Brainbow para etiquetar muchos clones dentro del cerebro, se recopilan datos adicionales sobre el comportamiento clonal en relación con las técnicas que etiquetan un solo clon11. Es importante destacar que los enfoques descritos aquí se pueden ampliar para generar comparaciones de desarrollo entre peces que han sufrido diferentes manipulaciones genéticas o farmacológicas18. En general, estas ventajas hacen que las imágenes confocales in vivo de barcoiris expresen el tamaño de los peces cebra sean ideales para investigadores que exploran el desarrollo del sistema nervioso de los vertebrados, en particular aquellos interesados en el papel de los clones.
Protocolo
Los procedimientos relacionados con sujetos de animales han sido aprobados por el Comité Institucional de Cuidado y Uso de Animales (IACUC) en Lewis & Clark College.
1. Microinyección de embriones de pez cebra
- Establecer el tipo salvaje, pez cebra adulto en tanques de apareamiento segregados por sexo la tarde antes de realizar microinyecciones39,40.
- Prepare la solución de ADN en la mañana de las microinyecciones. Hspdiluido :Zebrabow11 plásmido ADN a una concentración de 10 ng /L en 0,1 mM de KCl, junto con 2,5% de color rojo fenol y 3,75U Cre recombinase enzima.
- Realizar microinyecciones de solución de ADN en embriones de pez cebra de una célula dentro de 45 min de fertilización39,41. Inyectar aproximadamente 4,2 nL de esta solución en cada embrión, equivalente a 42 pg de ADN plásmido.
NOTA: El paso de microinyección se puede omitir si en su lugar se aparea una línea transgénica de pez cebra que expresa a Brainbow, como Tg(ubi:Zebrabow)15 y Tg(neurod:Zebrabow)18. Realizar microinyecciones puede ser ventajoso porque permite a los investigadores valorar el número de copia y la densidad de etiquetado. Además, una segunda construcción de ADN que etiqueta o manipula un producto genético específico se puede inyectar junto con Brainbow si se desea (por ejemplo, una proteína fluorescente de color rojo lejano que complementa Brainbow18). - Mantener los embriones inyectados en platos Petri de E3 medio39 en una incubadora de 28oC durante 24 h.
2. Choque de calor para inducir la expresión de Brainbow
NOTA: Si el ADN de plásmido inyectado o el transgén expresado no utiliza el promotor hsp70 para impulsar la expresión, se puede omitir el paso de choque térmico, y los embriones sanos deben transferirse inmediatamente a fenilationa (PTU) a las 24 h después de la fertilización (hpf).
- A 24 hpf, se hunden embriones muertos y deformes del grupo de embriones inyectados. A continuación, transfiera los embriones sanos a un tubo de 50 ml, con hasta 20 embriones/tubo.
- Llene los tubos con 10 ml de E3. Coloque una tapa en la parte superior de cada tubo, pero no cierre bien.
- Coloque el portatubos que contiene los tubos de 50 ml en posición vertical en un baño de agua de 37 oC. Asegúrese de que el nivel de agua en el baño es más alto que el nivel del E3 en los tubos y déjelos durante 80 a 90 minutos.
- Retire el portatubos con los embriones del baño de agua y vuelva a la incubadora de 28oC en posición vertical. Deje que hasta 1 h para que el E3 se enfríe y los embriones se vuelvan a aclimatar gradualmente a la temperatura. A continuación, transferir los embriones a platos Petri de 0,2 mM de PTU en E3 calentados en la incubadora para evitar la pigmentación de embriones.
PRECAUCION: La PTU es un producto químico tóxico que debe manipularse con cuidado y eliminarse adecuadamente. Se sugiere usar guantes.
3. Detección de embriones para la expresión de Brainbow
- A las 2 a 4 h después del choque de calor, examine los embriones bajo un microscopio de disección de fluorescencia estándar para la expresión de CFP o YFP, lo que indica una recombinación exitosa de Brainbow (de la expresión predeterminada de dTomato).
NOTA: Si hay varias opciones de filtro de fluorescencia disponibles, la detección de CFP es la forma más eficiente de identificar peces bien recombinados. Si los filtros CFP y YFP no están disponibles, YFP también se puede visualizar débilmente bajo filtros de proteína fluorescente verde (GFP) debido a la superposición en sus perfiles espectrales. - Seleccione embriones con una expresión de FP robusta en todo el tiempo y transfiera a un plato separado con PTU. Imagen de embriones a 1 día después de la fertilización (dpf; 28 hpf o posterior) o mantener embriones en PTU e imagen a 2 o 3 dpf.
4. Montaje de embriones para imágenes in Vivo
-
Antes del día del experimento, prepare la cámara de imágenes y la herramienta de manipulación de embriones.
- Prepare la cámara de imágenes superpegando cuidadosamente un anillo de plástico en el centro de un plato Petri de 60 mm.
- Opcionalmente, prepare un manipulador de embriones hecho a medida para mover embriones dentro del plato y orientar los embriones durante el montaje. Construir el manipulador supergluing una pequeña longitud de la línea de pesca de nylon (1 /2 pulg., 6 libras) hasta el final de un bastón de hisopo de algodón de madera que se ha cortado a un 4 de longitud.
- Si es necesario (es decir, imágenes a 2 dpf o antes), antes de montar el decotermonato de embriones utilizando jeringas bajo un microscopio de disección.
-
Anestetizar embriones de pez cebra.
- Llene un plato Petri de 60 mm hasta la mitad con E3 y agregue 5-6 gotas de 4 mg/ml MS-222 Tricaine-S a una concentración final de 0,2 mM. Gire para mezclar.
PRECAUCION: La tricaína debe manipularse con cuidado y desecharse adecuadamente. - Transfiera embriones de la PTU a la solución de tricaína, asegurando que se transfiera la menor PTU posible. Mientras que los reflejos de los peces deben cesar después de 1-2 min, deje los embriones en tricaína hasta 10 minutos para asegurar una anestesia completa para experimentos de imágenes de lapso de tiempo.
- Confirme que los embriones están correctamente anestesiados. Evalúe el reflejo del sobresalto tocando suavemente las colas del embrión con el manipulador de embriones. Si se observa un movimiento reflejo, agregue una gota adicional de tricaína al plato lo más lejos posible de los embriones y gire para mezclar. Observe la frecuencia cardíaca del embrión bajo un alcance de disección. Si es anormalmente lento, añadir gotas adicionales de E3 al plato para reducir la concentración de tricaína.
- Llene un plato Petri de 60 mm hasta la mitad con E3 y agregue 5-6 gotas de 4 mg/ml MS-222 Tricaine-S a una concentración final de 0,2 mM. Gire para mezclar.
-
Montar embriones anestesiados en agarosa.
- Transfiera el pescado al centro del anillo de plástico dentro de la cámara de imágenes y retire tanto exceso de E3 como sea posible utilizando una pipeta de transferencia de punta fina.
- Con una pipeta de transferencia limpia, cubra el pescado con agarosa de fusión baja (LMA) al 1,0% en E3 almacenada a 42 oC y llene todo el anillo de plástico con una fina capa de agarosa. Utilice una pipeta de transferencia para tirar suavemente de los embriones hacia arriba en la punta de la pipeta y de nuevo en la agarosa sin introducir burbujas de aire.
- Antes de que la agarosa se endurezque, utilice rápidamente un manipulador de embriones para orientar a los peces adecuadamente. Si toma imágenes con un microscopio vertical, coloque los embriones lo más cerca posible de la superficie superior de la agarosa. Coloque los embriones paralelos a la parte inferior de la cámara de imágenes con la cola recta.
NOTA: Para las imágenes posteriores del cerebro, los embriones se pueden colocar en una vista dorsal o sagital. Se debe tener cuidado de asegurar que las cabezas de los embriones no se hundan mientras la agarosa sigue siendo líquida. Otras orientaciones pueden ser apropiadas para atacar otros tejidos en desarrollo para la toma de imágenes. Para los microscopios invertidos, el montaje se haría de manera diferente, normalmente colocando el pez cerca de la parte inferior de un plato Petri con fondo de vidrio.
- Espere a que la agarosa se endurezque y, a continuación, llene la cámara de imágenes con E3. Agregue tanto E3 como sea posible para tener en cuenta la evaporación en el transcurso de la imagen.
5. Imágenes confocales de lapso de tiempo del desarrollo del pez cebra Hindbrain
-
Coloque la cámara de imágenes en el microscopio confocal en preparación para la toma de imágenes.
- Coloque la cámara de imágenes con el pez y E3 en el microscopio confocal.
- Seleccione un objetivo con una alta apertura numérica y una larga distancia de trabajo, como un objetivo de inmersión en agua 20x (1,0 NA).
- Encuentre una región con un etiquetado adecuado y denso y brillante de los tipos de celda de interés.
NOTA: También se puede utilizar un escenario/aparato con temperatura controlada en el microscopio para regular la temperatura y la humedad durante largas sesiones de diagnóstico por imágenes. Esto puede disminuir la evaporación.
-
Configure los parámetros de adquisición para la imagen de Brainbow.
- Imagen de cada canal FP secuencialmente. Si se utiliza un software comercial (por ejemplo, software Zen), esto se hace mediante la preparación de tres "pistas". Utilice un láser de argón para excitar CFP a 458 nm y YFP a 514 nm. Utilice un láser DPSS 561 nm para excitar dTomato. Recoger emisiones entre 463–509 nm para CFP, 519–555 nm para YFP, y 566–691 nm para dTomato.
- Para la visualización en pantalla, codifique CFP como azul, YFP como amarillo y dTomato como rojo.
NOTA: Estos ajustes variarán dependiendo de qué líneas láser y otras características estén disponibles en el microscopio confocal. Para aumentar la velocidad de la imagen, los canales CFP y dTomato se pueden crear imágenes simultáneamente sin sangrado apreciable. Si también se está creando una imagen de UN FP independiente, como un FP de color rojo lejano, esto se puede crear una imagen secuencial o simultáneamente con YFP. - Configure los parámetros generales de imagen para tomar imágenes de 16 bits con una resolución de al menos 1024 x 1024 y promediar dos veces. Ajuste el zoom dependiendo de la región y el tipo de celda de interés (es decir, para imágenes de cerebro trasero con un objetivo de 20x, el zoom oscilará entre 1.0 y 2.5). Maximizar el campo de visión para permitir el crecimiento de los peces a lo largo del tiempo.
- Visualización de una pista a la vez (y desactivación de otros láseres para evitar el fotoblanqueo), optimice los ajustes de adquisición para cada FP individualmente (por ejemplo, potencia láser, tamaño del agujero, ganancia del tubo del fotomultiplicador, etc.).
- Seleccione Rango de pila Z para crear una imagen.
NOTA: Para largas sesiones de imágenes (>2 h), tenga en cuenta que el pez seguirá creciendo a lo largo del curso de la toma de imágenes, por lo que tanto el campo XY como el rango de la pila Z deben tener en cuenta esto con espacio adicional. Si toma imágenes del cerebro trasero, incluya espacio en el campo para que el tejido se mueva rostrally durante el período de diagnóstico por imágenes. También es necesario incluir espacio para el crecimiento en la dimensión Z. Incluir en la imagen aproximadamente 10-20 m por encima de la región de interés, si el pez está posicionado para la vista sagital o dorsal.
-
Configure los parámetros para la creación de imágenes de lapso de tiempo.
- Seleccione un intervalo de tiempo. La longitud del intervalo oscila entre 10-30 min para rastrear eventos mitoticos y apoptoticos en el cerebro trasero en desarrollo. La longitud del intervalo variará dependiendo de la velocidad de los eventos de interés y el tiempo total que se tarda en capturar una sola pila Z.
- Seleccione la duración de la sesión de imágenes. Las sesiones de imágenes de lapso de tiempo generalmente ocurren durante la noche y pueden durar al menos 16 horas.
-
Ejecuta el experimento.
NOTA: El pescado puede ser eutanasiado inmediatamente después de la toma de imágenes o diseccionado cuidadosamente de la agarosa usando jeringas y devuelto a E3 para recuperarse. Los peces recuperados se pueden volver a tomar una imagen en un momento posterior, aunque la posición y la profundidad de las celdas cambiarán durante este período debido al crecimiento general.
6. Gestión de archivos de lapso de tiempo
-
Importe el archivo de imagen en el software de análisis.
- Si utiliza el software Zen, guarde el archivo en formato .czi una vez completada la adquisición de la imagen. Asegúrese de guardar los datos sin procesar en un formato compatible con Fiji42 y/u otro software.
- Importe al software de Fiji utilizando BioFormats Importer.
NOTA: Los archivos de lapso de tiempo serán grandes y pueden requerir cantidades significativas de tiempo y memoria para abrir para el análisis. Por lo tanto, es útil ser estratégicos en la forma en que se guardan y se abren. Puede ser útil para guardar una versión de menor calidad que se puede abrir más fácilmente para buscar eventos de interés a simple vista.
- Si utiliza Fiji, cree subconjuntos más pequeños y manejables de archivos de lapso de tiempo. Genere subconjuntos por tiempo (por ejemplo, viendo la pila Z completa en el primer punto de tiempo) o por profundidad (por ejemplo, viendo las primeras 50 secciones Z en cada punto de tiempo) haciendo clic en Imagen. Pilas? Herramientas? Hacer Substack.
7. Análisis cuantitativo del color clonal de imágenes de Arco iris
- Mida los valores medios de intensidad rojo, verde y azul (RGB) para las celdas de interés en Fiji.
NOTA: Estas instrucciones son específicas para el análisis de imágenes en Fiji. Sin embargo, los investigadores pueden preferir obtener valores medios de intensidad RGB en un programa de software alternativo.- Asegúrese de que el archivo esté recortado correctamente para que no haya espacio negro alrededor de los bordes de la imagen. El espacio negro reducirá artificialmente las mediciones de intensidad mínima necesarias para el análisis. Si es necesario recortar el archivo, seleccione el botón Selección de rectángulo y dibuje una región de interés (ROI) alrededor del campo de visión, excluyendo todo el espacio negro. Haga clic en Imagen ( Image) Recortar.
- Establezca la herramienta de medición para que tome medidas de valor medio, mínimo y máximo de valor gris. Haga clic en Analizar (Analizar) Establezca Medidas. Asegúrese de que las casillas de verificación Valor mínimo y máximo de gris y Valor gris medio estén seleccionados.
- Al ver los archivos de datos sin procesar o las pilas Z de subconjuntos en Fiji, busque celdas de interés para el análisis de color clonal. En el cerebro trasero, los clones putativos pueden ser identificados visualmente por su tono compartido, así como por su orientación radial. No seleccione celdas que estén en estrecho contacto con las celdas vecinas de otro color y, por lo tanto, difíciles de resolver desde ellas. Puede ser difícil cuantificar su tono.
- Busque el plano Z central de la celda de interés utilizando la barra de desplazamiento Z. Haga clic con el botón derecho en el botón Selección ovalada y seleccione el botón Selección elíptica. Otras herramientas de selección serán adecuadas para diferentes tipos de celdas. Dibuje un ROI elíptico alrededor de la central de 2/3 del cuerpo de la célula.
- Con la barra de desplazamiento En C,seleccione el canal rojo (dTomato). Tome la medición de intensidad media para este canal seleccionando Analizar. Medir. Repita con el mismo ROI y plano focal para los canales verde (YFP) y azul (CFP) y guarde todos los valores de intensidad media.
- Haga clic en cualquier parte de la imagen para eliminar el ROI elíptico en la celda de interés.
- Para medir los niveles de fondo para la normalización, utilice la barra de desplazamiento C y seleccione el canal rojo (dTomato). Tome la medición de intensidad mínima para todo el campo en este canal seleccionando Analizar. Medir. Repita con el mismo plano focal para los canales verde (YFP) y azul (CFP) y guarde todos los valores de intensidad mínimos.
- Calcular pesos de canal RGB relativos para las celdas de interés.
NOTA: El cálculo de los pesos de canal RGB relativos a partir de estos valores de intensidad se puede realizar en una variedad de programas de software, como Microsoft Excel o R43.- Normalice la medición de intensidad media del ROI celular para cada canal restando la intensidad mínima de todo el campo en ese canal y plano focal.
- Sume los valores medios normalizados de intensidad RGB de cada canal para encontrar la intensidad RGB normalizada total para la celda.
- Calcule el peso del canal relativo para cada canal dividiendo el valor de intensidad media normalizada para ese canal por la intensidad RGB normalizada total.
- Visualice los pesos de canal RGB relativos como trazados ternarios utilizando el paquete Ternary para R43,44. Las gráficas ternarias son útiles para visualizar la agrupación en clústeres de colores similares en el espacio RGB 3D.
- Calcule el coeficiente de dispersión de color para cuantificar la diferencia entre los colores de celda.
- Calcule la distancia euclidiana entre los dos colores en el espacio RGB 3D utilizando la siguiente ecuación:
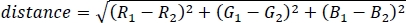
donde R, G y B son los pesos de canal RGB relativos calculados en 7.2. - Para facilitar la comprensión, normalice la distancia de color dividiendo por 2, la distancia máxima posible entre los colores, para obtener un coeficiente de dispersión de color entre 0 y 1, donde 0 indica exactamente el mismo color y 1 indica colores completamente diferentes (por ejemplo, rojo puro y azul puro).
- Calcule la distancia euclidiana entre los dos colores en el espacio RGB 3D utilizando la siguiente ecuación:
Resultados
En esta sección se ilustran ejemplos de resultados que se pueden obtener utilizando el enfoque de imágenes de lapso de tiempo multicolor in vivo que se describe aquí. Mostramos que Brainbow clona de células codificadas por colores en la zona ventricular proliferativa del cerebro trasero de pez cebra en desarrollo14 (Figura 1).
Normalmente, cuando las celdas etiquetadas con Brainbow se organizaban a lo largo de una fibra radial determinada, compartían el mismo color(Figura 1D),que podría cuantificarse como pesos de canal RGB relativos(Figura 1E). Esto sugiere que estos grupos radiales eran clones de células divisorias y que su color similar podría ser utilizado para identificarlos como relacionados clonalmente, como se observó en estudios anteriores en peces cebra, ratón, y polluelo14,,15. Cuando la densidad de etiquetado de Brainbow es relativamente escasa, esta codificación de color se puede utilizar para visualizar y rastrear claramente muchos clones radiales distintos dentro de regiones proliferativas del cerebro vertebrado vivo.
Un análisis cuantitativo de color mostró que las células hijas expresaban el mismo color que su célula madre (progenitora)(Figura 1F),pero que los cúmulos radiales vecinos de células se pueden distinguir entre sí11 (Figura 1E). Esto significa que numerosos clones de células relacionadas se pueden seguir simultáneamente durante horas in vivo(Figura 2),lo que permite un análisis y comparación del linaje multiplex. La cuantificación de la expresión de color en clones a 2 dpf y luego de nuevo a 3 dpf(Figura 2A-B)mostró que la expresión Brainbow también se mantuvo relativamente constante de 2 a 3 días en vivo11. Por lo tanto, las células individuales pueden ser identificadas y rastreadas en tiempo real durante períodos de tiempo relativamente largos durante el desarrollo.
Las imágenes de lapso de tiempo revelaron numerosas células sometidas a migración nuclear intercinética y división celular durante el período de 1-2 dpf(Figura 2C–F)11, lo que permite el estudio del ciclo celular. Usando un intervalo de lapso de tiempo de 30 min, no siempre capturamos la figura mitotica durante la división celular. Esto introdujo un error potencial de hasta 30 minutos para el tiempo asignado de la división celular. Además, observamos que algunos clones que parecen contener dos celdas progenitoras (por ejemplo, la Figura 2F). Dentro de estas restricciones, se puede medir la longitud del ciclo celular. Mediante el seguimiento de progenitores individuales etiquetados por Brainbow a lo largo del tiempo calculamos un ciclo celular promedio de 8,4 x 1,5 h, comparable a las mediciones anteriores en peces cebra1,45,46. Además, utilizando la técnica de imagen de lapso de tiempo Brainbow, también pudimos observar células individuales sometidas a los cambios morfológicos estereotipados asociados con la apoptosis (Figura 3)11, como la manchado de membrana y la fragmentación celular47,48.
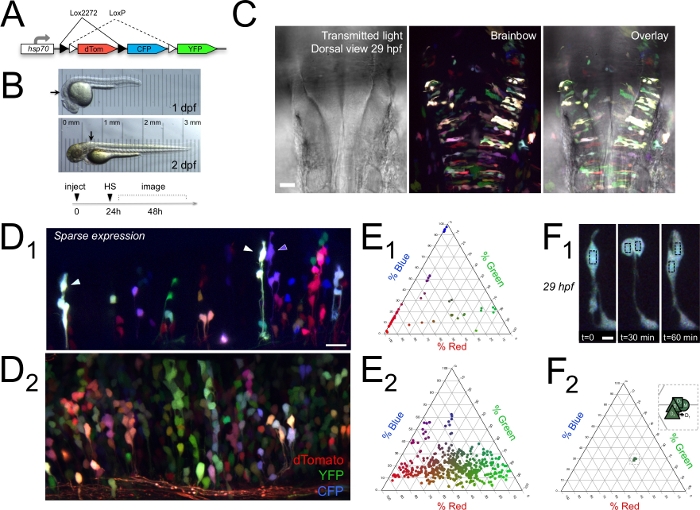
Figura 1: Brainbow etiquetado clonalmente relacionados con cúmulos de células divisorias en el cerebro trasero de pez cebra en desarrollo. (A) Esquema de ADN hsp:Zebrabow (Brainbow) expresado transitoriamente a clones de código de color. (B) Imágenes de luz transmitida in vivo del pez cebra en desarrollo a 1 y 2 dpf; flechas indican la región general del cerebro trasero destinada a la toma de imágenes. La translucidez de los embriones se demuestra mediante un micrómetro situado debajo de cada pez. Línea de tiempo experimental que muestra inyecciones en una etapa de una célula, choque de calor (HS) a 24 hpf e imágenes de 1-3 dpf. (C) Vista dorsal del cerebro trasero del pez cebra de 29 hpf etiquetado con Brainbow. El canal de luz transmitido mostró la morfología del cerebro trasero y el ventrículo superpuesta con la máxima proyección de intensidad de clones marcados por Brainbow que representan 165 m. Rostral se ha levantado. (D) Expresión in vivo barco iris en el cerebro trasero, que se muestra en proyecciones de intensidad máxima que representan 41 m en peces cebra de 51 hp(D1),y 81 m en peces cebra de 63,5 hpf (D2). En ambos paneles, dorsal está hacia arriba y rostral está a la izquierda. (E) El color de las celdas en D1 y D2 se cuantificó como pesos de canal relativos en las gráficas ternarias correspondientes (n a 54 celdas E1; 461 celdas E2). (F) El color celular se mantuvo relativamente constante en las celdas hijas después de la división celular. Serie de puntos de tiempo que muestran eventos mitoticos in vivo en el cerebro trasero de 29 hpf Brainbow-etiquetado pez cebra sobre 1 h(F1, proyecciones de intensidad máxima, 30 m de profundidad). El color de las celdas madre e hija, indicado por las cajas negras en F1, se representa como pesos de canal relativos en la gráfica ternaria en F2. El recuadro muestra un zoom del mismo trazado para mostrar colores de celda estrechamente agrupados (M es madre; D1 y D2 son hijas a 30 min/cuadrados, y 60 min/triángulos). Barras de escala a 30 m (C), 20 m (D1), 16 m (D2) y 5 m (F1). En C, D y F, dTomato se codifica como rojo, YFP se codifica como verde y CFP se codifica como azul. Imágenes reimpresas con permiso de Brockway et al.11. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Imágenes de lapso de tiempo in vivo combinadas con la codificación de color Brainbow distinguieron múltiples clones vecinos de células sometidas a migración nuclear intercinética y división. (A) Racimos radiales codificados por colores etiquetados en el cerebro trasero de un pez que expresa Brainbow a 2 dpf (55 hpf; A1) y 3 dpf (71 hpf; A2); proyecciones de intensidad máxima que representan 62 m y 47 m respectivamente. Tres clústeres radiales codificados por colores se identifican con puntas de flecha en cada punto de tiempo. (B) El color de todas las celdas etiquetadas por Brainbow en A1 y A2 se cuantificó como pesos de canal relativos en las gráficas ternarias correspondientes en B1 y B2 (n a 106 celdas, 119 celdas). (C) cerebro trasero de pez cebra a 35 hpf etiquetado con Brainbow (proyección de intensidad máxima que muestra 24 m); el recuadro indicado con el cuadro blanco discontinuo es el primer punto de tiempo que se muestra en la serie D. (D) de puntos de tiempo tomados a intervalos de 30 minutos en el cerebro trasero de C. Se muestra un período de >9,5 h. Las células etiquetadas fueron objeto de migración nuclear intercinética; las puntas de flecha blancas indican una celda en la superficie apical. El redondeo del soma celular en la superficie apical y un aumento correspondiente en el número de clon (por ejemplo, glóbulos rojos a 5,5 h y luego 8 h) se consideró un evento mitótico. (E) Vista dorsolateral del cerebro trasero del pez cebra a 30,5 hpf etiquetado con Brainbow, donde la línea blanca discontinua y punteada muestra el límite aproximado de la cabeza, y el cuadro con guiones blancos indica el recuadro mostrado en el primer punto de tiempo de F. (F) Serie de puntos de tiempo tomados a intervalos de 30 minutos en el cerebro trasero de E. Durante un período de 1,5 h, se pudieron observar dos células azules indicadas por las puntas de flecha blancas moviéndose a la superficie apical y suponiendo mitosis, lo que sugiere un clon que contiene dos células progenitoras. Barra de escala a 20 m (A), 25 m (C) y 20 m (F). En A-D, dorsal está hacia arriba y rostral está a la izquierda. En E y F, rostral está a la izquierda. En todas las imágenes, dTomato se codifica como rojo, YFP se codifica como verde y CFP se codifica como azul. Imágenes reimpresas con permiso de Brockway et al.11. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Las imágenes de Brainbow de lapso de tiempo muestran que las células de la zona ventricular sufren la muerte celular in vivo. (A) cerebro trasero de pez cebra a 31 hpf etiquetado con Brainbow (proyección de intensidad máxima que representa 36 m). El cuadro discontinuo blanco indica el recuadro mostrado en el primer punto de tiempo en la serie B. (B) de puntos de tiempo que muestran el cerebro trasero en A a intervalos de 30 minutos. La punta de flecha blanca en el primer panel muestra una celda de lavanda que sufrió apoptosis como se muestra en los paneles posteriores, indicado por la fragmentación celular seguida de un aclaramiento gradual de cuerpos apoptoticos. Las células etiquetadas vecinas parecían saludables en todas las instalaciones. Dorsal está arriba y rostral está a la izquierda. Barra de escala a 20 m (B). En todas las imágenes, dTomato se codifica como rojo, YFP se codifica como verde y CFP se codifica como azul. Imágenes reimpresas con permiso de Brockway et al.11. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Este protocolo describe un método para visualizar clones de células progenitoras y neuronas en el cerebro trasero de pez cebra en desarrollo y seguirlos in vivo utilizando Brainbow y la microscopía confocal de lapso de tiempo11. La principal ventaja de este protocolo en comparación con los estudios in vitro o ex vivo es la capacidad de observar directamente la zona proliferativa del cerebro vertebrado en su entorno natural a lo largo del tiempo. Esta técnica se basa en estudios anteriores que etiquetaron a un solo clon utilizando vectores retrovirales. Por el contrario, el uso de hsp:Zebrabow construye códigos de color muchos clones simultáneamente, lo que permite el trazado de linaje multiplex y un enfoque en la dinámica entre los clones11. Este protocolo se puede modificar para centrarse en el desarrollo en otros sistemas o tipos de células. Por ejemplo, diferentes construcciones de Brainbow pueden expresarse en embriones de pez cebra. El uso del promotor hsp70 de pez cebra en hsp:Zebrabow dará lugar al etiquetado de mosaico de una variedad de tipos de células después del choque de calor, incluidos los progenitores neuronales y las neuronas11,y da a los investigadores control temporal sobre la expresión génica29. Cabe señalar que el uso del promotor de choque térmico etiqueta constantemente clones codificados por colores, mientras que otros promotores pueden no delinear claramente el linaje celular de esta manera. Cuando la identidad clonal no es un factor de interés, las construcciones que utilizan otros promotores se pueden utilizar para etiquetar tipos de celda específicos. Por ejemplo, el promotor neuroD se puede utilizar para etiquetar las neuronas inmaduras18,49. Además, en lugar de realizar microinyecciones, los investigadores pueden optar por utilizar el pez cebra Brainbow transgénico, incluyendo líneas como Tg(ubi:Zebrabow)15 y Tg(neurod:Zebrabow)18. En este caso, los cruces a las líneas que expresan Cre recombinase, como Tg(hsp:Crea134))15, producen embriones no inyectados que se recogen y mantienen de manera similar. Mientras que este protocolo está diseñado para fotodar peces entre 1-3 dpf, con el fin de coincidir con el período principal de neurogénesis del desarrollo50, el pez cebra puede mantenerse durante más tiempo dependiendo de la etapa de desarrollo de interés. Sin embargo, el choque térmico para inducir la expresión de Brainbow antes de 24 hpf puede ser más perjudicial para los embriones51. Dependiendo de la edad y el tejido de interés, diferentes estrategias de montaje serán apropiadas para tener el tejido objetivo orientado correctamente.
Hay una serie de pasos que pueden requerir la solución de problemas, en particular si se realizan modificaciones en el protocolo. La concentración de ADN, el tamaño del bolo inyectado y la cantidad total de ADN entregado por embrión pueden ajustarse si la expresión de Brainbow es tenue o escasa o si los embriones no parecen sanos. Además, la longitud y la sincronización del paso de choque térmico pueden modificarse si no se logra la expresión deseada. Los parámetros de adquisición utilizados en la toma de imágenes variarán en función de las líneas láser y los filtros disponibles, así como del brillo de la expresión, el montaje y el tipo de análisis deseado. Además, los parámetros de lapso de tiempo deben ajustarse en función de la velocidad de los eventos de interés y el tiempo necesario para adquirir una sola pila Z. Uno de los pasos más críticos en el protocolo es el montaje adecuado de los peces en agarosa: si el ángulo del pez es inapropiado o si el pez está cubierto por demasiada agarosa, no será posible obtener imágenes óptimas. Optimizar esta técnica puede tomar algo de práctica. Además, a menudo es útil montar varios peces antes de la toma de imágenes, ya que puede ser difícil determinar si el montaje es apropiado antes de ver la expresión FP en el propio microscopio confocal. Un paso crítico adicional es la selección y selección de los peces específicos que se van a tomar imágenes. Si se realizan microinyecciones, el brillo, la densidad de etiquetado y el número de copia de Brainbow pueden variar significativamente entre los peces. Las imágenes tomadas en peces con etiquetado tenue, etiquetado demasiado denso o un número de copia bajo que resulta en pocos colores pueden ser más difíciles de analizar.
Con este protocolo, hemos sido capaces de imaginar continuamente peces cebra vivos hasta 16 h11. Este período de tiempo puede verse limitado por la evaporación E3 de la cámara de imágenes, el crecimiento de los peces fuera del plano de imagen, la muerte o el movimiento de los peces, o las restricciones del usuario en el tiempo del microscopio confocal. La evaporación podría disminuir con el uso de un accesorio de temperatura controlada en la etapa del microscopio. Cabe señalar que los conjuntos de datos 5D generados con este enfoque tienden a ser archivos grandes que requieren un espacio significativo en el disco duro (por ejemplo, hasta 100 GB para un lapso de tiempo durante la noche) y una potencia informática adecuada para analizar.
Este protocolo guía a los investigadores para visualizar clones codificados por colores dentro de una población de progenitores neuronales y neuronas hijas en el cerebro de pez cebra en desarrollo y realizar un seguimiento de su dinámica a lo largo del tiempo. Una posible expansión es la combinación de imágenes de lapso de tiempo Brainbow con etiquetado de FP de color rojo lejano para evaluar las funciones de genes específicos en el desarrollo18. De esta manera, los clones que expresan genes manipulados pueden visualizarse en un color distinto, rastrearse a lo largo del tiempo y compararse con clones vecinos no manipulados con etiqueta Brainbow. Las imágenes multicolores in vivo se pueden utilizar para abordar preguntas mecanicistas importantes sobre cómo se forma y funciona el sistema nervioso.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a Y. A. Pan, J. Livet y Z. Tobias por sus contribuciones técnicas e intelectuales. Este trabajo fue apoyado por la National Science Foundation (Premio 1553764) y el M.J. Murdock Charitable Trust.
Materiales
| Name | Company | Catalog Number | Comments |
| 1.5 mL transfer pipette (fine tip) | Globe Scientific, Inc. | 134020 | |
| 1-phenyl-2-thiourea (PTU) | Alfa Aesar | L06690 | Diluted to 0.2 mM in E3 to prevent embryo pigmentation |
| 50 mL conical tubes | Corning | 352070 | For heat shocking embryos |
| 6 lb nylon fishing line | SecureLine | NMT250 | For making embryo manipulators |
| 7.5 mL transfer pipet | Globe Scientific, Inc. | 135010 | |
| CaCl2 | Sigma | C3881 | For E3 |
| Cotton swabs | Puritan | 867-WC NO GLUE | For making embryo manipulators |
| Cre recombinase | New England Biolabs | M0298M | |
| Digital dry bath | Genemate | 490016-616 | Used to store LMA at 42 °C |
| Epifluorescence dissection scope | |||
| Glass capillary tubes | World Precision Instruments | TW100F-4 | |
| Incubator | Forma Scientific | 3158 | To maintain embryos at 28 °C |
| Injection plate molds | Adaptive Science Tools | TU-1 | |
| Isotemp water bath | Fisher Scientific | 2320 | For heat shocking embryos |
| KCl | AMRESCO | 0395 | For E3 and for DNA solution for injections |
| Laser-scanning confocal microscope | Zeiss | LSM710 | |
| LE agarose | Genemate | E3120 | To create agarose injection plates |
| Low-melt agarose (LMA) | AMRESCO | J234 | |
| Mating tanks | Aquaneering, Inc. | ZHCT100 | |
| Methylene blue | Sigma | M9140 | For E3 |
| MgSO4 | Sigma | 9397 | For E3 |
| Micromanipulator | World Precision Instruments | M3301 | |
| Micropipette Puller | Sutter Instrument Co. | P-97 | |
| MS-222 Tricaine-S | Western Chemical, Inc. | Stock made at 4 mg/mL in reverse osmosis (RO) water, then added dropwise to E3 to final concentration of 0.2 mM to anesthetize embryos | |
| NaCl | J.T. Baker | 4058-01 | For E3 |
| Petri dishes (90 mm, 60 mm) | Genesee Scientific | 32-107G | To house embryos and create imaging chamber (60 mm) |
| Phenol red | Sigma | P0290 | |
| Soft stitch ring markers | Clover Needlecraft, Inc. | 354 | For creating imaging chamber with Petri dish |
| Super glue (Ultra gel control) | Loctite | 1363589 | For making embryo manipulators |
| Syringe needles | Beckton Dickinson | BD329412 | For dechorionating embryos |
Referencias
- Lyons, D. A., Guy, A. T., Clarke, J. D. W. Monitoring neural progenitor fate through multiple rounds of division in an intact vertebrate brain. Development. 130, 3427-3436 (2003).
- Noctor, S. C., Flint, A. C., Weissman, T. A., Dammerman, R. S., Kriegstein, A. R. Neurons derived from radial glial cells establish radial units in neocortex. Nature. 409 (6821), 714-720 (2001).
- Kriegstein, A., Alvarez-Buylla, A. The glial nature of embryonic and adult neural stem cells. Annual Review of Neuroscience. 32, 149-184 (2009).
- Florio, M., Huttner, W. B. Neural progenitors, neurogenesis and the evolution of the neocortex. Development. 141, 2182-2194 (2014).
- Mcconnell, S. K. Constructing the Cerebral Cortex: Neurogenesis and Fate Determination Review. Neuron. 15, 761-768 (1995).
- Schmidt, R., Strähle, U., Scholpp, S. Neurogenesis in zebrafish - from embryo to adult. Neural Development. 8, 3(2013).
- Chenn, A., Walsh, C. Regulation of Cerebral Cortical Size by Control of Cell Cycle Exit in Neural Precursors. Science. 297 (5580), 365-369 (2002).
- Lui, J. H., Hansen, D. V., Kriegstein, A. R. Development and Evolution of the Human Neocortex. Cell. 146 (1), 18-36 (2011).
- Nonaka-Kinoshita, M., et al. Regulation of cerebral cortex size and folding by expansion of basal progenitors. The EMBO Journal. 32, 1817-1828 (2013).
- Homem, C. C. F., Repic, M., Knoblich, J. A. Proliferation control in neural stem and progenitor cells. Nature Reviews Neuroscience. 16 (11), 647-659 (2015).
- Brockway, N. L., et al. Multicolor lineage tracing using in vivo time-lapse imaging reveals coordinated death of clonally related cells in the developing vertebrate brain. Developmental Biology. 453 (2), 130-140 (2019).
- Livet, J., et al. Transgenic strategies for combinatorial expression of fluorescent proteins in the nervous system. Nature. 450 (7166), 56-62 (2007).
- Weissman, T. A., Pan, Y. A. Brainbow: New resources and emerging biological applications for multicolor genetic labeling and analysis. Genetics. 199 (2), 293-306 (2015).
- Loulier, K., et al. Multiplex Cell and Lineage Tracking with Combinatorial Labels. Neuron. 81 (3), 505-520 (2014).
- Pan, Y. A., et al. Zebrabow: multispectral cell labeling for cell tracing and lineage analysis in zebrafish. Development. 140, 2835-2846 (2013).
- Ko, S. K., Chen, X., Yoon, J., Shin, I. Zebrafish as a good vertebrate model for molecular imaging using fluorescent probes. Chemical Society Reviews. 40, 2120(2011).
- Kesavan, G., Hammer, J., Hans, S., Brand, M. Targeted knock-in of CreERT2 in zebrafish using CRISPR/Cas9. Cell and Tissue Research. 372, 41-50 (2018).
- Cook, Z. T., et al. Combining near-infrared fluorescence with Brainbow to visualize expression of specific genes within a multicolor context. Molecular Biology of the Cell. 30 (4), 491-505 (2019).
- Kuwata, M., Nikaido, M., Hatta, K. Local heat-shock mediated multi-color labeling visualizing behaviors of enteric neural crest cells associated with division and neurogenesis in zebrafish gut. Developmental Dynamics. 248, 437-448 (2019).
- Kinkhabwala, A., et al. A structural and functional ground plan for neurons in the hindbrain of zebrafish. Proceedings of the National Academy of Sciences. 108 (3), 1164-1169 (2011).
- Heap, L. A., Goh, C. C., Kassahn, K. S., Scott, E. K. Cerebellar Output in Zebrafish: An Analysis of Spatial Patterns and Topography in Eurydendroid Cell Projections. Frontiers in Neural Circuits. 7, 53(2013).
- Robles, E., Filosa, A., Baier, H. Precise Lamination of Retinal Axons Generates Multiple Parallel Input Pathways in the Tectum. The Journal of Neuroscience. 33 (11), 5027-5039 (2013).
- Dirian, L., et al. Spatial Regionalization and Heterochrony in the Formation of Adult Pallial Neural Stem Cells. Developmental Cell. 30, 123-136 (2014).
- Chen, X. A., et al. Behavioral/Cognitive QRFP and Its Receptors Regulate Locomotor Activity and Sleep in Zebrafish. The Journal of Neuroscience. 36 (6), 1823-1840 (2016).
- Albadri, S., De Santis, F., Di Donato, V., Del Bene, F. CRISPR/Cas9-Mediated Knockin and Knockout in Zebrafish. Genome Editing in Neurosciences. Jaenisch, R., Zhang, F., Gage, F. , Springer. Cham, Switzerland. 41-49 (2017).
- Furlan, G., et al. Life-Long Neurogenic Activity of Individual Neural Stem Cells and Continuous Growth Establish an Outside-In Architecture in the Teleost Pallium. Current Biology. 27, 3288-3301 (2017).
- Herget, U., Arturo Gutierrez-Triana, J., Salazar Thula, O., Knerr, B., Ryu, S. Single-cell reconstruction of oxytocinergic neurons reveals separate hypophysiotropic and encephalotropic subtypes in larval zebrafish Brainbow-guided morphology of oxytocinergic cells. eNeuro. 4 (1), (2017).
- Halloran, M. C., et al. Laser-targeted gene expression. Development. 127, 1953-1960 (2000).
- Shoji, W., Sato-Maeda, M. Application of heat shock promoter in transgenic zebrafish. Development, Growth & Differentiation. 50, 401-406 (2008).
- Luskin, M. B., Pearlman, A. L., Sanes, J. R. Cell lineage in the cerebral cortex of the mouse studied in vivo and in vitro with a Recombinant Retrovirus. Neuron. 1 (8), 635-647 (1988).
- Price, J., Thurlow, L. Cell lineage in the rat cerebral cortex: a study using retroviral-mediated gene transfer. Development. 104, 473-482 (1988).
- Walsh, C., Cepko, C. L. Widespread dispersion of neuronal clones across functional regions of the cerebral cortex. Science. 255 (5043), 434-441 (1992).
- Walsh, C., Cepko, C. L. Clonal dispersion in proliferative layers of developing cerebral cortex. Nature. 362 (6421), 632-635 (1993).
- Cai, L., Hayes, N. L., Nowakowski, R. S. Synchrony of Clonal Cell Proliferation and Contiguity of Clonally Related Cells: Production of Mosaicism in the Ventricular Zone of Developing Mouse Neocortex. The Journal of Neuroscience. 17 (6), 2088-2100 (1997).
- Reznikov, K., Acklin, S. E., Van Der Kooy, D. Clonal Heterogeneity in the Early Embryonic Rodent Cortical Germinal Zone and the Separation of Subventricular From Ventricular Zone Lineages. Developmental Dynamics. 210, 328-343 (1997).
- Qian, X., Goderie, S., Shen, Q., Stern, J., Temple, S. Intrinsic programs of patterned cell lineages in isolated vertebrate CNS ventricular zone cells. Development. 125, 3143-3152 (1998).
- McCarthy, M., Turnbull, D. H., Walsh, C. A., Fishell, G. Telencephalic Neural Progenitors Appear to Be Restricted to Regional and Glial Fates before the Onset of Neurogenesis. The Journal of Neuroscience. 21 (17), 6772-6781 (2001).
- Yu, Y. C., Bultje, R. S., Wang, X., Shi, S. H. Specific synapses develop preferentially among sister excitatory neurons in the neocortex. Nature. 458 (7237), 501-504 (2009).
- Nusslein-Volhard, C., Dahm, R. Zebrafish. , Oxford University Press. Oxford, UK. (2002).
- Nasiadka, A., Clark, M. D. Zebrafish Breeding in the Laboratory Environment. ILAR Journal. 53 (2), 161-168 (2012).
- Rosen, J. N., Sweeney, M. F., Mably, J. D. Microinjection of Zebrafish Embryos to Analyze Gene Function. Journal of Visualized Experiments. 25, e1115(2009).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- R Core Team. R: A language and environment for statistical computing. , (2017).
- Smith, M. R. Ternary: an R package to generate ternary plots. , (2017).
- Baye, L. M., Link, B. A. Development/Plasticity/Repair Interkinetic Nuclear Migration and the Selection of Neurogenic Cell Divisions during Vertebrate Retinogenesis. The Journal of Neuroscience. 27 (38), 10143-10152 (2007).
- Leung, L., Klopper, A. V., Grill, S. W., Harris, W. A., Norden, C. Apical migration of nuclei during G2 is a prerequisite for all nuclear motion in zebrafish neuroepithelia. Development. 139, 2635(2012).
- Kerr, J. F. R., Wyllie, A. H., Curriet, A. R. Apoptosis: A Basic Biological Phenomenon with Wide-ranging Implications in Tissue Kinetics. British Journal of Cancer. 26, 239(1972).
- Cole, L. K., Ross, L. S. Apoptosis in the Developing Zebrafish Embryo. Developmental Biology. 240, 123-142 (2001).
- Liao, J., He, J., Yan, T., Korzh, V., Gong, Z. A Class of NeuroD-Related Basic Helix-Loop-Helix Transcription Factors Expressed in Developing Central Nervous System in Zebrafish. DNA and Cell Biology. 18 (4), 333-344 (1999).
- Trevarrow, B., Marks, D. L., Kimmel, C. B. Organization of hindbrain segments in the zebrafish embryo. Neuron. 4 (5), 669-679 (1990).
- Yabu, T., Todoriki, S., Yamashita, M. Stress-induced apoptosis by heat shock, UV and gamma-ray irradiation in zebrafish embryos detected by increased caspase activity and whole-mount TUNEL staining. Fisheries Science. 67, 333-340 (2001).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados