Method Article
Visualisierung des sich entwickelnden Gehirns bei lebenden Zebrafischen mit Brainbow und Zeitraffer-Konfokalbildnis
In diesem Artikel
Zusammenfassung
In vivo Imaging ist ein leistungsfähiges Werkzeug, das verwendet werden kann, um die zellulären Mechanismen zugrunde liegenden Entwicklung des Nervensystems zu untersuchen. Hier beschreiben wir eine Technik zur Verwendung von Zeitraffer-Konfokalmikroskopie, um eine große Anzahl von mehrfarbigen Brainbow-markierten Zellen in Echtzeit innerhalb des sich entwickelnden Zebrafischnervensystems zu visualisieren.
Zusammenfassung
Die Entwicklung des Wirbeltiernervensystems erfordert eine präzise Koordination komplexer zellulärer Verhaltensweisen und Wechselwirkungen. Der Einsatz hochauflösender in vivo-Bildgebungstechniken kann ein klares Fenster in diese Prozesse im lebenden Organismus geben. Zum Beispiel können zellteilungsmittelteilungende Zellen und ihre Nachkommen in Echtzeit verfolgt werden, wenn sich das Nervensystem bildet. In den letzten Jahren haben technische Fortschritte in multicolor-Techniken die Arten von Fragen erweitert, die untersucht werden können. Der mehrfarbige Brainbow-Ansatz kann nicht nur verwendet werden, um zwischen gleichartigen Zellen zu unterscheiden, sondern auch mehrere verschiedene Klone verwandter Zellen, die jeweils aus einer Vorläuferzelle stammen, zu kodieren. Dies ermöglicht eine Multiplex-Linienanalyse vieler verschiedener Klone und deren Verhalten gleichzeitig während der Entwicklung. Hier beschreiben wir eine Technik zur Verwendung von Zeitraffer-Konfokalmikroskopie, um eine große Anzahl von mehrfarbigen Brainbow-markierten Zellen in Echtzeit innerhalb des sich entwickelnden Zebrafischnervensystems zu visualisieren. Dies ist besonders nützlich für die Verfolgung zellulärer Wechselwirkungen zwischen ähnlichen Zellen, die mit traditionellen, von Einem Promoter angetriebenen Farben nur schwer differenziell zu kennzeichnen sind. Unser Ansatz kann verwendet werden, um Linienbeziehungen zwischen mehreren verschiedenen Klonen gleichzeitig zu verfolgen. Die großen Datensätze, die mit dieser Technik generiert werden, liefern umfangreiche Informationen, die quantitativ über genetische oder pharmakologische Manipulationen hinweg verglichen werden können. Letztlich können die generierten Ergebnisse helfen, systematische Fragen zu beantworten, wie sich das Nervensystem entwickelt.
Einleitung
In den frühen Entwicklungsphasen teilen sich Pools spezialisierter Vorläuferzellen wiederholt in proliferativeZonen und produzieren verschiedene Arrays von Tochterzellen. Die Zellen, die während dieser Entwicklungsperiode geboren werden, werden sich dann differenzieren und reisen, um die entstehenden Organe zu bilden. Im Nervensystem führen Vorläufer wie radiale Glia zu unreifen Neuronen in ventrikulären Zonen. Wenn Neuronen von Ventrikeln wegwandern und reifen, bildet das expandierende Gewebe schließlich die hochkomplexen Strukturen des Gehirns1,2,3,4,5,6. Die Koordination zwischen Derliganundierung und Differenzierung und Migration von Neuronen wird die endgültige Größe, Form und damit Funktion des Gehirns bestimmen, direkt beeinflussenverhalten7,8,9,10. Während eine strenge Kontrolle über diese Prozesse eindeutig entscheidend für die normale Gehirnentwicklung ist, sind die globalen Mechanismen, die diese Dynamik regulieren, nicht gut verstanden. Hier beschreiben wir ein Werkzeug, um die Entwicklung des Nervensystems mit einer zellulären Auflösung zu untersuchen, so dass Forscher Progenitorzellen und Neuronen in vivo im sich entwickelnden Zebrafischhirn mit Brainbow visualisieren und ihr Verhalten im Laufe der Zeit über die Zeitraffer-Konfokalmikroskopie11verfolgen können. Der Ansatz kann auch angepasst werden, um andere Teile des sich entwickelnden Embryos zu visualisieren.
Um zellenimimim entwickelnden Zebrafischhirn zu beobachten und zu unterscheiden, haben wir die Brainbow-Zellbeschriftungstechnik11angepasst. Brainbow verwendet die zufällig ermittelte, kombinatorische Expression von drei verschiedenen fluoreszierenden Proteinen (FPs), um eine Population von Zellen zu kennzeichnen. Während die Standardexpression für brainbow Expression die rote FP dTomato ist, führt die Rekombination durch das Enzym Cre recombinase zur Expression von mCerulean (Cyan fluoreszierendes Protein, CFP) oder gelbem fluoreszierendem Protein (YFP)12,13. Die kombinierte Menge jedes FP, die in einer Zelle ausgedrückt wird, verleiht ihm einen einzigartigen Farbton, der eine klare visuelle Unterscheidung von benachbarten Zellen ermöglicht. Wenn sich eine Vorläuferzelle teilt, erbt jede Tochterzelle die Farbe von ihrer Mutterzelle, erzeugt farbcodierte Klone und ermöglicht es Forschern, die Zelllinie11,14nachzuverfolgen. Während ursprünglich verwendet, um neuronale Schaltkreise bei Mäusen12zu analysieren, Brainbow wurde seitdem in einer Vielzahl von Modellorganismen exprimiert, einschließlich Zebrafisch15.
Unsere Technik baut auf früheren mehrfarbigen Etikettierungs- und Bildgebungsmethoden auf, um mehrere farbcodierte Klone im Laufe der Zeit direkt in lebenden Zebrafischen abzubilden. Aufgrund ihrer optischen Transparenz als Embryonen eignen sich Zebrafische gut für bildgebende Experimente16, und frühere Studien haben Brainbow in Zebrafischen verwendet, um eine Vielzahl von Geweben zu studieren, einschließlich des Nervensystems11,15,17,18,19,20,21,22,23,24,25, 26,27. Die Fähigkeit, sich direkt in den lebenden Organismus einbilden zu können, zusammen mit ihrer schnellen Ex-Utero-Entwicklung, machen Zebrafische zu einem wertvollen Modell der Wirbeltierentwicklung. Im Gegensatz zum Säugetierhirn ist die gesamte proliferative Zone des Zebrafisch-Hinterhirns leicht für die Bildgebung ohne Störung seiner endogeneUmgebung6 verfügbar. Dies ermöglicht Experimente im lebenden Organismus, anstatt in vitro oder festen Gewebepräparaten. Im Gegensatz zu festen bildgebenden Experimenten ermöglichen In-vivo-Studien ein Längsdesign, das stundenlange Datenergebnisse erzeugt, die auf Muster analysiert werden können, wodurch die Wahrscheinlichkeit erhöht wird, relativ seltene Ereignisse zu beobachten. Je nach Geschwindigkeit und Länge der Ereignisse von Interesse können die Forscher kurze (1-2 h) oder lange (bis zu 16 h) Zeitraffer-Bildgebungsexperimente durchführen. Durch die Verwendung des Zebrafisch-Wärmeschockpromotors 70 (hsp70, hsp) kann der Brainbow-Ausdruck zeitlich gesteuert werden28,29. Darüber hinaus eignet sich der von diesem Promotor induzierte Mosaikausdruck gut zum Etikettieren und Verfolgen vieler Klone11.
Die Fähigkeit, mehrere Klone im lebenden Gehirn visuell zu identifizieren, ist ein Vorteil dieser Methode. Wichtige frühere Studien, die die Rolle von Klonen in der Entwicklung des Nervensystems untersuchten, nutzten retrovirale Vektoren, um eine einzelne Vorläuferzelle und ihre Nachkommen mit einem einzigen FP oder einem anderen leicht visualisierten Protein zu kennzeichnen. Eine solche Kennzeichnung ermöglicht es Forschern, einen einzelnen Klon im Laufe der Zeit zu beobachten, entweder in vitro oder in vivo2,30,31,32,33,34,35,36,37,38. Im Gegensatz zu Methoden, um das Verhalten von Zellen innerhalb eines Klons zu verfolgen, ermöglichen die unterschiedlichen Farben von Brainbow forschern, die Dynamik zwischen Klonen zu beobachten. Darüber hinaus werden durch die Verwendung von Brainbow, um viele Klone im Gehirn zu kennzeichnen, zusätzliche Daten über das klonale Verhalten im Verhältnis zu Techniken gesammelt, die einen einzelnen Klon11kennzeichnen. Wichtig ist, dass die hier beschriebenen Ansätze erweitert werden können, um Entwicklungsvergleiche zwischen Fischen zu erzeugen, die unterschiedliche genetischen oder pharmakologischen Manipulationen unterzogen wurden18. Insgesamt machen diese Vorteile die Zeitraffer-in-vivo-Konfokalbildgebung von Brainbow-exprimierenden Zebrafischen ideal für Forscher, die die Entwicklung des Wirbeltiernervensystems erforschen, insbesondere für diejenigen, die an der Rolle von Klonen interessiert sind.
Protokoll
Verfahren, an denen tiertierische Personen beteiligt sind, wurden vom Institutional Animal Care and Use Committee (IACUC) am Lewis & Clark College genehmigt.
1. Mikroinjektion von Zebrafisch-Embryonen
- Richten Sie wilde Art, erwachsene Zebrafische in sex-getrennten Paarungtanks am Nachmittag vor der Durchführung von Mikroinjektionen39,40.
- Bereiten Sie die DNA-Lösung am Morgen der Mikroinjektionen vor. Verdünnung hsp:Zebrabow11 Plasmid-DNA in einer Konzentration von 10 ng/l in 0,1 mM KCl, zusammen mit 2,5% Phenolrot und 3,75U Cre Rekombinantase-Enzym.
- Führen Sie Mikroinjektionen von DNA-Lösung in einzellige Zebrafisch-Embryonen innerhalb von 45 min der Befruchtung39,41. Injizieren Sie etwa 4,2 nL dieser Lösung in jeden Embryo, was 42 pg Plasmid-DNA entspricht.
HINWEIS: Der Mikroinjektionsschritt kann weggelassen werden, wenn stattdessen eine transgene, Brainbow-extierende Zebrafischlinie gepaart wird, wie Tg(ubi:Zebrabow)15 und Tg(neurod:Zebrabow)18. Die Durchführung von Mikroinjektionen kann von Vorteil sein, da es Forschern ermöglicht, die Kopiernummer und DieBeschriftungsdichte zu tittieren. Darüber hinaus kann ein zweites DNA-Konstrukt, das ein bestimmtes Genprodukt markiert oder manipuliert, auf Wunsch neben Brainbow injiziert werden (z. B. ein weitrotes fluoreszierendes Protein, das Brainbow18ergänzt). - Injizierte Embryonen in Petrischalen von E3 medium39 in einem 28 °C-Inkubator für 24 h aufbewahren.
2. Hitzeschock, um Brainbow Expression zu induzieren
HINWEIS: Wenn die injizierte Plasmid-DNA oder das transgene Exprimierte den hsp70-Promotor nicht zum Antrieb der Expression verwendet, kann der Hitzeschockschritt übersprungen werden, und gesunde Embryonen sollten sofort bei 24 h nach der Befruchtung (hpf) auf Phenylthiourea (PTU) übertragen werden.
- Bei 24 hpf, keulen tote und deformierte Embryonen aus der Gruppe der injizierten Embryonen. Übertragen Sie dann die gesunden Embryonen in ein 50 ml-Rohr mit bis zu 20 Embryonen/Rohr.
- Füllen Sie die Rohre mit 10 ml E3. Legen Sie eine Kappe auf jede Röhre, aber nicht fest schließen.
- Legen Sie das Rohrträger mit den 50 ml Rohren aufrecht in ein 37 °C Wasserbad. Stellen Sie sicher, dass der Wasserstand im Bad höher ist als der Niveau des E3 in den Rohren und lassen Sie sie für 80 bis 90 min.
- Entfernen Sie das Rohrgestell mit den Embryonen aus dem Wasserbad und kehren Sie aufrecht zum 28 °C-Inkubator zurück. Lassen Sie bis zu 1 h für die E3 abkühlen und die Embryonen allmählich wieder auf die Temperatur zu akklimatisieren. Dann übertragen Sie die Embryonen auf Petrischalen von 0,2 mM PTU in E3 erwärmt im Inkubator, um Pigmentierung von Embryonen zu verhindern.
ACHTUNG: PTU ist eine giftige Chemikalie, die mit Sorgfalt behandelt und entsprechend entsorgt werden sollte. Es wird empfohlen, Handschuhe zu tragen.
3. Screening-Embryonen für Brainbow Expression
- Untersuchen Sie die Embryonen nach dem Hitzeschock 2 bis 4 h nach dem Hitzeschock unter einem Standardfluoreszenz-Dissektionsmikroskop auf expression von GFP oder YFP, was auf eine erfolgreiche Brainbow-Rekombination (von der Standardexpression von dTomato) hinweist.
HINWEIS: Wenn mehrere Fluoreszenzfilteroptionen verfügbar sind, ist das Screening auf CFP die effizienteste Möglichkeit, gut rekombinierte Fische zu identifizieren. Wenn GFP- und YFP-Filter nicht verfügbar sind, kann YFP aufgrund der Überlappung in ihren Spektralprofilen auch unter GFP-Filtern (Green Fluorescent Protein) nur wenig visualisiert werden. - Wählen Sie Embryonen mit robuster FP-Expression durch und übertragen Sie sie auf eine separate Schale mit PTU. Bild-Embryonen bei 1 Tag nach der Befruchtung (dpf; 28 hpf oder höher) oder pflegen Embryonen in PTU und Bild bei 2 oder 3 dpf.
4. Montage von Embryonen für In Vivo Imaging
-
Bereiten Sie vor dem Tag des Experiments die Bildgebungskammer und das Werkzeug zur Embryonenmanipulation vor.
- Bereiten Sie die Bildkammer vor, indem Sie einen Kunststoffring sorgfältig in die Mitte einer 60 mm Petrischale überkleben.
- Optional bereiten Sie einen maßgeschneiderten Embryomanipulator vor, um Embryonen innerhalb der Schale zu bewegen und Embryonen während der Montage zu orientieren. Konstruieren Sie den Manipulator, indem Sie eine kleine Länge der Nylon-Angelschnur (1,5 000, 6 lb) bis zum Ende eines hölzernen Wattestäbchens, der auf eine Länge von 4 Dollar geschnitten wurde, überkleben.
- Falls erforderlich (d.h. Bildgebung bei 2 dpf oder früher), bevor die Embryonen mit Spritzen unter einem Seziermikroskop montiert werden.
-
Anästhesisieren Zebrafisch Embryonen.
- Füllen Sie eine 60 mm Petrischale auf halbem Weg mit E3 und fügen Sie 5–6 Tropfen von 4 mg/ml MS-222 Tricaine-S zu einer Endkonzentration von 0,2 mM hinzu. Wirbel nieren.
ACHTUNG: Tricaine sollte mit Sorgfalt behandelt und entsprechend entsorgt werden. - Übertragen Sie Embryonen von der PTU auf die Tricain-Lösung, um sicherzustellen, dass so wenig PTU wie möglich übertragen wird. Während Fischreflexe nach 1–2 min aufhören sollten, lassen Sie die Embryonen in Tricain bis zu 10 min, um eine gründliche Anästhesie für Zeitraffer-Bildgebungsexperimente zu gewährleisten.
- Bestätigen Sie, dass die Embryonen richtig anästhesisiert sind. Bewerten Sie den Erdreflex, indem Sie die Embryoschwänze sanft mit dem Embryomanipulator berühren. Wenn eine Reflexbewegung beobachtet wird, fügen Sie der Schale einen zusätzlichen Tropfen Tricain so weit wie möglich von den Embryonen hinzu und wirbeln Sie zum Mischen. Beobachten Sie die Embryo-Herzfrequenz unter einem Sezierbereich. Wenn es ungewöhnlich langsam ist, fügen Sie zusätzliche Tropfen E3 in die Schale, um die Tricain-Konzentration zu reduzieren.
- Füllen Sie eine 60 mm Petrischale auf halbem Weg mit E3 und fügen Sie 5–6 Tropfen von 4 mg/ml MS-222 Tricaine-S zu einer Endkonzentration von 0,2 mM hinzu. Wirbel nieren.
-
Mount anästhesierte Embryonen in Agarose.
- Übertragen Sie den Fisch in die Mitte des Kunststoffrings innerhalb der Bildkammer und entfernen Sie so viel überschüssiges E3 wie möglich mit einer feingekippten Transferpipette.
- Mit einer sauberen Transferpipette die Fische mit 1,0% schmelzarmer Agarose (LMA) in E3 bei 42 °C lagern und den gesamten Kunststoffring mit einer dünnen Agaroseschicht füllen. Verwenden Sie eine Transferpipette, um die Embryonen sanft in die Pipettespitze und zurück in die Agarose zu ziehen, ohne Luftblasen einzuführen.
- Bevor die Agarose verhärtet, verwenden Sie schnell einen Embryo-Manipulator, um den Fisch angemessen auszurichten. Wenn Sie die Bilder mit einem aufrechten Mikroskop abbilden, positionieren Sie die Embryonen so nah wie möglich an der oberen Oberfläche der Agarose. Positionieren Sie die Embryonen parallel zur Unterseite der Bildkammer mit dem Geraden des Schwanzes.
HINWEIS: Für die Hinterhirnbildgebung können die Embryonen in einer dorsalen oder sagittalen Ansicht positioniert werden. Es sollte darauf geachtet werden, dass die Köpfe der Embryonen nicht sinken, während die Agarose noch flüssig ist. Andere Orientierungen können geeignet sein, andere sich entwickelnde Gewebe für die Bildgebung zu zielen. Bei invertierten Mikroskopen würde die Montage anders erfolgen, da der Fisch in der Regel in der Nähe des Bodens einer Glasboden-Petrischale positioniert wird.
- Warten Sie, bis die Agarose aushärtet, und füllen Sie dann die Bildkammer mit E3. Fügen Sie so viel E3 wie möglich hinzu, um die Verdunstung im Verlauf der Bildgebung zu berücksichtigen.
5. Zeitraffer Konfokalbildgebung der Entwicklung zebrafish Hindbrain
-
Legen Sie die Bildgebungskammer in Vorbereitung auf die Bildgebung auf das konfokale Mikroskop.
- Legen Sie die Bildkammer mit dem Fisch und E3 auf das konfokale Mikroskop.
- Wählen Sie ein Ziel mit einer hohen numerischen Blende und einem langen Arbeitsabstand, z. B. ein 20-faches Wassertauchziel (1,0 NA).
- Suchen Sie einen Bereich mit entsprechend dichter und heller Beschriftung des/der Zelltyps von Interesse.
HINWEIS: Ein temperaturgeregeltes Stadium/Gerät am Mikroskop kann auch verwendet werden, um Temperatur und Luftfeuchtigkeit während langer Bildgebungssitzungen zu regulieren. Dies kann die Verdunstung verringern.
-
Richten Sie die Erfassungsparameter für die Brainbow-Bildgebung ein.
- Bildn jeder FP-Kanal sequenziell. Bei Verwendung einer kommerziellen Software (z.B. Zen-Software) erfolgt dies durch die Vorbereitung von drei "Tracks". Verwenden Sie einen Argon-Laser, um CFP bei 458 nm und YFP bei 514 nm zu begeistern. Verwenden Sie einen DPSS 561 nm Laser, um dTomato zu begeistern. Sammeln Sie Emissionen zwischen 463–509 nm für CFP, 519–555 nm für YFP und 566–691 nm für dTomato.
- Für die Bildschirmanzeige code CFP als blau, YFP als gelb und dTomato als rot.
HINWEIS: Diese Einstellungen variieren je nachdem, welche Laserlinien und andere Funktionen auf dem konfokalen Mikroskop verfügbar sind. Um die Geschwindigkeit der Bildgebung zu erhöhen, können CFP- und dTomato-Kanäle gleichzeitig ohne nennenswerte Durchblutung abgebildet werden. Wenn auch ein separates FP abgebildet wird, z. B. ein rot-rotes FP, kann dies sequenziell oder gleichzeitig mit YFP abgebildet werden. - Richten Sie die allgemeinen Bildgebungsparameter ein, um 16-Bit-Bilder mit einer Auflösung von mindestens 1024 x 1024 und einer durchschnittlichen Gesamtauflösung von zwei Personen aufzunehmen. Passen Sie den Zoom abhängig von der Region und dem Zelltyp des Interesses an (d. h. für die Hinterhirnbildgebung mit einem 20-fachen Objektiv reicht der Zoom von 1,0 bis 2,5). Maximieren Sie das Sichtfeld, um das Wachstum der Fische im Laufe der Zeit zu ermöglichen.
- Anzeigen einer Spur nach der anderen (und Ausschalten anderer Laser, um Photobleichungen zu verhindern), optimieren Sie die Erfassungseinstellungen für jedes FP einzeln (z. B. Laserleistung, Lochgröße, Photomultiplierrohrverstärkung usw.).
- Wählen Sie Z-Stack-Bereich zum Abbild aus.
HINWEIS: Bei langen Bildgebungssitzungen (>2 h) sollten Sie berücksichtigen, dass die Fische im Laufe der Bildgebung weiter wachsen werden, so dass sowohl das XY-Feld als auch der Z-Stack-Bereich dies mit zusätzlichem Raum berücksichtigen sollten. Wenn die Abbildung des Hinterhirns, enthalten Raum im Feld für das Gewebe rostral während der Bildgebungsphase zu bewegen. Auch für das Wachstum in der Z-Dimension muss Der Raum einbezogen werden. Fügen Sie in das Bild ca. 10–20 m über dem Interessengebiet ein, unabhängig davon, ob der Fisch für sagittale oder dorsale Sicht positioniert ist.
-
Richten Sie Parameter für die Zeitraffer-Bildgebung ein.
- Wählen Sie ein Zeitintervall aus. Intervalllänge reicht zwischen 10 und 30 min, um mitotische und apoptotische Ereignisse im sich entwickelnden Hinterhirn zu verfolgen. Die Intervalllänge hängt von der Geschwindigkeit der Ereignisse ab, die von Interesse sind, und der Gesamtzeit, die zum Erfassen eines einzelnen Z-Stacks benötigt wird.
- Wählen Sie die Länge der Imaging-Sitzung aus. Zeitraffer-Bildgebungssitzungen treten in der Regel über Nacht auf und können mindestens 16 h dauern.
-
Führen Sie das Experiment aus.
HINWEIS: Der Fisch kann unmittelbar nach der Bildgebung eingeschläfert oder vorsichtig von der Agarose mit Spritzen seziert und zur Genesung an E3 zurückgegeben werden. Zurückgewonnene Fische können dann zu einem späteren Zeitpunkt wieder abgebildet werden, obwohl sich Die Zellposition und -tiefe während dieses Zeitraums aufgrund des Gesamtwachstums verschieben wird.
6. Zeitraffer-Dateiverwaltung
-
Importieren Sie die Bilddatei in die Analysesoftware.
- Wenn Sie Zen-Software verwenden, speichern Sie die Datei im .czi-Format, sobald die Bildaufnahme abgeschlossen ist. Stellen Sie sicher, rohdaten in einem Format zu speichern, das mit Fidschi42 und/oder anderer Software kompatibel ist.
- Importieren Sie in Fidschi-Software mit BioFormats Importer.
HINWEIS: Zeitrafferdateien sind groß und können erhebliche Zeit- und Arbeitsspeichermengen erfordern, um sie für die Analyse zu öffnen. Daher ist es hilfreich, strategisch zu sein, wie sie gespeichert und geöffnet werden. Es kann nützlich sein, eine version mit geringerer Qualität zu speichern, die leichter geöffnet werden kann, um nach Ereignissen von Interesse per Auge zu suchen.
- Wenn Sie Fidschi verwenden, erstellen Sie kleinere, leichter verwaltbare Teilmengen von Zeitrafferdateien. Generieren Sie Teilmengen entweder nach Zeit (z. B. Anzeigen des vollständigen Z-Stacks beim ersten Zeitpunkt) oder nach Tiefe (z. B. Anzeigen der ersten 50 Z-Abschnitte zu jedem Zeitpunkt), indem Sie auf Bild| Stapel| Werkzeuge| Machen Sie Substack.
7. Quantitative Clonal Color Analysis of Brainbow Images
- Messen Sie die mittleren roten, grünen und blauen (RGB) Intensitätswerte für die Zellen, die in Fidschi von Interesse sind.
HINWEIS: Diese Anweisungen sind spezifisch für die Bildanalyse in Fidschi. Jedoch, Forscher können es vorziehen, mittlere RGB-Intensitätswerte in einem alternativen Software-Programm zu erhalten.- Stellen Sie sicher, dass die Datei korrekt zugeschnitten ist, sodass an den Rändern des Bildes kein schwarzer Raum vorhanden ist. Schwarzer Raum senkt die für die Analyse erforderlichen Minimalintensitätsmessungen künstlich. Wenn die Datei zugeschnitten werden muss, wählen Sie die Rechteckauswahlschaltfläche aus, und zeichnen Sie einen Bereich von Interesse (ROI) um das Sichtfeld, ohne den gesamten schwarzen Raum. Klicken Sie auf Bild| Ernte.
- Legen Sie das Messwerkzeug so fest, dass Mittelwert-, Minimal- und maximaler Grauwert gemessen werden. Klicken Sie auf Analysieren| Stellen Sie Messungen ein. Stellen Sie sicher, dass die Kontrollkästchen für Min & Max Gray Value und Mean Gray Value ausgewählt sind.
- Anzeigen der Rohdatendateien oder subsetted Z-Stacks in Fidschi, finden Sie Zellen von Interesse für klonale Farbanalyse. Im Hinterhirn können vermeintliche Klone visuell durch ihren gemeinsamen Farbton sowie ihre radiale Ausrichtung identifiziert werden. Wählen Sie keine Zellen aus, die in engem Kontakt mit benachbarten Zellen einer anderen Farbe stehen und daher schwer aufzulösen sind. Es kann schwierig sein, ihren Farbton zu quantifizieren.
- Mit der Z-Scrollbarfinden Sie die mittlere Z-Ebene der Zelle von Interesse. Klicken Sie mit der rechten Maustaste auf die Schaltfläche Ovale Auswahl, und wählen Sie die Schaltfläche "Elliptische Auswahl"aus. Andere Auswahlwerkzeuge sind für verschiedene Zelltypen geeignet. Zeichnen Sie einen elliptischen ROI um die zentrale Nr. 2/3 des Zellkörpers.
- Wählen Sie mit der C-Scrollbarden roten (dTomato) Kanal aus. Nehmen Sie die mittlere Intensitätsmessung für diesen Kanal, indem Sie Analysieren| Maßnahme. Wiederholen Sie dies mit demselben ROI und der gleichen Fokusebene für die grünen (YFP) und blauen (CFP) Kanäle und speichern Sie alle mittleren Intensitätswerte.
- Klicken Sie auf eine beliebige Stelle auf dem Bild, um den elliptischen ROI in der betreffenden Zelle zu entfernen.
- Um Hintergrundebenen für die Normalisierung zu messen, verwenden Sie die C-Scrollbar und wählen Sie den roten (dTomato) Kanal aus. Nehmen Sie die minimale Intensitätsmessung für das gesamte Feld in diesem Kanal, indem Sie Analysieren| Maßnahme. Wiederholen Sie dies mit derselben Brennweite für die grünen (YFP) und blauen (GFP) Kanäle, und speichern Sie alle Minimalintensitätswerte.
- Berechnen Sie relative RGB-Kanalgewichtungen für die von Interesse sindden Zellen.
HINWEIS: Die Berechnung der relativen RGB-Kanalgewichtungen aus diesen Intensitätswerten kann in einer Vielzahl von Softwareprogrammen wie Microsoft Excel oder R43durchgeführt werden.- Normalisieren Sie die mittlere Intensitätsmessung des Zell-ROI für jeden Kanal, indem Sie die minimale Intensität vom gesamten Feld in diesem Kanal und der Brennebenen subtrahieren.
- Summiert die normalisierten mittleren RGB-Intensitätswerte aus jedem Kanal, um die gesamte normalisierte RGB-Intensität für die Zelle zu ermitteln.
- Berechnen Sie die relative Kanalgewichtung für jeden Kanal, indem Sie den normalisierten mittleren Intensitätswert für diesen Kanal durch die gesamte normalisierte RGB-Intensität dividieren.
- Relative RGB-Kanalgewichtungen als ternäre Plots mit dem Ternary-Paket für R43,44anzeigen. Ternäre Plots sind nützlich, um das Clustering ähnlicher Farben im 3D-RGB-Raum zu visualisieren.
- Berechnen Sie den Farbstreukoeffizient, um die Differenz zwischen den Zellenfarben zu quantifizieren.
- Berechnen Sie den euklidischen Abstand zwischen den beiden Farben im 3D-RGB-Raum mit der folgenden Gleichung:
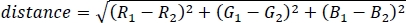
wobei R, G und B die relativen RGB-Kanalgewichtungen sind, die in 7.2 berechnet werden. - Um das Verständnis zu erleichtern, normalisieren Sie den Farbabstand, indem Sie den maximal möglichen Abstand zwischen den Farben durch den maximal möglichen Abstand zwischen den Farben dividieren, um einen Farbstreukoeffizienten zwischen 0 und 1 zu erhalten, wobei 0 genau die gleiche Farbe und 1 völlig unterschiedliche Farben anzeigt (z. B. rein rot und reinblau).
- Berechnen Sie den euklidischen Abstand zwischen den beiden Farben im 3D-RGB-Raum mit der folgenden Gleichung:
Ergebnisse
In diesem Abschnitt werden Beispiele für Ergebnisse veranschaulicht, die mit dem hier beschriebenen mehrfarbigen Zeitraffer-Bildgebungsansatz in vivo erzielt werden können. Wir zeigen, dass Brainbow farbcodierte Klone von Zellen in der proliferativen ventrikulären Zone des sich entwickelnden Zebrafisch-Hinterhirns14 (Abbildung 1).
Wenn Brainbow-markierte Zellen entlang einer bestimmten radialen Faser angeordnet waren, teilten sie in der Regel dieselbe Farbe (Abbildung 1D), die wie die relativen RGB-Kanalgewichtungen quantifiziert werden konnte (Abbildung 1E). Dies deutet darauf hin, dass diese radialen Gruppen Klone von teilenden Zellen waren und dass ihre ähnliche Farbe verwendet werden könnte, um sie als klonal verwandt zu identifizieren, wie in früheren Studien an Zebrafischen, Mäusen und Küken14,15beobachtet wurde. Wenn die Brainbow-Etikettierungsdichte relativ spärlich ist, kann diese Farbcodierung verwendet werden, um viele verschiedene radiale Klone innerhalb proliferativer Regionen des lebenden Wirbeltierhirns klar zu visualisieren und zu verfolgen.
Eine quantitative Farbanalyse zeigte, dass Tochterzellen die gleiche Farbe wie ihre Mutterzelle (Vorläufer) exprimierten (Abbildung 1F), aber dass benachbarte radiale Zellhaufen voneinander unterschieden werden können11 (Abbildung 1E). Dies bedeutet, dass zahlreiche Klone verwandter Zellen gleichzeitig über Stunden in vivo verfolgt werden können (Abbildung 2), was eine Multiplex-Linienanalyse und einen Vergleich ermöglicht. Die Quantifizierung des Farbausdrucks in Klonen bei 2 dpf und dann wieder bei 3 dpf (Abbildung 2A-B ) zeigte, dass derBrainbow-Ausdruckauch von 2–3 Tagen in vivo11relativ konstant blieb. Einzelne Zellen können so während der Entwicklung relativ lange in Echtzeit identifiziert und nachverfolgt werden.
Die Zeitraffer-Bildgebung zeigte zahlreiche Zellen, die sich in der Zeit von 1–2 dpf (Abbildung 2C–F)11einer interkinetischen Kernmigration und Zellteilung unterzogen haben, was eine Untersuchung des Zellzyklus ermöglicht. Mit einem Zeitrafferintervall von 30 min haben wir die mitotische Figur nicht immer während der Zellteilung erfasst. Dadurch wurde ein potenzieller Fehler von bis zu 30 min zur zugewiesenen Zeit der Zellteilung eingeführt. Zusätzlich beobachteten wir einige Klone, die zwei Vorläuferzellen enthielten (z. B. Abbildung 2F). Innerhalb dieser Einschränkungen kann die Länge des Zellzyklus gemessen werden. Durch die Verfolgung einzelner Brainbow-markierter Vorläufer im Zeitverlauf berechneten wir einen durchschnittlichen Zellzyklus von 8,4 x 1,5 h, vergleichbar mit früheren Messungen in Zebrafischen1,45,46. Darüber hinaus konnten wir mit der Brainbow-Zeitraffer-Bildgebungstechnik auch einzelne Zellen beobachten, die den stereotypen morphologischen Veränderungen im Zusammenhang mit Apoptose unterzogen wurden (Abbildung 3)11, wie Membranblessing und Zellfragmentierung47,48.
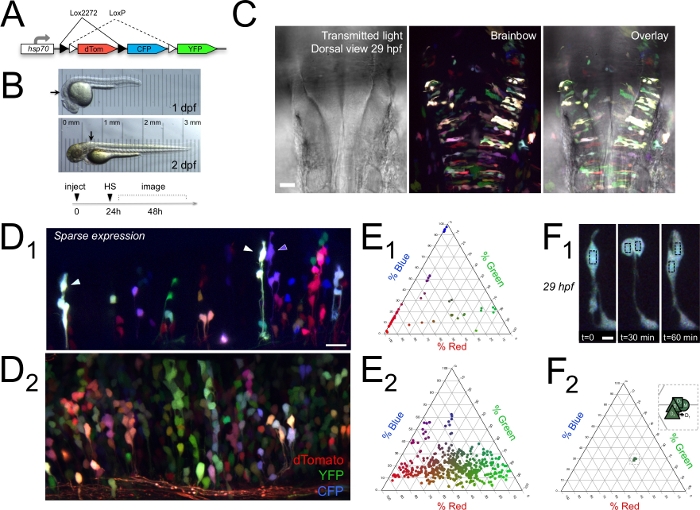
Abbildung 1: Brainbow beschriftet klonal verwandte Cluster von teilenden Zellen im sich entwickelnden Zebrafisch-Hinterhirn. (A) Schematische ssp:Zebrabow (Brainbow) DNA vorübergehend in Farbcode-Klone ausgedrückt. (B) In vivo übertragene Lichtbilder von sich entwickelnden Zebrafischen bei 1 und 2 dpf; Pfeile zeigen die allgemeine Hinterhirnregion an, die für die Bildgebung bestimmt ist. Die Transluzenz der Embryonen wird durch ein Mikrometer unter jedem Fisch nachgewiesen. Experimentelle Zeitleiste mit Injektionen im Einzellstadium, Hitzeschock (HS) bei 24 hpf und Bildgebung von 1–3 dpf. (C) Dorsale Ansicht von 29 hpf Zebrafisch Hinterhirn mit Brainbow gekennzeichnet. Der übertragene Lichtkanal zeigte die Morphologie des Hinterhirns und des Ventrikels, die mit einer maximalen Intensitätsprojektion von Brainbow-markierten Klonen überlagert wurden, die 165 m darstellen. Rostral ist oben. (D) In vivo Brainbow-Expression im Hinterhirn, dargestellt in maximalen Intensitätsprojektionen, die 41 m in spärlich beschrifteten 51-hpf-Zebrafischen (D1) und 81 m in 63,5-h-Zebrafischen (D2) darstellen. In beiden Paneelen ist dorsal oben und rostral ist auf der linken Seite. (E) Die Farbe der Zellen in D1 undD2 wurde als relative Kanalgewichte in entsprechenden ternären Diagrammen quantifiziert (n = 54 Zellen E1; 461 Zellen E2). (F) Die Zellfarbe blieb in Dendaughter-Zellen nach der Zellteilung relativ konstant. Reihe von Zeitpunkten, die in vivo mitotische Ereignisse im Hinterhirn von 29 hpf Brainbow-markierten Zebrafischen über 1 h zeigen (F1, maximale Intensitätsprojektionen, 30 m Tiefe). Die Farbe der Mutter- und Tochterzellen, angegeben durch die schwarzen Kästchen in F1, wird als relative Kanalgewichte im ternären Plot in F2dargestellt. Der Einset zeigt einen Zoom des gleichen Diagramms, um eng gruppierte Zellenfarben anzuzeigen (M ist Mutter; D1 und D2 sind Töchter bei 30 min/Quadrat und 60 min/Dreiecke). Skalenstäbe = 30 m (C), 20 m (D1), 16 m (D2) und 5 m (F1). In C, D und F ist dTomato rot, YFP als grün und GFP als blau codiert. Bilder mit Genehmigung von Brockway et al.11nachgedruckt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Zeitraffer-in-vivo-Bildgebung in Kombination mit Brainbow-Farbcodierung zeichnete mehrere benachbarte Klone von Zellen aus, die sich einer interkinetischen Kernmigration und -teilung unterziehen. (A) Farbkodierte Radialcluster, die im Hinterhirn eines Fisches beschriftet sind, der Brainbow bei 2 dpf (55 hpf; A1) und 3 dpf (71 hpf; A2); maximale Intensitätsprojektionen, die 62 m bzw. 47 m ausmachen. Drei farbcodierte Radialcluster werden zu jedem Zeitpunkt mit Pfeilspitzen identifiziert. (B) Die Farbe aller Brainbow-markierten Zellen in A1 undA2 wurde als relative Kanalgewichte in den entsprechenden ternären Diagrammen in B1 undB2 quantifiziert (n = 106 Zellen, 119 Zellen). (C) Zebrafisch-Hinterhirn bei 35 hpf mit Brainbow beschriftet (maximale Intensitätsprojektion mit 24 m); Inset, der mit gestricheltem weißen Kasten angezeigt wird, ist der erste Zeitpunkt, der in D. (D) Serie von Zeitpunkten angezeigt wird, die in 30 min Intervallen im Hinterhirn von C genommen werden. Es wird eine Periode von >9,5 h angezeigt. Die markierten Zellen wurden interkinetisch einer nuklearen Migration unterzogen; weiße Pfeilspitzen zeigen eine Zelle an der apikalen Oberfläche an. Das Aufrunden des Zellsoms an der apikalen Oberfläche und eine entsprechende Erhöhung der Klonzahl (z.B. rote Zelle bei 5,5 h und dann 8 h) wurde als mitotisches Ereignis betrachtet. (E) Dorsolaterale Ansicht des Zebrafisch-Hinterhirns bei 30,5 hpf mit Brainbow beschriftet, wobei die weiße gestrichelte und gepunktete Linie die ungefähre Grenze des Kopfes anzeigt und das weiße gestrichelte Feld den einweichenden Einset wert ist, der im ersten Zeitpunkt von F. (F) Serie von Zeitpunkten in 30 min Intervallen im Hinterhirn von E aufgenommen wurde. Über einen Zeitraum von 1,5 h konnten zwei blaue Zellen beobachtet werden, die von den weißen Pfeilspitzen angezeigt wurden und sich auf die apikale Oberfläche bewegten und sich einer Mitose unterziehen, was auf einen Klon hindeutet, der zwei Vorläuferzellen enthält. Skala bar = 20 'm (A), 25 'm (C) und 20 'm (F). In A-D ist dorsal oben und rostral ist auf der linken Seite. In E und F ist rostral auf der linken Seite. In allen Bildern ist dTomato rot, YFP als grün und CFP als blau codiert. Bilder mit Genehmigung von Brockway et al.11nachgedruckt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Die Zeitraffer-Brainbow-Bildgebung zeigt, dass Zellen in der ventrikulären Zone in vivo zelldeathieren. (A) Zebrafisch-Hinterhirn bei 31 hpf mit Brainbow beschriftet (maximale Intensitätsprojektion für 36 m). Weiße gestrichelte Box zeigt Inset im ersten Zeitpunkt in B. (B) Reihe von Zeitpunkten zeigt Hindbrain in A in 30 min Intervallen. Weiße Pfeilspitze im ersten Panel zeigt eine Lavendelzelle, die einer Apoptose unterzogen wurde, wie in den nachfolgenden Panels gezeigt, die durch Zellfragmentierung und anschließende allmähliche Clearance von apoptotischen Körpern angezeigt wird. Benachbarte markierte Zellen erschienen durchweg gesund. Dorsal ist oben und rostral ist auf der linken Seite. Skalenbalken = 20 m (B). In allen Bildern ist dTomato rot, YFP als grün und CFP als blau codiert. Bilder mit Genehmigung von Brockway et al.11nachgedruckt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Dieses Protokoll beschreibt eine Methode zur Visualisierung von Klonen von Vorläuferzellen und Neuronen im sich entwickelnden Zebrafisch-Hindbrain und folgt ihnen in vivo mit Brainbow und Zeitraffer-Konfokalmikroskopie11. Der große Vorteil dieses Protokolls im Vergleich zu In-vitro- oder Ex-vivo-Studien ist die Fähigkeit, die proliferative Zone des Wirbeltierhirns in seinem natürlichen Milieu im Laufe der Zeit direkt zu beobachten. Diese Technik baut auf früheren Studien auf, die einen einzelnen Klon mit retroviralen Vektoren kennzeichnen. Im Gegensatz dazu, die Verwendung der hsp:Zebrabow konstruieren Farbcodes viele Klone gleichzeitig, so dass Multiplex-Linienverfolgung und einen Fokus auf Dynamik unter den Klonen11. Dieses Protokoll kann geändert werden, um sich auf die Entwicklung in anderen Systemen oder Zelltypen zu konzentrieren. Beispielsweise können verschiedene Brainbow-Konstrukte in Zebrafischembryonen ausgedrückt werden. Verwendung des Zebrafischs hsp70 Promotor in hsp:Zebrabow wird in Mosaik-Etikettierung einer Vielzahl von Zelltypen nach Hitzeschock führen, einschließlich neuronale Vorläufer und Neuronen11, und gibt Forschern zeitliche Kontrolle über die Genexpression29. Es sollte beachtet werden, dass die Verwendung des Hitzeschock-Promotors farbcodierte Klone konsequent kennzeichnet, während andere Promotoren die Zelllinie auf diese Weise möglicherweise nicht klar abgrenzt. Wenn die klonale Identität kein Faktor von Interesse ist, können Konstrukte, die andere Promotoren verwenden, verwendet werden, um bestimmte Zelltypen zu kennzeichnen. Zum Beispiel kann der NeuroD-Promotor verwendet werden, um unreife Neuronen18,49zu kennzeichnen. Zusätzlich, anstatt Mikroinjektionen durchzuführen, Forscher können wählen, transgene Brainbow Zebrafische zu verwenden, einschließlich Linien wie Tg(ubi:Zebrabow)15 und Tg(neurod:Zebrabow)18. In diesem Fall entstehen Kreuze zu Linien, die Cre Recombinase exdrücken, wie Tg(hsp:Crea134)15, nicht injizierte Embryonen, die in ähnlicher Weise gesammelt und gepflegt werden. Während dieses Protokoll entworfen wurde, um Fische zwischen 1–3 dpf abzubilden, um mit der Hauptperiode der Entwicklungsneurogenese50zusammenzufallen, können Zebrafische je nach Entwicklungsstadium länger gepflegt werden. Hitzeschock, um Brainbow-Expression vor 24 hpf zu induzieren, kann jedoch für Embryonen schädlicher sein51. Je nach Alter und Gewebe sind unterschiedliche Montagestrategien geeignet, um das gezielte Gewebe richtig zu orientieren.
Es gibt eine Reihe von Schritten, die eine Fehlerbehebung erfordern können, insbesondere wenn Änderungen am Protokoll vorgenommen werden. Die Konzentration der injizierten DNA, der Bolusgröße und der Gesamtmenge der pro Embryo abgegebenen DNA kann angepasst werden, wenn die Brainbow-Expression schwach oder spärlich ist oder wenn Embryonen nicht gesund erscheinen. Zusätzlich können die Länge und das Timing des Hitzeschockschritts geändert werden, wenn der gewünschte Ausdruck nicht erreicht wird. Die erfassungsparameter, die in der Bildgebung verwendet werden, variieren je nach den verfügbaren Laserlinien und Filtern sowie der Helligkeit von Ausdruck, Montage und Art der gewünschten Analyse. Zusätzlich müssen die Zeitrafferparameter in Abhängigkeit von der Geschwindigkeit der Ereignisse von Interesse und dem Zeitbedarf für den Erwerb eines einzelnen Z-Stacks angepasst werden. Einer der wichtigsten Schritte im Protokoll ist die entsprechende Montage der Fische in Agarose: Wenn der Winkel des Fisches ungeeignet ist oder wenn der Fisch von zu viel Agarose bedeckt ist, ist eine optimale Bildgebung nicht möglich. Die Optimierung dieser Technik kann einige Übung nehmen. Darüber hinaus ist es oft nützlich, mehrere Fische vor der Bildgebung zu montieren, da es schwierig sein kann, festzustellen, ob die Montage angemessen ist, bevor die FP-Expression auf dem konfokalen Mikroskop selbst betrachtet wird. Ein weiterer kritischer Schritt ist das Screening und die Auswahl der zu bebildernden spezifischen Fische. Wenn Mikroinjektionen, Helligkeit, Beschriftungsdichte und Brainbow Kopiernummer können alle erheblich zwischen Fischen variieren. Bilder, die in Fischen mit schwacher Beschriftung, zu dichter Beschriftung oder einer geringen Kopierzahl aufgenommen wurden, die zu wenigen Farben führt, können schwieriger zu analysieren sein.
Mit diesem Protokoll konnten wir lebende Zebrafische bis 16 h11kontinuierlich abbilden. Diese Zeitspanne kann durch E3-Verdunstung aus der Bildkammer, das Wachstum der Fische aus der Ebene der Bildgebung, Tod oder Bewegung der Fische oder Benutzerbeschränkungen auf konfokaler Mikroskopzeit begrenzt werden. Die Verdunstung konnte durch den Einsatz eines temperaturgeregelten Zubehörs auf der Mikroskopstufe verringert werden. Es sollte beachtet werden, dass die 5D-Datasets, die mit diesem Ansatz generiert werden, in der Regel große Dateien sind, die erheblichen Festplattenspeicher erfordern (z. B. bis zu 100 GB für einen Zeitraffer über Nacht) und ausreichende Rechenleistung für die Analyse.
Dieses Protokoll führt Forscher, farbcodierte Klone innerhalb einer Population von neuronalen Vorläufern und Tochterneuronen im sich entwickelnden Zebrafischhirn zu visualisieren und ihre Dynamik im Laufe der Zeit zu verfolgen. Eine mögliche Erweiterung ist die Kombination von Brainbow-Zeitraffer-Bildgebung mit weit-rotem FP-Tagging, um die Rolle bestimmter Gene in der Entwicklung zu bewerten18. Auf diese Weise können Klone, die manipulierte Gene exemiten, in einer bestimmten Farbe visualisiert, im Laufe der Zeit verfolgt und mit Brainbow-markierten, nicht manipulierten benachbarten Klonen verglichen werden. In vivo Multicolor Imaging kann verwendet werden, um wichtige mechanistische Fragen darüber zu beantworten, wie sich das Nervensystem bildet und funktioniert.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Wir danken Y. A. Pan, J. Livet und Z. Tobias für ihre technischen und intellektuellen Beiträge. Diese Arbeit wurde von der National Science Foundation (Preis 1553764) und dem M.J. Murdock Charitable Trust unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 1.5 mL transfer pipette (fine tip) | Globe Scientific, Inc. | 134020 | |
| 1-phenyl-2-thiourea (PTU) | Alfa Aesar | L06690 | Diluted to 0.2 mM in E3 to prevent embryo pigmentation |
| 50 mL conical tubes | Corning | 352070 | For heat shocking embryos |
| 6 lb nylon fishing line | SecureLine | NMT250 | For making embryo manipulators |
| 7.5 mL transfer pipet | Globe Scientific, Inc. | 135010 | |
| CaCl2 | Sigma | C3881 | For E3 |
| Cotton swabs | Puritan | 867-WC NO GLUE | For making embryo manipulators |
| Cre recombinase | New England Biolabs | M0298M | |
| Digital dry bath | Genemate | 490016-616 | Used to store LMA at 42 °C |
| Epifluorescence dissection scope | |||
| Glass capillary tubes | World Precision Instruments | TW100F-4 | |
| Incubator | Forma Scientific | 3158 | To maintain embryos at 28 °C |
| Injection plate molds | Adaptive Science Tools | TU-1 | |
| Isotemp water bath | Fisher Scientific | 2320 | For heat shocking embryos |
| KCl | AMRESCO | 0395 | For E3 and for DNA solution for injections |
| Laser-scanning confocal microscope | Zeiss | LSM710 | |
| LE agarose | Genemate | E3120 | To create agarose injection plates |
| Low-melt agarose (LMA) | AMRESCO | J234 | |
| Mating tanks | Aquaneering, Inc. | ZHCT100 | |
| Methylene blue | Sigma | M9140 | For E3 |
| MgSO4 | Sigma | 9397 | For E3 |
| Micromanipulator | World Precision Instruments | M3301 | |
| Micropipette Puller | Sutter Instrument Co. | P-97 | |
| MS-222 Tricaine-S | Western Chemical, Inc. | Stock made at 4 mg/mL in reverse osmosis (RO) water, then added dropwise to E3 to final concentration of 0.2 mM to anesthetize embryos | |
| NaCl | J.T. Baker | 4058-01 | For E3 |
| Petri dishes (90 mm, 60 mm) | Genesee Scientific | 32-107G | To house embryos and create imaging chamber (60 mm) |
| Phenol red | Sigma | P0290 | |
| Soft stitch ring markers | Clover Needlecraft, Inc. | 354 | For creating imaging chamber with Petri dish |
| Super glue (Ultra gel control) | Loctite | 1363589 | For making embryo manipulators |
| Syringe needles | Beckton Dickinson | BD329412 | For dechorionating embryos |
Referenzen
- Lyons, D. A., Guy, A. T., Clarke, J. D. W. Monitoring neural progenitor fate through multiple rounds of division in an intact vertebrate brain. Development. 130, 3427-3436 (2003).
- Noctor, S. C., Flint, A. C., Weissman, T. A., Dammerman, R. S., Kriegstein, A. R. Neurons derived from radial glial cells establish radial units in neocortex. Nature. 409 (6821), 714-720 (2001).
- Kriegstein, A., Alvarez-Buylla, A. The glial nature of embryonic and adult neural stem cells. Annual Review of Neuroscience. 32, 149-184 (2009).
- Florio, M., Huttner, W. B. Neural progenitors, neurogenesis and the evolution of the neocortex. Development. 141, 2182-2194 (2014).
- Mcconnell, S. K. Constructing the Cerebral Cortex: Neurogenesis and Fate Determination Review. Neuron. 15, 761-768 (1995).
- Schmidt, R., Strähle, U., Scholpp, S. Neurogenesis in zebrafish - from embryo to adult. Neural Development. 8, 3(2013).
- Chenn, A., Walsh, C. Regulation of Cerebral Cortical Size by Control of Cell Cycle Exit in Neural Precursors. Science. 297 (5580), 365-369 (2002).
- Lui, J. H., Hansen, D. V., Kriegstein, A. R. Development and Evolution of the Human Neocortex. Cell. 146 (1), 18-36 (2011).
- Nonaka-Kinoshita, M., et al. Regulation of cerebral cortex size and folding by expansion of basal progenitors. The EMBO Journal. 32, 1817-1828 (2013).
- Homem, C. C. F., Repic, M., Knoblich, J. A. Proliferation control in neural stem and progenitor cells. Nature Reviews Neuroscience. 16 (11), 647-659 (2015).
- Brockway, N. L., et al. Multicolor lineage tracing using in vivo time-lapse imaging reveals coordinated death of clonally related cells in the developing vertebrate brain. Developmental Biology. 453 (2), 130-140 (2019).
- Livet, J., et al. Transgenic strategies for combinatorial expression of fluorescent proteins in the nervous system. Nature. 450 (7166), 56-62 (2007).
- Weissman, T. A., Pan, Y. A. Brainbow: New resources and emerging biological applications for multicolor genetic labeling and analysis. Genetics. 199 (2), 293-306 (2015).
- Loulier, K., et al. Multiplex Cell and Lineage Tracking with Combinatorial Labels. Neuron. 81 (3), 505-520 (2014).
- Pan, Y. A., et al. Zebrabow: multispectral cell labeling for cell tracing and lineage analysis in zebrafish. Development. 140, 2835-2846 (2013).
- Ko, S. K., Chen, X., Yoon, J., Shin, I. Zebrafish as a good vertebrate model for molecular imaging using fluorescent probes. Chemical Society Reviews. 40, 2120(2011).
- Kesavan, G., Hammer, J., Hans, S., Brand, M. Targeted knock-in of CreERT2 in zebrafish using CRISPR/Cas9. Cell and Tissue Research. 372, 41-50 (2018).
- Cook, Z. T., et al. Combining near-infrared fluorescence with Brainbow to visualize expression of specific genes within a multicolor context. Molecular Biology of the Cell. 30 (4), 491-505 (2019).
- Kuwata, M., Nikaido, M., Hatta, K. Local heat-shock mediated multi-color labeling visualizing behaviors of enteric neural crest cells associated with division and neurogenesis in zebrafish gut. Developmental Dynamics. 248, 437-448 (2019).
- Kinkhabwala, A., et al. A structural and functional ground plan for neurons in the hindbrain of zebrafish. Proceedings of the National Academy of Sciences. 108 (3), 1164-1169 (2011).
- Heap, L. A., Goh, C. C., Kassahn, K. S., Scott, E. K. Cerebellar Output in Zebrafish: An Analysis of Spatial Patterns and Topography in Eurydendroid Cell Projections. Frontiers in Neural Circuits. 7, 53(2013).
- Robles, E., Filosa, A., Baier, H. Precise Lamination of Retinal Axons Generates Multiple Parallel Input Pathways in the Tectum. The Journal of Neuroscience. 33 (11), 5027-5039 (2013).
- Dirian, L., et al. Spatial Regionalization and Heterochrony in the Formation of Adult Pallial Neural Stem Cells. Developmental Cell. 30, 123-136 (2014).
- Chen, X. A., et al. Behavioral/Cognitive QRFP and Its Receptors Regulate Locomotor Activity and Sleep in Zebrafish. The Journal of Neuroscience. 36 (6), 1823-1840 (2016).
- Albadri, S., De Santis, F., Di Donato, V., Del Bene, F. CRISPR/Cas9-Mediated Knockin and Knockout in Zebrafish. Genome Editing in Neurosciences. Jaenisch, R., Zhang, F., Gage, F. , Springer. Cham, Switzerland. 41-49 (2017).
- Furlan, G., et al. Life-Long Neurogenic Activity of Individual Neural Stem Cells and Continuous Growth Establish an Outside-In Architecture in the Teleost Pallium. Current Biology. 27, 3288-3301 (2017).
- Herget, U., Arturo Gutierrez-Triana, J., Salazar Thula, O., Knerr, B., Ryu, S. Single-cell reconstruction of oxytocinergic neurons reveals separate hypophysiotropic and encephalotropic subtypes in larval zebrafish Brainbow-guided morphology of oxytocinergic cells. eNeuro. 4 (1), (2017).
- Halloran, M. C., et al. Laser-targeted gene expression. Development. 127, 1953-1960 (2000).
- Shoji, W., Sato-Maeda, M. Application of heat shock promoter in transgenic zebrafish. Development, Growth & Differentiation. 50, 401-406 (2008).
- Luskin, M. B., Pearlman, A. L., Sanes, J. R. Cell lineage in the cerebral cortex of the mouse studied in vivo and in vitro with a Recombinant Retrovirus. Neuron. 1 (8), 635-647 (1988).
- Price, J., Thurlow, L. Cell lineage in the rat cerebral cortex: a study using retroviral-mediated gene transfer. Development. 104, 473-482 (1988).
- Walsh, C., Cepko, C. L. Widespread dispersion of neuronal clones across functional regions of the cerebral cortex. Science. 255 (5043), 434-441 (1992).
- Walsh, C., Cepko, C. L. Clonal dispersion in proliferative layers of developing cerebral cortex. Nature. 362 (6421), 632-635 (1993).
- Cai, L., Hayes, N. L., Nowakowski, R. S. Synchrony of Clonal Cell Proliferation and Contiguity of Clonally Related Cells: Production of Mosaicism in the Ventricular Zone of Developing Mouse Neocortex. The Journal of Neuroscience. 17 (6), 2088-2100 (1997).
- Reznikov, K., Acklin, S. E., Van Der Kooy, D. Clonal Heterogeneity in the Early Embryonic Rodent Cortical Germinal Zone and the Separation of Subventricular From Ventricular Zone Lineages. Developmental Dynamics. 210, 328-343 (1997).
- Qian, X., Goderie, S., Shen, Q., Stern, J., Temple, S. Intrinsic programs of patterned cell lineages in isolated vertebrate CNS ventricular zone cells. Development. 125, 3143-3152 (1998).
- McCarthy, M., Turnbull, D. H., Walsh, C. A., Fishell, G. Telencephalic Neural Progenitors Appear to Be Restricted to Regional and Glial Fates before the Onset of Neurogenesis. The Journal of Neuroscience. 21 (17), 6772-6781 (2001).
- Yu, Y. C., Bultje, R. S., Wang, X., Shi, S. H. Specific synapses develop preferentially among sister excitatory neurons in the neocortex. Nature. 458 (7237), 501-504 (2009).
- Nusslein-Volhard, C., Dahm, R. Zebrafish. , Oxford University Press. Oxford, UK. (2002).
- Nasiadka, A., Clark, M. D. Zebrafish Breeding in the Laboratory Environment. ILAR Journal. 53 (2), 161-168 (2012).
- Rosen, J. N., Sweeney, M. F., Mably, J. D. Microinjection of Zebrafish Embryos to Analyze Gene Function. Journal of Visualized Experiments. 25, e1115(2009).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- R Core Team. R: A language and environment for statistical computing. , (2017).
- Smith, M. R. Ternary: an R package to generate ternary plots. , (2017).
- Baye, L. M., Link, B. A. Development/Plasticity/Repair Interkinetic Nuclear Migration and the Selection of Neurogenic Cell Divisions during Vertebrate Retinogenesis. The Journal of Neuroscience. 27 (38), 10143-10152 (2007).
- Leung, L., Klopper, A. V., Grill, S. W., Harris, W. A., Norden, C. Apical migration of nuclei during G2 is a prerequisite for all nuclear motion in zebrafish neuroepithelia. Development. 139, 2635(2012).
- Kerr, J. F. R., Wyllie, A. H., Curriet, A. R. Apoptosis: A Basic Biological Phenomenon with Wide-ranging Implications in Tissue Kinetics. British Journal of Cancer. 26, 239(1972).
- Cole, L. K., Ross, L. S. Apoptosis in the Developing Zebrafish Embryo. Developmental Biology. 240, 123-142 (2001).
- Liao, J., He, J., Yan, T., Korzh, V., Gong, Z. A Class of NeuroD-Related Basic Helix-Loop-Helix Transcription Factors Expressed in Developing Central Nervous System in Zebrafish. DNA and Cell Biology. 18 (4), 333-344 (1999).
- Trevarrow, B., Marks, D. L., Kimmel, C. B. Organization of hindbrain segments in the zebrafish embryo. Neuron. 4 (5), 669-679 (1990).
- Yabu, T., Todoriki, S., Yamashita, M. Stress-induced apoptosis by heat shock, UV and gamma-ray irradiation in zebrafish embryos detected by increased caspase activity and whole-mount TUNEL staining. Fisheries Science. 67, 333-340 (2001).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten