Method Article
Imagerie 3D haute résolution de l'infection par le virus de la rage dans un tissu cérébral éclairci par solvant
Dans cet article
Résumé
Les nouvelles techniques de compensation des tissus immunostaining-compatibles comme l'imagerie 3D ultime des organes éclaircis par solvant permettent la visualisation 3D de l'infection cérébrale du virus de la rage et de son environnement cellulaire complexe. D'épaisses tranches de tissu cérébral étiquetées par anticorps sont rendues optiquement transparentes afin d'augmenter la profondeur d'imagerie et de permettre l'analyse 3D par microscopie de balayage laser confocal.
Résumé
La visualisation des processus d'infection dans les tissus et les organes par immunolabeling est une méthode clé dans la biologie moderne d'infection. La capacité d'observer et d'étudier la distribution, le tropisme et l'abondance des agents pathogènes à l'intérieur des tissus d'organes fournit des données cruciales sur le développement et la progression de la maladie. À l'aide de méthodes de microscopie conventionnelles, l'immunolabeling est principalement limité aux sections minces obtenues à partir d'échantillons incrustés de paraffine ou congelés. Cependant, le plan d'image 2D limité de ces sections minces peut mener à la perte de l'information cruciale sur la structure complexe d'un organe infecté et le contexte cellulaire de l'infection. Les techniques modernes de compensation des tissus multicolores et immunostaining-compatibles offrent maintenant un moyen relativement rapide et peu coûteux d'étudier des piles d'images 3D à fort volume de tissus d'organes infectés par le virus. En exposant le tissu à des solvants organiques, il devient optiquement transparent. Cela correspond aux indices de réfraction de l'échantillon et conduit finalement à une réduction significative de la diffusion de la lumière. Ainsi, en combinaison avec de longs objectifs de distance de travail libre, de grandes sections de tissu jusqu'à 1 mm de taille peuvent être imaged par microscopie de balayage laser confocal e conthique conventionnelle (CLSM) à haute résolution. Ici, nous décrivons un protocole pour appliquer l'imagerie de tissu profond après dégagement de tissu pour visualiser la distribution de virus de rage dans les cerveaux infectés afin d'étudier des sujets comme la pathogénie de virus, la propagation, le tropisme, et la neuroinvasion.
Introduction
Les techniques d'histologie conventionnelles reposent principalement sur de fines sections de tissus d'organes, qui peuvent intrinsèquement fournir seulement des aperçus 2D dans un environnement 3D complexe. Bien que faisable en principe, la reconstruction 3D à partir de sections minces en série nécessite des pipelines techniques exigeants à la fois pour le découpage et l'alignement silico suivant des images acquises1. En outre, la reconstruction transparente des z-volumes après le découpage de microtome est critique car les artefacts mécaniques et computationnels peuvent rester en raison de l'enregistrement d'image sous-optimal causé par des plans d'image non overlapping, les variations de coloration, et physique destruction des tissus par, par exemple, la lame du microtome. En revanche, le découpage optique pur des échantillons intacts de tissu épais permet l'acquisition des plans d'image de chevauchement (suréchantillonnage) et, de ce fait, facilite la reconstruction 3D. Ceci, à son tour, est très bénéfique pour l'analyse des processus d'infection dans les populations cellulaires complexes (par exemple, les réseaux neuronaux dans le contexte des cellules gliales et immunitaires environnantes). Cependant, les obstacles inhérents des sections épaisses de tissu incluent la diffusion légère et la pénétration limitée d'anticorps dans le tissu. Ces dernières années, une variété de techniques a été développée et optimisée pour surmonter ces problèmes2,3,4,5,6,7,8 , 9 (en) , 10 Ans et plus , 11 Ans, états-unis ( , 12 Ans, états-unis , 13. Essentiellement, les tissus cibles sont devenus optiquement transparents par traitement avec soit aqueuse2,3,4,5,6,7 ,8,9 ou à base de solvants organiques10,11,12,13 solutions. L'introduction de 3DISCO (imagerie 3D d'organes éclaircis par solvant)11,12 et son successeur uDISCO (imagerie 3D ultime d'organes éclaircis par solvant)13 a fourni un outil relativement rapide, simple et peu coûteux avec d'excellentes capacités de compensation. Les principaux constituants du protocole de compensation sont les solvants organiques tert-butanol (TBA), l'alcool de benzyl (BA), le benzoate de benzyl (BB), et l'éther de diphenyl (DPE). Le développement et l'ajout d'iDISCO (imagerie 3D immunolabeling d'organes éclaircis par solvant)14, un protocole d'immunostaining compatible, ont constitué un autre avantage par rapport aux méthodes existantes et ont permis l'étiquetage des tissus profonds des antigènes d'intérêt, ainsi que le stockage à long terme d'échantillons immunostained. Ainsi, la combinaison de l'iDISCO14 et de l'uDISCO13 permet l'imagerie à haute résolution de protéines étiquetées par anticorps dans de grandes sections de tissus (jusqu'à 1 mm) à l'aide du CLSM conventionnel.
La préservation de la structure complexe d'un organe dans les trois dimensions est particulièrement importante pour le tissu cérébral. Les neurones constituent une sous-population cellulaire très hétérogène avec des morphologies 3D très diverses basées sur leurs projections neurites (revue par Masland15). En outre, le cerveau se compose d'un certain nombre de compartiments et de compartiments, chacun composé de différentes sous-populations cellulaires et les ratios de celui-ci, y compris les cellules gliales et les neurones (examiné par von Bartheld et al.16). En tant que virus neurotrope, le virus de la rage (RABV, examiné par Fooks et coll.17) infecte principalement les neurones, utilisant leurs machines de transport pour voyager dans la direction rétrograde le long des axones du site primaire de l'infection au système nerveux central (SNC). Le protocole décrit ici (figure 1A) permet la détection et la visualisation immunostaining-assistées des cellules RABV et RABV-infectées dans de grandes piles cohérentes d'image obtenues à partir du tissu cérébral infecté. Cela permet une évaluation impartiale et 3D à haute résolution de l'environnement infectieux. Il s'applique aux tissus cérébraux d'une variété d'espèces, peut être effectué immédiatement après la fixation ou après le stockage à long terme d'échantillons de paraformaldéhyde (PFA), et permet le stockage et la réimagerie des échantillons tachés et défrichés pendant des mois.
Protocole
Le matériel cérébral archivé RABV-infecté et PFA-fixe a été employé. Les études expérimentales animales respectives ont été évaluées par le comité responsable de soins, d'utilisation et d'éthique des animaux du Bureau d'État de l'agriculture, de la sécurité alimentaire et de la pêche en Mecklembourg-Poméranie occidentale (LALFF M-V) et ont obtenu l'approbation avec les autorisations 7221.3-2.1-002/11 (souris) et 7221.3-1-068/16 (ferrets). Les soins généraux et les méthodes utilisées dans les expériences sur les animaux ont été effectués conformément aux lignes directrices approuvées.
CAUTION: Ce protocole utilise diverses substances toxiques et / ou nocives, y compris pfA, méthanol (MeOH), peroxyde d'hydrogène (H2O2), azide de sodium (NaN3), TBA, BA, BB, et DPE. MeOH et TBA sont très inflammables. Évitez l'exposition en portant un équipement de protection individuelle approprié (une blouse de laboratoire, des gants et une protection oculaire) et en menant des expériences dans une hotte de fumée. Recueillir les déchets séparément dans des conteneurs appropriés et les éliminer conformément à la réglementation locale. Le virus de la rage est classé comme agent pathogène de niveau de biosécurité (BSL)-2 et peut donc généralement être manipulé dans des conditions De BSL-2. Certaines activités, y compris les procédures qui peuvent générer des aérosols, travailler avec des concentrations élevées de virus, ou de travailler avec de nouveaux lyssavirus, peuvent nécessiter la classification BSL-3. La prophylaxie pré-exposition est recommandée pour le personnel à risque élevé, y compris les gardiens d'animaux et les travailleurs de laboratoire18,19. Consultez les règlements des autorités locales.
1. Fixation du tissu cérébral et de la sectionnement
- Fixez des échantillons de cerveau dans un volume approprié de 4 % de PFA dans la saline tamponnée en phosphate (PBS [pH 7.4]) pendant au moins 48 h à 4 oC (avec un rapport tissu-fixatif approximatif de 1:10 [v/v]).
- Laver les échantillons de tissus 3x dans le PBS pendant au moins 30 min chaque lavage et les stocker dans 0,02% NaN3/PBS à 4 oC jusqu'à l'utilisation.
- Sectionnez le tissu en sections de 1 mm d'épaisseur à l'aide d'un vibratome (taux d'alimentation de la lame : 0,3 à 0,5 mm/s, amplitude : 1 mm, épaisseur de la tranche : 1 000 m).
- Pour conserver la séquence de tranche correcte, entreposez chaque section de tissu séparément dans un puits d'une plaque de culture de cellules multiwell. Ajouter 0,02 % NaN3/PBS et conserver les sections de tissus à 4 oC jusqu'à ce qu'elles soient utilisées.
2. Prétraitement d'échantillon avec le méthanol
REMARQUE : Effectuez toutes les étapes d'incubation avec une oscillation douce et, si elle n'est pas indiquée autrement, à température ambiante. Protégez les échantillons de la lumière. Le prétraitement de l'échantillon sert à améliorer la diffusion des anticorps et à réduire l'autofluorescence tissulaire par exposition à MeOH et H2O2, respectivement14.
- Préparer 20 % (v/v), 40 %, 60 % et 80 % de solutions MeOH dans de l'eau distillée. Par exemple, pour 20 % de MeOH, ajouter 10 ml de 100 % de MéOH à 40 ml d'eau distillée dans un récipient étanche approprié et mélanger en l'inversant.
- Transférer les échantillons à des récipients de taille raisonnable (p. ex., tubes de réaction de 5 ml). Prenez soin d'utiliser des matériaux chimiquement résistants aux réactifs utilisés dans ce protocole. Par exemple, notez que même si le polypropylène convient, le polystyrène ne l'est pas.
REMARQUE : Les spécifications de volume de ce protocole se réfèrent à des tubes de réaction de 5 ml. Si un autre navire est utilisé, ajustez les volumes en conséquence. - Incuber les échantillons en 4 ml de chaque concentration de la série préparée de solutions MeOH en ordre croissant pour 1 h chacun.
- Incuber les échantillons 2x pour 1 h chacun en pur (100%) MeOH.
- Refroidir les échantillons à 4 oC (p. ex., dans un réfrigérateur sécuritaire pour le laboratoire).
- Préparer une solution de blanchiment (5% H2O2 dans MeOH) en diluant, par exemple, une solution de 30% H2O2 stock à 1:6 en pur (100%) MeOH, et réfrigérer à 4 oC.
- Retirer le 100% MeOH des échantillons réfrigérés et ajouter 4 ml de solution de blanchiment préréfrigérée (5% H2O2 dans MeOH). Incuber toute la nuit à 4 oC.
- Échangez la solution de blanchiment pour 4 ml de 80 % De MeOH et incubez-vous pendant 1 h. Continuez d'utiliser la série préparée de solutions MeOH en ordre décroissant pour 1 h chacune jusqu'à ce que les échantillons aient été incubés pendant 1 h dans 4 ml de 20 % de MeOH.
- Laver les échantillons 1x pour 1 h avec 4 ml de PBS.
3. Immunostaining
REMARQUE : Effectuez toutes les étapes d'incubation avec une oscillation douce et, si elle n'est pas indiquée autrement, à température ambiante. Protégez les échantillons de la lumière. Pour prévenir la croissance microbienne, ajoutez NaN3 à une concentration finale de 0,02 % aux solutions de cette section. Les échantillons de tissus sont encore perméabilisés par le traitement avec des détergents nonionic Triton X-100 et Tween 20. Le sérum normal est utilisé pour bloquer la liaison d'anticorps non spécifique. Glycine et héparine sont ajoutés pour réduire le fond d'immunolabeling14.
- Laver les échantillons 2x pour 1 h chacun dans 4 ml de 0,2% Triton X-100/PBS.
- Perméabilisez les échantillons pendant 2 jours à 37 oC avec 4 ml de 0,2 % Triton X-100/20 % DMSO/0,3 M glycine/PBS.
- Bloquer la liaison non spécifique des anticorps en couvant les échantillons pendant 2 jours à 37 oC dans 4 ml de 0,2 % De 0,2 % Triton X-100/10 % DMSO/6 % sérum normal/PBS.
REMARQUE : Utilisez le sérum normal de la même espèce que l'anticorps secondaire a été élevé pour obtenir des résultats de blocage idéaux. - Incuber les échantillons dans 2 ml de solution d'anticorps primaire (3 % de sérum normal/5 % DMSO/PTwH [PBS-Tween 20 avec héparine] - anticorps/anticorps primaires) pendant 5 jours à 37 oC. Rafraîchissez la solution d'anticorps primaire après 2,5 jours.
- Pour PTwH, reconstituer le sel de sodium héparindans dans de l'eau distillée pour faire une solution de stock de 10 mg/mL (stocker cette solution, aliquoted, à 4 oC). Ajouter la solution de stock à 0,2% Tween 20/PBS à une concentration finale de 10 g/mL.
REMARQUE : Le choix de la dilution correcte des anticorps peut nécessiter une optimisation. En général, les concentrations standard d'immunohistochimie sont un bon point de départ.
- Pour PTwH, reconstituer le sel de sodium héparindans dans de l'eau distillée pour faire une solution de stock de 10 mg/mL (stocker cette solution, aliquoted, à 4 oC). Ajouter la solution de stock à 0,2% Tween 20/PBS à une concentration finale de 10 g/mL.
- Laver les échantillons pendant 1 jour dans 4 ml de PTwH, échanger le tampon de lavage au moins 4x à 5x au cours de la journée et laisser le lavage final pendant la nuit.
- Incuber les échantillons dans 2 ml de solution d'anticorps secondaire (3 % de sérum normal/PTwH - anticorps/anticorps secondaires) pendant 5 jours à 37 oC. Rafraîchissez la solution d'anticorps secondaire après 2,5 jours.
- Diluer les anticorps/anticorps secondaires à 1:500 dans la solution secondaire d'anticorps (c.-à-d., 4 l dans 2 mL).
- Laver les échantillons pendant 1 jour comme décrit à l'étape 3.5, en laissant le lavage final pendant la nuit.
4. Taches nucléaires
REMARQUE : Effectuez toutes les étapes d'incubation avec une oscillation douce et, si elle n'est pas indiquée autrement, à température ambiante. Protégez les échantillons de la lumière. Si aucune coloration nucléaire n'est requise ou si le spectre de longueur d'onde/émission d'excitation de TO-PRO-3 est nécessaire pour l'excitation ou la détection d'un autre fluorophore, sautez cette étape.
- Diluer la tache d'acide nucléique TO-PRO-3 à 1:1,000 dans PTwH et couver les échantillons dans 4 ml de solution de coloration nucléaire pendant 5 h.
- Laver les échantillons pendant 1 jour comme décrit à l'étape 3.5, en laissant le lavage final pendant la nuit.
REMARQUE : Après les lavages, les échantillons peuvent être stockés dans le PBS à 4 oC jusqu'à ce que le défrichement optique.
5. Défrichement des tissus
REMARQUE : Effectuez toutes les étapes d'incubation avec une oscillation douce et, si elle n'est pas indiquée autrement, à température ambiante. Protégez les échantillons de la lumière. Les échantillons de tissus sont déshydratés dans une série de solutions TBA graduées. Comme l'immunostaining nécessite des solutions aqueuses, toutes les procédures de coloration doivent être terminées avant le défrichement des tissus. Le dégagement optique et l'appariement d'index réfractif sont réalisés par traitement avec un mélange de BA, BB, et DPE. La solution de compensation est complétée par DL-tocophérol comme antioxydant13.
- Préparer 30 % (v/v), 50 %, 70 %, 80 %, 90 % et 96 % des solutions TBA dans l'eau distillée. Par exemple, pour 30 % d'ACT, ajouter 15 ml de 100 % de TBA à 35 ml d'eau distillée dans un récipient étanche approprié et mélanger par inversion.
REMARQUE : L'ACT a un point de fusion de 25 à 26 oC; ainsi, il tend à être solide à la température ambiante. Afin de préparer des solutions TBA, chauffer la bouteille bien scellée à 37 oC dans un incubateur ou un bain d'eau. - Déshydrater les échantillons avec 4 ml de chaque concentration de la série préparée de solutions TBA en ordre croissant pour 2 h chacune. Laissez le 96% TBA sur la nuit.
- Déshydrater davantage les échantillons en pur (100%) TBA pour 2 h.
- Préparer la solution de compensation BABB-D15.
REMARQUE: BABB-D15 est une combinaison de BA et BB (BABB) qui est mélangé avec DPE à un rapport de x:1, où x est spécifié dans le nom de la solution, dans ce cas 15.- Pour BABB, mélanger une partie BA avec deux parties BB.
- Mélanger BABB et DPE à un ratio de 15:1.
- Ajouter 0,4 vol% DL-Tocophérol (vitamine E).
REMARQUE : Par exemple, pour 20 ml de BABB-D15, mélangez 6,25 ml de BA avec 12,5 ml de BB. Ajouter 1,25 ml de DPE et le compléter avec 0,08 ml de DL-tocophérol.
- Effacer les échantillons dans la solution de compensation jusqu'à ce qu'ils soient optiquement transparents (2 à 6 h).
- Les échantillons peuvent être stockés à 4 oC dans BABB-D15, à l'abri de la lumière, jusqu'à ce que le montage et l'imagerie.
6. Montage d'échantillon
- À l'aide d'une imprimante 3D, imprimez la chambre d'imagerie et le couvercle (matériel : copolyester [CPE], buse : 0,25 mm, hauteur de la couche : 0,06 mm, épaisseur du mur : 0,88 mm, nombre de murs : 4, remplissage : 100 %, pas de structure de support ; le correspondant . Le fichier STL se trouve dans les documents supplémentaires de ce protocole).
- Assembler la chambre d'imagerie (figure 2).
- Montez un couvercle rond (diamètre : 30 mm) sur la chambre d'imagerie avec du caoutchouc en silicone RTV-1 (une pièce-température-vulcanisation). Retirer l'excès de caoutchouc de silicone à l'eau et guérir toute la nuit.
- Montez un couvercle rond (diamètre : 22 mm) sur le couvercle avec du caoutchouc en silicone RTV-1. Retirer l'excès de caoutchouc de silicone à l'eau et guérir toute la nuit.
- Placez l'échantillon dans une chambre d'imagerie, ajoutez un petit volume de BABB-D15 et insérez le couvercle. Remplissez la chambre de BABB-D15 à travers l'en-plein, à l'aide d'une aiguille hypodermique (27 G x 3/4 po [0,40 mm x 20 mm]).
- Branchez l'inlet et scellez la chambre d'imagerie avec du caoutchouc en silicone RTV-1. Cure toute la nuit dans l'obscurité.
7. Imagerie et traitement d'image
- Configurez l'acquisition d'image en sélectionnant les lignes laser respectives pour correspondre aux fluorophores utilisés. Ajuster les plages de détection de chaque détecteur pour éviter le chevauchement du signal entre les canaux.
REMARQUE : Les plages de détection exemplaires pour Alexa Fluor 488, Alexa Fluor 568 et TO-PRO-3 sont de 500 à 550 nm, 590 à 620 nm et 645 à 700 nm, respectivement. - Choisissez les paramètres d'acquisition, définissez la bordure supérieure et inférieure de la z-stack et acquérez la pile d'images.
REMARQUE : Les paramètres d'acquisition exemplaires sont un scan séquentiel d'une taille de pixel de 60-90 nm, une taille z-étape de 0,5 m, une moyenne de ligne de 1, une vitesse d'analyse de 400 Hz, et une taille de sténopé de 1 unité Airy. - Traiter la pile d'images à l'aide d'un logiciel d'analyse d'image approprié (p. ex., Fidji) pour générer des projections 3D ou effectuer des analyses approfondies.
REMARQUE : En raison de la grande taille des fichiers d'image acquis, l'utilisation d'un poste de travail est habituellement nécessaire.- Ouvrez le fichier d'acquisition ou d'image (s) aux Fidji (Fichier Ouvert à l'ouvert Sélectionnez les fichiers).
- Si vous utilisez, par exemple, . Fichiers LIF, sélectionnez ou désélectionnez les options souhaitées dans la fenêtre de dialogue Bio-Formats. Afficher la pile avec Hyperstack. A part cela, aucune sélection ou tique spécifique n'est nécessaire. Appuyez sur OK.
- Si le fichier contient plusieurs piles d'images, sélectionnez ceux à analyser et confirmez en appuyant sur OK.
- Effectuer la correction de l'eau de Javel en divisant l'image fusionnée en canaux individuels (Image La couleur Outil de canaux, puis sélectionnez Plus Split Channels). Pour chaque canal, sélectionnez correction de l'eau de Javel(Image Ajuster Correction de blanchiment) et choisissez Simple Ratio (intensité de fond : 0,0).
REMARQUE : Dans certains cas, par exemple, lorsqu'il n'y a pas de décomposition linéaire du signal ou que le signal est trop faible dans l'ensemble, Simple Ratio peut échouer. Sinon, essayez Exponential Fit ou sautez la correction de l'eau de Javel. - Ajuster la luminosité et le contraste pour chaque canal à l'aide des curseurs (Image Ajuster Luminosité de l'année Contraste).
- Fusionner les canaux (Image La couleur Merge Channels), faire un composite (Image La couleur Outil de canaux, puis sélectionnez Plus Make Composite), et le convertir en format RGB (Image La couleur Outil de canaux, puis sélectionnez Plus Convertir en RGB).
- Si nécessaire, redimensionnez la pile d'images pour réduire le temps de calcul et la taille du fichier (Image Ajuster Taille,les deux options cochées plus interpolation bilinear).
- Générer une projection 3D (Image Piles ( Projet 3D). Choisissez Brightest Point comme méthode de projection et configurez l'espacement de tranche pour correspondre à la taille z-step de la pile d'image acquise. Pour une qualité maximale, fixez l'incrément de l'angle de rotation à 1 et activez l'interpolation. Modifier la rotation totale, les seuils de transparence et l'opacité au besoin.
- Si nécessaire, réajuster le contraste et la luminosité en reconverti la projection 3D en format 8 bits (Image Type de type 8 bits). Utilisez les curseurs respectifs (Image Ajuster Luminosité de l'année Contraste) et reconvertir la pile d'images en format RGB tel que décrit à l'étape 7.3.4.
- Enregistrer la projection 3D comme . Fichier TIF (format de fichier d'image) et . Fichier AVI (format de fichier vidéo).
- Ouvrez le fichier d'acquisition ou d'image (s) aux Fidji (Fichier Ouvert à l'ouvert Sélectionnez les fichiers).
Résultats
La combinaison de l'iDISCO14 et de l'uDISCO13 couplée à la Haute résolution CLSM fournit des aperçus profonds dans la résolution spatiotemporale et la plasticité de l'infection DE RABV du tissu cérébral et le contexte cellulaire environnant.
En utilisant l'immunostaining de la phosphoprotéine RABV (P), des couches complexes de cellules neuronales infectées peuvent être visualisées dans des sections épaisses du cerveau de souris (Figure 3). Par la suite, les projections 3D transparentes des piles d'images acquises peuvent être reconstruites (Figure 3A, B, panneau droit; Figure animée 1). Il faut prendre soin de l'utilisation d'anticorps primaires et d'anticorps secondaires contre l'espèce dont provient le matériel organique. L'utilisation d'anticorps IgG anti-souris sur le tissu cérébral de la souris a entraîné une coloration distincte du système vasculaire (figure3B, panneau gauche). En raison de la haute résolution avec laquelle les piles d'images sont acquises, l'infection peut être évaluée jusqu'à un niveau unicellulaire (figure 4), permettant des affirmations sur, par exemple, l'abondance et la distribution de l'antigène dans la cellule (Figure 4C ).
Mis à part le tissu cérébral de souris, le protocole peut également être appliqué auxtissus cérébraux d'autres espèces animales (p. ex. furets) (figure 5; Figure animée 2). Les sections prélevées dans différents compartiments d'un cerveau de furet infecté ont révélé un degré variable d'infection par le VRAB (figure5A-D).
Comme le cerveau comprend de nombreuses sous-populations cellulaires différentes, la différenciation entre ces populations est vitale. À l'aide d'anticorps dirigés contre les marqueurs cellulaires, l'évaluation de l'identité cellulaire des cellules infectées et voisines est possible. Par exemple, les astrocytes peuvent être différenciés par l'expression de la protéine acide fibrillaire gliale (GFAP) (figure6A,C; Figure animée 3), tandis que les neurites peuvent être spécifiquement tachées pour les protéines 2 associées aux microtubules (MAP2) (figure6B,D; Figure animée 4). Simultanément, les protéines virales, dans ce cas, la nucléoprotéine RABV (N), peuvent être costained pour évaluer la relation entre les cellules infectées et la sous-population cellulaire mise en évidence.

Figure 1 : Principe de base et flux de travail du protocole. (A) Représentation graphique du flux de travail basée sur les protocoles de Renier et coll.14 et Pan et al.13. (B) Deux tranches de cerveau de furet exemplaires, une avant (panneau gauche) et une après traitement avec des solvants organiques (panneau droit). Le défrichement rend le tissu optiquement transparent comme observable par le texte alors lisible. La tranche de cerveau dégagée est incorporée dans une chambre d'imagerie imprimée en 3D (panneau droit). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
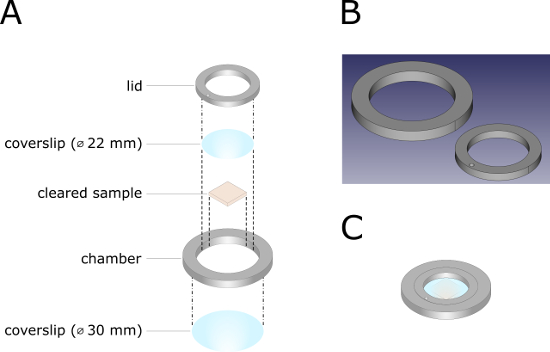
Figure 2 : Illustration technique de la chambre d'imagerie imprimée en 3D. (A) Dessin de vue explosé de la chambre d'imagerie. Les lignes point-dashed mettent en évidence les composants qui doivent être montés les uns sur les autres à l'aide de caoutchouc en silicone RTV-1. Les lignes pointillées représentent des instructions pour l'assemblage ultérieur de la chambre. (B) CAO (conception assistée par ordinateur) de la chambre d'imagerie. Le correspondant . Le fichier STL pour imprimer la chambre d'imagerie se trouve dans les Matériaux Supplémentaires. (C) Chambre d'imagerie entièrement assemblée. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 3 : Imagerie des tissus profonds du tissu cérébral de souris infecté par le RABV. (A et B) Les souris ont été infectées par un virus recombinant de la rue vulpine. La coloration RABV P (verte) révèle des couches neuronales infectées avec de grandes projections de neurite s'emmêler à l'intérieur du tissu cérébral effacé. Les noyaux ont été contrecarrés avec TO-PRO-3 (bleu). (B) L'utilisation d'anticorps secondaires IgG antisouris étiquetés fluorophore (rouge) sur le tissu de la souris donne lieu à un étiquetage distinct du système vasculaire. La reconstruction 3D des piles d'images acquises permet l'observation sous différents angles de vision (A et B, panneaux droits). Barres d'échelle de 60 m. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 4 : L'acquisition d'images haute résolution permet des évaluations complexes jusqu'à un niveau unicellulaire. Le cerveau a été disséqué d'une souris Infectée Par le SAD L16. (A) RABV P coloration (vert) met en évidence un neurone infecté individuellement. (B et C) Des images et des projections détaillées démontrent que des analyses approfondies de l'abondance, de la distribution et de la localisation des antigènes peuvent être effectuées. Les noyaux ont été contrecarrés avec TO-PRO-3 (bleu). Barres d'échelle de 20 m (A), 5 m (C). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
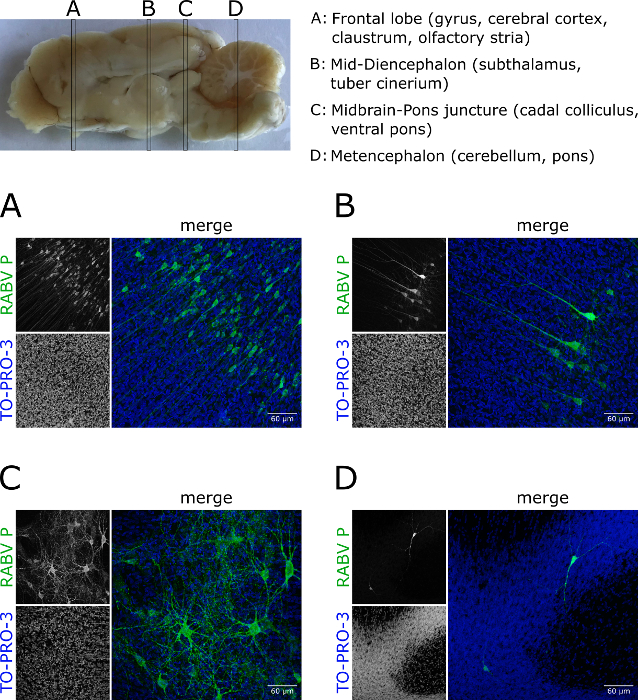
Figure 5 : Imagerie des tissus profonds du tissu cérébral du furet infecté par le RABV. Les furets ont été infectés par la rue canine RABV. (A-D) Des tranches provenant de certaines zones du cerveau ont été prélevées, immunostained pour RABV P (vert), et optiquement dégagées. Les projections démontrent que les cellules infectées dans différentes parties du cerveau diffèrent en quantité et en morphologie. En outre, ils mettent en évidence l'applicabilité du protocole aux tissus autres que la souris dérivée. Les noyaux ont été contrecarrés avec TO-PRO-3 (bleu). Barres d'échelle de 60 m. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 6 : L'immunofluorescence multicolore permet la costaining des marqueurs cellulaires. Des tranches de tissu cérébral provenant de furets infectés par la rue canine RABV ont été utilisées et coimmunostained pour RABV N (rouge) et soit (A et C) GFAP (vert) ou (B et D) MAP2 (vert). Alors que le GFAP est un marqueur d'astrocytes, MAP2 met spécifiquement en évidence les neurites. Les noyaux ont été contrecarrés avec TO-PRO-3 (bleu). (C et D) En bas, des extractions à tranche unique des vues détaillées énumérées des projections fusionnées sont représentées. Barres d'échelle de 15 m (A et B) 10 m (C et D). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure animée 1 : reconstructions 3D et projections détaillées des piles d'images à partir de cerveaux de souris infectés par le RABV. Les projections ont été générées à partir des piles d'images décrites dans la figure 3. (A et C) Animations de l'ensemble des z-stacks respectifs. (B) Une projection détaillée d'une reconstruction 3D d'une partie de la z-pile des zones mises en évidence au début de la vidéo. (D) Une projection détaillée d'un tomogramme d'une partie de la z-pile des zones mises en évidence au début de la vidéo. Vert rabOUd P; rouge et souris IgG; bleu et noyaux. Veuillez cliquer ici pour visionner cette vidéo. (Clic droit pour télécharger.)

Figure animée 2 : Reconstructions 3D de piles d'images acquises à partir d'un cerveau de furet infecté par le RABV. Les projections ont été générées à partir des piles d'images décrites dans la figure 5. (A-D) Les annotations se réfèrent au même chiffre et décrivent les zones respectives du cerveau à partir destranches. Vert rabOUd P; bleu et noyaux. Veuillez cliquer ici pour visionner cette vidéo. (Clic droit pour télécharger.)

Figure animée 3 : Projection graduelle des différents canaux d'une reconstruction 3D d'un cerveau de furet RABV-infecté costained avec le marqueur d'astrocyte GFAP. Les projections ont été générées à partir de la pile d'images décrite à la figure 6A. L'ajout progressif des canaux commence par RABV N (rouge), après quoi suivre les noyaux cellulaires (bleu) et, enfin, GFAP (vert), tandis que la soustraction enlève d'abord RABV N, puis les noyaux cellulaires, et finalement GFAP. Veuillez cliquer ici pour visionner cette vidéo. (Clic droit pour télécharger.)

Figure animée 4 : Projection graduelle des différents canaux d'une reconstruction 3D d'un cerveau de furet RABV-infecté costained avec le marqueur neuronal MAP2. Les projections ont été générées à partir de la pile d'images décrite dans la figure 6B. L'ajout progressif des canaux commence par RABV N (rouge), après quoi suivre les noyaux cellulaires (bleu) et, enfin, MAP2 (vert), tandis que la soustraction enlève d'abord RABV N, puis les noyaux cellulaires, et finalement MAP2. Veuillez cliquer ici pour visionner cette vidéo. (Clic droit pour télécharger.)
Discussion
La résurgence et le développement ultérieur des techniques de défrichement des tissus au cours des dernières années2,3,4,5,6,7,8, 9 (en) , 10 Ans et plus , 11 Ans, états-unis ( , 12 Ans, états-unis , 13 (en) , 14 ont ouvert de nombreuses nouvelles possibilités pour obtenir des piles d'images à volume élevé de tissu d'organes. Cela a fourni un outil inégalé et puissant pour étudier, entre autres sujets, l'infection par le virus. La reconstruction 3D subséquente de ces piles d'images permet des affirmations sophistiquées sur, par exemple, le tropisme de virus, l'abondance, et le cours de temps de l'infection. Ce protocole décrit la visualisation immunolabeling-assistée de l'infection de RABV dans le tissu de cerveau solvant-éclairci.
Il y a plusieurs étapes critiques dans la préparation et l'acquisition des piles d'image des tissus immunolabeled, solvant-dédouanés. L'exposition prolongée à PFA peut masquer des épitopes et, par conséquent, entraîner une diminution de l'antigénicité20,21,22. Il est donc important de limiter les temps de fixation au minimum nécessaire et de transférer les échantillons vers une solution appropriée (p. ex., PBS complété par 0,02 % NaN3 pour le stockage à long terme). Cependant, toutes les images et projections représentatives fournies ici ont été acquises à partir d'échantillons de cerveau archivés, dont certains avaient été stockés dans PFA pendant des semaines, soulignant l'applicabilité de la technique au matériel d'organe qui a été exposé à la PFA pour périodes prolongées. Des résultats similaires ont été montrés pour des échantillons de cerveau humain13. Une autre étape critique, sinon la plus critique, est l'incubation des anticorps. Les concentrations d'anticorps doivent être choisies avec soin. Tandis que dans la plupart des cas, les concentrations standard de CiSE sont un bon point de départ pour l'étiquetage d'anticorps de tissu profond, certains antigènes peuvent exiger l'optimisation additionnelle de la concentration d'anticorps. Ce protocole démontre que rabV N et P sont facilement détectables par les anticorps utilisés. Tandis que le prétraitement de MeOH améliore habituellement l'immunolabeling, quelques antigènes sont incompatibles avec ce traitement. Pour ceux-ci, Renier et ses collègues14 ont fourni un autre prétraitement de l'échantillon sans MeOH. L'analyse des piles d'images acquises nécessite une puissance de calcul adéquate. En raison de la grande taille des fichiers allant jusqu'à plusieurs gigaoctets par pile, des ordinateurs puissants sont nécessaires pour traiter les images. Le posttraitement comprend souvent la soustraction du bruit de fond et une correction de l'eau de Javel pour compenser les effets de blanchiment associés à l'acquisition. Lors de la mesure des distances, il faut garder à l'esprit que, comme un effet secondaire, le tissu se rétrécit au cours du processus de compensation.
Comparativement à d'autres plates-formes de microscopie, comme la microscopie par fluorescence par feuille de lumière (LSFM) ou la microscopie à balayage laser à deux photons (2PLSM), cLSM a la distance de travail la plus limitée. Par conséquent, il est nécessaire de prétrancher les organes à 1 mm d'épaisseur pour l'imagerie. En outre, CLSM a la vitesse d'acquisition la plus lente en raison de sa haute résolution d'imagerie. L'utilisation d'un microscope à feuilles de lumière permettrait une imagerie plus rapide de plus grands volumes jusqu'à l'imagerie du cerveau entier, tout en sacrifiant simultanément la résolution de l'image. Une autre limitation est l'augmentation inhérente de l'autofluorescence tissulaire avec la longueur d'onde décroissante. Cela rend l'utilisation de fluorophores et de colorants excités par la ligne laser à 405 nm, y compris les colorants Hoechst, impossible à impossible.
En ce qui concerne d'autres techniques de compensation tissulaire, l'hybride d'iDISCO14 et uDISCO13 combine les attributs les plus positifs : il est très compatible avec l'immunostaining, très polyvalent, relativement rapide et peu coûteux, il a d'excellentes capacités de compensation, et il est possible avec un microscope confocal standard. En outre, ce protocole n'est pas limité à l'immunostaining et le dégagement du tissu cérébral, mais peut également être appliqué à une variété d'autres tissus mous et pathogènes, à la fois neuro-invasive et non-neuroinvasive. En conclusion, le protocole décrit ici représente un pipeline pour la formation image 3D à haute résolution du tissu cérébral RABV-infecté. Les reconstructions 3D du tissu cérébral infecté peuvent être employées pour répondre à une série de questions concernant la progression de la maladie de RABV, la pathogénie, et la neuroinvasion.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Les auteurs remercient Thomas C. Mettenleiter et Verena te Kamp d'avoir lu le manuscrit de façon critique. Ce travail a été soutenu par l'Initiative fédérale d'excellence de Mecklembourg Poméranie occidentale et le Fonds social européen (FSE) Grant KoInfekt (ESF/14-BM-A55-0002/16) et une subvention de recherche collaborative intra-muros sur les Lyssavirus à la Friedrich-Loeffler-Institut (Ri-0372).
matériels
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Benzyl alcohol | Alfa Aesar | 41218 | Clearing reagent |
| Benzyl benzoate | Sigma-Aldrich | BB6630-500ML | Clearing reagent |
| Dimethyl sulfoxide | Carl Roth | 4720.2 | Various buffers |
| Diphenyl ether | Sigma-Aldrich | 240834-100G | Clearing reagent |
| DL-α-Tocopherol | Alfa Aesar | A17039 | Antioxidant |
| Donkey serum | Bio-Rad | C06SBZ | Blocking reagent |
| Glycine | Carl Roth | 3908.2 | Background reduction |
| Goat serum | Merck | S26-100ML | Blocking reagent |
| Heparin sodium salt | Carl Roth | 7692.1 | Background reduction |
| Hydrogen peroxide solution (30 %) | Carl Roth | 8070.2 | Sample bleaching |
| Methanol | Carl Roth | 4627.4 | Sample pretreatment |
| Paraformaldehyde | Carl Roth | 0335.3 | Crystalline powder to make fixative solution |
| Sodium azide | Carl Roth | K305.1 | Prevention of microbial growth in stock solutions |
| tert-Butanol | Alfa Aesar | 33278 | Sample dehydration for tissue clearing |
| TO-PRO-3 | Thermo Fisher | T3605 | Nucleic acid stain |
| Triton X-100 | Carl Roth | 3051.2 | Detergent |
| Tween 20 | AppliChem | A4974,0500 | Detergent |
| Miscellaneous | |||
| 5 mL reaction tubes | Eppendorf | 0030119401 | Sample tubes |
| Coverslip, circular (diameter: 22 mm) | Marienfeld | 0111620 | Part of imaging chamber |
| Coverslip, circular (diameter: 30 mm) | Marienfeld | 0111700 | Part of imaging chamber |
| Hypodermic needle (27 G x ¾” [0.40 mm x 20 mm]) | B. Braun | 4657705 | Filling of the imaging chamber with clearing solution |
| RTV-1 silicone rubber | Wacker | Elastosil E43 | Adhesive for the assembly of the imaging chamber |
| Ultimaker CPE 2.85 mm transparent | Ultimaker | 8718836374869 | Copolyester filament for 3D printer to print parts of the imaging chamber |
| Technical equipment and software | |||
| 3D printer | Ultimaker | Ultimaker 2+ | Printing of imaging chamber |
| Automated water immersion system | Leica | 15640019 | Software-controlled water pump |
| Benchtop orbital shaker | Elmi | DOS-20M | Sample incubation at room temperature (~ 150 rpm) |
| Benchtop orbital shaker, heated | New Brunswick Scientific | G24 Environmental Shaker | Sample incubation at 37 °C (~ 150 rpm) |
| Confocal laser scanning microscope | Leica | DMI 6000 TCS SP5 | Inverted confocal microscope for sample imaging |
| Fiji | NIH (ImageJ) | open source software (v1.52h) | Image processing package based on ImageJ |
| Long working distance water immersion objective | Leica | 15506360 | HC PL APO 40x/1.10 W motCORR CS2 |
| Vibratome | Leica | VT1200S | Sample slicing |
| Workstation | Dell | Precision 7920 | CPU: Intel Xeon Gold 5118 GPU: Nvidia Quadro P5000 RAM: 128 GB 2666 MHz DDR4 SSD: 2 TB |
| Primary antibodies | |||
| Goat anti-RABV N | Friedrich-Loeffler-Institut | Monospecific polyclonal goat anti-RABV N serum, generated by goat immunization with baculovirus-expressed and His-tag-purified RABV nucleoprotein N Dilution: 1:400 | |
| Rabbit anti-GFAP | Dako | Z0334 | Polyclonal antibody (RRID:AB_10013382) Dilution: 1:100 |
| Rabbit anti-MAP2 | Abcam | ab32454 | Polyclonal antibody (RRID:AB_776174) Dilution: 1:250 |
| Rabbit anti-RABV P 160-5 | Friedrich-Loeffler-Institut | Monospecific polyclonal rabbit anti-RABV P serum, generated by rabbit immunization with baculovirus-expressed and His-tag-purified RABV phosphoprotein P (see reference 23: Orbanz et al., 2010) Dilution: 1:1,000 | |
| Secondary antibodies | |||
| Donkey anti-goat IgG | Thermo Fisher Scientific | depending on conjugated fluorophore | Highly cross-absorbed Dilution: 1:500 |
| Donkey anti-mouse IgG | Thermo Fisher Scientific | depending on conjugated fluorophore | Highly cross-absorbed Dilution: 1:500 |
| Donkey anti-rabbit IgG | Thermo Fisher Scientific | depending on conjugated fluorophore | Highly cross-absorbed Dilution: 1:500 |
| Goat anti-mouse IgG | Thermo Fisher Scientific | depending on conjugated fluorophore | Highly cross-absorbed Dilution: 1:500 |
| Goat anti-rabbit IgG | Thermo Fisher Scientific | depending on conjugated fluorophore | Highly cross-absorbed Dilution: 1:500 |
Références
- Pichat, J., Iglesias, J. E., Yousry, T., Ourselin, S., Modat, M. A Survey of Methods for 3D Histology Reconstruction. Medical Image Analysis. 46, 73-105 (2018).
- Chung, K., et al. Structural and molecular interrogation of intact biological systems. Nature. 497 (7449), 332-337 (2013).
- Hama, H., et al. ScaleS: an optical clearing palette for biological imaging. Nature Neuroscience. 18 (10), 1518-1529 (2015).
- Ke, M. T., Fujimoto, S., Imai, T. SeeDB: a simple and morphology-preserving optical clearing agent for neuronal circuit reconstruction. Nature Neuroscience. 16 (8), 1154-1161 (2013).
- Kuwajima, T., et al. ClearT: a detergent- and solvent-free clearing method for neuronal and non-neuronal tissue. Development. 140 (6), 1364-1368 (2013).
- Susaki, E. A., et al. Whole-brain imaging with single-cell resolution using chemical cocktails and computational analysis. Cell. 157 (3), 726-739 (2014).
- Susaki, E. A., et al. Advanced CUBIC protocols for whole-brain and whole-body clearing and imaging. Nature Protocols. 10 (11), 1709-1727 (2015).
- Yang, B., et al. Single-cell phenotyping within transparent intact tissue through whole-body clearing. Cell. 158 (4), 945-958 (2014).
- Treweek, J. B., et al. Whole-body tissue stabilization and selective extractions via tissue-hydrogel hybrids for high-resolution intact circuit mapping and phenotyping. Nature Protocols. 10 (11), 1860-1896 (2015).
- Dodt, H. U., et al. Ultramicroscopy: three-dimensional visualization of neuronal networks in the whole mouse brain. Nature Methods. 4 (4), 331-336 (2007).
- Erturk, A., et al. Three-dimensional imaging of the unsectioned adult spinal cord to assess axon regeneration and glial responses after injury. Nature Medicine. 18 (1), 166-171 (2011).
- Erturk, A., et al. Three-dimensional imaging of solvent-cleared organs using 3DISCO. Nature Protocols. 7 (11), 1983-1995 (2012).
- Pan, C., et al. Shrinkage-mediated imaging of entire organs and organisms using uDISCO. Nature Methods. 13 (10), 859-867 (2016).
- Renier, N., et al. iDISCO: a simple, rapid method to immunolabel large tissue samples for volume imaging. Cell. 159 (4), 896-910 (2014).
- Masland, R. H. Neuronal cell types. Current Biology. 14 (13), 497-500 (2004).
- von Bartheld, C. S., Bahney, J., Herculano-Houzel, S. The search for true numbers of neurons and glial cells in the human brain: A review of 150 years of cell counting. The Journal of Comparative Neurology. 524 (18), 3865-3895 (2016).
- Fooks, A. R., et al. Rabies. Nature Reviews Disease Primers. 3, 17091 (2017).
- WHO. WHO Expert Consultation on Rabies, Third Report. WHO Technical Report Series. , (2018).
- CDC. . Biosafety in Microbiological and Biomedical Laboratories, 5th Edition. US Department of Health and Human Services. , (2009).
- Arnold, M. M., et al. Effects of fixation and tissue processing on immunohistochemical demonstration of specific antigens. Biotechnic & Histochemistry. 71 (5), 224-230 (1996).
- Webster, J. D., Miller, M. A., Dusold, D., Ramos-Vara, J. Effects of prolonged formalin fixation on diagnostic immunohistochemistry in domestic animals. Journal of Histochemistry and Cytochemistry. 57 (8), 753-761 (2009).
- Werner, M., Chott, A., Fabiano, A., Battifora, H. Effect of formalin tissue fixation and processing on immunohistochemistry. The American Journal of Surgical Pathology. 24 (7), 1016-1019 (2000).
- Orbanz, J., Finke, S. Generation of recombinant European bat lyssavirus type 1 and inter-genotypic compatibility of lyssavirus genotype 1 and 5 antigenome promoters. Archives of Virology. 155 (10), 1631-1641 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon